Method Article
Пивоваренные дрожжи Метаболические маркировки с 4-thiouracil и количественная оценка недавно синтезированных мРНК как прокси для РНК-полимераза II деятельности
В этой статье
Резюме
Протокол, описанные здесь основан на количественное определение генома общесистемной вновь синтезированной мРНК, очищенного от дрожжевых клеток с 4-thiouracil. Этот метод позволяет измерять синтез мРНК, расцеплено от гниения мРНК и, таким образом, обеспечивает точное измерение II транскрипции РНК-полимеразы.
Аннотация
Глобальные дефекты в транскрипции РНК полимеразой II может игнорировать транскриптомики исследований, анализа установившегося РНК. Действительно было показано глобальное снижение синтеза мРНК компенсироваться одновременным снижением деградации мРНК для восстановления нормального уровня устойчивого состояния. Следовательно количественного определения генома широкий синтез мРНК, независимо от распада мРНК, является лучшим прямым отражением РНК полимеразой II транскрипционный анализ деятельности. Здесь мы рассмотрим метод, с помощью не нарушается метаболизм маркировки зарождающейся РНК в Saccharomyces cerevisiae (S. cerevisiae). Конкретно клетки культивировали на 6 мин с урацила аналоговые, 4-thiouracil, и обозначенные недавно транскрипции РНК очищенный и количественно определить показатели синтеза всех отдельных мРНК. Кроме того, с помощью помечены Делящиеся дрожжи клетки как внутренний стандарт позволяет, сравнение синтез мРНК в различных S. cerevisiae штаммов. Используя этот протокол и установку данных с динамической кинетической модели, соответствующие показатели распада мРНК могут быть определены.
Введение
Клетки реагируют на эндогенных и экзогенных подсказки, через динамические изменения их программы выражения гена. В последние годы огромное развитие генома общесистемной методологии позволяет точное и всеобъемлющее описание транскриптом изменения в различных условиях. В большинстве исследований транскриптомики microarray гибридизации или высок объём последовательности используются для количественного определения уровня РНК от общей доли РНК установившегося. Transcriptional изменений под конкретные возмущения может отображать широкий спектр возможных результатов, с большой спектр генов вверх - или downregulated или изменения выражения определенного гена. Экспрессия генов является результатом точного равновесия — или стационарном — между синтез РНК, РНК-полимеразы и других процессов, влияющих на уровень РНК. Транскрипции РНК-полимераза II, включая его три этапа (инициации, элонгации и прекращение), высоко и неразрывно связан с обработки, цитоплазматических экспорта, перевод и деградации мРНК.
Несколько недавних исследований показали, что синтез мРНК и распада спаренных механизмы и показал, что транскрипционный анализ эффектов после мутации или под стимулы могут быть пропущены при количественной оценки общего устойчивого состояния РНК. Во-первых обнаружение transcriptional изменений путем анализа установившегося уровни mRNA всегда зависит от mRNAs half-life. Как только вводится возмущений, устойчивого состояния уровня мРНК с долгий период полураспада чем те мРНК с короткие периоды полураспада затронет гораздо меньше. Таким образом обнаружения изменений в синтез РНК является сильно предвзятым в пользу недолго стенограммы, в то время как анализ видов повериям мРНК могут не выявить динамических изменений курса транскрипции. Во-вторых несколько докладов показали, что, как в дрожжей и млекопитающих, могут игнорировать глобальные изменения в транскрипции, при анализе установившегося уровни mRNA. Это, вероятно, за счет механизмов, которые связывают синтез мРНК и деградации, что приводит к буферизации мРНК. Это побудило разработки новых протоколов для количественного определения синтез мРНК, расцеплено от деградации, путем анализа вновь трансляции мРНК. В последние годы были представлены несколько вариантов, включая глобальные выбега последовательности (GRO-seq)1и родной удлинение Стенограмма виртуализации (NET-seq)2,3. Здесь мы представляем протокол изначально разработанный в mammalian клетках4,5,6 и затем адаптирована к дрожжи7,8,9,10, 11, которая основана на РНК, маркировки с thiolated нуклеозидов или базовый аналоговый, 4-thiouridine (4СУ) или 4-thiouracil (4tU), соответственно.
Этот метод специально очищает недавно транскрипции РНК из клеток, в которых РНК, пульс помечены 4СУ практически без вмешательства в гомеостаза клетки. Таким образом после того, как клетки подвергаются 4СУ, молекула является быстро освоен, фосфорилированных 4СУ трифосфата и включены в РНК транскрипции. После того, как пульс меткой, это можно извлечь всего клеточной РНК (соответствующий уровень установившегося РНК), и, впоследствии, фракция 4СУ меченых РНК тиоловых специально изменены, приводит к образованию дисульфидных связей между биотина и Недавно транскрипции РНК4,5. Однако 4СУ только может быть освоен, выражая нуклеозидный транспортер, как человека equilibrative нуклеозидный транспортер (hENT1), предотвращая ее немедленного использования бутонизации или деления дрожжевой клетки. Хотя одно может выразить hENT1 S. pombe или S. cerevisiae, более простой подход может быть достигнуто с помощью измененный базовый 4tU, так как клетки дрожжей может занять до 4tU, без необходимости выражения нуклеозидный транспортер10, 11 , 12 , 13. В самом деле, метаболизм 4tU требует активности фермента урацила phosphoribosyltransferase (UPRT). В нескольких организмов, в том числе дрожжи, но не млекопитающих UPRT имеет важное значение для пути спасения пиримидина, переработка урацила в уридина монофосфат.
Важное значение смещения в транскриптомики исследования могут быть введены по нормализации отношений между различными образцы, проанализированы параллельно. Действительно, многие иные факторы могут повлиять на сравнительный анализ транскриптом мутант и дикого штаммов: эффективность лизис клеток, различия в добыче и восстановления РНК, и разницы в калибровка сканера для microarray анализ , среди других. Как говорилось выше, такие различия может быть особенно заблуждение, когда глобальные последствия для транскрипции РНК полимеразой II, как ожидается. Элегантный значит точно сравнить ставки синтез мРНК между различными образцами был разработан с использованием дрожжей отдаленно связанные деления Делящиеся дрожжи как внутренний стандарт. Для этого, фиксированное количество помечены S. pombe ячеек добавляется образцы S. cerevisiae , одичал тип или мутант клетки, до лизис клеток и РНК добыча10. Впоследствии как статических, так и заново синтезированные РНК от S. pombe и S. cerevisiae количественно RT-ПЦР или через использование высокопроизводительного секвенирования10или обломоки microarray. Сочетание этих данных с кинетического моделирования, абсолютное ставки мРНК синтеза и распада в многообещающий дрожжей могут быть измерены.
В рамках этой рукописи мы покажем, как анализ недавно транскрипции РНК позволил выявить глобальную роль для комплексов coactivator Сага и TFIID в транскрипции РНК полимеразой II в многообещающий дрожжей,14,15 16. Важно отметить, что последние исследования количественно уровни mRNA установившегося в S. cerevisiae и предложил, что сага играет функцию преобладающим на ограниченный набор генов дрожжей, которые сильно пострадавших от мутации в Сага, но относительно устойчив к TFIID мутации17,18,19. Удивительно Сага ферментативную деятельность были продемонстрированы действовать в целом транскрипции генома, предлагая более широкую роль для этого совместного активатор транскрипции РНК полимеразой II. Снижение РНК полимеразой II найма на наиболее выраженное генов было отмечено по инактивации Сага или TFIID, предполагая, что эти коактиваторы работать вместе на большинство генов. Следовательно количественная оценка недавно трансляции мРНК показали, что Сага и TFIID необходимы для транскрипции почти всех генов РНК полимеразы II14,,1516. Осуществление компенсационных механизмов возникает как способ для клетки, чтобы справиться с глобальным снижением синтеза мРНК, который буферизуется одновременное снижение глобальной деградации мРНК. Сага добавляет в список факторов, имеющих глобальный эффект на транскрипции РНК полимеразой II, например, РНК Pol II подразделения10, посредник coactivator комплекс20, Генеральный транскрипционным фактором TFIIH21,22 и косвенно, элементы10,9,механизм деградации мРНК23. В Сага мутантов, приходится скромные и ограниченные изменения в стационарном уровни mRNA несмотря на глобальных и серьезным снижением синтеза мРНК14универсально наблюдались такие компенсационные мероприятия. Были также подобные анализы в BRE1 исключить деформации, что приводит к полной потере гистона H2B ubiquitination. Интересно, что гораздо мягче, но последовательно глобальный эффект на транскрипции РНК полимеразой II могут быть обнаружены в отсутствие Bre1, указав, что метаболические маркировки недавно транскрипции РНК в дрожжи можно обнаружить и количественно оценить широкий спектр изменений в мРНК синтез ставки.
протокол
1. клетки культивирования и истощение Rapamycin субъединицы Сага
- Для каждого штамма S. cerevisiae и репликации включая одичал тип или управления штаммов, прививать единую колонию из свежих пластину на 5 мл среды YPD (2% Пептон, экстракт дрожжей 1% и 2% раствор глюкозы).
- Рост клеток S. cerevisiae ночь в 30 ° C с постоянным перемешиванием (150 об/мин).
- Измеряют оптическую плотность на 600 Нм (600ОД) и развести культуры ОД600 приблизительно 0,1 в 100 мл YPD среднего и пусть растут до од600 составляет около 0,8.
- Параллельно, прививать единой колонии клеток S. pombe из свежих пластину на 50 мл среды Да (0,5% экстракт дрожжей; 250 мг/Л аденин, гистидина, урацила, лейцин и лизин; 3% глюкозы) и расти клетки на ночь на 32 ° C с постоянным перемешиванием (150 об/мин).
- Измерить ОД600 S. pombe ночь культуры и разбавить культуры ОД600 приблизительно 0,1 в 500 мл среды да и пусть растут до од600 примерно 0,8.
- Для каждого штамма S. cerevisiae якорь прочь и репликации включая одичал тип или управления штаммов, прививать единую колонию из свежих пластину на 5 мл среды YPD (2% Пептон, экстракт дрожжей 1% и 2% раствор глюкозы).
- Рост клеток на ночь на 30 ° C с постоянным перемешиванием (150 об/мин).
- На следующее утро, измерить ОД600, разбавить культуры ОД600 приблизительно 0,1 в 100 мл YPD среднего и пусть растут до OD600≈ 0.8.
- 100 мкл rapamycin культуре от Стоковый раствор 1 мг/мл (окончательный rapamycin концентрация составляет 1 мкг/мл) и пусть Инкубируйте на 30 ° C с постоянным перемешиванием на время, необходимое для протеина интереса к условно истощены от ядра (клетки как правило 30 мин адекватной). Для элемента управления используйте аналогичные культуры дрожжей, но вместо добавления rapamycin, добавьте эквивалентный объем диметилсульфоксида (ДМСО).
2. 4tU маркировки с S. pombe как Спайк в (подсчет)
-
Готовить свежий раствор 2 M 4-thiouracil. После подготовки, держите его при комнатной температуре и вдали от света.
- Точно взвесить 64,1 мг 4-thiouracil для каждой культуры S. cerevisiae и растворить его в 250 мкл диметилформамида (DMF) или 250 мкл ДМСО.
- Для культуры S. pombe использоваться как Спайк в весят 320.5 мг 4-thiouracil и растворить его в 1250 мкл ДМСО.
- Добавьте решение 4-thiouracil S. cerevisiae и S. pombe культур для конечной концентрации 5 мм и инкубировать их за 6 мин с постоянным перемешиванием в 30 ° C до 32 ° C, соответственно.
- Через 6 мин удалите небольшой Алиготе каждой культуры для подсчета клеток. Подсчет количества ячеек, используя счетчик автоматическое клеток или Нойбауэр камеры.
- Собрать клетки путем центрифугирования (2500 x g) за 5 мин при 4 ° C.
- Отбросить супернатанта, вымыть клетки с ледяной ПБС и снова центрифуги (2500 x g, 4 ° C, 5 мин).
- Рассчитайте общее количество клеток в каждом из образцов S. cerevisiae и S. pombe .
- Ресуспензируйте клетки в 5 мл ледяной ПБС и смешайте S. cerevisiae с S. pombe клетки с соотношении 3:1.
- Центрифуга клетки (2500 x g, 4 ° C, 5 мин), удалите PBS, флэш замораживание клеток в жидком N2и хранить образца при температуре-80 ° C до дальнейшего использования.
3. РНК добыча и обработка DNase
- Оттепель клетки на льду для приблизительно 20 – 30 мин.
- Продолжите извлечение RNA, с использованием дрожжей РНК добыча комплект (Таблица материалов) с несколько адаптаций.
- За каждого образца наливайте 750 мкл ледяной циркония бисера в 1,5 мл колпачок трубки поставляется с комплектом. Имейте в виду, что за каждой трубы, могут быть эффективно извлечены РНК от9 до 10 клеток. Следовательно Подготовьте количество трубок, необходимых для каждого образца. Например, S. cerevisiae культуры по 100 мл (OD600≈ 0,8) может оказать9 около 2 x 10-3 х 109 клеток, вплоть до в общей сложности 2,7 х 109 -4 x 109 клеток в общей сложности (после всплеска в треть S. pombe клетки). В этом случае будет потребоваться до 3-4 реакции трубок каждого образца/состояние/мутант/репликацию.
- За каждый 1 x 109 клеток добавьте 480 мкл буфера lysis, комплект, 48 мкл 10% SDS и 480 мкл фенола: хлороформ: изоамилового спирта (25:24:1, v/v/v).
- Смешать ячейки, используя вихревой смеситель и перенести их в пробирки, содержащие ледяные шарики циркония.
- Размещения труб на вихревой смеситель адаптер, повернуть vortex на максимальной скорости и бить в течение 10 минут, чтобы лизировать клетки дрожжей (в комнате на 4 ° C). Кроме того выполните лизис клеток в автоматической шарик колотушки.
- Центрифуга для трубы на 16000 x g 5 мин при комнатной температуре и тщательно собирать Верхний этап (этап РНК содержащих) свежие 15 мл трубки сокола. Обычно, объем, восстановленные в каждую пробирку составляет около 500-600 мкл.
- Для 15 мл пробирки, содержащие частично очищенный РНК добавьте привязки буфер, предоставленный с комплектом и смесь тщательно. За каждый 100 мкл раствора РНК, мкл 350 привязки буфера (то есть, когда объем водной фазе раствора составляет 600 мкл, 2.1 мл буфера привязки должны быть добавлены).
- К предыдущей смеси добавить 100% этанола и тщательно перемешать. За каждый 100 мкл раствора РНК, мкл 235 100% этанола (то есть, когда объем водной фазе раствора составляет 600 мкл, добавить 1.41 мл этанола).
- Примените до 700 мкл в смеси с шагом 3,9 картриджа фильтра собранные в коллекции трубку, предоставлены в комплект.
- Центрифуги для 1 мин на 16000 x g. Если продолжительность центрифугирования не было достаточно для общего объема пройти через фильтр, повторите центрифугирования для 30 s.
- Отказаться от потока через и повторно же коллекции трубки. Добавьте еще 700 мкл RNA-связывая буфера этанола раствор для фильтрации и центрифугирования снова на 16000 x g за 1 мин.
- Отказаться от потока через и повторите шаги 3.11 и 3.12 до тех пор, пока раствор РНК закончена.
- Промойте фильтр 2 x с 700 мкл концентрированного моющего раствора 1. Собирать стиральной решение через центрифугирования в 16000 x g за 1 мин и всегда держать трубку коллекции.
- Промойте фильтр 2 x с 500 мкл концентрированного моющего раствора 2. Собирать стиральной решение через центрифугирования в 16000 x g за 1 мин и всегда держать трубку коллекции.
- Центрифуга трубы еще раз на 16000 x g за 1 мин до полностью высушите фильтр.
- Передать окончательный коллекции трубки (РНК соответствующие трубки) картриджа фильтра и элюировать РНК с 50 мкл DEPC-лечение, бесплатные РНКазы H2O (разогретую до 100 ° C).
- Центрифуги для 1 мин на 16000 x g.
- Элюировать РНК снова (чтобы же трубки) с 50 мкл разогретой DEPC-лечение, бесплатные РНКазы H2O. Убедитесь в том, что весь объем прошло через фильтр; в противном случае центрифуги для более длительных периодов.
- Если используются несколько трубок для одного единого образца, объединить их все в одной из труб.
- Количественно оценить и проверить чистоту образца с помощью соответствующего оборудования.
Примечание: Хотя 4tU только включены в недавно синтезированных РНК, есть шанс мелкие загрязнения с ДНК. По этой причине, это всегда целесообразно рассматривать образцы с DNase I. Для этого используйте реагенты, РНК добыча комплект (Таблица материалов) следуя рекомендациям изготовителя.
4. тиоловых конкретных Biotinylation недавно синтезированных РНК
- Регулировка концентрации РНК, полученные с разделом 3 Протокола до 2 мг/мл. Аликвота 200 мкг всего РНК, тепло его на 10 мин при 60 ° C и сразу же охладить на льду на 2 мин.
-
Для РНК Алиготе, добавьте реагентов, упомянутых ниже в следующем порядке: 600 мкл DEPC-лечение, бесплатные РНКазы H2O, 100 мкл буфера biotinylation (100 мм трис-HCl [pH 7.5] и 10 мм ЭДТА, в DEPC-лечение, RNase свободный H2O) и 200 мкл биотин HPDP от запас биотин HPDP 1 мг/мл в ДМСО или функцию DMF.
- В некоторых ситуациях биотин HPDP решение, как правило, осадок, вероятно из-за его низкой растворимостью в воде. В этой ситуации, увеличить объем ДМСО/DMF до 40% от объема реакции (на пример РНК, мкл 400 DEPC-лечение H2O, 100 мкл буфера biotinylation и 400 мкл биотин HPDP из запаса 0.5 мг/мл).
- Проинкубируйте образцы при комнатной температуре и защищены от света для 3 h, с нежным агитации.
- После инкубации добавьте примерно равное количество метилхлороформа трубы и смесь энергично.
- Спиновые образца на 13 000 x g за 5 мин, при 4 ° C. Этот шаг позволяет удаление избыточного биотина, что сделал не biotinylate РНК. Кроме того выполните этот шаг, используя фазы lock трубы (тяжелый). Для этого спина вниз трубы упора фаза 1 мин на 13 000 x g, добавьте смесь РНК и равное количество метилхлороформа, смешать их энергично и центрифуги их на 13 000 x g за 5 мин, при 4 ° C.
- Тщательно перенесите Верхний этап в новые трубы 2 мл.
- Добавить одну десятую от объема 5 М NaCl и перемешайте образец.
- Добавить равное количество изопропиловый спирт, тщательно перемешать образца и спина его на 13 000 x g для по крайней мере 30 мин, при 4 ° C.
- Осторожно удалить супернатант и добавьте 1 mL ледяной 75% этанола.
- Спина на 13 000 x g за 10 мин., на 4 ° C.
- Тщательно удалите супернатанта, быстро спин трубки и остальные этанола раствор. Убедитесь, что гранулы РНК не сухой.
- Приостановить РНК в 100 мкл DEPC-лечение, бесплатные РНКазы H2O.
5. Очистка фракции заново синтезированные от всего и непомеченного РНК с помощью стрептавидина покрытием магнитные бусы
- Тепла биотинилированным РНК в течение 10 минут при температуре 65 ° C и затем охладить образцы на льду на 5 мин.
- Добавьте 100 мкл стрептавидина покрытием магнитные бусы биотинилированным РНК (на окончательный объем 200 мкл). В частности рекомендуется использовать бусы, указано в Таблице материалов, поскольку, после разговоров с другими лабораториями, эти, казалось, чтобы быть более последовательным и надежным.
- Инкубируйте образца с небольшой тряски 90 мин, при комнатной температуре.
- Разместите столбцы, комплект (Таблица материалов) в магнитного стенд.
- 900 мкл-комнатной Отмывающий буфер (100 мм трис-HCl [pH 7.5], ЭДТА 10 мм, 1 M NaCl и 0,1% Tween-20, в DEPC-лечение, RNase свободный H2O) со столбцами (предварительно запустить и сбалансировать).
- Применять смесь бусы/РНК (200 мкл) к столбцам.
- Сбор потока через в 1,5 мл пробирок и применить его вновь на один и тот же магнитный столбец. При необходимости, держите этот поток через, как он представляет собой немеченого фракция РНК.
- Вымойте колонки 5 x с увеличением объемов отмывающего буфера (600, 700, 800, 900 и 1000 мкл).
- Элюировать заново синтезированные РНК с 200 мкл 0,1 м DTT.
- Выполните второй элюции, 3 минуты позже, с равным объемом 0,1 М DTT.
- После элюирующие РНК, добавить 0,1 объема 3 M NaOAc (рН 5.2), 3 тома ледяной 100% этанола и 2 мкл 20 мг/мл гликогена (РНК класс) и пусть РНК осадка на ночь, при-20 ° C.
- Восстановить РНК путем центрифугирования (13 000 x g 10 минут, при температуре 4 ° C) и Ресуспензируйте в 15 мкл DEPC-лечение, бесплатные РНКазы H20. Ожидается, что доля РНК помечены всего около 2% - 4% (как правило, больше в нижнем конце), оказание около 2,0 мкг недавно синтезированных РНК. Этого количества достаточно, чтобы сделать несколько ПЦР экспериментов, а также для анализа microarray/последовательности.
6. RT-ПЦР проверки различных фракций
- Синтез cDNA от 2 мкг всего РНК или 10 мкл помечены РНК, с использованием случайных hexamers и обратная транскриптаза выбора, согласно инструкциям производителя (Таблица материалов).
- Увеличьте cDNA на ПЦР в реальном времени, используя стандартный протокол (Таблица материалов).
Примечание: Все образцы должны выполняться в трех экземплярах из как минимум двух биологических реплицирует. Исправьте все необработанные значения для выражения тубулин S. pombe .
7. Microarray гибридизации
- Гибридизируйте образцов РНК на обломоки microarray предпочтительным согласно инструкциям производителя (для этого конкретного протокола, см. Таблицу материалы). Вкратце, подготовить биотинилированным cRNA цели от 150 нг РНК с помощью премьер комплекта амплификации РНК (Таблица материалов), согласно инструкциям производителя. Гибридизируйте 4 мг фрагментированных cRNAs для 16 h 45 ° c и 60 rpm на обломоки microarray.
- Вымойте, пятно и сканировать фишки, используя указанные станции и сканера (Таблица материалов). Извлечение исходных данных (CEL интенсивности файлов) из отсканированных изображений, с помощью командной консоли (AGCC, версия 4.1.2).
- Далее процесс CEL файлы с версией программного обеспечения консоли выражение 1.4.1 для вычисления зонда набор интенсивности сигнала, с использованием алгоритмов на основе статистики MAS 5.0 с параметрами по умолчанию и Глобальный Скейлинг как метод нормализации.
Примечание: Подстриженные средний целевой интенсивности каждого чипа был произвольно равным 100. Выполнение всех экспериментов с использованием по крайней мере двух независимых биологических реплицирует. Нормализовать необработанные данные на S. pombe сигнал и рассчитать раза изменения уровня общей и заново синтезированные РНК.
8. анализ данных с помощью существующего трубопровода R
- Рассчитать синтеза и распада ставки с помощью конвейера и R/Bioconductor пакета публично доступны, как описано в8,10.
Результаты
При выполнении метаболических маркировки недавно транскрипции РНК, некоторые аспекты необходимо контролировать: время и эффективности маркировки, Спайк в пропорции, извлечение протокола и biotinylation эффективности (включая соотношение сигнал шум), среди других. Эти условия были широкие и методически показаны другие7,10,11. Здесь мы главным образом сосредоточиться на интерпретации и немедленного анализа, которые могут быть выполнены после того, как образцы были обработаны, RT-ПЦР, microarray дна или последовательности. Анализ различных мутантных штаммов продемонстрировать мощь метода для выявления не только резкое снижение глобальный синтез мРНК, как и в случае с Сага мутант S. cerevisiae штаммов, но и очень мягкий уменьшения РНК полимеразой II деятельности по подавление гистона H2B monoubiquitination.
Наш анализ Сага ферментативной деятельности предлагает широкий набор хроматина15, которая не была выявлена путем анализа установившегося мРНК уровня в Сага мутантных штаммов. Как РНК полимеразой II набора была нарушена после Сага инактивации, мы решили проанализировать ли ставки синтез мРНК глобально пострадает. Следовательно одичал тип или мутантных штаммов S. cerevisiae были подвержены 4tU продолжительностью в 6 мин, чтобы обозначить недавно транскрипции РНК. После смешивания с Спайк в помеченных клеток (S. pombe) в пропорции 3:1, всего РНК был извлечен, и недавно синтезированных РНК биотинилированным и очищенный согласно протоколу, представленные здесь, как и интеренет, показанный на рисунке 1. Помечены РНК были очищены от в общей сложности 200 мкг РНК, обеспечивая, что количество очищенного продукта будет достаточно для любого приложения, ниже по течению. Первоначальный и систематического шага перед любой анализ генома широкий недавно синтезированных РНК очистки был подтвержден RT-ПЦР. Гены были выбраны согласно различных параметров, включая уровень выражения, нормативные пути и зависимость на различных РНК-полимеразы.
Чтобы подтвердить, что этот протокол специально очищает помечены РНК, мы количественно уровни стенограммы в долях, очищенного от дикого типа клеток, которые были культивируемых с или без 4tU. Незначительный фоновый уровень анализируемого RNAs были обнаружены из клеток, которые не были подвержены 4tU(рис. 2). Далее вновь транскрипции РНК очистки был подтвержден наблюдается обогащение Интрон содержащих 1 акт пре мРНК (данные не показаны). После проверки качества образцов мы проверили ли синтез мРНК будут затронуты после удаления SPT20, который известен сорвать комплекса Ассамблеи Сага. Как сообщили другие, квантификация мРНК на общее (устойчивого состояния) РНК от штамма Δ spt20показали основном неизменным или умеренно снижение уровней для протестированных генов (рис. 2B). Аналогичные результаты были получены для штаммов, удалены для BRE1 (рис. 2B). В противоположность этому, анализ новых транскрипции РНК от штамма Δ spt20показал резкое снижение синтеза мРНК на три-в пять раз для всех протестированных генов (рис. 2C). Потеря Bre1 привело к более сдержанный, но еще видны снижение уровня вновь синтезированной мРНК для изученных генов (рис. 2C). Хорошо согласуется с ролью Сага и Bre1 в транскрипции РНК полимеразой II, потеря Spt20 или Bre1 не влияет на выражение RDN25 или snR6 генов, транскрибируются РНК полимеразой я и РНК полимеразой III, соответственно ( Рисунок 2C).
Однако штаммы, удаленных структурных подразделений комплекса, как SPT7 или SPT20, Сага отображения тяжелой фенотипов медленного роста, которые могут стать причиной наблюдаемых transcriptional изменений. Чтобы исключить нежелательные вторичные последствия, мы условно обедненного Spt7 из ядра, с помощью анкера прочь, S. cerevisiae штаммов14,24. После 60 мин rapamycin лечения, до пульс маркировки с 4tU (см. Рисунок 1 схематическое представление), недавно трансляции мРНК уровни были сокращены же степени, что наблюдается в исключить деформации (Рисунок 2D и -2E ). Этот анализ, таким образом, подтвердил наш бывший результаты и проверить протокол для этой системы индуцибельной истощения. В анализе времени курс, где клетки подвергаются rapamycin за время охватывающие от 0 до 240 мин, снижение выражение проявляется сразу же после 15 минут воздействия препарата. Более интересно уровни mRNA устойчивого состояния, как правило, первоначально уменьшить, но вернулся к нормальному после 60 мин, признак, что компенсационный механизм происходит в то же время (рис. 2E).
В целом маркировки и количественной оценки недавно синтезированных РНК позволило выявить новые нормативной роли для комплекса Сага. Описывается протокол мог бы также выявить умеренное воздействие на РНК полимеразой II деятельности и был успешно применяется для условного истощения штаммов дрожжей.
Один из нисходящие приложения для очищенного заново синтезированные РНК является геном общесистемной количественную оценку стенограммы с помощью microarray гибридизации или последовательности (4tU-seq). В то время как высокопроизводительного секвенирования более количественный, чувствительной и информативным, microarray гибридизации может быть очень полезно определить, изменены ли глобальные уровни mRNA. В этом контексте, где важна нормализации мы добавили Спайк в организм в образец, который мы стремились проанализировать. В частности Мы смешали S. cerevisiae клетки на S. pombe ячейки в соотношении 3:1, оба ранее подвергается 4tU. Когда очищенная, обозначенные РНК подвергаются высокопроизводительного секвенирования, можно использовать протокол подготовки любой стандартной библиотеки, чаще всего после истощения рибосомной РНК. Нормализации между 4tU-seq данных из различных образцов была выполнена путем добавления либо помечены РНК из22 различных видов (т.е., смешивая S. cerevisiae и S. pombe клетки как выше) или в пробирке- транскрипции, thiolated Спайк в РНК12,25,,2627. Обломоки microarray коммерчески доступных содержат зонды для всей транскриптом как начинающим, так и деления дрожжей, позволяя количественная оценка mRNA от обоих организмов в один один эксперимент. Гибридизациях microarray дна были выполнены с общей и заново синтезированные РНК, подтверждены RT-ПЦР, как указывалось выше (одичал тип, spt20Δ и bre1Δ). Кроме того мы включили штамм, который не поддерживает monoubiquitination гистона H2B, через Точечная мутация остатков ubiquitinable (K123R). Поскольку точное же долю S. pombe клетки были использованы в различных образцов и реплицирует, возможна линейно масштаб в массивы света таким образом, чтобы общая и помечены S. pombe зонды имеют той же средней интенсивности. Эта нормализации, или масштабирование, могут быть выполнены с помощью Bioconductor пакет, разработанный в лаборатории Патрик Крамер и используется параллельно во всех проанализированных пробах, Общая и помечены фракций и дикого типа и мутантов штаммов8. Вкратце ввод этого конвейера является файл Excel, содержащий датчики и их интенсивность (MAS5 или RMA) для всех образцов и фракций. После исключения зондами, которые находятся за пределами диапазона обнаружения, является ремасштабирована интенсивности сигнала, принимая во внимание значения интенсивности зондов, S. pombe . Наконец можно сопоставить зонды бутонизации и деления дрожжевой геномов, заканчивая матрицы, содержащие значения выражение, нормированный для всплеска в. Эти значения могут быть дальнейшей обработки (см. следующий раздел) или используется как. В следующем примере мы определили, что раз изменить каждый Стенограмма между мутант и дикого штамма.
Анализы были проведены для обоих установившемся или недавно синтезированных уровни РНК и заговор против их статистической значимости (p-значение) (рис. 3). По согласованию с другими исследованиями, когда общий уровень РНК были проанализированы, только несколько генов были их выражения изменены либо вверх - или downregulated (Рисунок 3А-3 C). Однако анализ новых транскрипции РНК привело к поразительно различные выводы. Вновь переписаны уровни mRNA более чем 4000 генов были значительно сокращены по крайней мере два раза после удаления SPT20, предлагая глобальное позитивное воздействие саги на транскрипции РНК полимеразой II в многообещающий дрождей (рис. 3D ). Кроме того, в bre1Δ и K123R мутантов, результаты были более сдержанный: большинство генов, по-видимому, их выражение сокращены, но степень Даунрегуляция и количество значительно пострадавших генов (≈ 300-500) был действительно более Limited (Рисунок 3E и 3F).
Как ранее упоминалось, стационарном или общего уровня мРНК продиктованы жесткие равновесия между синтеза и распада (рис. 4A). Когда транскрипции РНК полимеразой II глобально нарушается, можно на фото два сценария: (i) либо всего мРНК уровни уменьшаться глобально, в ответ на снижение синтеза, но постоянные распада, или деградации мРНК (ii) уменьшается в той же степени, полученный в основном неизменным уровни mRNA устойчивого состояния. Второй сценарий сообщалось несколько условий, в том числе в контексте Сага или TFIID нарушения9,10,14,16,22. Процедура называется анализ сравнительных динамических транскриптом (cDTA) на основе 4tU маркировки и динамических кинетического моделирования позволяет нам определить синтез мРНК и вывести распада ставки за каждую запись8,10. Еще раз мы воспользовались данные, собранные для ранее упомянутых штаммов (одичал тип, spt20Δ, bre1Δ и K123R). Как и ожидалось, после удаления SPT20, мы наблюдали с одновременным снижением мРНК синтеза и деградации ставки по сравнению с одичал тип деформации. В этот мутантный штамм компенсация была почти оптимальное, с среднем снижение синтеза 3.8-fold и среднем снижение распада 4.1-fold (рис. 4B), подкрепляющих почему могут быть лишь ограниченные transcriptional изменений обнаружен на общий уровень мРНК (рисA). В двух других мутантных штаммов (bre1Δ и K123R), также наблюдались сопутствующие изменения в синтез мРНК и распада, но изменения были в гораздо меньших масштабах и более дисперсионные (Рисунок 4 c и 4 D).
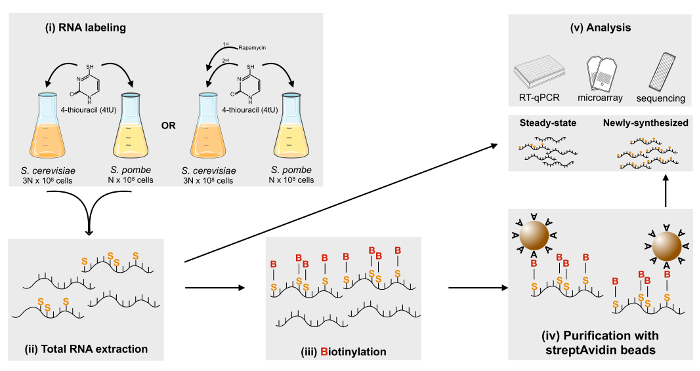
Рисунок 1 : Схематическое изображение метаболических маркировки РНК с помощью 4tU. Свежеприготовленные 4tU добавляется к питательной среды и клетки помечены на 6 мин Приклеенные этикетку S. cerevisiae и S. pombe клетки смешивают в соотношении 3:1 и общая РНК добывается. Потом недавно синтезированных РНК биотинилированным, могут быть очищены с помощью стрептавидина покрытием магнитные бусы. Наконец, всего РНК (устойчивого состояния) и помечены заново синтезированные РНК может использоваться в различных нисходящие приложения, включая RT-ПЦР и microarray гибридизации или последовательности. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
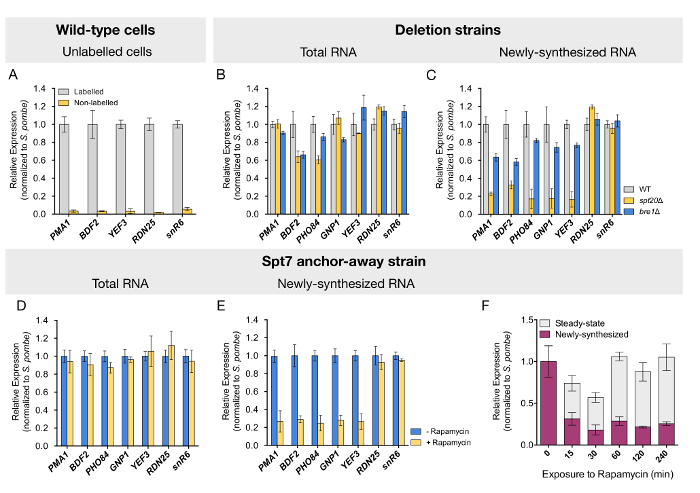
Рисунок 2 : Анализ, изображающие transcriptional изменений в обоих установившемся и вновь транскрипции РНК, определяется RT-ПЦР. (A) РНК уровни пяти различных генов были количественно помечены фракции РНК, очищенного от одичал тип клеток, которые были либо предоставлены или не 4tU. Следующие две панели показывают (B) общей и (C) недавно синтезированных РНК квантификации по RT-ПЦР для одичал тип (WT), spt20Δ и bre1Δ дрожжевых клеток. Якорь прочь штаммов Spt7, не лечить или лечить с rapamycin 60 мин, были помечены 4tU, и (E) или (D) общей недавно транскрипции РНК была количественно оценена по RT-ПЦР. (F) этой группы показывает, что время курс анализа изменений в стационарном состоянии и недавно синтезированных мРНК на Spt7 ядерных истощения. Для всех образцов уровни mRNA для пяти РНК полимеразой II гены были количественно по RT-ПЦР. РНК-полимераза I и РНК полимеразой III генов (RDN25 и snR6, соответственно) были использованы в качестве контроля. Значения выражений (означает SD ± трех независимых экспериментов) были нормализованы с шипами в S. pombe сигнал и установите 1 в примере элемента управления. Были изменены панели D-F от Баптиста и др. 14. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
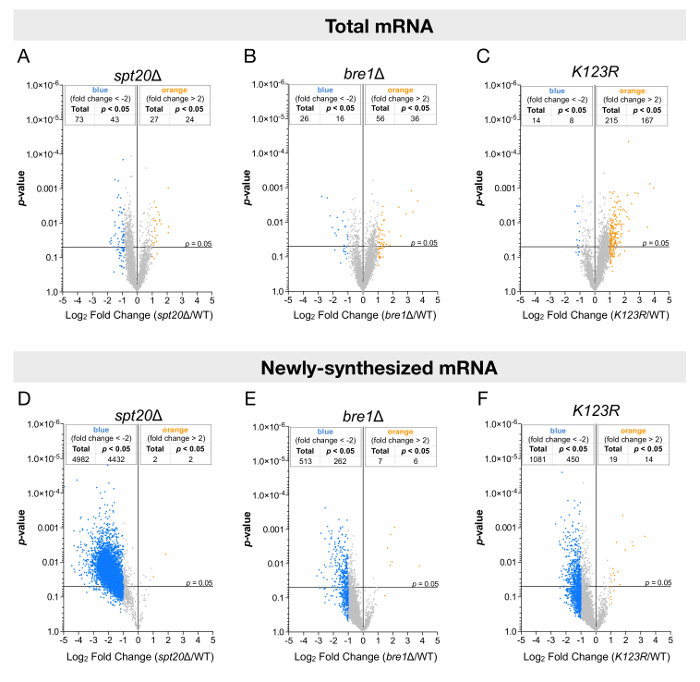
Рисунок 3 : Анализ генома общесистемной уровни mRNA, используя полное или помечены РНК фракций. Эти панели показывают вулкан участков показаны раз изменения уровня мРНК устойчивого состояния (A-C) или (D-F) недавно синтезированных уровни mRNA по отношению к их значимости (p-значение). Раз изменений (FC) рассчитывались как log2 отношение значения выражения каждого гена после нормализации на S. pombe сигнал в (A и D) spt20Δ, Δ bre1(B и E) или () C и F) K123R штамм против значение выражения такого же гена в одичал типа S. cerevisiae. В общей сложности 5385 генов были проанализированы, и изменить пороговые значения двойной (синяя точка: более чем двукратное уменьшение; желтые точки: более чем двукратное увеличение) и 0,05 p-значения были рассмотрены. Панели и D были изменены от Баптиста и др. 14. пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
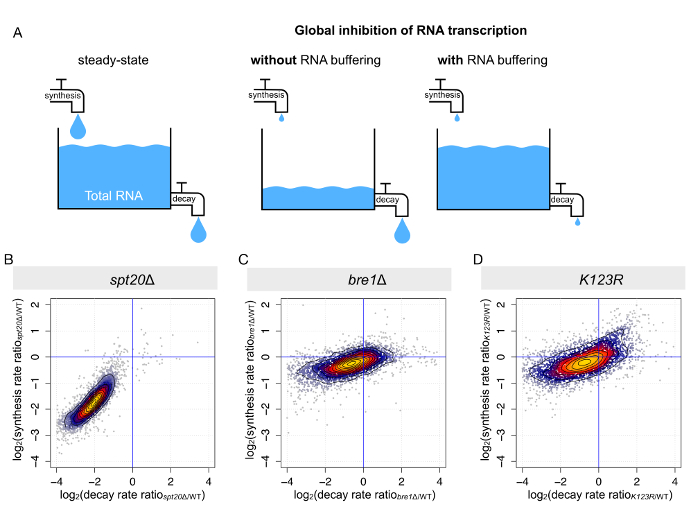
Рисунок 4 : Параллельные изменения в синтез мРНК и мРНК распада приводит к буферизации мРНК. (A) Эта группа показывает схематическое представление итогов возмущений синтез РНК на стационарном уровнях РНК. (B-D) Эти панели показывают расчет ставок распада из анализа общего и заново синтезированные РНК и синтез мРНК. Синтез и распад ставки были определены для каждого S. cerevisiae Стенограмма (B) spt20Δ, Δ bre1(C) и (D) K123R. Изменения (рассчитывается как log2 соотношение между мутант и одичал тип) синтез ставок были заговоре против изменения ставок распада. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
В то время как генома общесистемной инструментов для анализа изменений в транскрипции по-прежнему улучшается, единственным анализ транскриптом путем количественной оценки устойчивого состояния уровня РНК не точно может отражать изменения РНК полимеразой II деятельности. Действительно уровни mRNA регулируются не только синтез РНК, но и их созревания и деградации. Чтобы измерить синтез мРНК, расцеплено от деградации мРНК, различные протоколы были разработаны в последние годы для анализа зарождающейся транскрипции в дрожжей и млекопитающих.
Одним из наиболее широко используемых протоколов для количественного определения нарождающихся транскрипции — GRO-seq1. Хотя главным преимуществом GRO-seq является его способность раскрыть транскрипционно заинтересованное и активное полимеразы на низких фоновых и высоким разрешением, он имеет два различных точек, которые снижают его привлекательность: (i) она является технически сложным и требует изоляции ядра и, (ii) ядер манипуляции ввести некоторые возмущениям системы28. Другой альтернативой является NET-seq, которая опирается на последовательность 3' концы стенограммы, получены по иммунопреципитации чрезвычайно стабильной троичного комплекса формируется между зарождающейся РНК, шаблон ДНК и РНК-полимераза II. Этот подход позволяет характеристика зарождающейся РНК с разрешением пары и могут исследовать мРНК обработки событий через иммунопреципитации модифицированных РНК-полимераза II (фосфорилирование Серина-5, к примеру)2,3 , 29. Тем не менее, он представляет некоторые препятствия, которые мы выделяем три. Во-первых в то время как антитела, ориентация РНК-полимераза II, как правило, весьма специфический, это зависит от эффективности антитела. Во-вторых, РНК могут быть подвержены деградации в инкубационный период, таким образом ведет нас обратно к предыдущей точке (менее эффективным антител может потребоваться больше инкубации раз, оставляя более подвержены деградации МРНК)29. Третье и особенно в млекопитающих, MNase пищеварение и выбор размера может исключить уникальную последовательность выравнивание29,30.
Здесь мы представили подробный протокол для метаболических маркировки заново синтезированные РНК с помощью 4tU в S. cerevisiae , что имеет различные преимущества по сравнению с другими имеющихся подходов. Потому что транскрипции очень чувствительна к возмущениям, клетки следует сохранить в наиболее физиологических условиях. К примеру ГРУ seq предполагает арест транскрипционно занимается РНК-полимераза II путем воздействия ядер клеток sarkosyl. Однако лечение sarkosyl был описан как тормозящий несколько клеточных процессов11. В этом случае метаболические маркировки с 4tU или 4СУ в описанных концентрации не нарушается и не влияет на заметно гомеостаза клетки, особенно для коротких периодов воздействия. В отличие от других методов, с помощью ингибирование транскрипции для измерения mRNA полураспада cDTA или 4tU-seq и фитинги с динамическое моделирование определяет темпы деградации каждого одной мРНК в невозмущенной клеток. Следовательно один единственный метод может одновременно решать синтез и распад темпы весь транскриптом на конкретные ячейки типа. Так как cDTA или 4tU-seq использует надежные нормализации методов, а именно через использование Спайк в различных наборов данных можно проанализированы вместе и непосредственно сравнивать. Наконец метаболические маркировки и количественной оценки недавно синтезированных РНК является метод, который может быть легко реализован в любой лаборатории молекулярной биологии, как он не требует какого-либо конкретного оборудования. Это вероятно объясняет довольно большое распространение этой техники, которая имеет тенденцию к более и более систематически использоваться для изучения производства РНК и деградации дрожжей, а также в высших эукариот.
РНК могут быть помечены в живых клетках, используя другие Нуклеозидные аналоги, а именно 5-bromouridine (BrU)31,32 или 5-ethynyluridine (ЕС)33,34. Изоляции BrU пульс меченых РНК опирается на Очищение антитела анти BrdU, которые могут иметь различные эффективности между экспериментов. ЕС меченых РНК можно ковалентно конъюгированных для биотина с помощью нажмите химии, ведущих к несимметричных конъюгации в отличие от тиоловых модификации, которые могут быть отменены с восстановителями. Бру и маркировки ЕС были использованы в mammalian клетках для определения генома общесистемной РНК распада ставки31,32 или оценить зарождающейся РНК синтеза34,35. Однако Брю или ЕС маркировки не был описан в S. cerevisiae. Действительно поглощение Нуклеозидные аналоги требуется выражение нуклеозидный транспортер, и, таким образом, эти методы кажутся менее гибкими в многообещающий дрожжей, чем маркировки с 4tU.
Продолжительность 4tU маркировки может быть адаптирована и зависит вопрос. Когда оценки синтез мРНК в S. cerevisiae, короткий импульс маркировки 6 мин гарантирует, что недавно транскрибируется уровни mRNA минимально подвержены деградации РНК. Учитывая, что время задержки, прежде чем измененные нуклеотидов может быть включена в зарождающейся РНК — менее 1 мин, этот 6-Минимальная продолжительность маркировки гарантирует, что разумное количество вновь синтезированных РНК могут быть очищены. Такой протокол, как определено Миллер и др. 11, был использован в различных S. cerevisiae мутантов, чтобы выявить глобальные изменения в синтез мРНК и РНК распад9,10,22,26,27, 36. продолжительностью больше маркировки (3 h) был использован в метаболических пульс Чейз маркировки с 4tU для определения ставок распад всех мРНК в S. cerevisiae12. Наконец чрезвычайно короткие (от 1,5 мин) маркировки 4tU был использован для изучения кинетики обработки РНК в бутонизации и деления дрожжевой13,,2537.
Тем не менее этот метод имеет некоторые ограничения, например, относительно низкая доходность помечены РНК, восстановленные в конце всего протокола. Хотя в дрожжи, нет никаких ограничений в исходный материал, и это может быть проблемой при анализе многоклеточные клетки, специально если этот протокол будучи использованы в vivo. Одним из ограничивающих действия протокола является biotinylation помечены РНК пула, эффективность которого является далеко не полным и оценивалась изменить один из трех остатков 4СУ в обозначенные РНК5. Он недавно показали, что использование methanethiosulfonate (МТС)-биотин, вместо биотин HPDP, увеличивает урожайность восстановленные РНК38. Однако по словам неопубликованные результаты этой исследовательской группы и результаты от Рутковский и Dölken, МТС биотин не полностью тиоловых конкретным, ведущих к очищению немеченого РНК, которая является особенно проблематичным, когда низкое количество помечены РНК очищенный39. Еще одним ограничением метаболических маркировки с помощью 4tU/4СУ является присущие скорость, на которой РНК полимеразы удлиняется. Средняя скорость РНК-полимераза II составляла около 3,5 КБ/мин40,,4142. Следовательно в 6 мин маркировки, полимеразы будет иметь способность записать 15 – 20 КБ. Таким образом, в эксперименте с короткого импульса маркировки с 4tU/4СУ, только 3' конца частью недавно транскрипции РНК обозначена, а 5' конца регионов были ранее существовавших перед добавлением 4tU/4СУ. Таким образом очистка помечены РНК также обогащает существующие 5' концы стенограмм, особенно для длинные стенограммы как в mammalian клетках. Для преодоления этого предубеждения, в изысканной протокол под названием TT-seq, извлеченные помечены РНК фрагментирована, sonication до очистки заново синтезированные видов43.
Вообще 4tU-seq является отличным способом для транскрипционный анализ изменения адреса в данном контексте. Тем не менее и хотя протокол довольно прост, он состоит из нескольких различных шагов, будучи в конечном счете относительно обширные. Во-первых поскольку этот протокол обрабатывает РНК от начала до конца, это обязательно будут выполнены все требования, необходимые для образца чистой и свободной от деградации. Следовательно все реагенты следует РНКазы бесплатно, и все материалы должны быть посвящены манипулирования РНК, очистки все тщательно и регулярно раствором РНКазы обеззараживания. Во-вторых в то время как биотин реакция весьма специфические, избыток биотин HPDP, которые не реагируют должны удаляться из образца (чтобы избежать насыщения стрептавидина бусины с биотина, что не обязан ковалентно РНК, например). В-третьих, как описано в протоколе, настоятельно рекомендуется использовать указанные стрептавидина покрытием магнитные бусы, поскольку несколько исследовательских групп, у нас говорили все указали, что эти бусы привело к снижению фоновых уровней. В-четвертых, соответствующие качеству и экспериментальных элементов должны использоваться. Включает образцы, полученные из клеток, которые не были подвержены 4tU/4СУ. Эти образцы будут иметь большую ценность для адрес фоновый уровень, и чтобы убедиться, что во время эксперимента, не происходит загрязнения с-меченых РНК. Кроме того, еще одна отличная альтернатива проверить ли образец обогащается для недавно синтезированных РНК — выполнить RT-ПЦР с праймерами против интро содержащие ген. Праймеры должно дополнять 3' конца и 5' концу двух последовательных экзона и Интрон или наоборот. Наконец до последовательности microarray гибридизации, проверка образцов по RT-ПЦР. Для этого различных генов с разными уровнями выражение и регулирования следует выбрать и проанализированы. Оптимально это следует выполнять для двух различных фракций РНК (устойчивого состояния и заново синтезированные РНК).
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим Tora Ласло за его поддержку и V. Фишер, K. Schumacher и F. El Saafin для их обсуждения. Т.б. была поддержана Мари Кюри-ITN стипендий (PITN-GA-2013-606806, NR-NET) и Фонд дуги. Эта работа была поддержана средств от Agence Nationale de la Recherche (АНР-15-CE11-0022 SAGA2). Это исследование было также поддержано АНР-10-LABX-0030-INRT, французский государственный фонд управляется Agence Nationale de la Recherche под рамки программы Investissements будущее АНР-10-IDEX-0002-02.
Материалы
| Name | Company | Catalog Number | Comments |
| 4-Thiouracil | Sigma-Aldrich | Cat# 440736 | |
| Rapamycin | Euromedex | Cat# SYN-1185 | |
| Countess II FL Automated Cell Counter | ThermoFisher | N/A | |
| RiboPure RNA Purification kit, yeast | ThermoFisher | Cat# AM1926 | |
| NanoDrop 2000 Spectrophotometer | ThermoFisher | ND-2000 | |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | |
| EZ-Link HPDP Biotin | ThermoFisher | Cat# 21341 | |
| Thiolutin | Abcam | ab143556 | |
| µMACS Streptavidin kit | Miltenyi Biotec | Cat# 130-074-101 | |
| Transcriptor Reverse Transcriptase | Roche | 03 531 295 001 | |
| SYBR Green I Master | Roche | 4707516001 | |
| GeneChip Yeast Genome 2.0 | ThermoFisher | 900555 | |
| GeneChip Fluidics Station 450 | ThermoFisher | 00-0079 | |
| GeneChip Scanner 3000 7G | ThermoFisher | 00-0210 |
Ссылки
- Gardini, A. Global Run-On Sequencing (GRO-Seq). Methods in Molecular Biology. 1468, 111-120 (2017).
- Churchman, L. S., Weissman, J. S. Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature. 469 (7330), 368-373 (2011).
- Churchman, L. S., Weissman, J. S. Native elongating transcript sequencing (NET-seq). Current Protocols in Molecular Biology. , Chapter 4 Unit 4 11-17 (2012).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23 (2), 232-237 (2005).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14 (9), 1959-1972 (2008).
- Kenzelmann, M., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proceedings of the National Academy of Sciences of the United States of America. 104 (15), 6164-6169 (2007).
- Radle, B., et al. Metabolic labeling of newly transcribed RNA for high resolution gene expression profiling of RNA synthesis, processing and decay in cell culture. Journal of Visualized Experiments. (78), e50195(2013).
- Schwalb, B., et al. Measurement of genome-wide RNA synthesis and decay rates with Dynamic Transcriptome Analysis (DTA). Bioinformatics. 28 (6), 884-885 (2012).
- Sun, M., et al. Global analysis of eukaryotic mRNA degradation reveals Xrn1-dependent buffering of transcript levels. Molecular Cell. 52 (1), 52-62 (2013).
- Sun, M., et al. Comparative dynamic transcriptome analysis (cDTA) reveals mutual feedback between mRNA synthesis and degradation. Genome Research. 22 (7), 1350-1359 (2012).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Munchel, S. E., Shultzaberger, R. K., Takizawa, N., Weis, K. Dynamic profiling of mRNA turnover reveals gene-specific and system-wide regulation of mRNA decay. Molecular Biology of the Cell. 22 (15), 2787-2795 (2011).
- Eser, P., et al. Determinants of RNA metabolism in the Schizosaccharomyces pombe genome. Molecular Systems Biology. 12 (2), 857(2016).
- Baptista, T., et al. SAGA Is a General Cofactor for RNA Polymerase II Transcription. Molecular Cell. 68 (1), 130-143 (2017).
- Bonnet, J., et al. The SAGA coactivator complex acts on the whole transcribed genome and is required for RNA polymerase II transcription. Genes & Development. 28 (18), 1999-2012 (2014).
- Warfield, L., et al. Transcription of Nearly All Yeast RNA Polymerase II-Transcribed Genes Is Dependent on Transcription Factor TFIID. Molecular Cell. 68 (1), 118-129 (2017).
- Huisinga, K. L., Pugh, B. F. A genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA in Saccharomyces cerevisiae. Molecular Cell. 13 (4), 573-585 (2004).
- Lee, T. I., et al. Redundant roles for the TFIID and SAGA complexes in global transcription. Nature. 405 (6787), 701-704 (2000).
- Lenstra, T. L., et al. The specificity and topology of chromatin interaction pathways in yeast. Molecular Cell. 42 (4), 536-549 (2011).
- Plaschka, C., et al. Architecture of the RNA polymerase II-Mediator core initiation complex. Nature. 518 (7539), 376-380 (2015).
- Helenius, K., et al. Requirement of TFIIH kinase subunit Mat1 for RNA Pol II C-terminal domain Ser5 phosphorylation, transcription and mRNA turnover. Nucleic Acids Research. 39 (12), 5025-5035 (2011).
- Rodriguez-Molina, J. B., Tseng, S. C., Simonett, S. P., Taunton, J., Ansari, A. Z. Engineered Covalent Inactivation of TFIIH-Kinase Reveals an Elongation Checkpoint and Results in Widespread mRNA Stabilization. Molecular Cell. 63 (3), 433-444 (2016).
- Haimovich, G., et al. Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. Cell. 153 (5), 1000-1011 (2013).
- Haruki, H., Nishikawa, J., Laemmli, U. K. The anchor-away technique: rapid, conditional establishment of yeast mutant phenotypes. Molecular Cell. 31 (6), 925-932 (2008).
- Neymotin, B., Athanasiadou, R., Gresham, D. Determination of in vivo RNA kinetics using RATE-seq. RNA. 20 (10), 1645-1652 (2014).
- Shetty, A., et al. Spt5 Plays Vital Roles in the Control of Sense and Antisense Transcription Elongation. Molecular Cell. 66 (1), 77-88 (2017).
- Xu, Y., et al. Architecture of the RNA polymerase II-Paf1C-TFIIS transcription elongation complex. Nature Communications. 8, 15741(2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews. Genetics. 13 (10), 720-731 (2012).
- Nojima, T., Gomes, T., Carmo-Fonseca, M., Proudfoot, N. J. Mammalian NET-seq analysis defines nascent RNA profiles and associated RNA processing genome-wide. Nature Protocols. 11 (3), 413-428 (2016).
- Storvall, H., Ramskold, D., Sandberg, R. Efficient and comprehensive representation of uniqueness for next-generation sequencing by minimum unique length analyses. PloS One. 8 (1), 53822(2013).
- Tani, H., et al. Identification of hundreds of novel UPF1 target transcripts by direct determination of whole transcriptome stability. RNA Biology. 9 (11), 1370-1379 (2012).
- Tani, H., et al. Genome-wide determination of RNA stability reveals hundreds of short-lived noncoding transcripts in mammals. Genome Research. 22 (5), 947-956 (2012).
- Jao, C. Y., Salic, A. Exploring RNA transcription and turnover in vivo. by using click chemistry. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15779-15784 (2008).
- Palozola, K. C., et al. Mitotic transcription and waves of gene reactivation during mitotic exit. Science. 358 (6359), 119-122 (2017).
- Ardehali, M. B., et al. Polycomb Repressive Complex 2 Methylates Elongin A to Regulate Transcription. Molecular Cell. 68 (5), 872-884 (2017).
- Schulz, D., et al. Transcriptome surveillance by selective termination of noncoding RNA synthesis. Cell. 155 (5), 1075-1087 (2013).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Duffy, E. E., et al. Tracking Distinct RNA Populations Using Efficient and Reversible Covalent Chemistry. Molecular Cell. 59 (5), 858-866 (2015).
- Rutkowski, A. J., Dolken, L. High-Resolution Gene Expression Profiling of RNA Synthesis, Processing, and Decay by Metabolic Labeling of Newly Transcribed RNA Using 4-Thiouridine. Methods in Molecular Biology. 1507, 129-140 (2017).
- Darzacq, X., et al. In vivo dynamics of RNA polymerase II transcription. Nature Structural & Molecular Biology. 14 (9), 796-806 (2007).
- Fuchs, G., et al. Simultaneous measurement of genome-wide transcription elongation speeds and rates of RNA polymerase II transition into active elongation with 4sUDRB-seq. Nature Protocols. 10 (4), 605-618 (2015).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16 (11), 1128-1133 (2009).
- Schwalb, B., et al. TT-seq maps the human transient transcriptome. Science. 352 (6290), 1225-1228 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены