Method Article
שמר האפייה מטבולית תיוג עם 4-thiouracil, כימות של לאחרונה מסונתז mRNA כמדד לפעילות RNA פולימראז II
In This Article
Summary
הפרוטוקול המתואר כאן מבוסס על כימות ברמת הגנום של mRNA החדש מסונתז מטוהרים מתאי שמרים עם 4-thiouracil תוויות. שיטה זו מאפשרת למדוד את סינתזת mRNA חשיפות מהשחתה mRNA ומספק, לפיכך, מדידה מדויקת של RNA פולימראז II שעתוק.
Abstract
אולי אפשר להתעלם ליקויים גלובלית-RNA פולימראז II שעתוק על ידי מחקרים transcriptomic ניתוח מצב יציב RNA. אכן, הפחתת הגלובלית סינתזת mRNA הוכח פיצוי כספי על ידי ירידה בו זמנית ב- mRNA השפלה כדי לשחזר את רמות נורמליות של מצב יציב. ומכאן, כימות ברמת הגנום של סינתזת mRNA, באופן עצמאי מהשחתה mRNA, הוא הטוב ביותר השתקפות ישירה של RNA פולימראז II פעילות גנים ברמת השעתוק. כאן, אנו נדון שיטה באמצעות שאינם perturbing מטבולית תיוג של RNAs nascent ב האפייה (cerevisiae ס). באופן ספציפי, התאים מתורבתים במשך 6 דקות עם אורציל אנלוגי, 4-thiouracil, ואת RNAs לאחרונה משועתקים שכותרתו הם מטוהרים לכמת כדי לקבוע את קצבי סינתזת mRNA בודדות כל. יתר על כן, באמצעות תווית שמר הביקוע תאים כמו תקן פנימי מאפשר השוואת סינתזה mRNA שונה cerevisiae ס זנים. באמצעות פרוטוקול זה, הולם את הנתונים באמצעות מודל קינטי דינמי, יכול להיקבע mRNA הניוון המתאימים.
Introduction
התאים מגיבים רמזים אנדוגני, אקסוגני, דרך שינוי דינמי של התוכנית ביטוי גנים. בשנים האחרונות, התפתחות עצומה של הגנום כולו מתודולוגיות מאפשר תיאור מקיף ומדויק של transcriptome שינויים בתנאים שונים. במחקרים transcriptomic רוב, רצף הכלאה או תפוקה גבוהה microarray משמשים כדי לכמת את רמות ה-RNA מ שבריר הכולל של RNA מצב יציב. שינויים תעתיק תחת ההפרעות ספציפי באפשרותך להציג מגוון רחב של תוצאות אפשריות, עם שינויים בביטוי גנים ספציפיים או מגוון גדול של גנים להיות למעלה - או downregulated. ביטוי גנים הוא התוצאה של שיווי משקל מכויל — או מצב יציב – בין סינתזת RNA על ידי RNA polymerases ותהליכים אחרים המשפיעים על רמות ה-RNA. שעתוק rna פולימראז II, כולל שלושה שלבי ברורים שלה (חניכה, התארכות, סיום), קשורה מאוד, מסובכת mRNA עיבוד ייצוא cytoplasmic, תרגום, השפלה.
מספר מחקרים שנעשו לאחרונה הראו כי mRNAs סינתזה ודעיכה הם מנגנונים בשילוב והראו כי ניתן להתעלם תעתיק אפקטים על מוטציה או תחת גירויים בעת לכימות הכולל RNA מצב יציב. ראשית, הגילוי של שינויים גנים ברמת השעתוק דרך הבדיקות של רמות מצב יציב של mRNA תמיד תלוי mRNAs half-life. פעם אחת הוא הציג את ההפרעות, רמות מצב יציב mRNAs עם זמן מחצית החיים יושפעו הרבה פחות מאלה של mRNAs עם מחצית החיים הקצר. לכן, detectability של השינויים סינתזה RNA חריפה מוטה לטובת תעתיקים קצרת ימים, בזמן הניתוח של מינים mRNA longer-lived עשוי להיכשל לחשוף שינוי דינמי בקצב שעתוק. שנית, מספר דיווחים הראו כי, גם שמרים וגם יונקים, שינויים כלליים בתעתיק עשוי להיות להתעלם בעת ניתוח רמות מצב יציב של mRNA. . זה ככל הנראה בגלל המנגנון לקשר סינתזת mRNA והשפלה וכתוצאה מכך mRNA אגירה. זה הוביל לפיתוח של פרוטוקולים חדשים לכמת סינתזת mRNA חשיפות מן השפלה, דרך הניתוח של mRNA החדש משועתקים. בשנים האחרונות, מספר חלופות הוצגו, כולל ריצה הכללית רצף (GRO-seq)1ותעתיק מקורי התארכות רצף (NET-seq)2,3. כאן, אנחנו מציגים את פרוטוקול שפותחה לראשונה ב תאים בתרבית של4,5,6 ומותאמים ואז השמרים7,8,9,10, 11, אשר מבוסס על ה-RNA תיוג עם thiolated nucleoside או אנלוגי בסיס, 4-thiouridine (4sU) או 4-thiouracil (4tU), בהתאמה.
שיטה זו במיוחד מטהרת RNA משועתקים לאחרונה את התאים ב- RNA אשר נמצאים המסומנת הדופק 4sU עם כמעט ללא התערבות הומאוסטזיס תא. לכן, ברגע התאים נחשפים 4sU, המולקולה היא במהירות uptaken, phosphorylated אל 4sU-טריפוספט, שולבו RNAs עיבד. ברגע דופק עם התווית, זה אפשרי לחלץ סך RNA הסלולר (תואם לרמות מצב יציב של RNA), והיא, לאחר מכן, השבר RNA התווית על-ידי 4sU תיול-במיוחד משתנה, שמוביל להיווצרות קשר דיסולפידי בין ביוטין ו 4,לאחרונה משועתקים רנ א5. עם זאת, 4sU יכולה להיות רק uptaken על ידי התאים לבטא נהג nucleoside, כמו המשגר האנושי nucleoside equilibrative (hENT1), מניעת השימוש המיידי בו ניצני ביקוע או שמרים. בעוד אחד יכול להביע את hENT1 ס הביקוע או cerevisiae ס, גישה קלה יכולה להיות מושגת באמצעות 4tU את הבסיס שונה, שכן תאי שמרים עשויה להימשך עד 4tU, ללא הצורך של ביטוי של טרנספורטר nucleoside10, 11 , 12 , 13. למעשה, חילוף החומרים של 4tU דורש פעילות אנזים אורציל phosphoribosyltransferase (UPRT). מספר אורגניזמים, כולל שמרים אבל לא יונקים, UPRT הוא חיוני עבור מסלול הניצולת פירימידין, מיחזור אורציל כדי uridine monophosphate.
הטיה חשוב במחקרים transcriptomic יכול להיות הציג נירמול בין מדגמים שונים ניתח במקביל. אכן, גורמים deviating רבים יכולים להשפיע על ניתוח השוואתי של transcriptome של מוטציה וללחצים פראי-סוג: היעילות של פירוק התא, הבדלים החילוץ, שחזור של RNA וסטיות בתוך הסורק כיול עבור ניתוחים microarray , בין היתר. כפי שצוין לעיל, וריאציות כזה יכול להיות מטעה במיוחד כאשר צפויים אפקטים הכללית ב- RNA פולימראז II שעתוק. אומר אלגנטי להשוות במדויק mRNA מחירים סינתזה בין מדגמים שונים תוכנן על-ידי שימוש שמרים ביקוע קרובים רחוקים שמר הביקוע תקן פנימי. בשביל זה, מספר קבוע שכותרתו ס הביקוע תאים מתווסף הדגימות cerevisiae ס , או פראי-סוג או מוטציה בתאים, לפני פירוק התא ל- RNA החילוץ10. לאחר מכן, גם מצב יציב וגם לאחרונה מסונתז RNAs ס הביקוע , cerevisiae ס הם לכמת RT-qPCR או באמצעות שימוש microarray צ'יפס או רצף תפוקה גבוהה10. שילוב נתונים אלה עם מידול קינטי, שיעורי מוחלטת של סינתזת mRNA ודעיכה של ניצני שמרים ניתן למדוד.
במסגרת של כתב יד זה, נראה כיצד הניתוח של RNA לאחרונה משועתקים מותר לחשוף תפקיד גלובאלי עבור מתחמי coactivator SAGA, TFIID בתעתיק RNA פולימראז II בניצני שמרים14,15, 16. חשוב לציין, מחקרים שנעשו בעבר לכמת את רמות ה-mRNA מצב יציב cerevisiae ס והציע זה הסאגה ממלא פונקציה השולט על קבוצה מוגבלת של גנים שמרים אשר חריפה המושפעים על ידי מוטציות בסאגה, אבל יחסית עמידים בפני TFIID מוטציות17,18,19. באופן מפתיע, פעילויות אנזימטיות הסאגה הוצגו לפעול על הגנום כולו משועתקים, רומז תפקיד רחב יותר עבור activator זה שותף ב- RNA פולימראז II שעתוק. ירידה RNA פולימראז II גיוס-גנים ביטוי ביותר נצפתה על איון של הסאגה או TFIID, רומז כי אלה coactivators לעבוד ביחד-רוב הגנים. לפיכך, כימות של mRNA משועתקים לאחרונה חשף כי הסאגה, TFIID נדרשים עבור שעתוק של גנים כמעט כל RNA פולימראז II14,15,16. מימוש מנגנוני פיצוי מתגלה דרך עבור התאים להתמודד עם ירידה גלובלית סינתזת mRNA אשר נאגרת על ידי ירידה גלובלית סימולטני mRNA השפלה. הסאגה מוסיף לרשימה של גורמים שיש השפעה גלובלית על ה-RNA פולימראז II שעתוק, כגון ה-RNA Pol II subunits10, מתחם coactivator20את המתווך, הגנרל שעתוק מקדם TFIIH21,22 , בעקיפין, יסודות ה-mRNA השפלה מכונות9,10,23. הסאגה מוטציות, והיוו השינויים צנוע ומוגבל ברמות ה-mRNA מצב יציב למרות ירידה גלובלית וחמור סינתזת mRNA14אוניברסלית זו נצפתה אירועים כאלה הבולמוס. ניתוחים דומות בוצעו גם בזן מחיקה BRE1 , וכתוצאה מכך אובדן מוחלט של היסטון ויזת עבודה H2B ubiquitination. מעניין, הרבה מתון יותר אך עקבי הכללית השפעה על ה-RNA פולימראז II תמלול יכול להתגלות, בהעדר Bre1, המציין כי תיוג מטבולית של RNA משועתקים לאחרונה ב שמרים ניתן לזהות ולכמת מגוון רחב של שינויים ב- mRNA המחירים סינתזה.
Protocol
1. תא Culturing ו Rapamycin דלדול של יחידת משנה סאגה
- לכל זן cerevisiae ס או שכפול, לרבות פראי-סוג או שליטה זנים, לחסן מושבה בודדת מצלחת טריים על 5 מ של YPD בינונית (2% Peptone, תמצית שמרים 1% ו-2% גלוקוז).
- לגדל תאים cerevisiae ס בן לילה ב 30 ° C עם עצבנות מתמדת (150 סל ד).
- מודדים את צפיפות אופטית על 600 nm (OD600), לדלל את תרבות יתר600 של 0.1 ל-100 מ"ל YPD בינוני, תני לו לצמוח עד ה OD600 הוא סביב 0.8.
- במקביל, לחסן מושבה בודדת של תאים ס הביקוע מצלחת טריים על 50 מ של כן בינוני (0.5% תמצית שמרים; 250 mg/L אדנין, היסטידין, אורציל, לאוצין, ליזין; 3% גלוקוז) לגדל את התאים בן לילה ב 32 ° C עם עצבנות מתמדת (150 סל ד).
- למדוד את יתר600 של התרבות לילה ס הביקוע , לדלל את תרבות יתר600 של 0.1 ב 500 מ של מדיום. כן, תני לו לצמוח עד ה OD600 הוא כ 0.8.
- לכל זן עוגן-משם cerevisiae ס או שכפול, לרבות פראי-סוג או שליטה זנים, לחסן מושבה בודדת מצלחת טריים על 5 מ של YPD בינונית (2% Peptone, תמצית שמרים 1% ו-2% גלוקוז).
- לגדל את התאים בן לילה ב 30 ° C עם עצבנות מתמדת (150 סל ד).
- למחרת בבוקר, למדוד את יתר600, לדלל את תרבות יתר600 של 0.1 ל-100 מ"ל של מדיום YPD ולתת לו לגדול עד יתר600≈ 0.8.
- להוסיף 100 µL של rapamycin התרבות מתוך פתרון מניות של 1 מ"ג/מ"ל (ריכוז rapamycin הסופי של µg 1/mL) ולתת את התאים דגירה ב 30 ° C עם עצבנות מתמדת במשך הזמן הדרוש עבור החלבון לעניין להיות מותנה מרוקנים מ (גרעין בדרך כלל, 30 דקות היא נאותה). עבור הפקד, להשתמש תרבות שמרים דומה, אך במקום הוספת rapamycin, להוסיף נפח המקבילה של דימתיל סולפוקסיד (דימתיל סולפוקסיד).
2. 4tU תיוג עם ס' הביקוע ספייק-in (ספירת)
-
להכין פתרון טריים של 2 מ' 4-thiouracil. ברגע מוכן, לשמור את זה בטמפרטורת החדר ולא מן האור.
- במדויק שוקל 64.1 מ ג 4-thiouracil עבור כל תרבות cerevisiae ס , להמיס 250 µL של dimethylformamide (DMF) או 250 µL של דימתיל סולפוקסיד.
- בגלל התרבות הביקוע ס לשמש ספייק-in, שוקל 320.5 מ"ג 4-thiouracil, לפזר זאת בתוך µL 1,250 של דימתיל סולפוקסיד.
- הוסף את הפתרון 4-thiouracil לתרבויות cerevisiae ס ו ס הביקוע על ריכוז הסופי של 5 מ מ, דגירה אותם במשך 6 דקות עם עצבנות מתמדת של 30 מעלות צלזיוס, 32 ° C, בהתאמה.
- לאחר 6 דקות, להסיר aliquot קטן של כל תרבות לספירת תאים. לספור את התאים באמצעות מונה הניתן תא אוטומטי או תא Neubauer.
- לאסוף את התאים באמצעות צנטריפוגה (2,500 x g) עבור 5 דקות ב 4 º C.
- למחוק את תגובת שיקוע, לשטוף את התאים עם PBS קר כקרח 1 x, צנטריפוגה שוב (x 2,500 g, 4 ° C, 5 דקות).
- לחשב את המספר הכולל של תאים בכל אחת הדגימות cerevisiae ס ו ס הביקוע .
- Resuspend התאים 5 מ של PBS קר כקרח 1 x ומערבבים cerevisiae ס עם ס' הביקוע תאים עם יחס של 3:1.
- Centrifuge התאים (2,500 x g, 4 ° C, 5 דקות) להסיר את PBS, הקפאה התאים N נוזלי2, לאחסן את הדגימה ב-80 מעלות צלזיוס עד שימוש נוסף.
3. RNA חילוץ וטיפול DNase
- להפשיר את התאים על קרח למשך כ 20-30 דקות.
- להמשיך עם מיצוי ה-RNA באמצעות ערכת חילוץ שמרים-RNA (טבלה של חומרים) עם כמה עיבודים.
- עבור כל דגימה, יוצקים µL 750 של חרוזי קריסטל קרח לתוך צינור 1.5-mL בורג מכסה מסופק עם הקיט. זכור כי לכל כל שפופרת, מתאי9 עד 10 ביעילות רנ א ניתן. לפיכך, הכן את מספר צינורות הכרחי עבור כל דגימה. לדוגמה, תרבות cerevisiae ס של 100 מ ל (OD600≈ 0.8) יכול לדקלם בסביבות 2 x 109 3 x 109 תאים, שהובילו בסך 2.7 x 109 4 x 10 תאים9 סה כ (אחרי ה ספייק-in עם שליש הביקוע ס תאים). במקרה זה, עד 3-4 צינורות התגובה לכל כל מדגם/מצב/חשבונאי/שכפול יהיה צורך.
- לכל כל עונה 1 פרק 109 תאים, להוסיף µL 480 המאגר פירוק מסופק עם הקיט, µL 48 למען חברה דמוקרטית 10% ו- 480 µL של אלכוהול פנול: כלורופורם: isoamyl (25:24:1, וי/v/v).
- לערבב את התאים באמצעות מערבל מערבולת, להעביר אותם הצינורות המכילים את החרוזים זירקונים קר כקרח.
- להכיל את הצינורות על מתאם מיקסר מערבולת, להפוך את המערבולת במהירות המרבית, היכו במשך 10 דקות lyse תאי שמרים (בחדר ב 4 ° C). לחלופין, לבצע פירוק של התאים מכה חרוז אוטומטית.
- צנטריפוגה צינורות-16,000 x g עבור 5 דקות בטמפרטורת החדר, בזהירות לאסוף את השלב העליון (שלב המכילות RNA) לרכבת התחתית בז טריים 15 מ"ל. בדרך כלל, שאמצעי האחסון התאושש למחזור כל סביב 500-600 µL.
- כדי הצינורות 15 מ"ל המכיל את הרנ א מטוהרים חלקית, להוסיף למאגר איגוד מסופק עם הקיט, לערבב ביסודיות. לפי כל µL 100 של פתרון ה-RNA, להוסיף 350 µL מחייב מאגר (כלומר, כאשר אמצעי האחסון פאזה מימית פתרון µL 600, mL 2.1 המאגר איגוד אמור להתווסף).
- לתערובת הקודמת, מוסיפים האתנול 100% ומערבבים היטב. לפי כל µL 100 של פתרון ה-RNA, להוסיף µL 235 של 100% אתנול (כלומר, כאשר אמצעי האחסון פאזה מימית פתרון 600 µL, להוסיף 1.41 מ"ל אתנול).
- החל µL עד 700 של תערובת מהשלב 3.9 כדי מחסנית מסנן התאספו בשפופרת איסוף, שניהם מסופק עם הקיט.
- צנטריפוגה עבור 1 דקות ב 16,000 x g. אם משך הזמן צנטריפוגה לא היה מספיק עבור אמצעי האחסון הכולל לעבור דרך המסנן, חזור על צנטריפוגה עבור 30 s.
- למחוק את הזרימה דרך ולהשתמש ברכבת התחתית אוסף אותו. להוסיף עוד 700 µL של פתרון מאגר-אתנול RNA מחייב המסנן ואת צנטריפוגה שוב ב 16,000 x g עבור 1 דקות.
- למחוק את הזרימה דרך ' וחזור על שלבים 3.11 ו- 3.12, עד גמר את פתרון ה-RNA.
- שטוף את המסנן 2 x עם 700 µL לרחוץ את פתרון 1. לאסוף את הכביסה פתרון באמצעות צנטריפוגה-16,000 x g עבור 1 דקות ולשמור תמיד את הצינור אוסף.
- שטוף את המסנן 2 x עם 500 µL לרחוץ את פתרון 2. לאסוף את הכביסה פתרון באמצעות צנטריפוגה-16,000 x g עבור 1 דקות ולשמור תמיד את הצינור אוסף.
- Centrifuge צינורות שוב ב x 16,000 g עבור 1 דקות להתייבש לחלוטין את המסנן.
- להעביר את מחסנית מסנן הצינור אוסף סופי (הצינור המתאים RNA), elute RNA עם 50 µL שטופלו DEPC, ללא RNase H2O (טרופה עד 100 ° C).
- צנטריפוגה עבור 1 דקות ב 16,000 x g.
- Elute רנ א שוב (כדי הצינור אותו) עם µL 50 של טרופה שטופלו DEPC, ללא RNase H2O. לוודא כי כל אמצעי האחסון עבר דרך המסנן; אחרת, צנטריפוגה לתקופות ארוכות.
- אם צינורות מרובים עבור דגימה בודדת אחת נעשה שימוש, בריכת אותם בכל שפופרת אחת.
- לכמת וסמנו את הטוהר של המדגם שימוש בציוד המתאים.
הערה: בזמן 4tU מעוגנת רק בתוך RNA לאחרונה מסונתז, יש סיכוי לזיהום קטן עם ה-DNA. מסיבה זו, זה תמיד רצוי להתייחס הדגימות עם DNase אני. לשימוש זה, ריאגנטים מסופק עם הקיט רנ א-הפקת (טבלה של חומרים) בעקבות ההמלצות של היצרן.
4. תיול ספציפי Biotinylation של RNA מסונתז לאחרונה
- התאם את ריכוז RNA שהושג עם סעיף 3 פרוטוקול 2 מ"ג/מ"ל. Aliquot µg 200 של RNA הכולל, אחמם את זה 10 דקות ב 60 מעלות צלזיוס, מיד תרגעו על קרח למשך 2 דקות.
-
כדי aliquot ה-RNA, הוסף את ריאגנטים המוזכרים להלן לפי הסדר הבא: 600 µL של שטופלו DEPC, ללא RNase H2O, 100 µL biotinylation מאגר (100 מ מ טריס-HCl [pH 7.5] ו 10 מ מ EDTA, שטופלו DEPC, נטולת RNase H2O), 200 µL של ביוטין-HPDP מתוך מלאי של 1 מ"ג/מ"ל ביוטין-HPDP דימתיל סולפוקסיד או DMF.
- במצבים מסוימים, הפתרון ביוטין-HPDP נוטה לזרז, ככל הנראה עקב שלה מסיסות נמוכה במים. במצב זה, להגביר את עוצמת דימתיל סולפוקסיד/DMF עד 40% של אמצעי האחסון התגובה (לדגימת RNA, להוסיף 400 µL שטופלו DEPC H2O, µL 100 biotinylation מאגר ולאחר µL 400 של ביוטין-HPDP מתוך מלאי 0.5 מ"ג/מ"ל).
- דגירה המדגם בטמפרטורת החדר, מוגן מפני אור במשך 3 שעות, עם עצבנות עדין.
- לאחר דגירה, להוסיף אמצעי שווים בקירוב של כלורופורם הצינורות, מערבבים היטב.
- ספין המדגם ב x 13,000 g למשך 5 דקות, ב 4 º C. שלב זה מאפשר את הסרת עודפי ביוטין שעשה לא biotinylate הרנ א. לחלופין, לבצע שלב זה באמצעות צינורות שלב נעילה (כבד). בשביל זה, ספין לטמיון שלב נעילה עבור 1 דקות ב 13,000 x g, להוסיף את התערובת RNA וכמות זהה של סם הרדמה, מערבבים אותם היטב, ו centrifuge אותם ב 13,000 x g למשך 5 דקות, ב 4 º C.
- להעביר בזהירות השלב העליון לתוך צינורות 2 mL החדש.
- מוסיפים עשירית נפח של 5 M NaCl ומערבבים את הדגימה.
- הוסף אמצעי שווה של אלכוהול איזופרופיל, לערבב את הדגימה ביסודיות, ולסובב את זה ב 13,000 x g למשך 30 דקות לפחות, ב 4 º C.
- בזהירות להסיר את תגובת שיקוע והוסף 1 מ"ל אתנול 75% קר כקרח.
- ספין-x 13,000 g למשך 10 דקות, ב 4 º C.
- בזהירות להסיר את תגובת שיקוע, מהיר-ספין הצינור, ולהסיר את הפתרון אתנול הנותרים. ודא כי בגדר RNA לא יבשה.
- להשעות את הרנ א ב 100 µL שטופלו DEPC, ללא RNase H2O.
5. טיהור של שבר מסונתז לאחרונה מ- RNA הכולל, ללא תווית באמצעות Beads מגנטי מצופה Streptavidin
- מחממים את biotinylated RNA 10 דקות ב 65 ° C ולהירגע ואז את הדוגמאות על קרח למשך 5 דקות.
- להוסיף 100 µL של beads מגנטי מצופה streptavidin biotinylated RNA (בווליום הסופי של 200 µL). באופן ספציפי, מומלץ להשתמש את החרוזים המצוין בטבלה של חומרים, מאז, לאחר שיחות עם מעבדות אחרות, אלה נראה תהיה יותר עקבית ומהימנה.
- דגירה הדגימה עם מהרעידות במשך 90 דקות, בטמפרטורת החדר.
- מקם את העמודות מסופק עם הקיט (טבלה של חומרים) ביציע מגנטי.
- הוסף 900 µL מאגר כביסה בטמפרטורת החדר (100 מ מ טריס-HCl [pH 7.5], 10 מ מ EDTA, 1 M NaCl, ו 0.1% Tween 20, שטופלו DEPC, נטולת RNase H2O) לעמודות (להפעיל מראש ו- equilibrate).
- חרוזים/RNA תערובת (200 µL) חלות על העמודות.
- לאסוף את הזרימה דרך צינורות 1.5-mL ולהחיל אותו שוב כדי באותה עמודה מגנטי. במידת הצורך, לשמור זה הזרימה דרך כמו זה מייצג את השבר RNA ללא תווית.
- לשטוף את העמודות 5 x עם הגדלת נפחי שטיפה מאגר (600, 700, 800, 900 ו 1,000 µL).
- Elute את הרנ א מסונתז לאחרונה עם 200 µL של 0.1 M DTT.
- ביצוע של שני • תנאי, 3 דקות מאוחר יותר, עם אמצעי אחסון שווה של 0.1 M DTT.
- לאחר eluting את ה-RNA, להוסיף 0.1 כרכים של 3 מ' NaOAc (pH 5.2), שלושה כרכים של אתנול 100% קר כקרח, 2 µL של 20 מ"ג/מ"ל גליקוגן (RNA-כיתה) ואפשר הלילה precipitate RNA, ב-20 ° C.
- לשחזר את הרנ א על ידי צנטריפוגה (13,000 x g למשך 10 דקות, ב 4 ° C), resuspend אותו ב- µL 15 שטופלו DEPC, ללא RNase H20. חלקם של RNA הנקרא-כדי-סיכום צפוי להיות בסביבות 2% - 4% (בדרך כלל יותר לכיוון הקצה התחתון), עיבוד כ 2.0 µg של RNA לאחרונה מסונתז. כמות זו מספיקה כדי לעשות מספר ניסויים qPCR, כמו גם עבור ניתוחים microarray/רצף.
6. RT-qPCR אימות של שברים שונים
- לסנתז cDNA µg 2 של RNA הכולל או 10 µL של RNA שכותרתו באמצעות hexamers אקראית של רוורס טרנסקריפטאז של בחירה, לפי הוראות היצרן (טבלה של חומרים).
- להגביר את cDNA מאת qPCR בזמן אמת באמצעות פרוטוקול תקשורת (טבלה של חומרים).
הערה: הדוגמאות צריכה לפעול שהפקידים לפחות שני ביולוגי משכפל לתקן כל הערכים גולמיים עבור הביטוי של ס' הביקוע טובולין.
7. Microarray הכלאה
- Hybridize דגימות ה-RNA על גבי שבבי microarray מועדף על פי הוראות היצרן (עבור פרוטוקול ספציפי זה, ראה טבלה של חומרים). להכין בקצרה, biotinylated מטרות קרנה מ 150 ng של RNA באמצעות Premier RNA הגברה הערכה (טבלה של חומרים), על פי הוראות היצרן. Hybridize מ ג של cRNAs מפוצלים עבור 16 h ב 45 ° C ו- 60 סל ד על שבבי microarray.
- לשטוף את כתם, לסרוק את שבבי באמצעות תחנת המצוין סורק (טבלה של חומרים). לחלץ את הנתונים הגולמיים (קבצי. צ'ל בעוצמה) מתמונות סרוקות באמצעות מסוף הפקודה (AGCC, גירסה 4.1.2).
- בהמשך לעבד את הקבצים צ'ל עם גרסת תוכנה מסוף בביטוי כדי לחשב בדיקה להגדיר אות בעוצמות, באמצעות האלגוריתמים המבוסס על סטטיסטיקה MAS 5.0 עם הגדרות ברירת המחדל ואת גלובלי קנה מידה שיטה לנרמול 1.4.1.
הערה: האינטנסיביות החתוך היעד הממוצע של כל שבב באופן שרירותי נקבע ל- 100. לבצע כל ניסויים באמצעות לפחות שני עצמאית ביולוגי משכפל. לנרמל את הנתונים הגולמיים לאות ס' הביקוע ולחשב הקיפול שינויים ברמות ה-RNA מוחלטת ובלתי לאחרונה מסונתז.
8. נתוני ניתוח באמצעות צינור R הקיים
- לחשב סינתזה, ריקבון המחירים באמצעות צינור R/Bioconductor החבילה זמין לציבור, כפי שתואר לעיל8,10.
תוצאות
בעת ביצוע תיוג מטבולית של RNA לאחרונה משועתקים, מספר היבטים צריך להיות תחת פיקוח: הזמן ואת היעילות של תיוג שיעור בספייק, פרוטוקול החילוץ, היעילות biotinylation (כולל יחס אות לרעש), בין היתר. תנאים אלה בהרחבה ובאופן שיטתי הוכחו על ידי אחרים7,10,11. כאן אנו מתמקדים בעיקר פרשנות וניתוחים מיידית שיכולים להתבצע פעם אחת הדגימות עובדו, או על ידי RT-qPCR, microarray או רצף. הניתוחים של זנים מוטנטים שונים להדגים את הכוח של השיטה כדי לזהות לא רק ירידה גלובלית דרמטי ב- mRNA סינתזה, כמו במקרה של מוטציה הסאגה cerevisiae ס זנים, אלא גם ירידה מתונה מאוד של RNA פולימראז II פעילות על דיכוי של היסטון ויזת עבודה H2B monoubiquitination.
שלנו ניתוחים של פעילויות אנזימטיות הסאגה הציע גיוס רחב כרומטין15, אשר לא נתגלה על ידי הניתוח של מצב יציב mRNA רמות זנים מוטציה הסאגה. כמו ה-RNA פולימראז II הגיוס היה לקוי על הסאגה איון, החלטנו לבדוק אם המחירים סינתזת mRNA יושפעו באופן גלובלי. לפיכך, פראי-סוג או מוטציה זנים cerevisiae ס נחשפו 4tU לתקופה 6 דקות, לתייג RNAs לאחרונה משועתקים. לאחר ערבוב עם ספייק-ב שכותרתו בתאים (ס' הביקוע) שיעור של 3:1, הכולל RNA שהופק, RNA מסונתז לאחרונה היה biotinylated וטיהרו לפי הפרוטוקול המובאים כאן, כמו chronogram המוצג באיור1. RNAs שכותרתו היו טהור מתוך סכום כולל של 200 µg של RNA, המבטיח כי כמות המוצר מטוהרים יהיה מספיק עבור כל יישום במורד הזרם. כשלב ראשוני ושיטתית לפני כל ניתוח הגנום כולו, טיהור RNA לאחרונה מסונתז שהוכח על ידי נסיונות RT-qPCR. גנים נבחרו על פי פרמטרים שונים, כולל רמה של הביטוי, מסלולים הרגולציה של התלות polymerases RNA שונים.
כדי לאשר פרוטוקול זה מטהר במיוחד RNA שכותרתו, אנחנו לכמת את הרמות של תעתיקים בחלקים מטוהרים פראי-סוג התאים היו תרבותי עם או בלי 4tU. רמות הרקע זניח RNAs שנותחה אותרו התאים לא נחשפו 4tU (איור 2א). לאחרונה משועתקים RNA טיהור נוסף שהוכח על ידי נסיונות העשרת הנצפה של המכילים אינטרון ACT1 pre-mRNA (נתונים לא מוצג). לאחר אימות של האיכות של הדגימות, בדקנו סינתזת mRNA יושפעו על מחיקה של SPT20, אשר ידוע לשבש את מכלול מורכב של הסאגה. כפי שדווח על ידי אחרים, כימות mRNA שבוצעה ב- RNA (מצב יציב) הכולל מן המתח Δ spt20חשף רמות ללא שינוי או מופחתת במתינות ברובו על הגנים שנבדקו (איור 2B). תוצאות דומות התקבלו עבור זנים שנמחקו עבור BRE1 (איור 2B). לעומת זאת, הניתוח של RNA משועתקים לאחרונה בגלל הלחץ Δ spt20חשף ירידה דרמטית סינתזת mRNA ב-3-נבדק עד למטר עבור כל הגנים (איור 2C). האובדן של Bre1 הוביל לירידה יותר דיסקרטי אך עדיין גלויה ברמות ה-mRNA החדש מסונתז על הגנים למדה (איור 2C). הסכם טוב עם תפקיד של הסאגה ו Bre1 ב RNA פולימראז II שעתוק, אובדן או Spt20 או Bre1 לא השפיע על הביטוי של גנים RDN25 או snR6 , עיבד על ידי RNA פולימראז ו- RNA פולימראז השלישי, בהתאמה ( איור 2C).
עם זאת, זנים למחוק subunits מבנית של הסאגה מורכבים, כמו SPT7 או SPT20, להציג פנוטיפים חמורים הגידול אשר עשוי להסביר השינוי תעתיק שנצפו. כדי לשלול תופעות לוואי משנית, שאנחנו מותנית דלה Spt7 מהגרעין, באמצעות עוגן-משם ס' cerevisiae זנים14,24. על 60 דקות של טיפול rapamycin, לפני הדופק-תיוג עם 4tU (ראה איור 1 . ייצוג סכמטי), רמות ה-mRNA החדש משועתקים הצטמצם במידה דומה עד כדי כך שנצפתה המתח מחיקה (איור 2D and 2E ). ניתוח זה, לפיכך, אישר את תוצאות לשעבר שלנו ואומת הפרוטוקול עבור מערכת זו דלדול inducible. ב ניתוח מסלול הזמן, שבו תאים נחשפו rapamycin עבור הזמן פורש מ-0 מינימום 240, ביטוי מופחתת היה עדות מיד לאחר 15 דקות של חשיפה הסם. יותר מעניין, מצב יציב mRNA רמות נטו בתחילה לצמצם אך חזר לרמות נורמליות לאחר 60 דקות, אינדיקציה כי מנגנון פיצוי מתרחש בינתיים (איור 2E).
בסך הכל, תיוג, כימות של RNA לאחרונה מסונתז מותר חשיפת תפקידים חדשים רגולטוריות עבור המתחם הסאגה. הפרוטוקול המתואר גם יכול לחשוף תופעות מתון על ה-RNA פולימראז II פעילות והיה דלדול בהצלחה שימושית כדי מותנה זני שמרים.
אחד היישומים במורד הזרם עבור הרנ א לאחרונה מסונתז מזוכך הוא כימות ברמת הגנום של תעתיקים באמצעות microarray הכלאה או רצף (4tU-seq). בעוד רצף תפוקה גבוהה יותר כמותיים, רגיש, אינפורמטיבי, הכלאה microarray עשויים להיות מועילים במיוחד לקבוע אם הם שינו את רמות ה-mRNA גלובלית. בהקשר זה איפה מרכזי נורמליזציה, הוספנו אורגניזם ספייק-in המדגם שאנחנו שמטרתו לנתח. באופן ספציפי, אנחנו מעורבים cerevisiae ס תאים לתאים ס הביקוע על יחס של 3:1, שניהם בעבר נחשפים 4tU. כאשר RNAs מטוהרים, שכותרתו הם נתון רצף תפוקה גבוהה, בכל פרוטוקול הכנה ספרייה תקנית יכול לשמש, לרוב בעקבות המחסור ribosomal RNA. נורמליזציה בין נתונים 4tU-seq מדגימות שונות בוצעה על-ידי הוספת גם הנקרא RNA מינים שונים (קרי, ערבוב ס' cerevisiae תאים וס' הביקוע כמו לעיל)22 או חוץ גופית- עיבד, thiolated ספייק ב- RNA12,25,26,27. צ'יפס microarray זמינים מסחרית מכילים זונדי כל transcriptome של שמרים ניצני והן ביקוע, המאפשר כימות של mRNA של שני אורגניזמים בניסוי בודד אחד. Microarray hybridizations בוצעו עם ה-RNA מוחלטת ובלתי לאחרונה מסונתז אומת על-ידי RT-qPCR כמצוין לעיל (פראי-סוג, spt20Δ וδ bre1). בנוסף, אנו כלל מאמץ זה לא תומך monoubiquitination של היסטון ויזת עבודה H2B, דרך נקודת מוטציה של השאריות ubiquitinable (K123R). כי פרופורציה המדויק של תאים ס' הביקוע שימשו דוגמאות שונות, משכפל, זה אפשרי באופן ליניארי לשינוי של מערכי של עוצמות כך הכולל ומתויגים ס הביקוע רגשים יש באותה אינטנסיביות החציוני. זו נורמליזציה, או לשנות קנה מידה, ניתן לבצע באמצעות חבילת Bioconductor שפותחה על ידי המעבדה של פטריק קרמר להשתמש במקביל כל הדגימות שנותחה, הכולל ויש שכותרתו זנים שברים, פראי-סוג, מוטציות8. בקצרה, הקלט של צינור זה הוא קובץ של Excel המכיל את הגששים והעוצמה שלהם (MAS5 או RMA) עבור כל דגימות, שברים. לאחר למעט הגששים הנמצאים מחוץ לטווח של זיהוי, עוצמת אות ישתנה, לוקח בחשבון את ערכי העוצמה של הגששים ס הביקוע . לבסוף, באפשרותך למפות את הגששים ניצני, ביקוע שמרים הגנום, המסתיימים מטריצה המכילה את הערכים בביטוי מנורמל את ספייק-אין. ערכים אלה יכולים להיות עוד יותר מעובד (ראה סעיף הבא) או בשימוש כמו. בדוגמה הבאה, קבענו שלקפל את שינוי כל תעתיק בין המתח פראי-סוג המוטציה.
הבדיקות נערכו שני מצב יציב או לאחרונה מסונתז רמות של RNA, לעומת שלהם מובהקות סטטיסטית (p-ערך) (איור 3). מסכים עם מחקרים אחרים, כאשר רמות ה-RNA סה כ נותחו, רק כמה גנים היה הביטוי שלהם שונה, או להמציא - או downregulated (איור 3 אג-3). עם זאת, הניתוח של RNA לאחרונה משועתקים הובילה למסקנות שונה. רמות ה-mRNA משועתקים החדש של יותר מ-4,000 גנים הצטמצמו באופן משמעותי על ידי לפחות כפולה בעת מחיקה של SPT20, המציעה את הכללית השפעה חיובית של הסאגה על ה-RNA פולימראז II שעתוק של ניצני שמרים (איור 3D ). בנוסף, bre1Δ, מוטציות K123R , התוצאות היו קצת יותר דיסקרטיים: רוב הגנים הופיע יש לביטוי מופחת, אך מידת downregulation ומספר הגנים מושפעת באופן משמעותי (≈ 300-500) היה אכן יותר מוגבלת (איור 3E , 3F).
כפי שהוזכרו, מצב יציב או בעבר הכולל רמות של mRNA מוכתבים על ידי האיזון הדוק בין סינתזה ודעיכה (איור 4א). כאשר RNA פולימראז II שעתוק פגום באופן גלובלי, יכול להיות בתמונה שני תרחישים: (i) או mRNA הכולל רמות להקטין ברחבי העולם, בתגובה סינתזה מופחתת אבל דעיכה קבוע, או השפלה (ii) mRNA הוא ירד באותה מידה, וכתוצאה מכך ברמות ברובו ללא שינוי מצב יציב mRNA. התרחיש השני דווח על מספר תנאים, לרבות בהקשר של הסאגה או TFIID הפרעה9,10,14,16,22. הליך שנקרא ניתוח השוואתי transcriptome דינמי (cDTA) המבוסס על תיוג 4tU, מידול קינטית דינאמית ומאפשרת לנו לקבוע סינתזת mRNA, להסיק הניוון כל תעתיק8,10. שוב, אנחנו ניצל הנתונים שנאספו עבור זנים כאמור (פראי-סוג, Δ spt20, bre1Δ ו- K123R). כצפוי, עם מחיקה של SPT20, הבחנו בירידה mRNA בו זמנית המחירים סינתזה והשפלה בהשוואה המתח פראי-סוג. זן זה מוטציה, הפיצוי היה כמעט אופטימלי, עם ממוצע הדירי סינתזה של 3.8-fold, ממוצע הדירי דעיכה של 4.1-fold (איור 4B), בעדות למה שינויים תעתיק מוגבלים יכול להיות זוהה על mRNA הכולל רמות (איור 3א). ב שתי המוטציות זנים אחרים (Δbre1ו- K123R), במקביל לשינויים סינתזת mRNA ודעיכה נצפו גם, אבל השינויים היו בקנה מידה קטן יותר, יותר התפזרו (איור 4C וד' 4).
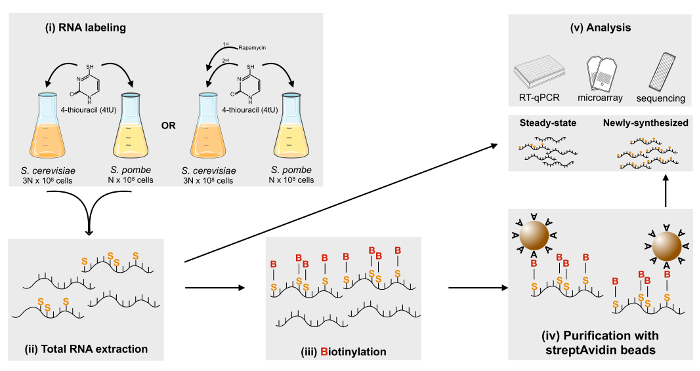
איור 1 : ייצוג סכמטי של תיוג מטבולית של RNA באמצעות 4tU. 4tU הטרי מתווסף המדיום התרבות תאים מסומנים עבור 6 מינימלית Labeled cerevisiae ס ו ס הביקוע התאים מעורבבים על יחס של 3:1 ו RNA הכולל מופק. לאחר מכן, שזה עתה מסונתז RNA biotinylated, אפשר לטהר באמצעות מצופים streptavidin beads מגנטי. לבסוף, סה כ (מצב יציב) RNA, שכותרתו החדש מסונתז RNA ניתן להשתמש במגוון רחב של יישומים במורד הזרם, כולל RT-qPCR ו- microarray הכלאה או רצף. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
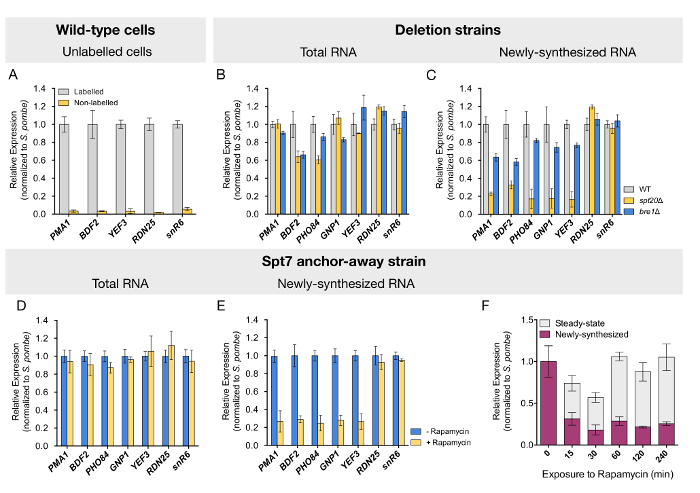
איור 2 : ניתוח המתארים שינויים תעתיק שני מצב יציב, שזה עתה עיבד RNA נקבע על ידי RT-qPCR. (א) RNA רמות של חמישה גנים שונים היו לכמת מן השבר RNA שכותרתו מטוהרים פראי-סוג התאים נחשפו גם או לא 4tU. הלוחות שני הבא להראות (B) הכולל, (ג) החדש מסונתז RNA כמת על ידי RT-qPCR פראי-סוג (WT), Δ spt20של תאי שמרים Δ bre1. Spt7 עוגן-משם זנים, לא מטופל או שטופלו rapamycin במשך 60 דקות, עם תווית עם 4tU, סה כ (ד) או (E) החדש עיבד RNA היה לכמת מאת RT-qPCR. (F) לוח זה מציג שניתוח זמן-קורס של שינויי מצב יציב, שזה עתה מסונתז mRNA על דלדול גרעיני Spt7. עבור כל הדגימות, רמות ה-mRNA עבור חמישה רנ א פולימראז II הגנים היו לכמת מאת RT-qPCR. Rna פולימראז ו- RNA פולימראז השלישי גנים (RDN25 ו- snR6, בהתאמה) שימשו השליטה. ביטוי ערכים (אומר ± SD של 3 ניסויים עצמאית) היו מנורמל ל עלה ס' הביקוע אות ו מוגדר כ- 1 במדגם שליטה. לוחות D-F שונו מ בטיסטה. et al. 14. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
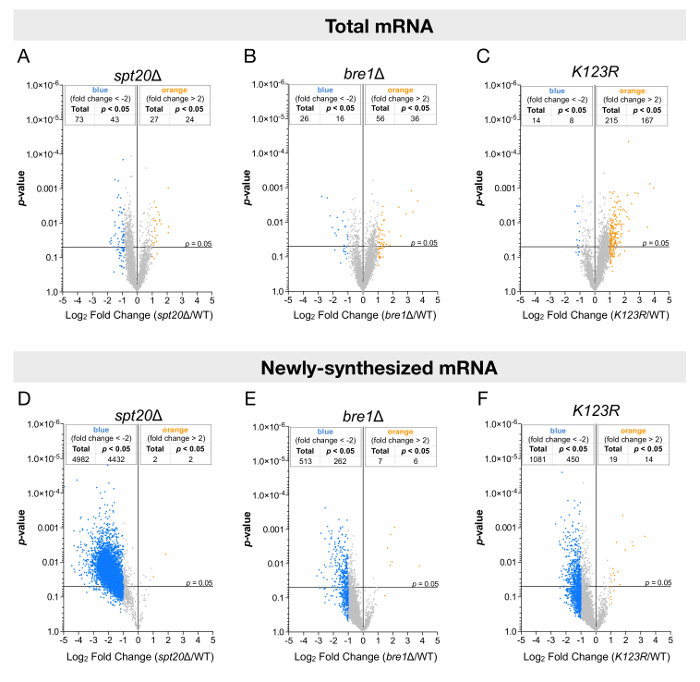
איור 3 : ניתוח ברמת הגנום של רמות ה-mRNA באמצעות שברים RNA מוחלט או שכותרתו. אלה מראות הר הגעש מתווה בסה כ מציג הקיפול שינויים ברמות ה-mRNA מצב יציב (A-C) או (D-F) לאחרונה מסונתז mRNA רמות יחסית את משמעותם (p-ערך). השינויים הקיפול (FC) חושבו ככל log2 היחס בין ערך הביטוי של כל גן לאחר נורמליזציה לאות ס' הביקוע בδ spt20(A ו- D), Δ bre1(B ו- E), או ( C ו- F) K123R זן לעומת הערך ביטוי של אותו גן בפראי-סוג cerevisiae ס. סך של גנים 5,385 נותחו ושנה סף של כפולה (נקודה כחולה: יותר כפולה ירידה; צהוב נקודות: יותר מאשר עלייה כפולה) ו- 0.05 p-ערכים נחשבו. לוחות A ו- D שונו מ בטיסטה. et al. 14. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
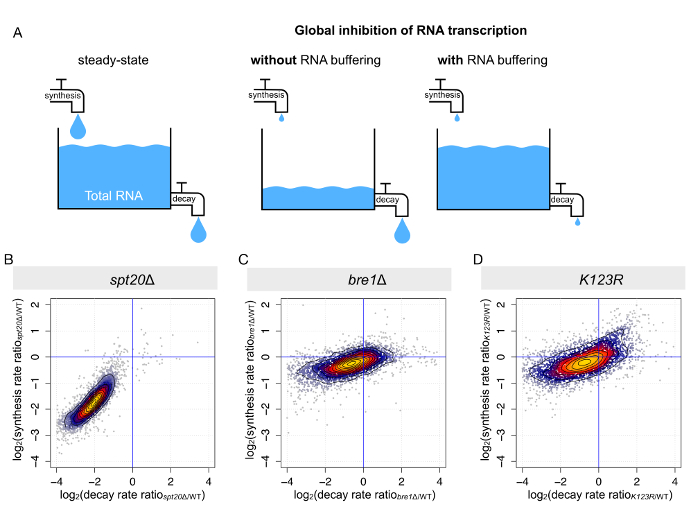
איור 4 : במקביל לשינויים סינתזת mRNA ותוצאות דעיכה mRNA ב mRNA אגירה. (א) לוח זה מציג ייצוג סכמטי של התוצאה של ההפרעות סינתזה RNA על רמות ה-RNA מצב יציב. (B-D) לוחות אלה להראות את החישוב של סינתזת mRNA ואת הניוון מן הניתוחים של RNA מוחלטת ובלתי לאחרונה מסונתז. התעריפים סינתזה ודעיכה היו נחושים עבור כל תעתיק cerevisiae ס ב Δ spt20(B), (ג) bre1Δ ו- K123R (D). שינויים (מחושבת log2 היחס בין מוטציה פראי-סוג) בשיעורי סינתזה שורטטו נגד שינויים הניוון. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
בעוד הגנום כולו כלים לנתח את השינויים בתעתיק עדיין מוכיחה, ניתוח הבלעדית של transcriptome באמצעות כימות של רמות מצב יציב של RNA אולי אינן משקפות במדויק את שינויים בפעילות השנייה של RNA פולימראז. אכן, רמות ה-mRNA מוסדרים לא רק על ידי סינתזה RNA, אלא גם על ידי שלהם התבגרות והשפלה. כדי למדוד את סינתזת mRNA חשיפות מן ה-mRNA השפלה, הוכנו נהלים ברורים בשנים האחרונות לניתוח של שעתוק nascent שמרים והן יונקים.
הוא אחד מהפרוטוקולים הנפוצה ביותר עבור כימות של שעתוק nascent GRO-seq1. היתרון העיקרי של GRO-seq הוא יכולתה לחשוף פולימראז transcriptionally מעורבת ופעילה ברזולוציה גבוהה ועל רקע נמוך, יש לו שתי נקודות נפרדות להקטין את האטרקטיביות שלה: (i) זה הוא טכנית מאתגר ודורש בידוד של גרעינים ומניפולציות, (ii) גרעינים להציג כמה לפליטת המערכת28. אלטרנטיבה אחרת היא נטו-seq, הנשענת על הרצף של 3' קצות תעתיקים שהושג על immunoprecipitation של המתחם ternary מאד יציב בין nascent RNA, תבנית ה-DNA ו- RNA פולימראז II. גישה זו מאפשרת אפיון nascent RNAs ברזולוציה זוג בסיסים, יכולים לחקור אירועים עיבוד mRNA דרך immunoprecipitation של ששונה RNA פולימראז II (זרחון סרין-5, למשל)2,3 , 29. בכל זאת, הוא מציג כמה מכשולים, שממנו אנחנו להדגיש שלושה. ראשית, בעוד נוגדנים מיקוד RNA פולימראז II הם בדרך כלל ספציפי מאוד, זה תלוי נוגדן יעילות. שנית, RNA יכול להיות נוטה השפלה בתקופות דגירה, ובכך מוביל אותנו חזרה לנקודה הקודמת (נוגדנים פחות יעיל עשויים לדרוש דגירה ארוך יותר פעמים, עוזב את הרנ א רגישים יותר השפלה)29. שלישית, במיוחד בתוך יונקים, MNase עיכול ובחירת גודל עשוי לכלול רצף ייחודי יישור29,30.
כאן אנחנו הציגה פרוטוקול מפורט עבור תיוג מטבולית של RNA מסונתז לאחרונה באמצעות 4tU ב cerevisiae ס כי יש יתרונות שונים לעומת גישות אחרות זמינים. מכיוון תמלול היא רגישה מאוד לפליטת, התאים צריכים להישמר בתנאים פיזיולוגיים ביותר. למשל, GRO-seq מרמז על מעצרו של transcriptionally עוסקת RNA פולימראז II בחשיפה של התאים/הגרעינים sarkosyl. עם זאת, הטיפול sarkosyl מתוארת כ מעכבות תהליכים תאיים מספר11. במקרה זה, תיוג מטבולית עם 4tU או 4sU-שמתואר הריכוז שאינם perturbing, בעליל לא משפיעה על הומאוסטזיס של התא, בעיקר לתקופות קצרות של חשיפה. בניגוד לשיטות אחרות באמצעות עיכוב שעתוק כדי למדוד mRNA מחצית החיים, cDTA או 4tU-seq, התאמה עם מודלים דינמיים קובע שיעורי השפלה של כל יחיד mRNA בתאים בשלוות נפש. לפיכך, שיטה יחידה אחת יכולה בו זמנית כתובת הסינתזה והן הניוון של transcriptome כל על סוג תא מסוים. מאז cDTA או 4tU-seq לוקח את היתרונות של שיטות נורמליזציה ואמינים, כלומר דרך הקישורים של ספייק-in, נתונים (datasets) שונות יכול להיות ניתח ביחד, והשוו ישירות. לבסוף, תיוג מטבולית, כימות של RNA מסונתז לאחרונה היא טכניקה אשר ניתן ליישם בקלות במעבדה בביולוגיה מולקולרית כלשהו כפי שהוא אינו מצריך כל ציוד ספציפי. זה כנראה מסביר הפצת די גדול של טכניקה זו, אשר נוטה לשמש באופן שיטתי יותר ויותר לחקור ייצור RNA והשפלה שמרים, כמו גם פרוקריוטים גבוה יותר.
RNA יכול להיקרא בתאים חיים באמצעות nucleoside תחליפי, כלומר 5-bromouridine (אחי)31,32 או 5-ethynyluridine (EU)33,34אחרים. בידודו של ברו הדופק-הנקרא RNA מסתמך על טיהור נוגדן anti-BrdU אשר ייתכן יעילות שונות בין ניסויים. התווית על-ידי האיחוד האירופי RNA יכול להיות מצומדת covalently ל ביוטין באמצעות לחץ על כימיה שמוביל ההטיה בלתי-הפיך לעומת השינוי תיול, אשר יכול להיות הפוך עם צמצום סוכנים. ברו ולקרוא האיחוד האירופי נעשה שימוש בתרבית של תאים כדי לברר הגנום כולו RNA דעיכה המחירים31,32 או להעריך nascent RNA סינתזה34,35. עם זאת, ברו או האיחוד האירופי תיוג לא תואר ב cerevisiae ס. אכן, ספיגת של תחליפי nucleoside דורש את הביטוי של nucleoside טרנספורטר ו, לכן, שיטות אלה מופיעים וגמיש פחות בניצני שמרים תיוג עם 4tU.
משך הזמן של תיוג 4tU ניתן להתאים ובסוללה ומשתנה לפי השאלה. בעת הערכת mRNA סינתזה cerevisiae ס, דופק תיוג קצרה של 6 דקות מבטיחה רמות ה-mRNA החדש משועתקים מושפעים מינימלית RNA השפלה. בהתחשב בעובדה זמן ההשהיה לפני נוקלאוטיד ששונה ניתן לשלב את הרנ א nascent היא פחות מ- 1 דקות, משך תיוג זה 6-מין צווי כמות סבירה של RNA לאחרונה מסונתז יכול להיטהר. פרוטוקול כזה, כפי שהוגדרו על ידי מילר. et al. 11, שימש ב שונים cerevisiae ס המוטציות לחשוף שינויים כלליים סינתזת mRNA ו RNA ריקבון9,10,22,26,27, 36. משך זמן תיוג (3h) שימש במרדף מטבולית הדופק-תיוג עם 4tU כדי לקבוע את הניוון של כל mRNAs cerevisiae ס12. לבסוף, קצרה ביותר (מ- 1.5 דקות) 4tU תיוג שימש לבחון קינטיקה עיבוד RNA ניצני, ביקוע שמרים13,25,37.
ובכל זאת, לשיטה זו יש מגבלות מסוימות, כגון התשואה נמוכה יחסית של RNA שכותרתו התאושש בסוף הפרוטוקול כל. בעוד שמרים, יש ללא הגבלה, חומר המוצא, זה יכול להוות בעיה בעת ניתוח תאים metazoan, במיוחד אם הוא פרוטוקול זה להיות מנוצל בתוך vivo. אחד הצעדים המגביל של הפרוטוקול הוא biotinylation של מאגר ה-RNA שכותרתו, רחוקה מלהיות מלאה ויעילות אשר הוערך לשינוי בשלושה שאריות 4sU RNA שכותרתו5. זה לאחרונה הראה כי השימוש methanethiosulfonate (MTS)-ביוטין, במקום ביוטין-HPDP, מגדילה את היבול של RNA התאושש38. עם זאת, על פי תוצאות שלא פורסמו מקבוצת המחקר הזה נובע Rutkowski, Dölken, MTS-ביוטין אינה לחלוטין תיול ספציפיים, הם מובילים הטיהור של RNA ללא תווית, אשר הוא בעייתי במיוחד כאשר נמוך כמויות של RNA שכותרתו מטוהרים39. הגבלה נוספת של תיוג מטבולית באמצעות 4tU/4sU הוא מהירות הגלום ב אילו RNA פולימראז לתרמיל. המהירות הממוצעת של RNA פולימראז II היה מוערך כ 3.5 kb/דקה40,41,42. לפיכך, תיוג 6 דקות, פולימראז תהיה את היכולת לתמלל 15 – 20 kb. לכן, בניסוי עם קצר הדופק-תיוג עם 4tU/4sU, רק 3' סוף חלק RNA לאחרונה משועתקים תוצמד, בעוד 5' סוף האזורים היו קיימים לפני התוספת של 4tU/4sU. לפיכך, הטיהור של RNAs שכותרתו מעשיר גם קיימים 5' קצוות תעתיקים, במיוחד עבור תעתיקים ארוכים כמו בתרבית של תאים. כדי להתגבר על הנטיה הזו, ב פרוטוקול מעודן שנקרא TT-seq, חילוץ הנקרא RNA פיצול קבצים על-ידי sonication לפני הטיהור של מינים לאחרונה מסונתז43.
בסך הכל, 4tU-seq היא דרך מצוינת לשינויים תעתיק כתובת בהקשר נתון. למרות זאת, וזה בעוד הפרוטוקול הוא פשוט למדי, כולל מספר שלבים שונים, להיות בסופו של דבר יחסית נרחב. בראש ובראשונה, מאז פרוטוקול זה מטפל RNA מתחילתו ועד סופו, זה חובה כי הכרחי עבור מדגם נקי וללא השפלה כל הדרישות ימולאו. לפיכך, ריאגנטים כל צריך להיות נטול RNase, כל החומרים צריכים להיות מוקדש מניפולציה RNA, ניקוי הכל ביסודיות באופן קבוע עם פתרון טיהור RNase. שנית, בעוד התגובה ביוטין הוא ספציפי מאוד, עודף של ביוטין-HPDP זה לא הגיבו להסירם מן המדגם (כדי למנוע את הרוויה של חרוזים streptavidin עם ביוטין המאוגד לא covalently RNA, לדוגמה). שלישית, כפי שמתואר הפרוטוקול, השימוש של חרוזים המצוין מגנטי מצופה streptavidin מומלץ בחום, מאז מחקר כמה קבוצות יש לנו דיבר כל ציינו כי אלה חרוזים הוביל הרקע לרמות נמוכות יותר. הרביעית, המתאימים ואיכותי ופקדים נסיוני אמור לשמש. כולל דוגמאות שמקורם תאים לא נחשפו 4tU/4sU. הדוגמיות הללו יהיה בעל ערך רב לרמות רקע כתובת, כדי לוודא זאת, במהלך הניסוי, לא יתרחש זיהום עם לא התווית על-ידי ה-RNA. בנוסף, חלופה מצוינת כדי לבדוק אם המדגם הוא מועשר עבור RNA מסונתז החדש הוא לבצע של RT-qPCR באמצעות primers נגד ג'ין המכיל הקדמה. צבעי בסיס צריך להיות משלימים קצה 3' ו- 5' סוף אקסון שני ברציפות אינטרון, או להיפך. לבסוף, לפני הרצף של הכלאה microarray, לאמת את הדגימות מאת RT-qPCR. בשביל זה, גנים שונים עם רמות שונות של ביטוי ובקרת גנים צריך להיות נבחר, ניתח. בצורה אופטימלית, זו צריכה להתבצע עבור שני שברים שונים של RNA (מצב יציב, שזה עתה מסונתז RNA).
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנו מודים לזלו תורה התמיכה שלו, נגד פישר, שומאכר ק ו פ Saafin El לדיונים שלהם. שחפת נתמכה על ידי מלגת מארי קירי-ITN (PITN-GA-2013-606806, נ-NET) ואת הקשת Fondation. עבודה זו נתמכה על ידי קרנות מ- סוכנות הידיעות נאסיונאל דה לה רשרש (ANR-15-CE11-0022 SAGA2). מחקר זה נתמך גם על ידי ANR-10-LABX-0030-INRT, קרן צרפתית המדינה ניהלה את סוכנות הידיעות נאסיונאל דה לה רשרש תחת d'Avenir Investissements תוכנית מסגרת ANR-10-IDEX-0002-02.
Materials
| Name | Company | Catalog Number | Comments |
| 4-Thiouracil | Sigma-Aldrich | Cat# 440736 | |
| Rapamycin | Euromedex | Cat# SYN-1185 | |
| Countess II FL Automated Cell Counter | ThermoFisher | N/A | |
| RiboPure RNA Purification kit, yeast | ThermoFisher | Cat# AM1926 | |
| NanoDrop 2000 Spectrophotometer | ThermoFisher | ND-2000 | |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | |
| EZ-Link HPDP Biotin | ThermoFisher | Cat# 21341 | |
| Thiolutin | Abcam | ab143556 | |
| µMACS Streptavidin kit | Miltenyi Biotec | Cat# 130-074-101 | |
| Transcriptor Reverse Transcriptase | Roche | 03 531 295 001 | |
| SYBR Green I Master | Roche | 4707516001 | |
| GeneChip Yeast Genome 2.0 | ThermoFisher | 900555 | |
| GeneChip Fluidics Station 450 | ThermoFisher | 00-0079 | |
| GeneChip Scanner 3000 7G | ThermoFisher | 00-0210 |
References
- Gardini, A. Global Run-On Sequencing (GRO-Seq). Methods in Molecular Biology. 1468, 111-120 (2017).
- Churchman, L. S., Weissman, J. S. Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature. 469 (7330), 368-373 (2011).
- Churchman, L. S., Weissman, J. S. Native elongating transcript sequencing (NET-seq). Current Protocols in Molecular Biology. , Chapter 4 Unit 4 11-17 (2012).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23 (2), 232-237 (2005).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14 (9), 1959-1972 (2008).
- Kenzelmann, M., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proceedings of the National Academy of Sciences of the United States of America. 104 (15), 6164-6169 (2007).
- Radle, B., et al. Metabolic labeling of newly transcribed RNA for high resolution gene expression profiling of RNA synthesis, processing and decay in cell culture. Journal of Visualized Experiments. (78), e50195(2013).
- Schwalb, B., et al. Measurement of genome-wide RNA synthesis and decay rates with Dynamic Transcriptome Analysis (DTA). Bioinformatics. 28 (6), 884-885 (2012).
- Sun, M., et al. Global analysis of eukaryotic mRNA degradation reveals Xrn1-dependent buffering of transcript levels. Molecular Cell. 52 (1), 52-62 (2013).
- Sun, M., et al. Comparative dynamic transcriptome analysis (cDTA) reveals mutual feedback between mRNA synthesis and degradation. Genome Research. 22 (7), 1350-1359 (2012).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Munchel, S. E., Shultzaberger, R. K., Takizawa, N., Weis, K. Dynamic profiling of mRNA turnover reveals gene-specific and system-wide regulation of mRNA decay. Molecular Biology of the Cell. 22 (15), 2787-2795 (2011).
- Eser, P., et al. Determinants of RNA metabolism in the Schizosaccharomyces pombe genome. Molecular Systems Biology. 12 (2), 857(2016).
- Baptista, T., et al. SAGA Is a General Cofactor for RNA Polymerase II Transcription. Molecular Cell. 68 (1), 130-143 (2017).
- Bonnet, J., et al. The SAGA coactivator complex acts on the whole transcribed genome and is required for RNA polymerase II transcription. Genes & Development. 28 (18), 1999-2012 (2014).
- Warfield, L., et al. Transcription of Nearly All Yeast RNA Polymerase II-Transcribed Genes Is Dependent on Transcription Factor TFIID. Molecular Cell. 68 (1), 118-129 (2017).
- Huisinga, K. L., Pugh, B. F. A genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA in Saccharomyces cerevisiae. Molecular Cell. 13 (4), 573-585 (2004).
- Lee, T. I., et al. Redundant roles for the TFIID and SAGA complexes in global transcription. Nature. 405 (6787), 701-704 (2000).
- Lenstra, T. L., et al. The specificity and topology of chromatin interaction pathways in yeast. Molecular Cell. 42 (4), 536-549 (2011).
- Plaschka, C., et al. Architecture of the RNA polymerase II-Mediator core initiation complex. Nature. 518 (7539), 376-380 (2015).
- Helenius, K., et al. Requirement of TFIIH kinase subunit Mat1 for RNA Pol II C-terminal domain Ser5 phosphorylation, transcription and mRNA turnover. Nucleic Acids Research. 39 (12), 5025-5035 (2011).
- Rodriguez-Molina, J. B., Tseng, S. C., Simonett, S. P., Taunton, J., Ansari, A. Z. Engineered Covalent Inactivation of TFIIH-Kinase Reveals an Elongation Checkpoint and Results in Widespread mRNA Stabilization. Molecular Cell. 63 (3), 433-444 (2016).
- Haimovich, G., et al. Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. Cell. 153 (5), 1000-1011 (2013).
- Haruki, H., Nishikawa, J., Laemmli, U. K. The anchor-away technique: rapid, conditional establishment of yeast mutant phenotypes. Molecular Cell. 31 (6), 925-932 (2008).
- Neymotin, B., Athanasiadou, R., Gresham, D. Determination of in vivo RNA kinetics using RATE-seq. RNA. 20 (10), 1645-1652 (2014).
- Shetty, A., et al. Spt5 Plays Vital Roles in the Control of Sense and Antisense Transcription Elongation. Molecular Cell. 66 (1), 77-88 (2017).
- Xu, Y., et al. Architecture of the RNA polymerase II-Paf1C-TFIIS transcription elongation complex. Nature Communications. 8, 15741(2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews. Genetics. 13 (10), 720-731 (2012).
- Nojima, T., Gomes, T., Carmo-Fonseca, M., Proudfoot, N. J. Mammalian NET-seq analysis defines nascent RNA profiles and associated RNA processing genome-wide. Nature Protocols. 11 (3), 413-428 (2016).
- Storvall, H., Ramskold, D., Sandberg, R. Efficient and comprehensive representation of uniqueness for next-generation sequencing by minimum unique length analyses. PloS One. 8 (1), 53822(2013).
- Tani, H., et al. Identification of hundreds of novel UPF1 target transcripts by direct determination of whole transcriptome stability. RNA Biology. 9 (11), 1370-1379 (2012).
- Tani, H., et al. Genome-wide determination of RNA stability reveals hundreds of short-lived noncoding transcripts in mammals. Genome Research. 22 (5), 947-956 (2012).
- Jao, C. Y., Salic, A. Exploring RNA transcription and turnover in vivo. by using click chemistry. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15779-15784 (2008).
- Palozola, K. C., et al. Mitotic transcription and waves of gene reactivation during mitotic exit. Science. 358 (6359), 119-122 (2017).
- Ardehali, M. B., et al. Polycomb Repressive Complex 2 Methylates Elongin A to Regulate Transcription. Molecular Cell. 68 (5), 872-884 (2017).
- Schulz, D., et al. Transcriptome surveillance by selective termination of noncoding RNA synthesis. Cell. 155 (5), 1075-1087 (2013).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Duffy, E. E., et al. Tracking Distinct RNA Populations Using Efficient and Reversible Covalent Chemistry. Molecular Cell. 59 (5), 858-866 (2015).
- Rutkowski, A. J., Dolken, L. High-Resolution Gene Expression Profiling of RNA Synthesis, Processing, and Decay by Metabolic Labeling of Newly Transcribed RNA Using 4-Thiouridine. Methods in Molecular Biology. 1507, 129-140 (2017).
- Darzacq, X., et al. In vivo dynamics of RNA polymerase II transcription. Nature Structural & Molecular Biology. 14 (9), 796-806 (2007).
- Fuchs, G., et al. Simultaneous measurement of genome-wide transcription elongation speeds and rates of RNA polymerase II transition into active elongation with 4sUDRB-seq. Nature Protocols. 10 (4), 605-618 (2015).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16 (11), 1128-1133 (2009).
- Schwalb, B., et al. TT-seq maps the human transient transcriptome. Science. 352 (6290), 1225-1228 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved