Method Article
Saccharomyces cerevisiae Metabólica de rotulagem com 4-thiouracil e a quantificação dos recém sintetizado mRNA como um Proxy para a RNA polimerase II atividade
Neste Artigo
Resumo
O protocolo descrito aqui baseia-se a quantificação de todo o genoma de mRNA recentemente sintetizadas purificada de células de levedura rotuladas com 4-thiouracil. Este método permite medir a síntese de mRNA desacoplada do decaimento do mRNA e, portanto, fornece uma medição precisa da transcrição do RNA polimerase II.
Resumo
Globais defeitos na transcrição do RNA polimerase II podem ser ignorados pelos estudos de transcriptomic, analisando o estado estacionário do RNA. Com efeito, a diminuição global da síntese de mRNA foi mostrada para ser compensado por uma diminuição simultânea na degradação do mRNA para restaurar os níveis normais de estado estacionário. Portanto, a quantificação de todo o genoma de síntese de mRNA, independentemente do decaimento do mRNA, é o melhor reflexo direto da atividade transcricional de RNA polimerase II. Aqui, vamos discutir um método usando não-distorçer metabólica rotulagem de RNAs nascentes em Saccharomyces cerevisiae (S. cerevisiae). Especificamente, as células são cultivadas por 6 min com uma uracila analógico, 4-thiouracil, e os rotulado RNAs recém transcritos são purificados e quantificados para determinar as taxas de síntese de mRNA individuais todos os. Além disso, usando rotulado Schizosaccharomyces pombe células como padrão interno permite comparar a síntese de mRNA em S. cerevisiae de diferentes linhagens. Usando este protocolo e encaixe os dados com um modelo cinético dinâmico, as taxas de decaimento do mRNA correspondente podem ser determinadas.
Introdução
As células respondem a sinais endógenos e exógenos, através da alteração dinâmica do seu programa de expressão do gene. Nos últimos anos, um enorme desenvolvimento das metodologias de todo o genoma permite a descrição precisa e abrangente de transcriptoma mudanças em condições diferentes. Na maioria dos estudos transcriptomic, sequenciamento de hibridação ou elevado-throughput de microarray são usadas para quantificar os níveis de RNA de uma fração de RNA total estado estacionário. Transcriptional mudanças sob uma perturbação específica podem exibir uma ampla gama de resultados possíveis, com um amplo espectro de genes sendo - up ou ativador ou mudanças de expressão de genes específicos. Expressão do gene é o resultado de um equilíbrio de aperfeiçoá-lo — ou estado estacionário — entre a síntese de RNA pela ARN-polimerase e outros processos que afetam os níveis de RNA. Transcrição de RNA polimerase II, incluindo suas três fases distintas (iniciação, alongamento e término), é altamente e intricadamente associado com processamento, exportação citoplasmática, tradução e degradação do mRNA.
Vários estudos recentes demonstraram que a decadência e a síntese de mRNAs são mecanismos acoplados e mostraram que efeitos transcriptional sobre mutação ou sob estímulos podem ser negligenciados quando quantificar o RNA total de estado estacionário. Em primeiro lugar, a detecção de alterações transcricionais através da análise dos níveis de estado estacionário de mRNA sempre depende de mRNAs Half-Life. Uma vez que a perturbação é introduzida, os níveis de estado estacionário de mRNAs com meia-vida longa serão muito menos afetados do que aqueles dos mRNAs com meia-vida curta. Portanto, a capacidade de detecção das alterações na síntese do RNA é fortemente tendencioso em favor de transcrições de curta duração, enquanto a análise das espécies de mRNA caudais pode falhar para revelar alterações dinâmicas na taxa de transcrição. Em segundo lugar, vários relatos têm demonstrado que, tanto em leveduras e mamíferos, mudanças globais na transcrição podem ser negligenciadas ao analisar os níveis de estado estacionário de mRNA. Isto é provavelmente devido os mecanismos que vinculam a síntese de mRNA e degradação, resultando em buffer de mRNA. Isto levou ao desenvolvimento de novos protocolos para quantificar a síntese de mRNA desacoplada da degradação, através da análise do mRNA recentemente transcrito. Nos últimos anos, foram apresentadas várias alternativas, incluindo sequenciamento pperigoso global (GRO-seq)1e transcrição de alongamento nativo sequenciamento (NET-seq)2,3. Aqui, apresentamos um protocolo desenvolvido inicialmente em células de mamíferos4,5,6 e em seguida adaptado para levedura7,8,9,10, 11, que é baseado no RNA etiquetando com um nucleosídeo thiolated ou base analógica, 4-thiouridine (4sU) ou 4-thiouracil (4tU), respectivamente.
Esse método especificamente purifica recém transcrito de RNA das células no qual ARN são pulso-etiquetadas com 4sU com virtualmente nenhuma interferência na homeostase da célula. Portanto, uma vez que as células são expostas a 4sU, a molécula rapidamente é captação, fosforilada para 4sU-trifosfato e incorporada em RNAs sendo transcritos. Uma vez rotulado de pulso, é possível extrair o RNA celular total (correspondente aos níveis de estado estacionário do RNA), e, posteriormente, a fração de RNA 4sU-rotulado thiol-especificamente modificado, levando à formação de uma ligação dissulfureto entre biotina e a recentemente transcrito de RNA4,5. No entanto, 4sU só pode ser a captação pelas células expressando um transporte de nucleosídeo, como o transporte de nucleosídeo equilibrative humano (hENT1), impedindo a sua utilização imediata em leveduras brotamento ou fissão. Enquanto um pode expressar a hENT1 em S. pombe ou S. cerevisiae, uma abordagem mais fácil pode ser alcançada usando o 4tU base modificado, uma vez que as células de levedura podem demorar até 4tU, sem a necessidade de expressão de um transporte de nucleosídeos10, 11 , 12 , 13. na verdade, o metabolismo do 4tU exige a atividade da enzima uracila Fosforibosiltransferase (UPRT). Em vários organismos, incluindo o fermento mas não mamíferos, UPRT é essencial para uma via de salvamento de pirimidina, reciclagem uracil para monofosfato de uridina.
Um viés importante em estudos de transcriptomic pode ser introduzido pela normalização entre diferentes amostras analisadas em paralelo. De fato, muitos fatores diferentes podem afetar a análise comparativa da transcriptoma do mutante e as estirpes de tipo selvagem: a eficiência do lysis da pilha, as diferenças na extração e recuperação de RNA e desvios na calibração de scanner para análise de microarray , entre outros. Como discutido acima, essas variações podem ser particularmente enganosas, quando são esperados efeitos globais na transcrição do RNA polimerase II. Uma elegante significa precisão comparar taxas de síntese de mRNA entre diferentes amostras foi projetada usando o fermento de fissão distantemente relacionados Schizosaccharomyces pombe como padrão interno. Por isso, um número fixo de rotulado S. pombe células é adicionado para as amostras de S. cerevisiae , células selvagem-tipo ou mutantes, antes da lise celular e de extração de RNA10. Posteriormente, RNAs de estado estacionário e recentemente sintetizadas de S. pombe e S. cerevisiae são quantificados por RT-qPCR ou através do uso de chips de microarray ou arranjar em sequência do elevado-throughput10. Combinando estes dados com modelagem cinética, absolutas taxas de síntese de mRNA e decadência em leveduras brotamento podem ser medidas.
No âmbito do presente manuscrito, vamos mostrar como a análise do RNA transcrito recentemente autorizados a revelar um papel global para os complexos coactivator SAGA e TFIID na transcrição de RNA polimerase II em brotamento fermento14,15, 16. Importante, após estudos quantificar os níveis de RNAm de estado estacionário em S. cerevisiae e sugeriram que a SAGA desempenha uma função predominante em um conjunto limitado de genes de fermento que são fortemente afetadas por mutações na SAGA, mas relativamente resistente à TFIID mutações17,18,19. Surpreendentemente, as atividades enzimáticas SAGA foram mostradas para agir sobre o genoma inteiro transcrito, sugerindo um papel mais amplo para esse ativador co na transcrição do RNA polimerase II. Observou-se diminuição da recrutamento de II de polimerase de RNA em genes mais expressos mediante a inativação da SAGA ou TFIID, sugerindo que estes coactivators trabalham juntos na maioria dos genes. Portanto, a quantificação de mRNA recentemente transcrito revelou que a SAGA e TFIID são necessários para a transcrição dos genes quase todos pela RNA polimerase II14,15,16. A implementação de mecanismos compensatórios emerge como uma maneira para que as células lidar com uma diminuição global da síntese de mRNA que é armazenado em buffer por uma simultânea redução global na degradação do mRNA. SAGA adiciona à lista de fatores, tendo um efeito global na transcrição do RNA polimerase II, a tais como subunidades de RNA Pol II10, o mediador coactivator complexo20, o general transcrição fator TFIIH21,22 e indirectamente, de elementos de degradação do mRNA máquinas9,10,23. Tais eventos compensatórios foram observados universalmente em mutantes SAGA, contabilidade para as modestas e limitadas alterações nos níveis de RNAm de estado estacionário apesar de uma diminuição global e grave na síntese de mRNA14. Análises semelhantes também foram realizadas em uma cepa de exclusão BRE1 , resultando em uma perda completa da ubiquitination histona H2B. Curiosamente, um tanto mais suave mas consistente efeito global na transcrição do RNA polimerase II poderia ser detectado na ausência de Bre1, indicando que a rotulagem metabólica do RNA recém transcrito no fermento pode detectar e quantificar uma vasta gama de mudanças no mRNA taxas de síntese.
Protocolo
1. célula de cultivo e rapamicina depleção de uma subunidade da SAGA
- Para cada cepa de S. cerevisiae e replicar, incluindo o selvagem-tipo ou cepas de controle, inocular uma única colônia de um prato fresco em 5 mL de meio YPD (2% de peptona, extrato de levedura 1% e 2% de glicose).
- Desenvolvem-se células de S. cerevisiae durante a noite a 30 ° C, com agitação constante (150 rpm).
- Medir a densidade óptica em 600 nm (OD600) e diluir a cultura para uma OD600 de aproximadamente 0.1 em 100 mL de meio YPD e deixe crescer até que o OD600 é em torno de 0,8.
- Em paralelo, inocular uma única colônia de células de S. pombe de um prato fresco em 50 mL de meio de Sim (extrato de levedura 0,5%; 250 mg/L adenina, uracila, histidina, leucina e lisina; 3% de glicose) e crescer as células durante a noite a 32 ° C, com agitação constante (150 rotações por minuto).
- Medir o OD600 de cultura durante a noite de S. pombe e diluir a cultura para uma OD600 de aproximadamente 0.1 em 500 mL de meio de Sim e deixá-lo crescer até o OD600 é aproximadamente 0,8.
- Para cada estirpe âncora-afastado de S. cerevisiae e replicar, incluindo o selvagem-tipo ou cepas de controle, inocular uma única colônia de um prato fresco em 5 mL de meio YPD (2% de peptona, extrato de levedura 1% e 2% de glicose).
- Crescem as células durante a noite a 30 ° C, com agitação constante (150 rpm).
- Na manhã seguinte, medir a OD600, diluir a cultura para uma OD600 de aproximadamente 0.1 em 100 mL de meio YPD e deixá-lo crescer até o OD600≈ 0,8.
- Adicionar 100 µ l de rapamicina para a cultura de uma solução stock de 1 mg/mL (concentração final de rapamicina de 1 µ g/mL) e incubar a 30 ° C, com agitação constante durante o tempo necessário para a proteína de interesse para ser condicionalmente empobrecido do núcleo (deixar que as células Normalmente, 30 min é adequada). Para o controle, use uma cultura de levedura semelhante, mas em vez de adicionar rapamicina, adicionar o volume equivalente de Dimetilsulfóxido (DMSO).
2. 4tU rotulagem com S. pombe Spike-in (contagem)
-
Prepare uma solução fresca de 2 M. 4-thiouracil. Uma vez preparados, mantê-lo em temperatura ambiente e longe da luz.
- Com precisão pesar 64,1 mg de 4-thiouracil de cada cultura de S. cerevisiae e dissolvê-lo em 250 µ l de dimetilformamida (DMF) ou em 250 µ l de DMSO.
- Para a cultura de S. pombe ser usado como spike-no, pesa 320,5 mg de 4-thiouracil e dissolvê-lo em 1.250 µ l de DMSO.
- Adicionar a solução de 4-thiouracil de S. cerevisiae e S. pombe culturas para uma concentração final de 5 mM e incube-os por 6 min com agitação constante a 30 ° C e 32 ° C, respectivamente.
- Após 6 min, retire uma pequena alíquota de cada cultura para a contagem de células. Conte as células usando um contador automático de células ou uma câmara de Neubauer.
- Coletar as células através de centrifugação (2.500 x g) por 5 min a 4 ° C.
- Desprezar o sobrenadante, lavar as células com PBS 1x gelada e centrifugar novamente (2.500 x g, 4 ° C, 5 min).
- Calcule o número total de células em cada uma das amostras de S. cerevisiae e S. pombe .
- Ressuspender as células em 5 mL de PBS 1x gelada e misture S. cerevisiae com células de S. pombe , com uma proporção de 3:1.
- Centrifugar as células (2.500 x g, 4 ° C, 5 min), remover a PBS, congelar as células no líquido N2e armazenar a amostra a-80 ° C até utilização posterior.
3. o RNA extração e tratamento de DNase
- Descongele as células no gelo por aproximadamente 20 a 30 min.
- Proceda com a extração do RNA usando um kit de extração de RNA de leveduras (Tabela de materiais) com algumas adaptações.
- Por cada amostra, despeje 750 µ l de grânulos de Zirconia gelados em um tubo de 1,5 mL de tampão de parafuso fornecido com o kit. Lembre-se que por cada tubo, RNA de até 109 células pode ser eficientemente extraído. Portanto, prepare-se o número de tubos necessários para cada amostra. Por exemplo, uma cultura de S. cerevisiae de 100 mL (OD600≈ 0,8) pode processar em torno de 2 x 109 3 x 109 células, levando até um total de 2.7 x 109 4 x 109 células no total (após o pico-com um terço da S. pombe células). Neste caso, até 3-4 tubos de reação por cada amostra/condição/mutante/replicar seria necessária.
- Por cada 1 x 109 células, adicione 480 µ l do lysis buffer fornecido com o kit, 48 µ l de SDS 10% e 480 µ l de álcool isoamílico: fenol: clorofórmio (25:24:1, v/v/v).
- Misture as células usando um misturador do vortex e transferi-los para os tubos contendo os grânulos de Zirconia gelados.
- Acomodar os tubos em um adaptador de misturador de vórtice, vire o vórtice na velocidade máxima e bata por 10 min lisar as células de levedura (em um quarto a 4 ° C). Como alternativa, realize a Lise das células em um grânulo-batedor automático.
- Centrifugar tubos a 16.000 x g durante 5 min à temperatura ambiente e cuidadosamente, coletar a fase superior (fase contendo RNA) para um tubo de Falcão de 15 mL. Normalmente, o volume recuperado por cada tubo é em torno de 500 – 600 µ l.
- Para os tubos de 15 mL contendo o RNA parcialmente purificado, adicione o buffer de ligação fornecido com o kit e misturar cuidadosamente. Por cada 100 µ l de solução de RNA, adicionar 350 µ l de tampão de ligação (ou seja, quando o volume da fase aquosa solução é 600 µ l, 2,1 mL de tampão de ligação deve ser adicionado).
- Para a mistura anterior, adicione o etanol 100% e misture bem. Por cada 100 µ l de solução de RNA, acrescentar 235 µ l de etanol 100% (ou seja, quando o volume da fase aquosa solução é 600 µ l, adicionar 1,41 mL de etanol).
- Aplica até 700 µ l da mistura da etapa 3,9 para um cartucho de filtro montado em um tubo de coleta, ambos fornecidos com o kit.
- Centrífuga para 1 min a 16.000 x g. Se a duração da centrifugação não foi suficiente para o volume total de passar através do filtro, repetir a centrifugação por 30 s.
- Descartar o escoamento e reutilizar o tubo da mesma coleção. Adicione outro 700 μL de solução de tampão-etanol de RNA-obrigatórias para o filtro e centrifugar novamente 16.000 x g por 1 min.
- Descartar o escoamento e repita os passos 3.11 e 3.12 até a solução do RNA é terminada.
- Lavar o filtro 2 x com 700 µ l de solução 1 de lavagem. Recolher a lavagem solução através de centrifugação a 16.000 x g por 1 min e mantenha sempre o tubo de coleta.
- Lavar o filtro 2 x com 500 µ l de solução 2 de lavagem. Recolher a lavagem solução através de centrifugação a 16.000 x g por 1 min e mantenha sempre o tubo de coleta.
- Centrifuga os tubos mais uma vez em 16.000 x g por 1 min secar completamente o filtro.
- Transferir o refil do filtro para o tubo de coleta final (tubo apropriado de RNA) e eluir RNA com 50 µ l de DEPC tratada, livre de RNase H2O (pré-aquecido a 100 ° C).
- Centrífuga para 1 min a 16.000 x g.
- Eluir RNA novamente (para o mesmo tubo) com 50 µ l de pré-aquecido DEPC tratada, livre de RNase H2O. Certifique-se de que todo o volume passou através do filtro; caso contrário, centrifugue durante períodos mais longos.
- Se forem utilizados tubos múltiplos para uma única amostra, piscina-los todos em um tubo.
- Quantificar e verificar a pureza da amostra usando o equipamento apropriado.
Nota: Enquanto 4tU só é incorporado dentro RNA recém sintetizado, há uma chance de menor contaminação com DNA. Por esse motivo, é sempre aconselhável para tratar as amostras com DNase eu. Para isso, use os reagentes fornecidos com o kit de extração de RNA-(Tabela de materiais), seguindo as recomendações do fabricante.
4. tiol específicas Biotinylation do RNA recém sintetizado
- Ajuste a concentração do RNA obtido com a seção 3 do protocolo de 2 mg/mL. Alíquota de 200 microgramas de RNA total, aqueça-a durante 10 min a 60 ° C e chill-lo imediatamente no gelo por 2 min.
-
A alíquota do RNA, adicionar os reagentes mencionados abaixo na seguinte ordem: 600 µ l de DEPC tratada, livre de RNase H2O, 100 µ l de tampão de biotinylation (100 mM Tris-HCl [pH 7.5] e 10 mM de EDTA e de DEPC tratada, livre de RNase H2O) e 200 µ l de Biotina-HPDP de um estoque de 1 mg/mL biotina-HPDP em DMSO ou DMF.
- Em algumas situações, a solução de biotina-HPDP tende a precipitar, provavelmente devido à sua baixa solubilidade em água. Nesta situação, aumentar o volume de DMSO/DMF até 40% sobre o volume de reação (com a amostra de RNA, adicione 400 µ l de DEPC tratada H2O, 100 µ l de tampão de biotinylation e 400 µ l de biotina-HPDP de um estoque de 0,5 mg/mL).
- Incubar a amostra em temperatura ambiente e protegido da luz por 3 h, com leve agitação.
- Após a incubação, adicionar um volume aproximadamente igual de clorofórmio para os tubos e misture vigorosamente.
- Girar a amostra a 13.000 x g por 5 min, a 4 ° C. Esta etapa permite a remoção de excesso biotina que fez não biotinylate o RNA. Como alternativa, execute esta etapa usando tubos de captura de fase (pesados). Por isso, spin para baixo os tubos de captura de fase por 1 min a 13.000 x g, adicione a mistura de RNA e uma quantidade igual de clorofórmio, misturá-los vigorosamente e centrifugá-los a 13.000 x g durante 5 min, a 4 ° C.
- Transferi com cuidado a fase superior para novos tubos de 2 mL.
- Adicionar um décimo do volume de 5 M NaCl e misturar a amostra.
- Adicionar um volume igual de isopropanol, homogeneizar bem a amostra e girá-lo em 13.000 x g , durante pelo menos 30 min, a 4 ° C.
- Cautelosamente, remover o sobrenadante e adicionar 1 mL de etanol gelado de 75%.
- Girar a 13.000 x g durante 10 minutos, a 4 ° C.
- Cuidadosamente remova o sobrenadante, rápido-rotação do tubo e a restante da solução de etanol. Certifique-se que a pelota do RNA não seca.
- Suspender o RNA em 100 µ l de DEPC tratada, livre de RNase H2O.
5. purificação da fração recentemente sintetizada de RNA Total e sem rótulo usando grânulos magnéticos Streptavidin-revestido
- Aqueça o biotinilado RNA durante 10 minutos a 65 ° C e em seguida relaxar as amostras no gelo por 5 min.
- Adicione 100 µ l de grânulos magnéticos streptavidin-revestido para o RNA biotinilado (em um volume final de 200 µ l). Especificamente, é recomendável usar os grânulos indicados na Tabela de materiais, desde que, após conversas com outros laboratórios, estes parecidos ser a mais consistente e confiável.
- Incube a amostra com ligeira agitação por 90 min, à temperatura ambiente.
- Coloque as colunas fornecidas com o kit (Tabela de materiais), no suporte magnético.
- Adicionar 900 µ l de tampão de lavagem temperatura (100 mM Tris-HCl [pH 7,5], EDTA 10 mM, 1 M NaCl e 0,1% de Tween 20, em DEPC tratada, livre de RNase H2O) para as colunas (pre-executar e equilibrar).
- Aplique a mistura de grânulos/RNA (200 µ l) para as colunas.
- Coletar o escoamento em tubos de 1,5 mL e aplicá-lo novamente para a mesma coluna magnética. Se necessário, manter esta passagem como ele representa a fração de RNA sem rótulo.
- Lavar as colunas 5 x com crescentes volumes de tampão de lavagem (600, 700, 800, 900 e 1.000 µ l).
- Eluir o RNA recém sintetizado com 200 µ l de 0,1 M de TDT.
- Execute uma segunda eluição, 3 min mais tarde, com igual volume de 0,1 M de TDT.
- Após a eluição do RNA, adicionar 0,1 volumes de NaOAc 3 M (pH 5.2), 3 volumes de gelada 100% de etanol e 2 µ l de glicogênio 20 mg/mL (RNA-classe) e deixe a RNA precipitado durante a noite, a-20 ° C.
- Recuperar o RNA por centrifugação (13.000 x g durante 10 minutos, a 4 ° C) e Resuspenda-lo em 15 µ l de DEPC tratada, livre de RNase H20. A proporção de rotulado-para total do RNA é esperada para ser ao redor 2% - 4% (geralmente mais em direção a extremidade inferior), renderização de aproximadamente 2,0 µ g de RNA recém sintetizado. Esta quantidade é suficiente para fazer várias experiências de qPCR, bem como para análises microarray/sequenciamento.
6. RT-qPCR validação das diferentes frações
- Sintetiza o cDNA de 2 µ g de RNA total ou 10 µ l de RNA etiquetado usando hexâmeros aleatórios e o transcriptase reversa de escolha, de acordo com as instruções do fabricante (Tabela de materiais).
- Amplifica o cDNA por qPCR em tempo real, usando um protocolo padrão (Tabela de materiais).
Nota: Todas as amostras devem ser executadas em triplicado desde um mínimo de duas repetições biológicas. Corrigi todos os valores brutos para a expressão de S. pombe tubulina.
7. Microarray hibridização
- Hibridizam amostras de RNA em fichas de microarray preferencial de acordo com as instruções do fabricante (para este protocolo específico, consulte Tabela de materiais). Brevemente, preparar biotinilado cRNA alvos de 150 ng do RNA usando o Premier RNA Amplification Kit (Tabela de materiais), de acordo com as instruções do fabricante. Hibridizam 4mg de cRNAs fragmentada para 16 h a 45 ° C e 60 rpm em chips de microarray.
- Lavar, mancha e digitalizar as fichas usando a estação indicada e scanner (Tabela de materiais). Extrai os dados brutos (arquivos de intensidade CEL) das imagens digitalizadas usando o console de comando (AGCC, versão 4.1.2).
- Mais processe os arquivos do CEL com versão de software do Console de expressão 1.4.1 para calcular a sonda conjunto intensidades de sinal, usando os algoritmos baseados em estatísticas MAS 5.0 com as configurações padrão e a escala global, como método de normalização.
Nota: A intensidade de alvo média aparada de cada chip foi arbitrariamente definida como 100. Execute todos os experimentos usando pelo menos duas repetições biológicas independentes. Normalizar os dados brutos para o sinal de S. pombe e calcular prega mudanças nos níveis de RNA totais e recém sintetizadas.
8. dados análise usando um Pipeline R existente
- Calcular a síntese e decadência taxas usando um pipeline e o pacote R/Bioconductor publicamente disponível, como descrito anteriormente8,10.
Resultados
Ao realizar a rotulagem metabólica do RNA recém transcrito, vários aspectos precisam ser controlados: o tempo e a eficiência da rotulagem, a proporção de pico-no, o protocolo de extração e a eficácia de biotinylation (incluindo relação sinal-ruído), entre outros. Estas condições foram extensivamente e metodicamente mostradas por outros7,10,11. Aqui focamos principalmente sobre a interpretação e a análise imediata que pode ser executada, uma vez que as amostras foram processadas, por RT-qPCR, microarray ou sequenciamento. As análises de diferentes estirpes mutantes demonstram o poder do método para detectar não apenas uma diminuição dramática global na síntese de mRNA, como no caso de mutante SAGA S. cerevisiae cepas, mas também uma redução muito suave actividade RNA polimerase II em cima supressão da monoubiquitination de histona H2B.
Nossas análises de actividades enzimáticas SAGA sugeriram um recrutamento amplo a cromatina15, que não foi revelado pela análise dos níveis de mRNA de estado estacionário em cepas mutantes de SAGA. Como RNA polimerase II recrutamento foi prejudicado após inativação de SAGA, nós decidimos analisar se taxas de síntese de mRNA seria afetadas globalmente. Daí, selvagem-tipo ou mutante cepas de S. cerevisiae foram expostas ao 4tU durante um período de 6 min, a gravadora RNAs recém transcritos. Após a mistura com as spike-em pilhas etiquetadas (S. pombe) em uma proporção de 3:1, o RNA total foi extraído, e RNA recém sintetizado foi biotinilado e purificado de acordo com o protocolo apresentado aqui, como no cronograma mostrado na Figura 1. Rotulado de RNAs foram depuradas, de um total de 200 µ g de RNA, garantindo que a quantidade de produto purificado seria suficiente para qualquer aplicação a jusante. Como um passo inicial e sistemático antes de qualquer análise de todo o genoma, purificação de RNA recém sintetizada foi validada por RT-qPCR. Os genes foram selecionados de acordo com parâmetros diferentes, incluindo um nível de expressão, vias reguladoras e uma dependência da ARN-polimerase diferente.
Para confirmar que este protocolo especificamente purifica etiquetado RNA, podemos quantificar os níveis de transcritos em fracções purificados do selvagem-tipo células que foram cultivadas com ou sem 4tU. Níveis insignificantes dos RNAs analisados foram detectados de células que não foram expostas ao 4tU(Figura 2). Purificação de RNA recém transcrita mais foi validada pelo enriquecimento observado do intron-contendo ACT1 pre-mRNA (dados não mostrados). Após a validação da qualidade das amostras, nós testamos se síntese de mRNA seria afetado após a exclusão de SPT20, que é conhecido para interromper a montagem complexa da SAGA. Conforme relatado por outros, quantificação de mRNA realizada no RNA (estado estacionário) total da estirpe Δ spt20revelou níveis principalmente inalterados ou levemente reduzidos para os genes testados (Figura 2B). Resultados semelhantes foram obtidos por estirpes excluídas para BRE1 (Figura 2B). Em contraste, a análise do RNA recém transcrito da estirpe Δ spt20revelou uma redução dramática na síntese do mRNA por três-testaram para cinco vezes para todos os genes (Figura 2C). A perda de Bre1 levou a uma diminuição de mais discreta, mas ainda visível em níveis de mRNA recentemente sintetizadas para os genes estudados (Figura 2C). Bom de acordo com um papel de SAGA e Bre1 na transcrição do RNA polimerase II, a perda de Spt20 ou Bre1 não afetou a expressão dos genes RDN25 ou snR6 , transcrito por RNA polimerase I e RNA polimerase III, respectivamente ( Figura 2C).
No entanto, cepas eliminadas por subunidades estruturais da SAGA complexo, como SPT7 ou SPT20, exibem fenótipos graves de crescimento lento que podem explicar as alterações observadas transcriptional. Para descartar os efeitos secundários indesejáveis, que nós condicionalmente empobrecido Spt7 do núcleo, usar âncora-afastado s. cerevisiae cepas de14,24. Após 60 min de tratamento de rapamicina, antes a pulso-rotulagem com 4tU (ver Figura 1 para uma representação esquemática), os níveis de mRNA recentemente transcrito foram reduzidos de forma similar como observada na estirpe de exclusão (Figura 2D e 2E ). Nesta análise, assim, confirmado nossos resultados antigos e validado o protocolo para este sistema de esgotamento inducible. Em um tempo-curso de análise, onde as células foram expostas a rapamicina para uma abrangência de tempo de 0 a 240 min, evidenciou-se reduzida expressão imediatamente após 15 min de exposição à droga. Mais interessante, os níveis do mRNA de estado estacionário tendem a diminuir inicialmente mas retornado aos níveis normais após 60 min, uma indicação de que um mecanismo compensatório ocorre entretanto(Figura 2).
No total, a rotulagem e a quantificação do RNA recém sintetizado permitiram revelar novas funções reguladoras para o complexo SAGA. O protocolo descrito também poderia revelar efeitos moderados na atividade do RNA polymerase II e foi aplicada com sucesso para conditional depleção de cepas de leveduras.
Uma das aplicações a jusante para o RNA recém sintetizado purificada é uma quantificação de todo o genoma de transcrições usando microarray hibridização ou sequenciamento (4tU-seq). Enquanto o sequenciamento de alto rendimento é mais quantitativa, sensível e informativo, microarray hibridização pode ser muito útil para determinar se os níveis do mRNA global são alterados. Neste contexto, onde a normalização é fundamental, nós adicionamos um pico no organismo para a amostra que tivemos como objetivo analisar. Especificamente, misturado S. cerevisiae células às células de S. pombe em uma proporção de 3:1, ambos sendo exposto anteriormente a 4tU. Quando os purificado, etiquetados RNAs são submetidas a sequenciamento de alta produtividade, pode ser usado qualquer protocolo de preparação de biblioteca padrão, mais frequentemente após depleção de RNA ribossomal. Normalização entre 4tU-seq dados de diferentes amostras foi realizada adicionando também rotulado RNA de uma espécie diferente (i.e., mistura s. cerevisiae e s. pombe células como acima)22 ou em vitro- transcrito, thiolated pico-no RNA12,25,26,27. Microarray comercialmente disponíveis fichas contêm sondas para a transcriptoma inteira de fermento tanto brotamento e fissão, permitindo a quantificação de mRNA de ambos os organismos em uma única experiência. Hibridação de Microarray foram realizadas com o RNA total e recentemente sintetizado, validado por RT-qPCR como indicado acima (tipo selvagem, spt20Δ e bre1Δ). Além disso, incluímos uma estirpe que não oferece suporte a monoubiquitination de histona H2B, através do ponto de mutação do resíduo ubiquitinable (K123R). Porque a mesma proporção exata das células de s. pombe foram utilizados nas diferentes amostras e repetições, é possível redimensionar linearmente as matrizes intensidades para que o total e etiquetados S. pombe sondas têm a mesma intensidade mediana. Esta normalização, ou rescaling, pode ser realizada usando um pacote de Bioconductor desenvolvido pelo laboratório de Patrick Cramer e é usado em paralelo em todas as amostras analisadas, totais e rotulado de frações e tipo selvagem e mutantes cepas8. Brevemente, a entrada desse pipeline é um arquivo do Excel que contenham as sondas e sua intensidade (MAS5 ou RMA) para todas as amostras e as frações. Após a exclusão de sondas que estão fora do intervalo da deteção, a intensidade do sinal é redimensionada, tendo em conta os valores de intensidade das sondas S. pombe . Finalmente, você pode mapear as sondas para o brotamento e genomas de levedura de fissão, terminando com uma matriz que contém os valores de expressão normalizados para a espiga. Esses valores podem ainda ser processados (consulte a próxima seção) ou usado como é. No exemplo a seguir, determinamos que a dobra muda de cada transcrição entre os mutantes e a estirpe selvagem-tipo.
As análises foram realizadas para ambos estado estacionário ou recém sintetizado níveis de RNA e plotados contra sua significância estatística (p-valor) (Figura 3). De acordo com outros estudos, quando os níveis de RNA totais foram analisados, apenas alguns genes tinham sua expressão alterada, ou up - ou ativador (Figura 3A-3 C). No entanto, a análise do RNA recém transcrito levou a conclusões muito diferentes. Os níveis do mRNA recentemente transcrito de mais de 4.000 genes foram significativamente reduzidos pelo menos duplo sobre exclusão de SPT20, sugerindo um efeito positivo global da SAGA na transcrição de RNA polimerase II em leveduras brotamento (Figura 3D ). Além disso, no bre1Δ e K123R mutantes, os resultados foram mais discretos: a maioria dos genes pareciam ter sua expressão reduzida, mas a extensão do downregulation e o número de genes significativamente afetados (≈ 300-500) era de fato mais Limited (Figura 3E e 3F).
Como anteriormente mencionados, estado estacionário ou totais os níveis do mRNA são ditados pelo apertado equilíbrio entre síntese e degradação(Figura 4). Quando a transcrição do RNA polimerase II é globalmente prejudicada, dois cenários podem ser retratados: níveis de (i) ou o mRNA total diminuem globalmente, como uma resposta à síntese reduzida mas constante deterioração, ou degradação do mRNA (ii) é diminuída na mesma medida, resultante na maior parte inalterados os níveis do mRNA de estado estacionário. O segundo cenário tem sido relatado para várias condições, incluindo no contexto da SAGA ou TFIID interrupção9,10,14,16,22. Um procedimento chamado análise comparativa transcriptome dinâmico (CDT) com base em 4tU rotular e modelagem cinética dinâmica nos permite determinar a síntese de mRNA e inferir as taxas de decomposição de cada transcrição8,10. Mais uma vez, aproveitamos os dados coletados para as cepas mencionadas anteriormente (tipo selvagem, spt20Δ, bre1Δ e K123R). Como esperado, após a exclusão de SPT20, observamos uma diminuição simultânea do mRNA taxas de síntese e degradação quando comparado com a estirpe selvagem-tipo. Nesta estirpe mutante, a compensação foi quase ideal, com uma média o diminuição da síntese de 3.8-fold e média diminuição da decadência de 4.1-fold (Figura 4B), corroborando porque apenas alterações transcricionais limitadas podem ser detectado em níveis de mRNA total(Figura 3). Nas dois outras mutantes cepas (bre1Δ e K123R), também foram observadas alterações concomitantes na síntese do mRNA e decadência, mas as mudanças foram em uma escala muito menor e mais dispersaram (Figura 4 e 4 D).
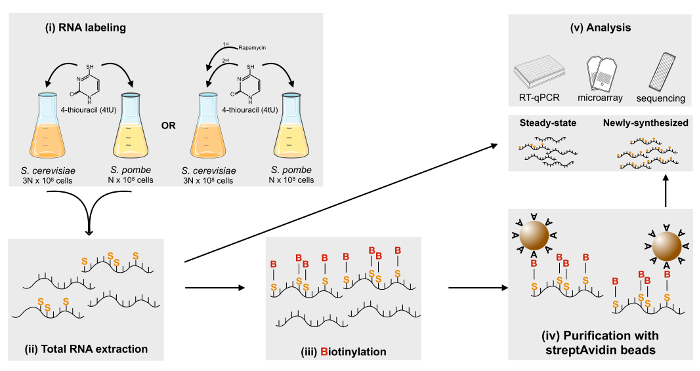
Figura 1 : Representação esquemática da rotulagem metabólica do RNA usando 4tU. 4tU recentemente preparada é adicionado ao meio de cultura e as células são rotuladas de 6 min. etiquetados S. cerevisiae e S. pombe células são misturadas em uma proporção de 3:1 e RNA total é extraído. Depois, o RNA recém sintetizado é biotinilado e pode ser purificado usando grânulos magnéticos streptavidin-revestido. Finalmente, total do RNA (estado estacionário) e rotulado recém sintetizada RNA pode ser usado em uma variedade de aplicações a jusante, incluindo sequenciamento ou hibridização RT-qPCR e microarray. Clique aqui para ver uma versão maior desta figura.
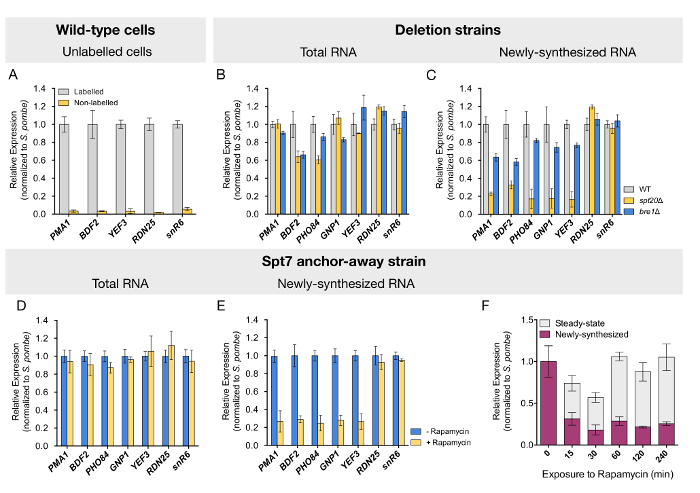
Figura 2 : Análise retratando alterações transcricionais em ambos estado estacionário e recém transcrito de RNA determinado por RT-qPCR. Níveis de (A), RNA de cinco diferentes genes foram quantificados da fracção etiquetada RNA purificada a partir de células de tipo selvagem que também foram expostas ou não a 4tU. Os próximos dois painéis mostram (B) total e (C) recém sintetizado quantificação de RNA por RT-qPCR para selvagem-tipo (WT), spt20Δ e células de levedura Δ de bre1. Spt7 cepas âncora-afastado, não tratada ou tratada com rapamicina por 60 min, foram rotuladas com 4tU e total (D) ou (E) recém transcrito de RNA foi quantificada por RT-qPCR. (F), este painel mostra que o tempo-curso de análise de mudanças no estado estacionário e recém sintetizado mRNA em cima Spt7 nuclear depleção. Para todas as amostras, os níveis do mRNA para cinco genes II de RNA polimerase foram quantificados por RT-qPCR. RNA polimerase I e genes de RNA polimerase III (RDN25 e snR6, respectivamente) foram utilizados como controle. Valores de expressão (média ± SD de três experimentos independentes) foram normalizados para o cravado em S. pombe de sinal e definido como 1 na amostra controle. Os painéis de D-F foram modificados de Baptista et al . 14. clique aqui para ver uma versão maior desta figura.
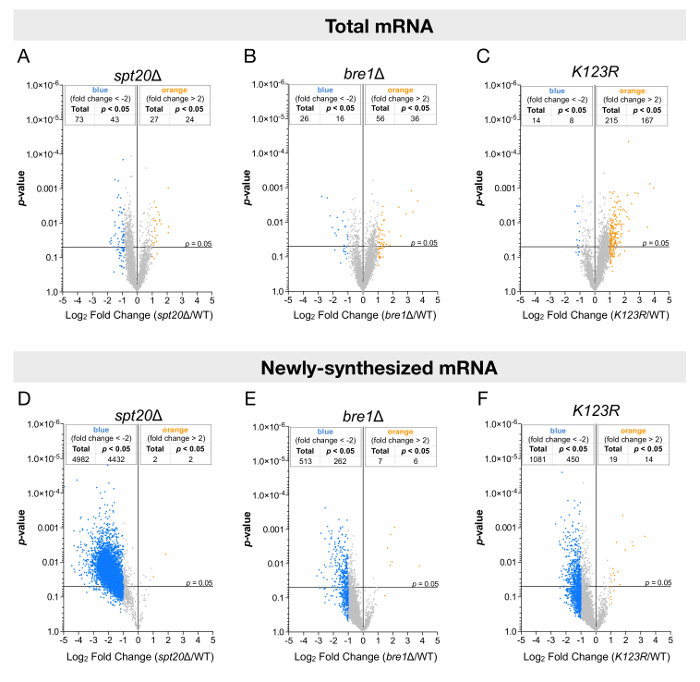
Figura 3 : Análises de todo o genoma dos níveis de mRNA usando frações de RNA totais ou rotuladas. Estes painéis mostram vulcão parcelas apresentando dobra as alterações nos níveis de RNAm de estado estacionário (A-C) ou (D-F) recém sintetizados os níveis do mRNA em relação ao seu significado (p-valor). As alterações de dobra (FC) foram calculadas como o log2 da relação entre o valor da expressão de cada gene após a normalização do sinal de S. pombe no Δ de spt20(A e D), Δ de bre1(B e E), ou ( C e F) K123R tensão contra o valor da expressão do gene da mesmo no selvagem-tipo S. cerevisiae. Um total de 5.385 genes foram analisados, e alterar os limites da dupla (ponto azul: mais do que uma dupla diminuir; amarelo pontos: mais do que um aumento de dupla) e 0,05 p-valores foram considerados. Os painéis A e D foram modificados de Baptista et al . 14. clique aqui para ver uma versão maior desta figura.
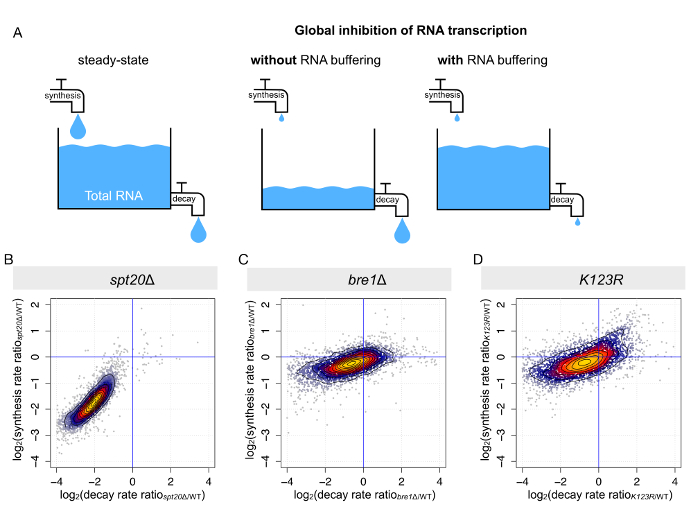
Figura 4 : Alterações na síntese do mRNA e resultados de decaimento do mRNA em mRNA buferização paralelo. (A), este painel mostra uma representação esquemática do resultado da perturbação de síntese de RNA em níveis de RNA do estado estacionário. (B-D) Estes painéis mostram o cálculo da síntese do mRNA e as taxas de deterioração das análises de RNA total e recém sintetizado. As taxas de síntese e degradação foram determinadas para cada transcrição de S. cerevisiae em Δ de spt20(B) e (C) bre1Δ K123R (D). (Calculadas como o log2 da proporção entre mutantes e selvagem-tipo) de alterações nas taxas de síntese foram plotadas contra alterações nas taxas de decomposição. Clique aqui para ver uma versão maior desta figura.
Discussão
Enquanto ainda estão a melhorar todo o genoma de ferramentas para analisar as alterações na transcrição, a análise exclusiva da transcriptoma através da quantificação dos níveis de estado estacionário de RNA pode não refletir com precisão as alterações na atividade do RNA polymerase II. Com efeito, os níveis do mRNA são regulados não só pela síntese do RNA, mas também pela sua maturação e degradação. Para medir a síntese de mRNA desacoplada da degradação do mRNA, protocolos distintos foram desenvolvidos nos últimos anos para a análise da transcrição nascente em levedura e mamíferos.
Um dos protocolos mais amplamente utilizados para a quantificação da transcrição nascente é GRO-seq1. Enquanto a principal vantagem do GRO-seq é sua capacidade de desvendar a polimerase transcricionalmente engajado e ativo em alta resolução e baixo fundo, ele tem dois pontos distintos que diminuem a sua atractividade: (i) tecnicamente é um desafio e requer a isolamento dos núcleos e manipulações de núcleos (ii), apresentar algumas perturbações para o sistema28. Outra alternativa é o NET-seq, que depende o sequenciamento das extremidades 3' dos transcrições obtidas mediante imunoprecipitação do complexo ternário extremamente estável formada entre RNA nascente, modelo DNA e RNA polimerase II. Esta abordagem permite a caracterização dos RNAs nascentes em uma resolução de pares de base e pode explorar eventos de processamento do mRNA através da imunoprecipitação de modificados RNA polimerase II (fosforilação da serina-5, por exemplo)2,3 , 29. no entanto, apresenta alguns obstáculos, da qual destacamos três. Em primeiro lugar, enquanto anticorpos direcionamento RNA polimerase II são geralmente altamente específicos, depende de eficiência de anticorpo. Em segundo lugar, RNA pode ser propenso a degradação durante os períodos de incubação, conduzindo-nos assim para o ponto anterior (menos eficientes anticorpos podem exigir mais tempo incubação vezes, deixando o RNA mais suscetíveis à degradação)29. Terceiro e especialmente em mamíferos, a digestão de MNase e seleção de tamanho podem excluir a sequência única alinhamento29,30.
Aqui apresentamos um protocolo detalhado para a rotulagem metabólica do RNA recém sintetizado usando 4tU em S. cerevisiae que tem diferentes vantagens em comparação com outras abordagens disponíveis. Porque a transcrição é altamente sensível a perturbações, as células devem ser mantidas em condições fisiológicas mais. Por exemplo, o GRO-seq implica a prisão de transcricionalmente engajado RNA polimerase II através da exposição dos núcleos/células de sarkosyl. No entanto, tratamento sarkosyl tem sido descrito como inibidor de vários processos celulares11. Neste caso, etiquetando metabólica com 4tU ou 4sU na concentração descrita é não-distorçer e visivelmente não afeta a homeostase celular, especialmente para períodos curtos de exposição. Em contraste com outros métodos usando inibição da transcrição para medir mRNA meias-vidas, CDT ou 4tU-seq e encaixe com modelagem dinâmica determina taxas de degradação de cada único mRNA em células imperturbável. Portanto, um único método simultaneamente pode abordar tanto a síntese e as taxas de decomposição da transcriptoma toda em um tipo específico de célula. Desde CDT ou 4tU-seq aproveita-se de métodos altamente confiável de normalização, nomeadamente através da utilização de espigão-no, diferentes conjuntos de dados podem ser analisados em conjunto e comparados diretamente. Finalmente, rotulagem metabólicos e quantificação do RNA recém sintetizado é uma técnica que pode ser facilmente implementada em qualquer laboratório de biologia molecular como não exige nenhum equipamento específico. Isto provavelmente explica a grande difusão desta técnica, que tende a ser usado cada vez mais sistematicamente para explorar a produção de RNA e degradação em leveduras, bem como em eucariontes superiores.
RNA pode ser rotulado em células vivas, usando outros análogos de nucleosídeos, ou seja,31,5-bromouridine (BrU)32 ou,5-ethynyluridine (UE)3334. O isolamento de BrU pulso-etiquetados RNA baseia-se na purificação do anticorpo anti-BrdU que pode ter diferentes eficiências entre experiências. EU-etiquetados RNA pode ser conjugado covalentemente a biotina usando clique química, levando a uma conjugação de irreversível em contraste com a modificação do tiol, que pode ser revertida com agentes redutores. BrU e EU rotulagem tem sido usados em células de mamíferos determinar todo o genoma RNA decadência taxas31,32 ou avaliar de34,de síntese de RNA nascente35. No entanto, BrU ou UE rotulagem não foi descrito no S. cerevisiae. Com efeito, absorção de análogos de nucleosídeos requer a expressão do transportador de nucleosídeos e, assim, esses métodos aparecem menos flexíveis em leveduras brotamento de rotulagem com 4tU.
A duração da 4tU rotulagem pode ser adaptada e varia de acordo com a pergunta. Quando avaliar a síntese de mRNA em S. cerevisiae, um curto pulso de rotulagem de 6 min garante que os níveis do mRNA recentemente transcrito são minimamente afetados pela degradação do RNA. Considerando que o tempo de atraso antes que o nucleotídeo modificado pode ser incorporado o RNA nascente é menos de 1 min, esta duração de rotulagem-6min garante que uma quantidade razoável de RNA recém sintetizado pode ser purificada. Tal protocolo, tal como definido por Miller et al . 11, tem sido usado em diferentes S. cerevisiae mutantes para revelar mudanças globais na síntese do mRNA e RNA decaem9,10,22,26,27, 36. uma duração mais rotulagem (3 h) foi usada em uma perseguição de pulso metabólica etiquetando com 4tU para determinar as taxas de decomposição de todos os mRNAs em S. cerevisiae12. Finalmente, extremamente curto (de 1,5 min) 4tU rotulagem tem sido usado para examinar cinética de processamento do RNA em brotamento e fissão fermento13,25,37.
No entanto, este método tem algumas limitações, tais como o rendimento relativamente baixo de etiquetados RNA recuperado no final do protocolo de todo. Enquanto no fermento, não há nenhuma limitação em matérias-primas, e isso pode ser um problema quando analisando células de metazoários, especificamente se este protocolo é ser utilizado em vivo. Uma das etapas limitantes do protocolo é biotinylation da piscina etiquetada RNA, cuja eficiência está longe de ser completa e foi estimada para modificar um em cada três 4sU resíduos em etiquetados RNA5. Recentemente mostrou que o uso de methanethiosulfonate (MTS)-biotina, em vez de biotina-HPDP, aumenta o rendimento do recuperado RNA38. No entanto, de acordo com resultados inéditos deste grupo de pesquisa e resultados de Rutkowski e Dölken, MTS-biotina não é totalmente tiol específicos, levando à purificação do RNA sem rótulo, que é particularmente problemáticas quando baixas quantidades de RNA etiquetado são purificada,39. Outra limitação a rotulagem metabólicos usando 4tU/4sU é a velocidade inerente no qual RNA polimerase alonga. A velocidade média da RNA polimerase II foi estimada em aproximadamente 3,5 kb/min40,41,42. Portanto, em uma rotulagem de 6 min, o polymerase terá capacidade para transcrever 15 – 20 kb. Assim, em um experimento com um pulso curto-etiquetando com 4tU/4sU, apenas a 3' terminar parte do RNA recém transcrito é rotulado, enquanto as regiões de extremidade 5' foram pre-existentes antes da adição de 4tU/4sU. Assim, a purificação de RNAs rotulados também enriquece pré-existentes 5' extremidades de transcrições, particularmente para transcrições longas como em células de mamíferos. A fim de superar este preconceito, em um refinado protocolo chamado TT-seq, o rotulado RNA extraído é fragmentado por sonication antes da purificação da espécie recentemente sintetizadas43.
No total, 4tU-seq é uma excelente maneira de endereço transcriptional alterações em um determinado contexto. No entanto, e enquanto o protocolo é bastante simples, consiste em várias etapas diferentes, sendo em última análise, relativamente extenso. Primeiro e acima de tudo, desde que este protocolo trata do RNA do início ao fim, é obrigatório que todos os requisitos necessários para uma amostra limpa e livre de degradação forem atendidos. Portanto, todos os reagentes devem ser RNase-livre, e todos os materiais devem ser dedicados para manipular RNA, limpeza tudo cuidadosamente e regularmente com solução de descontaminação de RNase. Em segundo lugar, enquanto a reação de biotina é altamente específica, o excesso de biotina-HPDP que não reagiu deve ser retirado da amostra (para evitar a saturação dos grânulos streptavidin com biotina, o que não é covalentemente ligado a RNA, por exemplo). Terceiro, conforme descrito no protocolo, o uso dos grânulos magnéticos streptavidin-revestido indicados é altamente recomendado, desde que os vários grupos temos de pesquisa falou que todas indicado que estes grânulos levaram a baixar os níveis de plano de fundo. Quarta, adequada qualidade e controles experimentais devem ser usados. Inclui amostras derivadas de células que não foram expostas a 4tU/4sU. Essas amostras serão de grande valia para os níveis de fundo endereço e para certificar-se que, durante o experimento, não ocorre uma contaminação com RNA não-rotulados. Além disso, é outra excelente alternativa para testar se a amostra é enriquecida por RNA recém sintetizado para executar um RT-qPCR usando as primeiras demão contra um gene contendo intro. Os primers devem ser complementares à extremidade 3' e 5' final de duas consecutivas exon e intrão ou vice-versa. Finalmente, antes do sequenciamento da hibridação de microarray, valide as amostras por RT-qPCR. Por isso, genes diferentes com diferentes níveis de expressão e regulamento devem ser selecionados e analisados. Idealmente, isto deve ser executado para as dois diferentes frações de RNA (RNA recém sintetizado e de estado estacionário).
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos a Laszlo Tora pelo seu apoio e V. Fisher, K. Schumacher e F. El Saafin para suas discussões. Tuberculose foi apoiado por uma bolsa Marie Curie-ITN (documento PITN-GA-2013-606806, NR-NET) e o arco de Fondation. Este trabalho foi financiado por fundos da Agence Nationale de la Recherche (SAGA2 ANR-15-CE11-0022). Este estudo foi suportado também por ANR-10-LABX-0030-INRT, um fundo de Estado francês, gerido pela Agence Nationale de la Recherche sob o quadro programa Investissements d'Avenir ANR-10-IDEX-0002-02.
Materiais
| Name | Company | Catalog Number | Comments |
| 4-Thiouracil | Sigma-Aldrich | Cat# 440736 | |
| Rapamycin | Euromedex | Cat# SYN-1185 | |
| Countess II FL Automated Cell Counter | ThermoFisher | N/A | |
| RiboPure RNA Purification kit, yeast | ThermoFisher | Cat# AM1926 | |
| NanoDrop 2000 Spectrophotometer | ThermoFisher | ND-2000 | |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | |
| EZ-Link HPDP Biotin | ThermoFisher | Cat# 21341 | |
| Thiolutin | Abcam | ab143556 | |
| µMACS Streptavidin kit | Miltenyi Biotec | Cat# 130-074-101 | |
| Transcriptor Reverse Transcriptase | Roche | 03 531 295 001 | |
| SYBR Green I Master | Roche | 4707516001 | |
| GeneChip Yeast Genome 2.0 | ThermoFisher | 900555 | |
| GeneChip Fluidics Station 450 | ThermoFisher | 00-0079 | |
| GeneChip Scanner 3000 7G | ThermoFisher | 00-0210 |
Referências
- Gardini, A. Global Run-On Sequencing (GRO-Seq). Methods in Molecular Biology. 1468, 111-120 (2017).
- Churchman, L. S., Weissman, J. S. Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature. 469 (7330), 368-373 (2011).
- Churchman, L. S., Weissman, J. S. Native elongating transcript sequencing (NET-seq). Current Protocols in Molecular Biology. , Chapter 4 Unit 4 11-17 (2012).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23 (2), 232-237 (2005).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14 (9), 1959-1972 (2008).
- Kenzelmann, M., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proceedings of the National Academy of Sciences of the United States of America. 104 (15), 6164-6169 (2007).
- Radle, B., et al. Metabolic labeling of newly transcribed RNA for high resolution gene expression profiling of RNA synthesis, processing and decay in cell culture. Journal of Visualized Experiments. (78), e50195(2013).
- Schwalb, B., et al. Measurement of genome-wide RNA synthesis and decay rates with Dynamic Transcriptome Analysis (DTA). Bioinformatics. 28 (6), 884-885 (2012).
- Sun, M., et al. Global analysis of eukaryotic mRNA degradation reveals Xrn1-dependent buffering of transcript levels. Molecular Cell. 52 (1), 52-62 (2013).
- Sun, M., et al. Comparative dynamic transcriptome analysis (cDTA) reveals mutual feedback between mRNA synthesis and degradation. Genome Research. 22 (7), 1350-1359 (2012).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Munchel, S. E., Shultzaberger, R. K., Takizawa, N., Weis, K. Dynamic profiling of mRNA turnover reveals gene-specific and system-wide regulation of mRNA decay. Molecular Biology of the Cell. 22 (15), 2787-2795 (2011).
- Eser, P., et al. Determinants of RNA metabolism in the Schizosaccharomyces pombe genome. Molecular Systems Biology. 12 (2), 857(2016).
- Baptista, T., et al. SAGA Is a General Cofactor for RNA Polymerase II Transcription. Molecular Cell. 68 (1), 130-143 (2017).
- Bonnet, J., et al. The SAGA coactivator complex acts on the whole transcribed genome and is required for RNA polymerase II transcription. Genes & Development. 28 (18), 1999-2012 (2014).
- Warfield, L., et al. Transcription of Nearly All Yeast RNA Polymerase II-Transcribed Genes Is Dependent on Transcription Factor TFIID. Molecular Cell. 68 (1), 118-129 (2017).
- Huisinga, K. L., Pugh, B. F. A genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA in Saccharomyces cerevisiae. Molecular Cell. 13 (4), 573-585 (2004).
- Lee, T. I., et al. Redundant roles for the TFIID and SAGA complexes in global transcription. Nature. 405 (6787), 701-704 (2000).
- Lenstra, T. L., et al. The specificity and topology of chromatin interaction pathways in yeast. Molecular Cell. 42 (4), 536-549 (2011).
- Plaschka, C., et al. Architecture of the RNA polymerase II-Mediator core initiation complex. Nature. 518 (7539), 376-380 (2015).
- Helenius, K., et al. Requirement of TFIIH kinase subunit Mat1 for RNA Pol II C-terminal domain Ser5 phosphorylation, transcription and mRNA turnover. Nucleic Acids Research. 39 (12), 5025-5035 (2011).
- Rodriguez-Molina, J. B., Tseng, S. C., Simonett, S. P., Taunton, J., Ansari, A. Z. Engineered Covalent Inactivation of TFIIH-Kinase Reveals an Elongation Checkpoint and Results in Widespread mRNA Stabilization. Molecular Cell. 63 (3), 433-444 (2016).
- Haimovich, G., et al. Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. Cell. 153 (5), 1000-1011 (2013).
- Haruki, H., Nishikawa, J., Laemmli, U. K. The anchor-away technique: rapid, conditional establishment of yeast mutant phenotypes. Molecular Cell. 31 (6), 925-932 (2008).
- Neymotin, B., Athanasiadou, R., Gresham, D. Determination of in vivo RNA kinetics using RATE-seq. RNA. 20 (10), 1645-1652 (2014).
- Shetty, A., et al. Spt5 Plays Vital Roles in the Control of Sense and Antisense Transcription Elongation. Molecular Cell. 66 (1), 77-88 (2017).
- Xu, Y., et al. Architecture of the RNA polymerase II-Paf1C-TFIIS transcription elongation complex. Nature Communications. 8, 15741(2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews. Genetics. 13 (10), 720-731 (2012).
- Nojima, T., Gomes, T., Carmo-Fonseca, M., Proudfoot, N. J. Mammalian NET-seq analysis defines nascent RNA profiles and associated RNA processing genome-wide. Nature Protocols. 11 (3), 413-428 (2016).
- Storvall, H., Ramskold, D., Sandberg, R. Efficient and comprehensive representation of uniqueness for next-generation sequencing by minimum unique length analyses. PloS One. 8 (1), 53822(2013).
- Tani, H., et al. Identification of hundreds of novel UPF1 target transcripts by direct determination of whole transcriptome stability. RNA Biology. 9 (11), 1370-1379 (2012).
- Tani, H., et al. Genome-wide determination of RNA stability reveals hundreds of short-lived noncoding transcripts in mammals. Genome Research. 22 (5), 947-956 (2012).
- Jao, C. Y., Salic, A. Exploring RNA transcription and turnover in vivo. by using click chemistry. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15779-15784 (2008).
- Palozola, K. C., et al. Mitotic transcription and waves of gene reactivation during mitotic exit. Science. 358 (6359), 119-122 (2017).
- Ardehali, M. B., et al. Polycomb Repressive Complex 2 Methylates Elongin A to Regulate Transcription. Molecular Cell. 68 (5), 872-884 (2017).
- Schulz, D., et al. Transcriptome surveillance by selective termination of noncoding RNA synthesis. Cell. 155 (5), 1075-1087 (2013).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Duffy, E. E., et al. Tracking Distinct RNA Populations Using Efficient and Reversible Covalent Chemistry. Molecular Cell. 59 (5), 858-866 (2015).
- Rutkowski, A. J., Dolken, L. High-Resolution Gene Expression Profiling of RNA Synthesis, Processing, and Decay by Metabolic Labeling of Newly Transcribed RNA Using 4-Thiouridine. Methods in Molecular Biology. 1507, 129-140 (2017).
- Darzacq, X., et al. In vivo dynamics of RNA polymerase II transcription. Nature Structural & Molecular Biology. 14 (9), 796-806 (2007).
- Fuchs, G., et al. Simultaneous measurement of genome-wide transcription elongation speeds and rates of RNA polymerase II transition into active elongation with 4sUDRB-seq. Nature Protocols. 10 (4), 605-618 (2015).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16 (11), 1128-1133 (2009).
- Schwalb, B., et al. TT-seq maps the human transient transcriptome. Science. 352 (6290), 1225-1228 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados