Method Article
Saccharomyces cerevisiae Beschriftung mit 4-Thiouracil und Quantifizierung der metabolischen synthetisiert neu mRNA als Proxy für RNA-Polymerase II Aktivität
In diesem Artikel
Zusammenfassung
Das hier beschriebene Protokoll basiert auf der genomweiten Quantifizierung der neugebildete mRNA von Hefezellen mit 4-Thiouracil beschriftet gereinigt. Diese Methode ermöglicht die Messung der mRNA-Synthese von mRNA Decay abgekoppelt und schafft somit eine genaue Messung der RNA-Polymerase II Transkription.
Zusammenfassung
Globale Mängel in der RNA-Polymerase II Transkription könnte durch transkriptomischen Studien analysieren stationären RNA überblickt werden. Der weltweite Rückgang der mRNA-Synthese ist in der Tat nachweislich durch eine gleichzeitige Abnahme der mRNA Abbau zu normalen stationären wiederherstellen kompensiert werden. Daher ist die genomweite Quantifizierung der mRNA-Synthese, unabhängig von mRNA Decay, die beste direkte Reflexion der RNA-Polymerase II transkriptionelle Aktivität. Hier diskutieren wir eine Methode verwenden, nicht störend metabolische Kennzeichnung der im Entstehen begriffenen RNAs in Saccharomyces Cerevisiae (S. Cerevisiae). Insbesondere die Zellen sind für 6 min. mit einem Uracil analog, 4-Thiouracil kultiviert, und die beschrifteten neu transkribierten RNAs sind gereinigt und quantifiziert, um die Synthese von allen einzelnen mRNA zu bestimmen. Darüber hinaus mit Schizosaccharomyces Pombe Zellen beschriftet, als interner Standard ermöglicht Vergleich der mRNA-Synthese in verschiedenen S. Cerevisiae Stämme. Mithilfe dieses Protokolls und Anpassung der Daten mit einem dynamischen kinetischen Modell, können die entsprechende mRNA Decay Tarife ermittelt werden.
Einleitung
Zellen reagieren auf endogene und exogene Signale durch die dynamische Veränderung ihrer Gen-Ausdruck-Programm. In den letzten Jahren ermöglicht eine enorme Entwicklung der genomweiten Methoden die genaue und umfassende Beschreibung der Transkriptom Änderungen unter verschiedenen Bedingungen. In den meisten transkriptomischen Studien Microarray-Hybridisierung oder Hochdurchsatz-Sequenzierung werden verwendet, um RNA Levels von einem gesamten stationären RNA Bruchteil zu quantifizieren. Transcriptional Änderungen unter einer bestimmten Störung können eine Vielzahl von Möglichkeiten, mit Veränderungen der spezifischen Genexpression oder ein großes Spektrum von Genen wird entweder auf- oder herunterreguliert anzeigen. Gen-Ausdrucks ergibt sich aus einem fein abgestimmten Gleichgewicht – oder stationären — zwischen RNS-Synthese von RNA-Polymerasen und andere Prozesse, die RNA-Ebene. RNA-Polymerase II Transkription, einschließlich seiner drei Phasen (Einleitung, Verlängerung und Kündigung) ist hoch und aufwendig mit mRNA Verarbeitung, zytoplasmatischen Export, Übersetzung und Abbau verbunden.
Mehrere neuere Studien gezeigt, dass mRNAs Synthese und Verfall gekoppelten Mechanismen und zeigte, dass transkriptionelle Effekte auf Mutation oder unter Reize bei der Quantifizierung der gesamten stationären RNA übersehen werden können. Erstens hängt die Erkennung von transcriptional Änderungen durch die Analysen der Steady-State Ebenen der mRNA immer mRNAs Halbwertszeit. Sobald die Störung eingeführt wird, werden die Steady-State Ebenen der mRNAs mit langen Halbwertszeiten viel weniger als die der mRNAs mit kurzen Halbwertszeiten betroffen sein. Daher ist die Nachweisbarkeit der Veränderungen in der RNS-Synthese stark zu Gunsten der kurzlebigen Transkripte, voreingenommen, während die Analyse der langlebigeren mRNA-Spezies Scheitern könnte, um dynamische Veränderungen der Transkription Rate zu offenbaren. Zweitens: mehrere Berichte haben gezeigt, dass sowohl in Hefe und Säugetiere, globale Veränderungen in der Transkription übersehen werden könnten, wenn die Steady-State Ebenen der mRNA zu analysieren. Dies ist wahrscheinlich auf die Mechanismen, die mRNA-Synthese und Degradation mRNA Pufferung zu verknüpfen. Dies veranlasste die Entwicklung neuer Protokolle, mRNA-Synthese, die losgelöst vom Abbau durch die Analyse der neu transkribierte mRNA zu quantifizieren. In den letzten Jahren wurden mehrere Alternativen vorgestellt, darunter globale Nachlauf Sequenzierung (GRO-Seq)1und native Dehnung Transkript Sequenzierung (NET-Seq)2,3. Hier präsentieren wir ein Protokoll zunächst in Säugerzellen4,5,6 entwickelt und dann an Hefe7,8,9,10, 11, basiert auf RNA, die Kennzeichnung mit einem Thiolated Nukleosid oder base Analog, 4-Thiouridine (4sU) oder 4-Thiouracil (4tU), beziehungsweise.
Diese Methode reinigt speziell neu transkribierten RNA aus den Zellen in die RNA mit 4sU mit praktisch keine Einmischung in die Zelle Homöostase Puls gekennzeichnet sind. Daher, sobald die Zellen 4sU ausgesetzt sind, ist das Molekül schnell Uptaken, um 4sU-Triphosphat phosphoryliert, und in RNA transkribiert wird. Sobald Puls-Label, es ist möglich, insgesamt zelluläre RNA (entsprechend Steady-State Ebenen der RNA) extrahieren und anschließend mit der Bezeichnung von 4sU RNA ist Thiol-speziell modifiziert, was zur Bildung einer Disulfid Bindung zwischen Biotin und die neu transkribierten RNA4,5. 4sU kann jedoch nur Uptaken durch die Zellen einen Nukleosid-Transporter, wie der menschliche equilibrative Nukleosid-Transporter (hENT1), verhindert den unmittelbaren Einsatz in Knospung oder Spaltung Hefe zum Ausdruck zu bringen. Während eines hENT1 in S. Pombe oder S. Cerevisiaeausdrücken könnte, kann ein einfacher Ansatz erreicht werden durch die modifizierte base 4tU da 4tU, ohne die Notwendigkeit des Ausdrucks eines Nukleosid-Transporter-10, Hefe-Zellen aufnehmen kann 11 , 12 , 13. In der Tat, der Stoffwechsel von 4tU erfordert die Aktivität des Enzyms Uracil-Phosphoribosyltransferase (UPRT). In einigen Organismen, einschließlich Hefe aber keine Säugetiere ist UPRT für eine Pyrimidine Salvage Pathway recycling Uracil, Uridin Monophosphate.
Eine wichtige Voreingenommenheit in transkriptomischen Studien kann durch die Normalisierung zwischen verschiedenen Proben analysiert parallel eingeführt werden. In der Tat können viele abweichende Faktoren beeinflussen die vergleichende Analyse des transkriptoms Mutanten und Wildtyp Stämme: die Effizienz der Zelle Lysis, Unterschiede in der Gewinnung und Verwertung von RNA und Abweichungen in der Scanner-Kalibrierung für Microarray Analysen , unter anderem. Wie oben besprochen, können solche Schwankungen besonders irreführend sein, wenn globale Auswirkungen auf RNA-Polymerase II Transkription zu erwarten sind. Eine eleganteste Mittel genau mRNA Synthese Preise zwischen verschiedenen Proben vergleichen wurde mithilfe der weitläufig verwandten Spalthefe Schizosaccharomyces Pombe als interner Standard entwickelt. Dafür eine feste Anzahl von S. Pombe beschriftet S. Cerevisiae Proben, Wildtyp oder mutierte Zellen vor der Zelle Lysis und RNA-Extraktion10Zellen hinzugefügt wird. Anschließend werden Steady-State und neugebildete RNAs von S. Pombe und S. Cerevisiae durch RT-qPCR oder über die Verwendung von Microarray-Chips oder Hochdurchsatz-Sequenzierung10quantifiziert. Zusammenführung dieser Daten mit kinetische Modellierung, können absolute Preise der mRNA-Synthese und des Verfalls in der angehenden Hefe gemessen werden.
Im Rahmen dieser Handschrift zeigen wir, wie die Analyse der neu transkribierten RNA erlaubt, um eine globale Rolle zu offenbaren, weil die Coactivator komplexe SAGA und TFIID RNA Polymerase II Transkription in angehende-14,15Hefe, 16. Wichtig ist, frühere Studien Steady-State mRNA-Niveaus in S. Cerevisiae quantifiziert und vorgeschlagen, dass SAGA spielt eine vorherrschende Funktion auf eine begrenzte Anzahl von Hefe Gene die stark betroffenen durch Mutationen im SAGA, aber relativ resistent gegenüber TFIID Mutationen,17,18,19. Überraschenderweise zeigten die SAGA enzymatischen Aktivitäten auf dem gesamten transkribierten Genom, was auf eine größere Rolle für diese Coaktivator in RNA Polymerase II Transkription handeln. Verminderte RNA Polymerase II Rekrutierung an am meisten exprimierten Genen wurde beobachtet, bei der Inaktivierung von SAGA oder TFIID, was darauf hindeutet, dass diese Koaktivatoren auf die meisten Gene zusammenarbeiten. Daher zeigte die Quantifizierung der neu transkribierte mRNA, SAGA und TFIID für die Transkription von fast allen Genen durch RNA-Polymerase II14,15,16erforderlich. Die Umsetzung der Kompensationsmechanismen entpuppt sich als eine Möglichkeit für die Zellen mit einem globalen Rückgang der mRNA-Synthese durch eine gleichzeitige weltweite Abnahme der mRNA Abbau gepuffert ist zu kämpfen. SAGA fügt in die Liste der Faktoren, die eine globale Auswirkung auf RNA Polymerase II Transkription, wie z. B. RNA Pol II Untereinheiten10, der Mediator Coactivator Komplex20, die allgemeine Transkription Faktor TFIIH21,22 , und indirekt, Elemente der mRNA Abbau Maschinen9,10,23. In SAGA Mutanten, Bilanzierung von bescheiden und begrenzte Änderungen im Steady-State mRNA-Niveaus trotz eines globalen und schweren Rückgangs der mRNA-Synthese-14wurden allgemein solche kompensatorische Ereignisse beobachtet. Ähnliche Untersuchungen wurden auch in einem BRE1 Löschung Stamm, was zu einem vollständigen Verlust des Histon H2B Ubiquitination durchgeführt. Interessanterweise konnte wesentlich milder, aber konsequente globale Auswirkungen auf RNA-Polymerase II Transkription nachgewiesen werden, bei fehlender Bre1, darauf hinweist, dass metabolische Kennzeichnung neu transkribierten RNA in Hefe erkennen und eine Vielzahl von Änderungen in mRNA zu quantifizieren Synthese-Preise.
Protokoll
(1) Zelle Kultivierung und Rapamycin Erschöpfung der SAGA-Untereinheit
- Für jede S. Cerevisiae -Stamm und replizieren impfen einschließlich der Wildtyp oder vergleichslinien, eine einzige Kolonie von einem frischen Teller auf 5 mL YPD Medium (2 % Pepton, Hefeextrakt 1 % und 2 % Glukose).
- S. Cerevisiae Zellen über Nacht bei 30 ° C mit ständiger Bewegung (150 u/min) wachsen.
- Messung die optische Dichte bei 600 nm (OD600) und verdünnen Sie die Kultur, die eine OD600 von ca. 0,1 in 100 mL YPD Medium und lassen es wachsen, bis die OD600 um 0,8 ist.
- Parallel, impfen eine einzige Kolonie von S. Pombe Zellen aus einem frischen Teller auf 50 mL ja Medium (0,5 % Hefeextrakt; 250 mg/L Adenin, Histidin, Uracil, Leucin und Lysin; 3 % Glukose) und wachsen die Zellen über Nacht bei 32 ° C mit ständiger Bewegung (150 u/min).
- Messen die OD600 S. Pombe über Nacht Kultur und verdünnen Sie die Kultur, die eine OD600 von ca. 0,1 in 500 mL ja Medium und lassen es wachsen, bis die OD600 ca. ist 0,8.
- Für jeden Anker-away S. Cerevisiae -Stamm und replizieren impfen einschließlich der Wildtyp oder vergleichslinien, eine einzige Kolonie von einem frischen Teller auf 5 mL YPD Medium (2 % Pepton, Hefeextrakt 1 % und 2 % Glukose).
- Wachsen Sie die Zellen über Nacht bei 30 ° C mit ständiger Bewegung (150 u/min).
- Am nächsten Morgen Messen Sie OD600, verdünnen Sie die Kultur, die eine OD600 von ca. 0,1 in 100 mL YPD Medium und lassen Sie es wachsen, bis die OD600≈ 0,8.
- Die Kultur von einer Stammlösung von 1 mg/mL (endgültige Rapamycin Konzentration von 1 µg/mL) 100 µL Rapamycin hinzu und lassen Sie die Zellen, die bei 30 ° C mit ständiger Bewegung für die notwendige Zeit für das Protein von Interesse für bedingt aufgebraucht sein, aus dem Zellkern (inkubieren in der Regel reicht 30 min). Verwenden Sie für das Steuerelement eine ähnliche Hefekultur, aber anstatt Rapamycin hinzuzufügen, fügen Sie die gleiche Menge an Dimethyl Sulfoxid (DMSO).
2. 4tU Etikettierung mit S. Pombe als Spike-in (zählen)
-
Bereiten Sie eine frische Lösung von 2 M 4-Thiouracil. Nachdem vorbereitet, halten Sie es bei Raumtemperatur und vor Licht.
- Genau wiegen Sie 64,1 mg 4-Thiouracil für jede Kultur S. Cerevisiae und lösen Sie es in 250 µL Dimethylformamid (DMF) oder 250 µL von DMSO.
- Für die S. Pombe Kultur als Spike-in verwendet werden wiegen 320,5 mg 4-Thiouracil und in 1.250 µL von DMSO auflösen.
- S. Cerevisiae und S. Pombe Kulturen für eine Endkonzentration von 5 mM 4-Thiouracil-Lösung hinzu und inkubieren sie 6 min mit ständiger Bewegung bei 30 ° C und 32 ° C, beziehungsweise.
- Entfernen Sie nach 6 min eine kleine Aliquot der jeweiligen Kultur für Zellzählung. Zählen Sie die Zellen mit einer automatischen Zelle Zähler oder eine Neubauer-Kammer.
- Sammeln der Zellen durch Zentrifugation (2.500 X g) für 5 min bei 4 ° C.
- Verwerfen Sie den überstand, waschen Sie die Zellen mit eiskalten 1 X PBS und Zentrifuge wieder (2.500 X g, 4 ° C, 5 min).
- Die Gesamtzahl der Zellen in jedem der S. Cerevisiae und S. Pombe Proben zu berechnen.
- Die Zellen in 5 mL eiskaltes 1 X PBS aufschwemmen und S. Cerevisiae mit S. Pombe Zellen mit einem Verhältnis von 3:1 mischen.
- Zentrifugieren der Zellen (2.500 x g, 4 ° C, 5 min), entfernen Sie die PBS Schockfrosten der Zellen in flüssigem N2und die Probe bei-80 ° C bis zur weiteren Verwendung zu speichern.
(3) RNA-Extraktion und DNase-Behandlung
- Auftauen der Zellen für ca. 20-30 min auf Eis.
- Fahren Sie mit der RNA-Extraktion mit einem Hefe-RNA-Extraktion-Kit (Table of Materials) mit ein paar Anpassungen.
- Pro jede Probe Gießen Sie 750 µL eiskalte Zirkonia Perlen in ein 1,5 mL Schraubverschluss-Röhrchen mit dem Kit geliefert. Denken Sie daran, dass pro jedes Rohr RNA aus bis zu 109 Zellen effizient extrahiert werden kann. Bereiten Sie daher die Anzahl der Rohre notwendig für jede Probe. Beispielsweise kann eine Kultur von S. Cerevisiae 100 ml (OD600≈ 0,8) ca. 2 x 10-9 , 3 x 10-9 -Zellen, im Vorfeld insgesamt 2,7 x 10 Rendern9 , 4 x 10-9 -Zellen insgesamt (nach dem Spike-in einem Drittel der S. Pombe Zellen). In diesem Fall wäre bis zu 3-4 Reaktionsgefäßen pro jede Probe/Zustand/Mutant/Replikation erforderlich.
- Fügen Sie pro jeweils 1 x 10-9 -Zellen 480 µL des Puffers Lyse zur Verfügung gestellt mit dem Kit, 48 µL 10 % SDS und 480 µL Phenol: Chloroform: Isoamyl Alkohol (25:24:1, V/V/V).
- Mischen Sie die Zellen mit einem Vortex-Mixer und übertragen Sie sie auf das Röhrchen mit der eiskalten Zirkonia Perlen.
- Platz für die Rohre auf einen Vortex-Mixer-Adapter, drehen Sie den Wirbel mit maximaler Geschwindigkeit und schlagen für 10 min in die Hefezellen (in einem Raum bei 4 ° C) zu lösen. Alternativ führen Sie Lyse der Zellen in eine automatische Wulst-Schläger.
- Zentrifugieren Sie Rohre bei 16.000 X g für 5 min bei Raumtemperatur und sammeln Sie sorgfältig die obere Phase (RNA-haltigen Phase) zu einem frischen 15 mL Falcon-Röhrchen. Das Volume wiederhergestellt pro jedes Rohr ist in der Regel um 500 – 600 µL.
- Die 15 mL-Röhrchen mit teilweise gereinigtes RNA, die Bindung Puffer zur Verfügung gestellt mit dem Kit und Mischung gründlich. Pro jeweils 100 µL RNA-Lösung hinzufügen 350 µL Puffer Bindung (d. h., wenn die wässrige Phase Lösung Volumen 600 µL, 2,1 mL des Puffers Bindung sollte hinzugefügt werden).
- Zu den vorherigen Mischung die 100 % Ethanol und mischen Sie gründlich. Pro jeweils 100 µL RNA-Lösung hinzufügen 235 µL 100 % Ethanol (d. h., wenn die wässrige Phase Lösung Volumen 600 µL addieren 1,41 mL Ethanol).
- Bewerben Sie bis zu 700 µL der Mischung aus Schritt 3,9 zu eine Filterpatrone montiert in einem Sammelrohr, beide mit dem Kit versehen.
- Zentrifuge für 1 min bei 16.000 X g. Wenn die Zentrifugation Dauer nicht genug für das Gesamtvolumen der Filter durchlaufen wurde, wiederholen Sie die Zentrifugation für 30 s.
- Die Flow-through zu verwerfen Sie und wiederverwenden Sie, die gleiche sammelröhrchen. Am Filter und Zentrifuge wieder bei 16.000 X g für 1 min eine weitere 700 µL RNA-bindende Puffer-Ethanol-Lösung hinzufügen.
- Verwerfen Sie der durchströmten und wiederholen Sie die Schritte 3.11 und 3.12 bis die RNA-Lösung abgeschlossen ist.
- Waschen Sie den Filter 2 X mit 700 µL Waschlösung 1. Sammeln Sie waschen Lösung per Zentrifugation bei 16.000 X g für 1 min zu und behalten Sie immer das sammelröhrchen.
- Waschen Sie den Filter 2 X mit 500 µL Waschlösung 2. Sammeln Sie waschen Lösung per Zentrifugation bei 16.000 X g für 1 min zu und behalten Sie immer das sammelröhrchen.
- Zentrifugieren Sie die Rohre wieder bei 16.000 X g für 1 min den Filter vollständig trocknen.
- Übertragen Sie die Filterpatrone auf dem endgültigen Sammelrohr (RNA-geeignete Röhre) und eluieren Sie RNA mit 50 µL DEPC-behandeltem, RNase-freie H2O (bis 100 ° C vorgewärmt).
- Zentrifuge für 1 min bei 16.000 X g.
- Eluieren Sie RNA wieder (mit dem gleichen Rohr) mit 50 µL vorgewärmten DEPC-behandeltem, RNase-freie H2O. Stellen Sie sicher, dass das Volumen des Filters durchlaufen hat; Zentrifugieren Sie andernfalls über einen längeren Zeitraum.
- Wenn mehrere Rohre für ein einzelnes Sample verwendet werden, bündeln sie alle in eine Röhre.
- Quantifizieren Sie und überprüfen Sie die Reinheit der Probe mit der entsprechenden Ausrüstung.
Hinweis: Während 4tU nur innerhalb neu synthetisierte RNA eingebaut wird, gibt es eine Chance, kleine Verunreinigungen mit DNA. Aus diesem Grund, es ist immer ratsam, die Proben mit DNase behandeln ich. Verwenden Sie die Reagenzien zur Verfügung gestellt mit dem RNA-Extraktion Kit (Table of Materials) nach den Empfehlungen des Herstellers.
4. Thiol-spezifische Biotinylierungen neu synthetisierte RNA
- Passen Sie die Konzentration der RNA mit Abschnitt 3 des Protokolls bis 2 mg/mL erzielt. Aliquoten 200 µg von Gesamt-RNS, für 10 min bei 60 ° C erhitzen und kühlen es sofort für 2 min auf Eis.
-
Hinzufügen der RNA aliquoten, Reagenzien, die in der folgenden Reihenfolge unten genannten: 600 µL DEPC-behandeltem, RNase-freie H2O, 100 µL biotinylierungen-Puffer (100 mM Tris-HCl [pH 7.5] und 10 mM EDTA, in DEPC-behandeltem, RNase-freie H2O) und 200 µL Biotin-HPDP aus einem Bestand von 1 mg/mL Biotin-HPDP in DMSO oder DMF.
- In einigen Situationen tendenziell die Biotin-HPDP-Lösung auszufällen, wahrscheinlich aufgrund seiner geringen Löslichkeit in Wasser. In diesem Fall erhöhen Sie die Lautstärke von DMSO/DMF bis zu 40 % das Reaktionsvolumen (RNS-Probe hinzufügen 400 µL DEPC-behandeltem H2O, 100 µL biotinylierungen-Puffer und 400 µL Biotin-HPDP aus einem Bestand von 0,5 mg/mL).
- Die Probe bei Raumtemperatur inkubieren und lichtgeschützt für 3 h, mit sanften Agitation.
- Nach der Inkubation der Rohre ein etwa gleiches Volumen von Chloroform hinzu und mischen Sie kräftig.
- Drehen Sie die Probe bei 13.000 X g für 5 min bei 4 ° C. Dieser Schritt erlaubt die Entfernung von überschüssigen Biotin, die nicht Biotinylate die RNA hat. Alternativ führen Sie diesen Schritt mit Phase-Lock Röhren (schwer). Dafür spin-down Phase-Lock Röhren für 1 min bei 13.000 X g, fügen Sie die RNA-Mischung und eine gleiche Menge von Chloroform, mischen sie kräftig und Zentrifugieren sie 13.000 x g für 5 min bei 4 ° C.
- Übertragen Sie die obere Phase in neuen 2 mL Röhrchen sorgfältig.
- Ein Zehntel des Volumens von 5 M NaCl und Mischen der Probenmaterials.
- Fügen Sie ein gleiches Volumen von Isopropanol, durchmischen der Probenmaterials und drehen es mit 13.000 X g für mindestens 30 min, bei 4 ° c
- Vorsichtig entfernen Sie den überstand zu und fügen Sie 1 mL eiskaltem 75 % Ethanol.
- Drehen Sie mit 13.000 X g für 10 min bei 4 ° C.
- Sorgfältig überstand, Quick-Spin den Schlauch entfernen Sie und die restlichen Ethanol-Lösung. Stellen Sie sicher, dass die RNA-Pellet nicht eintrocknet.
- Hängen Sie die RNA in 100 µL DEPC-behandeltem, RNase-freie H2O.
5. Reinigung der neugebildete Bruchteil von insgesamt und unbeschriftete RNA mit magnetischen Beads Streptavidin beschichteten
- Erhitzen Sie biotinylierte RNA für 10 min bei 65 ° C und dann kühlen Sie die Proben für 5 min auf Eis.
- Die biotinylierte RNA (bei einem Endvolumen von 200 µL) 100 µL Streptavidin beschichteten magnetische Beads hinzufügen. Insbesondere empfiehlt es sich, die Perlen, die in der Tabelle der Materialien, da nach Gesprächen mit anderen Labors, diese schien angegeben verwenden, um die konsistente und zuverlässige.
- Inkubieren Sie die Probe mit leichten Schütteln für 90 min bei Raumtemperatur.
- Legen Sie die Spalten mit dem Kit (Table of Materials) in den Magnetstativ zur Verfügung gestellt.
- Fügen Sie 900 µL Raumtemperatur waschen Puffer (100 mM Tris-HCl [pH 7.5], 10 mM EDTA, 1 M NaCl, und 0,1 % Tween 20 in DEPC-behandeltem, RNase-freie H2O) auf die Spalten (Pre-laufen und equilibrate).
- Perlen/RNA Mischung (200 µL) auf die Spalten anwenden.
- Der Durchfluss in 1,5 mL Röhrchen sammeln und wieder auf die gleiche magnetische Spalte anwenden. Bei Bedarf halten Sie diese durchströmten da es den unbeschrifteten RNA-Anteil darstellt.
- Waschen Sie die Spalten 5 X mit steigender Datenmengen Waschpuffer (600, 700, 800, 900 und 1.000 µL).
- Eluieren Sie die neugebildete RNA mit 200 µL 0.1 M DTT.
- Führen Sie eine zweite Elution, 3 min später mit ein gleiches Volumen von 0,1 M DTT.
- Nach eluierenden RNA, 0,1 Volumen von 3 M NaOAc (pH 5,2), 3 Bände von eiskalten 100 % Ethanol und 2 µL von 20 mg/mL Glykogen (RNA-Klasse) und lassen Sie die RNA Niederschlag über Nacht, bei-20 ° C.
- Wiederherstellen der RNS durch Zentrifugieren (13.000 X g für 10 min bei 4 ° C) und in 15 µL DEPC-behandeltem, RNase-freie H20 aufzuwirbeln. Der Anteil der beschriftet, Total RNA wird voraussichtlich rund 2 % - 4 % (in der Regel mehr am unteren Ende), ca. 2,0 µg neu synthetisierte RNA rendern. Diese Menge reicht mehrere qPCR-Experimente, sowie für Microarray/Sequenzierung Analysen machen.
(6) RT-qPCR Validierung der verschiedenen Fraktionen
- CDNA von 2 µg Gesamt-RNS oder 10 µL beschrifteten RNA mit zufälligen Hexamers und die Reverse Transkriptase Wahl, nach den Anweisungen des Herstellers (Table of Materials) zu synthetisieren.
- Verstärken Sie die cDNA durch Echtzeit-qPCR Standardprotokolls (Table of Materials).
Hinweis: Alle Beispiele sollten in dreifacher Ausfertigung von einem Minimum von zwei biologischen Wiederholungen ausgeführt werden. Korrigieren Sie alle Rohwerte für den Ausdruck von S. Pombe Tubulin.
(7) Microarray-Hybridisierung
- Hybridisieren RNA-Proben auf bevorzugte Microarray-Chips nach den Anweisungen des Herstellers (für dieses spezielle Protokoll Siehe Tabelle of Materials). Kurz, bereiten biotinylierte cRNA Ziele von 150 ng RNA mit dem Premier RNA Amplifikation Kit (Table of Materials), gemäß den Anweisungen des Herstellers. Hybridisieren Sie 4 mg fragmentierten cRNAs für 16 h bei 45 ° C und 60 u/min auf Microarray-Chips.
- Waschen Sie, Färben Sie, und Scannen Sie die Chips unter Verwendung der angegebenen Station und Scanner (Table of Materials). Extrahieren Sie die Rohdaten (CEL-Intensität-Dateien) aus den gescannten Bildern unter Verwendung der Befehlskonsole (AGCC, Version 4.1.2).
- Die CEL-Dateien mit Ausdruck Konsole Softwareversion 1.4.1 berechnen Sonde Signalintensitäten, mit den Statistiken basierende Algorithmen MAS 5.0 mit Standardeinstellungen und global-scaling-als Normalisierung Methode gesetzt weiter zu verarbeiten.
Hinweis: Die getrimmte mittlere Ziel Intensität jeder Chip wurde willkürlich auf 100 festgelegt. Durchführen Sie alle Experimente mit mindestens zwei unabhängige biologische Wiederholungen. Raw-Daten mit dem S. Pombe Signal zu normalisieren und Falte Veränderungen im gesamten und neu synthetisierte RNA-Ebene zu berechnen.
8. Daten Analyse mit einer bestehenden R-Pipeline
- Berechnen Sie Synthese und Verfall der Preise mit einer Rohrleitung und R/Bioconductor Paket öffentlich zugänglich, wie zuvor beschrieben8,10.
Ergebnisse
Bei der metabolischen Kennzeichnung neu transkribierten RNA mehrere Aspekte kontrolliert werden müssen: die Zeit und Effizienz die Kennzeichnung der Spike-Anteil, die Extraktion-Protokoll und die biotinylierungen Wirksamkeit (einschließlich Signal-Rausch-Verhältnis), unter anderem. Diese Bedingungen wurden ausgiebig und methodisch von anderen7,10,11gezeigt. Hier konzentrieren wir uns hauptsächlich auf die Auslegung und sofortige Analysen, die ausgeführt werden können, sobald die Proben durch RT-qPCR, Microarray oder Sequenzierung bearbeitet wurde. Die Analysen der verschiedenen mutierte Stämme zeigen die Macht der Methode zu erkennen, nicht nur einen dramatischen globalen Rückgang der mRNA-Synthese, wie im Fall von SAGA Mutant S. Cerevisiae Stämme, sondern auch eine sehr milde Reduktion der RNA-Polymerase II Aktivität auf Unterdrückung von Histon H2B Monoubiquitination.
Unsere Analysen der SAGA enzymatischen Aktivitäten vorgeschlagen, eine breite Rekrutierung Chromatin15, was nicht durch die Analyse der stationären mRNA-Niveaus in SAGA mutierte Stämme herabgesandt wurde. Da RNA Polymerase II Rekrutierung auf SAGA Inaktivierung beeinträchtigt wurde, beschlossen wir zu analysieren, ob mRNA Synthese Preise weltweit betroffen wären. Daher Wildtyp oder mutierte S. Cerevisiae -Stämmen 4tU lang 6 min., neu transkribierten RNAs beschriften ausgesetzt waren. Nach dem Mischen mit der Spitze in beschrifteten Zellen (S. Pombe) in einem Verhältnis von 3:1, Gesamt-RNS extrahiert wurde, und neu synthetisierte RNA biotinylierte war und nach dem Protokoll präsentierte hier, wie die in Abbildung 1dargestellte Chronogramm gereinigt. Beschriftete RNAs wurden gereinigt, von insgesamt 200 µg RNA, um sicherzustellen, dass die Menge des gereinigten Produktes für alle nachgelagerten Anwendungen ausreichen würde. Als erste und systematische Schritt vor einer genomweiten Analyse wurde neu synthetisierte RNA-Reinigung von RT-qPCR validiert. Gene wurden nach verschiedenen Parametern, einschließlich ein Maß an Ausdruck, regulatorische Signalwege und eine Abhängigkeit von verschiedenen RNA-Polymerasen ausgewählt.
Um zu bestätigen, dass dieses Protokoll speziell markierte RNA reinigt, quantifizieren wir die Ebenen der Transkripte in Bruchteilen von Wildtyp Zellen, die mit oder ohne 4tU gezüchtet wurden gereinigt. Vernachlässigbar Hintergrundkonzentrationen von der analysierten RNAs gefunden aus Zellen, die nicht 4tU ausgesetzt wurden (Abb. 2A). Neu transkribierten RNA Reinigung wurde weiter durch die beobachtete Anreicherung der Intron-haltigen ACT1 Pre-mRNA (Daten nicht gezeigt) validiert. Nach der Validierung der Qualität der Proben haben wir getestet, ob mRNA Synthese beim Löschen von SPT20, betroffen wären, die bekannt ist, um die SAGA aufwendige Montage zu stören. Wie von anderen berichtet, offenbart mRNA Quantifizierung durchgeführt auf insgesamt (stationären) RNA aus dem spt20-Δ-Stamm meist unveränderte oder leicht reduzierte Ebenen für die getesteten Gene (Abbildung 2B). Ähnliche Ergebnisse wurden für die Stämme für BRE1 (Abbildung 2B) gelöscht. Im Gegensatz dazu die Analyse der neu transkribierten RNA aus dem spt20-Δ-Stamm ergab einen dramatischen Rückgang der mRNA-Synthese durch drei-zu 5er-Kombi für alle getestet Gene (Abbildung 2C). Der Verlust von Bre1 führte zu ein diskreter, aber noch sichtbaren Rückgang der neugebildete mRNA-Niveaus für die untersuchten Gene (Abbildung 2C). In guter Übereinstimmung mit einer Rolle von SAGA und Bre1 in RNA Polymerase II Transkription, der Verlust von Spt20 oder Bre1 hatte keinen Einfluss auf den Ausdruck RDN25 oder snR6 Gene transkribiert von RNA-Polymerase I und RNA-Polymerase III, bzw. ( Abbildung 2C).
Allerdings zeigen Belastungen gelöscht für strukturelle Untereinheiten der SAGA Komplex, wie SPT7 oder SPT20, schwere langsam wachsenden Phänotypen, die für die beobachteten transcriptional Änderungen ausmachen können. Um unerwünschte Nebenwirkungen auszuschließen wir bedingt Spt7 vom Kern erschöpft, Stämme mit Anker-away S. Cerevisiae 14,24. Nach 60 min von Rapamycin Behandlung, vor der die Puls-Kennzeichnung mit 4tU (siehe Abbildung 1 für eine schematische Darstellung), neu transkribierte mRNA-Niveaus verringerten sich in ähnlichem Ausmaß wie die beobachtet in der Löschung Belastung (Abb. 2D und 2E ). Diese Analyse so bestätigt unsere frühere Ergebnisse und das Protokoll für dieses induzierbaren Erschöpfung System validiert. In einer Zeit-Kurs-Analyse, wo Zellen Rapamycin für eine Zeit überspannt von 0 bis 240 min ausgesetzt waren, wurde reduzierte Ausdruck unmittelbar nach 15 min Exposition auf das Medikament bewiesen. Noch interessanter ist, stationäre mRNA-Niveaus zunächst verringern tendenziell kehrte aber auf ein normales Niveau nach 60 min, Indiz dafür, dass ein Ausgleichsmechanismus in der Zwischenzeit (Abb. 2E) stattfindet.
Insgesamt erlaubt die Kennzeichnung und Quantifizierung der neugebildete RNA enthüllt neue regulatorische Rollen für den SAGA-Komplex. Das beschriebene Protokoll könnte auch moderate Auswirkungen auf die RNA-Polymerase II Aktivität zeigen und wurde erfolgreich angewandte, bedingte Erschöpfung Hefestämme.
Die downstream-Anwendungen für die gereinigten neu synthetisierte RNA gehört eine genomweite Quantifizierung der Transkripte mittels Microarray-Hybridisierung oder Sequenzierung (4tU-Seq). Hochdurchsatz-Sequenzierung quantitative, empfindlicher und informativ ist, kann Microarray-Hybridisierung sehr hilfreich sein, festzustellen, ob die globale mRNA-Niveaus verändert werden. In diesem Zusammenhang, wo Normalisierung ausschlaggebend ist, haben wir einen Spike-Organismus der Probe, die wir analysieren soll. Insbesondere haben wir S. Cerevisiae Zellen S. Pombe Zellen in einem Verhältnis von 3:1, beide zuvor ausgesetzt 4tU gemischt. Wenn der gereinigte, beschriftete RNAs Hochdurchsatz-Sequenzierung unterliegen, kann Standardbibliothek Vorbereitung Protokoll verwendet werden, am häufigsten nach Erschöpfung der ribosomalen RNA. Normalisierung zwischen 4tU-Seq-Daten aus verschiedenen Proben wurde durch Zugabe von durchgeführt, entweder mit der Bezeichnung RNA aus einer verschiedenen Arten (z.B. mischen S. Cerevisiae und S. Pombe Zellen als oben)22 oder in Vitro- transkribiert, Thiolated Spike-in RNA12,25,26,27. Handelsübliche Microarray-Chips enthalten Sonden für das ganze Transkriptom sowohl Kernspaltung als auch angehende Hefe, erlauben die Quantifizierung der mRNA von beiden Organismen in einem einzigen Experiment. Microarray Hybridisierungen wurden mit insgesamt und neu synthetisierte RNA durch RT-qPCR validiert, wie erwähnt (Wildtyp, spt20Δ und bre1Δ) durchgeführt. Darüber hinaus enthalten wir eine Sorte, die nicht Monoubiquitination von Histon H2B, durch Punktmutation Ubiquitinable Rückstände (K123R) unterstützt. Weil die genau gleichen Verhältnis von S. Pombe Zellen dienten in den verschiedenen Proben und Wiederholungen, es ist möglich, die Arrays linear skalieren Intensitäten damit die Gesamt- und beschrifteten S. Pombe Sonden weisen die gleiche mittlere Intensität. Diese Normalisierung oder Skalierung, mit einem Bioconductor -Paket entwickelt im Labor von Patrick Cramer durchgeführt werden kann und parallel in allen analysierten Proben, insgesamt genutzt und Brüche und Wildtyp und Mutanten Stämme8gekennzeichnet. Kurz gesagt, ist die Eingabe dieser Pipeline eine Excel-Datei mit den Sonden und deren Intensität (MAS5 oder RMA) für alle Proben und Brüche. Nach Ausschluss von Sonden, die außerhalb des Bereichs der Nachweisgrenze liegen, ist die Signalintensität skaliert, unter Berücksichtigung der Intensitätswerte der S. Pombe Sonden. Schließlich können Sie die Sonden die angehenden und Kernspaltung Hefe Genome, zuordnen endet mit einer Matrix mit den Ausdruck-Werten für den Spike-in normalisiert. Diese Werte können weitere werden (siehe nächsten Abschnitt) verarbeitet oder genutzt, da ist. Im folgenden Beispiel haben wir festgestellt, dass die Falte jede Niederschrift zwischen den Mutanten und Wildtyp Stamm ändern.
Die Analysen wurden für beide Fließgleichgewicht durchgeführt oder neu Ebenen der RNA synthetisiert und aufgetragen gegen ihre statistische Signifikanz (p-Wert) (Abbildung 3). In Übereinstimmung mit anderen Studien, wenn total RNA Ebenen analysiert wurden, nur wenige Gene ihren Ausdruck verändert, hatte entweder auf- oder herunterreguliert (Abb. 3A-3 C). Allerdings führte die Analyse neu transkribierten RNA zu auffallend unterschiedliche Schlussfolgerungen. Neu transkribierte mRNA-Niveaus von mehr als 4.000 Genen wurden deutlich reduziert durch mindestens zweifach beim Löschen des SPT20, was auf eine globale positive Wirkung von SAGA auf RNA Polymerase II Transkription in knospenden Hefen (Abbildung 3D ). Darüber hinaus in den bre1Δ und K123R -Mutanten, die Ergebnisse waren diskreter: die meisten Gene ihren Ausdruck reduziert zu haben schien, aber das Ausmaß der Downregulation und die Anzahl der stark betroffenen Gene (≈ 300-500) war in der Tat mehr Limited (Abbildung 3E und 3F).
Wie zuvor erwähnte, stationäre oder total Ebenen der mRNA durch die engen Gleichgewicht zwischen Synthese und Verfall (Abb. 4A) diktiert werden. Bei der RNA-Polymerase II Transkription weltweit beeinträchtigt ist, können zwei Szenarien abgebildet werden: (i) entweder die gesamte mRNA-Spiegel sinkt weltweit als Reaktion auf reduzierte Synthese aber ständigen Verfall, oder (Ii) mRNA Abbau wird im gleichen Maße verringert sich daraus ergebenden in meist unverändert Steady-State mRNA-Niveaus. Das zweite Szenario wurde für mehrere Bedingungen, einschließlich der im Zusammenhang mit SAGA oder TFIID Unterbrechung9,10,14,16,22berichtet. Eine Prozedur namens vergleichende dynamische Transkriptom-Analyse (cDTA) basierend auf 4tU Kennzeichnung und dynamische kinetische Modellierung ermöglicht es uns, mRNA-Synthese zu bestimmen und den Verfall Preise für jedes Protokoll8,10ableiten. Wieder einmal nutzten wir die Daten für die oben erwähnten Stämme (Wildtyp spt20Δ, bre1Δ und K123R). Wie erwartet, beim Löschen von SPT20, beobachteten wir eine gleichzeitige Abnahme der mRNA Synthese und Degradation Preise im Vergleich zu Wildtyp Stamm. In dieser Mutante der Entschädigung war fast optimal, mit durchschnittlich Rückgang der Synthese von 3.8-fold und eine durchschnittlichen Rückgang der Zerfall des 4.1-fold (Abbildung 4B), erhärten warum nur begrenzte transcriptional Änderungen werden könnte auf insgesamt mRNA-Niveaus(Abbildung 3)erkannt. In die beiden anderen mutierte Stämme (bre1Δ und K123R), damit einhergehende Veränderungen der mRNA-Synthese und Verfall wurden auch beobachtet, aber die Änderungen wurden auf einem viel kleineren Maßstab und mehr zerstreut (Abbildung 4 und 4 D).
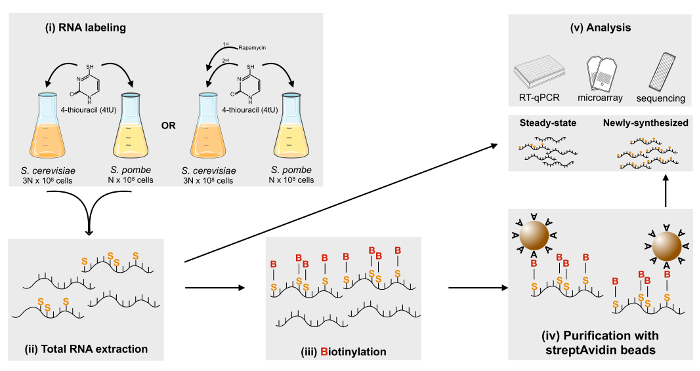
Abbildung 1 : Schematische Darstellung der metabolischen Beschriftung der RNA mit 4tU. Frisch zubereitete 4tU wird hinzugefügt, um das Nährmedium und Zellen sind für 6 min. etikettierte S. Cerevisiae beschriftet und S. Pombe Zellen werden in einem Verhältnis von 3:1 gemischt und Gesamt-RNS wird extrahiert. Danach neu synthetisiert RNA ist biotinylierte und mit magnetischen Beads Streptavidin beschichteten gereinigt werden kann. Schließlich, total RNA (stationären) und mit der Bezeichnung neu synthetisierte RNA kann in einer Vielzahl von downstream-Anwendungen, einschließlich RT-qPCR und Microarray-Hybridisierung oder Sequenzierung verwendet werden. Bitte klicken Sie hier für eine größere Version dieser Figur.
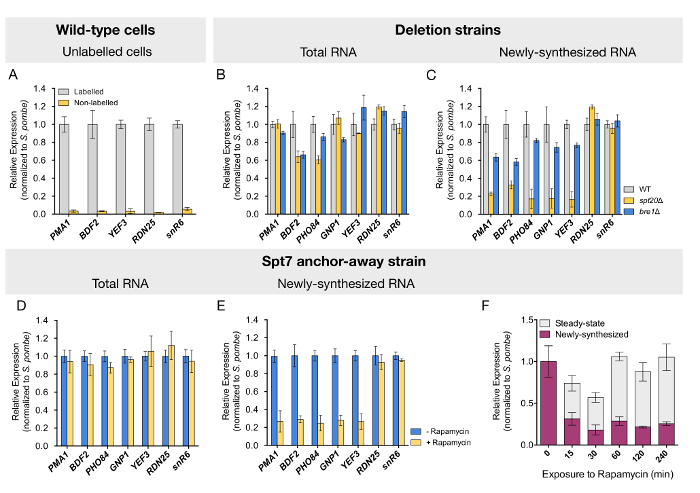
Abbildung 2 : Analyse, Darstellung transcriptional Änderungen in beiden stationären und neu transkribiert RNA durch RT-qPCR ermittelt. (A) RNA-Levels von fünf verschiedene Gene wurden aus der beschrifteten RNA-Fraktion gereinigt aus Wildtyp Zellen, die entweder ausgesetzt waren oder nicht 4tU quantifiziert. Die nächsten beiden Fenster zeigen (B) insgesamt und (C) neu synthetisiert Quantifizierung der RNA durch RT-qPCR für Wildtyp (WT), spt20Δ und bre1Δ Hefezellen. Spt7 Anker-away Stämme, unbehandelt oder mit Rapamycin für 60 min, behandelt mit 4tU beschriftet wurden, und (D) insgesamt oder (E) neu transkribiert RNA wurde durch RT-qPCR quantifiziert. (F) dieses Panel zeigt die Zeitverlauf Analyse der Veränderungen in den stationären und mRNA auf nukleare Erschöpfung Spt7 synthetisiert. Für alle Proben waren mRNA-Niveaus für fünf RNA Polymerase II Gene durch RT-qPCR quantifiziert. RNA-Polymerase I und RNA-Polymerase III Gene (RDN25 und snR6, beziehungsweise) dienten als Kontrolle. Ausdruckswerte (Mittelwert ± SD von drei unabhängigen Experimenten) wurden normalisiert, versetzt in S. Pombe signalisieren und 1 in der Kontrollprobe. Paneele D-F wurden von Baptista Et Al. geändert 14. Bitte klicken Sie hier für eine größere Version dieser Figur.
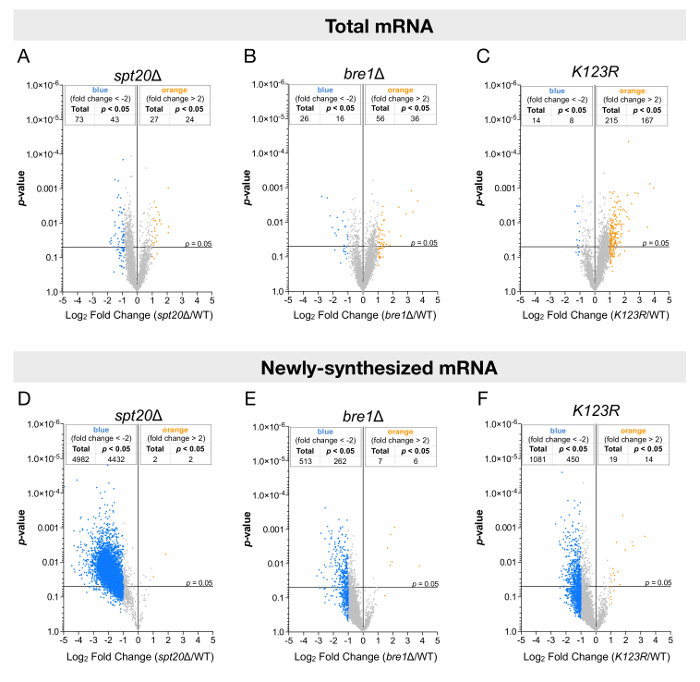
Abbildung 3 : Genomweite Analysen der mRNA-Niveaus mit insgesamt oder beschrifteten RNA-Fraktionen. Diese Tafeln zeigen Vulkan Grundstücke zeigen Falten Änderungen im Steady State-mRNA-Niveaus (A-C) oder (D-F) neu synthetisiert mRNA-Niveaus im Verhältnis zu ihrer Bedeutung (p-Wert). Die Falte Änderungen (FC) wurden als die log2 des Verhältnisses der Wert des Ausdrucks der jedes Gen nach Normalisierung der S. Pombe -Signal in der (A und D) spt20Δ, berechnet (B und E) bre1Δ oder () C und F) K123R Belastung im Vergleich zu den Ausdruckswert des gleichen Gens in Wildtyp S. Cerevisiae. Insgesamt 5.385 Gene wurden analysiert und Schwellen der doppelten ändern (blauer Punkt: mehr als ein zweifaches verringern; gelbe Punkte: mehr als eine Verdoppelung) und 0,05 p-Werte galten. Panels A und D wurden von Baptista Et Al. modifiziert 14. Bitte klicken Sie hier für eine größere Version dieser Figur.
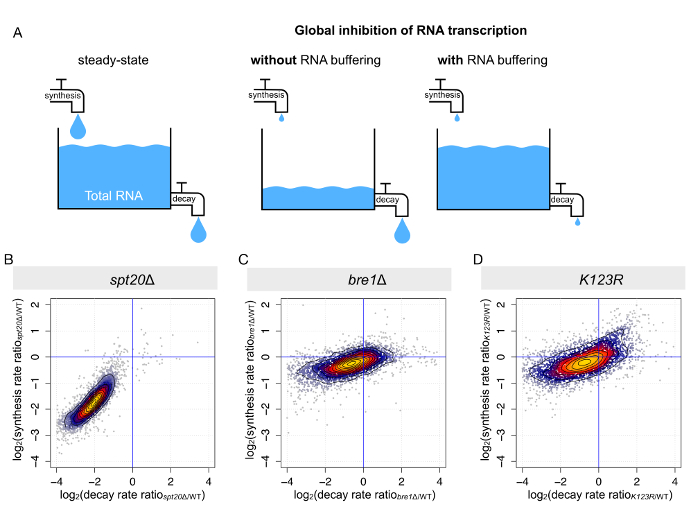
Abbildung 4 : Parallele Änderungen in der mRNA-Synthese und mRNA Decay Ergebnisse in mRNA Pufferung. (A) dieses Panel zeigt eine schematische Darstellung der Ergebnisse der RNA-Synthese-Störung auf stationären RNA Ebenen. (B-D) Diese Tafeln zeigen die Berechnung der mRNA-Synthese und der Zerfall Sätze aus den Analysen der Gesamt- und neu synthetisierte RNA. Die Synthese und Verfall Preise wurden für jedes Skript S. Cerevisiae (B) spt20Δ, (C) bre1Δ und (D) K123R bestimmt. Steuersatzänderungen (berechnet als die log2 des Verhältnisses zwischen Mutanten und Wildtyp) Synthese wurden gegen Zerfall Steuersatzänderungen geplottet. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Während genomweite Tools, um Veränderungen in der Transkription zu analysieren noch zu verbessern sind, könnte die einzige Analyse des transkriptoms durch die Quantifizierung der Steady-State Ebenen der RNA nicht Änderungen in RNA Polymerase II Aktivität widerspiegeln. MRNA-Niveaus sind in der Tat nicht nur durch die RNA-Synthese, sondern auch durch ihre Reifung und Abbau geregelt. Zur Messung der mRNA-Synthese von mRNA Abbau abgekoppelt wurden unterschiedliche Protokolle in den letzten Jahren für die Analyse der im Entstehen begriffenen Transkription in Hefe und Säugetieren entwickelt.
Eines der am häufigsten verwendeten Protokolle für die Quantifizierung der im Entstehen begriffenen Transkription ist GRO-Seq1. Während GRO-Seq Hauptvorteil seine Fähigkeit ist, transcriptionally engagierte und aktive Polymerase bei hoher Auflösung und niedrigen Hintergrund zu enthüllen, es hat zwei verschiedene Punkte, die ihre Attraktivität verringern: (i) Es ist technisch anspruchsvoll und erfordert die Isolierung von Kernen und (Ii) Kerne Manipulationen führen einige Störungen in der System-28. Eine weitere Alternative ist NET-Seq, die stützt sich auf die Sequenzierung der 3' Enden der Transkripte bezogen auf Immunopräzipitation des äußerst stabilen ternären komplexes zwischen entstehenden RNA, Schablone DNA und RNA-Polymerase II gebildet. Dieser Ansatz ermöglicht die Charakterisierung der im Entstehen begriffenen RNAs mit einer Auflösung von Basenpaaren und erkunden mRNA Verarbeitung von Ereignissen durch Immunopräzipitation der modifizierten RNA-Polymerase II (Serin-5 Phosphorylierung, zum Beispiel)2,3 , 29. Dennoch stellt es einige Hindernisse, von denen wir drei hervorheben. Erstens, während Antikörper gezielt RNA-Polymerase II in der Regel sehr spezifisch sind, kommt es auf Antikörper Effizienz. Zweitens, RNA möglicherweise anfällig für Abbau während Inkubationszeiten, so führt uns wieder auf den vorherigen Punkt (weniger effiziente Antikörper erfordern längere Inkubationszeit Zeiten, verlassen die RNA anfälliger für Abbau)29. Dritte und vor allem in Säugetieren, die MNase Verdauung und Größenauswahl können eindeutige Sequenz Ausrichtung29,30ausschließen.
Hier präsentierten wir ein detailliertes Protokoll zur metabolischen Kennzeichnung von neu synthetisierte RNA mit 4tU in S. Cerevisiae , die verschiedene Vorteile im Vergleich zu anderen verfügbaren Ansätze hat. Weil Transkription sehr empfindlich auf Störungen reagiert, sollten die Zellen in die meisten physiologischen Bedingungen beibehalten werden. GRO-Seq bedeutet zum Beispiel die Verhaftung von transcriptionally engagierte RNA-Polymerase II durch Einwirkung der Zellen/Kerne Sarkosyl. Jedoch wurde Sarkosyl Behandlung als hemmende verschiedene zelluläre Prozesse11beschrieben. In diesem Fall metabolische Markierung mit 4tU oder 4sU bei der beschriebenen Konzentration ist nicht störend und wirkt sich die Homöostase der Zelle, vor allem bei kurzzeitiger Exposition nicht sichtbar. Im Gegensatz zu anderen Methoden mit Transkription Hemmung zu mRNA Halbwertzeiten Messen bestimmt cDTA oder 4tU-Seq und Armatur mit dynamischen Modellierung Abbauraten von jeder einzelnen mRNA in unbeirrt Zellen. Daher kann eine einzige Methode gleichzeitig sowohl die Synthese und die Verwesung das ganze Transkriptom auf einem bestimmten Zelltyp adressieren. Da cDTA oder 4tU-Seq hochzuverlässige Normalisierung Methoden nutzt, nämlich durch den Einsatz von Spike-in können verschiedenen Datasets gemeinsam analysiert und direkt verglichen. Zu guter Letzt ist metabolische Kennzeichnung und Quantifikation der neu synthetisierte RNA eine Technik, die leicht in jedem molekularbiologischen Labor umgesetzt werden kann, da es keine keine spezielle Ausrüstung erforderlich ist. Das erklärt wahrscheinlich die recht große Verbreitung dieser Technik, die tendenziell mehr systematisch verwendet werden, um RNA Produktion und Abbau in Hefen sowie in höheren Eukaryoten zu erkunden.
RNA kann in lebenden Zellen mit anderen Nukleosid-Analoga, nämlich 5-Bromouridine (BrU)31,32 oder 5-Ethynyluridine (EU)33,34beschriftet werden. Die Isolation der BrU Puls-Label RNA stützt sich auf Anti-BrdU Antikörper Reinigung die unterschiedlichen Wirkungsgrade zwischen Experimente haben können. EU-Label RNA kann kovalent an Biotin mit Click-Chemie führt zu eine irreversible Konjugation im Gegensatz zu Thiol-Modifikation, die mit Reduktionsmitteln rückgängig gemacht werden kann konjugiert werden. BrU und EU Kennzeichnung in Säugetierzellen genomweiten RNA Verfall Preise31,32 bestimmen oder entstehende RNA Synthese34,35zu bewerten haben. Jedoch wurde BrU oder EU-Kennzeichnung nicht in S. Cerevisiaebeschrieben. In der Tat, Aufnahme von Nukleosid-Analoga erfordert die Expression von Nukleosid-Transporter und diese Methoden scheinen daher weniger flexibel in der angehenden Hefe als Beschriftung mit 4tU.
Die Dauer der 4tU Kennzeichnung kann angepasst werden und variiert je nach der Frage. Bei Beurteilung der mRNA-Synthese in S. Cerevisiae, ein kurzer Impuls der Kennzeichnung von 6 min sorgt dafür, dass die neu transkribierte mRNA-Niveaus minimal durch RNA-Abbau betroffen sind. Man bedenkt, dass die Verzögerungszeit bevor die modifizierte Nukleotid in die entstehende RNA integriert werden kann weniger als 1 min, sichert diese 6-minütiger Kennzeichnung Dauer zu, dass eine angemessene Menge an neugebildete RNA gereinigt werden kann. Solches Protokoll definiert durch Miller Et al. 11, wird in verschiedenen S. Cerevisiae Mutanten zu globale Veränderungen in der mRNA-Synthese und RNA offenbaren Verfall9,10,22,26,27, 36. eine mehr Kennzeichnung Dauer (3 h) diente in einer metabolischen Puls-Verfolgungsjagd Kennzeichnung mit 4tU Zerfall der alle mRNAs in S. Cerevisiae12. bestimmen Schließlich wurde extrem kurz (von 1,5 min) 4tU Kennzeichnung verwendet, RNA-Verarbeitung-Kinetik in angehende und Kernspaltung Hefe13,25,37zu untersuchen.
Diese Methode hat jedoch einige Einschränkungen, z. B. die relativ geringe Ausbeute an beschrifteten RNA, die am Ende des ganzen Protokolls wiederhergestellt. Während in der Hefe, keine Einschränkung im Ausgangsmaterial ist, und dies ein Problem sein kann, bei der Analyse der Zellen, insbesondere, wenn dieses Protokoll ist genutzt wird in Vivo. Ein limitierender Schritt des Protokolls ist biotinylierungen des beschrifteten RNA Pools, deren Effizienz ist bei weitem nicht vollständig und wurde schätzungsweise ein Drittel 4sU Rückstände in beschrifteten RNA5ändern. Es zeigte, dass vor kurzem die Verwendung von Methanethiosulfonate (MTS)-Biotin, anstelle von Biotin-HPDP, erhöht den Ertrag der wiederhergestellten RNA-38. Allerdings ist laut unveröffentlichte Ergebnisse aus dieser Forschungsgruppe und Ergebnisse von Rutkowski und Dölken, MTS-Biotin nicht vollständig Thiol-spezifisch, zu der Reinigung der unbeschrifteten RNA, die besonders problematisch, wenn geringe Mengen von beschrifteten RNA ist sind gereinigte39. Eine weitere Einschränkung der metabolischen Beschriftung mit 4tU/4sU ist die inhärente Geschwindigkeit an die RNA Polymerase verlängert. Die durchschnittliche Geschwindigkeit der RNA-Polymerase II war schätzungsweise ca. 3,5 kb/min40,41,42. Daher haben die Polymerase in ein 6 min Kennzeichnung, die Kapazität um 15 – 20 kb zu transkribieren. So in einem Experiment mit einem kurzen Puls-Etikettierung mit 4tU/4sU, nur die 3' Ende Teil neu transkribierten RNA bezeichnet, während die 5' Ende Regionen bereits vorhandene vor der Zugabe von 4tU/4sU waren. Die Reinigung des beschrifteten RNAs bereichert so auch bereits vorhandene 5' Enden der Transkripte, besonders für lange Transkripte in Säugerzellen. Um diese Verzerrung in einer raffinierten Protokoll namens TT-FF, zu überwinden ist der extrahierten RNA gekennzeichnet durch Ultraschallbehandlung vor der Reinigung der neugebildete Arten43fragmentiert.
4tU-Seq ist insgesamt eine hervorragende Möglichkeit um transcriptional Änderungen in einem bestimmten Kontext. Dennoch, und das Protokoll ist ziemlich einfach, es besteht aus mehreren verschiedenen Schritten, wird letztlich relativ umfangreich. Vor allem, da dieses Protokoll RNA von Anfang bis Ende behandelt, ist es zwingend erforderlich, dass alle Voraussetzungen für eine saubere und Abbau-freie Probe erfüllt sind. Daher sollten alle Reagenzien RNase-freie, und alle Materialien gewidmet sein, RNA, Reinigung alles gründlich und regelmäßig mit RNase Dekontaminationslösung zu manipulieren. Zweitens während Biotin Reaktion sehr spezifisch ist, sollte das Übermaß an Biotin-HPDP, die nicht reagierte aus der Probe (zur Vermeidung von der Sättigung des Streptavidin-Perlen mit Biotin, die nicht kovalent an RNA, z. B. gebunden ist) entfernt werden. Dritte, wie das Protokoll unter Verwendung der angegebenen Streptavidin beschichteten magnetische Beads wird dringend empfohlen, da die verschiedenen Forschungsgruppen wir haben gesprochen, alle haben darauf hingewiesen, dass diese Perlen geführt, um Hintergrund-Niveaus zu senken. Vierte, entsprechende Qualität und experimentelle Kontrollen sollten verwendet werden. Sind Proben abgeleitet aus Zellen, die 4tU/4sU nicht ausgesetzt waren. Diese Proben werden von großem Wert für Adresse Hintergrundkonzentrationen und um sicherzustellen, dass, während des Versuchs, eine Kontamination mit nicht-markierten RNA tritt nicht auf. Auch ist eine andere gute Alternative zu testen, ob die Probe für neu synthetisierte RNA angereichert ist eine RT-qPCR durchführen mit Primer gegen ein Intro-haltigen gen. Die Primer sollte ergänzend zu Ende 3' und 5'-Ende von zwei aufeinander folgenden Exon und Intron oder umgekehrt. Schließlich, vor der Sequenzierung von Microarray-Hybridisierung, überprüfen Sie die Proben von RT-qPCR. Hierzu sollten verschiedene Gene mit unterschiedlichem Ausdruck und Verordnung ausgewählt und analysiert werden. Im Idealfall sollte dies für zwei verschiedenen Fraktionen der RNA (Steady-State und neu synthetisierte RNA) durchgeführt werden.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken Laszlo Tora für seine Unterstützung und V. Fisher, K. Schumacher und F. El Saafin für ihre Gespräche. T.B. wurde unterstützt durch ein Stipendium der Marie Curie-ITN (PITN-GA-2013-606806, NR-NET) und der Fondation Bogen. Diese Arbeit wurde mit Mitteln der Agence Nationale De La Recherche (ANR-15-CE11-0022 SAGA2) unterstützt. Diese Studie wurde auch von ANR-10-LABX-0030-INRT, französische Staat von der Agence Nationale De La Recherche unter den Rahmen Programm Investissements Avenir ANR-10-IDEX-0002-02 verwalteten Fonds unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 4-Thiouracil | Sigma-Aldrich | Cat# 440736 | |
| Rapamycin | Euromedex | Cat# SYN-1185 | |
| Countess II FL Automated Cell Counter | ThermoFisher | N/A | |
| RiboPure RNA Purification kit, yeast | ThermoFisher | Cat# AM1926 | |
| NanoDrop 2000 Spectrophotometer | ThermoFisher | ND-2000 | |
| TURBO DNA-free Kit | ThermoFisher | AM1907 | |
| EZ-Link HPDP Biotin | ThermoFisher | Cat# 21341 | |
| Thiolutin | Abcam | ab143556 | |
| µMACS Streptavidin kit | Miltenyi Biotec | Cat# 130-074-101 | |
| Transcriptor Reverse Transcriptase | Roche | 03 531 295 001 | |
| SYBR Green I Master | Roche | 4707516001 | |
| GeneChip Yeast Genome 2.0 | ThermoFisher | 900555 | |
| GeneChip Fluidics Station 450 | ThermoFisher | 00-0079 | |
| GeneChip Scanner 3000 7G | ThermoFisher | 00-0210 |
Referenzen
- Gardini, A. Global Run-On Sequencing (GRO-Seq). Methods in Molecular Biology. 1468, 111-120 (2017).
- Churchman, L. S., Weissman, J. S. Nascent transcript sequencing visualizes transcription at nucleotide resolution. Nature. 469 (7330), 368-373 (2011).
- Churchman, L. S., Weissman, J. S. Native elongating transcript sequencing (NET-seq). Current Protocols in Molecular Biology. , Chapter 4 Unit 4 11-17 (2012).
- Cleary, M. D., Meiering, C. D., Jan, E., Guymon, R., Boothroyd, J. C. Biosynthetic labeling of RNA with uracil phosphoribosyltransferase allows cell-specific microarray analysis of mRNA synthesis and decay. Nature Biotechnology. 23 (2), 232-237 (2005).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14 (9), 1959-1972 (2008).
- Kenzelmann, M., et al. Microarray analysis of newly synthesized RNA in cells and animals. Proceedings of the National Academy of Sciences of the United States of America. 104 (15), 6164-6169 (2007).
- Radle, B., et al. Metabolic labeling of newly transcribed RNA for high resolution gene expression profiling of RNA synthesis, processing and decay in cell culture. Journal of Visualized Experiments. (78), e50195(2013).
- Schwalb, B., et al. Measurement of genome-wide RNA synthesis and decay rates with Dynamic Transcriptome Analysis (DTA). Bioinformatics. 28 (6), 884-885 (2012).
- Sun, M., et al. Global analysis of eukaryotic mRNA degradation reveals Xrn1-dependent buffering of transcript levels. Molecular Cell. 52 (1), 52-62 (2013).
- Sun, M., et al. Comparative dynamic transcriptome analysis (cDTA) reveals mutual feedback between mRNA synthesis and degradation. Genome Research. 22 (7), 1350-1359 (2012).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Munchel, S. E., Shultzaberger, R. K., Takizawa, N., Weis, K. Dynamic profiling of mRNA turnover reveals gene-specific and system-wide regulation of mRNA decay. Molecular Biology of the Cell. 22 (15), 2787-2795 (2011).
- Eser, P., et al. Determinants of RNA metabolism in the Schizosaccharomyces pombe genome. Molecular Systems Biology. 12 (2), 857(2016).
- Baptista, T., et al. SAGA Is a General Cofactor for RNA Polymerase II Transcription. Molecular Cell. 68 (1), 130-143 (2017).
- Bonnet, J., et al. The SAGA coactivator complex acts on the whole transcribed genome and is required for RNA polymerase II transcription. Genes & Development. 28 (18), 1999-2012 (2014).
- Warfield, L., et al. Transcription of Nearly All Yeast RNA Polymerase II-Transcribed Genes Is Dependent on Transcription Factor TFIID. Molecular Cell. 68 (1), 118-129 (2017).
- Huisinga, K. L., Pugh, B. F. A genome-wide housekeeping role for TFIID and a highly regulated stress-related role for SAGA in Saccharomyces cerevisiae. Molecular Cell. 13 (4), 573-585 (2004).
- Lee, T. I., et al. Redundant roles for the TFIID and SAGA complexes in global transcription. Nature. 405 (6787), 701-704 (2000).
- Lenstra, T. L., et al. The specificity and topology of chromatin interaction pathways in yeast. Molecular Cell. 42 (4), 536-549 (2011).
- Plaschka, C., et al. Architecture of the RNA polymerase II-Mediator core initiation complex. Nature. 518 (7539), 376-380 (2015).
- Helenius, K., et al. Requirement of TFIIH kinase subunit Mat1 for RNA Pol II C-terminal domain Ser5 phosphorylation, transcription and mRNA turnover. Nucleic Acids Research. 39 (12), 5025-5035 (2011).
- Rodriguez-Molina, J. B., Tseng, S. C., Simonett, S. P., Taunton, J., Ansari, A. Z. Engineered Covalent Inactivation of TFIIH-Kinase Reveals an Elongation Checkpoint and Results in Widespread mRNA Stabilization. Molecular Cell. 63 (3), 433-444 (2016).
- Haimovich, G., et al. Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. Cell. 153 (5), 1000-1011 (2013).
- Haruki, H., Nishikawa, J., Laemmli, U. K. The anchor-away technique: rapid, conditional establishment of yeast mutant phenotypes. Molecular Cell. 31 (6), 925-932 (2008).
- Neymotin, B., Athanasiadou, R., Gresham, D. Determination of in vivo RNA kinetics using RATE-seq. RNA. 20 (10), 1645-1652 (2014).
- Shetty, A., et al. Spt5 Plays Vital Roles in the Control of Sense and Antisense Transcription Elongation. Molecular Cell. 66 (1), 77-88 (2017).
- Xu, Y., et al. Architecture of the RNA polymerase II-Paf1C-TFIIS transcription elongation complex. Nature Communications. 8, 15741(2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews. Genetics. 13 (10), 720-731 (2012).
- Nojima, T., Gomes, T., Carmo-Fonseca, M., Proudfoot, N. J. Mammalian NET-seq analysis defines nascent RNA profiles and associated RNA processing genome-wide. Nature Protocols. 11 (3), 413-428 (2016).
- Storvall, H., Ramskold, D., Sandberg, R. Efficient and comprehensive representation of uniqueness for next-generation sequencing by minimum unique length analyses. PloS One. 8 (1), 53822(2013).
- Tani, H., et al. Identification of hundreds of novel UPF1 target transcripts by direct determination of whole transcriptome stability. RNA Biology. 9 (11), 1370-1379 (2012).
- Tani, H., et al. Genome-wide determination of RNA stability reveals hundreds of short-lived noncoding transcripts in mammals. Genome Research. 22 (5), 947-956 (2012).
- Jao, C. Y., Salic, A. Exploring RNA transcription and turnover in vivo. by using click chemistry. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15779-15784 (2008).
- Palozola, K. C., et al. Mitotic transcription and waves of gene reactivation during mitotic exit. Science. 358 (6359), 119-122 (2017).
- Ardehali, M. B., et al. Polycomb Repressive Complex 2 Methylates Elongin A to Regulate Transcription. Molecular Cell. 68 (5), 872-884 (2017).
- Schulz, D., et al. Transcriptome surveillance by selective termination of noncoding RNA synthesis. Cell. 155 (5), 1075-1087 (2013).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Duffy, E. E., et al. Tracking Distinct RNA Populations Using Efficient and Reversible Covalent Chemistry. Molecular Cell. 59 (5), 858-866 (2015).
- Rutkowski, A. J., Dolken, L. High-Resolution Gene Expression Profiling of RNA Synthesis, Processing, and Decay by Metabolic Labeling of Newly Transcribed RNA Using 4-Thiouridine. Methods in Molecular Biology. 1507, 129-140 (2017).
- Darzacq, X., et al. In vivo dynamics of RNA polymerase II transcription. Nature Structural & Molecular Biology. 14 (9), 796-806 (2007).
- Fuchs, G., et al. Simultaneous measurement of genome-wide transcription elongation speeds and rates of RNA polymerase II transition into active elongation with 4sUDRB-seq. Nature Protocols. 10 (4), 605-618 (2015).
- Singh, J., Padgett, R. A. Rates of in situ transcription and splicing in large human genes. Nature Structural & Molecular Biology. 16 (11), 1128-1133 (2009).
- Schwalb, B., et al. TT-seq maps the human transient transcriptome. Science. 352 (6290), 1225-1228 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten