Method Article
Interactome-Seq: Протокол для строительства Domainome Библиотека, проверки и отбора Фаговые отображения и следующего поколения последовательности
В этой статье
Резюме
Протоколы, описанные разрешить строительство, характеристика и выбор (против целевого выбора) из любого источника ДНК библиотеки «domainome». Это достигается путем исследования трубопровода, который сочетает в себе различные технологии: фага дисплей, складной репортером и следующего поколения последовательности с веб-инструмент для анализа данных.
Аннотация
Складной журналисты являются белки с легко идентифицировать фенотипы, например антибиотикам, складные и функции которого нарушается, когда сливается с плохо складывания белки или случайных открытом чтения фреймов. Мы разработали стратегию, где, используя ТЭМ-1 β-лактамаз (фермент, присвоении ампициллин сопротивления) в геномной масштабе, мы можем выбрать коллекции правильно сложенный протеин доменов из кодирования части ДНК любой intronless генома. Фрагменты белка, полученные на этот подход, так называемые «domainome», будет хорошо выражена и растворимые, что делает их пригодными для структурно функциональных исследований.
Клонирование и отображение «domainome» непосредственно в систему отображения фага, мы показали, что это можно выбрать конкретные белка домены с желаемой привязки свойств (например, другие белки или антитела), обеспечивая тем самым важным экспериментальной информации для идентификации генов аннотации или антигена.
Выявление наиболее обогащенного клонов в выбранной поликлональных населения может быть достиган с использованием Роман секвенирование нового поколения технологий (НГС). По этим причинам мы представляем глубокую последовательности анализа самой библиотеки и выбора мероприятий предоставлять полную информацию о разнообразии, изобилия и точное сопоставление каждого выбранного фрагмента. Протоколы, представленные здесь показать основные шаги для строительства библиотеки, характеристик и проверки.
Введение
Здесь мы описываем метод высокой пропускной способностью для строительства и выбор библиотек складывается и растворимого белка доменов из любого источника, генной/геномной начиная. Этот подход сочетает в себе три различные технологии: фага дисплей, использование складной репортером и следующего поколения последовательности (НГС) с конкретным веб-инструмент для анализа данных. Методы могут использоваться во многих различных контекстах белка на основе исследований, для идентификации и аннотации новых доменов белков/белков, характеристика структурных и функциональных свойств известных белков, а также определение белок взаимодействие сети.
Многие открытые вопросы по-прежнему присутствуют в исследованиях на основе белков и разработки методов для оптимального белок производства является важной потребность в нескольких областях расследования. Например несмотря на наличие тысяч геномов прокариот и эукариот1, соответствующий карта относительной протеомов аннотацию прямого закодированного белков и пептидов отсутствует до сих пор подавляющее большинство организмов. Каталог полный протеомов становится сложной цели, требующие огромных усилий в плане времени и ресурсов. Золотой стандарт для экспериментальных заметки остается клонирования все открытые рамки считывания (ORFs) генома, строительство так называемой «ORFeome». Обычно функции гена назначается на основании гомологии связанных генов известных деятельности, но этот подход является недостаточно точной из-за наличия многих неправильные аннотации в справочных баз данных2,3,4, 5. Кроме того, даже для белков, которые были выявлены и комментариями, дополнительные исследования необходимы для достижения характеристика с точки зрения обилия, выражений шаблонов в разных контекстах, включая структурные и функциональные свойства, а также взаимодействие сетей.
Кроме того поскольку белки состоят из разных доменов, каждый из них показаны специфические особенности и вклад в по-разному функций белков, изучение и точное определение этих доменов может позволить более полную картину, как на сингл гена и на уровне полного генома. Все необходимая информация делает исследования белков на широкой и сложной области.
В этой перспективе важный вклад могут предоставляться на беспристрастной и высок объём методы для производства белка. Однако успех таких подходов, рядом значительных инвестиций, необходимых, опирается на способность производить растворимый/стабильный белка конструкции. Это основные ограничения фактор, так как было подсчитано, что только около 30% белков может успешно выразил и производится на достаточных уровнях экспериментально полезным6,,78. Подход для преодоления этого ограничения основан на использовании случайно фрагментированных ДНК, чтобы произвести различных полипептидов, которые вместе обеспечивают перекрывающихся фрагментов представление отдельных генов. Только небольшой процент случайных фрагментов ДНК являются функциональные ORFs, хотя подавляющее большинство из них являются нефункциональные (благодаря наличию стоп-кодонов внутри их последовательностей) или кодировать для неестественное (ORF в кадре оригинал) полипептиды с биологического смысла.
Для решения всех этих вопросов, наша группа разработала высок объём белка выражение и взаимодействие анализ платформу, которая может быть использована на геномной масштаб9,10,и11,12. Эта платформа включает следующие методики: 1) метод для выбора коллекции правильно сложенный протеин доменов из кодирования части ДНК от любого организма; 2 технология отображения фага для выбора партнеров взаимодействия; 3) NGS полностью характеризовать весь interactome стадии изучения и выявления клонов интерес; и 4) веб-инструмент для анализа данных для пользователей без каких-либо биоинформатики или навыков программирования для выполнения анализа Interactome-Seq в простым и удобным способом.
Использование этой платформы обеспечивает важные преимущества по сравнению альтернативных стратегий расследования; Прежде всего этот метод полностью беспристрастной, высокой пропускной способности и модульные для исследования, начиная от одного гена до всего генома. Первый шаг конвейера является создание библиотеки от случайно фрагментированных ДНК исследуемых, который характеризуется затем глубоко NGS. Эта библиотека генерируется с использованием вектора инженерии где генов/фрагменты интерес клонируются между последовательность сигнала для секреции белков в Периплазматическое пространство (т.е., лидер Sec) и гена β-лактамаз ТЭМ1. Синтез белка будет наделять ампициллин сопротивления и способность выжить под давлением ампициллин, только в том случае, если клонированные фрагменты в кадр с этими элементами и результате синтез белка является правильно сложенные10,13 ,14. Все клоны спасли после антибиотика выбора, так называемые «фильтруют клонов», ORFs, подавляющее большинство из них (более 80%) и являются производными от реальных генов9. Кроме того сила этой стратегии заключается в выводах, что все клоны фильтрации ORF кодирования для правильно сложить/растворимые белки/домены15. Как много клонов, присутствует в библиотеке и картирования в том же регионе или домена, имеют разные начальные и конечные точки, это позволяет беспристрастной, единый этапа идентификации минимальная фрагментов, которые могут привести к растворимых продуктов.
Дальнейшее совершенствование технологии предоставляется с помощью NGS характеризуют библиотеки. Сочетание этой платформы и конкретных веб-инструмент для анализа данных дает важные объективную информацию на точное нуклеотидных последовательностей и расположение выбранных ORFs на ссылку ДНК исследуемых без необходимости дальнейших обширных анализов или экспериментальные работы.
Domainome библиотеки могут передаваться в контекст выделения и используется в качестве универсального инструмента для выполнения функциональных исследований. Высок объём белка выражение и взаимодействие анализ платформы что мы интегрированы и что мы называли Interactome-Seq использует преимущества технологий отображения Фаговые путем передачи фильтрованного ORF в phagemid вектор и создания ФАГ ORF Библиотека. После повторного клонирован в контекст отображения фага, белка, домены отображаются на поверхности частиц M13; Таким образом domainome библиотек можно непосредственно выбрать для фрагментов гена кодирования домены с конкретными ферментативную деятельность или привязки свойств, позволяя interactome сети профилирования. Этот подход был первоначально описан Zacchi соавт. 16 и позднее используется в нескольких других контексте13,17,18.
По сравнению с другими технологиями, используется для изучения взаимодействия протеин протеина (включая дрожжей два гибридной системы и масс-спектрометрии19,20), одно из главных преимуществ является усиление связывание партнера, что происходит во время ФАГ Отображение нескольких раундов выделения. Это увеличивает выбор чувствительности, таким образом позволяя выявление низкой обильные связывания белков доменов присутствует в библиотеке. Эффективность отбора выступал с ORF-фильтрации библиотеки далее увеличивается из-за отсутствия нефункциональные клонов. Наконец технология позволяет выбор выполняться против белков и не протеинового приманки21,,2223,24,25.
Фаговые выборки с использованием библиотеки domainome ФАГ может производиться с использованием антител, исходя из сыворотки больных с различных патологических состояний, например аутоиммунных заболеваний13, рака или инфекции заболеваний как приманку. Этот подход используется для получения так называемого «антитело подпись» болезни под исследования, позволяющие массово идентифицировать и охарактеризовать антигены/epitopes конкретно признаны больных антител в то же время. По сравнению с другими методами использование фага дисплей позволяет идентификации как линейной, так и конформационные антигенных эпитопам. Выявление конкретных подписи потенциально может иметь важное значение для понимания патогенеза, новый дизайн вакцины, выявление новых терапевтических целей и развития новых и конкретных инструментов, диагностические и прогностические. Кроме того когда исследование сосредоточено на инфекционные заболевания, основным преимуществом является, что открытие иммуногенных белков зависит от выращивания патогена.
Наш подход подтверждает, что складной журналистам может использоваться в геномной масштабе для выбора «domainome»: коллекция правильно сложенные, хорошо выраженной, растворимые белки доменов от кодирования части ДНК или cDNA от любого организма. Когда изолированные фрагменты белка полезны для многих целей, предоставляя основные экспериментальной информации для гена аннотации, а что касается структурных исследований, антитела epitope сопоставления, выявления антигена, и т.д. Полнота высок объём данных, предоставленных NGS позволяет анализ сложных образцов, таких как Фаговые отображения библиотек и имеет потенциал, чтобы обойти традиционной трудоемкой собирание и тестирование отдельных Фаговые спасли клонов.
В то же время благодаря функции отфильтрованных библиотеки и крайняя чувствительность и мощности NGS анализа можно определить ответственность каждого взаимодействия непосредственно на начальном экране, без необходимости создания домена протеина дополнительные библиотеки для каждого неизбежно белка. NGS позволяет получить всеобъемлющее определение всей domainome любого генно/геномной начиная источника и данных анализа веб-инструмент позволяет получение весьма специфические характеристики с качественной и количественной точки зрения interactome белки доменов.
протокол
1. Строительство ORF библиотеки (рис. 1)
- Подготовка Вставка ДНК
-
Подготовка фрагментов из синтетических или геномной ДНК
- Экстракт/очищают ДНК, используя стандартные методы26.
- Фрагмент ДНК в sonication. Если с помощью стандартного sonicator, как общее предложение начать с 30 s импульсов на 100% выходной мощности.
Примечание: Пилот экспериментов должно быть сделано с различной мощности и sonication раз, чтобы задать оптимальные условия для подготовки ДНК. После каждого испытания определяют размер фрагментов ДНК электрофорезом геля агарозы. - Загрузите sonicated ДНК на 1,5% агарозном геле, вместе с 100 лестница ДНК bp. Выполните короткий электрофорез на 5 V/см 15 мин и вырезать часть гель, содержащий мазок фрагментированных ДНК.
- Очищение дна вставки с комплектом извлечения геля на основе столбцов и измерять концентрацию, используя Спектрофотометр UV.
Примечание: по крайней мере 500 нг очищенный вставок должны быть получены после этого шага, чтобы быть лигируют с 1 мкг переваривается вектора, как описано в шаге 1.3. Проверьте качество подготовки фрагментов, оценки260nm/A280nm и коэффициенты 230nm/a260nm, поскольку низкого качества образца будут влиять на эффективность перевязки. - Лечить до 5 мкг вставок с 1 мкл быстрый притупления комплект фермента смеси, согласно инструкциям производителя. Инактивации ферментов нагреванием при 70 ° C, за 10 мин образцы можно хранить при температуре-20 ° C до использования.
-
Подготовка фрагментов из cDNA
- Извлечь РНК с стандартными методами (например, с помощью Тризол или аналогичных реагенты).
- Фрагмент мРНК при нагревании до выполнения обратной транскрипции. Окончательный длина фрагмента ДНК контролируется мРНК кипения времени и концентрации случайного праймера. Например тепла образец 6 мин на 95 ° C.
- Подготовьте cDNA, с использованием случайных праймеров с любых имеющихся комплекта после производителя протокол.
- Разрушающим cDNA поли dT хвосты по гибридизации с биотинилированным поли да за 3 ч при 37 ° C и отделить на стрептавидина магнитные шарики, как описано в Carninci et al. 13
- Восстановить свободный материал и очистки на основе столбцов ДНК очистки комплект следуя инструкциям производителя. Измерьте концентрацию, используя Спектрофотометр UV. Смотрите примечание в шаге 1.1.1.4.
-
Подготовка фрагментов из синтетических или геномной ДНК
- Подготовка фильтрации вектора
- Digest 5 мкг очищенный клонирования вектор pFILTER312 с 10 U EcoRV энзима ограничения, следующие производителя протокол.
- Загрузка 2 мкл (200 нг) переваривается вектора, вместе с 100 нг непереваренных вектора и 1 k bp молекулярных маркеров, на геле агарозы 1%, для проверки правильного пищеварения. Тепла инактивирует энзима ограничения.
- Добавить 1/10 объема 10 x буфер фосфатазы и 1 мкл (5 ед) фосфатазы и инкубировать при 37 ° C на 15 мин тепловой инактивации за 5 мин на 65 ° C.
- Очистить переваривается плазмиды путем извлечения из геля агарозы и измерять концентрацию, используя Спектрофотометр UV. Образцы можно хранить при температуре от-20 ° C до использования.
- Перевязка и преобразования
- Выполняют лигирование следующим: 1 мкг переваривается плазмида добавить 400 нг фосфорилированных вставок (плазмида: Вставка молярное соотношение 1:5), 10 мкл 10 x буфер для T4 ДНК лигаза, 2 мкл высокой концентрации Т4 ДНК лигаза в окончательный объем 100 мкл. Инкубируйте реакции на густыми 16 ° C GHT. Тепла инактивирует при 65 ° C для 10 мин.
- Осадок продукт перешнуровки, добавив 1/10 объема раствора ацетата натрия (3 M, рН 5.2) и 2,5 томов 100% этанола. Смешать и заморозить при температуре-80 ° C для 20 мин.
- Центрифуга для максимальной скоростью 20 мин при 4 ° C. Выбросите супернатант.
- 500 мкл холодный 70% этанола в гранулах и центрифуги на максимальной скорости на 20 мин при 4 ° C. Выбросите супернатант.
- Воздух сухой гранулы. Ресуспензируйте осажденный ДНК в 10 мкл воды.
- Выполняйте электропорации бактериальной клетки.
Примечание: Требуется использование высокоэффективных клеток (выше 5 х 109 трансформантов за мкг ДНК). Мы предлагаем использовать Escherichia coli DH5αF' (F'/ endA1 hsd17 (РК − МК +) supE44 Тхи 1recA1 gyrA (Nalr) relA1 (lacZYA-argF) U169 deoR (F80dlacD-(lacZ)M15) производится в доме или приобретенные от нескольких производителей.- Место соответствующее количество microcentrifuge трубок и 0,1 см электропорация кюветы на льду. 1 мкл очищенный лигирование раствора (в воде ди) для 25 мкл клеток и Флик трубки в несколько раз.
- Передача смеси ДНК клеточной холодной кювета, нажмите на столешницу 2 x, протрите воды от экстерьера кювета, место в пульс электропорации модуль и нажмите.
- Выполняют электропорации с машиной стандартного electroporator, с использованием 25 МКФ, 200 Ω и 1,8 кв. Постоянная времени должен быть 4-5 мс.
- Сразу же добавить 1 мл жидких 2xYT среды без любой антибиотик, передать тюбик 10 мл и позволяют выращивать при 37 ° C, тряски на 220 rpm для 1 h.
- Пластина превращается DH5αF' на 15 см 2xYT плиты агара дополнена 34 мкг/мл Хлорамфеникол (pFILTER сопротивление) и 25 мкг/мл ампициллин (селективный маркер для ORFs) и Инкубируйте на ночь на 30 ° C.
- Пластина разведений библиотеки на 10 см 2xYT плиты агара дополнены с хлорамфеникол + Ампициллин и хлорамфеникол только, для выполнения библиотеки титрования. Инкубируйте на ночь на 30 ° C.
- pFILTER-ORF библиотеки проверки
- Испытания 15-20 колоний от хлорамфеникола и хлорамфеникол/ампициллин пластины для оценки распределения размера вставки. Выбрать один колоний с наконечником и развести их отдельно в 100 мкл 2xYT среды без антибиотиков. Используйте 0.5 мкл этого решения в качестве шаблона дна для реакции PCR, с любой стандартной полимеразы TaqDNA производителя протоколом.
- Выполнение 25 циклов усиления с помощью T отжиг 55 ° C и времени расширение 40 s при 72 ° C. Грунтовка последовательности приведены в Таблице материалов.
- Загрузка продуктов ПЦР на 1,5% агарозном геле, вместе с 100 bp лестница ДНК и запуск.
- Коллекция библиотеки pFILTER-ORF
- Собирать бактерий от 150 мм пластин, добавив 3 мл свежего 2xYT среды и жать их с стерильных скребок, тщательно перемешать, дополнить их с 20% стерильный глицерина и хранить при температуре-80 ° C в небольших аликвоты.
- Очистить плазмида ДНК из одной Алиготе библиотеки (перед добавлением глицерин) с использованием комплекта добычи на основе столбцов плазмида, следуя инструкциям производителя.
- Измерьте концентрацию с Спектрофотометр UV. Образцы можно хранить при температуре-20 ° C до использоваться для подготовки библиотека phagemid и/или характеристика NGS.
2. subcloning из отфильтрованных ORFs в векторе Phagemid (рис. 2)
-
Подготовка ORF фильтрации фрагментов ДНК
- Настройка Пищеварение энзима ограничения 5 мкг очищенный вектора из вектора библиотека pFILTER-ORF, добавление 10 U BssHII и инкубации согласно протоколу производителя. Инактивирует фермент и дайджест с 10 U NheI.
- Загрузите переваривается ДНК на 1,5% агарозном геле, вместе с 100 лестница ДНК bp. Выполнение короткого электрофорез на 5 V/см 15 мин или просто достаточно отличить мазок подакцизным фрагментов и вырезать часть геля, содержащего их.
- Очищение дна вставки с комплектом гель колонке база добычи и измерять концентрацию, используя Спектрофотометр UV.
-
Подготовка phagemid ДНК
- Настройка Пищеварение энзима ограничения 5 мкг27 очищенный pDAN5 для вставки.
- Очистить переваривается плазмида, идущий переваривается ДНК на 0,75% агарозном геле и извлекать из геля с набором на основе столбцов.
- Измерьте концентрацию, используя Спектрофотометр UV. Образцы можно хранить при температуре от-20 ° C до использования.
-
Перешнуровка Библиотека, трансформации и коллекции
- Выполняют лигирование и преобразования, как описано для pFILTER вектора.
- Пластина превращается DH5αF' на 150 мм 2xYT плиты агара дополнена 100 мкг/мл Ампициллин и Инкубируйте на ночь на 30 ° C.
- Пластина разведений библиотеки на 100 мм 2xYT плиты агара дополнена 100 мкг/мл ампициллин, чтобы определить размер библиотеки.
- Выполните проверку библиотека методом ПЦР случайно выбранный клонов, как описано в шаге 1.4.
- Собирать библиотеку phagemid-ORF, уборки бактерий от 150 мм пластин, тщательно перемешать, дополнить их с 20% стерильный глицерина и хранить при температуре-80 ° C в небольших аликвоты.
- Очищайте плазмида ДНК из одной Алиготе библиотеки, используя набор для добычи на основе столбцов плазмида, следуя инструкциям производителя.
- Измерение концентрации в спектрофотометре УФ. Образцы можно хранить при температуре-20 ° C до использоваться для характеризации NGS.
3. Фаговые библиотеки подготовка и процедура отбора
- Фаговые производство
- Разбавить запасов Алиготе библиотеки phagemid в 10 мл жидкого бульона 2xYT дополнена 100 мкг/мл ампициллин для того чтобы иметь ОД600nm = 0,05.
- Растут 5 - 10 раз больше, чем исходный объем разреженных библиотеки в стерильную колбу, при 37 ° C при встряхивании в 220 об/мин, до тех пор, пока достигнет OD600nm = 0.5.
- Заразить бактерий с помощником фаг (например M13K07) в разносторонности инфекции 20:1. Оставьте при 37 ° C на 45 мин с случайные агитации (каждые 10 минут).
- Центрифуга бактерий на 4000 x g 10 мин при комнатной температуре. Отменить супернатанта, вновь приостановить Пелле бактерий в 40 мл жидкого бульона 2xYT дополнена ампициллин 100 мкг/мл и 50 мкг/мл канамицин и расти на 28 ° C с встряхивания на 220 об/мин для ночлега.
- На следующий день после, центрифуги бактерий на 4000 x g 20 мин при 4 ° C. Соберите супернатанта, содержащие бактериофаги.
- ПЭГ высыпание бактериофаги.
- Добавить 1/5 объем 0,22 мкм фильтруемый PEG/NaCl решение (20% w/v PEG 6000, 2,5 М NaCl) очищается бактериофаги и инкубировать на льду за 30-60 мин.
Примечание: Решение стало дымный после нескольких минут, указывающий успешное Фаговые осадков. Облачность решения будет увеличиваться со временем инкубации. - Центрифуга на 4000 x g 15 мин при 4 ° C. Белый Малый Пелле бактериофаги лягут.
- Ресуспензируйте в 1 мл стерильного PBS. Трансфер в 1,5 мл трубки и центрифуги на 4 ° C на 10 мин на максимальной скорости, чтобы удалить загрязнения бактерий. Станут коричневые Пелле.
- Супернатант передачи, содержащие бактериофаги к новой пробке. Держите бактериофаги на льду для выбора последовательных титрования и ФАГ.
- Добавить 1/5 объем 0,22 мкм фильтруемый PEG/NaCl решение (20% w/v PEG 6000, 2,5 М NaCl) очищается бактериофаги и инкубировать на льду за 30-60 мин.
- Фаговые титрования
- Подготовьте серийных разведений Фаговые решения. Положите 10 мкл раствора ФАГ в 990 мкл PBS для получения 10-2 разрежения. Разбавьте снова этот препарат, чтобы сделать 10-4 и от этого получить разбавления 10-6 .
- Расти DH5αF' клеток бактерий в жидкой среде 2xYT при 37 ° C при встряхивании до од600nm = 0,5 достигается. Передача 1 мл подготовленный бактерий в 1,5 мл трубку и сразу же заразить с 1 мкл Фаговые разбавления 10-4 . Инкубируйте без тряски при 37 ° C на 45 мин повторить ту же процедуру для разбавления 10-6 .
- Пластина разведений зараженных бактерий в 100 мм 2xYT пластины. Установите пластину на 30 ° C на ночь.
- Пластина 100 мкл неинфицированных DH5αF' на табличке агар 2xYT дополнена 100 мкг/мл ампициллин проверить отсутствие загрязнения в подготовке.
- На следующий день после подсчитать количество колоний и рассчитать титр ФАГ. Экспресс титр как количество бактериофаги/мл. Ожидаемые титр составляет 10-12-13 бактериофаги/мл.
- Фаговые выбор
-
Фаговые выбор, используя в качестве наживки очищенные антитела
- Saturate бактериофаги путем разбавления 200 мкл препарата ФАГ в равным объемом PBS - 4% сухого обезжиренного молока и инкубировать 1 час при комнатной температуре в медленное вращение. Этот шаг позволяет настроить блокирование бактериофаги для неспецифической привязки. Передать 30 мкл белка G с покрытием магнитные бусы 1,5 мл.
- Мыть два раза следующим: 500 мкл PBS, инкубировать на колесе в медленное вращение на 2 мин при комнатной температуре, обратить бисер на одной стороне трубки с помощью магнита и удалить супернатант.
- Инкубируйте насыщенных бактериофаги промывают бисером на 30 минут при комнатной температуре с медленным вращением.
- Нарисуйте бисер с одной стороны, с помощью магнитного поля. Соберите супернатанта, содержащий бактериофаги использоваться для выбора шага.
- Подготовьте магнитные бусы при выполнении на предыдущем шаге, спряжение очищенных антител. Вымойте 30 мкл белка G с покрытием магнитные бусы, как описано выше. Разбавить 10 мкг очищенных антител в 500 мкл PBS, Добавить промытый бусины и инкубировать в медленное вращение при комнатной температуре 45 мин мыть дважды с PBS.
Примечание: Выполнение двух различных препаратов магнитных шариков: один с антителами интерес и один с управления антител, например, антитела, очищенного от здоровых доноров. Последовательность, антигенов, выбранные с контролем, что антитела вычитаются на этапе анализа результатов. В качестве альтернативы магнитные бусы, нагруженные антителами управления может использоваться для выполнения предварительной очистки шаг бактериофаги (следовать протокол для инкубации с ООН конъюгированных бисером). - Фаговые выбор: рисовать бусы на одной стороне трубки с помощью магнита, удалить последний мыть, добавить бактериофаги и проинкубируйте с медленного вращения при комнатной температуре за 90 минут Вымойте 5 раз с 500 мкл PBS-0.1% Tween-20 и 5 раз с PBS.
- Элюировать привязанных бактериофаги, представляющий результат выбора, путем смешивания бисер с 1 мл раствора DH5αF' клеток, выращенных на од600 = 0.5. Инкубируйте бактерий с бисером для 45 минут при 37 ° C встряхивании периодически (каждые 10 минут). Пластина вывода на 150 мм 2xYT агар тарелку с 100 мкг/мл ампициллина.
- Пластина 100 мкл неразбавленной и различных разведениях вывода (10-1 -10-5) для выполнения титрования. На следующий день после сбора бактерий от 150 мм пластин, добавив 3 мл свежего 2xYT среды и жать их с стерильных скребок, тщательно перемешать, дополнить их с 20% стерильный глицерина и хранить при температуре-80 ° C в небольших аликвоты.
- Растут один Алиготе снова, чтобы выполнить второй раунд отбора. Повторите все панорамирования процедуру, как описано выше, за исключением условия стирки. В этом случае мыть 10 раз с PBS - 1% Tween-20 (Залейте раствор в трубе и вылейте снова немедленно). Затем добавить 500 мкл PBS и инкубировать на вращение при комнатной температуре за 10 мин выполняют другие 10 стирок с PBS. Перейдите к шагу элюции как первый раунд отбора.
- Извлечь плазмида ДНК из одной Алиготе вывода с помощью набор на основе столбцов, следуя инструкциям производителя. Хранить плазмида при-20 ° C до тех пор, пока он будет использоваться для глубокого последовательности.
-
Фаговые выбор, используя в качестве наживки рекомбинантных белков
- Saturate бактериофаги путем разбавления 200 мкл препарата ФАГ в равным объемом PBS - 4% сухого обезжиренного молока и инкубировать 1 час при комнатной температуре в медленное вращение.
- 100 мкл стрептавидина магнитные бусы. Инкубируйте 1 час при комнатной температуре для выбора бактериофаги стрептавидина привязки. Удаление стрептавидина привязкой бактериофаги, нарисовав бисер в одну сторону с помощью магнита. Возьмите супернатанта из предыдущего шага и добавить биотинилированным белка (в концентрации 100-550 Нм) и инкубировать на ротор при комнатной температуре за 30 мин до 1 часа.
- Подготовить магнитные бусы: при выполнении предыдущего шага, мыть 100 мкл стрептавидина магнитные бусы с PBS, Ресуспензируйте в PBS 2% сухого обезжиренного молока и проинкубируйте с вращения при комнатной температуре за 30 мин до 1 часа.
- Фаговые выбор: рисовать бусы на одной стороне трубки с помощью магнита, удалить PBS - 2% молока и Ресуспензируйте бусины с ФАГ белка смесь. Проинкубируйте с медленного вращения при комнатной температуре 90 мин.
- Рисовать бусы на одной стороне трубки с помощью магнита, удалить супернатант и мыть их тщательно пять раз с 500 мкл PBS 0,1% Tween-20. Выполните элюции, как описано в предыдущей сессии.
-
Фаговые выбор, используя в качестве наживки очищенные антитела
4. Фаговые библиотека глубокую виртуализации платформы (рис. 3)
-
ДНК вставляет восстановления от pFILTER-ORF-библиотеки, pDAN5-ORF-библиотеки или выбран ФАГ библиотек
- Размораживание одного Алиготе библиотеки, количественно с помощью спектрофотометра, восстановить вставками ДНК путем усиления с конкретными грунтовки.
Примечание: Праймеры, используемый для спасения вставки связаны в конце их 5' для адаптеров последовательностей, таким образом позволяя последовательных индексации ампликон бассейнов получены и прямого секвенирования ДНК вставок восстановлены с помощью синтезаторов. В Таблица материалових последовательность. Сетевые адаптеры жирным шрифтом, и конкретные праймеры выделены курсивом. - 2.5 мкл (pFILTER/phagemid/выбран ФАГ) библиотеки можно используйте в качестве шаблона дна для реакции PCR.
- Используйте следующую программу: 95 ° C 3 мин; 25 циклов 95 ° c за 30 сек, 55 ° C за 30 сек, 72 ° C для 30 s; 72 ° C для удержания 5 мин при 4 ° C.
Примечание: на данном этапе рекомендуется для запуска 1 мкл продукта PCR на Bioanalyzer или TapeStation, чтобы проверить размер ампликонов и проверить, что они находятся в допустимом диапазоне.
- Размораживание одного Алиготе библиотеки, количественно с помощью спектрофотометра, восстановить вставками ДНК путем усиления с конкретными грунтовки.
-
Очистке PCR
- Магнитные бусы (например, AMPure) довести до комнатной температуры. Передать весь продукт PCR от ПЦР-пробирку 1,5 мл. Вихревые магнитные шарики для 30 s, чтобы убедиться, что бисер равномерно рассредоточены. 20 мкл магнитной бусины, к каждой трубе, содержащие продукт PCR, микс от нежно закупорить. Инкубации при комнатной температуре без встряхивания в течение 5 мин.
- Место пластину на магнитного стенд на 2 мин или до тех пор, пока расчистил супернатант. С продуктов ПЦР на магнитную подставку снимите и выбросьте супернатант.
- Вымойте бусины с свежеприготовленные 80% этанола, с продуктов ПЦР на магнитного стенд, следующим: 200 мкл свежеприготовленные 80% этанола для каждого образца хорошо; инкубировать пластину на магнитного стенд для 3 s; Осторожно снимите и выбросьте супернатант.
- Выполните второй мыть этанола, с продуктов ПЦР на магнитную подставку; в конце второй мыть тщательно удалить все этанола и позволяют бисером высохнуть за 10 мин.
- Удаление продуктов ПЦР из магнитного стенд, 17,5 мкл 10 мм трис рН 8,5 к каждой трубе, нежно Пипетка вверх и вниз в 10 раз, убедитесь, что бусины полностью высокомобильна. Инкубации при комнатной температуре на 2 мин.
- Поместите трубки на магнитного стенд на 2 мин или до тех пор, пока расчистил супернатанта, тщательно передачи 15 мкл супернатанта, содержащих чистые продукты PCR для новой 1,5 мл трубки. Храните очищенные продукты PCR при-15 ° C до-25 ° C до недели, если не приступить немедленно к индекс PCR.
-
Индекс PCR
Примечание: После очистки ПЦР, выполните индекс PCR. Используйте комплект Nextera XT индекс; Таким образом можно будет последовательность результате двойной индексированных библиотек в мультиплексированном Illumina бежит.- Перевести все 15 мкл, содержащие каждый продукт очищается в новой ПЦР-пробирку и настроить следующими содержащие реакции: 15 мкл очищенный ампликон продукта, 5 мкл индекс грунтовка 1 и 5 мкл индекс грунтовка 2, 25 мкл 2 x PCR смеси; Окончательный объем 50 мкл.
- Выполнять ПЦР на тепловая велосипедист, используя следующую программу: 95 ° C в течение 3 мин, 8 циклов 95 ° c за 30 сек, 55 ° C за 30 s, 72 ° C для 30 s; 72° C за 5 мин, затем провести на 4 ° C.
-
ПЦР-очистки 2
- Следовать за тот же протокол, описанные в разделе 4.2 для ПЦР убирать со следующими изменениями: в первом шаге мкл 56 магнитной бусины для каждого 50 мкл продукта PCR.
- Ресуспензируйте бисер в 27,5 мкл 10 мм трис рН 8,5 на заключительном этапе очищения и передачи 25 мкл новой трубки (это очищенный окончательный библиотека готовых для количественной оценки и затем последовательности).
- Храните пластины при-15 ° C до-25 ° C до недели, если не приступить к библиотеки количественной оценки.
-
Качественные и количественные оценки последовательности библиотеки
- После очистки, запустить 1 мкл 1:10 разрежения окончательный библиотеки на bioanalyzer, чтобы проверить размер и количественно это выбор региона окончательный Библиотека трассировки.
- Параллельно выполните библиотека количественной оценки в реальном масштабе времени PCR с помощью библиотеки комплекта количественной оценки за производителя протокол.
-
Библиотеки виртуализации
- Бильярд двойного индексированного библиотек, производится вместе с других библиотек двойного индексированного последовательности. Считывает последовательность, которую долго читает этот вид библиотеки, создавая, по крайней мере 250 bp парных конец с помощью Hiseq2500 или MiSeq инструментов для получения в первом случае 250bp PE читает и в втором случае 300 bp PE.
5. Bioinformatic данных анализа с помощью Interactome-Seq веб-инструмент
- Анализируйте читает, возникла из библиотеки pFILTER/phagemid/выбран ФАГ последовательности с Interactome-seq конвейер данных анализа. Веб-инструмент свободно доступен по следующему адресу: http://interactomeseq.ba.itb.cnr.it/
Результаты
Метод фильтрации схематизируются на рисунке 1. Каждый вид intronless ДНК может использоваться. На рисунке 1A представлена первая часть фильтрации подхода: после загрузки на геле агарозы или bioanalyzer, хороший фрагментация ДНК интерес появляется как мазок фрагментов с распределение длины в желаемый размер 150-750 ВР. Дается образ представителя виртуальный гель фрагментированной ДНК получены. Фрагменты загружены на геле агарозы затем восстановлен, конец ремонт и фосфорилированных и затем клонирован в ранее притупляются pFILTER вектор для создания библиотеки случайных фрагментов ДНК. Для получения хорошего качества библиотека с полный охват ДНК исследуемых требуется выполнять каждый шаг процедуры клонирования при оптимальных условиях.
На рисунке 1B фильтрации подход представлен: Библиотека выращивается в присутствии Хлорамфеникол (pFILTER сопротивление) самостоятельно или хлорамфеникола и ампициллин выбрать ORF-содержащих колоний. Только колоний, имея фрагмент ДНК, соответствующий ORF производства функциональных β-лактамаз и выжить при наличии антибиотика выбора. Рисунок 1 c показывает, как увеличение селективного давления позволяет выбор хороших папку ORFs против бедных папки из них. Ожидаемым результатом является уменьшение размера библиотеки около кратной. Большее количество сохранившихся клонов показывает недостаточно селективного давления.
ORF фрагменты могут быть легко восстановлены из отфильтрованного библиотеки для последующего применения; для исследования взаимодействия Наша стратегия использует преимущества технологий отображения ФАГ. В рисунке 2представлены основные шаги Фаговые библиотека строительства: адекватные библиотека готовится путем вырезания отфильтрованных фрагменты из вектора pFILTER и повторного клонирования в плазмиду phagemid в fusion с последовательности кодирования для Фаговые capside g3p белка. После того, как инфицированных вспомогательный фага, наличие вектора в клетки бактерий позволяет производстве фаговых частиц отображения ORF-g3p фьюжн продуктов на их поверхности, таким образом делая отфильтрованных библиотеки доступны для выбора отображения ФАГ и далее анализ.
Все библиотеки глубоко проанализированы NGS, а также выходы Фаговые выбор, как показано во второй части Рисунка3. Фрагменты ДНК были спасены от растущих колоний по амплификации PCR с конкретными олигонуклеотиды отжига на хребте плазмиды и перевозящих конкретные адаптеры для виртуализации. NGS производится и читает затем анализируются с помощью инструмента web Interactome-Seq данных анализа.
На рисунке 4 мы сообщали схематическое представление процедуры отбора библиотеки отображения отфильтрованных Фаговые ORF. Выбор в этом примере выполняется с помощью антител, присутствующих в sera от пациентов, пострадавших от различных патологий (т.е. инфекционной патологии, аутоиммунной патологии, рак). В этом случае библиотека Фаговые непосредственно взаимодействует с антител в сыворотки больных и таким образом, что предполагаемые конкретные антигены можно обогатить потому, что они признаются специфические антитела болезни. В такого рода эксперимент обычно Библиотека также выбирается с помощью контроля сывороток от здоровых пациентов для того чтобы иметь фонового сигнала, который будет использоваться для последующих процедур сравнения и нормализации.
Выбор выполняются с использованием сыворотки из того же типа пациентов обычно сгруппированы в различные пулы для того, чтобы уменьшить межличностная изменчивость сера титр антител. Каждый пул независимо используется для двух-трех последовательных раундов выделения, обогатить библиотека для иммунной реактивной клоны специфические для патологии изучается. Тестовый набор антител инкубируют с библиотека бактериофаги, иммунной комплексы восстанавливаются протеина A покрытием магнетизм бисер и связанные бактериофаги этого eluted путем стандартными процедурами. Выбор циклов выполняются с увеличением мойки и привязка жесткости.
Читает, порожденных NGS могут быть проанализированы с помощью Interactome-Seq веб-инструмент разработан специально для управлять такого рода данных. Interactome-Seq анализа данных рабочего процесса состоит из четырех последовательных шагов, которые, начиная с сырой последовательности читает, генерирует список предполагаемых доменов с геномной аннотации (Рисунок 5A). На первом шаге входные данные (Рисунок 5A - красное поле) Interactome-Seq проверяет входные файлы (сырые читает, ссылка генома, списке аннотаций) правильно отформатирован. На втором шаге Предварительная обработка (Рисунок 5A - Оранжевая коробка), низкого качества последовательности данных сначала удаляются с помощью28 Cutadapt в зависимости от показателей качества и читает с менее чем 100 баз в длину, отбрасываются. В последующем шаге читать выравнивание (Рисунок 5A - зеленый флажок) остальные читает выравниваются с29 blastn в геноме, позволяя до 5% несоответствия. SAM файл создается и считывает только с больше чем 30 показатель качества (Q > 30) обрабатываются с помощью SAMtools30 и преобразован в файл BAM. После выравнивания Interactome-Seq выполняет домены обнаружения (Рисунок 5A - blue box), вызов Bedtools31 для фильтрации читает перекрытия по крайней мере, для 80% от их длины внутри стенограмм; освещение, Макс глубина и фокус значения затем рассчитываются для каждой части ORF, охватываемых сопоставления читает. Покрытие представляет собой общее количество операций чтения, присвоенный ген; Глубина — максимальное количество операций чтения, охватывающих конкретные генно часть; основное внимание — это индекс, полученные из соотношения между Макс глубина и охват, и она может варьироваться от 0 до 1. Когда фокус находится выше, чем 0,8 и охват является выше, чем средний охват наблюдается во всех регионах сопоставления в файле BAM, компакт-диски часть классифицируется как предполагаемого домена/epitope. Последний шаг трубопровода Interactome-Seq – выход (Рисунок 5A - фиолетовый box), список доменов, предполагаемый генерируется в табличном формате раздельный. Interactome-Seq трубопровода был включен в веб инструмента, позволяющего пользователям без каких-либо биоинформатики или навыков программирования для выполнения анализа Interactome-Seq через графический интерфейс и получать их результаты в легкой и удобной для пользователей форме. Как показано на рисунке 5B, результаты анализа отображаются с помощью JBrowse32 визуализации и разведки. Interactome-Seq генерирует треков в браузере генома, соответствует предполагаемым доменов обнаружения и обеспечивает также классической Венна чтобы показать пересечений между общего предполагаемого доменами, обогащенный например выбор различных экспериментов.

Рисунок 1: Схематичный обзор основных шагов для строительства фильтрации ORF библиотеки
A) ДНК из различных источников sonicated и раскололась на случайных фрагментов длиной 150-750 bp. Фрагменты оправился от геля и клонирования как тупые в pFILTER вектор; B) фильтрация шаг, используя β-лактамаз складной репортером. Вектор, содержащий не ORF фрагменты негативно выбираются на ампициллин во время ORF Клонированные фрагменты позволяют колоний расти; C) применение все более селективного давления (ампициллин концентрация в СМИ твердого роста от 0 до > 100 мкг/мл) позволяют выбор лучше сложить фрагментов. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 2: Схематичный обзор основных шагов для строительства Фаговые библиотеки
A) фильтрации ORF фрагменты вырезали из отфильтрованного вектора с использованием определенных энзимов ограничения. После очистки и восстановления фрагменты клонирован в phagemid вектор и преобразован; B) phagemid бактериальной библиотека инфицирован вспомогательный ФАГ и, после ночи роста, бактериофаги PEG-осаждают и собраны. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 3: ORF библиотеки виртуализации
Последовательности выполняется на обоих оригинальные ORF выбранной библиотеки, а также на Фаговые отображения библиотеки; 1) на обоих случаях восстанавливаются колоний выросли и извлечь ДНК; 2) фрагменты ДНК восстанавливаются путем усиления с помощью конкретных грунты, связан с адаптеры для последовательности; 3-4) фрагменты восстановленные и глубоких виртуализации с помощью NGS; 5) данные анализируются с помощью Interactome-Seq конвейера. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
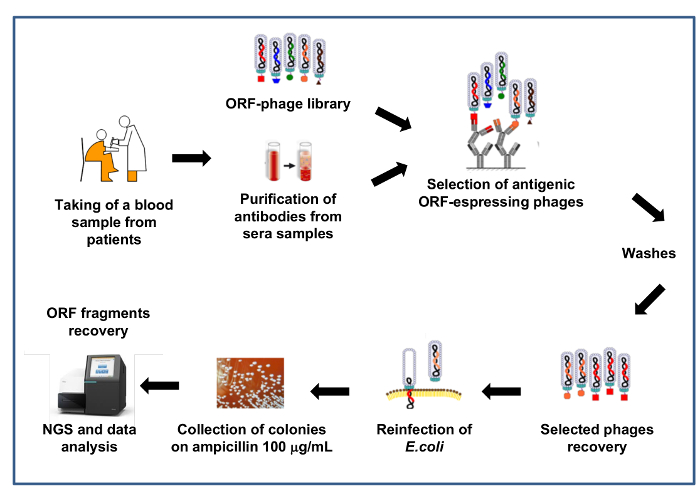
Рисунок 4: Схематичный обзор библиотеки отбора с использованием антител пациентов
Фаговые библиотека используется для выбора против антитела от пациентов сера. Антитела являются иммобилизованных на магнитной бусины, фаг библиотека захвата/выбор производится, выполняются три цикла мойки и впоследствии оправился и используется повторно заразить выбранных бактериофаги E. coli. Вновь инфицированных E. coli клетки покрыты селективного давления (ампициллин 100 мкг/мл). ORF фрагменты восстанавливаются путем усиления и бассейны ампликон затем Виртуализирован с NGS. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
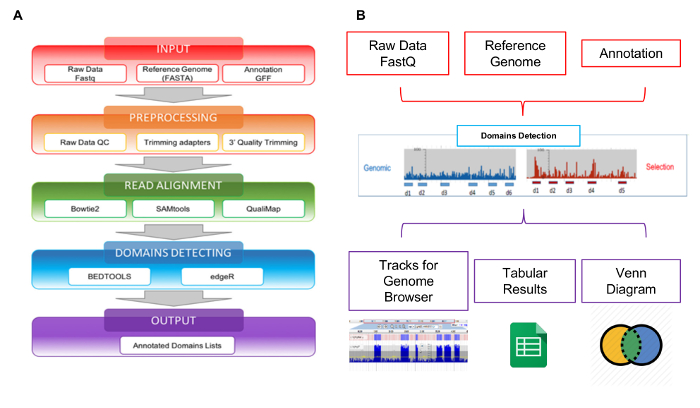
Рисунок 5: Схематичный обзор библиотеки анализа
A) представление рабочего процесса анализа данных, начиная с raw файлов FASTQ для окончательного аннотированный домены списки; B) схематическое представление входы и выходы Interactome-Seq веб-инструмент. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
Создание высокого качества весьма разнообразны ORFs отфильтрованных библиотеки является первым важным шагом в рамках всей процедуры так, как оно повлияет на все последующие шаги трубопровода.
Важной особенностью выгодно нашего метода является, что любой источник (intronless) ДНК (cDNA, геномной ДНК, ПЦР получены или синтетических ДНК) подходит для строительства библиотеки. Первый параметр, который следует принимать во внимание это, что длина фрагментов ДНК, клонированные в pFILTER вектор должен обеспечить представление всей коллекции областей генома или транскриптом, так называемые «domainome». Мы продемонстрировали, что доменов протеина могут быть успешно клонирован, выбран и окончательно определены, начиная от фрагментов ДНК с длиной распространения охватывающих от 150 до 750 bp33,34, и это соответствует тому, что сообщается в литература, показывая, что большинство доменов протеина 100 aa длины (в диапазоне от 50 до 200 aa)15.
ДНК, исходный материал должен раскололась на размер диапазон выбора и позднее клонированных в фильтрации вектор12 (pFILTER). Во время этих шагов можно избежать потенциальной предвзятости, максимальной эффективности все клонирование шаги реакций, включены в протокол, в частности фрагмент конец ремонт и фосфорилирования. Подготовка вектор является сложной задачей и следует при оптимальных условиях, а также, чтобы избежать плазмид деградации и/или загрязнение непереваренных вектор.
После того, как была создана библиотека, его следует «фильтровать» для того, чтобы сохранить только ORFs сложенном фрагментов. Ключевым параметром для модуляции этот шаг является селективным давление, которые могут быть изменены согласно строгости фильтрации желаемого. Выбор производится с использованием ампициллина: чем выше концентрация используется, тем меньше количество преобразованных бактерий колоний смогли выжить. Это отражает способность метода фильтрации для выбора хорошего-против бедных папка ORFs34. Это сокращение количество клонов уравновешивается увеличением складывания свойства выбранных фрагментов. Обычно ампициллин концентрация должна быть достаточно, чтобы сократить количество бактериальных колоний в отношении тех, которые могут быть получены растущей библиотеки на хлорамфеникол только до 1/20.
Библиотека проверки обычно делается путем амплификации PCR случайно выбранный колоний и их последовательности. Амплификации PCR некоторых колоний предложил для быстрой оценки качества библиотеки: длина вставки должна быть в ожидаемый диапазон 150-750 bp и различных колоний следует настоящему вставок с различными размер указанием хорошо Подготовка библиотеки в перспективе изменчивости. Эта обычных стратегия отбора, когда применяется как единственный метод для проверки библиотеки, не является всеобъемлющим и отнимает много времени, позволяя анализ лишь ограниченное количество колоний и имеющие высокий шанс пропавших без вести большую часть важных клонов. Наш подход основан на глубокой последовательности библиотеки, это обеспечивает полную информацию о разнообразии библиотеки и изобилия и точное сопоставление каждого из выбранных фрагментов.
Внедрение технологии NGS с фильтрации подход увеличивает глубину анализа на несколько порядков. Недавно мы оптимизированный протокол для секвенирования ORF библиотек с помощью платформе Illumina и разработали конкретные веб-инструмент для анализа данных, что делает анализ такого рода данных для каждого пользователя без каких-либо навыков программирования биоинформатики.
Библиотека «per se» представляет собой «универсальный инструмент» и может быть использована в различных контекстах для выражения протеина и/или отбора. Наш методологический подход основан на передаче производства ORFeome в контекст отображения ФАГ. Фрагменты белка выражаются на поверхности ФАГ и стал пригодным для последующего отбора.
Это делается спасать отфильтрованных ORFs из библиотеки pFILTER, пищеварение с определенных энзимов ограничения и повторно клонирования их в совместимый phagemid вектор, позволяя их слияние с g3p белка ФАГ.
После создания библиотеки phagemid-ORF, она может использоваться для выбора против различных целей, таких как предполагаемые связывания белков10 или очищенных антител35,36 как описано здесь. Так как Фаговые частицы будут отображаться на их поверхности, отфильтрованных ORFs, это результаты в гораздо более эффективные процедуры отбора в связи с отсутствием-отображение клоны, обычно обогнать его.
После выбора библиотеки ORF отображения ФАГ клоны вывода можно упорядочивать и проанализированы с помощью же конвейера. NGS может предоставить полный и статистически рейтинг наиболее часто выбраны ORFs, и это позволяет идентификации белков, главным образом взаимодействующих с приманки используется. Учитывая наличие множества различных версий каждого домена, отличающихся несколько аминокислоты, дублирования между различными виртуализированного клонов также определяет минимальный фрагмент/домен показаны свойства привязки. Наконец благодаря муфты генотип и фенотип информации в библиотеку фага, после того, как были определены домены выбор, последовательности ДНК могут быть легко спасены из библиотеки для дальнейших исследований, в пробирке и в естественных условиях Проверка и определение характеристик.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Эта работа была поддержана грант от министерства образования Италии и университета (2010P3S8BR_002 КТ).
Материалы
| Name | Company | Catalog Number | Comments |
| Sonopuls ultrasonic homogenizer | Bandelin | HD2070 | or equivalent |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | or equivalent |
| GeneRuler 1 kb DNA Ladder | Thermo Fisher Scientific | SM0311 | or equivalent |
| Molecular Biology Agarose | BioRad | 161-3102 | or equivalent |
| Green Gel Plus | Fisher Molecular Biology | FS-GEL01 | or equivalent |
| 6x DNA Loading Dye | Thermo Fisher Scientific | R0611 | or equivalent |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | or equivalent |
| Quick Blunting Kit | New England Biolabs | E1201S | |
| NanoDrop 2000 UV-Vis Spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368813 | |
| Streptavidin Magnetic Beads | New England Biolabs | S1420S | or equivalent |
| QIAquick PCR purification Kit | Qiagen | 28104 | or equivalent |
| EcoRV | New England Biolabs | R0195L | |
| Antarctic Phosphatase | New England Biolabs | M0289S | |
| T4 DNA Ligase | New England Biolabs | M0202T | |
| Sodium Acetate 3M pH5.2 | general lab supplier | ||
| Ethanol for molecular biology | Sigma-Aldrich | E7023 | or equivalent |
| DH5aF' bacteria cells | Thermo Fisher Scientific | ||
| 0,2 ml tubes | general lab supplier | ||
| 1,5 ml tubes | general lab supplier | ||
| 0,1 cm electroporation cuvettes | Biosigma | 4905020 | |
| Electroporator 2510 | Eppendorf | ||
| 2x YT medium | Sigma-Aldrich | Y1003 | |
| Ampicillin sodium salt | Sigma-Aldrich | A9518 | |
| Chloramphenicol | Sigma-Aldrich | C0378 | |
| DreamTaq DNA Polymerase | Thermo Fisher Scientific | EP0702 | |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | |
| 96-well thermal cycler (with heated lid) | general lab supplier | ||
| 150 mm plates | general lab supplier | ||
| 100 mm plates | general lab supplier | ||
| Glycerol | Sigma-Aldrich | G5516 | |
| BssHII | New England Biolabs | R0199L | |
| NheI | New England Biolabs | R0131L | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | or equivalent |
| M13KO7 Helper Phage | GE Healthcare Life Sciences | 27-1524-01 | |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K1377 | |
| Polyethylene glycol (PEG) | Sigma-Aldrich | P5413 | |
| Sodium Cloride (NaCl) | Sigma-Aldrich | S3014 | |
| PBS | general lab supplier | ||
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10003D | or equivalent |
| MagnaRack Magnetic Separation Rack | Thermo Fisher Scientific | CS15000 | or equivalent |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Nonfat dried milk powder | EuroClone | EMR180500 | |
| KAPA HiFi HotStart ReadyMix | Kapa Biosystems, Fisher Scientific | 7958935001 | |
| AMPure XP beads | Agencourt, Beckman Coulter | A63881 | |
| Nextera XT dual Index Primers | Illumina | FC-131-2001 or FC-131-2002 or FC-131-2003 or FC-131-2004 | |
| MiSeq or Hiseq2500 | Illumina | ||
| Spectrophotomer | Nanodrop | ||
| Agilent Bioanalyzer or TapeStation | Agilent | ||
| Forward PCR primer | general lab supplier | 5’ TACCTATTGCCTACGGCA GCCGCTGGATTGTTATTACTC 3’ | |
| Reverse PCR primer | general lab supplier | 5’ TGGTGATGGTGAGTACTA TCCAGGCCCAGCAGTGGGTTTG 3’ | |
| Forward primer for NGS | general lab supplier | 5’ TCGTCGGCAGCGTCAGA TGTGTATAAGAGACAGGCA GCAAGCGGCGCGCATGC 3’; | |
| Reverse primer for NGS | general lab supplier | 5’ GTCTCGTGGGCTCGGAGA TGTGTATAAGAGACAGGGG ATTGGTTTGCCGCTAGC 3’; |
Ссылки
- Loman, N. J., Pallen, M. J. Twenty years of bacterial genome sequencing. Nat Rev Microbiol. 13 (12), 787-794 (2015).
- Jones, C. E., Brown, A. L., Baumann, U. Estimating the annotation error rate of curated GO database sequence annotations. BMC Bioinformatics. 8 (1), 170 (2007).
- Andorf, C., Dobbs, D., Honavar, V. Exploring inconsistencies in genome-wide protein function annotations: a machine learning approach. BMC Bioinformatics. 8 (1), 284 (2007).
- Wong, W. -. C., Maurer-Stroh, S., Eisenhaber, F. More Than 1,001 Problems with Protein Domain Databases: Transmembrane Regions, Signal Peptides and the Issue of Sequence Homology. PLoS Comput Biol. 6 (7), e1000867 (2010).
- Bioinformatics, B., et al. Identification and correction of abnormal, incomplete and mispredicted proteins in public databases. BMC Bioinformatics. 9 (9), (2008).
- Phizicky, E., Bastiaens, P. I. H., Zhu, H., Snyder, M., Fields, S. Protein analysis on a proteomic scale. Nature. 422 (6928), 208-215 (2003).
- DiDonato, M., Deacon, A. M., Klock, H. E., McMullan, D., Lesley, S. A. A scaleable and integrated crystallization pipeline applied to mining the Thermotoga maritima proteome. J Struct Funct Genomics. 5 (1-2), 133-146 (2004).
- Nordlund, P., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Zacchi, P., Sblattero, D., Florian, F., Marzari, R., Bradbury, A. R. M. Selecting open reading frames from DNA. Genome Res. 13 (5), 980-990 (2003).
- Di Niro, R., et al. Rapid interactome profiling by massive sequencing. Nucleic Acids Res. 38 (9), e110 (2010).
- Gourlay, L. J., et al. Selecting soluble/foldable protein domains through single-gene or genomic ORF filtering: Structure of the head domain of Burkholderia pseudomallei antigen BPSL2063. Acta Crystallogr Sect D Biol Crystallogr. 71 (Pt 11), 2227-2235 (2015).
- D'Angelo, S., et al. Filtering "genic" open reading frames from genomic DNA samples for advanced annotation. BMC Genomics. 12 (Suppl 1), S5 (2011).
- D'Angelo, S., et al. Profiling celiac disease antibody repertoire. Clin Immunol. 148 (1), 99-109 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2009).
- Heger, A., Holm, L. Exhaustive enumeration of protein domain families. J Mol Biol. 328 (3), 749-767 (2003).
- Zacchi, P., Sblattero, D., Florian, F., Marzari, R., Bradbury, A. R. M. Selecting open reading frames from DNA. Genome Res. 13 (5), 980-990 (2003).
- Faix, P. H., Burg, M. A., Gonzales, M., Ravey, E. P., Baird, A., Larocca, D. Phage display of cDNA libraries: Enrichment of cDNA expression using open reading frame selection. Biotechniques. 36 (6), 1018-1029 (2004).
- Patrucco, L., et al. Identification of novel proteins binding the AU-rich element of α-prothymosin mRNA through the selection of open reading frames (RIDome). RNA Biol. 12 (12), 1289-1300 (2015).
- Collins, M. O., Choudhary, J. S. Mapping multiprotein complexes by affinity purification and mass spectrometry. Curr Opin Biotechnol. 19 (4), 324-330 (2008).
- Suter, B., Kittanakom, S., Stagljar, I. Two-hybrid technologies in proteomics research. Curr Opin Biotechnol. 19 (4), 316-323 (2008).
- Nakai, Y., Nomura, Y., Sato, T., Shiratsuchi, A., Nakanishi, Y. Isolation of a Drosophila gene coding for a protein containing a novel phosphatidylserine-binding motif. J Biochem. 137 (5), 593-599 (2005).
- Deng, S. J., et al. Selection of antibody single-chain variable fragments with improved carbohydrate binding by phage display. J Biol Chem. 269 (13), 9533-9538 (1994).
- Danner, S., Belasco, J. G. T7 phage display: A novel genetic selection system for cloning RNA-binding proteins from cDNA libraries. Proc Natl Acad Sci. 98 (23), 12954-12959 (2001).
- Gargir, A., Ofek, I., Meron-Sudai, S., Tanamy, M. G., Kabouridis, P. S., Nissim, A. Single chain antibodies specific for fatty acids derived from a semi-synthetic phage display library. Biochim Biophys Acta - Gen Subj. 1569 (1-3), 167-173 (2002).
- Patrucco, L., et al. Identification of novel proteins binding the AU-rich element of α-prothymosin mRNA through the selection of open reading frames (RIDome). RNA Biol. 12 (12), 1289-1300 (2015).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. Mol Biol. 1 (2), 146 (2003).
- Sblattero, D., Bradbury, A. Exploiting recombination in single bacteria to make large phage antibody libraries. Nat Biotechnol. 18, 75-80 (2000).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10 (2011).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10 (1), 421 (2009).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Quinlan, A. R. BEDTools: The Swiss-Army tool for genome feature analysis. Curr Protoc Bioinforma. , (2014).
- Skinner, M. E., Uzilov, A. V., Stein, L. D., Mungall, C. J., Holmes, I. H. JBrowse: A next-generation genome browser. Genome Res. 19 (9), 1630-1638 (2009).
- Gourlay, L. J., et al. Selecting soluble/foldable protein domains through single-gene or genomic ORF filtering: Structure of the head domain of Burkholderia pseudomallei antigen BPSL2063. Acta Crystallogr Sect D Biol Crystallogr. 71, 2227-2235 (2015).
- D'Angelo, S., et al. Filtering "genic" open reading frames from genomic DNA samples for advanced annotation. BMC Genomics. 12 (Suppl 1), S5 (2011).
- Di Niro, R., et al. Characterizing monoclonal antibody epitopes by filtered gene fragment phage display. Biochem J. 388 (Pt 3), 889-894 (2005).
- D'Angelo, S., et al. Profiling celiac disease antibody repertoire. Clin Immunol. 148 (1), 99-109 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены