Method Article
Interactome-Seq: פרוטוקול עבור ספריית Domainome ובניה, אימות בחירה Phage התצוגה ורצף הדור הבא
In This Article
Summary
הפרוטוקולים תיאר מאפשרים אפיון ובניה, מבחר (נגד המטרה של הבחירה) ספרייה "domainome" גרם מכל מקור ה-DNA. זו מושגת על ידי צינור מחקר המשלב טכנולוגיות שונות: phage התצוגה, כתב מתקפלים, רצף הדור הבא עם כלי אינטרנט לניתוח נתונים.
Abstract
כתבים מתקפלים הם חלבונים עם פנוטיפים קלות לזיהוי, כגון עמידות לאנטיביוטיקה, אשר מתקפל ותפקוד נפגעת כאשר דבוקה לקוי קיפול חלבונים או מסגרות קריאה פתוחה אקראי. פיתחנו אסטרטגיה שבו, על-ידי שימוש TEM-1 β לקטמאז (האנזים היוועצות ההתנגדות אמפיצילין) בסולם גנומית, אנחנו יכולים לבחור אוספים של תחומים חלבון מקופל כראוי מן החלק קידוד של ה-DNA של כל הגנום intronless. שברי חלבון מתקבל על ידי גישה זו, שנקרא "domainome", תהיה ביטוי היטב מסיסים, שהופך אותם למתאימים ללימודי מבני/תפקודית.
שיבוט ומציג את "domainome" ישירות במערכת התצוגה phage, אנחנו הראו שזה אפשרי לבחור תחומים חלבון ספציפי עם מאפייני האיגוד הרצוי (למשל, לחלבונים אחרים או נוגדנים), ובכך לספק חיוני ניסיוני מידע לזיהוי גנים ביאור או אנטיגן.
הזיהוי של שיבוטים מועשר ביותר בקרב אוכלוסיה polyclonal שנבחר יכולה להיות מושגת באמצעות טכנולוגיות הדור הבא רצפי הרומן (הגדרות). מסיבות אלו, אנו מציגים את רצף עמוק ניתוח של הספריה עצמה והתפוקות הבחירה כדי לספק מידע מלא על גיוון, שפע של מיפוי מדויק של כל אחד המקטע שנבחר. הפרוטוקולים המובאת כאן הצגת השלבים המפתח בניית ספרייה, אפיון של אימות.
Introduction
כאן, אנו מתארים שיטה תפוקה גבוהה הבנייה ואת הבחירה של ספריות של חלבון מקופל ולא מסיסים תחומים מכל מקור ההתחלה genic/גנומית. הגישה משלבת בשלוש טכנולוגיות שונות: phage לתצוגה, השימוש של כתב מתקפלים, רצף הדור הבא (הגדרות) עם כלי אינטרנט מסוימים עבור ניתוח נתונים. השיטות ניתן להשתמש בהקשרים שונים רבים של חלבון מבוססת מחקר, זיהוי, ביאור של דומיינים חלבונים/חלבונים חדשים, אפיון של מאפיינים מבניים ופונקציונליים של חלבונים הידועים, כמו גם ההגדרה של רשת אינטרקציה בין חלבונים.
שאלות פתוחות רבות עדיין קיימות במחקר המבוסס על חלבון ונמצא בפיתוח שיטות לייצור חלבון אופטימלי של צורך חשוב מספר שדות של החקירה. לדוגמה, למרות זמינותו של אלפי הגנום prokaryotic, האיקריוטים1, מפת המתאימה proteomes יחסית לביאור ישירה של מקודדת חלבונים ופפטידים חסר עדיין עבור הרוב הגדול של אורגניזמים. הקטלוג של proteomes מוחלט מתגלה כמטרה מאתגר הדורש מאמץ עצום במונחים של זמן ומשאבים. תקן זהב עבור ביאור ניסיוני נשאר השיבוט כל המסגרות פתח קריאה (ORFs) של הגנום, בבניין שנקרא "ORFeome". בדרך כלל פונקציה ג'ין מוקצה על סמך הומולוגיה ל גנים הקשורים בפעילות ידוע אבל גישה זו היא מדויקת לקוי בשל נוכחותם של רבים ביאורים שגוי הפניה מסדי נתונים2,3,4, 5. יתר על כן, אפילו עבור חלבונים מזוהה, מבואר, מחקרים נוספים נדרשים להשיג אפיון מבחינת שפע, דפוסי ביטוי בהקשרים שונים, כולל מאפיינים מבניים ופונקציונליים כמו גם אינטראקציה עם רשתות.
יתר על כן, מאז חלבונים מורכבים של תחומים שונים, כל אחד מהם מציג תכונות ספציפיות, ותרומה שונה פונקציות חלבון, המחקר ואת ההגדרה המדויקת של תחומים אלה יכול לאפשר תמונה מקיפה יותר, הן ברמה הסינגל ג'ין, ברמת הגנום המלא. כל המידע הנחוץ הזה הופך את חלבון מבוססת מחקר שדה רחב ומאתגר.
בפרספקטיבה הזו, יכול לקבל תרומה חשובה על ידי שיטות לא משוחד ואשר תפוקה גבוהה לייצור חלבונים. עם זאת, ההצלחה של גישות כאלה, לצד ההשקעה ניכרת נדרש, מסתמך על היכולת לייצר חלבון מסיס/יציבה בונה. . זה רב סרן הגבלת גורם שכן ההערכה היא כי רק כ- 30% של חלבונים יכולים להיות בהצלחה הביע והפיק ברמות מספיקות כדי להיות שימושי השפעול6,7,8. גישה זו על מנת להתגבר על מגבלה זו מבוססת על השימוש של DNA מפוצלים באופן אקראי כדי לייצר polypeptides שונים, אשר יחד מספקים קטעים חופפים, ייצוג גנים יחידניים. רק אחוז קטן של שברי DNA שנוצר באופן אקראי הינם פונקציונליים ORFs בעוד הרוב הגדול של אותם שאינם פונקציונליים (בשל נוכחותם של עצירה codons בתוך והרצפים שלהם) או קידוד עבור האו ם טבעי (ORF בתוך מסגרת שונה מהמקור) polypeptides ללא משמעות ביולוגית.
כדי לטפל בכל הנושאים האלה, הקבוצה שלנו פיתחה תפוקה גבוהה חלבון ביטוי ואינטראקציה ניתוח פלטפורמה שניתן להשתמש בהם על11,1210,9,מידה גנומית. פלטפורמה זו משלבת הטכניקות הבאות: 1) שיטה לבחירת אוספים של חלבון מקופל כראוי תחומים מתוך החלק קידוד של ה-DNA מכל יצור; 2 טכנולוגית תצוגה phage) בחירת שותפים של אינטראקציות; 3) המיתרים לחלוטין לאפיין את interactome כל שנבחנה ולזהות השיבוטים של עניין; . ו-4) כלי אינטרנט עבור ניתוח נתונים עבור משתמשים ללא ביואינפורמטיקה או בתכנות לבצע ניתוח Interactome-Seq בצורה קלה וידידותית.
השימוש של פלטפורמה זו מציע יתרונות חשובים על אסטרטגיות אלטרנטיביות של החקירה; מעל כל השיטה זו לחלוטין לא משוחד תפוקה גבוהה, מודולרי למחקר החל גן יחיד עד הגנום כולו. השלב הראשון של הצינור הוא היצירה של ספריה מ- DNA באופן אקראי מפוצלים שנבחנה, המתאפיינת ואז עמוקות המיתרים. ספריה זו נוצרת באמצעות וקטור הנדסה שבו גנים/קטעים מעניינים הם משובטים בין רצף אות על הפרשת חלבון לחלל periplasmic (כלומר, מנהיג שניה) הגן β לקטמאז TEM1. החלבון היתוך להתייעץ אמפיצילין ההתנגדות ואת היכולת לשרוד תחת לחץ אמפיצילין רק אם קטעים משוכפל במסגרת עם רכיבים אלה והן את חלבון כימרי וכתוצאה מכך הוא מקופל כראוי10,13 ,14. שיבוטים כל חולצה אחרי הבחירה אנטיביוטי, שנקרא מסוננים המשובטים", הינם ORFs, רוב גדול של אותם (יותר מ 80 אחוז), נגזרות אמיתי גנים9. יתר על כן, הכוח של אסטרטגיה זו טמון הממצאים כי כל השיבוטים ORF מסונן קידוד חלבונים כראוי מקופל/מסיסים/תחומים15. כמו שיבוטים רבים, נוכח הספריה ואת מיפוי האזור באותו/תחום, יש שונות להתחלה וסיום נקודות, זה מאפשר זיהוי לא משוחדת, צעד אחר צעד של השברים מינימלית כי צפויים להניב מוצרים מסיסים.
הטבה נוספת בטכנולוגיה ניתנת על ידי שימוש המיתרים לאפיין את הספרייה. השילוב של הפלטפורמה ושל כלי אינטרנט מסוימים עבור ניתוח נתונים נותן מידע לא משוחד חשוב על הרצפים נוקלאוטיד המדויק, על מיקומו של ORFs שנבחרו על ההפניה DNA שנבחנה ללא צורך עוד ניתוחים נרחב או מאמץ ניסיוני.
Domainome ספריות ניתן להעביר לתוך הקשר הנבחר, המשמש כמכשיר האוניברסלי לביצוע מחקרים פונקציונלי. תפוקה גבוהה חלבון ביטוי ואינטראקציה ניתוח הפלטפורמה כי אנו משולב, כי קראנו Interactome-Seq מנצל הטכנולוגיה התצוגה phage על-ידי העברת את ORF מסוננים לתוך וקטור phagemid ויצירת phage-ORF ספריה. פעם מחדש משובטים לתוך הקשר התצוגה phage חלבון תחומים מוצגים על-פני M13 חלקיקים; בדרך זו domainome ספריות ניתן לבחור ישירות עבור ג'ין שברי קידוד תחומים עם פעילויות אנזימטיות ספציפיות או מחייב מאפיינים, ומאפשר רשתות interactome פרופיל. גישה זו תוארה לראשונה על ידי. Zacchi et al. 16 ומאוחר יותר השתמש בכמה אחרים בהקשר13,17,18.
לעומת טכנולוגיות אחרות המשמשות ללימוד אינטראקציית חלבון-חלבון (כולל מערכת היברידית שני שמרים ספקטרומטר מסה19,20), אחד היתרונות העיקריים היא ההגברה של הזוג איגוד המתרחשת במהלך phage הצגת מספר סיבובים של בחירה. זה מגביר את הרגישות הבחירה ובכך מאפשר הזיהוי תחומים של איגוד בשפע נמוך חלבונים קיים בספריה. היעילות של הבחירה ביצע עם ספריה ORF-מסוננים הוא גדל עוד יותר בשל היעדר שיבוטים פונקציונלי. בסופו של דבר, הטכנולוגיה מאפשרת את הבחירה לבצע נגד חלבון והן הלא-חלבון פיתיונות21,22,23,24,25.
בחירות phage בעזרת הספריה domainome-phage יכול להתבצע באמצעות נוגדנים מגיע סרה של חולים עם מצבים פתולוגיים שונים, למשל מחלות אוטואימוניות13, סרטן או זיהום מחלות כפיתיון. גישה זו משמשת כדי להשיג שנקרא "נוגדנים החתימה" של המחלה שנבחנה המאפשר לזהות ולאפיין את אנטיגנים/epitopes במיוחד המוכרות נוגדנים של החולים במקביל בנפט. לעומת שיטות אחרות השימוש של phage התצוגה מאפשר זיהוי epitopes antigenic ליניאריות ולא הסתגלותי. הזיהוי של חתימה ספציפי יכול להיות פוטנציאל השפעה חשובה ההבנה פתוגנזה, עיצוב חיסון חדש, זיהוי של מטרות טיפוליות חדשות ופיתוח של כלי האבחון, prognostic חדשה וספציפי. יתר על כן, כאשר המחקר מתמקדת מחלות זיהומיות, היתרון העיקרי הוא הגילוי של חלבונים immunogenic הוא עצמאי מטיפוח הפתוגן.
הגישה שלנו מאשרת כי הכתבים מתקפלים יכולים לשמש בקנה מידה גנומית כדי לבחור את "domainome": אוסף של חלבון מקופל בצורה נכונה, ביטוי טוב, מסיסים תחומים מחלק קידוד ה-DNA ו/או cDNA מכל יצור. פעם מבודד שברי חלבון שימושיים למטרות רבות, מתן מידע נסיוני חיוני עבור ג'ין ביאור גם לגבי לימודי המבנית, נוגדן epitope מיפוי, זיהוי אנטיגן, וכו '. שלמות התרגום של תפוקה גבוהה נתונים שסופקו על-ידי הגדרות המאפשרת הניתוח של דגימות מאוד מורכבים, כגון ספריות התצוגה phage, ומחזיק את היכולת לעקוף האיסוף מפרך מסורתי ובדיקות של שיבוטים phage בודדים שניצלו.
באותו הזמן בזכות התכונות של הספריה מסוננים וכדי את רגישות קיצונית והכוח של ניתוח המיתרים, זה ניתן לזהות את התחום חלבון אחראי ישירות על המסך הראשוני, ללא צורך ליצור אינטראקציה ספריות נוספות עבור כל אחד מחויב חלבון. הגדרות מאפשר להשיג הגדרה מקיפה כל domainome של כל מקור התחלתי genic/גנומית ומאפשרת בכלי האינטרנט של ניתוח נתונים של הקניית ספציפי מאוד אפיון בשני כמותיים נקודת מבט של התחומים של interactome חלבונים.
Protocol
1. בניית הספרייה ORF (איור 1)
- הכנת הכנס ה-DNA
-
הכנה קטעים מ- DNA סינטטיים או גנומית
- תמצית/DNA תוך שימוש בשיטות הרגילות26לטהר.
- קטע דנ א על ידי sonication. אם משתמש של sonicator סטנדרטי, בתור התחלה הצעה כללית עם פולסים s 30 ב- 100% כוח פלט.
הערה: טייס הניסויים צריך להיעשות עם כוח שונים ושעות sonication להציב את התנאים אופטימליים עבור הכנת ה-DNA. אחרי כל מבחן לקבוע את הגודל של שברי DNA על ידי agarose בג'ל. - לטעון את ה-DNA sonicated על גבי ג'ל 1.5% agarose, יחד עם סולם הדנ א bp 100. ביצוע של אלקטרופורזה קצר לרוץ במהירות 5 V ס למשך 15 דקות וחותכים את החלק של הג'ל המכיל את השמצות של ה-DNA מפוצלים.
- לטהר את הדנ א של הוספה עם ערכת חילוץ ג'ל מבוססי עמודה ומדידת הריכוז באמצעות ספקטרופוטומטרים UV.
הערה: לפחות 500 ננוגרם של מוסיף מטוהרים צריכה להתקבל לאחר שלב זה, כדי להיות מאתרים עם µg 1 של וקטור מעוכל, כפי שמתואר בשלב 1.3. בדוק איכות של שברי הכנה על-ידי הערכת /A280nm של260nm, של יחסי230nm /A260nmמאז באיכות נמוכה של המדגם ישפיעו על היעילות מצדו. - לטיפול עד 5 μg של התוספות עם μL 1 של המיקס אנזים ערכת Blunting מהירה, על פי הוראות היצרן. בטל אנזימים על ידי חימום-70 מעלות צלזיוס, במשך 10 דקות דוגמאות שניתן לאחסן ב-20 ° C עד השימוש.
-
שברי הכנה של cDNA
- תמצית ה-RNA בשיטות הרגילות (למשל, באמצעות TRizol או ריאגנטים דומה).
- קטע mRNA על ידי חימום לפני שתבצע שעתוק במהופך. אורך קטע DNA הסופי נשלטת על ידי פריימר אקראי ריכוז וזמן רותחים mRNA. לדוגמה, חום דוגמת עבור 6 דקות ב 95 ° C.
- להכין cDNA באמצעות תחל אקראי עם כל ערכת הזמינים ע פ הפרוטוקול של היצרן.
- לרוקן cDNA של זנב poly-dT על ידי הכלאה עם פולי biotinylated-dA עבור h 3 ב 37 מעלות צלזיוס, ולהפריד על beads מגנטי streptavidin כפי שתואר על ידי. Carninci et al. 13
- לשחזר את החומר לא מאוגד ולטהר עם עמודה מבוסס DNA ערכת טיהור ההוראות של היצרן. למדוד את הריכוז באמצעות ספקטרופוטומטרים UV. ראה הערה בשלב 1.1.1.4.
-
הכנה קטעים מ- DNA סינטטיים או גנומית
- הכנה של וקטור סינון
- µG Digest 5 מטוהרים שיבוט וקטור pFILTER312 עם אנזים הגבלה U של EcoRV 10, הפרוטוקול של היצרן הבא.
- לטעון 2 µL (200 ng) של וקטור מעוכל, יחד עם 100 ננוגרם של הווקטור מעוכל, 1 k bp סמן מולקולרי, על ג'ל 1% agarose, כדי לבדוק אם ולעיכול תקין. חום להשבית את אנזים הגבלה.
- להוסיף נפח 1/10 של 10 x מאגר פוספטאז, µL 1 (5 U) של פוספטאז, דגירה ב 37 מעלות צלזיוס במשך 15 דקות חום בטל במשך 5 דקות ב 65 º C.
- לטהר פלסמיד מתעכל על ידי מיצוי של agarose ג'ל, למדוד את הריכוז באמצעות ספקטרופוטומטרים UV. דוגמאות ניתן לאחסן ב-20 ° C עד השימוש.
- מצדו וטרנספורמציה
- לבצע מצדו כדלקמן: 1 µg של פלסמיד מתעכל להוסיף 400 ננוגרם של phosphorylated מוסיף (פלסמיד: הוספה טוחנת יחס 1:5), µL 10 של 10 x למאגר T4 DNA ליגאז, 2 µL של ריכוז גבוה T4 DNA ליגאז באמצעי אחסון הסופי של µL 100. דגירה התגובה בבית overni 16 ° C . בסדר. חום בטל ב 65 מעלות צלזיוס למשך 10 דקות.
- לזרז את המוצר מצדו על-ידי הוספת 1/10 נפח של סודיום אצטט פתרון (3 מ', pH 5.2) ו- 2.5 בנפחים של 100% אתנול. לערבב, להקפיא ב-80 מעלות צלזיוס למשך 20 דקות.
- צנטריפוגה במהירות המרבית עבור 20 דקות ב 4 º C. למחוק את תגובת שיקוע.
- להוסיף 500 µL של אתנול 70% קר צניפה, צנטריפוגה במהירות המרבית עבור 20 דקות ב 4 º C. למחוק את תגובת שיקוע.
- האוויר יבש בגדר. Resuspend ה-DNA precipitated לתוך 10 µL של מים.
- לבצע אלקטרופורציה תא החיידק.
הערה: השימוש של תאים יעילות גבוהה (מעל 5 x 109 transformants לכל µg של ה-DNA) נדרש. אנו ממליצים להשתמש Escherichia coli DH5αF' (F'/ endA1 hsd17 (rK − ח"כ +) supE44 thi-1recA1 gyrA (Nalr) relA1 (lacZYA-argF) U169 deoR (F80dlacD-(lacZ)M15) מיוצר בבית או רכש של יצרנים שונים.- במקום מספר מתאים של צינורות microcentrifuge, 0.1 ס"מ-אלקטרופורציה וואקום על קרח. להוסיף 1 µL של פתרון מצדו מטוהרים (במים DI) 25 µL של התאים, קפיצי הצינור כמה פעמים.
- להעביר את התערובת DNA-תא cuvette קר, הקש על השיש 2 x, לנגב מים מן החיצוני של cuvette, הכנס הדופק של מודול ולחץ אלקטרופורציה.
- לבצע אלקטרופורציה עם מכונה electroporator רגיל באמצעות 25 µF, Ω 200 ו- 1.8 kV. קבוע הזמן חייב להיות 4-5 ms.
- מיד להוסיף 1 מ"ל של 2xYT נוזלי בינונית ללא כל אנטיביוטיקה, להעביר צינור 10 מ"ל ולאפשר לגדול ב 37 מעלות צלזיוס, רועדת-סל ד 220 לשעה.
- צלחת הפך DH5αF' על 15 ס מ 2xYT פלטות אגר בתוספת 34 כלורמפניקול µg/mL (pFILTER ההתנגדות) ו- µg 25/mL אמפיצילין (סלקטיבית סמן ORFs), דגירה בין לילה ב- 30 ° C.
- צלחת דילולים של הספרייה על 10 ס מ 2xYT אגר צלחות שיושלם עם כלורמפניקול + אמפיצילין ועם כלורמפניקול בלבד, כדי לבצע ספריית טיטור. דגירה בין לילה ב- 30 ° C.
- pFILTER-ORF ספריית אימות
- מבחן 15-20 מושבות של צלחות כלורמפניקול והן כלורמפניקול/אמפיצילין כדי לאמוד התפלגות גודל הוספה. לבחור אחת המושבות עם טיפ, לדלל אותם בנפרד ב- 100 µL 2xYT בינוני ללא אנטיביוטיקה. השתמש µL 0.5 של פתרון זה כתבנית DNA לתגובה PCR, עם כל פולימראז TaqDNA סטנדרטי, ע פ הפרוטוקול של היצרן.
- מבצע 25 מחזורי הגברה באמצעות T חישול של 55 מעלות וזמן הרחבה של 40 s ב- 72 מעלות צלזיוס. פריימר רצפים הינם מסופקים בכל טבלה של חומרים.
- לטעון את המוצרים PCR על 1.5% agarose ג'ל, יחד עם סולם הדנ א bp 100 ולהפעיל.
- אוסף הספריה pFILTER-ORF
- לאסוף חיידקים מן הלוחות 150 מ מ על-ידי הוספת 3 מ"ל של מדיום 2xYT טריים, לקצור אותם עם במגרדת סטרילי, לערבב ביסודיות, להשלים אותם עם 20% גליצרול סטרילי, לאחסן ב- 80 ° C ב- aliquots קטן.
- לטהר פלסמיד הדנ א aliquot אחד של הספרייה (לפני התוספת של גליצרול) באמצעות ערכת חילוץ פלסמיד מבוססי עמודה, ההוראות של היצרן.
- מודדים את הריכוז עם ספקטרופוטומטרים UV. דוגמאות שניתן לאחסן ב-20 ° C עד לשמש הכנה ספריית phagemid ו/או אפיון המיתרים.
2. subcloning של ORFs המסוננות ב וקטור Phagemid (איור 2)
-
הכנת ORF מסונן שברי DNA
- להגדיר את העיכול אנזים הגבלה של 5 µg של וקטור מטוהרים מפני נתיב ספריית pFILTER-ORF הוספת 10 U של BssHII ו המקננת לפי הפרוטוקול של היצרן. בטל את האנזים ואת תקציר עם 10 U של NheI.
- לטעון את ה-DNA מתעכל על 1.5% agarose ג'ל, יחד עם סולם הדנ א bp 100. ביצוע של אלקטרופורזה קצר עולה כ 5 V ס למשך 15 דקות. או מספיק רק להבדיל את השמצות של קטעים נכרת וחותכים את החלק של הג'ל המכילה אותם.
- לטהר את הדנ א של הוספה עם ערכת חילוץ ג'ל בסיס העמודה ומדידת הריכוז באמצעות ספקטרופוטומטרים UV.
-
הכנת phagemid דנ א
- להגדיר את העיכול אנזים הגבלה של 5 µg pDAN5 מטוהרים27 לגבי התוספות.
- לטהר פלסמיד מתעכל על ידי פועל דנ א מתעכל על 0.75% agarose ג'ל ולחלץ מ ג'ל עם ערכת מבוססי עמודה.
- למדוד את הריכוז באמצעות ספקטרופוטומטרים UV. דוגמאות ניתן לאחסן ב-20 ° C עד השימוש.
-
ספריית מצדו, שינוי, אוסף
- לבצע מצדו וטרנספורמציה כמתואר עבור וקטור pFILTER.
- צלחת הפך DH5αF' על 150 מ מ פלטות אגר 2xYT בתוספת 100 אמפיצילין µg/mL, דגירה בין לילה ב- 30 ° C.
- צלחת דילולים של הספרייה על 100 מ מ- 2xYT-אגר-צלחות בתוספת 100 אמפיצילין µg/mL כדי לקבוע גודל הספרייה.
- מבצע אימות לספריה על-ידי ה-PCR של שיבוטים שנבחרו באופן אקראי כפי שמתואר בשלב 1.4.
- לאסוף את הספריה phagemid-ORF בבציר חיידקים מן צלחות 150 מ מ, לערבב ביסודיות, להשלים אותם עם 20% סטרילי גליצרול וחנות ב-80 מעלות צלזיוס ב aliquots קטן.
- לטהר פלסמיד ה-DNA של aliquot אחד של הספרייה באמצעות ערכת חילוץ פלסמיד מבוססי עמודה, ההוראות של היצרן.
- למדוד את הריכוז-ספקטרופוטומטרים UV. דוגמאות שניתן לאחסן ב-20 ° C עד לשמש אפיון על-ידי הגדרות.
3. הליך בחירה והכנה של ספריית phage
- Phage הפקה
- לדלל עם aliquot מלאי של הספרייה phagemid לתוך 10 מ ל מרק נוזלי 2xYT בתוספת אמפיצילין µg/mL 100 על מנת יתר של600nm = 0.05.
- מגדל הספרייה מדולל בבקבוקון סטרילי 5 - 10 פעמים גדול יותר האחסון המקורי, ב 37 מעלות צלזיוס ברעידות-220 סל"ד עד מגיע לקבל מנת יתר-600nm = 0.5.
- להדביק חיידקים עם העוזר phage (למשל M13K07)-ריבוי של זיהום 20:1. יוצאים ב 37 מעלות צלזיוס במשך 45 דקות עם עצבנות ומדי פעם (כל 10 דקות).
- צנטריפוגה חיידקים ב 4000 g x 10 דקות בטמפרטורת החדר. למחוק את תגובת שיקוע, מחדש להשעות חיידקים גלולה ב- 40 מ ל מרק נוזלי 2xYT בתוספת אמפיצילין µg/mL 100 ו- 50 µg/mL kanamycin ולגדול ב 28 ° C ברעידות-220 סל"ד נלון.
- ביום לאחר צנטריפוגה חיידקים ב 4000 x g למשך 20 דקות ב 4 º C. לאסוף את תגובת שיקוע המכיל phages.
- פג-משקעים של phages.
- להוסיף נפח 1/5 מיקרומטר 0.22 מסוננים פג/NaCl פתרון (w 20% / פג 6000, v 2.5 M NaCl) phages שנוקה, דגירה על קרח למשך 30-60 דקות.
הערה: פתרון הפכה מעושן לאחר כמה דקות, המציין של משקעים phage מוצלחת. עכירות של הפתרון יגדל לאורך זמן הדגירה. - צנטריפוגה ב 4000 x g למשך 15 דקות ב 4 º C. גלולה קטנה לבן של phages יהוו.
- Resuspend זה ב 1 מ"ל של PBS סטרילי. להעביר צינור 1.5 mL, צנטריפוגה ב 4 מעלות צלזיוס למשך 10 דקות במהירות המרבית כדי להסיר חיידקים מזהם. גלולה חום יהוו.
- העברת תגובת שיקוע המכיל phages על צינור. שמור phages על הקרח עבור טיטור ובחירת phage רצופים.
- להוסיף נפח 1/5 מיקרומטר 0.22 מסוננים פג/NaCl פתרון (w 20% / פג 6000, v 2.5 M NaCl) phages שנוקה, דגירה על קרח למשך 30-60 דקות.
- Phage טיטור
- להכין דילולים טורי של הפתרון phage. לשים 10 µL של הפתרון phage 990 µL ל- PBS להשיג דילול-2 10. לדלל שוב בתכשיר לעשות 10-4 , מזה לקבל 10-6 לדילול.
- לגדול DH5αF "תאי חיידקים לתוך 2xYT בינוני נוזלי ב 37 ° C עם לרעוד עד OD600nm = 0.5 מתמלאת. להעביר 1 מ"ל של החיידק מוכן לתוך צינור 1.5 mL ולהדביק מיד עם µL 1 של דילול phage 10-4 . דגירה מבלי לרעוד ב 37 מעלות צלזיוס במשך 45 דקות חוזר באותו נוהל דילול-6 10.
- צלחת דילולים של החיידק נגוע לתוך צלחת 2xYT 100 מ מ. לשים את הצלחת ב 30 מעלות צלזיוס למשך הלילה.
- צלחת µL 100 של DH5αF נגוע ' על צלחת אגר 2xYT בתוספת 100 אמפיצילין µg/mL לבדוק העדר של זיהום בהכנה.
- יום אחרי לספור את מספר מושבות ולחשב את כייל phage. אקספרס כייל נוגדנים כמספר של phages/mL. כייל הצפוי הוא 1012-13 phages/mL.
- Phage בחירה
-
Phage בחירה באמצעות כפיתיון נוגדנים מטוהרים
- עיתוני phages על ידי µL 200 המדללת phage הכנה אל אמצעי אחסון שווה של חלב כחוש PBS - 4%, תקופת דגירה של h 1 בטמפרטורת החדר סיבוב איטי. שלב זה מאפשר חסימה של phages עבור איגוד לא ספציפי. להעביר 30 µL של חלבון G beads מגנטי מצופה צינור 1.5 מ.
- לשטוף פעמיים כדלקמן: להוסיף 500 µL ל- PBS, דגירה על הגלגל ב סיבוב איטי למשך 2 דקות בטמפרטורת החדר, לצייר את החרוזים לצד אחד של הצינור באמצעות מגנט ולהסיר את תגובת שיקוע.
- דגירה phages רוויים עם חרוזים שטף למשך 30 דקות בטמפרטורת החדר עם סיבוב איטי.
- לצייר את החרוזים לצד אחד באמצעות שדה מגנטי. לאסוף את תגובת שיקוע המכיל phages כדי לשמש עבור השלב הבחירה.
- להכין beads מגנטי בעת ביצוע השלב הקודם, על-ידי conjugating הנוגדנים מטוהרים. לשטוף 30 µL של חלבון G beads מגנטי מצופה כמתואר לעיל. לדלל 10 µg של נוגדנים מטוהרים ב µL 500 ל- PBS, להוסיף החרוזים שטף, דגירה הסיבוב איטי בטמפרטורת החדר במשך 45 דקות לשטוף פעמיים עם PBS.
הערה: בצע שני תכשירים שונים של beads מגנטי: אחת עם נוגדנים עניין ואחת עם שליטה נוגדנים, למשל, נוגדנים מטוהרים מתורמים מאוד בריאים. הרצף של אנטיגנים שנבחרו באמצעות שליטה נוגדנים אינם מופחתים במהלך השלב ניתוח של התוצרים. לחלופין, ניתן להשתמש beads מגנטי נטען עם שליטה נוגדנים לביצוע שלב קדם ניקוי כלשהו phages (בצע את פרוטוקול הדגירה עם חרוזים מצומדת האו ם). - Phage בחירה: לצייר חרוזים לצד אחד של הצינור באמצעות מגנט, להסיר בכביסה האחרונה, להוסיף phages, דגירה עם סיבוב איטי בטמפרטורת החדר במשך 90 דקות לשטוף 5 פעמים עם 500 µL של PBS-0.1% Tween-20, 5 פעמים עם PBS.
- Elute phages מאוגדים, המייצג את הפלט של הבחירה, על ידי ערבוב את החרוזים עם 1 מ"ל של DH5αF' התאים גדל OD600 = 0.5. דגירה חיידקים עם חרוזים למשך 45 דקות ב 37 ° C עם פה ושם רועד (כל 10 דקות). צלחת הפלט על צלחת אגר 2xYT 150 מ מ בתוספת 100 אמפיצילין µg/mL.
- צלחת 100 µL של מדולל ושל דילולים שונים של הפלט (10-1 כדי 10-5) כדי לבצע טיטור. יום אחרי לאסוף חיידקים מן הלוחות 150 מ מ על-ידי הוספת 3 מ"ל של מדיום 2xYT טריים, לקצור אותם עם במגרדת סטרילי, לערבב ביסודיות, להשלים אותם עם 20% גליצרול סטרילי, לאחסן ב- 80 ° C ב- aliquots קטן.
- לגדול aliquot אחד שוב לבצע שנייה של בחירה. חזור על כל ההליך השתוותם כמתואר לעיל למעט התנאים כביסה. לשטוף במקרה זה 10 פעמים עם PBS - 1% Tween-20 (שופכים פתרון בצינור, שופכים שוב מיד). לאחר מכן להוסיף 500 µL ל- PBS, דגירה בסבב בטמפרטורת החדר במשך 10 דקות לבצע אחרים שוטף 10 עם PBS. להמשיך עם הצעד • תנאי באשר בסיבוב הראשון של בחירה.
- לחלץ פלסמיד DNA מ- aliquot אחד של הפלט באמצעות ערכת מבוססי עמודה, בעקבות הוראות היצרנים. חנות פלסמיד ב-20 ° C עד שהוא ישמש עבור רצף עמוק.
-
הבחירה phage באמצעות חומרים פיתיון
- עיתוני phages על ידי µL 200 המדללת phage הכנה אל אמצעי אחסון שווה של חלב כחוש PBS - 4%, תקופת דגירה של h 1 בטמפרטורת החדר סיבוב איטי.
- להוסיף 100 µL של streptavidin beads מגנטי. תקופת דגירה של h 1 בטמפרטורת החדר כדי לבחור מחייב streptavidin phages. הסר את phages streptavidin-מחויב על ידי ציור החרוזים לצד אחד באמצעות מגנט. לקחת supernatant מהשלב הקודם להוסיף חלבון biotinylated (בריכוז של 100-550 ננומטר), דגירה על הרוטור בטמפרטורת החדר למשך 30 דקות כדי 1 h.
- להכין beads מגנטי: בעת ביצוע השלב הקודם, לשטוף µL 100 של חרוזים streptavidin-מגנטי עם PBS, resuspend ב- PBS 2% חלב כחוש, דגירה עם סיבוב בטמפרטורת החדר למשך 30 דקות כדי 1 h.
- Phage בחירה: לצייר חרוזים לצד אחד של הצינור באמצעות מגנט, להסיר חלב PBS - 2%, resuspend חרוזים במיקס phage-חלבון. דגירה עם סיבוב איטי בטמפרטורת החדר במשך 90 דקות.
- לצייר חרוזים לצד אחד של הצינור באמצעות מגנט, למחוק את תגובת שיקוע ולשטוף אותם בקפידה חמש פעמים עם µL 500 ל- PBS 0.1% Tween-20. לבצע • תנאי כפי שמתואר בהפעלה הקודמת.
-
Phage בחירה באמצעות כפיתיון נוגדנים מטוהרים
4. phage ספריית רצף עמוק פלטפורמה (איור 3)
-
דנ א מוסיף ההתאוששות מן pFILTER-ORF-ספרייה, pDAN5-ORF-ספרייה או נבחר-phage-ספריות
- להפשיר את אחד aliquot של הספרייה, לכמת את זה על-ידי שימוש ספקטרופוטומטרים, לשחזר מוסיף DNA על-ידי הגברה עם תחל ספציפיים.
הערה: צבעי בסיס המשמש כדי להציל את הכיסויים מקושרים בסוף 5' כדי רצפים מתאמים, ובכך מאפשר בניית אינדקס רצופים של הבריכות אמפליקון שהושג וחילצנו רצף ישירה של התוספות הדנ א באמצעות סקוונסרים. הרצף שלהם הוא טבלה של חומרים. מתאמים הם הצביעו על מודגש, תחל ספציפיים יצוינו בכתב נטוי. - השתמש µL 2.5 של הספרייה (pFILTER/phagemid/נבחר-phage) כתבנית DNA לתגובה PCR.
- השתמש התוכנית הבאה: 95 מעלות צלזיוס למשך 3 דקות; 25 מחזורי 95 ° C ל 30 s, 55 ° C ל 30 s, 72 ° C ל 30 s; 72 מעלות צלזיוס במשך 5 דק להחזיק על 4 מעלות צלזיוס.
הערה: בשלב זה מומלץ לפעול µL 1 של המוצר PCR Bioanalyzer או TapeStation כדי לוודא שגודל amplicons ולבדוק כי הם בטווח הנכון.
- להפשיר את אחד aliquot של הספרייה, לכמת את זה על-ידי שימוש ספקטרופוטומטרים, לשחזר מוסיף DNA על-ידי הגברה עם תחל ספציפיים.
-
ניקיון PCR
- להביא את החרוזים מגנטי (למשל, AMPure) בטמפרטורת החדר. העברה המוצר PCR מהצינור PCR צינור 1.5 מ. מערבולת beads מגנטי עבור 30 s כדי לוודא החרוזים מפוזרים באופן אחיד. הוסף 20 µL של beads מגנטי כל שפופרת המכילה את המוצר PCR, מיקס על ידי בעדינות pipetting. דגירה בטמפרטורת החדר בלי טלטולים למשך 5 דקות.
- מניחים את הצלחת על דוכן מגנטית למשך 2 דקות, או עד ניקה תגובת שיקוע. עם המוצרים PCR על הדוכן מגנטי, להסיר ולמחוק את תגובת שיקוע.
- לשטוף את החרוזים עם אתנול 80% טריות, עם המוצרים PCR על הדוכן מגנטי, כדלקמן: להוסיף 200 µL של אתנול 80% טריות כל מדגם טוב; דגירה את הצלחת על הדוכן מגנטי עבור 3 s; בזהירות להסיר ולמחוק את תגובת שיקוע.
- לבצע שטיפה אתנול השני, עם המוצרים PCR על הדוכן מגנטי; בסוף שטיפת השני בזהירות להסיר כל האתנול ולאפשר את החרוזים מילה נהדרת עבור 10 דקות.
- להסיר את המוצרים PCR מן הדוכן מגנטי, להוסיף µL 17.5 של 10 מ מ טריס pH 8.5 כל שפופרת, בעדינות פיפטה למעלה וודא למטה 10 פעמים, חרוזים הם resuspended באופן מלא. דגירה בטמפרטורת החדר למשך 2 דקות.
- למקם את הצינורית על הדוכן מגנטית למשך 2 דקות או עד ניקה תגובת שיקוע, להעביר בזהירות µL 15 של תגובת שיקוע המכיל את המוצרים PCR מטוהרים צינור 1.5 מ. לאחסן את המוצרים PCR מטוהרים ב-15 מעלות צלזיוס ל-25 מעלות צלזיוס במשך שבוע, אם אתה לא מיד להמשיך ל- PCR מדד.
-
אינדקס ה-PCR
הערה: לאחר ה-PCR לנקות, לבצע PCR מדד. להשתמש בערכה Nextera XT אינדקס; לכן זה ניתן יהיה רצף של ספריות אינדקס כפול וכתוצאה מכך בתוך מרובבת אילומינה פועל.- תעביר כל μL 15 המכיל כל מוצר מטוהרים לתוך צינור PCR חדש ולהגדיר את התגובה הבאה המכיל: 15 µL של המוצר אמפליקון מטוהרים, 5 µL של אינדקס פריימר 1 ו- 5 µL של אינדקס פריימר 2, 25 µL של תערובת x PCR 2; נפח 50 µL הסופי.
- לבצע PCR הצנטרפוגה תרמי באמצעות התוכנית הבאה: 95 מעלות צלזיוס למשך 3 דקות, 8 מחזורים של 95 ° C ל 30 s, 55 ° C ל 30 s, 72 ° C ל 30 s; 72 מעלות צלזיוס למשך 5 דקות, ואז תחזיק ב 4 º C.
-
PCR לנקות 2
- בצע באותו פרוטוקול המתואר בסעיף 4.2 עבור ה-PCR לנקות עם השינויים הבאים: בשלב הראשון להוסיף 56 µL של beads מגנטי כל µL 50 של מוצר ה-PCR.
- Resuspend את החרוזים ב- 27.5 µL של 10 מ מ טריס pH 8.5 בשלב הסופי של טיהור ולהעביר 25 µL צינור (זוהי הספרייה הסופי מטוהרים מוכן כימות ולאחר מכן קביעת רצף).
- אחסן את הצלחת ב-15 מעלות צלזיוס ל-25 מעלות צלזיוס במשך שבוע אם לא מתקדם אל ספריית כמת.
-
הערכה כמותיים של הספרייה רצף
- לאחר טיהור, לפעול µL 1 של 1:10 דילול של הספרייה הסופי על bioanalyzer כדי לוודא את הגודל ולכמת זאת בחירת האזור של המעקב ספריית הסופי.
- במקביל לבצע כימות הספרייה באמצעות PCR בזמן אמת באמצעות ערכת כימות ספריה לכל הפרוטוקול של היצרן.
-
ספריות רצף
- מאגר של ספריות אינדקס כפול הפיק יחד עם ספריות אחרות רצף אינדקס כפול. רצף שכזה של הספרייה על ידי יצירת זמן קורא, לפחות 250 bp סוף לזווג באמצעות הן את Hiseq2500 או כלי הנגינה MiSeq כדי להשיג את קריאות PE 250bp המקרה הראשון, לחץ דם המקרה 300 השני PE קורא.
5. ניתוח נתונים Bioinformatic באמצעות הכלי Web Interactome-Seq
- לנתח את קריאות שמקורם pFILTER/phagemid/נבחר-phage ספריית רצף עם הצינור ניתוח נתונים Interactome-seq. הכלי באינטרנט זמין באופן חופשי בכתובת: http://interactomeseq.ba.itb.cnr.it/
תוצאות
גישת הסינון הוא schematized באיור1. ניתן להשתמש בכל סוג של דנ א intronless. איור 1A החלק הראשון של הגישה סינון מיוצג: לאחר טעינה של ג'ל agarose או bioanalyzer, פיצול טוב של ה-DNA של הריבית מופיע כתם של קטעים עם התפלגות האורך בגודל הרצוי של לחץ דם 150-750. תמונת הנציגה ג'ל וירטואלי של ה-DNA מפוצלים שהושג ניתנת. שברי טעון על הג'ל agarose מכן הבריא, תוקן קצה, phosphorylated, ואז משובטים לתוך וקטור pFILTER בעבר קהות ליצור ספריה שברי DNA אקראי. מבצע כל שלב של ההליך שיבוט בתנאים מיטביים נדרש כדי לקבל איכות טובה ספריה עם כיסוי מוחלט של ה-DNA במחקר.
איור 1B גישת הסינון מיוצג: הספרייה הוא גדל בנוכחות כלורמפניקול (הצומחת pFILTER) לבד או כלורמפניקול אמפיצילין לבחירה המכילות ORF מושבות. מושבות היחיד שיש קטע DNA המקביל ORF לייצר פונקציונלי של β לקטמאז ולשרוד כאשר הבחירה אנטיביוטי קיים. איור 1C מראה איך הגדלת הלחץ הסלקטיבי מאפשר בחירה של התיקיה טוב ORFs נגד עניים תיקיות אלה. התוצאה הצפויה היא ירידה של גודל הספרייה של 20-fold. מספר גבוה יותר של שיבוטים ששרד מציין לא מספיקות הלחץ הסלקטיבי.
ORF קטעים שניתן יהיה לשחזר בקלות מן הספריה מסוננים ליישום הבאים; ללימודי אינטראקציה האסטרטגיה שלנו מנצל phage טכנולוגיית התצוגה. איור 2, מיוצגים השלבים העיקריים של בניית ספריה phage: ספריה נאותה מוכן על ידי חיתוך מתוך שברי מסוננים וקטור pFILTER, שיבוט מחדש לתוך פלסמיד phagemid בפיוז'ן עם רצף קידוד phage capside חלבון g3p. לאחר נגוע phage המסייע, הנוכחות של וקטור לתאי חיידקים מאפשר הייצור של חלקיקים phage הצגת מוצרים פיוז'ן ORF-g3p על פני השטח שלהם ובכך הספריה מסוננים זמינה לבחירה התצוגה phage ויותר ניתוח.
כל ספריות עמוקות מנותחים על-ידי הגדרות, כמו גם התוצרים של הבחירות phage, כפי שמוצג בחלק השני של איור 3. שברי DNA חולצו מן גידול. המושבות על-ידי ה-PCR הגברה עם oligonucleotides ספציפיים חישול עמוד שידרה פלסמיד. וממשיך מתאמי ספציפי עבור הרצף. המיתרים מבוצעת, קריאות הם מכן נותחו באמצעות הכלי web Interactome-Seq ניתוח נתונים.
באיור 4 דיווחנו ייצוג סכמטי של ההליך הבחירה של ספריית התצוגה המסוננת phage ORF. הבחירה בדוגמה זו מתבצע באמצעות נוגדנים נוכח sera מחולים מושפע הלקויות השונות (קרי זיהומיות פתולוגיות, פתולוגיות אוטואימונית, סרטן). במקרה זה הספרייה phage אינטראקציה ישירות עם נוגדנים הנוכחי סרה המטופלים, בדרך זו יכול להיות מועשר בשם אנטיגנים ספציפיים כי הם מוכרים על ידי נוגדנים ספציפיים למחלה. בסוג זה של הניסוי, בדרך כלל הספרייה נבחר גם באמצעות שליטה סרה מחולים בריא כדי לקבל אות רקע שישמש עבור תהליכי השוואה ונורמליזציה רצופים.
בחירות מבוצעות באמצעות סרה מאותו סוג? של מטופלים בדרך כלל יקובצו לתוך בריכות שונות על מנת להפחית את ההשתנות הבין-אישי של sera נוגדנים כייל נוגדנים. בכל בריכה משמש באופן עצמאי עבור שלושה סבבים רצופים של בחירה, להעשיר את הספרייה החיסון-תגובתי שיבוטים ספציפיות עבור פתולוגיה שנבחנה. בדיקת נוגדנים קבע מודגרת עם ספריית phages, החיסון-מתחמי התאוששו על ידי חלבון A מצופה magnetics-חרוזים, phages מאוגדים הם eluted על ידי הליכים סטנדרטיים. מחזורי הבחירה מבוצעים עם הגדלת שטיפה ואיגוד לכתחילה.
קריאות שנוצרו על-ידי הגדרות ניתן לנתח באמצעות הכלי web Interactome-Seq שפותחו במיוחד כדי לנהל נתונים מסוג זה. Interactome-Seq נתונים ניתוח זרימת עבודה מורכבת של ארבעה שלבים רציפים ש, החל מ- raw רצף קריאות, יוצר הרשימה של תחומים בשם עם ביאורים גנומית (איור 5A). בשלב הראשון קלט (איור 5A - התיבה האדומה), Interactome-Seq בודק אם הקבצים קלט (raw קריאות, הפניה רצף הגנום, ביאור רשימה) מעוצבים כראוי. בשלב השני PREPROCESSING (תיבת5A איור - כתום), באיכות נמוכה רצף נתונים נסגרים קודם באמצעות Cutadapt28 תלוי באיכות ציונים, מושמטות קריאות עם פחות מ- 100 בסיסים אורך. בשלב יישור לקרוא הבא (איור 5A - ירוק תיבת), קריאות הנותרים מיושרים עם blastn29 רצף הגנום ומאפשר עד 5% של אי התאמות. קובץ סם מופק וקורא רק עם ניקוד איכות גדול מ- 30 (Q > 30) מעובדים באמצעות SAMtools30 ו להמיר קובץ בם. לאחר יישור, Interactome-Seq מבצעת זיהוי תחומים (איור 5A - הקופסה הכחולה), הפעלת Bedtools31 לסינון קורא חופפים לפחות 80% של אורכם בתוך התמלילים; כיסוי, מקס עומק וערכים המוקד מחושבים ואז עבור כל חלק ORF מכוסה על-ידי מיפוי קריאות. הכיסוי מייצג את המספר הכולל של קריאות שהוקצו גן; העומק הוא המספר המרבי של קריאות מכסה חלק genic מסוים; המוקד הוא אינדקס המתקבל היחס בין מקס עומק כיסוי ולאחר זה יכול לנוע בין 0 ל-1. כאשר המוקד נמצא גבוה יותר מאשר 0.8 הכיסוי הוא גבוה יותר מאשר הכיסוי הממוצע שנצפו עבור כל מיפוי אזורים בקובץ ה-בום, החלק תקליטורים מסווגים בשם קבוצת המחשבים/epitope. השלב האחרון של הצינור Interactome-Seq הפלט (איור 5A - קופסא סגולה), רשימה של תחומים בשם נוצר בתבנית טבלאית מופרדים. הצינור Interactome-Seq נכללה כלי אינטרנט כדי לאפשר למשתמשים ללא ביואינפורמטיקה או בתכנות כדי לבצע ניתוח Interactome-Seq באמצעות ממשק גרפי וכדי להשיג את התוצאות שלהם בתבנית קלה וידידותית. כפי שמוצג באיור 5B, מוצגות התוצאות של ניתוח באמצעות JBrowse32 כדי לאפשר ויזואליזציה וחקירה. Interactome-Seq יוצר רצועות בדפדפן הגנום המתאים לתחומים בשם זוהה, ומספק גם מהדיאגרמות קלאסית להראות הצטלבויות בין תחומים בשם נפוץ מועשר לדוגמה בניסויים בחירות שונות.

איור 1: סקירה סכמטי של השלבים העיקריים להקמת הספרייה ORF-סינון
א) ה-DNA ממקור שונה sonicated, מקוטעת לתוך קטעים אקראיים באורך לחץ דם 150-750. שברי התאוששה ג'ל, משובטים כמו קהה לתוך וקטור pFILTER; B) סינון שלב באמצעות β לקטמאז ככתב מתקפלים. וקטור המכיל קטעים ORF לא נבחרו באופן שלילי לוקחת אמפיצילין תוך ORF שברי משובטים לאפשר מושבות לגדול; ג) יישום לחץ סלקטיבי גובר (אמפיצילין בריכוז בתקשורת גידול מוצק בין 0 ל- > 100 μg/mL) לאפשר בחירה שברי מקופלת כדאי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2: סקירה סכמטי של השלבים העיקריים להקמת הספרייה phage
א) שברי ORF-מסוננים הם לגזור מן הווקטור מסונן באמצעות אנזימי הגבלה ספציפית. לאחר השחזור, טיהור, שברי הם משובטים לתוך וקטור phagemid והפך; B) ספריית חיידקי phagemid נגוע phage עוזר בנוסף, לאחר צמיחה לילה, phages הם פג-זירז ומאופקים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3: ספריות ORF רצף
רצף מתבצע על שני המקורי ORF שנבחר הספרייה, כמו גם על הספריה התצוגה phage; 1)-בשני המקרים התאוששו מושבות גדל של ה-DNA; 2) שברי DNA התאוששו על ידי הגברה באמצעות תחל ספציפיים הקשורים מתאמים עבור רצף; 3-4) האם התאושש ועמוק וסודרו באמצעות הגדרות; 5) הנתונים מנותחים באמצעות הצינור Interactome-Seq. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
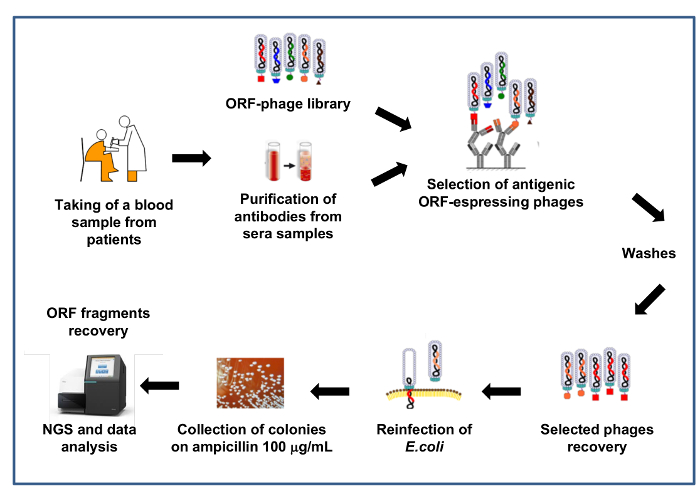
איור 4: סקירה סכמטי של ספריית בחירה באמצעות נוגדנים המטופלים
ספריית phage משמש עבור הבחירה נגד נוגדנים מן סרה המטופלים. נוגדנים הם ותשמרו על beads מגנטי phage ספריית לכידת/הבחירה מבוצעת, בשלושה מחזורים ממכוני מבוצעות, לאחר מכן phages שנבחר התאושש ומשמש להדביק מחדש e. coli. מחדש נגוע e. coli תאים הם מצופה בהלחץ הסלקטיבי (אמפיצילין 100 μg/mL). ORF שברי התאוששו על ידי הגברה, בריכות אמפליקון ואז הם וסודרו על-ידי הגדרות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
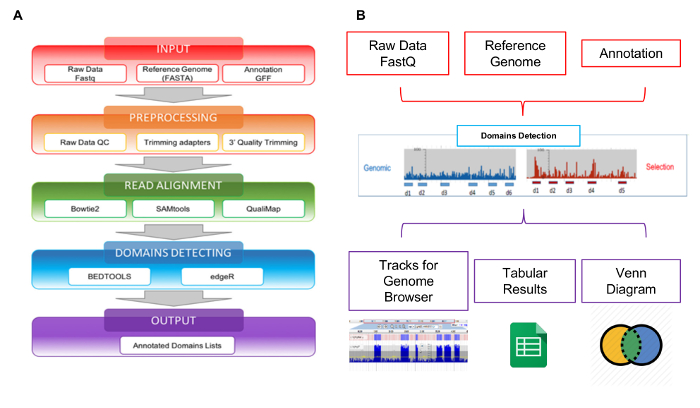
איור 5: סקירה סכמטי של ספריית ניתוח
א) ייצוג נתונים ניתוח זרימת העבודה, החל מ- FASTQ קבצי raw של הרשימות תחומים המבואר הסופי; ב) ייצוג סכמטי התשומות והתפוקות של הכלי web Interactome-Seq. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
הקמת ספרייה באיכות גבוהה המסוננות ORFs מגוונת מאוד הוא השלב הקריטי הראשון כל התהליך מאז זה ישפיע על כל השלבים הבאים של הצינור.
יתרון תכונה חשובה של השיטה שלנו היא כי כל מקור ה-DNA (intronless) (cDNA, הדנ א, ה-PCR נגזר או דנ א סינתטי) מתאים ספריית הבנייה. הפרמטר הראשון שצריך לקחת בחשבון הוא כי האורך של שברי DNA משובטים לתוך וקטור pFILTER צריך לספק ייצוג של האוסף כולו קבוצות המחשבים של גנום או של transcriptome, שנקרא "domainome". הראו כי תחומים חלבון יכול להיות משובטת בהצלחה, נבחר, מזוהה סוף סוף החל משברי DNA אורך ההפצה המתפרסות על-פני מ 150 ל bp 75033,34, וזה עולה בקנה אחד עם מה הוא דיווח ב הספרות מראה כי רוב תחומי חלבון הם באורך 100 aa (עם טווח שבין 50 ל-200 aa)15.
DNA מתחיל חומר חייב להיות מקוטעת לתוך הטווח גודל של בחירה ולאחר מכן משובטים לתוך הווקטור סינון12 (pFILTER). במהלך שלבים אלה, הטיה פוטנציאליים יוכלו להימנע למקסם את היעילות של כל השיבוט צעדים תגובות הכלולים הפרוטוקול, קטע מסוים סוף תיקון, זרחון. הכנת וקטור הוא מאתגר, צריכה להיעשות בתנאים אופטימליים, כדי להימנע הן פלסמיד השפלה ו/או זיהום על ידי וקטור מעוכל.
לאחר הספרייה נוצר, זה "לסנן" על מנת לשמר רק שברי מקופלת ORFs. פרמטר מפתח כדי לווסת את שלב זה הוא סלקטיבי הלחץ המופעל שניתן לשנות בהתאם לכתחילה של הסינון הרצויה. הבחירה מתבצעת באמצעות אמפיצילין: ריכוז גבוה יותר משתמשים, נמוך יותר מספר מושבות חיידקים טרנספורמציה מסוגל לשרוד. זה משקף את היכולת של פעולת סינון כדי לבחור טוב-מול העניים תיקיה ORFs34. הפחתה זו במספר של שיבוטים מאוזנת על ידי גידול קיפול מאפיינים של קטעים נבחרים. בדרך כלל, הריכוז אמפיצילין צריך להיות מספיק כדי לצמצם את 1/20 מספר מושבות חיידקים ביחס אלה יכול להיות מושגת גדל הספרייה על כלורמפניקול בלבד.
ספריית אימות מתבצע בדרך כלל באמצעות PCR הגברה של מושבות שנבחרו באופן אקראי ורצף שלהם. PCR הגברה של כמה מושבות המוצע על מנת לקבל את הערכה מהירה של האיכות של הספרייה: אורך השרוול צריך להיות בטווח הצפוי של 150-750 bp ועל מושבות שונות צריך מוסיף הנוכחי עם שונים בגודל טוב אנלוגיים ספריית הכנה לטווח של השתנות. אסטרטגיה זו המקובלת של ההקרנה, כאשר מורחים השיטה היחידה עבור ספריית אימות, אינה מקיפה, והוא זמן רב, המאפשר הניתוח של רק מספר מצומצם של המושבות ויש סיכוי גבוה של חסרים רוב שיבוטים חשוב. הגישה שלנו מבוסס על רצף עמוק של הספרייה, זה מספק מידע מלא על ספריית גיוון, שפע, מיפוי מדויק של כל אחד מהשברים שנבחרו.
היישום של טכנולוגיה המיתרים עם הגישה סינון מגדילה את עומק של הניתוח במספר סדרי גודל. לאחרונה, יש הפרוטוקול עבור קביעת רצף של ספריות ORF באמצעות פלטפורמת אילומינה ממוטב, ואנו פיתח כלי אינטרנט מסוימים עבור ניתוח נתונים שעושה הניתוח של אלה סוג הנתונים עבור כל משתמש ללא כל ביואינפורמטיקה יכולת תיכנות.
הספרייה "כשלעצמה" הוא "כלי אוניברסלי", שעלולים להיות מנוצלים לרעה בהקשרים שונים עבור ביטוי חלבון ו/או הבחירה. הגישה מתודולוגי שלנו מבוססת על העברת ORFeome המיוצר לתוך הקשר התצוגה phage. שברי חלבון באים לידי ביטוי על פני השטח phage והפך מתאים הבחירה הבאים.
זה עשוי להציל את ORFs מסוננים מהספריה pFILTER על ידי עיכול עם אנזימי הגבלה ספציפית, מחדש משבטים אותם לתוך וקטור phagemid תואם המאפשר שלהם פיוז'ן עם g3p חלבון phage.
לאחר יצירת ספריית phagemid-ORF, זה יכול לשמש עבור הבחירה נגד מטרות שונות, כגון חלבון בשם איגוד10 או נוגדנים מטוהרים35,36 כפי שמתואר כאן. מאז phage חלקיקים יציג על פני השטח ORFs מסוננים, התוצאה הליך בחירה הרבה יותר יעיל עקב העדרו של אי-הצגת שיבוטים שבדרך כלל לעקוף אותו.
לאחר הבחירה של הספרייה ORF phage התצוגה, שיבוטים פלט יכול להיות רציף וניתח עם הצינור אותו. המיתרים יכולה לספק פתרון מלא סטטיסטית הבכיר ביותר לעתים קרובות שנבחרו ORFs, זה מאפשר הזיהוי של החלבונים בעיקר אינטראקציה עם הפיתיון בשימוש. לאור נוכחותם של גרסאות רבות ומגוונות לכל תחום שונות על ידי כמה חומצות אמינו, החפיפה בין שיבוטים ברצף שונה גם מזהה את שבר/התחום המינימלי מציג את מאפייני האיגוד. בסופו של דבר, הודות המושבים של גנוטיפ פנוטיפ מידע לתוך הספריה phage, ברגע התחומים של הבחירה זוהו, רצף הדנ א יכול בקלות להינצל מהספריה עוד מחקרים במבחנה , בתוך vivo אימות ואפיון.
Disclosures
המחברים אין לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי מענק האיטלקי משרד החינוך ואוניברסיטת (2010P3S8BR_002 כדי CP).
Materials
| Name | Company | Catalog Number | Comments |
| Sonopuls ultrasonic homogenizer | Bandelin | HD2070 | or equivalent |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | or equivalent |
| GeneRuler 1 kb DNA Ladder | Thermo Fisher Scientific | SM0311 | or equivalent |
| Molecular Biology Agarose | BioRad | 161-3102 | or equivalent |
| Green Gel Plus | Fisher Molecular Biology | FS-GEL01 | or equivalent |
| 6x DNA Loading Dye | Thermo Fisher Scientific | R0611 | or equivalent |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | or equivalent |
| Quick Blunting Kit | New England Biolabs | E1201S | |
| NanoDrop 2000 UV-Vis Spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368813 | |
| Streptavidin Magnetic Beads | New England Biolabs | S1420S | or equivalent |
| QIAquick PCR purification Kit | Qiagen | 28104 | or equivalent |
| EcoRV | New England Biolabs | R0195L | |
| Antarctic Phosphatase | New England Biolabs | M0289S | |
| T4 DNA Ligase | New England Biolabs | M0202T | |
| Sodium Acetate 3M pH5.2 | general lab supplier | ||
| Ethanol for molecular biology | Sigma-Aldrich | E7023 | or equivalent |
| DH5aF' bacteria cells | Thermo Fisher Scientific | ||
| 0,2 ml tubes | general lab supplier | ||
| 1,5 ml tubes | general lab supplier | ||
| 0,1 cm electroporation cuvettes | Biosigma | 4905020 | |
| Electroporator 2510 | Eppendorf | ||
| 2x YT medium | Sigma-Aldrich | Y1003 | |
| Ampicillin sodium salt | Sigma-Aldrich | A9518 | |
| Chloramphenicol | Sigma-Aldrich | C0378 | |
| DreamTaq DNA Polymerase | Thermo Fisher Scientific | EP0702 | |
| Deoxynucleotide (dNTP) Solution Mix | New England Biolabs | N0447S | |
| 96-well thermal cycler (with heated lid) | general lab supplier | ||
| 150 mm plates | general lab supplier | ||
| 100 mm plates | general lab supplier | ||
| Glycerol | Sigma-Aldrich | G5516 | |
| BssHII | New England Biolabs | R0199L | |
| NheI | New England Biolabs | R0131L | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | or equivalent |
| M13KO7 Helper Phage | GE Healthcare Life Sciences | 27-1524-01 | |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K1377 | |
| Polyethylene glycol (PEG) | Sigma-Aldrich | P5413 | |
| Sodium Cloride (NaCl) | Sigma-Aldrich | S3014 | |
| PBS | general lab supplier | ||
| Dynabeads Protein G for Immunoprecipitation | Thermo Fisher Scientific | 10003D | or equivalent |
| MagnaRack Magnetic Separation Rack | Thermo Fisher Scientific | CS15000 | or equivalent |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Nonfat dried milk powder | EuroClone | EMR180500 | |
| KAPA HiFi HotStart ReadyMix | Kapa Biosystems, Fisher Scientific | 7958935001 | |
| AMPure XP beads | Agencourt, Beckman Coulter | A63881 | |
| Nextera XT dual Index Primers | Illumina | FC-131-2001 or FC-131-2002 or FC-131-2003 or FC-131-2004 | |
| MiSeq or Hiseq2500 | Illumina | ||
| Spectrophotomer | Nanodrop | ||
| Agilent Bioanalyzer or TapeStation | Agilent | ||
| Forward PCR primer | general lab supplier | 5’ TACCTATTGCCTACGGCA GCCGCTGGATTGTTATTACTC 3’ | |
| Reverse PCR primer | general lab supplier | 5’ TGGTGATGGTGAGTACTA TCCAGGCCCAGCAGTGGGTTTG 3’ | |
| Forward primer for NGS | general lab supplier | 5’ TCGTCGGCAGCGTCAGA TGTGTATAAGAGACAGGCA GCAAGCGGCGCGCATGC 3’; | |
| Reverse primer for NGS | general lab supplier | 5’ GTCTCGTGGGCTCGGAGA TGTGTATAAGAGACAGGGG ATTGGTTTGCCGCTAGC 3’; |
References
- Loman, N. J., Pallen, M. J. Twenty years of bacterial genome sequencing. Nat Rev Microbiol. 13 (12), 787-794 (2015).
- Jones, C. E., Brown, A. L., Baumann, U. Estimating the annotation error rate of curated GO database sequence annotations. BMC Bioinformatics. 8 (1), 170(2007).
- Andorf, C., Dobbs, D., Honavar, V. Exploring inconsistencies in genome-wide protein function annotations: a machine learning approach. BMC Bioinformatics. 8 (1), 284(2007).
- Wong, W. -C., Maurer-Stroh, S., Eisenhaber, F. More Than 1,001 Problems with Protein Domain Databases: Transmembrane Regions, Signal Peptides and the Issue of Sequence Homology. PLoS Comput Biol. 6 (7), e1000867(2010).
- Bioinformatics, B., et al. Identification and correction of abnormal, incomplete and mispredicted proteins in public databases. BMC Bioinformatics. 9 (9), (2008).
- Phizicky, E., Bastiaens, P. I. H., Zhu, H., Snyder, M., Fields, S. Protein analysis on a proteomic scale. Nature. 422 (6928), 208-215 (2003).
- DiDonato, M., Deacon, A. M., Klock, H. E., McMullan, D., Lesley, S. A. A scaleable and integrated crystallization pipeline applied to mining the Thermotoga maritima proteome. J Struct Funct Genomics. 5 (1-2), 133-146 (2004).
- Nordlund, P., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Zacchi, P., Sblattero, D., Florian, F., Marzari, R., Bradbury, A. R. M. Selecting open reading frames from DNA. Genome Res. 13 (5), 980-990 (2003).
- Di Niro, R., et al. Rapid interactome profiling by massive sequencing. Nucleic Acids Res. 38 (9), e110(2010).
- Gourlay, L. J., et al. Selecting soluble/foldable protein domains through single-gene or genomic ORF filtering: Structure of the head domain of Burkholderia pseudomallei antigen BPSL2063. Acta Crystallogr Sect D Biol Crystallogr. 71 (Pt 11), 2227-2235 (2015).
- D'Angelo, S., et al. Filtering "genic" open reading frames from genomic DNA samples for advanced annotation. BMC Genomics. 12 (Suppl 1), S5(2011).
- D'Angelo, S., et al. Profiling celiac disease antibody repertoire. Clin Immunol. 148 (1), 99-109 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2009).
- Heger, A., Holm, L. Exhaustive enumeration of protein domain families. J Mol Biol. 328 (3), 749-767 (2003).
- Zacchi, P., Sblattero, D., Florian, F., Marzari, R., Bradbury, A. R. M. Selecting open reading frames from DNA. Genome Res. 13 (5), 980-990 (2003).
- Faix, P. H., Burg, M. A., Gonzales, M., Ravey, E. P., Baird, A., Larocca, D. Phage display of cDNA libraries: Enrichment of cDNA expression using open reading frame selection. Biotechniques. 36 (6), 1018-1029 (2004).
- Patrucco, L., et al. Identification of novel proteins binding the AU-rich element of α-prothymosin mRNA through the selection of open reading frames (RIDome). RNA Biol. 12 (12), 1289-1300 (2015).
- Collins, M. O., Choudhary, J. S. Mapping multiprotein complexes by affinity purification and mass spectrometry. Curr Opin Biotechnol. 19 (4), 324-330 (2008).
- Suter, B., Kittanakom, S., Stagljar, I. Two-hybrid technologies in proteomics research. Curr Opin Biotechnol. 19 (4), 316-323 (2008).
- Nakai, Y., Nomura, Y., Sato, T., Shiratsuchi, A., Nakanishi, Y. Isolation of a Drosophila gene coding for a protein containing a novel phosphatidylserine-binding motif. J Biochem. 137 (5), 593-599 (2005).
- Deng, S. J., et al. Selection of antibody single-chain variable fragments with improved carbohydrate binding by phage display. J Biol Chem. 269 (13), 9533-9538 (1994).
- Danner, S., Belasco, J. G. T7 phage display: A novel genetic selection system for cloning RNA-binding proteins from cDNA libraries. Proc Natl Acad Sci. 98 (23), 12954-12959 (2001).
- Gargir, A., Ofek, I., Meron-Sudai, S., Tanamy, M. G., Kabouridis, P. S., Nissim, A. Single chain antibodies specific for fatty acids derived from a semi-synthetic phage display library. Biochim Biophys Acta - Gen Subj. 1569 (1-3), 167-173 (2002).
- Patrucco, L., et al. Identification of novel proteins binding the AU-rich element of α-prothymosin mRNA through the selection of open reading frames (RIDome). RNA Biol. 12 (12), 1289-1300 (2015).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. Mol Biol. 1 (2), 146(2003).
- Sblattero, D., Bradbury, A. Exploiting recombination in single bacteria to make large phage antibody libraries. Nat Biotechnol. 18, 75-80 (2000).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10(2011).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10 (1), 421(2009).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Quinlan, A. R. BEDTools: The Swiss-Army tool for genome feature analysis. Curr Protoc Bioinforma. , (2014).
- Skinner, M. E., Uzilov, A. V., Stein, L. D., Mungall, C. J., Holmes, I. H. JBrowse: A next-generation genome browser. Genome Res. 19 (9), 1630-1638 (2009).
- Gourlay, L. J., et al. Selecting soluble/foldable protein domains through single-gene or genomic ORF filtering: Structure of the head domain of Burkholderia pseudomallei antigen BPSL2063. Acta Crystallogr Sect D Biol Crystallogr. 71, 2227-2235 (2015).
- D'Angelo, S., et al. Filtering "genic" open reading frames from genomic DNA samples for advanced annotation. BMC Genomics. 12 (Suppl 1), S5(2011).
- Di Niro, R., et al. Characterizing monoclonal antibody epitopes by filtered gene fragment phage display. Biochem J. 388 (Pt 3), 889-894 (2005).
- D'Angelo, S., et al. Profiling celiac disease antibody repertoire. Clin Immunol. 148 (1), 99-109 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved