Method Article
В пробирке SUMOylation Assay для изучения сумо E3 лигаза активность
В этой статье
Резюме
В отличие от убиквитин ligases было выявлено несколько ligases E3 сумо. Это изменение в пробирке SUMOylation протокол имеет возможность идентифицировать Роман сумо E3 ligases assay растворения в пробирке .
Аннотация
Модификация маленький убиквитин как модификатор (сумо) является важным столб-поступательные изменения (ПТМ), которая опосредует сигнала прежде всего путем модулирования белок белковых взаимодействий. Подобно убиквитин модификации, SUMOylation режиссер последовательного фермента Каскад, включая E1-активация ферментов (SAE1/SAE2), E2-спряжение фермента (Ubc9), E3-лигаза (то есть, основные сборки взаимодействия семьи, RanBP2 и Pc2). Однако, в отличие от ubiquitination, E3 лигаза не является необходимым для реакции, но не обеспечивают точность и эффективность для сопряжения сумо. Белки, изменена SUMOylation может быть идентифицирован в vivo пробирного через иммунопреципитации субстрата специфических антител и immunoblotting с сумо специфические антитела. Однако демонстрация белка сумо E3 лигаза деятельности требует растворения в пробирке SUMOylation анализов с использованием очищенные ферменты, субстрат и сумо белков. Поскольку в реакциях, в пробирке , обычно SAE1/SAE2 и Ubc9, только достаточно для сумо спряжение, повышение SUMOylation по предполагаемым лигаза E3 не всегда легко обнаружить. Здесь мы описываем изменение в пробирке SUMOylation протокол, который последовательно идентифицирует сумо модификации с помощью системы в vitro воссоздана. Шаг за шагом протокол для очистки каталитически активных K-bZIP, вирусный лигаза E3 сумо-2/3, также представил. SUMOylation деятельность очищенный K-bZIP показываются на p53, известных целевых сумо. Этот протокол может быть использован не только для выяснения Роман сумо E3 ligases, но и для выявления их специфичности paralog сумо.
Введение
СУМО модификации первоначально была определена как реверсивные столб-поступательные изменения (ПТМ), который регулирует белок стабильности1. В дополнение к прямой спряжение сумо может быть присоединен к белку через non ковалентные взаимодействия по сумо взаимодействия мотивы (SIMs)2. Похож на тирозил-фосфорилирования молекул, укрывательство Src гомологии 2 (Ш2) или phosphotyrosine привязку (ПТБ) доменов3,4, сумо модификации предоставляет дополнительные взаимодействия платформу для селективного набор SIM-содержащих эффекторные белки5,6. Помимо регулирования передачи сигнала SUMOylation транскрипционный анализ факторов и chromatin remodeling молекул служат для модулировать ген выражение7,8. Исследования структуры генома всей SUMOylation показали, что сумо модификация связан с позитивным9,10 или отрицательные10,11,12 транскрипции регулирование, отчасти из-за специфики paralogue SUMOylation.
Существует три основных сумо изоформ белка, спряжение подарок в mammalian клетках; к ним относятся сумо-1 и весьма гомологичных сумо-2 и сумо-3 (упоминаемый как сумо-2/3)13. СУМО обычно спрягаются для остатков лизина (K) в ψKxD/E консенсус мотив в целевого белка. SIM-карты в сумо E3 ligases отвечает за специфики paralogue14. В отличие от ubiquitination путей, содержащих сотни E3 ligases там были лишь несколько сумо E3 ligases определены15. СУМО E3 ligases определяются свойствами, включая их способность (i) связывают Ubc9, (ii) связывают общие сумо через SIM домена и (iii) повышение сумо передачи от Ubc9 на подложке. Лигаза E3 не является абсолютно необходимым для сумо спряжение16, но обеспечивает субстрата и специфичность сумо paralogue. Поскольку обычно только малую часть всего субстрата белка, сумо изменение, обнаружение SUMOylated белков в vivo это всегда вызов. Таким образом точные и воспроизводимые анализы необходимы для того, чтобы выяснить точное предназначение сумо модификации.
В пробирке SUMOylation assay, который оценивает очищенный Ubc9 способность катализировать SUMOylation субстрата белков, является широко признанным assay для изучения сумо модификация17. Однако сумо E3 лигаза деятельность в большинстве случаев не могут быть обнаружены или могут быть обнаружены только с мутировал лигаза сумо с высокой активностью лигаза сумо E3 и низкой субстратная специфичность по стандартному протоколу из-за присутствия обильное количество Ubc918 . Общий успех этот assay в значительной степени зависит от тщательного титрования очищенный Ubc9. В vitro Пробирная SUMOylation описано здесь отличается от стандартного SUMOylation протокола (см. Таблицу материалы). Наблюдение за увеличение глобальной модификации сумо во время возобновления саркома Капоши связанные герпеса (KSHV) побудило нас выявления вирусной лигаза сумо E3, которая может нести ответственность за регулирование вверх SUMOylation. После небольших скрининг взаимодействия белков вирусной сумо E3 лигаза K-bZIP, p53 был признан Роман субстрата. В этом протоколе мы показать в деталях клетках насекомых, шаги, связанные в выражение и очистки одичал тип K-bZIP, вирусный лигаза сумо E3 с сумо-2/3 специфика. Очищенный K-bZIP способность увеличить SUMOylation p53, известный сумо субстрата, оценивается присутствии снижение количества E1 и E2 ферментов. Используя этот измененный SUMOylation assay, исследователи могут надежно определить способности сумо E3 лигаза SUMOylate роман или известных субстратов, который является основным шагом в изучении белков SUMOylation. Кроме того этот метод помогает идентифицировать Роман сумо E3 ligases с низкой лигаза активность, но высокий субстратная специфичность.
протокол
1. Подготовка бакуловирусы выражение конструкций
-
Очищать сумо E3 лигаза K-bZIP путем клонирования cDNA K-bZIP19 в вектор бакуловирусы двойного выражения (см. таблицу материалы) с N-терминальный epitope тегом. Мы имели успех с помощью тега octapeptide (указывается в протоколе как вектор тег K-bZIP).
Примечание: Шаблон для K-bZIP полимеразной цепной реакции (ПЦР) клонирования ДНК — cDNA обратной транскрипции от РНК, изолированных от клеток TREx F3H3-K-Rta BCBL-1, после лечения доксициклин20.- Передача полнометражного K-bZIP cDNA19 в двойной выражение вектор с СРОя клонирования сайтов. СРО Я дайджест продукт PCR и 1 мкг двойной выражение вектора при 37 ° C, 3 h21.
- Отдельные и извлечь переваривается ДНК, после электрофореза геля агарозы 0,8%22. Перевязать вставку (K-bZIP) и векторные T4 ДНК лигаза на 16 ° C в течение 30 мин в соответствии с инструкциями производителя (см. Таблицу материалы).
- 5 мкл лигирование ДНК до 50 мкл сведущее Escherichia coli клеток «А» (см. Таблицу материалы) в 1,5 мл. Положите на льду за 30 мин. Затем, инкубировать 1,5 мл в ванну воды 42 ° C для 45 s и немедленно положить трубку на льду на 3 мин.
- Добавить 600 мкл S.O.C. среднего (экстракт дрожжей 0,5% (w/v), 2% (w/v) Триптон, 10 мм NaCl, 2,5 мм KCl, 20 мм MgSO4и 4% (w/v) глюкозы) в трубку и инкубировать при 37 ° C в течение 1 ч. Затем центрифуга трубки при комнатной температуре 3 мин на 600 x g.
- Удалить супернатант, Ресуспензируйте лепешка с 60 мкл Лурия Бертани среднего (LB; триптон 1% (w/v), 0,5% (w/v) дрожжевой экстракт и 85 мм NaCl) и распространяются на плиты агара LB, содержащий ампициллина 100 мкг/мл. Инкубируйте плита агара при 37 ° C для 16 h23,24.
- Выберите 1 колонии для прививок в среде LB 5 мл, содержащие 100 мкг/мл Ампициллин и культуры при 37 ° C для 16 h с 250 rpm встряхивания. Затем извлеките плазмида, вектор тег K-bZIP, с плазмида добыча комплект согласно инструкциям производителя (см. Таблицу материалы).
-
Создания рекомбинантных bacmid, укрывательство тег K-bZIP путем преобразования вектор тег K-bZIP в сведущее Escherichia coli клетки «B» (см. таблицу материалы).
- Смесь 0,2 мкг вектор тег K-bZIP плазмиды и 100 мкл компетентным э. ИСЖ клетки «B» в 1,5 мл трубки и положить на лед на 30 мин инкубировать трубки в ванну воды 42 ° C для 45 s и немедленно положить трубку на льду на 3 мин.
- 900 мкл S.O.C. среды в трубку и инкубировать и при 37 ° C для 4 h с вращением на 50 об/мин. Далее принять 10 мкл среды, дополнительные 50 мкл S.O.C среды для распространения на плиты агара LB, содержащие 50 мкг/мл канамицин, гентамицин 7 мкг/мл, 10 мкг/мл тетрациклин, 100 мкг/мл галактозидазы субстрата и 40 мкг/мл изопропиловый β-D-1-тиогалактопиранозид (ИПТГ ) и инкубировать пластины для 48 ч при 37 ° C.
- Прививать один белый колонии в среде LB 5 мл, содержащие 50 мкг/мл канамицин, гентамицин 7 мкг/мл и 10 мкг/мл тетрациклин. Проинкубируйте при 37 ° C с 250 об/мин, пожимая на 16 h.
- Изолировать bacmid рекомбинантных ДНК, укрывательство тег K-bZIP, количественной оценки и вновь приостановить в концентрации 1 мкг/мкл25.
-
Генерировать рекомбинантных baculoviruses, выражая тег K-bZIP transfecting 1 мкг bacmid рекомбинантных ДНК, содержащие тег K-bZIP в Sf9 клетки в одной скважине 6-ну плиты.
- Семенной 2 x 106 Sf9 клетки (см. Таблицу материалы) в среде Насекомое 2 мл Грейс поставляется с 10% плода бычьим сывороточным (ФБС) и 1% гентамицина в каждом хорошо 6 хорошо пластины, а затем Инкубируйте на 27 ° C в течение 1 ч.
- Поддерживать культуру фазы журнала Sf9 клеток в среде насекомых Грейс поставляется с 10% FBS, гентамицин 1% и 1% моющего средства C (см. Таблицу материалов) при 27 ° C в орбитальных подвеска на 140 об/мин. Подсчет количества ячеек с помощью Горяева и пятнать Трипановый синий; жизнеспособность клеток должен превышать 95%. Поддержания клеток с использованием subcultivation соотношении 1:3 каждые 2-3 дня (минимальная плотность ~0.5 x 106 клеток/мл).
- Добавить 2 мкл bacmid ДНК (1 мкг/мкл) и 98 мкл сокращение сыворотке СМИ (см. Таблицу материалы) в 2-мл пробирку. Затем, добавить 4 мкл Реагента трансфекции (см. Таблицу материалы) для трубки и хорошо перемешать. Инкубации при комнатной температуре в течение 15 мин.
- Добавьте эту смесь трансфекции (104 мкл) в каждой скважине и Инкубируйте на 27 ° C в течение 12-16 ч.
- Удаление исчерпаны СМИ с нежным стремление и заменить 2 мл свежего Грейс насекомых среды поставляется с FBS и 1% гентамицин 10%. Sf9 клетки свободно придерживаться поверхности плиты и не будет нарушено с нежным стремление.
-
Собирать рекомбинантных бакуловирусы после transfection и усилить бакуловирусы для получения высокого титра запасов (P1) для дальнейших экспериментов.
- 72 ч после изменения среднего, скрип Sf9 ячеек с помощью стерильных ячейки стеклоподъёмника, собирать супернатант от каждой индивидуальной скважины в 1,5 мл трубки и вихревые для 10 s.
- Центрифуга на 4 ° C на 3 мин в 150 x g. сбор супернатанта как бакуловирусы P0 и Алиготе в 1,5 мл пробирок (1 мл бакуловирусы в каждой тюбике). Хранить при температуре-80 ° C.
- 7 Sf9 клетки семян 1 x 10 в 10 мл Грейс среднего насекомого поставляется с 10% FBS и 1% гентамицина в 10 см Петри и Инкубируйте на 27 ° C в течение 1 ч.
- Для усиления рекомбинантных baculoviruses выражая тег K-bZIP добавить 0,5 мл P0 бакуловирусы в сеяный клетки Sf9 и Инкубируйте на 27 ° C в течение 48 часов.
- Соберите супернатанта как P1 бакуловирусы в 15 мл трубки и хранить при 4 ° C до 1 месяца.
2. Очистка K-bZIP
- Поддерживать культуру фазы журнала Sf9 клеток в среде насекомых Грейс поставляется с 10% FBS, гентамицин 1% и 1% моющего средства C 27 ° C в орбитальных подвеска на 140 об/мин, как описано в шаге 1.3.2.
- В день трансдукции добавьте 1 mL бакуловирусы содержащих супернатант (P1) в 250 мл с плотностью 2 х 106 клеток/мл Sf9 клеток.
- Инкубировать Sf9 клетки за 72 ч при 27 ° C на орбитальный шейкер с 140 rpm тряски, а затем собрать центрифугированием в 2740 x g 15 мин при 4 ° C.
- Удалить супернатант и лизировать клетки окатышей в 10 мл буфера lysis (20 мм HEPES рН 7.9, 0.5 М NaCl, 1% моющего средства (см. Таблицу материалы), 2% глицерина, ингибитор протеазы коктейль) и поворот на 50 об/мин, с использованием подвеска смеситель на 4 ° C на 30 мин.
- Центрифуга ячейка lysate на 15000 x g 15 мин для сбора супернатант.
- Для того чтобы очистить тег K-bZIP, Проинкубируйте lysates клетки (10 мл) с 50 мкл антител тегами магнитные бусы в 14 мл полипропиленовые трубы и повернуть на 50 об/мин за 3 ч при 4 ° C, с помощью миксера подвеска. После захвата белка, Пелле бусы на 800 x g центрифуги для 30 s при 4 ° C. Удалить большую часть супернатанта, вновь приостановить бисер остаточного объема около 1 мл и передать 1,5 мл трубки для стирки.
- Вымойте захваченных белка, поместив трубка, содержащая бусы на магнитную подставку и удалить супернатант после магнитной бусины полностью присоединились к стороне трубки.
-
Вымойте бусины с буфера lysis четыре раза.
- Добавьте 1 mL литического буфера в 1,5 мл пробирок и инвертировать трубы 10 раз. Место 1,5 мл на магнитную подставку и удалить супернатант, как описано в 2.7.
- Повторите предыдущий шаг, еще 3 раза.
-
Один раз мыть бусины с-фосфатный буфер (PBS).
- Добавьте 1 mL PBS (137 мм NaCl, 2,7 мм KCl, 8 Na2HPO4, 1,5 мм KH2PO4) в 1,5 мл пробирок и инвертировать трубы 10 раз.
- 1,5 мл трубки надевают магнитного стенд и затем удалить супернатант.
- Элюировать тег K-bZIP белка от антител тегами магнитные бусы, добавив 100 мкл 150 мкг/мл octapeptide, разбавленных в PBS в 1,5 мл трубку. Поверните трубки для 10 мин на 50 об/мин, подвеска смеситель при комнатной температуре. Затем положите трубку на магнитного стенд и собирать K-bZIP содержащих супернатант в чистой 1,5 мл.
- Анализ 1-5 мкл очищенного тег K-bZIP белка, SDS-PAGE следуют Кумасси синим пятнать26. Загрузка 2 мкг, 1 мкг, и 0,5 мкг образцы бычьим сывороточным альбумином (БСА) на том же гель и использовать его для количественной оценки с помощью программы обработки изображений, как ImageJ. Для сравнения оценки K-bZIP концентрации BSA стандартам.
- Очищенный K-bZIP теперь готов для использования в каждом SUMOylation assay. Разбавляют K-bZIP в PBS до конечной концентрации 100 нг/мкл и хранить в небольших аликвоты-80 ° c.
3. SUMOylation Assay
- Мкл 3 100 нг/мкл очищенная тегом K-bZIP в мастер смесь каждого в пробирке SUMOylation реакции. Компоненты в мастер смеси содержат 1 мкл буфера белка, 1 x SUMOylation буфера, белок p53 0.5 мкл (0.5 мг/мл), 25 Нм E1 активации фермента (фондовых высококонцентрированные: 1 мкм), 50 Нм E2 спрягать фермента (фондовых высококонцентрированные: 10 мкм) и 250 Нм сумо пептидов (сумо-1 , Сумо-2, или сумо-3; запасов высококонцентрированные: 5 мкм) на окончательный объем 17 мкл.
- Осторожно перемешать содержимое и инкубировать реакции при 30 ° C на 3 ч. прекратить реакции, добавив 20 мкл 2 x SDS-PAGE загрузки буфера (100 мм трис-HCL pH 6.8, лаурилсульфат натрия 4%, 0,2% (w/v) бромфеноловый синий, 20% (v/v) глицерина и β-меркаптоэтанол 200 мм) и Денатурируйте образцы на 95 ° C за 5 мин.
-
Отдельные образцы на SDS-PAGE, передача разлученных белков на PVDF мембрану с помощью аппарата полусухим методом электрофореза передачи и анализировать, Западная помарка, с использованием антител анти p53.
- Настроить аппарат гель как шаг 2.11 и загрузить 20 мкл пример в гель скважин.
- Побегите гель 10% в постоянного тока на 80 V около 120 мин (до тех пор, пока краска группы достигает нижней части геля). После окончания электрофореза Выключите источник питания.
- Отключите аппарат гель и гель кассеты вывезти гель и плавать гель в полусухом передачи буфера (48 мм трис-HCl, 39 мм глицин, 20% метанола) за 5 мин.
- Возьмите другой контейнер и замочить PVDF мембрану в метаноле за 1 мин. Затем возьмите PVDF из метанола и помещаются в буфер передачи полусухое. Аккуратно агитируйте мембрана для 5 мин.
- Удалите крышку безопасности полусухое электрофоретической аппарата.
- Предварительно мокрый фильтр бумага и подготовить бутерброд гель на аноде дно платины следующим: фильтровальная бумага, PVDF мембраны, гель и фильтровальную бумагу.
- Безопасный катод пластины и безопасность Обложка, затем бежать пятно на 15 V постоянного тока для 90 минут повернуть источник питания, отключите аппарат полусухое и вывезти PVDF мембрану.
- Блок PVDF мембрану с блокирующий буфер (5% обезжиренное молоко в TBST буфер (137 мм2NaCl, 20 трис-HCl, 0.1% моющего средства B (см. Таблицу материалы), рН 7,6)) при комнатной температуре за 1 час с 30 об/мин, тряски, орбитальный шейкер.
- Гибридизируйте PVDF мембрану с анти p53 антитела разбавленной (1:1, 000) в блокирующем буфере для 12-16 ч при температуре 4 ° C встряхивании 30 об/мин с помощью миксера подвеска.
- Взять из PVDF мембрану в контейнер и поставьте TBST. Промойте PVDF мембрану с TBST вдвое больше.
- Замочите PVDF мембрану в TBST 30 мин с 45 rpm, тряски, орбитальный шейкер.
- Гибридизируйте PVDF мембрану с анти кролик антитело проспряганное с пероксидазы (ПХ) разбавляют (1:4, 000) в блокирующем буфере 1 ч при комнатной температуре с 30 об/мин, тряски, используя миксер подвеска.
- Вымойте PVDF мембрану с TBST 3 раза, как описано в шаге 3.4.10.
- Замочите PVDF мембрану в TBST 30 мин встряхивании 45 об/мин с помощью миксера подвеска. Удаление TBST и добавьте PBS для сохранения PVDF мембрану при 4 ° C до 12 ч.
- Перемешайте реагент расширенной Хемилюминесцентный субстрат (ECL субстрата) 1 и 2 (1:1) (см. Таблицу материалы). Удаление PVDF мембрану из PBS, кратко пятно с бумажным полотенцем или легких стеклоочистителя поглощать избыточную влагу, и сразу же добавить 400 мкл Реагента ECL на поверхности каждого мембраны для 3-5 мин удалить избыток ECL реагента, кратко промокательной, но не позволяют м embrane для полностью сухой.
- Разоблачить пятно с помощью люминесценции съемочной системы или авторадиографии фильм27.
Результаты
Согласно информации, представленной изготовителем, стандартное количество E1 и E2 фермента в SUMOylation assay-50 Нм до 500 Нм, соответственно. Минимальное количество E2 спрягать фермента Ubc9, которая способна SUMOylate p53 был сначала определяется в пробирке SUMOylation assay. Как низко как одной пятой от объема Ubc9, используемых в стандартных в пробирке SUMOylation протокол assay смогла эффективно SUMOylate p53 (рис. 1A). Таким образом половина суммы E1 фермента в сочетании с одной десятой от суммы Ubc9, используемых в стандартный протокол был использован для другого в пробирке SUMOylation assay. По сравнению с стандартный протокол (рис. 1Б) наблюдалось значительное снижение эффективности SUMOylation.
Таким образом сумо E3 лигаза K-bZIP способностью усиливать p53, SUMOylation был определен с помощью этих изменения в vitro условий SUMOylation. Меткой K-bZIP, очищенного от Sf9 клеток (рисA) работал в SUMOylation реакции, содержащие нижнюю E1 и E2 Ubc9 ферментов. В этих измененных условиях, K-bZIP может эффективно стимулировать SUMOylation p53 (рис. 2B). Immunoblotting с антителами anti-p53 подтвердила аналогичные суммы p53 в каждой реакции, а также обнаружены сумо изменения p53.

Рисунок 1 : Определение минимального количества сумо ферментов, используемых в assay SUMOylation в vitro . (A) p53, которую SUMOylation оценивалась в в пробирке SUMOylation assay который включал наполовину и одной пятой количество фермента Ubc9 используется в стандартный протокол. После 3 часов в пробирке SUMOylation реакции, SUMOylated p53 ферменты были рассмотрены Западная пятно антитела анти p53. (B) в пробирке SUMOylation р53 было дополнительно проанализировать с помощью половину суммы E1 фермента в сочетании с одной десятой количество Ubc9, используемых в стандартный протокол. Западная помарка была выполнена как описано как (A). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
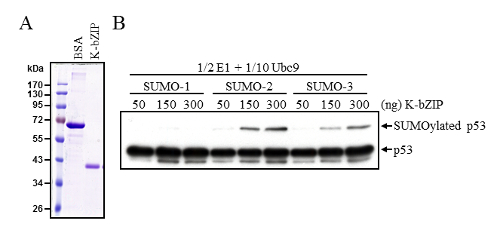
Рисунок 2 : Повышение SUMOylation p53, сумо E3 лигаза K-bZIP. (A) очищенной выразил бакуловирусы K-bZIP белка, анализируемой SDS-PAGE с Окрашивание Кумасси синий. 1 мкг BSA и 10 мкл очищенного K-bZIP загрузки для сравнительного количество белка. (B) K-bZIP Улучшено p53, которую SUMOylation оценивали с помощью в пробирке SUMOylation реакция с половиной количество фермента E1 и одну десятую количество рекомендуется Ubc9 в стандартный протокол. SUMOylated p53 расследуются Западная помарка, как описано в Рисунок 1A. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Обсуждение
В пробирке SUMOylation протокол, описанный здесь обычно используется для установления статуса SUMOylation выявленных Ubc9 субстратов. Основные ограничения, используя стандартный протокол для изучения SUMOylation функция сумо E3 лигаза является обилие активации сумо E1 и E2 спрягать ферментов Ubc9, которые максимально спряжение сумо в в vitro системах. Учитывая эту задачу мы считаем что титрование количество сумо E1 и E2 ферментов до уровня, что один едва можно обнаружить, что их деятельность в SUMOylation доступна только способ определить E3 сумо лигаза мероприятий каталитического характера с использованием этого в пробирке SUMOylation системы. После этой гипотезе мы успешно осветил деятельность лигаза E3 K-bZIP и определили его как Роман лигаза сумо E3 с специфичность к сумо-2/3.
Важным шагом в рамках протокола является включение нижнюю E1 и E2. С помощью этих нижнюю, как показано на рисунке 2, Роман сумо E3 ligases с низкой лигаза активности может быть возможность быть отождествлен с сохранением их характерность субстрата и сумо paralogues. Главным недостатком метода является условием высокого качества антитела, признавая сумо субстрата, поскольку лишь очень небольшая часть субстрата может быть изменен сумо.
Хотя многие белки показывают, потенциал для увеличения SUMOylation после гиперэкспрессия в естественных условиях, определение сумо E3 лигаза должна быть продемонстрирована восстановленный в пробирке SUMOylation системы с использованием очищенного E1, E2 и E3 ферменты. В отличие от сотни, возможно тысячи убиквитин E3 ligases определены до сих пор были найдены только несколько сумо E3 ligases. Это может быть отчасти объясняется использование традиционного стандарта в пробирке SUMOylation assay, которая максимизирует эффективность спряжение сумо и поэтому препятствует SUMOylation функции потенциальных сумо E3 ligases. Настоящий Протокол описывает простой и последовательной пробирного зондировать SUMOylation, усиливается лигаза сумо E3. Описан метод имеет важное значение для идентификации и определения характеристик Роман сумо E3 ligases.
Раскрытие информации
Авторы раскрывают не потенциальные конфликты интересов.
Благодарности
Эта работа была поддержана от грантов от министерства науки и технологии (большинство, 105-2320-B-010-007-MY3 КЦП), национального института здравоохранения исследований (НПУ-EX105-10215BC КЦП), министерства науки и технологии (наиболее 105-2314-Б-400-019 для ХИК) и от национального института медицинских исследований (НПЗУ мг-105-SP-10, НПЗУ мг-106-SP-10-ХИК.). Эта работа была также поддерживается частично с национальным университетом Yang Ming на публикации рукописи КЦП. Спонсоры имел никакой роли в дизайн исследования, сбор данных и анализ, решение опубликовать или возмещение рукописи.
Материалы
| Name | Company | Catalog Number | Comments |
| pFastBac | Invitrogen | 10359-016 | dual expression baculovirus vector |
| pCR2.1-TOPO vector | Invitrogen | PCR product vector | |
| competent cell E. coli DH5α | Yeastern Biotech | FYE678-80VL | competent E. coli cells A |
| E. coli DH10Bac | Invitrogen | 10359-016 | competent E. coli cells B |
| FugeneHD | Roche | 04709705001 | transfection reagent |
| Opti-MEM | Gibco | 31985062 | reduced serum media |
| T4 Ligase | NEW England BioLabs | M0202S | |
| CpoI (RsrII) | Thermo Scientific | ER0741 | |
| Bluo-gal | Thermo Scientific | B1690 | galactosidase substrate |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Grace’s Insect Medium | Gibco | 11605094 | |
| Fetal Bovine Serum | Gibco | 10082147 | |
| Gentamicin | Thermo Fisher | 15750060 | |
| Ampicillin | Sigma-Aldrich | A9393-25G | |
| Kanamycin | Sigma-Aldrich | K0254-20ML | |
| gentamicin | Gibco | 15710-064 | |
| tetracyclin | Sigma-Aldrich | 87128-25G | |
| LB Broth | Merk | 1.10285.0500 | |
| HEPES | Sigma-Aldrich | H4034-100G | |
| NaCl | Sigma-Aldrich | S9888-5KG | |
| KCl | Merk | 1.04936.1000 | |
| Na2HPO4 | Sigma-Aldrich | S5136-500G | |
| KH2PO4 | J.T.Baker | 3246-01 | |
| sodium dodecyl sulfate | Merk | 1.13760.1000 | |
| β-mercaptoethanol | Bio-Rad | 161-0710 | |
| TRIS (Base) | J.T.Baker | 4109-06 | |
| Non-fat milk | Fonterra | ||
| glycerol | J.T.Baker | 2136-01 | |
| Triton X-100 | Amresco | 0694-1L | detergent A |
| Tween 20 | Amresco | 0777-500ML | detergent B |
| Poloxamer 188 solution | Sigma-Aldrich | P5556-100ML | detergent C |
| Protease Inhibitor Cocktail Tablet | Roche | 04 693 132 001 | |
| 3x Flag peptide | Sigma | F4799 | |
| anti-FLAG m2 Magnetic beads | Sigma-Aldrich | M8823 | antibody-tagged magnetic beads |
| SUMOlink SUMO-1 Kit | Active Motif | 40120 | standard SUMOylation protocol |
| SUMOlink SUMO-2/3 Kit | Active Motif | 40220 | standard SUMOylation protocol |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAGEN Plasmid Mini Kit | QIAGEN | 12123 | plasmid extraction kit |
| Polypropylene tubes | Falcon | 352059 | |
| Petri Dish | Falcon | 351029 | |
| Cell lifter | Corning | CNG3008 | |
| Loading tip | Sorenson BioScience | 28480 | |
| PVDF | PerkinElmer | NEF1002 | |
| Blotting filter paper | Bio-Rad | 1703932 | |
| Mini slab gel apparatus (Bio-Rad Mini Protean II Cell) | Bio-Rad | 1658001 EDU | |
| Trans-Blot SD Semi-Dry Electrophoretic Transfer Cell | Bio-Rad | 1703940 | |
| Pierce ECL Western Blotting | Thermo | 32106 | ECL reagent |
| suspension mixer | Digisystem laboratory instruments Inc. | SM-3000 | |
| orbital shaker | Kansin instruments Co. | OS701 | |
| ImageQuant LAS 4000 biomolecular imager | GE Healthcare | 28955810 | |
| Sf9 | Thermo Scientific | B82501 | |
| anti-p53 antibody | Cell Signaling | #9282 | |
| anti-rabbit antibody | GE Healthcare | NA934-1ML |
Ссылки
- Bies, J., Markus, J., Wolff, L. Covalent attachment of the SUMO-1 protein to the negative regulatory domain of the c-Myb transcription factor modifies its stability and transactivation capacity. J Biol Chem. 277 (11), 8999-9009 (2002).
- Lin, D. Y., et al. Role of SUMO-interacting motif in Daxx SUMO modification, subnuclear localization, and repression of sumoylated transcription factors. Mol Cell. 24 (3), 341-354 (2006).
- Welsh, M., Jamalpour, M., Zang, G., Akerblom, B. The role of the Src Homology-2 domain containing protein B (SHB) in beta cells. J Mol Endocrinol. 56 (1), 21-31 (2016).
- Huang, W. Q., et al. Structure, function, and pathogenesis of SHP2 in developmental disorders and tumorigenesis. Curr Cancer Drug Targets. 14 (6), 567-588 (2014).
- Perry, J. J., Tainer, J. A., Boddy, M. N. A SIM-ultaneous role for SUMO and ubiquitin. Trends Biochem Sci. 33 (5), 201-208 (2008).
- Sun, H., Leverson, J. D., Hunter, T. Conserved function of RNF4 family proteins in eukaryotes: targeting a ubiquitin ligase to SUMOylated proteins. EMBO J. 26 (18), 4102-4112 (2007).
- Ouyang, J., Shi, Y., Valin, A., Xuan, Y., Gill, G. Direct binding of CoREST1 to SUMO-2/3 contributes to gene-specific repression by the LSD1/CoREST1/HDAC complex. Mol Cell. 34 (2), 145-154 (2009).
- Lyst, M. J., Stancheva, I. A role for SUMO modification in transcriptional repression and activation. Biochem Soc Trans. 35, 1389-1392 (2007).
- Liu, H. W., et al. Chromatin modification by SUMO-1 stimulates the promoters of translation machinery genes. Nucleic Acids Res. 40 (20), 10172-10186 (2012).
- Rosonina, E., Duncan, S. M., Manley, J. L. SUMO functions in constitutive transcription and during activation of inducible genes in yeast. Genes Dev. 24 (12), 1242-1252 (2010).
- Chang, P. C., et al. The chromatin modification by SUMO-2/3 but not SUMO-1 prevents the epigenetic activation of key immune-related genes during Kaposi's sarcoma associated herpesvirus reactivation. BMC Genomics. 14, 824 (2013).
- Neyret-Kahn, H., et al. Sumoylation at chromatin governs coordinated repression of a transcriptional program essential for cell growth and proliferation. Genome Res. 23 (10), 1563-1579 (2013).
- Tatham, M. H., et al. Polymeric chains of SUMO-2 and SUMO-3 are conjugated to protein substrates by SAE1/SAE2 and Ubc9. J Biol Chem. 276 (38), 35368-35374 (2001).
- Mattoscio, D., Segre, C. V., Chiocca, S. Viral manipulation of cellular protein conjugation pathways: The SUMO lesson. World J Virol. 2 (2), 79-90 (2013).
- Gareau, J. R., Lima, C. D. The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. Nat Rev Mol Cell Biol. 11 (12), 861-871 (2010).
- Bernier-Villamor, V., Sampson, D. A., Matunis, M. J., Lima, C. D. Structural basis for E2-mediated SUMO conjugation revealed by a complex between ubiquitin-conjugating enzyme Ubc9 and RanGAP1. Cell. 108 (3), 345-356 (2002).
- Knipscheer, P., et al. Ubc9 sumoylation regulates SUMO target discrimination. Mol Cell. 31 (3), 371-382 (2008).
- Vethantham, V., Manley, J. L. In vitro sumoylation of recombinant proteins and subsequent purification for use in enzymatic assays. Cold Spring Harb Protoc. 2009 (1), 5121 (2009).
- Lin, S. F., Robinson, D. R., Miller, G., Kung, H. J. Kaposi's sarcoma-associated herpesvirus encodes a bZIP protein with homology to BZLF1 of Epstein-Barr virus. J Virol. 73 (3), 1909-1917 (1999).
- Yang, W. S., Hsu, H. W., Campbell, M., Cheng, C. Y., Chang, P. C. K-bZIP Mediated SUMO-2/3 Specific Modification on the KSHV Genome Negatively Regulates Lytic Gene Expression and Viral Reactivation. PLoS Pathog. 11 (7), 1005051 (2015).
- Connor, C. D., Metcalf, E., Wrighton, C. J., Harris, T. J., Saunders, J. R. RsrII--a novel restriction endonuclease with a heptanucleotide recognition site. Nucleic Acids Res. 12 (17), 6701-6708 (1984).
- Helling, R. B., Goodman, H. M., Boyer, H. W. Analysis of endonuclease R-EcoRI fragments of DNA from lambdoid bacteriophages and other viruses by agarose-gel electrophoresis. J Virol. 14 (5), 1235-1244 (1974).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Gingold, E. B. Bacterial transformation. Methods Mol Biol. 2, 237-240 (1985).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. J Virol. 86 (18), 9708-9720 (2012).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Ahmed, S., Holt, M., Riedel, D., Jahn, R. Small-scale isolation of synaptic vesicles from mammalian brain. Nat Protoc. 8 (5), 998-1009 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены