Method Article
במבחנה SUMOylation Assay ללמוד סומו E3 ליגאז פעילות
In This Article
Summary
בניגוד ligases אוביקוויטין, זוהו מספר ligases סומו E3. ששונה זה במבחנה SUMOylation פרוטוקול הוא מסוגל לזהות ligases סומו E3 הרומן מאת assay שיחזור במבחנה .
Abstract
אוביקוויטין דמוי קטן משנה (סומו) השינוי הוא שינוי post-translational חשוב (PTM) המעביר אותות בעיקר דרך להתכוונן אינטראקציות חלבון חלבון. בדומה אוביקוויטין השינוי, SUMOylation הוא בבימויו של מפל אנזים רציפים לרבות E1-מפעיל אנזים (SAE1/SAE2), E2-ההטיה אנזים (Ubc9) ו- E3 ליגאז (קרי, משפחת PIAS, RanBP2, ו- Pc2). לעומת זאת, שונה ubiquitination, E3 ליגאז הוא שאינם חיוניים עבור התגובה אך האם לספק הדיוק והיעילות סומו ההטיה. חלבונים שונה על-ידי SUMOylation יכול להיות מזוהה על ידי ויוו assay ויה immunoprecipitation עם נוגדנים ספציפיים המצע, immunoblotting עם נוגדנים ספציפיים סומו. עם זאת, ההדגמה של פעילות החלבון סומו E3 ליגאז דורש במבחנה הכינון של מבחני SUMOylation באמצעות אנזימים מטוהרים, המצע סומו חלבונים. מאז בתגובות במבחנה , בדרך כלל SAE1/SAE2, Ubc9, לבד מספיק עבור סומו ההטיה, שיפור של SUMOylation על ידי ליגאז E3 בשם לא תמיד קל לזהות. כאן, אנו מתארים יישום ששונה במבחנה פרוטוקול SUMOylation שמזהה באופן עקבי סומו שינוי באמצעות מערכת במבחנה מחדש. פרוטוקול צעד אחר צעד לטהר פעיל catalytically K-bZIP, ליגאז ויראלי E3 סומו-2/3, מוצג גם. הפעילות SUMOylation של K-bZIP מטוהרים מוצגים ב- p53, מטרה ידועה של סומו. פרוטוקול זה יכול לא רק להיות מועסק שחקרתי הרומן ligases סומו E3, אלא גם חשיפת שלהם ירידה לפרטים paralog סומו.
Introduction
סומו השינוי זוהה בתחילה כמו שינוי post-translational הפיך (PTM) המסדירה את יציבות חלבון1. בנוסף נטיה ישירה, סומו ניתן גם לחבר חלבון תוך אינטראקציה אי-קוולנטיות על-ידי סומו אינטראקציה עם מוטיבים (סימס)2. דומה קשירה של tyrosyl-זרחון על ידי מולקולות מחסה Src הומולוגיה 2 (SH2) או איגוד phosphotyrosine (PTB) תחומים3,4, סומו שינוי מספק פלטפורמה אינטראקציה נוספת עבור סלקטיבית גיוס של ה-SIM המכיל אפקטור חלבונים5,6. בנוסף רגולציה של אותות, לשרת SUMOylation של כרומטין שיפוץ מולקולות וגורמי תעתיק לווסת את ג'ין-ביטוי-7,-8. מחקרים של הגנום כולו SUMOylation דפוסי גילו כי השינוי סומו קשורה חיובית9,10 או שלילי10,11,12 שעתוק . תקנה, בין היתר בשל יחודיות paralogue של SUMOylation.
ישנם שלושה איזופורמים סומו העיקריים עבור חלבון conjugating המתנה בתאי יונקים; אלה כוללים סומו-1, ואת מאוד הומולוגי סומו-2 סומו-3 (המכונה סומו-2/3)13. סומו הוא בדרך כלל מצומדת את השאריות ליזין (K) בתוך המוטיב ψKxD/E הסכמה חלבון המטרה. ה-SIM ב- E3 סומו ligases אחראית על ירידה לפרטים paralogue14. בניגוד ubiquitination מסלולים המכיל מאות E3 ligases, היו רק כמה E3 סומו ligases זיהה15. סומו E3 ligases מוגדרים על-ידי המאפיינים כולל את היכולת שלהם (i) לאגד Ubc9, (ii) לאגד את הסומו moiety דרך תחום ה-SIM ו- (iii) לשפר את העברת סומו Ubc9 על גבי המצע. E3 ליגאז אינה נדרשת לחלוטין סומו ההטיה16, אבל מספק המצע וספציפיות סומו-paralogue. מאז בדרך כלל רק חלק קטן של החלבון המצע הכולל הוא סומו-לאחרונה, זיהוי של SUMOylated חלבונים ויוו היא תמיד אתגר. לכן, מבחני לשחזור ומדויקים הדרושים כדי להבהיר הפונקציה מדויק של השינוי סומו.
ה במבחנה SUMOylation assay, הבודקת את היכולת של Ubc9 מטוהרים כדי לעודד SUMOylation של חלבונים המצע, הוא assay היטב מקובל ללמוד סומו השינוי17. עם זאת, סומו E3 ליגאז פעילות ברוב המקרים לא ניתן לאתרם או יכול רק להיות מזוהה עם ליגאז סומו מוטציה עם גבוהה סומו E3 ליגאז פעילות וספציפיות המצע נמוך לפי הפרוטוקול הסטנדרטי עקב נוכחות כמות שופעת של Ubc918 . ההצלחה הכוללת של assay זו תלויה במידה רבה טיטרציה זהירה של Ubc9 מטוהרים. במבחנה SUMOylation וזמינותו המתוארים כאן היא שונה SUMOylation הרגיל פרוטוקול (ראה טבלה של חומרים). התצפית של שינוי סומו הכללית מוגברת במהלך הפעלה מחדש הקשורים סרקומת קפוסי ההרפס (KSHV) בקשה לנו לזהות את ליגאז סומו E3 ויראלי, שעשויים להיות אחראי להסדרת-up של SUMOylation. בעקבות ההקרנה בקנה מידה קטן של החלבונים אינטראקציה של נגיפי סומו E3 ליגאז K-bZIP, p53 מזוהה כמו מצע הרומן. ב פרוטוקול זה, אנו מראים בפירוט בתאי חרקים השלבים המעורבים הביטוי ולטיהור פראי-סוג K-bZIP, נגיפי סומו E3 ליגאז עם ירידה לפרטים סומו-2/3. היכולת של מטוהרים K-bZIP כדי לשפר את SUMOylation של p53, מצע סומו ידועים, מחושבת בנוכחות כמות מופחתת של אנזימים E1, E2. שימוש assay ששונה זה SUMOylation, חוקרים יכול באופן אמין להגדיר את היכולות של סומו E3 ליגאז SUMOylate רומן או מצעים ידוע, וזה צעד ראשוני במחקר של חלבון SUMOylation. יתר על כן, שיטה זו מסייעת לזהות את הרומן ligases סומו E3 עם פעילות ליגאז נמוך אבל ירידה לפרטים המצע גבוהה.
Protocol
1. הכנת בונה הביטויים Baculovirus
-
לטהר סומו E3 ליגאז K-bZIP על-ידי שיבוט של cDNA של K-bZIP19 לתוך וקטור baculovirus ביטוי כפול (ראה טבלה של חומרים) עם epitope-תג N-מסוף. . היו לנו הצלחה באמצעות תג octapeptide (מסומן לאורך כל הפרוטוקול כמו וקטור-תג-K-bZIP).
הערה: תבנית ה-DNA של K-bZIP פולימראז תגובת שרשרת (PCR) שיבוט זה cDNA הפוכה עיבד מ- RNA מבודד מתאי TREx F3H3-K-Rta BCBL-1 בעקבות טיפול דוקסילין20.- העברה של cDNA באורך מלא K-bZIP19 לתוך הווקטור ביטוי כפול עם Cpoאני שיבוט אתרים. Cpo מעכל את המוצר PCR ו- µg 1 של וקטור ביטוי כפול ב 37 ° C 3 h21.
- להפריד ולחלץ את ה-DNA מתעכל בעקבות אלקטרופורזה על 0.8% agarose ג'ל22. מאתרים ומפסיקים את תותב (K-bZIP), וקטור על ידי T4 DNA ליגאז ב 16 מעלות צלזיוס למשך 30 דקות על-פי הוראות היצרן (ראה טבלה של חומרים).
- להוסיף 5 µL של מצדו דנ א כדי 50 µL המוסמכת e. coli תאים "A" (ראה טבלה של חומרים) צינור 1.5 מ ל. לשים על קרח למשך 30 דקות. לאחר מכן, דגירה הצינור 1.5 mL באמבט מים 42 מעלות צלזיוס במשך 45 s מיד את הצינורית על קרח למשך 3 דקות.
- הוסף µL 600 S.O.C. בינוני (0.5% (w/v) תמצית שמרים, 2% (w/v) טריפטון, 10 מ מ NaCl, 2.5 מ מ אשלגן כלורי, 20 מ מ MgSO4ו- 4% (w/v) גלוקוז) לתוך הצינור, דגירה ב 37 ° C עבור 1 h. לאחר מכן, centrifuge את הצינור בטמפרטורת החדר במשך 3 דקות ב 600 x g.
- להסיר את תגובת שיקוע, resuspend צנפה בינונית 60-לוריא-Bertani µL (ליברות; טריפטון 1% (w/v), 0.5% (w/v) שמרים לחלץ, ואת 85 מ מ NaCl) להתפשט על פלטות אגר LB המכיל אמפיצילין µg/mL 100. דגירה של צלחת אגר ב 37 ° C עבור 16 h23,24.
- בחר מושבת 1 לחסן ב 5 מ ל LB בינוני המכיל אמפיצילין µg/mL 100 ותרבות ב 37 ° C עבור 16 h עם סל"ד 250 רועדת. לאחר מכן, לחלץ את פלסמיד, וקטור-תג-K-bZIP, על-ידי ערכת חילוץ פלסמיד לפי הוראות היצרן (ראה טבלה של חומרים).
-
ליצור את bacmid רקומביננטי מסתירים את התג-K-bZIP על-ידי הפיכתו של וקטור-התג-K-bZIP לתוך המוסמכת e. coli תאים "B" (ראה טבלה של חומרים).
- לערבב 0.2 פלסמיד וקטור-תג-K-bZIP µg 100 µL המוסמכת אי coli תאים "B" צינור 1.5 mL, שים על קרח במשך 30 דקות דגירה הצינור באמבט מים 42 מעלות צלזיוס במשך 45 s מיד את הצינורית על קרח למשך 3 דקות.
- הוסף 900 µL בינוני S.O.C. לתוך הצינור, דגירה ב 37 מעלות צלזיוס במשך 4 שעות עם סיבוב ב-50 סל ד. בשלב הבא, קח 10 µL של המדיום, להוסיף את µL 50 נוספים של מדיום S.O.C למריחה על פלטות אגר LB המכיל 50 µg/mL kanamycin, גנטמיצין µg/mL 7, 10 µg/mL טטרציקלין, המצע galactosidase 100 של µg/mL, ו 40 µg/mL איזופרופיל β-D-1-thiogalactopyranoside (IPTG ), דגירה הלוחות עבור h 48-37 מעלות צלזיוס.
- לחסן המושבה לבן אחד מ ל LB בינוני המכיל 50 µg/mL kanamycin, גנטמיצין µg/mL 7 ו- 10 µg/mL טטרציקלין. דגירה ב 37 ° C עם 250 סל"ד רועד עבור 16 h.
- לבודד את bacmid רקומביננטי DNA מסתירים את התג-K-bZIP, לכמת, וכן להשעות מחדש-ריכוז של µg/µL 125.
-
צור baculoviruses רקומביננטי לבטא תג-K-bZIP על ידי transfecting 1 µg של recombinant bacmid DNA המכיל תגית-K-bZIP לתאים Sf9 אחד טוב של צלחת 6-. טוב.
- זרע 2 x 106 Sf9 תאים (ראה טבלה של חומרים) של 2 מ ל גרייס חרקים בינוני שסופק עם 10% סרום שור עוברית (FBS) ו 1% גנטמיצין בכל טוב של צלחת טוב 6, אז דגירה ב 27 ° C עבור 1 h.
- לשמור על תרבות שלב יומן של תאים Sf9 של גרייס חרקים בינוני שסופק עם 10% FBS גנטמיצין 1%, 1% דטרגנט C (ראה טבלה של חומרים) ב 27 ° C ההשעיה מסלולית-140 סל ד. לספור תאים באמצעות hemocytometer trypan blue מכתימה; תא הכדאיות צריך לחרוג מ- 95%. לשמור על תאים באמצעות subcultivation יחס של 1:3 כל 2-3 ימים (צפיפות מינימלית ~0.5 x 106 תאים/mL).
- להוסיף µL 2 bacmid DNA (µg 1/µL) 98 µL מופחת סרום מדיה (ראה טבלה של חומרים) לתוך צינור 2 מ"ל. לאחר מכן, הוסף 4 ריאגנט תרביות תאים µL (ראה טבלה של חומרים) שפופרת, לערבב היטב. דגירה בטמפרטורת החדר למשך 15 דקות.
- להוסיף תערובת זו תרביות תאים (104 µL) כל טוב, דגירה ב 27 ° C עבור h 12-16.
- להסיר את המדיה מותש עם השאיפה עדין, והחלף 2 מיליליטר חרקים בינוני של גרייס חדשה שסופק עם גנטמיצין FBS ו 1%-10%. התאים Sf9 לדבוק באופן רופף פני צלחת ולא תהיה מופרעת עם השאיפה עדין.
-
לאסוף baculovirus רקומביננטי לאחר תרביות תאים ומגבירים את baculovirus כדי לקבל מניות כייל גבוה (P1) לניסויים נוספים.
- 72 h לאחר שינוי בינוני, מגרדים Sf9 תאים באמצעות של מרים תא סטרילי, לאסוף את תגובת שיקוע מכל קידוח בודדים צינור 1.5 מ ל ו מערבולת 10 s.
- צנטריפוגה ב 4 מעלות צלזיוס למשך 3 דקות ב 150 ג x תגובת שיקוע לאסוף P0 baculovirus ו aliquot 1.5 mL צינורות (1 מ"ל baculovirus כל צינור). חנות ב-80 מעלות צלזיוס.
- התאים7 Sf9 זרע עונה 1 פרק 10 של 10 מ"ל גרייס חרקים בינוני שסופק עם גנטמיצין FBS ו 1% 10% ב- 10 ס מ פטרי, דגירה ב 27 ° C עבור 1 h.
- עבור הגברה של baculoviruses רקומביננטי לביטוי תג-K-bZIP, להוסיף 0.5 mL P0 baculovirus תאי Sf9 הזריעה, דגירה ב 27 מעלות צלזיוס במשך 48 שעות.
- לאסוף תגובת שיקוע ככל P1 baculovirus 15 mL שפופרת, חנות ב 4 ° C עד כחודש.
2. טיהור של K-bZIP
- לשמור על תרבות שלב יומן של תאים Sf9 של גרייס חרקים בינוני שסופק עם 10% FBS גנטמיצין 1%, 1% דטרגנט C-27 ° C ההשעיה מסלולית-140 סל ד, כפי שמתואר בשלב 1.3.2.
- ביום של התמרה חושית, להוסיף 1 מ"ל של תגובת שיקוע המכילים baculovirus (P1) 250 מ של תאים Sf9 עם צפיפות של 2 x 106 תאים למ"ל.
- דגירה Sf9 תאים עבור 72 h ב 27 ° C-תפקודי לב / נשימה 140 סל ד רועד ולאחר מכן לאסוף על ידי צנטריפוגה ב g x 2,740 למשך 15 דקות ב 4 º C.
- להסיר את תגובת שיקוע ואת lyse תא כדורי במאגר פירוק 10 מ"ל (20 מ מ HEPES pH 7.9, 0.5 M NaCl, דטרגנט 1% 2% (ראה טבלה של חומרים), גליצרול, מעכבי פרוטאז קוקטייל) וסיבוב ב-50 סל ד באמצעות מערבל ההשעיה ב 4 מעלות צלזיוס למשך 30 דקות.
- צנטריפוגה תא lysate ב g x 15,000 למשך 15 דקות. לאסוף את תגובת שיקוע.
- לטהר תג-K-bZIP, דגירה של תאים lysates (10 מ"ל) עם µL 50 של נוגדן מתויג beads מגנטי צינורות פוליפרופילן 14 מ"ל, וסובב ב-50 סל ד עבור h 3 ב 4 ° C באמצעות מערבל ההשעיה. בעקבות לכידת חלבון, גלולה את החרוזים-800 x g צנטריפוגה ב-30 s-4 מעלות צלזיוס. להסיר את מרבית תגובת שיקוע מחדש להשעות את החרוזים בהיקף שיורית של בערך 1 מ"ל, להעביר צינור 1.5 mL לרחצה.
- לשטוף את החלבון שנתפסו על ידי הנחת הצינור המכיל את החרוזים על דוכן מגנטי, ולהסיר את תגובת שיקוע ברגע החרוזים מגנטי יש מלא דבקה הצד של הצינור.
-
לשטוף את החרוזים עם מאגר פירוק ארבע פעמים.
- להוסיף 1 מ"ל פירוק מאגר לתוך צינורות 1.5 mL, היפוך הצינורות 10 פעמים. במקום הצינור 1.5 mL על דוכן מגנטי ולהסיר את תגובת שיקוע כמתואר ב- 2.7.
- חזור על השלב הקודם עוד 3 פעמים.
-
לשטוף את החרוזים בתמיסת פוספט buffered (PBS) פעם אחת.
- להוסיף 1 מ"ל PBS (137 מ"מ NaCl, 2.7 מ"מ אשלגן כלורי, 8 מ"מ Na2HPO4, 1.5 mM KH2PO4) לתוך צינורות 1.5 mL, היפוך הצינורות 10 פעמים.
- שמכניסים את נקז 1.5 mL על דוכן מגנטי ולאחר מכן להסיר את תגובת שיקוע.
- Elute תג-K-bZIP חלבון מ beads מגנטי מתויג נוגדן על-ידי הוספת 100 µL של octapeptide µg/mL 150 מדולל ב- PBS לתוך צינור 1.5 מ. סיבוב הצינור 10 דקות ב-50 סל ד ב- מיקסר התלוי בטמפרטורת החדר. לאחר מכן, למקם את הצינור בחזרה על הדוכן מגנטי ולאסוף את תגובת שיקוע K-bZIP-המכילה צינור נקי mL 1.5.
- ניתוח 1-5 µL מטוהרים תג-K-bZIP חלבון מאת מרחביות-דף ואחריו הכחול Coomassie מכתים26. לטעון 2 µg, 1 µg, µg 0.5, דוגמאות של אלבומין (BSA) על אותו ג'ל ולהשתמש בו עבור כימות עם תוכנית עיבוד התמונה, כמו ImageJ. מעריכים את ריכוז K-bZIP על ידי השוואה לסטנדרטים BSA.
- K-bZIP מטוהרים מוכן כעת לשמש כל assay SUMOylation. לדלל את bZIP-K ב- PBS כדי ריכוז סופי של 100 ng/µL ו חנות קטנה aliquots ב-80 מעלות צלזיוס.
3. SUMOylation Assay
- להוסיף 3 µL של 100 ng/µL של מטוהרים תג-K-bZIP לתערובת הראשית של כל במבחנה התגובה SUMOylation. מרכיבים בתמהיל מאסטר מכיל מאגר חלבון µL 1, 1 x SUMOylation מאגר, חלבון p53 µL 0.5 (0.5 מ"ג/מ"ל), 25 nM של E1 מפעיל אנזים (conc. מניות: 1 מיקרומטר), 50 nM של E2 conjugating אנזים (מניות conc.: 10 מיקרומטר), ל-250 ננומטר של פפטידים סומו (סומו-1 , סומו-2, או סומו-3; conc. מניות: 5 מיקרומטר) באמצעי אחסון הסופי של 17 µL.
- לערבב בעדינות את התוכן, דגירה התגובה ב 30 מעלות צלזיוס במשך 3 ח' לעצור את התגובה על-ידי הוספת 20 µL של 2 x מרחביות-דף טעינת מאגר (100 מ מ טריס-HCL pH 6.8, 4% נתרן גופרתי dodecyl, כחול bromophenol 0.2% (w/v), 20% (v/v) גליצרול ו-200 מ מ β-mercaptoethanol), denature את הדגימות ב 95 מעלות צלזיוס במשך 5 דקות.
-
דוגמאות נפרדות בדף מרחביות, להעביר את החלבונים מופרדים על גבי הממברנה PVDF באמצעות מנגנון העברה electrophoretic חצי יבש, ולנתח על ידי תספיג באמצעות נוגדן anti-p53.
- להרכיב את המערכת ג'ל כמו שלב 2.11, וטען µL 20 מדגם לתוך בארות ג'ל.
- להפעיל את הג'ל 10% קבוע הנוכחי 80 V עבור 120 דקות (עד הלהקה צבע מגיע לתחתית של הג'ל). לאחר השלמת אלקטרופורזה, לבטל את אספקת החשמל.
- נתק את המנגנון ג'ל ג'ל קלטות להוציא את הג'ל, ולממן את הג'ל לתוך מאגר העברה חצי יבש (48 מ מ טריס-HCl, 39 מ מ גליצין, 20% מתנול) במשך 5 דקות.
- קח עוד מיכל, לטבול את הקרום PVDF מתנול עבור 1 דקות. אז מוציאים PVDF מתנול ולשים מאגר העברה חצי יבש. בעדינות להתסיס את הקרום במשך 5 דקות.
- הסר את מכסה בטיחות של המנגנון electrophoretic חצי יבש.
- קדם מסנן נייר רטוב, ולהכין כריך ג'ל על האנודה התחתון פלטינה כדלקמן: נייר סינון PVDF ממברנה, ג'ל, נייר סינון.
- כיסוי צלחת ובטיחות קטודית המאובטח, והם בורחים כתם-15 קבוע V הנוכחי עבור 90 דקות התור אספקת החשמל, נתק מכשירים חצי יבש, להוציא את הקרום PVDF.
- ממברנה בלוק PVDF עם מאגר חסימה (5% שומן חלב במאגר TBST (137 מ מ NaCl2, 20 מ מ טריס-HCl, 0.1% דטרגנט B (ראה טבלה של חומרים), pH 7.6)) בטמפרטורת החדר במשך 1 h עם סל"ד 30 רועדת על ידי חומרים מסוכנים.
- Hybridize את קרום PVDF עם אנטי-p53 נוגדן מדולל (1:1, 000) במאגר חסימה עבור 12-16 h ב 4 ° C עם סל"ד 30 מנענע באמצעות מערבל ההשעיה.
- להוציא את הקרום PVDF לתוך מיכל ולשים TBST. לשטוף ממברנה PVDF עם TBST עוד פעמיים.
- משרים את הקרום PVDF, TBST למשך 30 דקות עם סל"ד 45 רועדת על ידי חומרים מסוכנים.
- Hybridize את קרום PVDF עם נוגדן אנטי-ארנב מצומדת עם חזרת peroxidase (HRP) מדולל (1:4, 000) במאגר החסימה לשעה בטמפרטורת החדר עם סל"ד 30 רועד באמצעות מערבל ההשעיה.
- לשטוף את הקרום PVDF עם TBST 3 פעמים כפי שמתואר בשלב 3.4.10.
- משרים את הקרום PVDF ב TBST למשך 30 דקות 45 rpm ייבוש באמצעות מערבל ההשעיה. להסיר TBST ולהוסיף PBS כדי לשמר את הקרום PVDF ב 4 ° C עבור עד 12 שעות.
- מערבבים הכימית המצע chemiluminescent משופרת (ECL המצע) 1 ו-2 (1:1) (ראה טבלה של חומרים). הסרת קרום PVDF מגניב, כתם בקצרה עם מגבת נייר או למטען קל קידמית סופג לחות עודפת, ולהוסיף מיד µL 400 של ריאגנט ECL אל פני השטח של כל הממברנה במשך 3-5 דקות להסיר עודפי ECL ריאגנט מאת בקצרה סופג, אך אינן מאפשרות של מ' embrane כדי יבש לחלוטין.
- לחשוף את האבן החשופה באמצעות מערכת הדמיה הפריה חוץ גופית או סרט autoradiography27.
תוצאות
על פי המידע שנמסר על ידי היצרן, כמות האנזים E1, E2 ב וזמינותו SUMOylation רגיל הוא 50 ננומטר, 500 ננומטר, בהתאמה. כמות מינימלית של E2 conjugating אנזים Ubc9 כי הוא מסוגל SUMOylate p53 נקבע לראשונה על ידי assay SUMOylation במבחנה . נמוך ככל חמישית מכמות Ubc9 המשמשים את תקן במבחנה SUMOylation assay פרוטוקול הצליח ביעילות SUMOylate p53 (איור 1א'). לכן, חצי מכמות האנזים E1 בשילוב עם עשירית מהסכום של Ubc9 בשימוש הפרוטוקול הסטנדרטי שימש עוד במבחנה assay SUMOylation. הפחתה משמעותית של יעילות SUMOylation נצפתה לעומת ההליך הרגיל (איור 1B).
לכן, היכולת של סומו E3 ליגאז K-bZIP כדי לשפר את p53 ש-sumoylation נקבע באמצעות אלה ששינה במבחנה SUMOylation תנאים. מתויג K-bZIP מטוהרים מתאי Sf9 (איור 2א) הועסק ב תגובה SUMOylation המכיל כמויות נמוכות של אנזימים E1, E2 Ubc9. בתנאים אלה ששונו, K-bZIP יכול ביעילות לעודד את SUMOylation של p53 (איור 2B). Immunoblotting עם נוגדן anti-p53 אישר את הכמויות הכולל דומה של p53 בתגובה לכל, גם סומו שזוהו ששינה p53.

איור 1 : קביעת הכמות המינימלית של אנזימים סומו בשימוש במבחנה וזמינותו SUMOylation. P53 (א) SUMOylation הוערך ב במבחנה SUMOylation assay הכלולות החמישי חצי אחד ואחד כמות האנזים Ubc9 להשתמש ב פרוטוקול סטנדרטי. לאחר 3 שעות של במבחנה SUMOylation, אנזימים p53 SUMOylated נבדקו על-ידי המערב כתם עם נוגדן anti-p53. (B) במבחנה SUMOylation של p53 בהמשך נותחו באמצעות חצי מכמות האנזים E1 בשילוב עם עשירית מכמות Ubc9 בשימוש הפרוטוקול הסטנדרטי. תספיג בוצע כמתואר כמו (A). אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
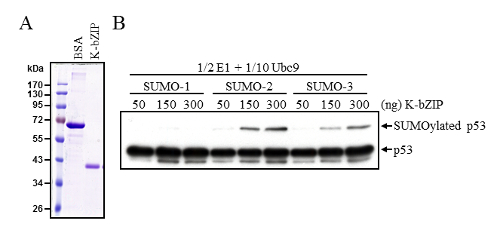
איור 2 : שיפור של p53 SUMOylation על ידי סומו E3 ליגאז K-bZIP- (א) מטוהרים הביע baculovirus חלבון K-bZIP, נותחו על ידי מרחביות-דף ואחריו עם Coomassie כחול מכתים. 1 µg BSA ו- 10 µL מטוהרים K-bZIP היו מעמיסים על השוואתי הכמות חלבון. (B) K-bZIP משופרת p53 ש-sumoylation הוערך על-ידי שימוש לתגובה SUMOylation במבחנה עם חצי כמות האנזים E1, עשירית מכמות Ubc9 להמליץ הפרוטוקול הסטנדרטי. SUMOylated p53 נחקרו על ידי תספיג כמתואר באיור 1א. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
במבחנה פרוטוקול SUMOylation המתוארים כאן באופן שגרתי משמש כדי לקבוע את מצב SUMOylation מזוהה Ubc9 סובסטרטים. המגבלה העיקרית באמצעות פרוטוקול סטנדרטי כדי לחקור את תפקוד SUMOylation סומו E3 ליגאז הוא השפע של הפעלת סומו E1, E2 conjugating אנזימים Ubc9 למקסם את ההטיה סומו במערכות במבחנה . בהתחשב באתגר זה, אנו מאמינים זה טיטור מהסכום של אנזימים סומו E1, E2 לשלב אחד בקושי יכול לזהות שאת הפעילות שלהם ב- SUMOylation זמין רק דרך לקבוע סומו E3 ליגאז קטליטי פעילויות באמצעות הזה חוץ גופית בתוך מערכת SUMOylation. בעקבות השערה זו, אנו בהצלחה מבואר E3 ליגאז הפעילות של K-bZIP, זיהה את זה כמו ליגאז סומו E3 הרומן עם ירידה לפרטים לקראת סומו-2/3.
השלב הקריטי בתוך הפרוטוקול הוא שילוב של כמויות נמוכות של E1, E2. באמצעות כמויות נמוכות אלה, כפי שמוצג באיור 2, הרומן סומו E3 ligases עם פעילות ליגאז נמוכה ייתכן תוכל להיות מזוהה עם שימור היחודיות שלהם כלפי המצע paralogues סומו. מגבלה משמעותית של הטכניקה היא הדרישה נוגדן באיכות גבוהה לזהות את המצע סומו, שכן רק אחוז קטן מאוד של המצע יכול להיות סומו שונה.
למרות חלבונים רבים ולהראות את הפוטנציאל להגדיל SUMOylation לאחר ביטוי ויוו, הגדרה של סומו E3 ליגאז עליך ניתן להדגים על ידי משוקם במבחנה SUMOylation מערכת באמצעות אנזימים E1, E2 ו- E3 מטוהרים. לעומת מאות, אולי אלפי אוביקוויטין E3 ligases מזוהה, עד עכשיו רק כמה ligases סומו E3 נמצאו. זה עשוי להיות בחלקו בשל השימוש המסורתי סטנדרטי במבחנה SUMOylation וזמינותו אשר ממקסם את יעילות ההטיה סומו, כתוצאה מכך מעכבת את תפקוד SUMOylation ligases סומו E3 פוטנציאליים. בפרוטוקול הנוכחי מתאר של assay פשוטה ועקבית כדי לחקור SUMOylation משופרת על ידי סומו E3 ליגאז. השיטה המתוארת חיוני זיהוי ואפיון של הרומן ligases סומו E3.
Disclosures
המחברים לחשוף אין פוטנציאל ניגודי עניינים.
Acknowledgements
עבודה זו נתמכה על ידי מענקים את משרד המדע והטכנולוגיה (רוב, 105-2320-B-010-007-MY3 כדי PCC), מן המכון מחקר הבריאות הלאומי (NHRI-EX105-10215BC כדי PCC), את משרד המדע והטכנולוגיה (ביותר 105-2314-B-400-019 כדי HJK) מ מכון המחקר הבריאות הלאומי (NHRI MG-105-SP-10, NHRI מ"ג-106-SP-10 כדי HJK). עבודה זו נתמכה גם חלקית עם האוניברסיטה הלאומית יאנג מינג על כתב היד את הפרסום PCC. התורמים שחיים היה אין תפקיד תכנון המחקר, איסוף נתונים, ניתוח, ההחלטה לפרסם או ותכליתם של כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| pFastBac | Invitrogen | 10359-016 | dual expression baculovirus vector |
| pCR2.1-TOPO vector | Invitrogen | PCR product vector | |
| competent cell E. coli DH5α | Yeastern Biotech | FYE678-80VL | competent E. coli cells A |
| E. coli DH10Bac | Invitrogen | 10359-016 | competent E. coli cells B |
| FugeneHD | Roche | 04709705001 | transfection reagent |
| Opti-MEM | Gibco | 31985062 | reduced serum media |
| T4 Ligase | NEW England BioLabs | M0202S | |
| CpoI (RsrII) | Thermo Scientific | ER0741 | |
| Bluo-gal | Thermo Scientific | B1690 | galactosidase substrate |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Grace’s Insect Medium | Gibco | 11605094 | |
| Fetal Bovine Serum | Gibco | 10082147 | |
| Gentamicin | Thermo Fisher | 15750060 | |
| Ampicillin | Sigma-Aldrich | A9393-25G | |
| Kanamycin | Sigma-Aldrich | K0254-20ML | |
| gentamicin | Gibco | 15710-064 | |
| tetracyclin | Sigma-Aldrich | 87128-25G | |
| LB Broth | Merk | 1.10285.0500 | |
| HEPES | Sigma-Aldrich | H4034-100G | |
| NaCl | Sigma-Aldrich | S9888-5KG | |
| KCl | Merk | 1.04936.1000 | |
| Na2HPO4 | Sigma-Aldrich | S5136-500G | |
| KH2PO4 | J.T.Baker | 3246-01 | |
| sodium dodecyl sulfate | Merk | 1.13760.1000 | |
| β-mercaptoethanol | Bio-Rad | 161-0710 | |
| TRIS (Base) | J.T.Baker | 4109-06 | |
| Non-fat milk | Fonterra | ||
| glycerol | J.T.Baker | 2136-01 | |
| Triton X-100 | Amresco | 0694-1L | detergent A |
| Tween 20 | Amresco | 0777-500ML | detergent B |
| Poloxamer 188 solution | Sigma-Aldrich | P5556-100ML | detergent C |
| Protease Inhibitor Cocktail Tablet | Roche | 04 693 132 001 | |
| 3x Flag peptide | Sigma | F4799 | |
| anti-FLAG m2 Magnetic beads | Sigma-Aldrich | M8823 | antibody-tagged magnetic beads |
| SUMOlink SUMO-1 Kit | Active Motif | 40120 | standard SUMOylation protocol |
| SUMOlink SUMO-2/3 Kit | Active Motif | 40220 | standard SUMOylation protocol |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAGEN Plasmid Mini Kit | QIAGEN | 12123 | plasmid extraction kit |
| Polypropylene tubes | Falcon | 352059 | |
| Petri Dish | Falcon | 351029 | |
| Cell lifter | Corning | CNG3008 | |
| Loading tip | Sorenson BioScience | 28480 | |
| PVDF | PerkinElmer | NEF1002 | |
| Blotting filter paper | Bio-Rad | 1703932 | |
| Mini slab gel apparatus (Bio-Rad Mini Protean II Cell) | Bio-Rad | 1658001 EDU | |
| Trans-Blot SD Semi-Dry Electrophoretic Transfer Cell | Bio-Rad | 1703940 | |
| Pierce ECL Western Blotting | Thermo | 32106 | ECL reagent |
| suspension mixer | Digisystem laboratory instruments Inc. | SM-3000 | |
| orbital shaker | Kansin instruments Co. | OS701 | |
| ImageQuant LAS 4000 biomolecular imager | GE Healthcare | 28955810 | |
| Sf9 | Thermo Scientific | B82501 | |
| anti-p53 antibody | Cell Signaling | #9282 | |
| anti-rabbit antibody | GE Healthcare | NA934-1ML |
References
- Bies, J., Markus, J., Wolff, L. Covalent attachment of the SUMO-1 protein to the negative regulatory domain of the c-Myb transcription factor modifies its stability and transactivation capacity. J Biol Chem. 277 (11), 8999-9009 (2002).
- Lin, D. Y., et al. Role of SUMO-interacting motif in Daxx SUMO modification, subnuclear localization, and repression of sumoylated transcription factors. Mol Cell. 24 (3), 341-354 (2006).
- Welsh, M., Jamalpour, M., Zang, G., Akerblom, B. The role of the Src Homology-2 domain containing protein B (SHB) in beta cells. J Mol Endocrinol. 56 (1), 21-31 (2016).
- Huang, W. Q., et al. Structure, function, and pathogenesis of SHP2 in developmental disorders and tumorigenesis. Curr Cancer Drug Targets. 14 (6), 567-588 (2014).
- Perry, J. J., Tainer, J. A., Boddy, M. N. A SIM-ultaneous role for SUMO and ubiquitin. Trends Biochem Sci. 33 (5), 201-208 (2008).
- Sun, H., Leverson, J. D., Hunter, T. Conserved function of RNF4 family proteins in eukaryotes: targeting a ubiquitin ligase to SUMOylated proteins. EMBO J. 26 (18), 4102-4112 (2007).
- Ouyang, J., Shi, Y., Valin, A., Xuan, Y., Gill, G. Direct binding of CoREST1 to SUMO-2/3 contributes to gene-specific repression by the LSD1/CoREST1/HDAC complex. Mol Cell. 34 (2), 145-154 (2009).
- Lyst, M. J., Stancheva, I. A role for SUMO modification in transcriptional repression and activation. Biochem Soc Trans. 35, 1389-1392 (2007).
- Liu, H. W., et al. Chromatin modification by SUMO-1 stimulates the promoters of translation machinery genes. Nucleic Acids Res. 40 (20), 10172-10186 (2012).
- Rosonina, E., Duncan, S. M., Manley, J. L. SUMO functions in constitutive transcription and during activation of inducible genes in yeast. Genes Dev. 24 (12), 1242-1252 (2010).
- Chang, P. C., et al. The chromatin modification by SUMO-2/3 but not SUMO-1 prevents the epigenetic activation of key immune-related genes during Kaposi's sarcoma associated herpesvirus reactivation. BMC Genomics. 14, 824 (2013).
- Neyret-Kahn, H., et al. Sumoylation at chromatin governs coordinated repression of a transcriptional program essential for cell growth and proliferation. Genome Res. 23 (10), 1563-1579 (2013).
- Tatham, M. H., et al. Polymeric chains of SUMO-2 and SUMO-3 are conjugated to protein substrates by SAE1/SAE2 and Ubc9. J Biol Chem. 276 (38), 35368-35374 (2001).
- Mattoscio, D., Segre, C. V., Chiocca, S. Viral manipulation of cellular protein conjugation pathways: The SUMO lesson. World J Virol. 2 (2), 79-90 (2013).
- Gareau, J. R., Lima, C. D. The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. Nat Rev Mol Cell Biol. 11 (12), 861-871 (2010).
- Bernier-Villamor, V., Sampson, D. A., Matunis, M. J., Lima, C. D. Structural basis for E2-mediated SUMO conjugation revealed by a complex between ubiquitin-conjugating enzyme Ubc9 and RanGAP1. Cell. 108 (3), 345-356 (2002).
- Knipscheer, P., et al. Ubc9 sumoylation regulates SUMO target discrimination. Mol Cell. 31 (3), 371-382 (2008).
- Vethantham, V., Manley, J. L. In vitro sumoylation of recombinant proteins and subsequent purification for use in enzymatic assays. Cold Spring Harb Protoc. 2009 (1), 5121 (2009).
- Lin, S. F., Robinson, D. R., Miller, G., Kung, H. J. Kaposi's sarcoma-associated herpesvirus encodes a bZIP protein with homology to BZLF1 of Epstein-Barr virus. J Virol. 73 (3), 1909-1917 (1999).
- Yang, W. S., Hsu, H. W., Campbell, M., Cheng, C. Y., Chang, P. C. K-bZIP Mediated SUMO-2/3 Specific Modification on the KSHV Genome Negatively Regulates Lytic Gene Expression and Viral Reactivation. PLoS Pathog. 11 (7), 1005051 (2015).
- Connor, C. D., Metcalf, E., Wrighton, C. J., Harris, T. J., Saunders, J. R. RsrII--a novel restriction endonuclease with a heptanucleotide recognition site. Nucleic Acids Res. 12 (17), 6701-6708 (1984).
- Helling, R. B., Goodman, H. M., Boyer, H. W. Analysis of endonuclease R-EcoRI fragments of DNA from lambdoid bacteriophages and other viruses by agarose-gel electrophoresis. J Virol. 14 (5), 1235-1244 (1974).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Gingold, E. B. Bacterial transformation. Methods Mol Biol. 2, 237-240 (1985).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. J Virol. 86 (18), 9708-9720 (2012).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Ahmed, S., Holt, M., Riedel, D., Jahn, R. Small-scale isolation of synaptic vesicles from mammalian brain. Nat Protoc. 8 (5), 998-1009 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved