Method Article
In Vitro Ensaio de SUMOylation para estudar a atividade de Ligase E3 sumô
Neste Artigo
Resumo
Ao contrário da ubiquitina ligases, foram identificados alguns ligases E3 sumô. Este modificado em vitro protocolo SUMOylation é capaz de identificar o romance sumô E3 ligases por um ensaio de reconstituição em vitro .
Resumo
Modificação de pequenos ubiquitin-como modificador (SUMO) é uma importante modificação pós-traducional (PTM) que medeia a transdução de sinal, principalmente através de interações da proteína-proteína de modulação. Semelhante à modificação da ubiquitina, SUMOylation é dirigido por uma cascata de enzima sequencial incluindo enzima E1-ativando (SAE1/SAE2), enzima E2-conjugação (Ubc9) e E3-ligase (i.e., família de PIAS, RanBP2 e Pc2). No entanto, diferente da ubiquitination, uma ligase E3 é não-essenciais para a reação, mas não fornecem precisão e eficácia para conjugação de sumô. Modificado por SUMOylation de proteínas podem ser identificadas por ensaio na vivo através da imunoprecipitação com anticorpos específicos do substrato e immunoblotting com anticorpos específicos de sumô. No entanto, a demonstração da atividade de ligase E3 sumô proteína requer em vitro reconstituição dos ensaios SUMOylation usando enzimas purificadas, substrato e proteínas de sumô. Desde que nas reações em vitro , geralmente SAE1/SAE2 e Ubc9, só são suficientes para conjugação de sumô, realce de SUMOylation por um putativo ligase E3 nem sempre é fácil de detectar. Aqui, descrevemos um modificado em vitro protocolo de SUMOylation que identifica consistentemente a modificação de sumô usando um sistema em vitro reconstituído. Um protocolo passo a passo para purificar cataliticamente ativo K-bZIP, um viral ligase E3 de sumô-2/3, também é apresentado. As atividades de SUMOylation da K-bZIP purificada são mostradas na p53, um destino bem conhecido do sumô. Este protocolo pode não apenas ser empregado para elucidar o romance sumô E3 ligases, mas também por revelar a sua especificidade de paralog de sumô.
Introdução
Modificação de sumô foi inicialmente identificada como uma modificação pós-traducional reversível (PTM) que regula a proteína estabilidade1. Além de conjugação direta, sumô também pode ser anexado a uma proteína através de interação não-covalente de sumô interação motivos (SIMs)2. Semelhante a vinculação de correntes-fosforilação por moléculas abrigando Src homologia 2 (SH2) ou ligação phosphotyrosine (PTB) domínios3,4, modificação de sumô fornece uma plataforma de interação adicional para seletiva recrutamento de SIM-contendo effector proteínas5,6. Além do Regulamento de transdução de sinal, SUMOylation de fatores transcricionais e cromatina remodelação moléculas servem para modular a expressão de gene7,8. Estudos dos padrões de SUMOylation de todo o genoma revelaram que a modificação de sumô está associada com positivo9,10 ou negativo10,11,12 transcrição Regulamento, em parte devido à especificidade de paralogue de SUMOylation.
Existem três principais isoformas de sumô para proteína, conjugando presente em células de mamíferos; Estes incluem SUMO-1 e altamente homólogos sumô-2 e 3-sumô (referido como SUMO-2/3)13. SUMÔ é normalmente conjugado com o resíduo de lisina (K) dentro do motivo de ψKxD/E consenso na proteína alvo. O cartão SIM no sumô E3 ligases é responsável para a especificidade de paralogue14. Em contraste com os caminhos da ubiquitination contendo centenas de E3 ligases, somente há alguns sumô E3 ligases identificados15. Ligases E3 de sumô são definidos pelas propriedades, incluindo a sua capacidade de (i) ligar Ubc9, (ii) ligar moiety sumô via um domínio SIM e (iii) melhorar a transferência SUMO de Ubc9 em carcaça. Ligase E3 não é absolutamente necessário para sumô conjugação16, mas fornece o substrato e a especificidade de sumô-paralogue. Desde geralmente apenas uma pequena fração da proteína total do substrato é sumô-modificado, detecção de SUMOylated proteínas na vivo é sempre um desafio. Portanto, ensaios precisos e reprodutíveis são necessárias a fim de elucidar a função precisa de modificação de sumô.
O em vitro ensaio de SUMOylation, que avalia a capacidade de Ubc9 purificado para catalisar SUMOylation das proteínas do substrato, é um ensaio bem aceitado para o estudo de modificação de sumô17. No entanto, a atividade de ligase E3 de sumô na maioria dos casos não pode ser detectada ou só pode ser detectada com mutantes ligase do sumô com alta atividade de ligase E3 de sumô e baixa especificidade pelo protocolo padrão devido à presença de uma quantidade abundante de Ubc918 . O sucesso deste teste depende em grande parte à titulação cuidadosa de Ubc9 purificado. O ensaio SUMOylation em vitro descrito aqui é modificado de uma SUMOylation padrão protocolo (consulte Tabela de materiais). A observação de aumento global modificação de sumô durante a reativação de herpesvírus associado sarcoma de Kaposi (KSHV) levou-na identificar o ligase E3 sumô viral que pode ser responsável para a regulação da SUMOylation. Após uma triagem em pequena escala das proteínas de interação de viral sumô E3 ligase do K-bZIP, p53 foi identificado como um substrato de romance. Neste protocolo, mostramos detalhadamente em células de inseto as etapas envolvidas na expressão e purificação do selvagem-tipo K-bZIP, uma ligase E3 sumô viral com especificidade de sumô-2/3. A capacidade do K-bZIP purificado de aumentar SUMOylation de p53, um substrato de sumô bem conhecido, é avaliada na presença de quantidades reduzidas de enzimas E1 e E2. Usando este ensaio SUMOylation modificado, investigadores podem confiantemente definir as habilidades de sumô E3 ligase do SUMOylate romance ou substratos conhecidos, que é uma etapa preliminar no estudo da proteína SUMOylation. Além disso, esse método ajuda a identificar romance sumô E3 ligases com ligase baixa atividade, mas alta especificidade.
Protocolo
1. preparação de Baculovirus expressão constrói
-
Purificar o sumô E3 ligase K-bZIP por clonagem do cDNA de K-bZIP19 em um vetor de baculovirus expressão dual (ver tabela de materiais) com uma epítopo-tag do N-terminal. Temos tido sucesso usando uma marca de Octapeptídeo (indicada em todo o protocolo como vetor-marca-K-bZIP).
Nota: O modelo de DNA para clonagem de reação em cadeia (PCR) K-bZIP polimerase é cDNA reverso transcrito de RNA isolado de células de TREx F3H3-K-Rta BCBL-1 doxiciclina tratamento20a seguir.- Transferi o longa-metragem de cDNA K-bZIP19 para o vetor de expressão dupla com Cpoeu clonagem de sites. CPO Eu digerir o produto do PCR e 1 µ g do vetor de expressão dual a 37 ° C por 3 h21.
- Separar e extrair o DNA digerido após eletroforese em gel de agarose 0,8%22. Ligate a inserção (K-bZIP) e vector por T4 DNA ligase a 16 ° C por 30 min, de acordo com as instruções do fabricante (ver Tabela de materiais).
- Adicionar 5 µ l de DNA de ligadura a 50 µ l competentes de Escherichia coli células "A" (ver Tabela de materiais) em um tubo de 1,5 mL. Ponha gelo durante 30 min. Em seguida, incubar o tubo de 1,5 mL em um banho de água de 42 ° C durante 45 s e imediatamente colocar o tubo no gelo por 3 min.
- Adicione 600 médio de S.O.C µ l (extrato de levedura 0,5% (p/v), triptona de 2% (p/v), 10 mM de NaCl, 2,5 mM KCl, 20 mM de MgSO4e 4% (p/v) glicose) para dentro do tubo e incubar a 37 ° C, durante 1 h. Em seguida, centrifugar o tubo em temperatura ambiente por 3 min a 600 x g.
- Remover o sobrenadante, resuspenda o pellet com meio de Luria-Bertani 60 µ l (LB; triptona 1% (p/v), 0,5% (p/v) do fermento extrato e 85 mM de NaCl) e distribuídos em placas de ágar LB contendo 100 ampicilina µ g/mL. Incube a placa de ágar a 37 ° C, durante 16 h23,24.
- Selecione 1 colônia para inocular em meio de LB de 5 mL contendo 100 µ g/mL ampicilina e cultura a 37 ° C durante 16 h com 250 rpm a tremer. Em seguida, extrair o plasmídeo, vetor-marca-K-bZIP, por um kit de extração de plasmídeo de acordo com as instruções do fabricante (ver Tabela de materiais).
-
Gerar o bacmid recombinante, abrigando a marca-K-bZIP pela transformação do vetor-marca-K-bZIP competentes de Escherichia coli células "B" (ver tabela de materiais).
- Misture o plasmídeo de vetor-marca-K-bZIP 0,2 µ g e 100 µ l competentes E. células de coli "B" em um tubo de 1,5 mL e colocar no gelo por 30 min. incubam o tubo em banho de água a 42 ° C por 45 s e imediatamente colocar o tubo no gelo por 3 min.
- Adicionar 900 µ l de meio S.O.C no tubo e incubar a 37 ° C por 4 h, com rotação a 50 rpm. Em seguida, tomar 10 µ l do meio, adicionar um adicionais 50 µ l de meio S.O.C para espalhar em placas de ágar LB contendo 50 µ g/mL canamicina, gentamicina 7 de µ g/mL, 10 tetraciclina µ g/mL, substrato de galactosidase 100 µ g/mL e 40 µ g/mL isopropílico β-D-1-thiogalactopyranoside (IPTG ) e incubar as placas por 48 h a 37 ° C.
- Inocule uma colônia branca em 5 mL de meio de LB contendo 50 µ g/mL canamicina, gentamicina 7 de µ g/mL e 10 tetraciclina µ g/mL. Incube a 37 ° C com 250 rpm agitação por 16 h.
- Isolar o bacmid recombinante DNA abrigando a marca-K-bZIP, quantificar e ressuspender em uma concentração de 1 µ g / µ l25.
-
Gere baculoviruses recombinante expressando marca-K-bZIP por transfecting, 1 µ g de bacmid recombinante DNA contendo marca-K-bZIP Sf9 células em um poço de uma placa de 6.
- Semente de 2 x 106 Sf9 células (ver Tabela de materiais) de 2ml Grace inseto médio fornecido com 10% de soro fetal bovino (FBS) e gentamicina 1% em cada um bem de uma placa bem 6 e, em seguida, incubam a 27 ° C, durante 1 h.
- Manter uma cultura de fase de registo de células Sf9 da Grace inseto médio fornecido com 10% FBS, gentamicina 1% e 1% de detergente C (ver Tabela de materiais) a 27 ° C em suspensão orbital a 140 rpm. Contar as células usando um hemocytometer e mancha azul trypan; viabilidade celular deve exceder 95%. Manter as células usando uma proporção de subcultura de 1:3 a cada 2-3 dias (densidade mínima ~0.5 x 106 células/mL).
- Adicionar 2 µ l bacmid DNA (1 µ g / µ l) e 98 µ l reduzidos meios de soro (ver Tabela de materiais) para um tubo de 2 mL. Em seguida, adicionar o reagente de transfeccao 4 µ l (veja a Tabela de materiais) para o tubo e misture bem. Incube a temperatura ambiente por 15 min.
- Adicione esta mistura de transfeccao (104 µ l) em cada poço e incubar a 27 ° C, durante 12-16 h.
- Remova a mídia exausta com aspiração suave e substitua com 2 mL do meio de inseto da Grace fresca fornecido com Gentamicina FBS e 1% de 10%. As células Sf9 aderem frouxamente a superfície da placa e não será perturbadas com aspiração suave.
-
Depois de transfeccao baculovirus recombinante e amplificam baculovirus para obter alta-título estoques (P1) para novas experiências.
- 72 h depois de mudar o meio, raspar o Sf9 células usando um levantador de pilha de estéril, recolher o sobrenadante de cada poço individual em um tubo de 1,5 mL e vórtice por 10 s.
- Centrifugar a 4 ° C por 3 min em 150 g. x sobrenadante cobrar como P0 baculovirus e alíquota em tubos de 1,5 mL (1 mL em cada tubo de baculovirus). Loja a-80 ° C.
- Semente de 1 x 107 Sf9 células inseto médio de 10ml Grace fornecido com Gentamicina FBS e 1% de 10% em uma placa de Petri de 10 cm e incuba a 27 ° C, durante 1 h.
- Para a amplificação de baculoviruses recombinante expressando marca-K-bZIP, adicionar 0,5 mL P0 baculovirus às células Sf9 semeadas e incubar a 27 ° C por 48 h.
- Recolha o sobrenadante como P1 baculovirus em uma loja a 4 ° C e o tubo de 15 mL até 1 mês.
2. purificação de K-bZIP
- Manter uma cultura de fase de registo de células Sf9 da Grace inseto médio fornecido com 10% FBS, gentamicina 1% e 1% de detergente C a 27 ° C em suspensão orbital a 140 rpm conforme descrito na etapa 1.3.2.
- No dia de transdução, adicione 1 mL do sobrenadante contendo baculovirus (P1) em 250 mL de Sf9 células com uma densidade de 2 x 106 células/mL.
- Incubar as células Sf9 por 72 h a 27 ° C em um agitador orbital com 140 rpm a tremer e, em seguida, recolher por centrifugação a 2.740 x g, durante 15 min a 4 ° C.
- Remover o sobrenadante e lyse pelotas de célula em 10 mL de tampão de lise (20 mM HEPES pH 7,9, 0,5 M de NaCl, 1% de detergente um (ver Tabela de materiais), 2% glicerol, coquetel de inibidor de protease) e gira a 50 rpm usando um misturador de suspensão a 4 ° C por 30 min.
- Lisado em 15.000 x g, durante 15 min recolher o sobrenadante celular de centrífuga.
- Para purificar a marca-K-bZIP, incubar os lisados celulares (10 mL) com 50 µ l de anticorpo marcado grânulos magnéticos em tubos de polipropileno de 14 mL e gira a 50 rpm por 3 h a 4 ° C, utilizando um misturador de suspensão. Após a captura de proteína, granular os grânulos em 800 x centrifugação g por 30 s a 4 ° C. Remover a maior parte do sobrenadante, re-suspender os grânulos do volume residual de cerca de 1 mL e transferir para um tubo de 1,5 mL para lavar.
- Lave a proteína capturada, colocando o tubo contendo os grânulos num suporte magnético e remover o sobrenadante, uma vez que os grânulos magnéticos aderiram totalmente ao lado do tubo.
-
Lave os grânulos com Lise quatro vezes.
- Adicionar 1 mL de tampão de Lise para tubos de 1,5 mL e inverter os tubos 10 vezes. Coloque o tubo de 1,5 mL num suporte magnético e remover o sobrenadante, conforme descrito em 2.7.
- Repita a etapa anterior outro 3 vezes.
-
Lave os grânulos com solução salina tamponada fosfato (PBS) uma vez.
- Adicione 1 mL de PBS (137 mM NaCl, 2,7 mM KCl, 8mm Na2HPO4, 1,5 mM KH2PO4) para tubos de 1,5 mL e inverter os tubos 10 vezes.
- Colocar o tubo de 1,5 mL num suporte magnético e em seguida, remover o sobrenadante.
- Eluir marca-K-bZIP proteína de grânulos magnéticos anticorpo marcado pela adição de 100 µ l de 150 Octapeptídeo µ g/mL, diluído em PBS em um tubo de 1,5 mL. Gire o tubo por 10 min a 50 rpm pelo misturador de suspensão em temperatura ambiente. Em seguida, coloque o tubo de volta a depor magnético e recolher o sobrenadante contendo K-bZIP em um tubo limpo 1,5 mL.
- Analise a 1-5 µ l purificado marca-K-bZIP proteína por SDS-PAGE seguido de azul de Coomassie coloração26. Carga de 2 µ g, 1 µ g, e 0,5 µ g amostras de albumina de soro bovino (BSA) no mesmo gel e usá-lo para quantificação com um programa de processamento de imagem, como o ImageJ. Estime a concentração de K-bZIP em comparação com os padrões de BSA.
- O K-bZIP purificado está agora pronto para ser usado em cada ensaio de SUMOylation. Diluir o K-bZIP em PBS para uma concentração final de 100 ng / µ l e loja em pequenas alíquotas a-80 ° C.
3. ensaio de SUMOylation
- Adicione 3 µ l de 100 ng / µ l da marca-K-bZIP purificada na mistura mestre de cada em vitro reação SUMOylation. Componentes na mistura mestre contêm tampão de proteína 1 µ l, 1 x buffer de SUMOylation, proteína de p53 0,5 µ l (0,5 mg/mL), 25 nM de E1 enzima de activação (conc. estoque: 1 µM), 50 nM de E2 enzima de conjugação (conc. estoque: 10 µM) e 250 nM cada dos peptides sumô (sumô-1 , Sumô-2, ou SUMO-3; conc. estoque: 5 µM) até um volume final de 17 µ l.
- Misturar o conteúdo suavemente e incubar a reação a 30 ° C por 3 h. pare a reação adicionando 20 µ l de 2x do amortecedor do carregamento SDS-PAGE (pH 100 mM Tris-HCL 6,8, 200mm β-Mercaptoetanol, azul de bromofenol 0,2% (p/v), 20% (v/v) glicerol e 4% de sódio Dodecil sulfato) e Desnature as amostras a 95 ° C por 5 min.
-
Amostras separadas em SDS-PAGE, transferir as proteínas separadas em membrana PVDF usando um aparelho de transferência eletroforética semi-seca e analisar por Western blot utilizando anticorpo anti-p53.
- Configurar o aparelho de gel como na etapa 2.11 e carregar 20 µ l de amostra no gel de poços.
- Funcione o gel de 10% em uma constante corrente à 80 V por cerca de 120 min (até a banda de corante atinge a parte inferior do gel). Após a conclusão da electroforese, desligue a fonte de alimentação.
- Desligue o aparelho de gel e gel de fitas para tirar o gel e flutuar o gel em buffer de transferência semi secos (48 mM Tris-HCl, glicina 39 milímetros, 20% metanol) por 5 min.
- Pegue outra vasilha e mergulhe a membrana PVDF em metanol por 1 min. Em seguida, tirar PVDF metanol e colocar no buffer de transferência semi secos. Agite suavemente a membrana por 5 min.
- Remova a tampa de segurança do aparelho eletroforética semi seco.
- Papel de filtro previamente molhado e preparar um sanduíche de gel no ânodo inferior platina como segue: papel de filtro, membrana PVDF, gel e papel de filtro.
- Cobertura de placa e segurança seguro de cátodo, e em seguida fugiu do borrão na corrente de 90 min. vire de 15 constante V fonte de alimentação, desconecte o aparelho semi seco e tirar a membrana PVDF.
- Membrana de PVDF de bloco com tampão de bloqueio (5% leite desnatado no buffer TBST (137 mM NaCl2, 20 mM Tris-HCl, detergente 0,1% B (ver Tabela de materiais), pH 7,6)) à temperatura ambiente durante 1 h com 30 rpm tremendo pelo agitador orbital.
- Cruzar a membrana PVDF com anti-p53 anticorpo diluído (1:1, 000) em tampão de bloqueio para 12-16 h a 4 ° C com agitação de 30 rpm usando um misturador de suspensão.
- Pegue a membrana PVDF em um recipiente e coloque em TBST. Lave a membrana PVDF com TBST mais duas vezes.
- Mergulhe a membrana PVDF no TBST por 30 min com 45 rpm tremendo pelo agitador orbital.
- Cruzar a membrana PVDF com anticoelho anticorpo conjugado com peroxidase de rábano (HRP) diluído (1:4, 000) em tampão de bloqueio por 1h à temperatura ambiente com 30 rpm usando um misturador de suspensão a tremer.
- Lave a membrana PVDF com TBST 3 vezes, conforme descrito na etapa 3.4.10.
- Mergulhe a membrana PVDF no TBST por 30 min com 45 rpm agitação usando um misturador de suspensão. Remover TBST e adicionar PBS para preservar a membrana PVDF a 4 ° C por até 12 h.
- Misture o reagente substrato quimioluminescente melhorado (substrato ECL) 1 e 2 (1:1) (ver Tabela de materiais). Retire a membrana PVDF a PBS, borre brevemente com uma toalha de papel ou limpador de ligeiros para absorver o excesso de umidade e imediatamente adicionar 400 µ l de reagente ECL à superfície de cada membrana para remover excesso ECL 3-5min reagente borrando brevemente, mas não permita que o m embrane para completamente seco.
- Expor a mancha usando um sistema de imagem de luminescência ou autoradiografia filme27.
Resultados
De acordo com as informações fornecidas pelo fabricante, a quantidade padrão de enzima E1 e E2 no ensaio de SUMOylation é de 50 nM e 500 nM, respectivamente. Primeiro, a quantidade mínima de E2, conjugando a enzima Ubc9 que é capaz de SUMOylate p53 foi determinada por um ensaio de SUMOylation em vitro . Tão baixo como um quinto da quantidade de Ubc9 usado no padrão em vitro SUMOylation protocolo de ensaio foi capaz de forma eficiente SUMOylate p53 (Figura 1A). Portanto, metade da quantidade de enzima E1 em combinação com um décimo da quantidade de Ubc9 usado no protocolo padrão foi usado para outra em vitro ensaio SUMOylation. Observou-se uma redução significativa da eficiência SUMOylation em comparação com o protocolo padrão (Figura 1B).
Assim, a capacidade de sumô E3 ligase K-bZIP de aumentar p53 que sumoylation foi determinada utilizando estas modificado em vitro SUMOylation condições. K-bZIP etiquetado purificado de células Sf9(Figura 2)foi empregado em uma reação de SUMOylation que contém menor quantidade de enzimas E1 e E2 Ubc9. Sob estas condições modificadas, K-bZIP eficientemente poderia catalisar o SUMOylation de p53 (Figura 2B). Immunoblotting com anticorpos anti-p53 confirmou as quantidades totais semelhantes de p53 em cada reação, e também detectado sumô modificado p53.

Figura 1 : Determinação da quantidade mínima de enzimas de sumô utilizados no ensaio em vitro SUMOylation. (A) p53 que sumoylation foi avaliada em um em vitro ensaio de SUMOylation que incluía um meia e um quinto a quantidade de enzima Ubc9 usada no protocolo padrão. Depois de 3 horas em vitro reação SUMOylation, SUMOylated p53 enzimas foram examinadas por Western blot com anticorpo anti-p53. (B) em vitro SUMOylation de p53 foi analisado ainda mais usando a metade da quantidade de enzima E1 em combinação com um décimo do quantidade de Ubc9 usado no protocolo padrão. Um Western blot foi realizado conforme descrito como em (A). Clique aqui para ver uma versão maior desta figura.
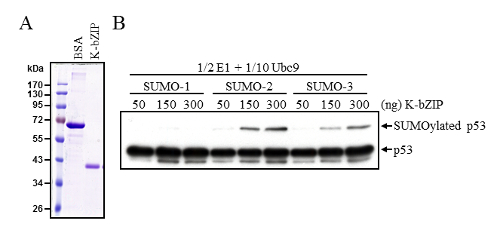
Figura 2 : Reforço da p53 SUMOylation pelo sumô E3 ligase bZIP-K. (A) purificado baculovirus-expressa proteína K-bZIP, analisada por SDS-PAGE seguido com coloração azul de Coomassie. 1 µ g BSA e 10 µ l purificado K-bZIP estavam carregando para comparativo da quantidade de proteína. (B) K-bZIP reforçada p53 que sumoylation foi avaliada por meio de uma reação de SUMOylation em vitro com metade da quantidade de enzima E1 e um décimo do quantidade de Ubc9 recomendo no protocolo padrão. SUMOylated p53 foram investigados por Western blot, conforme descrito na Figura 1A. Clique aqui para ver uma versão maior desta figura.
Discussão
O em vitro SUMOylation protocolo descrito aqui é rotineiramente utilizado para estabelecer o status SUMOylation de substratos de Ubc9 identificados. A limitação principal usando o protocolo padrão para estudar a função de SUMOylation de sumô E3 ligase é a abundância de ativação de sumô E1 e E2 conjugação enzimas Ubc9 que maximizam a conjugação de sumô em sistemas em vitro . Considerando esse desafio, nós acreditamos que essa titulação da quantidade de enzimas sumô E1 e E2 para o nível que um mal pode detectar que sua atividade em SUMOylation é o caminho disponível apenas para determinar atividades de catalítica de ligase E3 sumô usando esta em vitro Sistema de SUMOylation. Seguindo esta hipótese, conseguimos elucidado a atividade de ligase E3 de K-bZIP e identificou-o como um romance ligase E3 de sumô com especificidade para sumô-2/3.
O passo crítico no âmbito do protocolo é a incorporação de menor quantidade de E1 e E2. Usar estes montantes inferiores, como mostrado na Figura 2, romance sumô E3 ligases com atividade ligase baixa pode ser capaz de ser identificado com a preservação de sua especificidade para substrato e paralogues de sumô. Uma grande limitação da técnica é a exigência de um anticorpo de alta qualidade, reconhecendo o substrato de sumô, uma vez que apenas uma proporção muito pequena do substrato pode ser modificado de sumô.
Embora muitas proteínas mostram potencial para aumentar SUMOylation após superexpressão na vivo, definir uma ligase E3 sumô deve ser demonstrada por um sistema SUMOylation reconstituído em vitro usando enzimas purificadas de E1, E2 e E3. Em oposição às centenas e talvez milhares de ubiquitina E3 ligases identificados, até agora apenas alguns sumô E3 ligases foram encontrados. Isto pode ser devido em parte ao uso do ensaio tradicional padrão em vitro SUMOylation que maximiza a eficiência de conjugação de sumô e consequentemente dificulta a função SUMOylation do potencial sumô E3 ligases. O presente protocolo descreve um ensaio simples e consistente para sonda SUMOylation reforçada por uma ligase E3 de sumô. O método descrito é essencial para a identificação e caracterização do romance sumô E3 ligases.
Divulgações
Os autores não divulgar nenhuma potenciais conflitos de interesse.
Agradecimentos
Este trabalho foi financiado por doações do Ministério da ciência e tecnologia (a maioria, 105-2320-B-010-007-MY3 a PCC), do Instituto Nacional de pesquisa de saúde (INDH-EX105-10215BC, a PCC), do Ministério da ciência e tecnologia (mais 105-2314-B-400-019 para HJK) e do Instituto Nacional de pesquisas saúde (INDH MG-105-SP-10, INDH MG-106-SP-10 para HJK). Este trabalho também foi financiado em parte com a Universidade Nacional de Yang-Ming na publicação do manuscrito para o PCC. Os financiadores não tinham qualquer papel no projeto de estudo, coleta de dados e análise, a decisão de publicar ou reparação do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| pFastBac | Invitrogen | 10359-016 | dual expression baculovirus vector |
| pCR2.1-TOPO vector | Invitrogen | PCR product vector | |
| competent cell E. coli DH5α | Yeastern Biotech | FYE678-80VL | competent E. coli cells A |
| E. coli DH10Bac | Invitrogen | 10359-016 | competent E. coli cells B |
| FugeneHD | Roche | 04709705001 | transfection reagent |
| Opti-MEM | Gibco | 31985062 | reduced serum media |
| T4 Ligase | NEW England BioLabs | M0202S | |
| CpoI (RsrII) | Thermo Scientific | ER0741 | |
| Bluo-gal | Thermo Scientific | B1690 | galactosidase substrate |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Grace’s Insect Medium | Gibco | 11605094 | |
| Fetal Bovine Serum | Gibco | 10082147 | |
| Gentamicin | Thermo Fisher | 15750060 | |
| Ampicillin | Sigma-Aldrich | A9393-25G | |
| Kanamycin | Sigma-Aldrich | K0254-20ML | |
| gentamicin | Gibco | 15710-064 | |
| tetracyclin | Sigma-Aldrich | 87128-25G | |
| LB Broth | Merk | 1.10285.0500 | |
| HEPES | Sigma-Aldrich | H4034-100G | |
| NaCl | Sigma-Aldrich | S9888-5KG | |
| KCl | Merk | 1.04936.1000 | |
| Na2HPO4 | Sigma-Aldrich | S5136-500G | |
| KH2PO4 | J.T.Baker | 3246-01 | |
| sodium dodecyl sulfate | Merk | 1.13760.1000 | |
| β-mercaptoethanol | Bio-Rad | 161-0710 | |
| TRIS (Base) | J.T.Baker | 4109-06 | |
| Non-fat milk | Fonterra | ||
| glycerol | J.T.Baker | 2136-01 | |
| Triton X-100 | Amresco | 0694-1L | detergent A |
| Tween 20 | Amresco | 0777-500ML | detergent B |
| Poloxamer 188 solution | Sigma-Aldrich | P5556-100ML | detergent C |
| Protease Inhibitor Cocktail Tablet | Roche | 04 693 132 001 | |
| 3x Flag peptide | Sigma | F4799 | |
| anti-FLAG m2 Magnetic beads | Sigma-Aldrich | M8823 | antibody-tagged magnetic beads |
| SUMOlink SUMO-1 Kit | Active Motif | 40120 | standard SUMOylation protocol |
| SUMOlink SUMO-2/3 Kit | Active Motif | 40220 | standard SUMOylation protocol |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAGEN Plasmid Mini Kit | QIAGEN | 12123 | plasmid extraction kit |
| Polypropylene tubes | Falcon | 352059 | |
| Petri Dish | Falcon | 351029 | |
| Cell lifter | Corning | CNG3008 | |
| Loading tip | Sorenson BioScience | 28480 | |
| PVDF | PerkinElmer | NEF1002 | |
| Blotting filter paper | Bio-Rad | 1703932 | |
| Mini slab gel apparatus (Bio-Rad Mini Protean II Cell) | Bio-Rad | 1658001 EDU | |
| Trans-Blot SD Semi-Dry Electrophoretic Transfer Cell | Bio-Rad | 1703940 | |
| Pierce ECL Western Blotting | Thermo | 32106 | ECL reagent |
| suspension mixer | Digisystem laboratory instruments Inc. | SM-3000 | |
| orbital shaker | Kansin instruments Co. | OS701 | |
| ImageQuant LAS 4000 biomolecular imager | GE Healthcare | 28955810 | |
| Sf9 | Thermo Scientific | B82501 | |
| anti-p53 antibody | Cell Signaling | #9282 | |
| anti-rabbit antibody | GE Healthcare | NA934-1ML |
Referências
- Bies, J., Markus, J., Wolff, L. Covalent attachment of the SUMO-1 protein to the negative regulatory domain of the c-Myb transcription factor modifies its stability and transactivation capacity. J Biol Chem. 277 (11), 8999-9009 (2002).
- Lin, D. Y., et al. Role of SUMO-interacting motif in Daxx SUMO modification, subnuclear localization, and repression of sumoylated transcription factors. Mol Cell. 24 (3), 341-354 (2006).
- Welsh, M., Jamalpour, M., Zang, G., Akerblom, B. The role of the Src Homology-2 domain containing protein B (SHB) in beta cells. J Mol Endocrinol. 56 (1), 21-31 (2016).
- Huang, W. Q., et al. Structure, function, and pathogenesis of SHP2 in developmental disorders and tumorigenesis. Curr Cancer Drug Targets. 14 (6), 567-588 (2014).
- Perry, J. J., Tainer, J. A., Boddy, M. N. A SIM-ultaneous role for SUMO and ubiquitin. Trends Biochem Sci. 33 (5), 201-208 (2008).
- Sun, H., Leverson, J. D., Hunter, T. Conserved function of RNF4 family proteins in eukaryotes: targeting a ubiquitin ligase to SUMOylated proteins. EMBO J. 26 (18), 4102-4112 (2007).
- Ouyang, J., Shi, Y., Valin, A., Xuan, Y., Gill, G. Direct binding of CoREST1 to SUMO-2/3 contributes to gene-specific repression by the LSD1/CoREST1/HDAC complex. Mol Cell. 34 (2), 145-154 (2009).
- Lyst, M. J., Stancheva, I. A role for SUMO modification in transcriptional repression and activation. Biochem Soc Trans. 35, Pt 6 1389-1392 (2007).
- Liu, H. W., et al. Chromatin modification by SUMO-1 stimulates the promoters of translation machinery genes. Nucleic Acids Res. 40 (20), 10172-10186 (2012).
- Rosonina, E., Duncan, S. M., Manley, J. L. SUMO functions in constitutive transcription and during activation of inducible genes in yeast. Genes Dev. 24 (12), 1242-1252 (2010).
- Chang, P. C., et al. The chromatin modification by SUMO-2/3 but not SUMO-1 prevents the epigenetic activation of key immune-related genes during Kaposi's sarcoma associated herpesvirus reactivation. BMC Genomics. 14, 824(2013).
- Neyret-Kahn, H., et al. Sumoylation at chromatin governs coordinated repression of a transcriptional program essential for cell growth and proliferation. Genome Res. 23 (10), 1563-1579 (2013).
- Tatham, M. H., et al. Polymeric chains of SUMO-2 and SUMO-3 are conjugated to protein substrates by SAE1/SAE2 and Ubc9. J Biol Chem. 276 (38), 35368-35374 (2001).
- Mattoscio, D., Segre, C. V., Chiocca, S. Viral manipulation of cellular protein conjugation pathways: The SUMO lesson. World J Virol. 2 (2), 79-90 (2013).
- Gareau, J. R., Lima, C. D. The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. Nat Rev Mol Cell Biol. 11 (12), 861-871 (2010).
- Bernier-Villamor, V., Sampson, D. A., Matunis, M. J., Lima, C. D. Structural basis for E2-mediated SUMO conjugation revealed by a complex between ubiquitin-conjugating enzyme Ubc9 and RanGAP1. Cell. 108 (3), 345-356 (2002).
- Knipscheer, P., et al. Ubc9 sumoylation regulates SUMO target discrimination. Mol Cell. 31 (3), 371-382 (2008).
- Vethantham, V., Manley, J. L. In vitro sumoylation of recombinant proteins and subsequent purification for use in enzymatic assays. Cold Spring Harb Protoc. 2009 (1), 5121(2009).
- Lin, S. F., Robinson, D. R., Miller, G., Kung, H. J. Kaposi's sarcoma-associated herpesvirus encodes a bZIP protein with homology to BZLF1 of Epstein-Barr virus. J Virol. 73 (3), 1909-1917 (1999).
- Yang, W. S., Hsu, H. W., Campbell, M., Cheng, C. Y., Chang, P. C. K-bZIP Mediated SUMO-2/3 Specific Modification on the KSHV Genome Negatively Regulates Lytic Gene Expression and Viral Reactivation. PLoS Pathog. 11 (7), 1005051(2015).
- Connor, C. D., Metcalf, E., Wrighton, C. J., Harris, T. J., Saunders, J. R. RsrII--a novel restriction endonuclease with a heptanucleotide recognition site. Nucleic Acids Res. 12 (17), 6701-6708 (1984).
- Helling, R. B., Goodman, H. M., Boyer, H. W. Analysis of endonuclease R-EcoRI fragments of DNA from lambdoid bacteriophages and other viruses by agarose-gel electrophoresis. J Virol. 14 (5), 1235-1244 (1974).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Gingold, E. B. Bacterial transformation. Methods Mol Biol. 2, 237-240 (1985).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. J Virol. 86 (18), 9708-9720 (2012).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Ahmed, S., Holt, M., Riedel, D., Jahn, R. Small-scale isolation of synaptic vesicles from mammalian brain. Nat Protoc. 8 (5), 998-1009 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados