Method Article
In-vitro- Sumoylierung Assay SUMO E3 Ligase Tätigkeit zu studieren
In diesem Artikel
Zusammenfassung
Im Gegensatz zu Ubiquitin Ligases wurden einige E3 SUMO Ligases identifiziert. Dieses modifizierte in-vitro- Sumoylierung Protokoll ist in der Lage, neuartige SUMO E3 Ligases durch eine in-vitro- Rekonstitution Assay zu identifizieren.
Zusammenfassung
Kleine Ubiquitin-ähnliche Modifikator (SUMO) Modifikation ist eine wichtige Post-translationale Modifikation (PTM), die die Signaltransduktion in erster Linie durch Modulation der Protein-Protein-Wechselwirkungen vermittelt. Ähnlich wie Ubiquitin Modifikation Sumoylierung Regie eine sequenzielle Enzym Kaskade einschließlich E1-aktivierende Enzym (SAE1/SAE2), E2-Konjugation Enzym (Ubc9) und E3-Ligase (d.h., PIAS Familie, RanBP2 und Pc2). Jedoch anders als bei Ubiquitination, eine E3-Ligase ist unwesentlich für die Reaktion aber bieten Präzision und Wirksamkeit für SUMO Konjugation. Geändert von Sumoylierung Proteine können durch in-Vivo -Test über Immunopräzipitation mit Substrat-spezifische Antikörper und Immunoblotting mit SUMO-spezifische Antikörper identifiziert werden. Die Demonstration der Proteinaktivität SUMO E3 Ligase erfordert jedoch in Vitro Rekonstitution von Sumoylierung Assays mit gereinigtem Enzymen, Substrat und SUMO Proteine. Da in den in-vitro- Reaktionen in der Regel SAE1/SAE2 und Ubc9, allein für SUMO Konjugation ausreichen, ist Verbesserung der Sumoylierung durch eine vermeintliche E3-Ligase nicht immer leicht zu erkennen. Hier beschreiben wir eine modifizierte in Vitro Sumoylierung Protokoll, das konsequent SUMO-Modifikation mit einem in-vitro- rekonstituiert System identifiziert. Eine Schritt für Schritt Protokoll, katalytisch aktiven K-bZIP, eine virale SUMO-2/3-E3-Ligase zu reinigen wird ebenfalls dargestellt. Die Sumoylierung Aktivitäten der gereinigten K-bZIP werden p53, einem bekannten Ziel von SUMO angezeigt. Dieses Protokoll kann nicht nur für die neuartige SUMO E3 Ligases Aufklärung, sondern auch für die Preisgabe ihrer SUMO Paralog Spezifität eingesetzt werden.
Einleitung
SUMO-Umbau wurde zunächst als reversible Post-translationale Modifikation (PTM) identifiziert, die Protein Stabilität1regelt. Neben direkten Konjugation kann SUMO auch an ein Protein durch nicht-kovalente Wechselwirkungen durch SUMO Interaktion Motive (SIMs)2befestigt werden. Ähnlich wie die Bindung der Tyrosyl-Phosphorylierung von Molekülen beherbergen Src Homologie 2 (SH2) oder Phosphotyrosine Bindung (PTB) Domänen3,4, SUMO Modifikation bietet eine zusätzliche Interaktion Plattform für selektive Rekrutierung von SIM-haltigen Effektor Proteine5,6. Neben der Regulierung der Signaltransduktion Sumoylierung transcriptional Faktoren und Chromatin umgestalten Moleküle dienen gen Ausdruck7,8zu modulieren. Studien der genomweiten Sumoylierung Muster haben gezeigt, dass SUMO Änderung entweder positive9,10 oder negative10,11,12 Transkription zugeordnet ist Verordnung, teilweise aufgrund der Paralogue Besonderheit der Sumoylierung.
Es gibt drei große SUMO-Isoformen für Protein Konjugation Gegenwart in Säugerzellen; Dazu gehören SUMO-1, und die hochgradig homologen SUMO-2 und SUMO-3 (bezeichnet als SUMO-2/3)13. SUMO ist in der Regel an Lysin (K) Rückstände innerhalb der ψKxD/E Konsens-Motivs in das Zielprotein konjugiert. Die SIM-Karte im SUMO E3 Ligases ist verantwortlich für die Paralogue Spezifität14. Im Gegensatz zu Ubiquitination Wege mit Hunderten von E3 Ligases wurden nur wenige SUMO E3 Ligases identifiziert15. SUMO E3 Ligases zeichnen sich durch Eigenschaften, einschließlich ihrer Fähigkeit zur (i) Ubc9 binden, (Ii) binden SUMO glyko-über eine SIM-Domäne und (Iii) Förderung der SUMO-Übertragung von Ubc9 auf Substrat. E3-Ligase ist nicht zwingend erforderlich für SUMO Konjugation16, bietet aber Substrat und SUMO-Paralogue Spezifität. Da in der Regel nur ein kleiner Bruchteil des gesamten Substrat Proteins SUMO geändert ist, ist Erkennung der sumoyliert Proteine in Vivo immer eine Herausforderung. Deshalb sind genaue und reproduzierbare Tests erforderlich, um die genaue Funktion der SUMO Veränderung aufzuklären.
Die in-vitro- Sumoylierung Assay, die die Fähigkeit des gereinigten Ubc9, Sumoylierung Substrat Proteine katalysieren auswertet, ist ein gut angenommen Assay für SUMO Änderung17zu studieren. Jedoch SUMO E3-Ligase-Aktivität in den meisten Fällen nicht erkannt werden oder kann nur erkannt werden mit mutierten SUMO-Ligase mit hoher Aktivität der SUMO-E3-Ligase und geringe Substratspezifität durch das standard-Protokoll wegen der Anwesenheit von eine reichliche Menge von Ubc918 . Der Gesamterfolg des Assays hängt weitgehend die sorgfältige Titration der gereinigten Ubc9. Der in-vitro- Sumoylierung Test hier beschriebenen wird von einem standard Sumoylierung geändert (siehe Tabelle der Materialien)-Protokoll. Die Beobachtung der erhöhten globalen SUMO Änderung während Kaposi-Sarkom-assoziierte Herpesvirus (KSHV) Reaktivierung veranlasste uns, die virale SUMO E3-Ligase zu identifizieren, die für Up-Regulierung des Sumoylierung verantwortlich sein können. Nach einer kleinen Vorführung der interagierenden Proteine virale SUMO E3 Ligase K-bZIP wurde p53 als neuartiges Substrat identifiziert. In diesem Protokoll zeigen wir ausführlich in Insektenzellen, die Schritte in den Ausdruck und die Reinigung der Wildtyp K-bZIP, eine virale SUMO E3-Ligase mit SUMO-2/3 Spezifität beteiligt. Die Fähigkeit des gereinigten K-bZIP, Sumoylierung von p53, einem bekannten SUMO-Substrat zu verbessern wird in Anwesenheit von geringeren Mengen von E1 und E2 Enzyme ausgewertet. Mit dieser veränderten Sumoylierung Assay können Forscher zuverlässig definieren die Fähigkeiten der SUMO-E3-Ligase SUMOylate Roman oder bekannten Substrate, die primäre geht in die Untersuchung von Protein Sumoylierung. Darüber hinaus hilft diese Methode Roman SUMO E3 Ligases mit niedrigen Ligase Tätigkeit aber hohe Substratspezifität identifizieren.
Protokoll
1. Vorbereitung des Baculovirus Ausdruck Konstrukte
-
SUMO-E3-Ligase K-bZIP durch Klonen die cDNA von K-bZIP19 in eine duale Expressionsvektor Baculovirus zu reinigen (siehe Tabelle der Materialien) mit einem N-terminale Epitop-Tag. Wir hatten Erfolg mit einem Octapeptide Tag (angegeben im gesamten Protokoll als Vektor-Tag-K-bZIP).
Hinweis: Die DNA-Vorlage für K-bZIP Polymerase Kettenreaktion (PCR) Klonen ist cDNA reverse transkribiert von RNA isoliert von TREx F3H3-K-Rta BCBL-1 Zellen nach Doxycyclin Behandlung20.- Übertrage die Full-length K-bZIP cDNA19 in den dualen Ausdruck Vektor mit Cpoich Klonen Websites. CPO Das PCR-Produkt und 1 µg des dual Expressionsvektors bei 37 ° C für 3 h21zu verdauen.
- Trennen Sie und extrahieren Sie die verdaute DNA Elektrophorese im Anschluss an eine 0,8 % Agarose-Gel-22. Verbinden des Einsatzes (K-bZIP) und Vektor von T4 DNA-Ligase bei 16 ° C für 30 min nach Herstellerangaben (siehe Tabelle der Materialien).
- Fügen Sie 5 µL der Ligatur DNA zu 50 µL kompetente E. Coli Zellen "A" (siehe Tabelle of Materials) in einer 1,5 mL Tube. Für 30 min auf Eis gelegt. Dann inkubieren der 1,5 mL Tube im Wasserbad 42 ° C für 45 s und habe sofort das Rohr für 3 min auf Eis.
- Fügen Sie 600 µL S.O.C. Medium (0,5 % (w/V) Hefeextrakt, 2 % (w/V) Tryptone 10 mM NaCl, 2,5 mM KCl, 20 mM MgSO4und 4 % (w/V) Glukose) in die Röhre und Inkubation bei 37 ° C für 1 h. Dann Zentrifugieren Sie das Rohr bei Raumtemperatur für 3 min bei 600 X g.
- Entfernen Sie überstand, Aufschwemmen Pellet mit 60 µL Luria-Bertani Medium (LB; 1 % (w/V) Tryptone, 0,5 % (w/V) Hefe-Extrakt und 85 mM NaCl) und verteilt auf LB-Agar-Platten mit 100 µg/mL Ampicillin. Inkubieren Sie die Agarplatte bei 37 ° C für 16 h23,24.
- Wählen Sie 1 Kolonie in 5 mL LB-Medium mit 100 µg/mL Ampicillin und Kultur bei 37 ° C 16 h mit 250 u/min schütteln zu impfen. Extrahieren Sie das Plasmid, Vektor-Tag-K-bZIP, durch ein Plasmid Extraktion Kit gemäß den Anweisungen des Herstellers (siehe Tabelle der Materialien).
-
Generieren der rekombinanten Bacmid beherbergen die bZIP-K-Tag durch Umwandlung von Vektor-Tag-K-bZIP in kompetente E. Coli Zellen "B" (siehe Tabelle der Materialien).
- Mischen Sie 0,2 µg Vektor-Tag-K-bZIP Plasmid und 100 µL kompetente E. coli Zellen "B" in einem 1,5 mL-Tube und auf Eis für 30 min. Inkubieren Sie das Rohr in der 42 ° C Wasserbad für 45 s und habe sofort das Rohr für 3 min auf Eis.
- Fügen Sie 900 µL des S.O.C. Mediums in das Rohr und Inkubation bei 37 ° C für 4 h mit Rotation bei 50 Umdrehungen pro Minute. Als nächstes nehmen Sie 10 µL des Mediums, fügen Sie eine zusätzliche 50 µL S.O.C Medium auf LB-Agar-Platten mit 50 µg/mL Kanamycin, 7 µg/mL Gentamicin, 10 µg/mL Tetracyclin, 100 µg/mL Galaktosidase Substrat und 40 µg/mL Isopropyl β-D-1-Thiogalactopyranoside (IPTG verteilen ), und die Platten für 48 h bei 37 ° c inkubieren
- Eine weiße Kolonie in 5 mL LB-Medium mit 50 µg/mL Kanamycin, 7 µg/mL Gentamicin und 10 µg/mL Tetracyclin zu impfen. Inkubation bei 37 ° C mit 250 u/min schütteln für 16 h.
- Isolieren Sie der rekombinanten Bacmid DNA beherbergen die bZIP-K-Tag, quantifizieren Sie und erneut in einer Konzentration von 1 µg/µL25auszusetzen.
-
Rekombinante Baculoviren Tag K bZIP zu äußern, indem transfecting 1 µg der rekombinanten Bacmid DNA mit Tag-K-bZIP in Sf9 Zellen in einen Brunnen von einem 6-Well-Platte zu generieren.
- 2 x 106 Sf9 Samenzellen (siehe Tabelle der Materialien) in 2 mL Grace Insekt Medium geliefert mit 10 % fetalen bovine Serum (FBS) und 1 % Gentamicin in jedem der 6-well-Platte gut, dann Inkubation bei 27 ° C für 1 h.
- Pflegen eine Log-Phase-Kultur Sf9 Zellen Grace Insekt Medium mit 10 % FBS, 1 % Gentamicin und 1 % Waschmittel C geliefert (siehe Tabelle der Materialien) bei 27 ° C im orbital Aussetzung bei 140 u/min. Zählen von Zellen mit einem Hemocytometer und Trypan blau Färbung; Zellviabilität sollte 95 % überschreiten. Pflegen Sie Zellen mit einer Zellfläche-Verhältnis von 1:3 alle 2-3 Tage (minimale Dichte ~0.5 X 106 Zellen/mL).
- Fügen Sie 2 µL Bacmid DNA (1 µg/µL) und 98 µL reduziert Serum Media (siehe Tabelle der Materialien) in eine 2 mL-Tube. Fügen Sie 4 µL Transfection Reagens (siehe Tabelle der Materialien), Rohr und gut mischen. Bei Raumtemperatur für 15 min inkubieren.
- Diese Transfektion Mischung (104 µL) in jede Vertiefung, und Inkubation bei 27 ° C für 12-16 Uhr.
- Entfernen Sie erschöpfte Medien mit sanften Aspiration und ersetzen durch 2 mL frische Graces Insekt Medium mit 10 % FBS und 1 % Gentamicin geliefert. Die Sf9 Zellen halten locker an der Plattenoberfläche und nicht mit sanften Aspiration gestört werden.
-
Sammeln Sie rekombinante Baculovirus nach Transfektion und verstärken Sie Baculovirus um hohe Titer Bestände (P1) für weitere Experimente zu erhalten.
- 72 h nach dem Wechsel Medium, kratzen die Sf9 Zellen mit Hilfe einer sterilen Zelle Lifter, sammeln den Überstand aus jedem einzelnen Brunnen in einem 1,5 mL-Tube und Wirbel für 10 s.
- Zentrifugieren Sie bei 4 ° C für 3 min bei 150 X g. sammeln Überstand als P0 Baculovirus und Aliquot in 1,5 mL Röhrchen (1 mL Baculovirus in jedem Röhrchen). Shop bei-80 ° C.
- 1 x 107 Sf9 Samenzellen in 10 mL Grace Insekt Medium mit 10 % FBS und 1 % Gentamicin in einer 10 cm Petrischale geliefert und bei 27 ° C 1 h inkubieren.
- Zur Verstärkung des rekombinanten Baculoviren mit dem Ausdruck ihrer Tag-K-bZIP ausgesät Sf9 Zellen 0,5 mL P0 Baculovirus hinzu und Inkubation bei 27 ° C für 48 h.
- Sammeln Sie überstand als P1 Baculovirus in eine 15 mL-Tube und speichern bei 4 ° C für bis zu 1 Monat.
2. Reinigung des K-bZIP
- Erhalten Sie eine Protokoll Phase Kultur Sf9 Zellen Grace Insekt Medium mit 10 % FBS, 1 % Gentamicin und 1 % Waschmittel C bei 27 ° C im orbital Aussetzung bei 140 u/min geliefert, wie unter Punkt 1.3.2 beschrieben.
- Am Tag der Transduktion fügen Sie 1 mL Baculovirus-haltigen überstand (P1) in 250 mL Sf9 Zellen mit einer Dichte von 2 x 106 Zellen/mL.
- Inkubieren Sie Sf9 Zellen für 72 h bei 27 ° C auf einem Orbitalschüttler mit 140 u/min schütteln, dann sammeln Sie durch Zentrifugation bei 2.740 x g für 15 min bei 4 ° C.
- Entfernen Sie überstand und lyse der Zelle Pellets in 10 mL Lyse Puffer (20 mM HEPES pH 7,9, 0,5 M NaCl, 1 % Waschmittel ein (siehe Tabelle der Materialien), 2 % Glycerin, Protease-Inhibitor cocktail) und 50 u/min mit einer Federung Mixer bei 4 ° C für 30 min drehen.
- Zentrifuge Zelle lysate 15.000 x g für 15 min. Überstands zu sammeln.
- Tag-K-bZIP zu reinigen, brüten Sie Zelle Lysates (10 mL) mit 50 µL Antikörper Verschlagwortet mit magnetischen Beads in 14 mL Polypropylenröhrchen, und bei 50 u/min für 3 h bei 4 ° C mit einem Mixer Aufhängung drehen. Nach Einnahme von Protein, pellet-Perlen bei 800 x g Zentrifugation für 30 s bei 4 ° C. Entfernen die meisten des Überstands erneut aussetzen der Perlen in das Restvolumen von ca. 1 mL und übertragen auf einen 1,5 mL-Tube zum Waschen.
- Waschen Sie das aufgenommene Protein zu, indem man die Röhrchen mit den Perlen auf einem Magnetstativ, und entfernen Sie den Überstand zu, sobald die magnetische Beads vollständig auf die Seite des Rohres eingehalten haben.
-
Waschen Sie die Perlen mit Lyse Puffer viermal.
- Fügen Sie 1 mL Lyse Puffer in 1,5 mL Röhrchen und invertieren Sie die Rohre 10 Mal. Ein Magnetstativ setzen Sie der 1,5 mL Tube auf und entfernen Sie des Überstands wie unter 2.7 beschrieben.
- Wiederholen Sie den vorherigen Schritt noch 3 Mal.
-
Waschen Sie die Perlen mit Phosphat gepufferte Kochsalzlösung (PBS) einmal.
- Fügen Sie 1 mL PBS (137 mM NaCl, 2,7 mM KCl, 8 mM Na2HPO4, 1,5 mM KH2PO4) in 1,5 mL Röhrchen und invertieren Sie die Röhren 10-Mal.
- Ein Magnetstativ setzen Sie der 1,5 mL Tube auf und entfernen Sie dann den überstand.
- Eluieren Sie Tag K bZIP Protein aus der Antikörper-Verschlagwortet mit magnetischen Beads durch Zugabe von 100 µL 150 µg/mL Octapeptide mit PBS-Puffer in ein Rohr von 1,5 mL verdünnt. Drehen Sie das Rohr für 10 min bei 50 u/min durch Aussetzung Mischer bei Raumtemperatur. Dann setzen Sie den Schlauch wieder auf die Magnetstativ und sammeln des K-bZIP-haltigen Überstands in ein sauberes 1,5 mL-Tube.
- 1 bis 5 µL gereinigt Tag K bZIP Protein durch SDS-PAGE gefolgt von Coomassie blaue Färbung26zu analysieren. Laden Sie 2 µg, 1 µg und 0,5 µg Proben von Rinderserumalbumin (BSA) auf dem gleichen gel und verwenden es für die Quantifizierung mit einem Bildbearbeitungsprogramm wie ImageJ. Schätzen Sie K-bZIP Konzentration im Vergleich zu den BSA-Standards.
- Die gereinigten K-bZIP ist nun bereit, in jeder Sumoylierung Assay verwendet werden. Verdünnen Sie die K-bZIP mit PBS-Puffer, eine Endkonzentration von 100 ng/µL und Store in kleinen Aliquote bei-80 ° C.
(3) Sumoylierung Assay
- Fügen Sie 3 µL von 100 ng/µL des gereinigten Tag-K-bZIP in den master-Mix für jedes in-vitro- Sumoylierung Reaktion. Komponenten in master-Mix enthalten 1 µL Protein Puffer, 1 x Sumoylierung Puffer, 0,5 µL p53 Protein (0,5 mg/mL), 25 nM E1 aktiviert Enzym (Lager Konz.: 1 µM), 50 nM E2 Konjugation Enzym (Lager Konz.: 10 µM), und 250 nM von der SUMO-Peptiden (SUMO-1 , SUMO-2 oder SUMO-3; Lager Konz.: 5 µM) zu einem Endvolumen von 17 µL.
- Den Inhalt vorsichtig mischen und die Reaktion bei 30 ° C inkubieren, 3 h. Stop die Reaktion durch Zugabe von 20 µL 2 x SDS-PAGE laden Puffer (100 mM Tris-HCL pH 6,8, 4 % Sodium Dodecyl Sulfat, 0,2 % (w/V) Bromophenol Blue, 20 % (V/V) Glycerin und 200 mM β-Mercaptoethanol) und denaturieren Sie die Proben bei 95 ° C für 5 min.
-
Separate Proben auf SDS-PAGE, die getrennten Proteine auf PVDF Membran mithilfe eine halbtrockene elektrophoretische Transfer-Gerät übertragen und analysieren von Western-Blot mit Anti-p53-Antikörper.
- Richten Sie den Gel-Apparat wie in Schritt 2.11 und laden Sie 20 µL der Probe in Gel Brunnen.
- Führen Sie das 10 % Gel in einen konstanten Strom bei 80 V für ca. 120 min (bis die Farbstoff-Band das Ende des Gels erreicht). Schalten Sie nach Abschluss der Elektrophorese die Stromversorgung.
- Trennen Sie das Gel Apparat und Gel-Kassetten, nehmen Sie das Gel und schweben Sie das Gel in semi-dry Transfer Puffer (48 mM Tris-HCl, 39 mM Glycin, 20 % Methanol) für 5 min zu.
- Nehmen Sie einen anderen Container und Einweichen Sie die PVDF-Membran in Methanol für 1 min. Dann nehmen Sie PVDF aus Methanol und in semi-dry Transfer Puffer. Schütteln Sie sanft die Membran für 5 Minuten.
- Entfernen Sie die Sicherheitsabdeckung der halbtrockene elektrophoretischen Vorrichtung.
- Vornässen Filterpapier, und einen Gel-Sandwich auf der Unterseite Platin-Anode wie folgt vorbereiten: Filterpapier, PVDF-Membran, Gel und Filterpapier.
- Sichere Kathode Platte und Sicherheit Abdeckung, führen Sie dann Fleck bei 15 V Konstantstrom für 90 min. Drehen Sie die Stromversorgung trennen halbtrocken Apparat, und nehmen Sie die PVDF-Membran.
- Block-PVDF-Membran mit blockierenden Puffer (5 % fettfreie Milch in TBST Puffer (137 mM NaCl2, 20 mM Tris-HCl, 0,1 % Waschmittel B (siehe Tabelle der Materialien), pH 7.6)) bei Raumtemperatur 1 Stunde mit 30 u/min schütteln durch Orbitalschüttler.
- Hybridisieren Sie die PVDF-Membran mit Anti-p53 Antikörper verdünnt (1:1 000) in blocking-Puffer für 12-16 h bei 4 ° C mit 30 u/min schütteln mit einer Federung Mixer.
- Nehmen Sie die PVDF-Membran in einen Behälter und legte in TBST. Spülen Sie PVDF-Membran mit TBST zweimal.
- Einweichen Sie die PVDF-Membran in TBST für 30 min mit 45 u/min schütteln durch Orbitalschüttler.
- Hybridisieren Sie die PVDF-Membran mit Anti-Kaninchen-Antikörper konjugiert mit Meerrettich-Peroxidase (HRP) verdünnt (1:4 000) in blocking-Puffer für 1 h bei Raumtemperatur mit 30 u/min schütteln mit einer Aussetzung-Mixer.
- Waschen Sie die PVDF-Membran mit TBST 3 Mal wie in Schritt 3.4.10 beschrieben.
- Einweichen Sie die PVDF-Membran in TBST für 30 min mit 45 u/min schütteln mit einer Federung Mixer. Entfernen Sie TBST zu, und fügen Sie PBS Erhalt die PVDF-Membran bei 4 ° C für bis zu 12 h.
- Mischen Sie das verstärkte Chemilumineszenz Substrat (ECL Substrat) Reagenz 1 und 2 (1:1) (siehe Tabelle der Materialien). Entfernen Sie die PVDF-Membran aus der PBS, blot kurz mit einem Papiertuch oder leichte Wischer überschüssige Feuchtigkeit aufnehmen und sofort hinzufügen 400 µL der ECL-Reagenz an die Oberfläche jedes Membran für ca. 3-5 min. entfernen überschüssige ECL Reagenz durch kurz beflecken, aber erlauben nicht die m l, komplett trocken.
- Setzen Sie den Fleck mit einem Lumineszenz-imaging-System oder Autoradiographie Film27.
Ergebnisse
Nach den Angaben des Herstellers ist die Freigepäckmenge von E1 und E2 Enzym in der Sumoylierung Assay 50 nM und 500 nM, beziehungsweise. Die minimale Menge an E2 Konjugation Enzym Ubc9, das SUMOylate p53 wurde zuerst durch eine in-vitro- Sumoylierung Assay bestimmt. So niedrig wie ein Fünftel der Höhe der Ubc9 in der standard in Vitro Sumoylierung Assay-Protokoll verwendet effizient SUMOylate p53 konnte (Abb. 1A). Daher wurde die Hälfte des Betrags der E1-Enzym in Kombination mit einem Zehntel der Höhe des Ubc9 in das standard-Protokoll verwendet für ein weiterer in Vitro Sumoylierung Assay verwendet. Eine signifikante Reduktion der Sumoylierung Effizienz wurde im Vergleich zu standard-Protokoll (Abbildung 1B) beobachtet.
So verändert die Fähigkeit der SUMO-E3-Ligase K-bZIP, p53 von Sumoylierung bestimmt war, mit diesen zu erhöhen in Vitro Sumoylierung Bedingungen. Tagged K-bZIP von Sf9 Zellen (Abb. 2A) gereinigt wurde in einer Sumoylierung Reaktion enthalten geringere Mengen von E1 und E2 Ubc9 Enzyme eingesetzt. Unter diesen veränderten Bedingungen könnte K-bZIP effizient katalysieren die Sumoylierung von p53 (Abbildung 2B). Immunoblotting mit Anti-p53-Antikörper bestätigt die ähnliche Gesamtbeträge von p53 in jeder Reaktion und auch erkannte SUMO p53 geändert.

Abbildung 1 : Bestimmung der minimalen Höhe der SUMO-Enzyme, die in der in-vitro- Sumoylierung Assay verwendet. (A) p53, die Sumoylierung in einem in Vitro Sumoylierung Assay bewertet wurde, die eine Hälfte und ein Fünftel der Menge des Ubc9 Enzyms verwendet in standard-Protokoll enthalten. Nach 3 Stunden in Vitro Sumoylierung Reaktion, sumoyliert p53 Enzyme wurden untersucht, durch Western blot mit Anti-p53-Antikörper. (B) In Vitro Sumoylierung von p53 wurde weiter analysiert, indem die Hälfte der E1-Enzym in Kombination mit ein Zehntel der Menge an Ubc9 in das standard-Protokoll verwendet. Ein Western-Blot wurde durchgeführt, wie in (A) beschrieben. Bitte klicken Sie hier für eine größere Version dieser Figur.
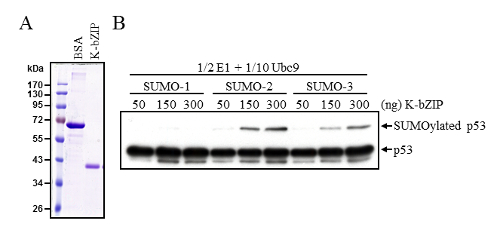
Abbildung 2 : Erweiterung des p53 Sumoylierung von SUMO E3 Ligase K-bZIP. (A) gereinigt Baculovirus ausgedrückt K-bZIP Protein, von SDS-PAGE folgte mit Coomassie blaue Färbung analysiert. 1 µg BSA und 10 µL gereinigt K-bZIP für vergleichende die Protein-Menge geladen wurden. (B) K-bZIP erhöht p53 Sumoylierung ausgewertet wurde, mithilfe einer in-vitro- Sumoylierung Reaktion mit halb so viel wie E1 Enzym und ein Zehntel der Menge des Ubc9 empfehlen in das standard-Protokoll. Sumoyliert p53 wurden durch Western-Blot untersucht, wie in Abbildung 1Abeschrieben. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Die in-vitro- Sumoylierung Protokoll hier beschriebenen wird routinemäßig eingesetzt, die Sumoylierung Status der identifizierten Ubc9 Substrate zu etablieren. Die größte Beschränkung des Standardprotokolls Sumoylierung Funktion des SUMO E3 Ligase zu studieren ist die Fülle der Aktivierung von SUMO E1 und E2 Konjugation Enzyme Ubc9, die die SUMO-Konjugation in in-vitro- Systemen zu maximieren. Angesichts dieser Herausforderung glauben wir, dass Titration von der Menge der SUMO E1 und E2 Enzyme auf das Niveau, dass man kaum, dass ihre Tätigkeit in Sumoylierung Weg ist nur verfügbar erkennen kann, SUMO E3 Ligase katalytische Tätigkeiten mit dieser in Vitro zu bestimmen Sumoylierung System. Nach dieser Hypothese wir erfolgreich die E3-Ligase Tätigkeit des K-bZIP aufgeklärt und es als eine neuartige SUMO E3-Ligase Spezifität gegenüber SUMO-2/3 zugeordnet.
Die entscheidende Schritt im Rahmen des Protokolls ist die Aufnahme geringer Mengen von E1 und E2. Diese geringeren Mengen verwenden, wie in Abbildung 2, Roman SUMO E3 Ligases mit niedrigen Ligase Tätigkeit möglicherweise mit der Erhaltung ihrer Spezifität gegenüber Substrat und SUMO Paralogues identifiziert werden. Eine große Einschränkung der Technik ist die Voraussetzung für eine qualitativ hochwertige Antikörper erkennen das SUMO-Substrat, da nur ein sehr kleiner Teil des Substrats SUMO geändert werden kann.
Obwohl viele Proteine das Potenzial Sumoylierung nach Überexpression in Vivozu erhöhen zeigen, muss definieren ein SUMO-E3-Ligase durch ein rekonstituierte in Vitro Sumoylierung-System unter Verwendung gereinigter E1, E2 und E3-Enzyme nachgewiesen werden. Im Gegensatz zu den Hunderten und vielleicht Tausende von E3 Ubiquitin Ligases identifiziert sind bis jetzt nur ein paar SUMO E3 Ligases gefunden worden. Dies kann zum Teil auf die Verwendung des traditionellen standard in Vitro Sumoylierung Assays, die SUMO Konjugation Effizienz maximiert und behindert somit die Sumoylierung Funktion des potenziellen SUMO E3 Ligases. Dieses Protokoll beschreibt einen einfachen und einheitlichen Assay Sumoylierung verstärkt durch ein SUMO-E3-Ligase Sonde. Das beschriebene Verfahren ist für die Identifizierung und Charakterisierung neuartiger SUMO E3 Ligases unerlässlich.
Offenlegungen
Die Autoren offenbaren keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde unterstützt durch Zuschüsse aus dem Ministerium für Wissenschaft und Technologie (MOST, 105-2320-B-010-007-MY3, PCC), von den National Health Research Institute (NMRI-EX105-10215BC, PCC), des Ministeriums für Wissenschaft und Technologie (die meisten 105-2314-B-400-019 zu HJK) und aus den National Health Research Institute (NMRI MG-105-SP-10, NMRI MG-106-SP-10 bis HJK). Diese Arbeit wurde auch teilweise mit National Yang-Ming University Manuskript Publikation PCC unterstützt. Die Geldgeber hatten keine Rolle beim Studiendesign, Datenerhebung und Analyse, Entscheidung, zu veröffentlichen oder Wiedergutmachung des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| pFastBac | Invitrogen | 10359-016 | dual expression baculovirus vector |
| pCR2.1-TOPO vector | Invitrogen | PCR product vector | |
| competent cell E. coli DH5α | Yeastern Biotech | FYE678-80VL | competent E. coli cells A |
| E. coli DH10Bac | Invitrogen | 10359-016 | competent E. coli cells B |
| FugeneHD | Roche | 04709705001 | transfection reagent |
| Opti-MEM | Gibco | 31985062 | reduced serum media |
| T4 Ligase | NEW England BioLabs | M0202S | |
| CpoI (RsrII) | Thermo Scientific | ER0741 | |
| Bluo-gal | Thermo Scientific | B1690 | galactosidase substrate |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Grace’s Insect Medium | Gibco | 11605094 | |
| Fetal Bovine Serum | Gibco | 10082147 | |
| Gentamicin | Thermo Fisher | 15750060 | |
| Ampicillin | Sigma-Aldrich | A9393-25G | |
| Kanamycin | Sigma-Aldrich | K0254-20ML | |
| gentamicin | Gibco | 15710-064 | |
| tetracyclin | Sigma-Aldrich | 87128-25G | |
| LB Broth | Merk | 1.10285.0500 | |
| HEPES | Sigma-Aldrich | H4034-100G | |
| NaCl | Sigma-Aldrich | S9888-5KG | |
| KCl | Merk | 1.04936.1000 | |
| Na2HPO4 | Sigma-Aldrich | S5136-500G | |
| KH2PO4 | J.T.Baker | 3246-01 | |
| sodium dodecyl sulfate | Merk | 1.13760.1000 | |
| β-mercaptoethanol | Bio-Rad | 161-0710 | |
| TRIS (Base) | J.T.Baker | 4109-06 | |
| Non-fat milk | Fonterra | ||
| glycerol | J.T.Baker | 2136-01 | |
| Triton X-100 | Amresco | 0694-1L | detergent A |
| Tween 20 | Amresco | 0777-500ML | detergent B |
| Poloxamer 188 solution | Sigma-Aldrich | P5556-100ML | detergent C |
| Protease Inhibitor Cocktail Tablet | Roche | 04 693 132 001 | |
| 3x Flag peptide | Sigma | F4799 | |
| anti-FLAG m2 Magnetic beads | Sigma-Aldrich | M8823 | antibody-tagged magnetic beads |
| SUMOlink SUMO-1 Kit | Active Motif | 40120 | standard SUMOylation protocol |
| SUMOlink SUMO-2/3 Kit | Active Motif | 40220 | standard SUMOylation protocol |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAGEN Plasmid Mini Kit | QIAGEN | 12123 | plasmid extraction kit |
| Polypropylene tubes | Falcon | 352059 | |
| Petri Dish | Falcon | 351029 | |
| Cell lifter | Corning | CNG3008 | |
| Loading tip | Sorenson BioScience | 28480 | |
| PVDF | PerkinElmer | NEF1002 | |
| Blotting filter paper | Bio-Rad | 1703932 | |
| Mini slab gel apparatus (Bio-Rad Mini Protean II Cell) | Bio-Rad | 1658001 EDU | |
| Trans-Blot SD Semi-Dry Electrophoretic Transfer Cell | Bio-Rad | 1703940 | |
| Pierce ECL Western Blotting | Thermo | 32106 | ECL reagent |
| suspension mixer | Digisystem laboratory instruments Inc. | SM-3000 | |
| orbital shaker | Kansin instruments Co. | OS701 | |
| ImageQuant LAS 4000 biomolecular imager | GE Healthcare | 28955810 | |
| Sf9 | Thermo Scientific | B82501 | |
| anti-p53 antibody | Cell Signaling | #9282 | |
| anti-rabbit antibody | GE Healthcare | NA934-1ML |
Referenzen
- Bies, J., Markus, J., Wolff, L. Covalent attachment of the SUMO-1 protein to the negative regulatory domain of the c-Myb transcription factor modifies its stability and transactivation capacity. J Biol Chem. 277 (11), 8999-9009 (2002).
- Lin, D. Y., et al. Role of SUMO-interacting motif in Daxx SUMO modification, subnuclear localization, and repression of sumoylated transcription factors. Mol Cell. 24 (3), 341-354 (2006).
- Welsh, M., Jamalpour, M., Zang, G., Akerblom, B. The role of the Src Homology-2 domain containing protein B (SHB) in beta cells. J Mol Endocrinol. 56 (1), 21-31 (2016).
- Huang, W. Q., et al. Structure, function, and pathogenesis of SHP2 in developmental disorders and tumorigenesis. Curr Cancer Drug Targets. 14 (6), 567-588 (2014).
- Perry, J. J., Tainer, J. A., Boddy, M. N. A SIM-ultaneous role for SUMO and ubiquitin. Trends Biochem Sci. 33 (5), 201-208 (2008).
- Sun, H., Leverson, J. D., Hunter, T. Conserved function of RNF4 family proteins in eukaryotes: targeting a ubiquitin ligase to SUMOylated proteins. EMBO J. 26 (18), 4102-4112 (2007).
- Ouyang, J., Shi, Y., Valin, A., Xuan, Y., Gill, G. Direct binding of CoREST1 to SUMO-2/3 contributes to gene-specific repression by the LSD1/CoREST1/HDAC complex. Mol Cell. 34 (2), 145-154 (2009).
- Lyst, M. J., Stancheva, I. A role for SUMO modification in transcriptional repression and activation. Biochem Soc Trans. 35, Pt 6 1389-1392 (2007).
- Liu, H. W., et al. Chromatin modification by SUMO-1 stimulates the promoters of translation machinery genes. Nucleic Acids Res. 40 (20), 10172-10186 (2012).
- Rosonina, E., Duncan, S. M., Manley, J. L. SUMO functions in constitutive transcription and during activation of inducible genes in yeast. Genes Dev. 24 (12), 1242-1252 (2010).
- Chang, P. C., et al. The chromatin modification by SUMO-2/3 but not SUMO-1 prevents the epigenetic activation of key immune-related genes during Kaposi's sarcoma associated herpesvirus reactivation. BMC Genomics. 14, 824(2013).
- Neyret-Kahn, H., et al. Sumoylation at chromatin governs coordinated repression of a transcriptional program essential for cell growth and proliferation. Genome Res. 23 (10), 1563-1579 (2013).
- Tatham, M. H., et al. Polymeric chains of SUMO-2 and SUMO-3 are conjugated to protein substrates by SAE1/SAE2 and Ubc9. J Biol Chem. 276 (38), 35368-35374 (2001).
- Mattoscio, D., Segre, C. V., Chiocca, S. Viral manipulation of cellular protein conjugation pathways: The SUMO lesson. World J Virol. 2 (2), 79-90 (2013).
- Gareau, J. R., Lima, C. D. The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. Nat Rev Mol Cell Biol. 11 (12), 861-871 (2010).
- Bernier-Villamor, V., Sampson, D. A., Matunis, M. J., Lima, C. D. Structural basis for E2-mediated SUMO conjugation revealed by a complex between ubiquitin-conjugating enzyme Ubc9 and RanGAP1. Cell. 108 (3), 345-356 (2002).
- Knipscheer, P., et al. Ubc9 sumoylation regulates SUMO target discrimination. Mol Cell. 31 (3), 371-382 (2008).
- Vethantham, V., Manley, J. L. In vitro sumoylation of recombinant proteins and subsequent purification for use in enzymatic assays. Cold Spring Harb Protoc. 2009 (1), 5121(2009).
- Lin, S. F., Robinson, D. R., Miller, G., Kung, H. J. Kaposi's sarcoma-associated herpesvirus encodes a bZIP protein with homology to BZLF1 of Epstein-Barr virus. J Virol. 73 (3), 1909-1917 (1999).
- Yang, W. S., Hsu, H. W., Campbell, M., Cheng, C. Y., Chang, P. C. K-bZIP Mediated SUMO-2/3 Specific Modification on the KSHV Genome Negatively Regulates Lytic Gene Expression and Viral Reactivation. PLoS Pathog. 11 (7), 1005051(2015).
- Connor, C. D., Metcalf, E., Wrighton, C. J., Harris, T. J., Saunders, J. R. RsrII--a novel restriction endonuclease with a heptanucleotide recognition site. Nucleic Acids Res. 12 (17), 6701-6708 (1984).
- Helling, R. B., Goodman, H. M., Boyer, H. W. Analysis of endonuclease R-EcoRI fragments of DNA from lambdoid bacteriophages and other viruses by agarose-gel electrophoresis. J Virol. 14 (5), 1235-1244 (1974).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Gingold, E. B. Bacterial transformation. Methods Mol Biol. 2, 237-240 (1985).
- Brulois, K. F., et al. Construction and manipulation of a new Kaposi's sarcoma-associated herpesvirus bacterial artificial chromosome clone. J Virol. 86 (18), 9708-9720 (2012).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Ahmed, S., Holt, M., Riedel, D., Jahn, R. Small-scale isolation of synaptic vesicles from mammalian brain. Nat Protoc. 8 (5), 998-1009 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten