Method Article
Isolamento suave de núcleos do tecido cerebral de Killifish turquesa africano adulto, um modelo naturalmente de curta duração para a pesquisa do envelhecimento
Neste Artigo
Resumo
Aqui apresentamos um protocolo para isolar núcleos dos cérebros do modelo de vertebrados de vida curta Nothobranchius furzeri para aplicações a jusante, como sequenciamento de RNA de núcleo único ou ensaio de núcleo único para cromatina acessível à transposase com sequenciamento (ATAC-seq).
Resumo
Estudar o envelhecimento cerebral na resolução de uma única célula em sistemas vertebrados continua a ser um desafio devido ao custo, tempo e restrições técnicas. Aqui, demonstramos um protocolo para gerar bibliotecas de sequenciamento de RNA de núcleo único (snRNA-seq) a partir dos cérebros do peixe-turquesa africano Nothobranchius furzeri, de vertebrados de vida naturalmente curta. O killifish turquesa africano tem uma vida útil de 4-6 meses e pode ser alojado de forma rentável, reduzindo assim as barreiras de custo e tempo para estudar o envelhecimento cerebral dos vertebrados. No entanto, são necessários protocolos personalizados para isolar núcleos de qualidade suficiente para experimentos unicelulares a jusante do cérebro de peixes jovens e idosos. Aqui, demonstramos um protocolo empiricamente otimizado para o isolamento de núcleos de alta qualidade do cérebro de killifish turquesa africano adulto, um passo crítico na geração de bibliotecas ômicas de núcleos únicos de alta qualidade. Além disso, mostramos que as etapas para reduzir o RNA de fundo contaminante são importantes para distinguir claramente os tipos de células. Em resumo, este protocolo demonstra a viabilidade de estudar o envelhecimento cerebral em organismos modelo de vertebrados não tradicionais.
Introdução
Compreender os mecanismos do envelhecimento cerebral dos vertebrados é fundamental para abordar doenças neurodegenerativas relacionadas à idade, como Alzheimer e demência1. O peixe-turquesa africano (Nothobranchus furzeri) é o vertebrado de vida mais curta que pode ser criado em cativeiro e, devido à sua curta vida útil e comprometimento cognitivo associado à idade, é um excelente modelo de envelhecimento cerebral 2,3,4,5. Recentemente, o advento de tecnologias "ômicas" de célula única, como RNA-seq de núcleos únicos (snRNA-seq) e ensaio de núcleos únicos para cromatina acessível à transposase com sequenciamento (snATAC-seq), permitiram aos pesquisadores interrogar o envelhecimento cerebral em uma resolução sem precedentes 6,7,8. Esses métodos dependem do isolamento de núcleos, uma vez que a recuperação de células cerebrais, como neurônios, é muitas vezes muito desafiadora para isolar 6,7,8,9,10. No entanto, a maioria dos protocolos de isolamento de núcleos publicados é otimizada para organismos modelo de mamíferos 11,12,13,14,15. Assim, como existe atualmente uma necessidade não atendida de isolar núcleos cerebrais no killifish como um novo organismo modelo em ascensão no campo da pesquisa sobre envelhecimento2, o objetivo deste protocolo é estabelecer um método para isolar núcleos de alta qualidade do tecido congelado do killifish cerebral.
Aqui, um fluxo de trabalho simplificado e robusto é estabelecido que usa materiais comumente disponíveis para isolar núcleos de alta qualidade de cérebros de killifish. Este protocolo foi modificado a partir de um protocolo de Genômica 10x para cérebros de camundongos para acomodar os cérebros com menor teor de mielina do killifish turquesa africano, a fragilidade do tecido congelado e a necessidade de reduzir o conteúdo de detritos ambientais para aplicações relacionadas ao sequenciamento16. De fato, protocolos previamente otimizados para tecido cerebral de mamíferos 17,18 levam a uma má qualidade dos núcleos (ou seja, sobrelise) e/ou alto teor de detritos quando usados em cérebros congelados de killifish, tornando-os inadequados para uso com snRNA-seq de acordo com as recomendações para RNA-seq de núcleos únicos usando microfluídica (Figura Suplementar 1).
Além do isolamento dos núcleos, demonstramos como avaliar a qualidade e o rendimento dos núcleos por microscopia e citometria de fluxo. Este artigo fornece exemplos de resultados ótimos e subótimos e discute a solução de problemas. Este protocolo foi projetado e otimizado para cérebros de killifish congelados, mas também pode ser usado sem grandes modificações em amostras de killifish recém-dissecadas. Os núcleos cerebrais de Killifish isolados usando este método foram otimizados para uso em RNA-seq de núcleo único (snRNA-seq) como uma aplicação a jusante, mas também devem ser passíveis de uso em snATAC-seq e ATAC-seq a granel.
Protocolo
O cuidado com os animais e a experimentação animal foram realizados de acordo com a IACUC da Universidade do Sul da Califórnia sob os protocolos aprovados #21215. Para qualquer trabalho que utilize este protocolo, é necessário obter a aprovação da IACUC da instituição antes de iniciar qualquer trabalho de investigação em animais vertebrados.
NOTA: Uma execução completa do protocolo a partir do tecido cerebral congelado por flash (a partir da etapa 3) deve levar ~ 2,5 h para seis amostras (Figura 1).
1. Dissecar cérebros de killifish
- Eutanasiar humanamente killifish usando uma solução de 1,5 g/L de metanossulfonato/tricadina (MS-222) na água do sistema.
- Use tesouras afiadas para decapitar rapidamente o killifish depois que todo o movimento do corpo e das brânquias tiver cessado.
- Dissecar a cabeça decapitada em uma placa de Petri limpa, conforme descrito anteriormente19.
- Coloque o prato sob um microscópio de dissecação (usando uma faixa de ampliação de 8x-35x). Vire a cabeça de tal forma que os raios branquiostegal estejam em foco
- Usando uma tesoura, corte a dentadura de tal forma que os raios branquiostegal e o crânio sejam revelados.
- Usando fórceps, segure os raios branquiostegal em ambos os lados do crânio. Use outro par de pinças para remover quaisquer fragmentos de tecido remanescentes do crânio. O quiasma óptico em forma de V que conecta o cérebro e os olhos deve se tornar visível.
- Corte o quiasma óptico usando uma tesoura para separar os dois olhos do cérebro. Use fórceps para liberar suavemente o crânio.
- Inverta o crânio e use fórceps para raspar o músculo do lado dorsal do crânio.
- Use um par de pinças para manter o crânio no lugar e outro par de pinças para remover suavemente os ossos do crânio, revelando o cérebro.
- O cérebro parece branco e macio. Uma vez visível, não é necessário remover todos os ossos cranianos. Remova o cérebro com pinça limpa, levantando e raspando suavemente.
- Flash congelar o cérebro, colocando-o em um tubo de microcentrífuga armazenado em gelo seco.
NOTA: O tecido cerebral congelado pode ser armazenado a -80 °C até que todas as amostras a serem processadas juntas tenham sido coletadas para limitar os efeitos em lote durante o processamento para geração de biblioteca. Embora as amostras armazenadas a -80 °C não possam ser armazenadas indefinidamente, este protocolo foi realizado com sucesso em cérebros de killifish armazenados por até 12 meses sem queda notável na qualidade.
2. Prepare tampões frescos
- Preparar tampão de lavagem de núcleos (albumina sérica bovina a 2% [BSA] em 1x PBS, solução salina tamponada com fosfato pH 7,4 e inibidor de RNase 0,2 U/μL) e armazenar no gelo. Prepare 250 mL de tampão de lavagem de núcleos para até oito amostras. Para 250 mL, use 50 mL de solução de estoque de BSA a 10%, 25 mL de uma solução-estoque de PBS 10x, 1,25 mL do estoque de inibidor de RNase de 40 U/μL e, em seguida, traga o volume para 250 mL com ddH2O livre de RNAse.
- Preparar tampão de lise de núcleos (10 mM Tris-HCl pH 7,4, 10 mM NaCl, 3 mM MgCl 2, 0,01875% v/v NP-40 e0,2 U/μL RNase inibidor) e armazenar no gelo. Prepare 10 mL de tampão de lise de núcleos para até oito amostras. Para 10 mL, use 100 μL de solução estoque de 1 M Tris-HCl pH 7,4, 20 μL de solução estoque de NaCl de 5 M, 30 μL de solução de estoque de 1 M de MgCl 2, 18,75 μL de uma solução-estoque de NP-40 a 10%, 50 μL do estoque de inibidor de RNase de 40 U/μL e, em seguida, traga o volume para 10 mL com ddH2 O livre de RNAse.
3. Isolar núcleos
- Se estiver usando amostras congeladas, descongele-as no gelo por 10 min. Se estiver usando cérebros recém-dissecados, prossiga diretamente para a etapa 3.2
NOTA: Embora este protocolo possa funcionar em cérebros únicos, amostras menores de peixes jovens (5-6 semanas de idade) terão um rendimento mais pobre. Rendimentos suficientes sempre podem ser obtidos pelo processamento de dois cérebros como uma amostra, independentemente da idade e do sexo do peixe (Tabela 1). Este protocolo isolou com sucesso núcleos de alta qualidade de peixes com idades entre 5 e 26 semanas, incluindo animais das cepas GRZ e ZMZ1001. Não há problemas esperados na aplicação deste protocolo a outras cepas. - Saltar o cérebro em 1 mL de tampão de lise de núcleos gelados em um homogeneizador Dounce de 2 mL. Mantenha o homogeneizador no gelo e evite gerar bolhas enquanto lisa a amostra. Salte a uma taxa aproximada de 0,5 s/downstroke e 0,5 s/upstroke com uma torção de 180° por ciclo. Salte usando o pilão solto por 15 golpes ou até que a mistura de tampão tecido/lise pareça homogênea se forem necessários traços adicionais.
NOTA: Os homogeneizadores de rejeição devem ser lavados, enxaguados com água destilada e esterilizados com ciclos de esterilização em autoclave seca padrão entre os usos.- Salte usando o pilão apertado por 30 braçadas com pequenas pausas, 30 s a 1 min, a cada 10 tacadas. Salte a uma taxa aproximada de 0,5 s/downstroke e 0,5 s/upstroke com uma torção de 180° por ciclo. Deixe a amostra descansar sobre o gelo no homogeneizador Dounce por 2 min.
NOTA: Este tempo de incubação garante lise adequada, mas deve ser encurtado se a sobrelise nuclear for observada.
- Salte usando o pilão apertado por 30 braçadas com pequenas pausas, 30 s a 1 min, a cada 10 tacadas. Salte a uma taxa aproximada de 0,5 s/downstroke e 0,5 s/upstroke com uma torção de 180° por ciclo. Deixe a amostra descansar sobre o gelo no homogeneizador Dounce por 2 min.
- Coe a amostra através de um filtro de células de 70 μm em um tubo cônico de 15 mL pré-revestido em BSA a 5%.
- Adicionar 4 ml de tampão de lavagem de núcleos à amostra pipetando o tampão de lavagem sobre o filtro de células de 70 μm da etapa 3.3 e misturar suavemente por inversão cinco vezes.
NOTA: Isso permite a recuperação de núcleos que podem estar presos no filtro e melhora o rendimento. - Centrífuga utilizando um rotor de caçamba oscilante a 500 x g, 4 °C, durante 10 min. Descarte o sobrenadante despejando suavemente em um recipiente de resíduos líquidos. Devolver a amostra para uma posição vertical e remover o amortecedor de lavagem restante utilizando uma pipeta P1000.
NOTA: É provável que o pellet dos núcleos seja visível quando se parte de dois cérebros em uma amostra. No entanto, ao processar cérebros únicos de animais jovens (5-6 semanas de idade), um rendimento menor é esperado, e os pellets nem sempre podem ser visíveis. Neste caso, o sobrenadante deve ser cuidadosamente removido enquanto assume a posição do pellet na parte inferior do tubo. - Ressussuscite imediatamente os núcleos em 1 mL de tampão de lavagem de núcleos com uma ponta de pipeta P1000 de furo largo.
- Levar a amostra a 5 mL adicionando 4 mL de tampão de lavagem de núcleos e misturar suavemente por inversão cinco vezes. Centrifugar utilizando um rotor de caçamba oscilante a 500 x g, 4 °C, durante 10 min, e eliminar o sobrenadante.
- Ressuspeite os núcleos em 1 mL de tampão de lavagem de núcleos com uma ponta de pipeta de furo largo e transfira para um tubo de citometria de fluxo (FACS).
- Adicionar 300 μL de solução de remoção de detritos (Tabela de Materiais) e misturar bem por pipetagem com uma ponta larga do furo até que a mistura fique homogénea.
- Sobrepor suavemente 1 ml de tampão de lavagem de núcleos sobre a solução de núcleos preparada no passo 3.9 utilizando uma pipeta P1000 e segurando o tubo num ligeiro ângulo (Figura 2 Suplementar). A fração da solução de remoção de detritos e a fração tampão de lavagem dos núcleos devem formar uma interface clara se colocadas em camadas corretamente.
- Centrífuga em rotor de caçamba oscilante a 3000 x g, 4 °C, durante 10 min. Descarte completamente as duas fases superiores usando uma pipeta P1000.
NOTA: O tubo FACS transparente permitirá a visualização das três fases distintas após a centrifugação (superior, interfase, inferior). Para facilitar a remoção máxima de detritos, recomenda-se ser mais agressivo do que conservador na remoção das duas fases superiores (ou seja, remover uma pequena quantidade da fase inferior é preferível a transportar qualquer uma das fases superiores; Figura 2 suplementar). Além disso, a camada interfásica pode ser muito fina para visualizar. Nesse caso, remova a camada superior (aparente), que contém a camada interfásica. A camada inferior que é retida deve conter ~ 1 mL de volume total. - Transfira a camada inferior para um tubo cônico de 15 mL pré-revestido com BSA a 5%.
- No tubo cônico, leve o volume para 15 mL com tampão de lavagem de núcleos e inverta três vezes para misturar.
- Centrífuga em rotor de caçamba oscilante a 1000 x g, 4 °C, durante 10 min. Remova o sobrenadante com cuidado.
- Ressuspeite imediatamente em 150 μL de tampão de lavagem de núcleos usando uma ponta de pipeta de furo largo.
- Mude para uma ponta de pipeta de furo padrão ajustada para ~300 μL. Absorva toda a amostra e, em seguida, prenda um filtro de 40 μm na ponta da ponta da pipeta, tomando cuidado para não expelir a amostra.
NOTA: Filtros na ponta são necessários para obter volumes menores, que são necessários para manter a concentração suficiente de núcleos para aplicações a jusante. Por exemplo, para snRNA-seq usando microfluídica, recomenda-se uma concentração mínima de >300 núcleos/μL, embora a faixa ideal seja de 700-1200 núcleos/μL18. Concentrações mais altas de núcleos também podem ser facilmente diluídas, se necessário. - Filtrar a amostra expelindo-a com força através do filtro para um tubo de microcentrífuga de 1,5 ml de ligação ao ADN. O volume final de eluição deve ser de 120-150 μL.
NOTA: Algumas espumantes podem resultar da filtração. Isso não parece afetar a qualidade dos núcleos.
4. Avaliar a qualidade dos núcleos por microscopia
- Num tubo de microcentrífuga separado, misturar 10 μL da amostra filtrada com 10 μL de solução azul de tripano a 0,4% por pipetagem suave. As amostras não requerem tempo de incubação prolongado e estão prontas para visualização imediatamente após a mistura com a solução azul de tripano.
- Deposite 10 μL dos núcleos corados em uma câmara de uma lâmina de câmara de contagem.
NOTA: Alternativamente, deposite 10 μL da amostra de núcleos corados em uma lâmina de hemocitômetro ou microscopia e cubra com uma folha de cobertura. - Avaliar a qualidade dos núcleos por microscopia de luz.
NOTA: Ampliações mais baixas só devem ser usadas para ampliar os núcleos, uma vez que os defeitos dos núcleos só serão visualmente aparentes em ampliações mais altas (ou seja, 60x ou mais). Esta etapa pode ser feita rapidamente para avaliar o sucesso da preparação dos núcleos e não requer análise secundária além da inspeção visual. Núcleos intactos e de alta qualidade terão uma borda20 bem definida (Figura 2A). Núcleos de baixa qualidade terão rompido membranas nucleares, coloração azul de tripano irregular perto da membrana nuclear indicativa de potencial vazamento de ácido nucleico e/ou evidência de blebbing20 (Figura 2B). Espera-se que os núcleos saudáveis tenham 10-15 μm de diâmetro.
5. Quantificar núcleos singletes, detritos e proporção múltipla por citometria de fluxo
NOTA: A terminologia específica e a interface do software do citômetro de fluxo podem diferir com base na marca da máquina, mas essas etapas devem ser facilmente adaptadas a outros sistemas, se necessário.
- Em um tubo FACS, ressuscite 10 μL da amostra de núcleos filtrados em 90 μL do tampão recomendado para citometria de fluxo para diluição de 1:10. Para tanto, utilizou-se neste estudo PBS esterilizada filtrada a 0,22 μm, pH 7,2, com EDTA de 2 mM e BSA a 0,5%.
NOTA: Embora essa diluição funcione para a maioria dos preparadores de núcleos, uma diluição mais baixa (por exemplo, 1:5) pode ser necessária para coletar amostras de núcleos suficientes para avaliação de qualidade no caso de uma amostra de menor rendimento (ou seja, <30 núcleos/μL na amostra diluída). - Coloração das amostras com iodeto de propídio (IP) na concentração final de 1 μg/mL. Nenhum tempo de incubação é necessário, e os núcleos podem ser analisados imediatamente.
NOTA: Alternativamente, corar as amostras com uma concentração final de 0,1 μg/mL de DAPI. Note que o DAPI deve ser lido em um canal de fluorescência diferente do PI (canal Vioblue-A V1-A; excitação a 405 nm, emissão a 450/50 nm). - Analise as amostras em um citômetro de fluxo da seguinte forma:
- Configure um espaço de trabalho:
- Tenha o citômetro de fluxo no modo de Aquisição. Clique em Arquivo e selecione Novo Espaço de Trabalho no menu suspenso. Na guia Experimento no painel esquerdo, insira o Nome do Projeto e a ID da Amostra em seus respectivos campos.
- Na barra de ferramentas superior esquerda, clique no botão Nova janela de análise (ícone de gráfico de dispersão). Clique na caixa com três gráficos (Plot 4t) no menu pop-up. Três enredos aparecerão na tela. Clique no botão Modo de Análise na barra de ferramentas superior esquerda (ícone A) para que fique cinza e não laranja.
- Configure gráficos de análise:
- Altere os eixos dos três gráficos da seguinte forma, ordenados como eixo X e eixo Y, respectivamente:
Gráfico 1: Eixo X: FSC-A, eixo Y: SSC-A
Gráfico 2: Eixo X: HDR-T, eixo Y: SSC-A
Parcela 3: Eixo X: PerCP-Vio700-A B3-A, Eixo Y: SSC-A - Altere os eixos clicando no rótulo do eixo e selecionando o canal desejado no menu suspenso.
- Defina os gráficos para serem codificados por cores de densidade clicando no ícone "i" cinza quadrado que aparece ao lado do canto superior direito de cada gráfico. Uma janela Propriedades é aberta com um painel de botões à esquerda. Na janela Propriedades, clique no ícone Gráfico de dispersão (segundo na parte superior à esquerda do painel). Clique em OK.
NOTA: O canal de fluorescência PerCP-Vio700-A B3-A corresponde a uma excitação a 488 nm e a uma emissão na faixa de 650-730 nm.
- Altere os eixos dos três gráficos da seguinte forma, ordenados como eixo X e eixo Y, respectivamente:
- Configure as configurações do instrumento:
- Clique na guia Canais no painel esquerdo. Uma lista de todos os canais aparecerá com três caixas à direita. Defina B3, FSC-A e SSC-A como Hlog clicando na seta para baixo em suas respectivas primeiras caixas à direita e selecionando Hlog no menu suspenso. Defina a tensão do SSC para 640 V, FSC para 300 V e B3 para 480 V digitando esses números em suas respectivas caixas do meio à direita e pressionando a tecla Enter . Observe que a terceira caixa à direita é uma alternância que pode ser usada alternadamente para digitação.
- No cabeçalho Trigger , defina o gatilho FSC como 4 e o gatilho SSC e B3 como 0 clicando na seta para baixo, selecionando-os no menu suspenso à esquerda, digitando o número na caixa à direita e pressionando a tecla Enter . Observe que há uma caixa de alternância à direita que pode ser usada alternativamente à digitação.
NOTA: As tensões PMT precisam ser otimizadas para cada máquina de citometria de fluxo específica, mas o gatilho para núcleos deve ser menor do que o que é normalmente usado para células. Além disso, a escala do eixo deve ser definida como h-log.
- Digite 100 μL na caixa de texto Volume de amostra e 50 μL na caixa de texto Volume de captação no lado esquerdo da janela do software de fluxo. Verifique se o parâmetro Mix Sample está definido como Mix Gentle para evitar a lise de núcleos nesta etapa. Pressione o triângulo dentro de um círculo (ícone Reproduzir) no canto inferior direito para iniciar o fluxo.
- Verifique a qualidade do fluxo:
- Use a área de dispersão lateral (SSC-A) versus HDR-T para verificar se há bolhas de ar ou aglomerados na amostra. Certifique-se de que este gráfico é um continuum suave de pontos. Bolhas de ar ou aglomerados resultarão em regiões em branco no gráfico SSC-A versus HDR-T.
- Verifique o gráfico de dispersão lateral (SSC-A) versus dispersão direta (FSC-A) para verificar se as tensões usadas estão corretas para a amostra.
NOTA: Ao contrário das células, os núcleos não se resolverão totalmente a partir de detritos em um gráfico de dispersão lateral versus dispersão para frente. No entanto, deve haver um aglomerado de eventos de alta densidade em direção ao meio do gráfico correspondente a núcleos (de cor mais quente) com uma densidade mais baixa de eventos (de cor mais fria) no fundo correspondente a detritos.
- Analise a contagem e a qualidade dos núcleos:
- Clique duas vezes no gráfico SSC-A vs. PerCp-Vio-700A para obter uma exibição ampliada. Clique no botão na barra de ferramentas superior esquerda com linhas perpendiculares (Quadrante) para selecionar uma porta de quadrante. Em seguida, clique em qualquer lugar no gráfico SSC-A vs. PerCp-Vio-700A e os quadrantes aparecerão dentro do gráfico.
- Clique em qualquer lugar nas linhas divisórias do quadrante e deslize o cursor para modificar o posicionamento do quadrante. Coloque os quadrantes de tal forma que o quadrante superior esquerdo contenha a população da extrema esquerda (detritos) e o quadrante superior direito contenha vários aglomerados em forma de lágrima (núcleos) (veja a Figura 3 para configuração do portão e exemplos de resultados de fluxo para resultados típicos com preparativos incluindo ou não uma etapa de remoção de detritos).
- Contagem de detritos versus núcleos:
- Clique no ícone "i" cinza quadrado no canto superior direito do gráfico para abrir a janela Propriedades . Clique na guia Funções da Região; listas de regiões e funções de plotagem serão exibidas.
- Na lista Regiões , verifique se há uma marca de seleção verde ao lado do item superior, indicando que todo o gráfico está selecionado. Na lista Funções , clique em Contar/μL para produzir uma marca de seleção verde. Clique em OK e uma métrica de contagens/μL aparecerá em cada quadrante.
- Para exibir contagens e porcentagens brutas, selecione Contagem e %-T na guia Funções de Região , se desejar. As contagens/μL nos quadrantes superior esquerdo e direito correspondem ao rendimento de detritos e núcleos, respectivamente.
- Singlet conta:
- O aglomerado em forma de lágrima mais à esquerda na porção inferior do quadrante superior direito representa singletos. Desenhe um portão poligonal ao redor dessa população clicando no botão de polígono (Polígono) na barra de ferramentas superior esquerda.
- Clique uma única vez, deslize o mouse para começar a desenhar e clique novamente para concluir um segmento linear do portão e começar o próximo. Clique duas vezes para terminar o portão. Clique na borda do portão e deslize o mouse para reposicioná-lo.
- Clique nos vértices da porta para modificar a forma. Serão apresentadas contagens/μL da população fechada (singletos). Esta é a concentração da preparação dos núcleos com a diluição inicial.
NOTA: A relação linear entre a área de dispersão para a frente e a altura 1:1, comumente usada para determinar a taxa de singletos ao fluir células, não se aplica aos núcleos e pode ser desconsiderada.
- Usando a concentração da preparação dos núcleos diluídos e o fator de diluição, calcule novamente a concentração da preparação inicial (por exemplo, para uma diluição de 1:10, multiplique a concentração observada por 10). Concentrações de >300 núcleos/μL são adequadas para snRNA-seq.
- Configure um espaço de trabalho:
Resultados
O protocolo de isolamento de núcleos cerebrais de killifish descrito aqui é otimizado especificamente para o killifish e está resumido na Figura 1. Além da extração de núcleos, o protocolo detalha diferentes métodos para avaliar a qualidade e a quantidade de núcleos isolados. A Figura 2 mostra exemplos de núcleos saudáveis (Figura 2A) e insalubres (Figura 2B), avaliados por microscopia de luz. Núcleos sadios adequados para análise a jusante (Figura 2A) apresentam-se como núcleos singletes com membranas intactas. Como mostra a Figura 2B, o modo de falha mais comum deste protocolo (e de outros protocolos de isolamento de núcleos) é a geração de núcleos sobrecarregados, que são caracterizados por membranas nucleares danificadas. Isso geralmente leva à aglomeração de núcleos e pode contribuir para sinais de fundo em aplicações a jusante devido ao vazamento de ácidos nucleicos dos núcleos rompidos. Se for observada aglomeração excessiva, recomendamos ser mais suave durante as etapas de rejeição e/ou reduzir os tempos de incubação em tampão de lise.
Este protocolo usa citometria de fluxo para quantificar o número de núcleos isolados e determinar a proporção relativa de núcleos múltiplos em uma amostra. Além disso, a citometria de fluxo pode ser usada para avaliar o conteúdo relativo de detritos contaminantes, fragmentos de núcleos frequentemente rompidos e outros detritos celulares que podem afetar negativamente os ensaios a jusante na amostra de núcleos. Os dados de fluxo representativos podem ser vistos na Figura 3. Um procedimento de isolamento de núcleos de alta qualidade produzirá: 1) uma pequena fração de detritos e 2) uma alta fração de núcleos singlete versus múltiplo (> 80% da fração núcleo). O uso da coloração PI, que cora ácidos nucleicos, permite que os núcleos singletes sejam segregados dos detritos e núcleos múltiplos. A Figura 3A é um exemplo de uma preparação contaminada por detritos, enquanto a Figura 3B é um exemplo de um experimento de alta qualidade.
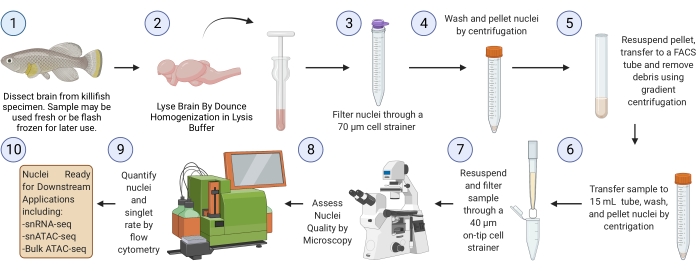
Figura 1: Fluxo de trabalho de isolamento dos núcleos cerebrais de Killifish. Fluxo de trabalho experimental para isolamento de núcleos de cérebros de killifish congelados ou frescos. Uma vez isolados e avaliados, os núcleos podem ser usados para várias análises "ômicas" a jusante. Por favor, clique aqui para ver uma versão maior desta figura.
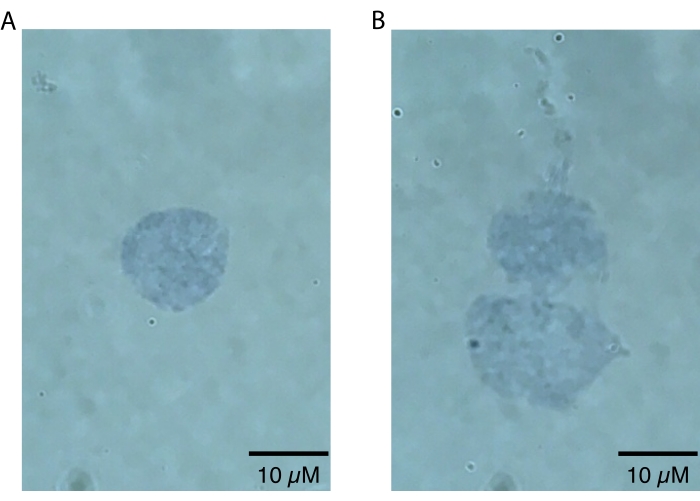
Figura 2: Avaliação da qualidade dos núcleos por microscopia. Os núcleos foram misturados 1:1 em uma solução de Azul de Tripano a 0,4% e visualizados em um microscópio Echo Revolve por imagem de campo brilhante a 60x. (A) Um exemplo de um núcleo de alta qualidade. O núcleo está presente como um singlete e a membrana nuclear está intacta. (B) Um exemplo de núcleos de baixa qualidade. As membranas nucleares estão danificadas e os núcleos estão se aglomerando em um dubleto, provavelmente devido ao vazamento de DNA pegajoso, que pode ser visto vazando do topo do núcleo mais alto. Por favor, clique aqui para ver uma versão maior desta figura.
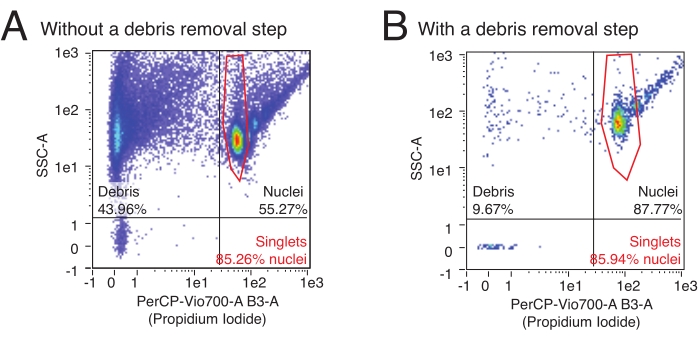
Figura 3: Quantificação de núcleos e avaliação da pureza por citometria de fluxo. As amostras de núcleos foram coradas com diluição de 1:100 de IP e executadas em citômetro de fluxo. Os núcleos estão presentes em formas singletas e múltiplas no quadrante superior direito de (A,B) com singlets como a menor nuvem de eventos seguida por duplos, trigêmeos, etc. em ordem crescente. Os detritos são marcados pelos eventos nos dois quadrantes mais à esquerda. (A) Um exemplo de uma preparação de núcleos de baixa qualidade, onde a etapa de remoção de detritos foi omitida e os detritos representam >40% dos eventos. (B) Um exemplo de uma amostra de núcleos de alta qualidade, com a etapa de remoção de detritos incluída. Neste exemplo, os detritos representam <10% de todos os eventos registrados pelo citômetro de fluxo. Por favor, clique aqui para ver uma versão maior desta figura.
| Tipo de amostra (2 cérebros) | Rendimento Médio (4 preparações independentes) |
| Cérebros masculinos de 5 semanas | 3,59 ± 1,76 x 105 |
| Cérebros masculinos de 10 semanas | 6,41 ± 1,33 x 105 |
| Cérebros masculinos de 15 semanas | 14,59 ± 2,05 x 105 |
| Cérebros femininos de 5 semanas | 2,54 ± 0,75 x 105 |
| Cérebros femininos de 10 semanas | 4,66 ± 1,29 x 105 |
| Cérebros femininos de 15 semanas | 7,95 ± 3,51 x 105 |
Tabela 1: Rendimentos médios esperados de dois cérebros de killifish turquesa africano ao longo do sexo e da idade. Os rendimentos médios expressos em 10a 5 núcleos ± erro padrão da média em quatro preparações de núcleos independentes em cada categoria.
Figura 1 Suplementar: Comparação da qualidade do isolamento de núcleos usando protocolos padrão e nosso protocolo otimizado em cérebros de killifish congelados. (A) Avaliação da qualidade dos núcleos por microscopia. Os núcleos foram misturados 1:1 em uma solução de Azul de Tripano a 0,4% e visualizados em microscópio por imagem de campo brilhante a 60x. (B) Quantificação da carga de núcleos e detritos por citometria de fluxo. As amostras de núcleos foram coradas com diluição de 1:100 de IP e executadas em citômetro de fluxo. (C) Resumo das métricas de avaliação da qualidade por microscopia e citometria de fluxo com os diferentes protocolos referenciados. Clique aqui para baixar este arquivo.
Figura 2 suplementar: Representação esquemática das etapas de remoção de detritos. (A) Esquema de remoção de detritos em camadas pré-centrifugação. (B) Esquema de remoção do sobrenadante após centrifugação. Clique aqui para baixar este arquivo.
Discussão
O protocolo aqui apresentado pode ser usado para gerar de forma reprodutível núcleos de alta qualidade a partir de cérebros de killifish. Este protocolo teve que ser projetado especificamente para o cérebro killifish, pois os protocolos típicos de isolamento de núcleos cerebrais baseados em mamíferos aplicados a cérebros de killifish resultaram consistentemente em baixa qualidade dos núcleos em nossas mãos. Suspeitamos que isso se deva ao menor teor relativo de mielina do cérebro de killifish em comparação com seus homólogos mamíferos, que lisariam e se aglomerariam em resposta às condições adversas necessárias para a lise de células cerebrais de mamíferos. Este protocolo é um avanço nos campos de envelhecimento e killifish, pois facilita a exploração do envelhecimento cerebral no nível de célula única em um modelo de custo e tempo eficaz do envelhecimento cerebral de vertebrados.
Este protocolo é robusto para amostras frescas ou congeladas, embora se deva considerar as aplicações a jusante ao usar tecido fresco ou congelado. O tecido congelado é muitas vezes conveniente, pois pode ser coletado e armazenado por meses enquanto as amostras são coletadas. Essas amostras podem ser usadas com segurança para aplicações como snRNA-seq. No entanto, o congelamento de amostras pode perturbar a estrutura nuclear e, portanto, a capacidade de medir com precisão a paisagem da cromatina por ATAC-seq21. Assim, para aplicações a jusante, como ATAC-seq em massa ou snATAC-seq, recomenda-se o uso de cérebros recém-dissecados em vez de cérebros congelados. Além disso, como todas as etapas após a homogeneização cerebral podem ser realizadas em paralelo, esse protocolo é passível de executar várias amostras em um período de tempo razoável, limitando assim a degradação do RNA causada pela incubação prolongada no gelo.
Além disso, é imperativo preparar tampões frescos (dentro de horas) após a realização da extração de núcleos. Descobrimos que detergentes, bem como BSA, devem ser adicionados aos buffers imediatamente antes do início do protocolo. Tampões contendo apenas sais (PBS, NaCl, etc.) podem ser feitos como concentrados, esterilizados por filtro (0,22 μm) e armazenados indefinidamente à temperatura ambiente. As soluções-mãe de BSA podem ser preparadas, esterilizadas e armazenadas a 4 °C no prazo de dias após a extracção dos núcleos (se preparadas a partir de um pó), mas devem ser sempre adicionadas aos tampões utilizados no presente protocolo imediatamente antes de o efectuarem. No entanto, recomendamos a preparação de soluções BSA no dia do protocolo. Se estiver usando soluções BSA pré-fabricadas de terceiros, é aconselhável usar garrafas frescas e fechadas. O uso de BSA fresco geralmente leva a um menor teor de detritos nos preparativos dos núcleos.
Se amostras frescas ou congeladas são usadas como entrada, é importante avaliar a qualidade dos núcleos após o isolamento dos núcleos. Embora este protocolo seja projetado especificamente para evitar a sobrelise, esta é a causa mais comum de perda de qualidade dos núcleos. A sobrelise pode resultar de muito tempo gasto no tampão de lise, manuseio excessivamente áspero dos núcleos, como pipetagem excessiva com uma ponta de pipeta de furo padrão, ou uma quantidade excessiva de tempo gasto entre o isolamento dos núcleos e as aplicações a jusante (>1 h). Núcleos excessivamente sobrecarregados muitas vezes terão periferias nucleares danificadas, que vazam DNA e causam aglomeração (Figura 2B). Isso levará a um aumento do número de múltiplos e contribuirá com ácidos nucleicos de fundo que interferirão nas aplicações a jusante, especialmente o snRNA-seq. Ambas as qualidades podem ser avaliadas por microscopia após o isolamento dos núcleos. Se for observada aglomeração nuclear excessiva, recomendamos tentar encurtar a incubação da etapa de lise para reduzir a chance de sobrelise. Como um método alternativo para aumentar os singletos dos núcleos, a classificação celular assistida por fluorescência (FACS) pode ser usada para enriquecer os singlets a jusante deste protocolo. No entanto, observamos que, ao trabalhar com núcleos já frágeis de tecido congelado, o estresse de cisalhamento que ocorre durante a triagem pode levar ao aumento da ruptura nuclear e, portanto, ao aumento do RNA/DNA ambiente. Além disso, observamos que o tempo necessário para executar uma classificação de rendimento FACS para núcleos ao processar várias amostras exigiria que todas as amostras de núcleos permanecessem no gelo por horas, enquanto outras amostras estão sendo classificadas. Assim, o aumento dos tempos de espera ao processar várias amostras em paralelo com a abordagem FACS também poderia levar à redução geral da qualidade dos núcleos e aumentar o risco de degradação do RNA. Assim, se o FACS for desejado para uma taxa de duplicidade reduzida, recomendamos que o teor de detritos seja verificado novamente após a classificação do rendimento e que a possível qualidade reduzida do RNA seja levada em conta para aplicações de RNA-seq de célula única como uma ressalva potencial.
Uma estimativa precisa da contagem de núcleos e da proporção de singletos é essencial para quase todas as aplicações "ômicas" a jusante e é de extrema importância. Devido à capacidade de facilmente portar e contar núcleos por tamanho, a citometria de fluxo é o método mais preciso de contagem de núcleos que avaliamos. Alternativamente, pode-se quantificar núcleos usando contadores de células como o Contador de Células Automatizadas Countess 2 FL da Invitrogen ou o Contador de Células Automatizadas DeNovix CellDrop . Para notar, o Contador Automatizado de Células Countess 2 FL da Invitrogen e, em muito menor grau, o DeNovix, tendem a superestimar as contagens de núcleos contando detritos como núcleos, o que significa que o tamanho manual pode ser necessário. Além disso, o citômetro de fluxo permite avaliar facilmente a pureza dos núcleos. Pode-se discernir a proporção relativa de núcleos singlete versus múltiplo de uma maneira quantitativa que é difícil pela microscopia. Isso é vital para o snRNA-seq e o snATAC-seq, uma vez que esses protocolos sofrerão de um excedente de amostras múltiplas, que devem ser excluídas das análises a jusante. Além dos múltiplos, a proporção relativa de "detritos" (núcleos fragmentados, detritos celulares) pode ser quantificada por citometria de fluxo e deve ser relativamente baixa, uma vez que este material geralmente contém ácidos nucleicos contaminantes que podem contribuir com um sinal de fundo e corromper dados "ômicos" de núcleo único.
Tal como acontece com os protocolos de isolamento de núcleos descritos anteriormente, as proporções de tipos celulares no tecido original não podem ser recapituladas fielmente na preparação dos núcleos19 e, portanto, devem ser interpretadas com cautela. Para notar, como todos os teleósteos, o killifish turquesa africano tem eritrócitos nucleados, que também devem ser representados na preparação dos núcleos. Esses núcleos podem ser identificados nos conjuntos de dados snRNA-seq e snATAC-seq pela maior expressão/acessibilidade dos genes da hemoglobina e podem ser excluídos computacionalmente, se desejado.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Alguns painéis foram gerados com BioRender.com. Este trabalho em nosso laboratório foi apoiado pela NIA T32 AG052374 Postdoctoral Training Grant to B.T., uma bolsa da Simons Foundation como parte da Simons Collaboration for Plasticity in the Aging Brain, uma bolsa piloto da Navigage Foundation e um prêmio da Família Hanson-Thorell para B.A.B.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan Blue solution | Gibco | 15250061 | |
| 10x PBS | Bioland | PBS01-03 | |
| 15 mL Conicle Centrifuge Tube | VWR | 89039-664 | |
| 2 mL Tissue Grinder | Kimble | 885300-0002 | Dounce Homogenizer |
| 5 mL Polystyrene Round-Bottom Tube | Falcon | 352054 | |
| 5M Sodium Chloride, Molecular Biology Grade | Promega | V4221 | |
| autoMACS Rinsing Solution | Miltenyi Biotec | 130-091-222 | This corresponds to 1x PBS pH 7.2, 2 mM EDTA (used for flow cytometry) |
| Debris Removal Solution | Miltenyi Biotec | 130-109-398 | |
| DNA LoBInd Tube | Eppendorf | 22431021 | |
| Echo Revolve microscope (fitted with 60x objective) | Echo | NA | No catalog number; Used to visually inspect nuclei. |
| Falcon Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352052 | FACS tube |
| FLOWMI Cell Strainers, 40 μM | SP Bel-Art | 136800040 | Referred to as on-tip filters in Protocol |
| Hydrochloric Acid | Sigma-Aldrich | 258146-500 mL | Used to lower TRIS pH from 8.0 to 7.4 |
| Leica EZ4 dissecting scope | Leica | NA | No catalog number |
| MACS BSA stock solution | Miltenyi Biotec | 130091376 | |
| MACS SmartStrainers (70 μM) | Miltenyi Biotec | 130-110-916 | |
| MACSQuant Analyzer 10 Flow Cytometer | Miltenyi Biotec | NA | No catalog number |
| Magnesium Chloride, Hexahydrate, Molecular Biology Grade (Powder) | Millipore | 442611 | |
| Megafuge 16R Centrifuge | ThermoScientific | 75003629 | |
| Micro Cover Glass | VWR | 48393081 | |
| Micro Slides Superfrost Plus | VWR | 48311-703 | |
| Nonidet P-40 Substitute | Roche | (Roche) 11332473001/ Catalog: 983739 P Code: -102368106 (Sigma Aldrich) | |
| NP-40 Surfact-Amps Detergent Solution | ThermoFisher | 85124 | |
| Nuclease-Free HyPure Molecular Biology Grade Water | HyClone | SH30538.02 | |
| NxGen RNase Inhibitor (50,000 U) | Lucigen | 30281-2 | |
| Propidium Iodide solution | MBL | FP00010020 | |
| PureBlu DAPI Nuclear Staining Dye | Biorad | 1351303 | |
| TipOne RPT Pipette Tips (Ultra low retention, filtered) in 10 µL, 20 µL, 200 µL, and 1000 µL sizes | USA Scientific | #1181-3810; #1180-1810; #1180-8810; #1182-1830 | |
| Tricaine-S (MS 222) | Syndel | Tricaine10G | Syndel is an FDA-approved provider for pharmaceutical grade Tricaine |
| TRIS, 1 M, pH 8.0 | VWR | E199-500 mL | |
| Wide Bore Pipet Tips | Axygen | T-1005-WB-C |
Referências
- Yankner, B. A., Lu, T., Loerch, P. The aging brain. Annual Review of Pathology. 3 (1), 41-66 (2008).
- Hu, C. K., Brunet, A. The African turquoise killifish: A research organism to study vertebrate aging and diapause. Aging Cell. 17 (3), 12757 (2018).
- McKay, A., et al. An automated feeding system for the African killifish reveals effects of dietary restriction on lifespan and allows scalable assessment of associative learning. bioRxiv. , (2021).
- Valenzano, D. R., Terzibasi, E., Cattaneo, A., Domenici, L., Cellerino, A. Temperature affects longevity and age-related locomotor and cognitive decay in the short-lived fish Nothobranchius furzeri. Aging Cell. 5 (3), 275-278 (2006).
- Vanhunsel, S., et al. The killifish visual system as an in vivo model to study brain aging and rejuvenation. NPJ Aging and Mechanisms of Disease. 7 (1), 22 (2021).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), 0209648 (2018).
- Ziffra, R. S., et al. Single-cell epigenomics reveals mechanisms of human cortical development. Nature. 598 (7879), 205-213 (2021).
- Grindberg, R. V., et al. RNA-sequencing from single nuclei. Proceedings of the National Academy of Sciences. 110 (49), 19802-19807 (2013).
- Kalish, B. T., et al. Single-nucleus RNA sequencing of mouse auditory cortex reveals critical period triggers and brakes. Proceedings of the National Academy of Sciences. 117 (21), 11744-11752 (2020).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Gaublomme, J. T., et al. Nuclei multiplexing with barcoded antibodies for single-nucleus genomics. Nature Communications. 10 (1), 2907 (2019).
- Narayanan, A., et al. Nuclei isolation from fresh frozen brain tumors for single-nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments. (162), e61542 (2020).
- Saunders, A., et al. Molecular diversity and specializations among the cells of the adult mouse brain. Cell. 174 (4), 1015-1030 (2018).
- . Isolation of Nuclei for Single Cell RNA Sequencing & Tissues for Single Cell RNA Sequencing Available from: https://support.10xgenomics.com/permalink/1dlB6Z91VqClgUmSC2OM8k (2021)
- Martin, C., et al. Frozen tissue nuclei extraction (for 10xV3 snSEQ) V.2. protocols.io. , (2020).
- Nuclei Isolation from Adult Mouse Brain Tissue for Single Cell RNA Sequencing. 10xGenomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/sample-prep/nuclei-isolation-from-adult-mouse-brain-tissue-for-single-cell-ma-sequencing (2022)
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- What are the best practices for working with nuclei samples for 3' single-cell gene expression. 10xGenomics Available from: https://kb.10xgenomics.com/hc/en-us/articles/360050780051-What-are-the-best-practices-for-working-with-nuclei-samples-for-3-single-cell-gene-expression (2022)
- Rocks, D., et al. Cell type-specific chromatin accessibility analysis in the mouse and human brain. Epigenetics. 17 (2), 202-219 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados