Method Article
Aislamiento suave de núcleos del tejido cerebral del pez killifish turquesa africano adulto, un modelo naturalmente de corta duración para la investigación del envejecimiento
En este artículo
Resumen
Aquí presentamos un protocolo para aislar núcleos de los cerebros del modelo de vertebrados de vida corta Nothobranchius furzeri para aplicaciones aguas abajo, como la secuenciación de ARN de núcleo único o el ensayo de núcleo único para cromatina accesible a transposasa con secuenciación (ATAC-seq).
Resumen
Estudiar el envejecimiento cerebral a resolución unicelular en sistemas de vertebrados sigue siendo un desafío debido al costo, el tiempo y las limitaciones técnicas. Aquí, demostramos un protocolo para generar bibliotecas de secuenciación de ARN de núcleo único (snRNA-seq) a partir de los cerebros del killi turquesa africano vertebrado de vida naturalmente corta Nothobranchius furzeri. El killi turquesa africano tiene una vida útil de 4-6 meses y se puede alojar de manera rentable, reduciendo así las barreras de costo y tiempo para estudiar el envejecimiento cerebral de los vertebrados. Sin embargo, se necesitan protocolos adaptados para aislar núcleos de calidad suficiente para experimentos unicelulares aguas abajo del cerebro de peces jóvenes y envejecidos. Aquí, demostramos un protocolo empíricamente optimizado para el aislamiento de núcleos de alta calidad del cerebro de killi turquesa africano adulto, un paso crítico en la generación de bibliotecas ómicas de núcleos únicos de alta calidad. Además, mostramos que los pasos para reducir el ARN de fondo contaminante son importantes para distinguir claramente los tipos de células. En resumen, este protocolo demuestra la viabilidad de estudiar el envejecimiento cerebral en organismos modelo vertebrados no tradicionales.
Introducción
Comprender los mecanismos del envejecimiento cerebral de los vertebrados es fundamental para abordar las enfermedades neurodegenerativas relacionadas con la edad, como el Alzheimer y la demencia1. El killi turquesa africano (Nothobranchus furzeri) es el vertebrado de vida más corta que se puede criar en cautiverio, y debido a su corta vida útil y deterioro cognitivo asociado a la edad, es un excelente modelo de envejecimiento cerebral 2,3,4,5. Recientemente, el advenimiento de las tecnologías "ómicas" de una sola célula, como el ARN-seq de un solo núcleo (snRNA-seq) y el ensayo de núcleos únicos para la cromatina accesible a transposasa con secuenciación (snATAC-seq), han permitido a los investigadores interrogar el cerebro envejecido a una resolución sin precedentes 6,7,8. Estos métodos se basan en el aislamiento de núcleos, ya que la recuperación de células cerebrales como las neuronas a menudo es demasiado difícil para aislar 6,7,8,9,10. Sin embargo, la mayoría de los protocolos de aislamiento de núcleos publicados están optimizados para organismos modelo mamíferos 11,12,13,14,15. Por lo tanto, como actualmente existe una necesidad insatisfecha de aislar núcleos cerebrales en el killifish como un nuevo organismo modelo prometedor en el campo de la investigación del envejecimiento2, el objetivo de este protocolo es establecer un método para aislar núcleos de alta calidad del tejido congelado del killifish cerebral.
Aquí, se establece un flujo de trabajo simplificado y robusto que utiliza materiales comúnmente disponibles para aislar núcleos de alta calidad de cerebros de killifish. Este protocolo se modificó a partir de un protocolo de genómica 10x para cerebros de ratones para acomodar los cerebros con menor contenido de mielina del killi turquesa africano, la fragilidad del tejido congelado y la necesidad de reducir el contenido de desechos ambientales para aplicaciones relacionadas con la secuenciación16. De hecho, los protocolos previamente optimizados para el tejido cerebral de mamíferos17,18 conducen a una mala calidad de los núcleos (es decir, sobrelisis) y / o alto contenido de desechos cuando se usan en cerebros congelados de killifish, lo que los hace inadecuados para su uso con snRNA-seq de acuerdo con las recomendaciones para núcleos individuales RNA-seq utilizando microfluídica (Figura suplementaria 1).
Además del aislamiento de núcleos, demostramos cómo evaluar la calidad y el rendimiento de los núcleos mediante microscopía y citometría de flujo. En este artículo se proporcionan ejemplos de resultados óptimos y subóptimos y se describe la solución de problemas. Este protocolo fue diseñado y optimizado para cerebros congelados de killifish, pero también se puede usar sin modificaciones importantes en muestras de killi recién diseccionadas. Los núcleos cerebrales de Killifish aislados utilizando este método se han optimizado para su uso en RNA-seq de núcleo único (snRNA-seq) como una aplicación posterior, pero también deben ser susceptibles de uso en snATAC-seq y ATAC-seq a granel.
Protocolo
El cuidado de los animales y la experimentación con animales se realizaron de acuerdo con la Universidad del Sur de California IACUC bajo los protocolos aprobados # 21215. Para cualquier trabajo que utilice este protocolo, es necesario obtener la aprobación del IACUC de la institución antes de comenzar cualquier trabajo de investigación en animales vertebrados.
NOTA: Una ejecución completa del protocolo a partir de tejido cerebral congelado rápidamente (a partir del paso 3) debe tomar ~ 2.5 h para seis muestras (Figura 1).
1. Diseccionar cerebros de killifish
- Sacrificar humanamente killifish usando una solución de 1.5 g / L de metanosulfonato / tricaína (MS-222) en el agua del sistema.
- Use tijeras afiladas para decapitar rápidamente a los killis después de que todo el movimiento del cuerpo y las branquias haya cesado.
- Diseccionar la cabeza decapitada en una placa de Petri limpia como se describió anteriormente19.
- Coloque el plato bajo un microscopio de disección (usando un rango de aumento de 8x-35x). Gire la cabeza de tal manera que los rayos branquiostegales estén enfocados
- Usando tijeras, corte a través del dentario de tal manera que se revelen los rayos branquiostegales y el cráneo.
- Usando fórceps, mantenga presionados los rayos branquiostegales a ambos lados del cráneo. Use otro par de fórceps para extraer cualquier fragmento de tejido restante del cráneo. El quiasma óptico en forma de V que conecta el cerebro y los ojos debe hacerse visible.
- Corta el quiasma óptico usando tijeras para separar ambos ojos del cerebro. Use fórceps para liberar suavemente el cráneo.
- Invierta el cráneo y use fórceps para raspar el músculo del lado dorsal del cráneo.
- Use un par de fórceps para mantener el cráneo en su lugar y otro par de fórceps para extraer suavemente los huesos del cráneo, revelando el cerebro.
- El cerebro aparece blanco y suave. Una vez visible, no es necesario extirpar todos los huesos craneales. Retire el cerebro con fórceps limpios levantando y raspando suavemente.
- Congele el cerebro colocándolo en un tubo de microcentrífuga almacenado en hielo seco.
NOTA: El tejido cerebral congelado se puede almacenar a -80 °C hasta que se hayan recogido todas las muestras que se procesarán juntas para limitar los efectos de lote al procesar para la generación de bibliotecas. Si bien las muestras almacenadas a -80 ° C no se pueden almacenar indefinidamente, este protocolo se ha realizado con éxito en cerebros de killi almacenados hasta 12 meses sin una caída notable en la calidad.
2. Prepara tampones nuevos
- Preparar tampón de lavado de núcleos (albúmina sérica bovina [BSA] al 2% en 1x PBS, solución salina tamponada con fosfato pH 7.4 e inhibidor de la RNasa 0.2 U/μL) y almacenar en hielo. Prepare 250 ml de tampón de lavado de núcleos para hasta ocho muestras. Para 250 ml, use 50 ml de solución madre BSA al 10%, 25 ml de una solución madre de PBS 10x, 1.25 ml de la cepa de inhibidores de RNasa de 40 U/μL y luego lleve el volumen a 250 ml con ddH2O libre de ARNasa.
- Preparar tampón de lisis de núcleos (10 mM Tris-HCl pH 7.4, 10 mM NaCl, 3 mM MgCl 2, 0.01875% v/v NP-40 y0.2 U/μL RNasa inhibidor) y almacenar en hielo. Prepare 10 ml de tampón de lisis de núcleos para hasta ocho muestras. Para 10 ml, utilizar 100 μL de solución madre de Tris-HCl pH 7,4 de 1 M, 20 μL de solución madre de NaCl 5 M, 30 μL de solución madre de 1 M de MgCl2 , 18,75 μL de una solución madre de NP-40 al 10%, 50 μL de la cepa de inhibidores de RNasa de 40 U/μL y, a continuación, llevar el volumen a 10 ml conddH2Olibre de ARNasa.
3. Aislar núcleos
- Si usa muestras congeladas, descongélelas en hielo durante 10 minutos. Si utiliza cerebros recién disecados, continúe directamente con el paso 3.2
NOTA: Aunque este protocolo puede funcionar en cerebros individuales, las muestras más pequeñas de peces jóvenes (5-6 semanas de edad) tendrán un rendimiento más pobre. Siempre se pueden obtener rendimientos suficientes procesando dos cerebros como una muestra, independientemente de la edad y el sexo del pez (Tabla 1). Este protocolo ha aislado con éxito núcleos de alta calidad de peces de 5 a 26 semanas, incluidos animales de las cepas GRZ y ZMZ1001. No se esperan problemas al aplicar este protocolo a otras cepas. - Rebote el cerebro en 1 ml de tampón de lisis de núcleos helados en un homogeneizador de 2 ml de rebote. Mantenga el homogeneizador en hielo y evite generar burbujas mientras lisa la muestra. Rebota a una velocidad aproximada de 0,5 s/carrera descendente y 0,5 s/carrera ascendente con un giro de 180° por ciclo. Rebote usando el mortero suelto durante 15 golpes, o hasta que la mezcla de tampón de tejido / lisis parezca homogénea si se necesitan trazos adicionales.
NOTA: Los homogeneizadores Dounce deben lavarse, enjuagarse con agua destilada y esterilizarse con ciclos estándar de esterilización en autoclave seco entre usos.- Rebote usando el mortero apretado durante 30 golpes con pequeños descansos, de 30 s a 1 minuto, cada 10 golpes. Rebota a una velocidad aproximada de 0,5 s/carrera descendente y 0,5 s/carrera ascendente con un giro de 180° por ciclo. Deje reposar la muestra sobre hielo en el homogeneizador Dounce durante 2 min.
NOTA: Este tiempo de incubación garantiza una lisis adecuada, pero debe acortarse si se observa una sobrelisis nuclear.
- Rebote usando el mortero apretado durante 30 golpes con pequeños descansos, de 30 s a 1 minuto, cada 10 golpes. Rebota a una velocidad aproximada de 0,5 s/carrera descendente y 0,5 s/carrera ascendente con un giro de 180° por ciclo. Deje reposar la muestra sobre hielo en el homogeneizador Dounce durante 2 min.
- Colar la muestra a través de un filtro celular de 70 μm en un tubo cónico de 15 ml prerecubierto en BSA al 5%.
- Añadir 4 ml de tampón de lavado de núcleos a la muestra pipeteando el tampón de lavado sobre el filtro de celda de 70 μm del paso 3.3 y mezclar suavemente por inversión cinco veces.
NOTA: Esto permite la recuperación de núcleos que pueden quedar atrapados en el filtro y mejora el rendimiento. - Centrífuga con rotor de cangilón oscilante a 500 x g, 4 °C, durante 10 min. Deseche el sobrenadante vertiéndolo suavemente en un recipiente de desechos líquidos. Devuelva la muestra a una posición vertical y retire el tampón de lavado restante con una pipeta P1000.
NOTA: Es probable que el pellet de núcleos sea visible cuando se parte de dos cerebros en una muestra. Sin embargo, cuando se procesan cerebros individuales de animales jóvenes (5-6 semanas de edad), se espera un rendimiento más bajo, y los pellets no siempre pueden ser visibles. En este caso, el sobrenadante debe retirarse cuidadosamente mientras asume la posición del pellet en el fondo del tubo. - Resuspender inmediatamente los núcleos en 1 ml de tampón de lavado de núcleos con una punta de pipeta P1000 de diámetro ancho.
- Lleve la muestra a 5 ml agregando 4 ml de tampón de lavado de núcleos y mezcle suavemente por inversión cinco veces. Centrifugar con un rotor de cangilón oscilante a 500 x g, 4 °C, durante 10 min, y desechar el sobrenadante.
- Resuspender los núcleos en 1 ml de tampón de lavado de núcleos con una punta de pipeta de diámetro ancho y transferirlos a un tubo de citometría de flujo (FACS).
- Añadir 300 μL de solución de eliminación de residuos (Tabla de materiales) y mezclar bien pipeteando con una punta de diámetro ancho hasta que la mezcla sea homogénea.
- Cubrir suavemente 1 ml de tampón de lavado de núcleos sobre la solución de núcleos preparada en el paso 3.9 utilizando una pipeta P1000 y sosteniendo el tubo en un ligero ángulo (figura complementaria 2). La fracción de solución de eliminación de residuos y la fracción tampón de lavado de núcleos deben formar una interfaz clara si se colocan correctamente en capas.
- Centrífuga en rotor de cangilón oscilante a 3000 x g, 4 °C, durante 10 min. Deseche completamente las dos fases superiores con una pipeta P1000.
NOTA: El tubo FACS transparente permitirá la visualización de las tres fases distintas después de la centrifugación (superior, interfase, inferior). Para facilitar la eliminación máxima de residuos, se recomienda ser más agresivo que conservador en la eliminación de las dos fases superiores (es decir, la eliminación de una pequeña cantidad de la fase inferior es preferible a la transferencia de cualquiera de las fases superiores; Figura complementaria 2). Además, la capa de interfase puede ser demasiado delgada para visualizarla. En este caso, elimine la capa superior (aparente), que contiene la capa de interfase. La capa inferior que se retiene debe contener ~ 1 ml de volumen total. - Transfiera la capa inferior a un tubo cónico de 15 ml prerecubierto con BSA al 5%.
- En el tubo cónico, llevar el volumen a 15 ml con tampón de lavado de núcleos e invertir tres veces para mezclar.
- Centrífuga en rotor de cangilón oscilante a 1000 x g, 4 °C, durante 10 min. Retire el sobrenadante con cuidado.
- Resuspender inmediatamente en 150 μL de tampón de lavado de núcleos utilizando una punta de pipeta de diámetro ancho.
- Cambie a una punta de pipeta de orificio estándar ajustada a ~300 μL. Tome toda la muestra y luego coloque un filtro de punta de 40 μm en el extremo de la punta de la pipeta, teniendo cuidado de no expulsar la muestra.
NOTA: Los filtros en punta son necesarios para obtener volúmenes más pequeños, que son necesarios para mantener una concentración de núcleos suficiente para aplicaciones posteriores. Por ejemplo, para snRNA-seq utilizando microfluídica, se recomienda una concentración mínima de >300 núcleos/μL, aunque el rango óptimo es de 700-1200 núcleos/μL18. Las concentraciones más altas de núcleos también se pueden diluir fácilmente si es necesario. - Filtrar la muestra expulsándola a través del filtro a través del filtro en un tubo de microcentrífuga de 1,5 ml de unión baja al ADN. El volumen final de elución debe ser de 120-150 μL.
NOTA: Algo de espuma puede resultar de la filtración. Esto no parece afectar la calidad de los núcleos.
4. Evaluar la calidad de los núcleos mediante microscopía
- En un tubo de microcentrífuga separado, mezclar 10 μL de la muestra filtrada con 10 μL de solución de azul de tripano al 0,4% mediante un pipeteo suave. Las muestras no requieren un tiempo de incubación prolongado y están listas para su visualización inmediatamente después de mezclarlas con la solución de azul de tripano.
- Depositar 10 μL de los núcleos teñidos en una cámara de un portaobjetos de cámara de conteo.
NOTA: Alternativamente, deposite 10 μL de la muestra de núcleos teñidos en un portaobjetos de hemocitómetro o microscopía y cúbrala con un cubreobjetos. - Evaluar la calidad de los núcleos mediante microscopía óptica.
NOTA: Los aumentos más bajos solo deben usarse para acercar los núcleos, ya que los defectos de los núcleos solo serán visualmente evidentes a aumentos más altos (es decir, 60x o más). Este paso se puede hacer rápidamente para evaluar el éxito de la preparación de núcleos y no requiere un análisis secundario más allá de la inspección visual. Los núcleos intactos de alta calidad tendrán un borde20 claramente definido (Figura 2A). Los núcleos de mala calidad tendrán membranas nucleares interrumpidas, tinción irregular de azul de tripano cerca de la membrana nuclear indicativa de posible fuga de ácido nucleico y / o evidencia de ampollo20 (Figura 2B). Se espera que los núcleos sanos tengan un diámetro de 10-15 μm.
5. Cuantificar núcleos singletes, desechos y proporción de multipletes por citometría de flujo
NOTA: La terminología específica y la interfaz del software del citómetro de flujo pueden diferir según la marca de la máquina, pero estos pasos deben adaptarse fácilmente a otros sistemas si es necesario.
- En un tubo FACS, resuspender 10 μL de la muestra de núcleos filtrados en 90 μL del tampón recomendado para la citometría de flujo para una dilución 1:10. Para este propósito, en este estudio se utiliza PBS esterilizado filtrado de 0,22 μm, pH 7,2, con 2 mM EDTA y 0,5% de BSA.
NOTA: Aunque esta dilución funcionará para la mayoría de las preparaciones de núcleos, puede ser necesaria una dilución más baja (por ejemplo, 1:5) para muestrear suficientes núcleos para la evaluación de la calidad en el caso de una muestra de menor rendimiento (es decir, <30 núcleos/μL en la muestra diluida). - Teñir las muestras con yoduro de propidio (PI) a una concentración final de 1 μg/ml. No se requiere tiempo de incubación y los núcleos se pueden analizar inmediatamente.
NOTA: Alternativamente, tiñe las muestras con una concentración final de 0,1 μg/ml de DAPI. Tenga en cuenta que DAPI debe leerse en un canal de fluorescencia diferente al de PI (canal Vioblue-A V1-A; excitación a 405 nm, emisión a 450/50 nm). - Analice las muestras en un citómetro de flujo de la siguiente manera:
- Configurar un espacio de trabajo:
- Tenga el citómetro de flujo en modo de adquisición. Haga clic en Archivo y, a continuación, seleccione Nuevo espacio de trabajo en el menú desplegable. En la pestaña Experimento en el panel izquierdo, ingrese el Nombre del proyecto y el ID de muestra en sus respectivos campos.
- En la barra de herramientas superior izquierda, haga clic en el botón Nueva ventana de análisis (icono de diagrama de dispersión). Haga clic en el cuadro con tres gráficos (Gráfico 4t) en el menú emergente. Aparecerán tres gráficos en la pantalla. Haga clic en el botón Modo de análisis en la barra de herramientas superior izquierda (icono A) para que sea gris y no naranja.
- Configurar gráficos de análisis:
- Cambie los ejes de los tres gráficos de la siguiente manera, ordenados como eje X y eje Y, respectivamente:
Gráfico 1: Eje X: FSC-A, Eje Y: SSC-A
Gráfico 2: Eje X: HDR-T, Eje Y: SSC-A
Gráfico 3: Eje X: PerCP-Vio700-A B3-A, Eje Y: SSC-A - Cambie los ejes haciendo clic en la etiqueta del eje y seleccionando el canal deseado en el menú desplegable.
- Establezca los gráficos para que estén codificados por colores de densidad haciendo clic en el icono gris cuadrado "i" que aparece junto a la esquina superior derecha de cada gráfico. Se abre una ventana Propiedades con un panel de botones a la izquierda. En la ventana Propiedades, haga clic en el icono Diagrama de dispersión (segundo desde la parte superior a la izquierda del panel). Haga clic en Aceptar.
NOTA: El canal de fluorescencia PerCP-Vio700-A B3-A corresponde a una excitación a 488 nm, y emisión en el rango de 650-730 nm.
- Cambie los ejes de los tres gráficos de la siguiente manera, ordenados como eje X y eje Y, respectivamente:
- Configure los ajustes del instrumento:
- Haga clic en la pestaña Canales en el panel izquierdo. Aparecerá una lista de todos los canales con tres cuadros a la derecha. Establezca B3, FSC-A y SSC-A en Hlog haciendo clic en la flecha hacia abajo en sus respectivos primeros cuadros a la derecha y seleccionando Hlog en el menú desplegable. Ajuste el voltaje de SSC a 640 V, FSC a 300 V y B3 a 480 V escribiendo estos números en sus respectivos cuadros centrales a la derecha y presionando la tecla Enter. Tenga en cuenta que el tercer cuadro a la derecha es un interruptor que se puede usar alternativamente para escribir.
- En el encabezado Trigger , establezca el desencadenador FSC en 4 y el desencadenador SSC y B3 en 0 haciendo clic en la flecha hacia abajo, seleccionándolos en el menú desplegable de la izquierda, escribiendo el número en el cuadro de la derecha y, a continuación, presionando la tecla Intro . Tenga en cuenta que hay un cuadro de alternancia a la derecha que se puede usar alternativamente a escribir.
NOTA: Los voltajes PMT deben optimizarse para cada máquina de citometría de flujo en particular, pero el disparador de los núcleos debe ser más bajo que el que se usa típicamente para las células. Además, la escala del eje debe establecerse en h-log.
- Escriba 100 μL en el cuadro de texto Volumen de muestra y 50 μL en el cuadro de texto Volumen de absorción en el lado izquierdo de la ventana del software de flujo. Compruebe que el parámetro Mix Sample esté establecido en Mix Gentle para evitar la lisis de núcleos en este paso. Presione el triángulo dentro de un círculo (icono Reproducir ) en la esquina inferior derecha para iniciar el flujo.
- Verifique la calidad del flujo:
- Use el área de dispersión lateral (SSC-A) en lugar de HDR-T para verificar si hay burbujas de aire o grumos en la muestra. Asegúrese de que esta gráfica sea un continuo suave de puntos. Las burbujas o grupos de aire darán como resultado regiones en blanco en el gráfico SSC-A versus HDR-T.
- Compruebe la gráfica de dispersión lateral (SSC-A) frente a dispersión directa (FSC-A) para verificar que los voltajes utilizados sean correctos para la muestra.
NOTA: A diferencia de las células, los núcleos no se resolverán completamente a partir de desechos en un gráfico de dispersión lateral frente a dispersión hacia adelante. Sin embargo, debe haber un grupo de eventos de alta densidad hacia la mitad de la parcela correspondiente a núcleos (de color más cálido) con una menor densidad de eventos (de color más frío) en el fondo correspondiente a los escombros.
- Analizar el recuento y la calidad de los núcleos:
- Haga doble clic en el gráfico SSC-A vs. PerCp-Vio-700A para obtener una vista ampliada. Haga clic en el botón de la barra de herramientas superior izquierda con líneas perpendiculares (cuadrante) para seleccionar una puerta de cuadrante. Luego, haga clic en cualquier lugar de la gráfica SSC-A vs. PerCp-Vio-700A y los cuadrantes aparecerán dentro de la gráfica.
- Haga clic en cualquier lugar de las líneas divisorias del cuadrante y deslice el cursor para modificar la ubicación del cuadrante. Coloque los cuadrantes de tal manera que el cuadrante superior izquierdo contenga la población del extremo izquierdo (escombros) y el cuadrante superior derecho contenga varios grupos en forma de lágrima (núcleos) (consulte la Figura 3 para la configuración de la puerta y ejemplos de resultados de flujo para obtener resultados típicos con preparaciones que incluyen o no incluyen un paso de eliminación de desechos).
- Recuentos de escombros versus núcleos:
- Haga clic en el icono gris cuadrado "i" en la esquina superior derecha de la parcela para abrir la ventana Propiedades . Haga clic en la pestaña Funciones de región ; Aparecerán listas de regiones y funciones de trazado.
- En la lista Regiones , asegúrese de que haya una marca de verificación verde junto al elemento superior, que indica que todo el trazado está seleccionado. En la lista Funciones , haga clic en Count/μL para producir una marca de verificación verde. Haga clic en OK y aparecerá una métrica de recuentos/μL en cada cuadrante.
- Para ver los recuentos sin procesar y los porcentajes, seleccione Recuento y %-T en la ficha Funciones de región si lo desea. Los recuentos/μL en los cuadrantes superior izquierdo y derecho se corresponden con el rendimiento de escombros y núcleos, respectivamente.
- Singlete cuenta:
- El grupo en forma de lágrima más a la izquierda en la parte inferior del cuadrante superior derecho representa singletes. Dibuje una puerta poligonal alrededor de esta población haciendo clic en el botón polígono (Polígono) en la barra de herramientas superior izquierda.
- Haga clic una sola vez, deslice el mouse para comenzar a dibujar y vuelva a hacer clic para terminar un segmento lineal de la puerta y comenzar el siguiente. Haga doble clic para finalizar la puerta. Haga clic en el borde de la puerta y deslice el ratón para cambiar la posición de la puerta.
- Haga clic en los vértices de la puerta para modificar la forma. Aparecerán recuentos/μL de la población cerrada (singletes). Esta es la concentración de la preparación de los núcleos con la dilución inicial.
NOTA: La relación lineal entre el área de dispersión directa y la altura 1:1, comúnmente utilizada para determinar la tasa de singlete cuando las células fluyen, no se aplica a los núcleos y puede ignorarse.
- Usando la concentración de la preparación de núcleos diluidos y el factor de dilución, calcule la concentración de la preparación inicial (por ejemplo, para una dilución 1:10, multiplique la concentración observada por 10). Las concentraciones de >300 núcleos/μL son adecuadas para snRNA-seq.
- Configurar un espacio de trabajo:
Resultados
El protocolo de aislamiento de núcleos cerebrales de killifish descrito aquí está optimizado específicamente para el killifish y se resume en la Figura 1. Además de la extracción de núcleos, el protocolo detalla diferentes métodos para evaluar la calidad y cantidad de núcleos aislados. La Figura 2 muestra ejemplos de núcleos sanos (Figura 2A) y no saludables (Figura 2B) evaluados por microscopía óptica. Los núcleos sanos adecuados para el análisis posterior (Figura 2A) se presentan como núcleos singletes con membranas intactas. Como se muestra en la Figura 2B, el modo de falla más común de este protocolo (y otros protocolos de aislamiento de núcleos) es la generación de núcleos sobrelisados, que se caracterizan por membranas nucleares dañadas. Esto a menudo conduce a la aglutinación de los núcleos y puede contribuir a las señales de fondo en aplicaciones posteriores debido a la fuga de ácidos nucleicos de los núcleos rotos. Si se observa una aglutinación excesiva, recomendamos ser más suave durante los pasos de rebote y / o reducir los tiempos de incubación en el tampón de lisis.
Este protocolo utiliza la citometría de flujo para cuantificar el número de núcleos aislados y determinar la proporción relativa de núcleos multipletes en una muestra. Además, la citometría de flujo se puede utilizar para evaluar el contenido relativo de desechos contaminantes, fragmentos de núcleos a menudo rotos y otros desechos celulares que pueden afectar negativamente los ensayos aguas abajo en la muestra de núcleos. Los datos de flujo representativos se pueden ver en la Figura 3. Un procedimiento de aislamiento de núcleos de alta calidad producirá: 1) una pequeña fracción de escombros, y 2) una alta fracción de núcleos singlete versus núcleos multipletes (> 80% de la fracción de núcleos). El uso de la tinción PI, que tiñe los ácidos nucleicos, permite separar los núcleos singletes de los núcleos de desechos y multipletes. La Figura 3A es un ejemplo de una preparación contaminada por escombros, mientras que la Figura 3B es un ejemplo de un experimento de alta calidad.
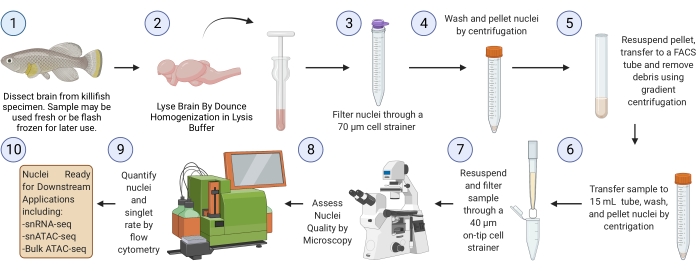
Figura 1: Flujo de trabajo de aislamiento de núcleos cerebrales de Killifish. Flujo de trabajo experimental para el aislamiento de núcleos de cerebros de killifish congelados o frescos. Una vez aislados y evaluados, los núcleos se pueden utilizar para varios análisis "ómicos" posteriores. Haga clic aquí para ver una versión más grande de esta figura.
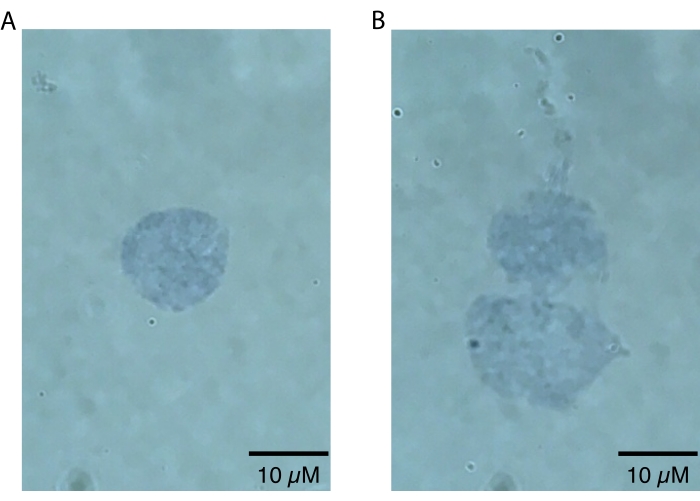
Figura 2: Evaluación de la calidad de los núcleos por microscopía. Los núcleos se mezclaron 1:1 en una solución de azul de tripano al 0,4% y se visualizaron en un microscopio Echo Revolve mediante imágenes de campo claro a 60x. (A) Un ejemplo de un núcleo de alta calidad. El núcleo está presente como un singlete y la membrana nuclear está intacta. (B) Un ejemplo de núcleos de mala calidad. Las membranas nucleares están dañadas y los núcleos se agrupan en un doblete, probablemente debido a la fuga de ADN pegajoso, que se puede ver filtrándose desde la parte superior del núcleo superior. Haga clic aquí para ver una versión más grande de esta figura.
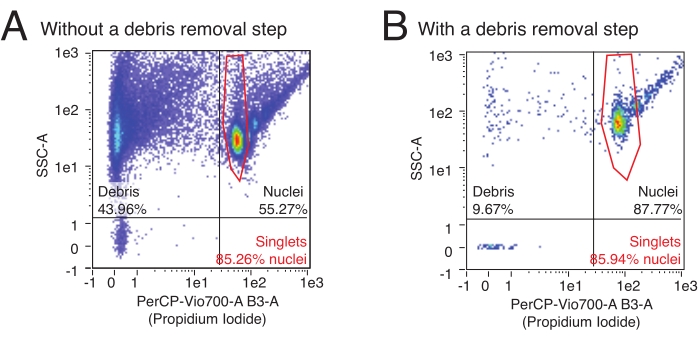
Figura 3: Cuantificación de núcleos y evaluación de la pureza por citometría de flujo. Las muestras de núcleos se tiñeron con una dilución 1:100 de PI y se ejecutaron en un citómetro de flujo. Los núcleos están presentes en formas singlete y multiplete en el cuadrante superior derecho de (A, B) con singletes como la nube más baja de eventos seguidos de dobletes, tripletes, etc. en orden ascendente. Los escombros están marcados por los eventos en los dos cuadrantes más a la izquierda. (A) Un ejemplo de una preparación de núcleos de baja calidad, donde se omitió el paso de eliminación de escombros y los desechos representan el >40% de los eventos. (B) Un ejemplo de una muestra de núcleos de alta calidad, con el paso de eliminación de desechos incluido. En este ejemplo, los desechos representan el <10% de todos los eventos registrados por el citómetro de flujo. Haga clic aquí para ver una versión más grande de esta figura.
| Tipo de muestra (2 cerebros) | Rendimiento promedio (4 preparaciones independientes) |
| Cerebros masculinos de 5 semanas | 3,59 ± 1,76 x 105 |
| Cerebros masculinos de 10 semanas | 6,41 ± 1,33 x 105 |
| Cerebros masculinos de 15 semanas | 14,59 ± 2,05 x 105 |
| Cerebros femeninos de 5 semanas | 2,54 ± 0,75 x 105 |
| Cerebros femeninos de 10 semanas | 4,66 ± 1,29 x 105 |
| Cerebros femeninos de 15 semanas | 7,95 ± 3,51 x 105 |
Tabla 1: Rendimientos promedio esperados de dos cerebros de killi turquesa africano a través del sexo y la edad. Los rendimientos medios expresados como 105 núcleos ± error estándar de la media sobre cuatro preparaciones de núcleos independientes en cada categoría.
Figura complementaria 1: Comparación de la calidad del aislamiento de núcleos utilizando protocolos estándar y nuestro protocolo optimizado en cerebros congelados de killifish. (A) Evaluación de la calidad de los núcleos por microscopía. Los núcleos se mezclaron 1:1 en una solución de azul de tripano al 0,4% y se visualizaron en un microscopio mediante imágenes de campo claro a 60x. (B) Cuantificación de la carga de núcleos y desechos mediante citometría de flujo. Las muestras de núcleos se tiñeron con una dilución 1:100 de PI y se ejecutaron en un citómetro de flujo. (C) Resumen de métricas de evaluación de la calidad por microscopía y citometría de flujo con los diferentes protocolos de referencia. Haga clic aquí para descargar este archivo.
Figura complementaria 2: Representación esquemática de los pasos de remoción de escombros. (A) Esquema de eliminación de escombros en capas antes de la centrifugación. (B) Esquema de eliminación de sobrenadante después de la centrifugación. Haga clic aquí para descargar este archivo.
Discusión
El protocolo presentado aquí se puede utilizar para generar de forma reproducible núcleos de alta calidad a partir de cerebros de killifish. Este protocolo tuvo que ser diseñado específicamente para el cerebro del killifish, ya que los protocolos típicos de aislamiento de núcleos cerebrales basados en mamíferos aplicados a los cerebros de killifish resultaron consistentemente en una mala calidad de los núcleos en nuestras manos. Sospechamos que esto se debe al menor contenido relativo de mielina del cerebro del killi en comparación con sus homólogos mamíferos, que se lisarían y agruparían en respuesta a las duras condiciones requeridas para la lisis de las células cerebrales de los mamíferos. Este protocolo es un avance en los campos de envejecimiento y killifish, ya que facilita la exploración del envejecimiento cerebral a nivel unicelular en un modelo rentable y rentable del envejecimiento cerebral de los vertebrados.
Este protocolo es robusto para muestras frescas o congeladas, aunque se deben considerar las aplicaciones posteriores cuando se usa tejido fresco o congelado. El tejido congelado a menudo es conveniente, ya que se puede recolectar y almacenar durante meses mientras se recolectan las muestras. Dichas muestras se pueden utilizar de forma segura para aplicaciones como snRNA-seq. Sin embargo, la congelación de muestras puede alterar la estructura nuclear y, por lo tanto, la capacidad de medir con precisión el paisaje de la cromatina mediante ATAC-seq21. Por lo tanto, para aplicaciones posteriores como ATAC-seq a granel o snATAC-seq, se recomienda utilizar cerebros recién diseccionados en lugar de cerebros congelados. Además, debido a que todos los pasos después de la homogeneización cerebral se pueden realizar en paralelo, este protocolo es susceptible de ejecutar múltiples muestras en un marco de tiempo razonable, limitando así la degradación del ARN causada por la incubación prolongada en hielo.
Además, es imperativo preparar tampones frescos (en cuestión de horas) de realizar la extracción de núcleos. Descubrimos que los detergentes y BSA deben agregarse a los tampones inmediatamente antes de comenzar el protocolo. Los tampones que contienen solo sales (PBS, NaCl, etc.) pueden fabricarse como concentrados, esterilizarse con filtro (0,22 μm) y almacenarse indefinidamente a temperatura ambiente. Las soluciones madre de BSA pueden prepararse, esterilizarse y almacenarse a 4 °C a los pocos días de la extracción de los núcleos (si se preparan a partir de un polvo), pero siempre deben añadirse a los tampones utilizados en este protocolo inmediatamente antes de realizar el protocolo. Sin embargo, recomendamos preparar las soluciones BSA el día del protocolo. Si utiliza soluciones BSA prefabricadas de un tercero, se recomienda utilizar botellas frescas y sin abrir. El uso de BSA fresco generalmente conduce a un menor contenido de desechos en las preparaciones de núcleos.
Ya sea que se utilicen muestras frescas o congeladas como entrada, es importante evaluar la calidad de los núcleos después del aislamiento de los núcleos. Aunque este protocolo está diseñado específicamente para evitar la sobrelisis, esta es la causa más común de pérdida de calidad de los núcleos. La sobrelisis puede ser el resultado de demasiado tiempo pasado en el tampón de lisis, un manejo demasiado brusco de los núcleos, como un pipeteo excesivo con una punta de pipeta de orificio estándar, o una cantidad excesiva de tiempo entre el aislamiento de los núcleos y las aplicaciones posteriores (>1 h). Los núcleos excesivamente lisados a menudo tendrán periferias nucleares dañadas, que filtran ADN y causan aglutinación (Figura 2B). Esto conducirá a un mayor número de multipletes y contribuirá con ácidos nucleicos de fondo que interferirán con las aplicaciones posteriores, especialmente snRNA-seq. Ambas cualidades pueden evaluarse mediante microscopía después del aislamiento de los núcleos. Si se observa una aglutinación nuclear excesiva, recomendamos tratar de acortar la incubación del paso de lisis para reducir la posibilidad de sobrelisis. Como método alternativo para aumentar los núcleos singletes, se puede utilizar la clasificación celular asistida por fluorescencia (FACS) para enriquecer los singletes aguas abajo de este protocolo. Sin embargo, observamos que, cuando se trabaja con núcleos ya frágiles de tejido congelado, el esfuerzo cortante que se produce durante la clasificación puede conducir a una mayor ruptura nuclear y, por lo tanto, a un aumento del ARN/ADN ambiental. Además, observamos que el tiempo necesario para ejecutar una clasificación de rendimiento de los núcleos del sistema de control de los bienes sobre el terreno cuando se procesan múltiples muestras requeriría que todas las muestras de núcleos permanecieran en hielo durante horas, mientras se clasifican otras muestras. Por lo tanto, el aumento de los tiempos de espera al procesar múltiples muestras en paralelo con el enfoque FACS también podría conducir a una reducción general de la calidad de los núcleos y aumentar el riesgo de degradación del ARN. Por lo tanto, si se desea que el sistema de control de los bienes sobre el terreno reduzca la tasa de dobletes, recomendamos que el contenido de residuos se verifique nuevamente después de la clasificación de rendimiento y que se tenga en cuenta la posible calidad reducida del ARN para las aplicaciones de RNA-seq de una sola célula como una posible advertencia.
Una estimación precisa de los recuentos de núcleos y la proporción de singletes es esencial para casi todas las aplicaciones "ómicas" posteriores y es de suma importancia. Debido a la capacidad de bloquear y contar fácilmente los núcleos por tamaño, la citometría de flujo es el método más preciso para contar núcleos que hemos evaluado. Alternativamente, se pueden cuantificar núcleos utilizando contadores de células como el contador de células automatizado Countess 2 FL de Invitrogen o el contador de células automatizado DeNovix CellDrop. Para tener en cuenta, el contador de células automatizado Countess 2 FL de Invitrogen, y en mucha menor medida el DeNovix, tienden a sobreestimar los recuentos de núcleos al contar los desechos como núcleos, lo que significa que puede ser necesario un control manual del tamaño. Además, el citómetro de flujo permite evaluar fácilmente la pureza de los núcleos. Se puede discernir la proporción relativa de núcleos singlete versus multipletes de una manera cuantitativa que es difícil por microscopía. Esto es vital para snRNA-seq y snATAC-seq, ya que esos protocolos sufrirán un excedente de muestras multiplete, que deben excluirse de los análisis posteriores. Además de los múltiplos, la proporción relativa de "desechos" (núcleos fragmentados, desechos celulares) puede cuantificarse mediante citometría de flujo y debe ser relativamente baja, ya que este material a menudo contiene ácidos nucleicos contaminantes que pueden contribuir con una señal de fondo y corromper los datos "ómicos" de un solo núcleo.
Al igual que con los protocolos de aislamiento de núcleos descritos anteriormente, las proporciones de tipos de células en el tejido original pueden no recapitularse fielmente en la preparación de núcleos19, y por lo tanto deben interpretarse con precaución. A tener en cuenta, como todos los teleósteos, el killi turquesa africano tiene eritrocitos nucleados, que también se espera que estén representados en la preparación de los núcleos. Estos núcleos pueden identificarse en conjuntos de datos snRNA-seq y snATAC-seq por la mayor expresión/accesibilidad de los genes de hemoglobina y pueden excluirse computacionalmente si se desea.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Algunos paneles se generaron con BioRender.com. Este trabajo en nuestro laboratorio fue apoyado por la NIA T32 AG052374 Postdoctoral Training Grant a B.T., una subvención de la Fundación Simons como parte de la Colaboración Simons para la Plasticidad en el Cerebro Envejecido, una subvención piloto de la Fundación Navigage y un premio de la Familia Hanson-Thorell a B.A.B.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.4% Trypan Blue solution | Gibco | 15250061 | |
| 10x PBS | Bioland | PBS01-03 | |
| 15 mL Conicle Centrifuge Tube | VWR | 89039-664 | |
| 2 mL Tissue Grinder | Kimble | 885300-0002 | Dounce Homogenizer |
| 5 mL Polystyrene Round-Bottom Tube | Falcon | 352054 | |
| 5M Sodium Chloride, Molecular Biology Grade | Promega | V4221 | |
| autoMACS Rinsing Solution | Miltenyi Biotec | 130-091-222 | This corresponds to 1x PBS pH 7.2, 2 mM EDTA (used for flow cytometry) |
| Debris Removal Solution | Miltenyi Biotec | 130-109-398 | |
| DNA LoBInd Tube | Eppendorf | 22431021 | |
| Echo Revolve microscope (fitted with 60x objective) | Echo | NA | No catalog number; Used to visually inspect nuclei. |
| Falcon Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352052 | FACS tube |
| FLOWMI Cell Strainers, 40 μM | SP Bel-Art | 136800040 | Referred to as on-tip filters in Protocol |
| Hydrochloric Acid | Sigma-Aldrich | 258146-500 mL | Used to lower TRIS pH from 8.0 to 7.4 |
| Leica EZ4 dissecting scope | Leica | NA | No catalog number |
| MACS BSA stock solution | Miltenyi Biotec | 130091376 | |
| MACS SmartStrainers (70 μM) | Miltenyi Biotec | 130-110-916 | |
| MACSQuant Analyzer 10 Flow Cytometer | Miltenyi Biotec | NA | No catalog number |
| Magnesium Chloride, Hexahydrate, Molecular Biology Grade (Powder) | Millipore | 442611 | |
| Megafuge 16R Centrifuge | ThermoScientific | 75003629 | |
| Micro Cover Glass | VWR | 48393081 | |
| Micro Slides Superfrost Plus | VWR | 48311-703 | |
| Nonidet P-40 Substitute | Roche | (Roche) 11332473001/ Catalog: 983739 P Code: -102368106 (Sigma Aldrich) | |
| NP-40 Surfact-Amps Detergent Solution | ThermoFisher | 85124 | |
| Nuclease-Free HyPure Molecular Biology Grade Water | HyClone | SH30538.02 | |
| NxGen RNase Inhibitor (50,000 U) | Lucigen | 30281-2 | |
| Propidium Iodide solution | MBL | FP00010020 | |
| PureBlu DAPI Nuclear Staining Dye | Biorad | 1351303 | |
| TipOne RPT Pipette Tips (Ultra low retention, filtered) in 10 µL, 20 µL, 200 µL, and 1000 µL sizes | USA Scientific | #1181-3810; #1180-1810; #1180-8810; #1182-1830 | |
| Tricaine-S (MS 222) | Syndel | Tricaine10G | Syndel is an FDA-approved provider for pharmaceutical grade Tricaine |
| TRIS, 1 M, pH 8.0 | VWR | E199-500 mL | |
| Wide Bore Pipet Tips | Axygen | T-1005-WB-C |
Referencias
- Yankner, B. A., Lu, T., Loerch, P. The aging brain. Annual Review of Pathology. 3 (1), 41-66 (2008).
- Hu, C. K., Brunet, A. The African turquoise killifish: A research organism to study vertebrate aging and diapause. Aging Cell. 17 (3), 12757 (2018).
- McKay, A., et al. An automated feeding system for the African killifish reveals effects of dietary restriction on lifespan and allows scalable assessment of associative learning. bioRxiv. , (2021).
- Valenzano, D. R., Terzibasi, E., Cattaneo, A., Domenici, L., Cellerino, A. Temperature affects longevity and age-related locomotor and cognitive decay in the short-lived fish Nothobranchius furzeri. Aging Cell. 5 (3), 275-278 (2006).
- Vanhunsel, S., et al. The killifish visual system as an in vivo model to study brain aging and rejuvenation. NPJ Aging and Mechanisms of Disease. 7 (1), 22 (2021).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), 0209648 (2018).
- Ziffra, R. S., et al. Single-cell epigenomics reveals mechanisms of human cortical development. Nature. 598 (7879), 205-213 (2021).
- Grindberg, R. V., et al. RNA-sequencing from single nuclei. Proceedings of the National Academy of Sciences. 110 (49), 19802-19807 (2013).
- Kalish, B. T., et al. Single-nucleus RNA sequencing of mouse auditory cortex reveals critical period triggers and brakes. Proceedings of the National Academy of Sciences. 117 (21), 11744-11752 (2020).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Gaublomme, J. T., et al. Nuclei multiplexing with barcoded antibodies for single-nucleus genomics. Nature Communications. 10 (1), 2907 (2019).
- Narayanan, A., et al. Nuclei isolation from fresh frozen brain tumors for single-nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments. (162), e61542 (2020).
- Saunders, A., et al. Molecular diversity and specializations among the cells of the adult mouse brain. Cell. 174 (4), 1015-1030 (2018).
- . Isolation of Nuclei for Single Cell RNA Sequencing & Tissues for Single Cell RNA Sequencing Available from: https://support.10xgenomics.com/permalink/1dlB6Z91VqClgUmSC2OM8k (2021)
- Martin, C., et al. Frozen tissue nuclei extraction (for 10xV3 snSEQ) V.2. protocols.io. , (2020).
- Nuclei Isolation from Adult Mouse Brain Tissue for Single Cell RNA Sequencing. 10xGenomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/sample-prep/nuclei-isolation-from-adult-mouse-brain-tissue-for-single-cell-ma-sequencing (2022)
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- What are the best practices for working with nuclei samples for 3' single-cell gene expression. 10xGenomics Available from: https://kb.10xgenomics.com/hc/en-us/articles/360050780051-What-are-the-best-practices-for-working-with-nuclei-samples-for-3-single-cell-gene-expression (2022)
- Rocks, D., et al. Cell type-specific chromatin accessibility analysis in the mouse and human brain. Epigenetics. 17 (2), 202-219 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados