Method Article
In vitro Caracterização de Chaperones de Histonas utilizando Ensaios Analíticos, Pull-Down e Chaperoning
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo descreve uma bateria de métodos que inclui cromatografia analítica de exclusão de tamanho para estudar a oligomerização e estabilidade da histona chaperona, ensaio pull-down para desvendar interações histona-histona-histona, AUC para analisar a estequiometria dos complexos proteicos e ensaio de chaperoning de histonas para caracterizar funcionalmente uma suposta histona chaperone in vitro.
Resumo
As proteínas histonas associam-se ao DNA para formar a cromatina eucariótica. A unidade básica da cromatina é um nucleossomo, composto por um octamero de histonas que consiste em duas cópias das histonas centrais H2A, H2B, H3 e H4, envolvidas pelo DNA. O octamero é composto por duas cópias de um dímero H2A/H2B e uma única cópia de um tetrâmero H3/H4. As histonas do núcleo altamente carregadas são propensas a interações inespecíficas com várias proteínas no citoplasma celular e no núcleo. As chaperonas de histona formam uma classe diversificada de proteínas que transportam histonas do citoplasma para o núcleo e auxiliam sua deposição no DNA, auxiliando assim o processo de montagem do nucleossomo. Algumas histonas acompanhantes são específicas para H2A/H2B ou H3/H4, e algumas funcionam como chaperonas para ambas. Este protocolo descreve como técnicas laboratoriais in vitro , como ensaios pull-down, cromatografia analítica de exclusão de tamanho, ultracentrifugação analítica e ensaio de chaperoning de histonas, podem ser usadas em conjunto para confirmar se uma determinada proteína é funcional como uma chaperona de histonas.
Introdução
Nucleossomos compostos de proteínas de DNA e histonas formam a unidade estrutural da cromatina e regulam vários eventos celulares críticos. Os nucleossomos são dinamicamente reposicionados e remodelados para tornar o DNA acessível a vários processos, como replicação, transcrição e tradução 1,2. As histonas altamente básicas tendem a interagir com proteínas ácidas no meio celular ou sofrem agregação, levando a vários defeitos celulares 3,4,5. Um grupo de proteínas dedicadas denominadas histonas chaperonas auxilia no transporte de histonas do citoplasma para o núcleo e previne eventos aberrantes de agregação histona-DNA 6,7. Fundamentalmente, a maioria das histonas acompanhantes armazena e transfere histonas para o DNA com força iônica fisiológica, auxiliando na formação de nucleossomos 8,9. Algumas histonas têm uma preferência definida pelos oligômeros de histonas H2A/H2B ou H3/H410.
As chaperonas de histona são caracterizadas com base em sua capacidade de montar nucleossomos dependentes ou independentes da síntese de DNA11. Por exemplo, o fator de montagem da cromatina-1 (CAF-1) é dependente, enquanto o regulador de histonas A (HIRA) é independente da síntese de DNA12,13. Da mesma forma, a família nucleoplasmina das histonas chaperonas está envolvida na descondensação da cromatina espermática e na montagem de nucleossomos14. Os membros da família da proteína de montagem de nucleossomos (NAP) facilitam a formação de estruturas semelhantes a nucleossomos in vitro e estão envolvidos no deslocamento de histonas entre o citoplasma e o núcleo15. As nucleoplasminas e as proteínas da família NAP são ambas chaperonas de histonas funcionais, mas não compartilham nenhuma característica estrutural. Essencialmente, nenhuma característica estrutural única permite a classificação de uma proteína como uma histona chaperona16. O uso de ensaios funcionais e biofísicos, juntamente com estudos estruturais, funciona melhor na caracterização de histonas acompanhantes.
Este trabalho descreve métodos bioquímicos e biofísicos para caracterizar uma proteína como uma chaperona de histonas que auxilia a montagem de nucleossomos. Primeiramente, foi realizada cromatografia analítica de exclusão de tamanho para analisar o estado oligomérico e a estabilidade das histonas chaperonas. Em seguida, um ensaio pull-down foi realizado para determinar as forças motrizes e a natureza competitiva das interações histona acompanhante-histona. No entanto, os parâmetros hidrodinâmicos dessas interações não puderam ser calculados com precisão usando cromatografia analítica de exclusão de tamanho devido à forma da proteína e seus complexos que afetam sua migração através da coluna. Para tanto, foi utilizada a ultracentrifugação analítica, que fornece propriedades macromoleculares em solução que incluem o peso molecular preciso, a estequiometria de interação e a forma das moléculas biológicas. Estudos anteriores utilizaram extensivamente o ensaio de chaperoning de histonas in vitro para caracterizar funcionalmente as chaperonas de histonas, como yScS116 17, DmACF18, ScRTT106p19, HsNPM120. O ensaio de chaperoning de histona também foi usado para caracterizar funcionalmente as proteínas como chaperonas de histonas.
Protocolo
1. Cromatografia analítica de exclusão de tamanho para elucidar o estado oligomérico e a estabilidade das chaperonas de histonas
- Análise do estado oligomérico das histonas chaperonas
- Equilibrar uma coluna de cromatografia analítica de exclusão de tamanho (SEC) de 24 mL com 1,2 volume de coluna (CV), ou seja, 28,8 mL de tampão SEC desgaseificado [20 mM de Tris-HCl (pH 7,5), 300 de mM de NaCl e 1 mM de β-mercaptoetanol (β-ME)] a 4 °C (ver Tabela de Materiais).
NOTA: O tipo de coluna, a composição do tampão e o pH do tampão podem ser selecionados com base na proteína de interesse. O volume de injeção da amostra não deve exceder 500 μL para uma coluna de 24 ml. Além disso, a pressão da coluna precisa ser mantida abaixo de 5 MPa. - A partir de uma solução de reserva de proteína de maior concentração, preparar 500 μL de amostra de proteína de 0,5 mg/mL em tampão SEC desgaseificado e injetá-la na coluna pré-equilibrada usando um loop de injeção de 500 μL. Permitir que a cromatografia prossiga a um caudal isocrático de 0,2-0,3 ml/min com o tampão SEC a 4 °C.
- Monitorize o perfil de eluição da proteína medindo a absorvância a um comprimento de onda de 280 nm. Ao lidar com proteínas sem resíduos aromáticos, meça a absorvância a 214 nm.
- Utilizar o volume de eluição da proteína para calcular o seu peso molecular aproximado em kDa utilizando a curva de calibração padrão21.
NOTA: A curva de calibração é preparada plotando o volume de retenção de proteínas de peso molecular conhecidas contra o log de seus respectivos pesos moleculares (log Mr), eluídas usando a mesma coluna.
- Equilibrar uma coluna de cromatografia analítica de exclusão de tamanho (SEC) de 24 mL com 1,2 volume de coluna (CV), ou seja, 28,8 mL de tampão SEC desgaseificado [20 mM de Tris-HCl (pH 7,5), 300 de mM de NaCl e 1 mM de β-mercaptoetanol (β-ME)] a 4 °C (ver Tabela de Materiais).
- Análise da estabilidade térmica das chaperonas de histonas
- Tomar 500 μL de 0,5 mg/ml da amostra proteica preparada em tampão SEC desgaseificado (o mesmo utilizado no ponto 1.1.1) em tubos de microcentrífuga individuais e aquecer cada tubo a uma temperatura específica que varia entre 20 °C e 90 °C (20 °C, 40 °C, 60 °C e 90 °C) durante 10 minutos em banho-maria.
- Subsequentemente, centrifugar as amostras tratadas termicamente a 16.200 x g durante 10 min a 4 °C, recolher o sobrenadante com uma micropipeta e injectar cada amostra individualmente utilizando uma alça de injecção de 500 μL na coluna analítica, pré-equilibrada com o tampão SEC a 4 °C.
- Permitir que a cromatografia prossiga a um caudal isocrático de 0,2-0,3 ml/min com o tampão SEC a 4 °C.
- Observe a posição e a altura dos picos de eluição e procure o aparecimento de picos adicionais para as diferentes amostras.
- Análise da estabilidade química das chaperonas histonas
- Para examinar a estabilidade salina das chaperonas de histonas, incubar 500 μL de amostra de proteína de 0,5 mg/mL preparada em tampão Tris [20 mM de Tris-HCl (pH 7,5) e 1 mM de β-ME] suplementado com concentrações crescentes de NaCl (300 mM, 600 mM, 1 M, 1,5 M e 2 M) em tubos de microcentrífuga separados por 30 min a 4 °C. Centrifugar as amostras a 16,200 x g durante 10 min a 4 °C e reter o sobrenadante.
- Em seguida, carregar as amostras de proteína em diferentes concentrações de NaCl individualmente, usando um loop de injeção de 500 μL na coluna analítica pré-equilibrada com 1,2 CV (28,8 mL) do respectivo tampão contendo concentrações crescentes de NaCl a 4 °C.
- Permitir que a cromatografia prossiga a um caudal isocrático de 0,2-0,3 ml/min com 1 CV (24 ml) do respectivo tampão a 4 °C.
- Observe a posição e a altura dos picos de eluição e procure o aparecimento de picos adicionais para as diferentes amostras.
- Da mesma forma, para análise de estabilidade da ureia, incubar 500 μL de amostra de proteína de 0,5 mg/mL preparada em tampão Tris [20 mM de Tris-HCl (pH 7,5) e 1 mM de β-ME] suplementado com concentrações crescentes de ureia (1 M, 2 M, 3 M, 4 M e 5 M) em tubos microcentrífugos separados por 16 h à temperatura ambiente. Centrifugar as amostras a 16.200 x g durante 10 min à temperatura ambiente e reter o sobrenadante.
- Em seguida, carregue as amostras de proteína tratadas com ureia individualmente usando um loop de injeção de 500 μL na coluna analítica pré-equilibrada com 1,2 CV (28,8 mL) do tampão correspondente contendo diferentes concentrações de ureia à temperatura ambiente.
- Permitir que a cromatografia prossiga a um caudal isocrático de 0,2-0,3 ml/min com 1 CV (24 ml) do respectivo tampão à temperatura ambiente.
CUIDADO: Não realize os experimentos com tampão contendo ureia a uma temperatura mais baixa, pois a ureia tende a cristalizar e danificar a coluna. - Observe a posição e a altura dos picos de eluição e procure o aparecimento de picos adicionais para as diferentes amostras.
2. Ensaios pull-down baseados em gradiente de sal para entender o tipo de interações que contribuem para a formação complexa entre oligômeros de histonas e uma chaperona de histonas
- Para cada reação de ensaio pull-down, pipetar 40 μL de resina Ni-NTA em uma coluna de rotação e lavar com água estéril de dupla destilação. Posteriormente, equilibre a resina com 100 CV (4 mL) de tampão de equilíbrio [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 10 mM de imidazol, 10 μg/mL de BSA e 1 mM de β-ME] (ver Tabela de Materiais).
NOTA: O pull-down também pode ser realizado em um tubo de microcentrífuga de 1,5 mL. - Preparar a amostra misturando 5 μM da chaperona de histona marcada com His com 20 μM de dímero H2A/H2B ou tetrâmero H3/H4 no tampão de equilíbrio. Incubar a amostra no gelo durante 1 h.
NOTA: O dímero H2A/H2B e o tetrâmero H3/H4 são preparados a partir de histonas humanas recombinantes21, e a integridade dos oligômeros é confirmada com base nas massas moleculares estimadas por ultracentrifugação analítica (AUC). Os mesmos oligômeros de histonas foram usados para todos os experimentos mencionados abaixo. - Carregar as amostras em colunas de rotação pré-equilibradas separadas com resina Ni-NTA a partir da etapa 2.1, cada uma rotulada para uma determinada concentração de sal, e manter as colunas durante 30 minutos a 4 °C. Centrifugar as colunas a 1000 x g durante 1 min.
- Em seguida, lave as colunas com 100 CV (4 mL) de tampão de lavagem [20 mM de Tris-HCl (pH 7,5), 50 mM de imidazol, 0,2% de Tween-20 e 1 mM de β-ME] contendo diferentes concentrações de sal (ou seja, 300 mM, 500 mM, 600 mM, 700 mM, 800 mM, 900 mM e 1 M de NaCl). Lavar cada coluna com um tampão com uma concentração de sal específica.
- Após a etapa de lavagem salina, eluir a proteína das diferentes colunas usando 100 μL de tampão de eluição [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 300 mM de imidazol e 1 mM de β-ME].
- Posteriormente, sujeitar as amostras eluídas a 18% de SDS-PAGE22 e visualizar o gel após coloração com Coomassie Brilliant Blue R250 (ver Tabela de Materiais). Alternativamente, você pode carregar diretamente a resina no gel SDS-PAGE em vez de eluír a proteína ligada da resina Ni-NTA.
NOTA: As composições do tampão de equilíbrio, lavagem e eluição e o pH podem ser modificados dependendo da proteína de interesse.
3. Ensaio pull-down competitivo para identificar a preferência de uma chaperona de histonas por H2A/H2B ou H3/H4
- Preparar a coluna de rotação conforme descrito na etapa 2.1
- Incubar 5 μM da chaperona de histonas com 20 μM de dímero H2A/H2B em 300 μL de tampão de equilíbrio (preparado na etapa 2.1) por 30 min sobre gelo.
NOTA: A proporção de oligômero de histona para chaperona de histona na reação pode ser escolhida com base em dados conhecidos de estequiometria de ligação; use cinco vezes mais histona em excesso se não houver informações disponíveis. - Centrifugar o complexo histona chaperona-H2A/H2B a 16,200 x g durante 5 min a 4 °C para remover qualquer precipitado. Em seguida, carregar a amostra na coluna de rotação pré-equilibrada com tampão de equilíbrio (preparado na etapa 2.1) e incubar durante 30 minutos a 4 °C.
- Lavar a coluna com 100 CV (4 mL) de tampão de lavagem [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 50 mM de imidazol, 0,2% de Tween-20 e 1 mM de β-ME] para remover o excesso de dímero H2A/H2B. Em seguida, misture o complexo histona chaperona-H2A/H2B com 20-60 μM de tetrâmero H3/H4 e incube por 30 min no gelo.
- Lavar novamente a coluna com 100 CV (4 ml) de tampão de lavagem (preparado na etapa 3.4) para remover qualquer tetrâmero H3/H4 não ligado e eluir a amostra utilizando tampão de eluição (preparado na etapa 2.5). Submeta as amostras eluídas a 18% de SDS-PAGE e visualize após a coloração com Coomassie Brilliant Blue R250.
NOTA: O ensaio pode ser invertido em que, primeiro, o tetrâmero H3/H4 pode ser misturado com a chaperona, o complexo pode se ligar a contas de Ni-NTA e o complexo então ser incubado com concentrações variáveis de dímero H2A/H2B.
4. Experimentos analíticos de ultracentrifugação - velocidade de sedimentação (AUC-SV) para analisar a estequiometria de ligação entre chaperonas de histonas e histonas
- Preparação da amostra para AUC
- Dialise o dímero H2A/H2B de histona reconstituído, o tetrâmero H3/H4 e a chaperona de histona separadamente através de um tubo de diálise de corte de 7 kDa 23, contra um tampão de diálise [20 mM de Tris (pH 7,5), 300 mM de NaCl e 1 mM de β-ME] (ver Tabela de Materiais). Para minimizar o erro de fundo devido à incompatibilidade do tampão, realize diálise extensivamente contra o tampão de diálise, de preferência três vezes durante um período de 24 horas.
NOTA: A OD 280 inicial das amostras de proteína deve ter um valor de duas a três vezes maior para alcançar uma OD280 final de 0,3-0,5. Isso é feito essencialmente para anular os efeitos da diluição. - Purificar o dímero H2A/H2B, o tetrâmero H3/H4 e a chaperona de histonas individualmente com o tampão de diálise, utilizando cromatografia analítica de exclusão de tamanho (conforme mencionado na etapa 1). Salve o buffer da corrida para preparar diluições adicionais mais tarde e para usar como referência na célula AUC.
- Dialise o dímero H2A/H2B de histona reconstituído, o tetrâmero H3/H4 e a chaperona de histona separadamente através de um tubo de diálise de corte de 7 kDa 23, contra um tampão de diálise [20 mM de Tris (pH 7,5), 300 mM de NaCl e 1 mM de β-ME] (ver Tabela de Materiais). Para minimizar o erro de fundo devido à incompatibilidade do tampão, realize diálise extensivamente contra o tampão de diálise, de preferência três vezes durante um período de 24 horas.
- Carregamento de amostra para AUC
- Misture as proteínas purificadas em um volume final de 450 μL usando tampão de diálise da etapa 4.1.1 para atingir um OD280 de 0,3-0,6. Misture a chaperona de histonas com o dímero H2A/H2B ou o tetrâmero H3/H4 para formação complexa em tubos de reação separados. Incubar as misturas de proteínas por 2-3 h.
NOTA: Alternativamente, os dados de sedimentação podem ser adquiridos com um sistema de varredura óptica de interferência na ultracentrífuga analítica. Separadamente, para misturar proteínas purificadas, fixar a concentração de histona chaperona e incubá-la com concentrações crescentes dos oligômeros de histonas para obter a estequiometria exata. - Montar a célula com uma peça central de setor duplo e janelas de quartzo para o experimento AUC-SV usando um detector de absorbância da ultracentrífuga analítica, conforme descrito anteriormente em detalhes24.
- Encher 400 μL da solução da amostra e 420 μL de tampão de diálise nos dois sectores (sectores da amostra e de referência, respectivamente) da célula.
NOTA: Um volume maior de tampão é usado no setor de referência para manter o menisco de referência acima do menisco da amostra. No entanto, ao usar um sistema de interferência óptica, preencha os dois setores com o mesmo volume. - Pese e equilibre com precisão as células e carregue-as em um rotor de titânio de quatro lugares (consulte Tabela de materiais). Alinhe as células usando as marcas fornecidas na parte inferior das células e do rotor. Carregue o rotor na centrífuga, feche a tampa e deixe desenvolver um vácuo até que a pressão caia para menos de 15 mícrons de Hg e a temperatura do rotor se estabilize para 20 °C (geralmente leva 2-2,5 h).
NOTA: Os parâmetros de operação da AUC incluem temperatura experimental, velocidade do rotor, o intervalo entre as varreduras e o número de varreduras a serem coletadas. No caso de experimentos de SV, o intervalo de varredura é dado de acordo com a massa molecular da proteína; proteínas menores requerem intervalos de tempo maiores entre os exames. A velocidade do rotor também é definida de acordo com a massa molecular esperada da proteína, e o experimento é conduzido a 20 °C. Os dados de absorbância são monitorados a 280 nm. - Para obter a estequiometria exata, mantenha a concentração de histona chaperona constante e incube com concentrações crescentes de oligômeros de histonas para atingir a saturação.
- Misture as proteínas purificadas em um volume final de 450 μL usando tampão de diálise da etapa 4.1.1 para atingir um OD280 de 0,3-0,6. Misture a chaperona de histonas com o dímero H2A/H2B ou o tetrâmero H3/H4 para formação complexa em tubos de reação separados. Incubar as misturas de proteínas por 2-3 h.
- Análise de dados AUC
- Realizar a análise dos dados conforme descrito anteriormente25. Resumidamente, calcule a densidade e a viscosidade dos componentes do buffer usando o programa SEDNTERP26 (consulte Tabela de Materiais). Da mesma forma, calcule o volume específico parcial da proteína com base em sua composição de aminoácidos, também usando SEDNTERP.
- Carregue os dados da máquina AUC no programa SEDFIT27 e defina o menisco (linha vermelha), a parte inferior da célula (linha azul) e os limites da análise de dados (linhas verdes). Escolha a distribuição C(s) contínua como modelo.
- Em seguida, defina a resolução máxima até 100; definir coeficiente(s) de sedimentação, s min: 0 e s max: 10-15; definir a razão de atrito para 1,2 inicialmente e optar por flutuar para derivar a razão dos dados; definir o nível de confiança (razão F; que determina a magnitude da regularização) para 0,68 para a regularização 1 sigma; definir valores parciais de volume específico, densidade do tampão e viscosidade do buffer obtidos do SEDNTERP.
- Pressione RUN para permitir que o software resolva a equação de Lamm27. Ajuste os parâmetros se houver uma incompatibilidade significativa de dados. Depois de ajustar os parâmetros, pressione FIT para refinar todos os parâmetros. Avalie a qualidade do ajuste com base no valor de desvio quadrado médio da raiz (RMSD), que deve ser inferior a 0,01 unidades de sinal.
- Estime as massas moleculares dos picos escolhendo a opção: mostrar o pico "Mw em c(s)" na função de exibição da barra de ferramentas principal, que fornecerá informações sobre os 's' da molécula/complexo.
5. Ensaio de superenrolamento plasmídico para confirmar a função de chaperoning de histonas
- Reação de montagem de nucleossomos
- Misture 2 μM de tetrâmero H3/H4 e 4 μM de dímero H2A/H2B com concentrações crescentes de chaperona de histona (1-6 μM) em um tampão de montagem [20 mM de Tris HCl (pH 7,5), 1 mM de TDT, 1 mM de MgCl2, 0,1 mg/mL de BSA e 100 mM de NaCl] até um volume final de 50 μL. Incubar a mistura a 4 °C por 30 min.
- Simultaneamente, numa reacção separada, pré-tratar 500 ng do plasmídeo pUC19 superenrolado negativamente com 1 μg de enzima topoisomerase I (ver Tabela de Materiais) no tampão de montagem num volume final de 50 μL e incubar a 30 °C durante 30 min.
NOTA: A topoisomerase I relaxa o DNA plasmidial de fita dupla superenrolado gerando um nick de fita simples. Poderia ser utilizada uma enzima topoisomerase I de origem eucariótica, como a topoisomerase I do gérmen de trigo comercialmente disponível ou a Drosophila melanogaster topoisomerase I expressa de forma recombinante. - Em seguida, combinar o tetrâmero H3/H4, o dímero H2A/H2B, a mistura de chaperona de histonas (a partir do passo 5.1.1), a mistura de reação de DNA plasmídico relaxado (a partir do passo 5.1.2) e incubar ainda mais a 30 °C durante 90 min.
NOTA: Configure duas reações de controle para o ensaio; um com a histona chaperona e o DNA plasmidial relaxado (mas não as histonas) e o outro com os oligômeros de histonas e o DNA plasmidial relaxado (mas não a chaperona de histonas). - Parar a reação de montagem adicionando 100 μL de tampão de parada 2x (40 mM de EDTA, 2% de SDS e 0,4 mg/mL de proteinase K) e incubar a 37 °C por 30 min.
NOTA: O tampão Stop desproteiniza o DNA plasmidial por desnaturação e proteólise de histonas ligadas.
- Extração de fenol-clorofórmio e precipitação de etanol
- Adicionar um volume igual de fenol saturado de Tris no tubo que contém a mistura de reacção da etapa 5.1.4 e misturar bem, seguido de centrifugação a 16,200 x g durante 10 min à temperatura ambiente.
- Recolha suavemente a fase aquosa superior com o ADN plasmídico com uma micropipeta e misture com um volume igual de clorofórmio. Vórtice da mistura e centrifugar a 16.200 x g durante 10 min à temperatura ambiente.
NOTA: O álcool isoamílico pode ser incluído nesta etapa para evitar uma interface difusa entre as fases aquosa e orgânica. - Em seguida, coletar a fase aquosa superior, adicionar 1/10 volume de acetato de sódio 3 M (pH 5,5) e 2,5 volumes de etanol gelado (ver Tabela de Materiais). Misture bem a solução invertendo o tubo 3-4 vezes e mantenha a mistura num congelador de -20 °C durante 30 min para uma precipitação completa do ADN plasmídico.
- Centrifugar a amostra da etapa 5.2.3 a 16200 x g durante 10 minutos e rejeitar suavemente o sobrenadante. Mantenha os tubos abertos à temperatura ambiente até que até mesmo vestígios de etanol evaporem, deixando o DNA plasmidial precipitado no tubo.
- Realize a eletroforese em gel de agarose para observar o efeito de superenrolamento do plasmídeo.
- Dissolver o ADN plasmídico precipitado a partir da etapa 5.2.4 em água estéril de dupla destilação.
- Resolver as amostras com um gel de agarose a 1% em 1x tampão Tris-acetato-EDTA (TAE) (40 mM de Tris, 20 mM de ácido acético e 1 mM de EDTA) (ver Tabela de Materiais).
- Coloração do gel com concentração de 0,2-0,5 μg/mL de brometo de etídio e observar sob UV para visualizar as bandas de DNA no gel.
Resultados
O domínio da nucleoplasmina N-terminal recombinante da proteína FKBP53 de Arabidopsis thaliana foi submetido à SEC analítica. O volume de pico de eluição foi plotado contra a curva padrão para identificar seu estado oligomérico. Os resultados analíticos da SEC revelaram que o domínio existe como pentâmero em solução, com massa molecular aproximada de 58 kDa (Figura 1A,B). Além disso, o domínio nucleoplasmina foi analisado quanto à estabilidade térmica e química em conjunto com a SEC analítica. A amostra de nucleoplasmina submetida a tratamento térmico até 90 °C não apresentou alteração aparente no volume de eluição e na altura de pico em relação às amostras mantidas a 20 °C, sugerindo que o domínio é altamente termoestável (Figura 1C). Da mesma forma, o domínio nucleoplasmina apresentou estabilidade salina de até 2 M de NaCl (Figura 1D) e estabilidade de ureia de até 4 M (Figura 1E). No entanto, o pentâmero nucleoplasmina começou a dissociar-se em concentrações mais elevadas de ureia.
Um ensaio pull-down foi realizado para determinar o tipo de interações que contribuem para a formação complexa entre a histona chaperona (domínio nucleoplasmina de AtFKBP53) e os oligômeros de histonas dímero H2A/H2B e tetrâmero H3/H4 usando uma lavagem salina gradiente. A interação do domínio nucleoplasmina com o dímero H2A/H2B manteve-se estável até uma concentração de sal de 0,4 M NaCl (Figura 2A). Em comparação, a associação com H3/H4 foi razoavelmente estável até 0,7 M NaCl (Figura 2B). A capacidade dos complexos chaperona-histona de suportar alta concentração de sal sugere o papel das interações hidrofóbicas na estabilização dos complexos. O complexo de chaperona com H3/H4 estável mesmo em altas concentrações de sal sugere um papel predominante das interações hidrofóbicas na formação do complexo. A menor estabilidade do complexo H2A/H2B-chaperona em altas concentrações de sais revela um papel significativo para as interações eletrostáticas na formação complexa. Em outro experimento, o ensaio pull-down foi usado para examinar se a chaperona prefere o dímero H2A/H2B ou o tetrâmero H3/H4. Os resultados revelaram que a chaperona liga-se ao dímero H2A/H2B e ao tetrâmero H3/H4 simultaneamente e independentemente da ordem em que são adicionados à chaperona (Figura 2C,D). Isso indicou que a chaperona possui locais separados para sua interação com os oligômeros de histonas.
Experimentos de AUC-SV (Figura 3) foram realizados para estudar a estequiometria de interação entre oligômeros de histonas e chaperonas. A análise dos dados da AUC-SV forneceu um valor de coeficiente de sedimentação de 5,40 S para o domínio nucleoplasmina AtFKBP53 em complexo com H2A/H2B que correspondeu a uma massa molecular de 104 kDa. O complexo do domínio nucleoplasmina com H3/H4 apresentou um valor de coeficiente de sedimentação de 7,35 S, correspondendo a 129 kDa. A massa molecular estimada dos complexos revela que a nucleoplasmina pentamérica forma complexo com dímero H2A/H2B e tetrâmero H3/H4 em uma estequiometria 1:1.
É essencial mostrar que a proteína pode depositar oligômeros de histonas no DNA para confirmar que é uma chaperona de histonas. Para tanto, adotou-se um ensaio de superenrolamento plasmídico (Figura 4). O plasmídeo circular relaxado foi incubado com os oligômeros de histonas H2A/H2B e H3/H4 com as histonas de plantas recombinantes da família NAP - AtNRP1 e AtNRP228. A presença da chaperona aumentou a quantidade de plasmídeo superenrolado, sugerindo que ele poderia depositar histonas no DNA para formar nucleossomos, causando superenrolamento do DNA.

Figura 1: Estado oligomérico e estabilidade do domínio nucleoplasmina de AtFBP53. (A) Perfil analítico de cromatografia de exclusão de tamanho do domínio nucleoplasmina AtFKBP53. (B) Curva de calibração obtida utilizando proteínas globulares de massa molecular conhecida. Os pontos azuis representam a massa molecular das proteínas conhecidas, enquanto o ponto vermelho representa o domínio da nucleoplasmina AtFKBP53. (440 kDa - ferritina, 158 kDa-aldolase, 75 kDa-con albumina, 44 kDa-ovalbumina, 6,5 kDa-aprotinina). (C) Cromatograma analítico de exclusão de tamanho de 500 μL de 0,5 mg/mL de domínio nucleoplasmina AtFKBP53 submetido a tratamento térmico em diferentes temperaturas: 20 °C (verde), 40 °C (laranja), 60 °C (preto), 90 °C (azul claro). (D) Cromatograma analítico de exclusão de tamanho de 500 μL de 0,5 mg/mL de domínio nucleoplasmina AtFKBP53 em tampões contendo diferentes concentrações de NaCl: 0,3 M (roxo), 0,6 M (vermelho), 1,0 M (azul claro), 1,5 M (verde), 2,0 M (preto). (E) Cromatograma analítico de exclusão de tamanho do domínio nucleoplasmina AtFKBP53 em tampões com diferentes concentrações de ureia: 0 M (controle; azul claro), 1,0 M (rosa), 2,0 M (preto), 3,0 M (azul escuro), 4,0 M (verde), 5,0 M (marrom). O pentâmero de nucleoplasmina apresenta alta estabilidade a condições de estresse térmico e químico. A figura é adaptada da Referência21. Por favor, clique aqui para ver uma versão maior desta figura.
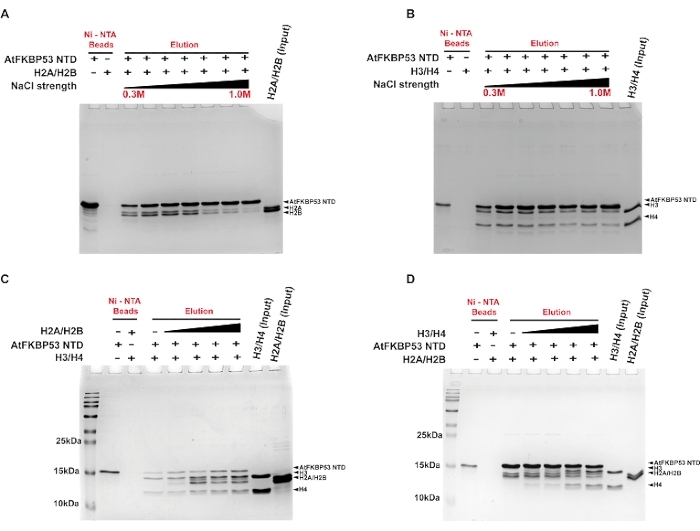
Figura 2: Ensaios pull-down para a interação do domínio nucleoplasmina de AtFKBP53 com oligômeros de histonas. Imagens SDS-PAGE de 18% das frações de eluição dos ensaios são apresentadas aqui. Ensaio pull-down para ( A ) 20 μM de dímero H2A/H2B e (B) tetrâmero H3/H4 de 20 μM com domínio de nucleoplasmina 5 μM AtFKBP53 em concentrações crescentes de NaCl na faixa de 0,3 M, 0,5 M, 0,6 M, 0,7 M, 0,8 M, 0,9 M e 1,0 M. 5 μM AtFKBP53 FKBD foi usado como controle negativo aqui. Para o experimento de ligação competitiva, ( C ) uma mistura de 5 μM de domínio de nucleoplasmina AtFKBP53 e tetrâmero de 20 μM H3/H4 incubada com uma faixa de 20-60 μM de dímero H2A/H2B e (D) uma mistura de 5 μM de domínio de nucleoplasmina AtFKBP53 e dímero de 20 μM H2A/H2B incubado com uma faixa de tetrâmero H3/H4 de 20-60 μM. O rótulo AtFKBP53 corresponde ao domínio nucleoplasmina do AtFKBP53. As frações de eluição mostram ligação simultânea de ambos os oligômeros de histonas à nucleoplasmina. A figura é adaptada da Referência21. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 3: Experimento de ultracentrifugação analítica - velocidade de sedimentação (AUC-SV) de oligômeros de histonas, domínio nucleoplasmina de AtFKBP53 e seus complexos. A distribuição de distância AUC vs. gráfico de coeficiente de sedimentação (S). Os valores do(s) coeficiente(s) de sedimentação obtido(s) e as massas moleculares também são fornecidos. O rótulo AtFKBP53 corresponde ao domínio nucleoplasmina do AtFKBP53. As massas moleculares estimadas revelam uma estequiometria de 1:1 para o domínio da nucleoplasmina AtFKBP53 com os oligômeros de histonas dímero H2A/H2B e tetrâmero H3/H4. 450 μL de todas as amostras de proteína com DO280 de 0,3-0,5 foram utilizados para os experimentos de AUC-SV. A figura é adaptada da Referência21. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 4: Ensaio de superenrolamento de plasmídeos. Ensaio de superenrolamento de plasmídeos para as chaperonas de histonas AtNRP1 e AtNRP2. 500 ng de DNA plasmidial pUC19 foram pré-tratados com 1 μg de topoisomerase I para o experimento. 4 μM AtNRP1, 4 μM AtNRP2 e uma mistura de 4 μM de dímero H2A/H2B e tetrâmero de 2 μM H3/H4 foram como controle que não mostra atividade de superenrolamento quando incubado com o DNA pUC19 pré-tratado. As faixas com uma mistura de 4 μM H2A/H2B de dímero e 2 μM H3/H4 de tetrâmero e 4 μM cada de AtNRP1 e AtNRP2 mostram a formação de DNA superenrolado após a incubação com o DNA pUC19 pré-tratado. Por favor, clique aqui para ver uma versão maior desta figura.
Discussão
Este trabalho demonstra e valida um conjunto abrangente de protocolos para a caracterização bioquímica e biofísica de uma suposta histona acompanhante. Aqui, proteínas da família NAP recombinantemente expressas e purificadas, AtNRP1 e AtNRP2, e o domínio N-terminal nucleoplasmina de AtFKBP53 foram utilizados para demonstrar os protocolos. O mesmo conjunto de experimentos poderia muito bem ser usado para delinear os atributos funcionais de acompanhantes de histonas previamente descaracterizadas de qualquer organismo.
A primeira parte da seção do protocolo envolve a investigação do estado oligomérico e da estabilidade de uma histona acompanhante. Vários relatos indicam que as histonas acompanhantes exibem uma diversidade considerável em seu estado oligomérico. Por exemplo, o CAF-1 humano existe como um monômero29. Os membros da família NAP existem como dímero ou tetrâmero 29,30,31. As nucleoplasminas revelam estados oligoméricos pentaméricos e frequentemente decaméricos32,33. Um experimento analítico SEC pode determinar o estado oligomérico de uma chaperona de histona, e experimentos AUC-SV podem confirmar o mesmo. Sabe-se que várias das histonas são altamente estáveis sob várias condições de estresse térmico e químico33,34. As características de estabilidade térmica e química das chaperonas de histonas também podem ser exploradas em conjunto com a SEC analítica. Além disso, a espectroscopia de dicroísmo circular poderia ser efetivamente usada para análise aprofundada das mudanças na estrutura secundária da chaperona quando submetida a temperaturas crescentes ou concentrações mais altas de um agente químico.
A segunda parte da seção do protocolo abrange ensaios pull-down que poderiam examinar as interações fundamentais que auxiliam a associação de oligômeros de histonas com a chaperona usando uma abordagem de gradiente de sal e um ensaio pull-down competitivo para identificar a preferência do oligômero de histona de um acompanhante. Se o complexo desmoronar com um ligeiro aumento na concentração de sal, isso sugeriria uma grande contribuição das interações eletrostáticas na estabilização do complexo. Um complexo intacto em alto teor de sal sugeriria um papel significativo para a hidrofobicidade na estabilização do complexo35. O ensaio pull-down competitivo poderia ser facilmente empregado para determinar a especificidade ou preferência de uma chaperona de histonas a uma classe específica de oligômeros de histonas. Com base em sua preferência por oligômeros de histonas, as chaperonas de histonas podem ser classificadas em três categorias, como chaperonas H2A/H2B, chaperonas H3/H4 e chaperonas H2A/H2B-H3/H410,36. Além disso, se necessário, a calorimetria de titulação isotérmica (ITC) poderia ser usada para entender a especificidade do oligômero de histonas de uma dada chaperona e as características termodinâmicas de suas interações.
A terceira parte da seção do protocolo abrange a investigação da estequiometria de interação entre uma histona chaperona e os oligômeros de histonas. Em geral, as diferentes famílias de histonas chaperonas diferem consideravelmente pela estequiometria de sua associação com H2A/H2B e/e H3/H4 21,28,37,38. O experimento AUC-SV auxilia na obtenção do(s) coeficiente(s) de sedimentação e massa molecular de uma proteína ou de seu complexo, o que se torna muito útil para estimar com precisão a estequiometria na formação complexa. Alternativamente, o ITC também pode ser usado para examinar a estequiometria.
A quarta parte da seção do protocolo se concentra na investigação da função de montagem de nucleossomos de histonas acompanhantes. As chaperonas de histona desempenham um papel crucial na montagem de nucleossomos, que regula processos celulares vitais, como replicação, transcrição e reparo de DNA39. O ensaio de superenrolamento de plasmídeos que é tipicamente empregado para a avaliação in vitro da atividade de chaperona de histonas de chaperonas de histonas é elaborado nesta seção.
Pode-se notar que nem todas as histonas acompanhantes estão totalmente estruturadas. Poucos são conhecidos por terem regiões intrinsecamente desordenadas40,41. Portanto, ensaios de estabilidade térmica e química podem não ser adequados para tais proteínas. Além disso, as histonas chaperonas de diferentes organismos têm diferentes estados oligoméricos e habilidades diferenciais para se ligar a histonas. Portanto, este protocolo pode ser um bom ponto de partida, mas implicaria modificações conforme necessário.
Divulgações
Não foi declarado qualquer conflito de interesses.
Agradecimentos
As subvenções extramuros a Dileep Vasudevan do Conselho de Pesquisa em Ciência e Engenharia, Governo da Índia [CRG/2018/000695/PS] e do Departamento de Biotecnologia, Ministério da Ciência e Tecnologia, Governo da Índia [BT/INF/22/SP33046/2019], bem como o apoio intramural do Instituto de Ciências da Vida, Bhubaneswar são muito reconhecidos. Agradecemos à Sra. Sudeshna Sen e à Sra. Annapurna Sahoo por sua ajuda com a preparação de histonas. As discussões com nossos colegas Dr. Chinmayee Mohapatra, Sr. Manas Kumar Jagdev e Dr. Shaikh Nausad Hossain também são reconhecidas.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acrylamide | MP Biomedicals | 814326 | |
| Agarose | MP Biomedicals | 193983 | |
| AKTA Pure 25M FPLC | Cytiva | 29018226 | Instrument for protein purification |
| Ammonium persulfate (APS) | Sigma | A3678 | |
| An-60Ti rotor | Beckman Coulter | 361964 | Rotor for analytical ultracentrifugation |
| Bovine serum albumin (BSA) | Sigma | A7030 | |
| Chloroform | Sigma | C2432 | |
| Coomassie brilliant blue R 250 | Sigma | 1.15444 | |
| Dialysis tubing (7 kDa cut-off) | Thermo Fisher | 68700 | For dialysing protein samples |
| Dithiothreitol (DTT) | MP Biomedicals | 100597 | |
| DNA Loading Dye | New England Biolabs | B7025S | |
| EDTA disodium salt | MP Biomedicals | 194822 | |
| Electronic balance | Shimadzu | ATX224R | |
| Ethanol | Sigma | E7023 | |
| Ethidium bromide (EtBr) | Sigma | E8751 | |
| Gel Doc System | Bio-Rad | 12009077 | For imaging gels after staining |
| Horizontal gel electrophoresis apparatus | Bio-Rad | 1704405 | Instrument for agarose gel electrophoresis |
| Hydrochloric acid (HCl) | Sigma | 320331 | |
| Imidazole | MP Biomedicals | 102033 | |
| Magnesium chloride (MgCl2) | Sigma | M8266 | |
| Micropipettes | Eppendorf | Z683779 | For pipetting of micro-volumes |
| Mini-PROTEAN electrophoresis system | Bio-Rad | 1658000 | Instrument for SDS-PAGE |
| N,N-methylene-bis-acrylamide | MP Biomedicals | 800172 | |
| Nano drop | Thermo Fisher | ND-2000 | For measurement of protein and DNA concentrations |
| Ni-NTA agarose | Invitrogen | R901-15 | Resin beads for pull-down assay |
| Optima AUC analytical ultracentrifuge | Beckman Coulter | B86437 | Instrument for analytical ultracentrifugation |
| pH Meter | Mettler Toledo | MT30130863 | |
| Phenol | Sigma | P4557 | |
| Plasmid isolation kit | Qiagen | 27104 | |
| Proteinase K | Sigma-Aldrich | 1.07393 | |
| pUC19 | Thermo Fisher | SD0061 | Plasmid for supercoiling assay |
| Refrigerated high-speed centrifuge | Thermo Fisher | 75002402 | |
| SDS-PAGE protein marker | Bio-Rad | 1610317 | |
| SEDFIT | Free software program for analytical ultracentrifugation data analysis | ||
| SEDNTERP | Free software program to estimate viscosity and density of buffer and partial specific volume of a protein | ||
| SigmaPrep Spin Columns | Sigma | SC1000 | For pull-down assay |
| Sodium acetate | Sigma | S2889 | |
| Sodium chloride (NaCl) | Merck | S9888 | |
| Sodium dodecyl sulfate (SDS) | MP Biomedicals | 102918 | |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | Column for analytical size-exclusion chromatography |
| Superdex 75 Increase 10/300 GL | Cytiva | 29148721 | Column for analytical size-exclusion chromatography |
| TEMED | Sigma | 1.10732 | |
| Topoisomerase I | Inspiralis | WGT102 | Enzyme used in plasmid supercoiling assay |
| Tris base | Merck | T1503 | |
| Tween-20 | Sigma | P1379 | |
| Urea | MP Biomedicals | 191450 | |
| Water bath | Nüve | NB 5 | For heat treatment of protein samples |
| β-mercaptoethanol (β-ME) | Sigma | M6250 |
Referências
- Hübner, M. R., Eckersley-Maslin, M. A., Spector, D. L. Chromatin organization and transcriptional regulation. Current Opinion in Genetics and Development. 23 (2), 89-95 (2013).
- Lai, W. K. M., Pugh, B. F. Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nature Reviews Molecular Cell Biology. 18 (9), 548-562 (2017).
- Kim, U. J., Han, M., Kayne, P., Grunstein, M. Effects of histone H4 depletion on the cell cycle and transcription of Saccharomyces cerevisiae. EMBO Journal. 7 (7), 2211-2219 (1988).
- Prado, F., Aguilera, A. Partial depletion of histone H4 increases homologous recombination-mediated genetic instability. Molecular and Cellular Biology. 25 (4), 1526-1536 (2005).
- Meeks-Wagner, D., Hartwell, L. H. Normal stoichiometry of histone dimer sets is necessary for high fidelity of mitotic chromosome transmission. Cell. 44 (1), 43-52 (1986).
- Groth, A., et al. Human Asf1 regulates the flow of S phase histones during replicational stress. Molecular Cell. 17 (2), 301-311 (2005).
- Laskey, R. A., Honda, B. M., Mills, A. D., Finch, J. T. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA. Nature. 275 (5679), 416-420 (1978).
- Das, C., Tyler, J. K., Churchill, M. E. A. The histone shuffle: histone chaperones in an energetic dance. Trends in Biochemical Sciences. 35 (9), 476-489 (2010).
- Akey, C. W., Luger, K. Histone chaperones and nucleosome assembly. Current Opinion in Structural Biology. 13 (1), 6-14 (2003).
- De Koning, L., Corpet, A., Haber, J. E., Almouzni, G. Histone chaperones: An escort network regulating histone traffic. Nature Structural and Molecular Biology. 14 (11), 997-1007 (2007).
- Eitoku, M., Sato, L., Senda, T., Horikoshi, M. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly. Cellular and Molecular Life Sciences. 65 (3), 414-444 (2008).
- Quivy, J. P., Grandi, P., Almouzni, G. Dimerization of the largest subunit of chromatin assembly factor 1: importance in vitro and during Xenopus early development. EMBO Journal. 20 (8), 2015-2027 (2001).
- Ray-Gallet, D., et al. HIRA is critical for a nucleosome assembly pathway independent of DNA synthesis. Molecular Cell. 9 (5), 1091-1100 (2002).
- Frehlick, L. J., Eirín-López, J. M., Ausió, J. New insights into the nucleophosmin/nucleoplasmin family of nuclear chaperones. Bioessays. 29 (1), 49-59 (2007).
- Ito, T., Bulger, M., Kobayashi, R., Kadonaga, J. T. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays. Molecular and Cellular Biology. 16 (6), 3112-3124 (1996).
- Elsässer, S. J., D'Arcy, S. Towards a mechanism for histone chaperones. Biochimica et Biophysica Acta. 1819 (3-4), 211-221 (2013).
- Rodríguez-Campos, A., Koop, R., Faraudo, S., Beato, M. Transcriptionally competent chromatin assembled with exogenous histones in a yeast whole cell extract. Nucleic Acids Research. 32 (13), 111(2004).
- Levenstein, M. E., Kadonaga, J. T. Biochemical analysis of chromatin containing recombinant Drosophila core histones. Journal of Biological Chemistry. 277 (10), 8749-8754 (2002).
- Huang, S., et al. Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing. Proceedings of the National Academy of Sciences of the United States of America. 102 (38), 13410-13415 (2005).
- Swaminathan, V., Kishore, A. H., Febitha, K. K., Kundu, T. K. Human histone chaperone nucleophosmin enhances acetylation-dependent chromatin transcription. Molecular and Cellular Biology. 25 (17), 7534-7545 (2005).
- Singh, A. K., Datta, A., Jobichen, C., Luan, S., Vasudevan, D. AtFKBP53: A chimeric histone chaperone with functional nucleoplasmin and PPIase domains. Nucleic Acids Research. 48 (3), 1531-1550 (2020).
- Scofield, B. T. K. H. Protein Electrophoresis. , (2012).
- Andrew, S. M., Titus, J. A., Zumstein, L. Dialysis and concentration of protein solutions. Current Protocols in Toxicology, Appendix 3. , 1-5 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. Journal of Visualized Experiments. (33), e1530(2009).
- Padavannil, A., Brautigam, C. A., Chook, Y. M. Molecular size analysis of recombinant importin-histone complexes using analytical ultracentrifugation. Bio-protocol. 10 (10), 3625(2019).
- Zhao, H., Brautigam, C. A., Ghirlando, R., Schuck, P. Overview of current methods in sedimentation velocity and sedimentation equilibrium analytical ultracentrifugation. Current Protocols in Protein Science. , Chapter 20, Unit20.12 (2013).
- Schuck, P. Size-distribution analysis of macromolecules by sedimentation velocity ultracentrifugation and Lamm equation modelling. Biophysical Journal. 78 (3), 1606-1619 (2000).
- Kumar, A., Kumar Singh, A., Chandrakant Bob de, R., Vasudevan, D. Structural characterization of Arabidopsis thaliana NAP1-related protein 2 (AtNRP2) and comparison with its homolog AtNRP1. Molecules. 24 (12), 2258(2019).
- Liu, W. H., Roemer, S. C., Port, A. M., Churchill, M. E. A. CAF-1-induced oligomerization of histones H3/H4 and mutually exclusive interactions with Asf1 guide H3/H4 transitions among histone chaperones and DNA. Nucleic Acids Research. 45 (16), 9809(2017).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Newman, E. R., et al. Large multimeric assemblies of nucleosome assembly protein and histones revealed by small-angle X-ray scattering and electron microscopy. Journal of Biological Chemistry. 287 (32), 26657-26665 (2012).
- Edlich-Muth, C., et al. The pentameric nucleoplasmin fold is present in Drosophila FKBP39 and a large number of chromatin-related proteins. Journal of Molecular Biology. 427 (10), 1949-1963 (2015).
- Franco, A., et al. Structural insights into the ability of nucleoplasmin to assemble and chaperone histone octamers for DNA deposition. Scientific Reports. 9 (1), 9487(2019).
- Xiao, H., Jackson, V., Lei, M. The FK506-binding protein, Fpr4, is an acidic histone chaperone. FEBS Letters. 580 (18), 4357-4364 (2006).
- Graziano, G. Role of hydrophobic effect in the salt-induced dimerization of bovine beta-lactoglobulin at pH 3. Biopolymers. 91 (12), 1182-1188 (2009).
- Burgess, R. J., Zhang, Z. Histone chaperones in nucleosome assembly and human disease. Nature Structural and Molecular Biology. 20 (1), 14-22 (2013).
- Donham, D. C., Scorgie, J. K., Churchill, M. E. The activity of the histone chaperone yeast Asf1 in the assembly and disassembly of histone H3/H4-DNA complexes. Nucleic Acids Research. 39 (13), 5449-5458 (2011).
- Avvakumov, N., Nourani, A., Côté, J. Histone chaperones: Modulators of chromatin marks. Molecular Cell. 41 (5), 502-514 (2011).
- Ransom, M., Dennehey, B. K., Tyler, J. K. Chaperoning histones during DNA replication and repair. Cell. 140 (2), 183-195 (2010).
- Chu, X., et al. Importance of electrostatic interactions in the association of intrinsically disordered histone chaperone Chz1 and histone H2A.Z-H2B. PLoS Computational Biology. 8 (7), 1002608(2012).
- Heidarsson, P. O., et al. Disordered proteins enable histone chaperoning on the nucleosome. bioRxiv. , (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados