Method Article
In vitro Charakterisierung von Histon-Chaperonen mittels analytischer, Pull-Down- und Chaperoning-Assays
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine Reihe von Methoden, die eine analytische Größenausschlusschromatographie zur Untersuchung der Histon-Chaperon-Oligomerisierung und -stabilität, den Pull-Down-Assay zur Entschlüsselung von Histon-Chaperon-Histon-Interaktionen, die AUC zur Analyse der Stöchiometrie der Proteinkomplexe und den Histon-Chaperoning-Assay zur funktionellen Charakterisierung eines mutmaßlichen Histon-Chaperons in vitro umfassen.
Zusammenfassung
Histonproteine assoziieren mit DNA, um das eukaryotische Chromatin zu bilden. Die Grundeinheit von Chromatin ist ein Nukleosom, das aus einem Histon-Oktamer besteht, das aus zwei Kopien der Kernhistone H2A, H2B, H3 und H4 besteht, die von der DNA umwickelt sind. Das Oktamer besteht aus zwei Kopien eines H2A/H2B-Dimers und einer einzigen Kopie eines H3/H4-Tetramers. Die hochgeladenen Kernhistonen neigen zu unspezifischen Wechselwirkungen mit mehreren Proteinen im zellulären Zytoplasma und im Zellkern. Histon-Chaperone bilden eine vielfältige Klasse von Proteinen, die Histone aus dem Zytoplasma in den Zellkern transportieren und deren Ablagerung auf der DNA unterstützen, wodurch der Nukleosomenzusammenbauprozess unterstützt wird. Einige Histonchaperone sind spezifisch für H2A / H2B oder H3 / H4, und einige fungieren als Chaperone für beide. Dieses Protokoll beschreibt, wie In-vitro-Labortechniken wie Pull-Down-Assays, analytische Größenausschlusschromatographie, analytische Ultrazentrifugation und Histon-Chaperoning-Assay zusammen verwendet werden können, um zu bestätigen, ob ein bestimmtes Protein als Histon-Chaperon funktionsfähig ist.
Einleitung
Nukleosomen, die aus DNA und Histonproteinen bestehen, bilden die strukturelle Einheit des Chromatins und regulieren mehrere kritische zelluläre Ereignisse. Nukleosomen werden dynamisch neu positioniert und umgestaltet, um DNA für verschiedene Prozesse wie Replikation, Transkription und Translation zugänglich zu machen 1,2. Sehr basische Histone neigen entweder dazu, mit sauren Proteinen im zellulären Milieu zu interagieren oder eine Aggregation zu durchlaufen, was zu verschiedenen zellulären Defekten führt 3,4,5. Eine Gruppe von dedizierten Proteinen, die Histon-Chaperone genannt werden, unterstützen den Transport von Histonen aus dem Zytoplasma zum Zellkern und verhindern aberrante Histon-DNA-Aggregationsereignisse 6,7. Grundsätzlich speichern und übertragen die meisten Histon-Chaperone Histone mit physiologischer Ionenstärke auf DNA, wodurch die Bildung von Nukleosomen unterstütztwird 8,9. Einige Histon-Chaperone haben eine deutliche Vorliebe für die Histon-Oligomere H2A/H2B oder H3/H410.
Histon-Chaperone werden aufgrund ihrer Fähigkeit charakterisiert, Nukleosomen abhängig oder unabhängig von der DNA-Synthesezu bilden 11. Zum Beispiel ist der Chromatin-Assemblierungsfaktor-1 (CAF-1) abhängig, während der Histonregulator A (HIRA) unabhängig von der DNA-Synthese12,13 ist. In ähnlicher Weise ist die Nukleoplasmin-Familie der Histon-Chaperone an der Spermienchromatin-Dekondensation und der Nukleosomenmontage beteiligt14. Die Mitglieder der Nukleosomen-Assemblierungsprotein-Familie (NAP) erleichtern die Bildung nukleosomenähnlicher Strukturen in vitro und sind am Pendeln von Histonen zwischen Zytoplasma und Kern15 beteiligt. Nukleoplasmine und Proteine der NAP-Familie sind beide funktionelle Histon-Chaperone, haben aber keine strukturellen Merkmale. Im Wesentlichen erlaubt kein einzelnes strukturelles Merkmal die Klassifizierung eines Proteins als Histon-Chaperon16. Die Verwendung von funktionellen und biophysikalischen Assays zusammen mit Strukturstudien funktioniert am besten bei der Charakterisierung von Histon-Chaperonen.
Diese Arbeit beschreibt biochemische und biophysikalische Methoden zur Charakterisierung eines Proteins als Histon-Chaperon, das die Nukleosomenbildung unterstützt. Zunächst wurde eine analytische Größenausschlusschromatographie durchgeführt, um den oligomeren Status und die Stabilität von Histon-Chaperonen zu analysieren. Als nächstes wurde ein Pull-Down-Assay durchgeführt, um die treibenden Kräfte und den Wettbewerbscharakter von Histon-Chaperon-Histon-Interaktionen zu bestimmen. Die hydrodynamischen Parameter dieser Wechselwirkungen konnten jedoch nicht genau mit der analytischen Größenausschlusschromatographie berechnet werden, da die Form des Proteins und seine Komplexe ihre Migration durch die Säule beeinflussen. Daher wurde eine analytische Ultrazentrifugation verwendet, die in Lösung makromolekulare Eigenschaften liefert, die ein genaues Molekulargewicht, die Stöchiometrie der Wechselwirkung und die Form der biologischen Moleküle umfassen. Frühere Studien haben ausgiebig In-vitro-Histon-Chaperoning-Assays verwendet, um Histon-Chaperone wie yScS116 17, DmACF 18, ScRTT106p 19, HsNPM120 funktionell zu charakterisieren. Der Histon-Chaperoning-Assay wurde auch verwendet, um die Proteine funktionell als Histon-Chaperone zu charakterisieren.
Protokoll
1. Analytische Größenausschlusschromatographie zur Aufklärung des oligomeren Status und der Stabilität von Histon-Chaperonen
- Analyse des oligomeren Status von Histon-Chaperonen
- Eine analytische 24-ml-Säule zur Größenausschlusschromatographie (SEC) mit einem Volumen von 1,2 Säulen (CV), d. h. 28,8 ml entgastem SEC-Puffer [20 mM Tris-HCl (pH 7,5), 300 mM NaCl und 1 mM β-Mercaptoethanol (β-ME)] bei 4 °C (siehe Materialtabelle).
HINWEIS: Säulentyp, Pufferzusammensetzung und Puffer-pH-Wert können basierend auf dem interessierenden Protein ausgewählt werden. Das Probeninjektionsvolumen sollte 500 μL für eine 24-ml-Säule nicht überschreiten. Außerdem muss der Säulendruck unter 5 MPa gehalten werden. - Aus einer höher konzentrierten Proteinstammlösung werden 500 μL 0,5 mg/ml Proteinprobe in entgastem SEC-Puffer hergestellt und unter Verwendung einer 500-μL-Injektionsschleife in die voräquilibrierte Säule injiziert. Lassen Sie den Chromatographielauf mit einer isokratischen Flussrate von 0,2-0,3 ml/min mit dem SEC-Puffer bei 4 °C ablaufen.
- Überwachen Sie das Elutionsprofil des Proteins, indem Sie die Absorption bei einer Wellenlänge von 280 nm messen. Bei Proteinen ohne aromatische Rückstände ist die Absorption bei 214 nm zu messen.
- Verwenden Sie das Elutionsvolumen des Proteins, um sein ungefähres Molekulargewicht in kDa unter Verwendung der Standardkalibrierkurve21 zu berechnen.
HINWEIS: Die Kalibrierkurve wird erstellt, indem das Retentionsvolumen bekannter Molekulargewichtsproteine gegen den Logarithmus ihrer jeweiligen Molekulargewichte (log Mr) aufgetragen wird, die unter Verwendung derselben Spalte eluiert werden.
- Eine analytische 24-ml-Säule zur Größenausschlusschromatographie (SEC) mit einem Volumen von 1,2 Säulen (CV), d. h. 28,8 ml entgastem SEC-Puffer [20 mM Tris-HCl (pH 7,5), 300 mM NaCl und 1 mM β-Mercaptoethanol (β-ME)] bei 4 °C (siehe Materialtabelle).
- Analyse der thermischen Stabilität der Histon-Chaperone
- 500 μl 0,5 mg/ml der in entgastem SEC-Puffer (wie in Abschnitt 1.1.1 verwendet) hergestellten Proteinprobe werden in einzelne Mikrozentrifugenröhrchen genommen und jedes Röhrchen auf eine bestimmte Temperatur zwischen 20 °C und 90 °C (20 °C, 40 °C, 60 °C und 90 °C) für 10 min in einem Wasserbad erhitzt.
- Anschließend zentrifugieren Sie die wärmebehandelten Proben bei 16.200 x g für 10 min bei 4 °C, sammeln den Überstand mit einer Mikropipette und injizieren jede Probe einzeln mit einer 500 μL Injektionsschleife in die analytische Säule, die mit dem SEC-Puffer bei 4 °C voräquilibriert wird.
- Lassen Sie den Chromatographielauf mit einer isokratischen Flussrate von 0,2-0,3 ml/min mit dem SEC-Puffer bei 4 °C ablaufen.
- Beobachten Sie die Position und Höhe der Elutionspeaks und achten Sie auf das Auftreten zusätzlicher Peaks für die verschiedenen Proben.
- Analyse der chemischen Stabilität der Histon-Chaperone
- Um die Salzstabilität von Histon-Chaperonen zu untersuchen, inkubieren Sie 500 μL einer 0,5 mg/ml Proteinprobe, die in einem Tris-Puffer [20 mM Tris-HCl (pH 7,5) und 1 mM β-ME] hergestellt wurde, ergänzt mit steigenden Konzentrationen von NaCl (300 mM, 600 mM, 1 M, 1,5 M und 2 M) in separaten Mikrozentrifugenröhrchen für 30 min bei 4 °C. Zentrifugieren Sie die Proben bei 16.200 x g für 10 min bei 4 °C und behalten Sie den Überstand bei.
- Anschließend werden die Proteinproben in verschiedenen NaCl-Konzentrationen einzeln geladen, wobei eine 500 μL-Injektionsschleife in die Analysesäule verwendet wird, die mit 1,2 CV (28,8 ml) des jeweiligen Puffers mit steigenden NaCl-Konzentrationen bei 4 °C voräquilibriert ist.
- Lassen Sie den Chromatographielauf mit einer isokratischen Flussrate von 0,2-0,3 ml/min mit 1 CV (24 ml) des jeweiligen Puffers bei 4 °C ablaufen.
- Beobachten Sie die Position und Höhe der Elutionspeaks und achten Sie auf das Auftreten zusätzlicher Peaks für die verschiedenen Proben.
- In ähnlicher Weise werden für die Harnstoffstabilitätsanalyse 500 μL einer 0,5 mg/ml Proteinprobe inkubiert, die in einem Tris-Puffer [20 mM Tris-HCl (pH 7,5) und 1 mM β-ME], ergänzt mit steigenden Harnstoffkonzentrationen (1 M, 2 M, 3 M, 4 M und 5 M) in separaten Mikrozentrifugenröhrchen für 16 h bei Raumtemperatur hergestellt wurde. Zentrifugieren Sie die Proben bei 16.200 x g für 10 min bei Raumtemperatur und behalten Sie den Überstand bei.
- Anschließend werden die harnstoffbehandelten Proteinproben einzeln mit einer 500-μL-Injektionsschleife in die Analysesäule geladen, die mit 1,2 CV (28,8 ml) des entsprechenden Puffers mit unterschiedlichen Harnstoffkonzentrationen bei Raumtemperatur voräquilibriert ist.
- Lassen Sie den Chromatographielauf mit einer isokratischen Flussrate von 0,2-0,3 ml/min mit 1 CV (24 ml) des jeweiligen Puffers bei Raumtemperatur ablaufen.
VORSICHT: Führen Sie die Experimente nicht mit einem harnstoffhaltigen Puffer bei einer niedrigeren Temperatur durch, da Harnstoff dazu neigt, zu kristallisieren und die Säule zu beschädigen. - Beobachten Sie die Position und Höhe der Elutionspeaks und achten Sie auf das Auftreten zusätzlicher Peaks für die verschiedenen Proben.
2. Salzgradienten-basierte Pulldown-Assays zum Verständnis der Art von Interaktionen, die zur komplexen Bildung zwischen Histon-Oligomeren und einem Histon-Chaperon beitragen
- Für jede Reaktion des Pulldown-Assays 40 μL Ni-NTA-Harz in eine Schleuderkolonne pipettieren und mit sterilem doppelt destilliertem Wasser waschen. Anschließend wird das Harz mit 100 CV (4 ml) Gleichgewichtspuffer [20 mM Tris-HCl (pH 7,5), 300 mM NaCl, 10 mM Imidazol, 10 μg/ml BSA und 1 mM β-ME] ausgeglichen (siehe Materialtabelle).
HINWEIS: Der Pull-Down kann auch in einem 1,5-ml-Mikrozentrifugenröhrchen durchgeführt werden. - Die Probe wird vorbereitet, indem 5 μM des His-markierten Histon-Chaperons entweder mit 20 μM Histon H2A/H2B-Dimer oder H3/H4-Tetramer im Gleichgewichtspuffer gemischt werden. Die Probe 1 h auf Eis inkubieren.
HINWEIS: H2A/H2B-Dimer und H3/H4-Tetramer werden aus rekombinanten menschlichen Histonen21 hergestellt, und die Integrität der Oligomere wird anhand der geschätzten Molekülmassen durch analytische Ultrazentrifugation (AUC) bestätigt. Die gleichen Histon-Oligomere wurden für alle unten genannten Experimente verwendet. - Laden Sie die Proben in separate voräquilibrierte Spinsäulen mit Ni-NTA-Harz aus Schritt 2.1, die jeweils für eine bestimmte Salzkonzentration gekennzeichnet sind, und halten Sie die Säulen 30 Minuten lang bei 4 °C. Zentrifugieren Sie die Säulen bei 1000 x g für 1 min.
- Als nächstes waschen Sie die Säulen mit 100 CV (4 ml) Waschpuffer [20 mM Tris-HCl (pH 7,5), 50 mM Imidazol, 0,2% Tween-20 und 1 mM β-ME] mit verschiedenen Salzkonzentrationen (d. h. 300 mM, 500 mM, 600 mM, 700 mM, 800 mM, 900 mM und 1 M NaCl). Waschen Sie jede Säule mit einem Puffer mit einer bestimmten Salzkonzentration.
- Nach dem Salzwaschschritt eluieren Sie das Protein aus den verschiedenen Säulen mit 100 μL Elutionspuffer [20 mM Tris-HCl (pH 7,5), 300 mM NaCl, 300 mM Imidazol und 1 mM β-ME].
- Anschließend die eluierten Proben 18% SDS-PAGE22 unterziehen und das Gel nach Färbung mit Coomassie Brilliant Blue R250 visualisieren (siehe Materialtabelle). Alternativ können Sie das Harz direkt auf das SDS-PAGE-Gel laden, anstatt das gebundene Protein aus dem Ni-NTA-Harz zu eluieren.
HINWEIS: Gleichgewichts-, Wasch- und Elutionspufferzusammensetzungen und der pH-Wert können je nach interessierendem Protein modifiziert werden.
3. Konkurrenzfähiger Pulldown-Assay zur Identifizierung der Präferenz eines Histon-Chaperons für H2A/H2B oder H3/H4
- Vorbereiten der Spin-Spalte wie in Schritt 2.1 beschrieben
- 5 μM des Histon-Chaperons mit 20 μM H2A/H2B-Dimer in 300 μL Gleichgewichtspuffer (hergestellt in Schritt 2.1) für 30 min auf Eis inkubieren.
HINWEIS: Das Verhältnis von Histon-Oligomer zu Histon-Chaperon in der Reaktion kann auf der Grundlage bekannter Bindungsstöchiometriedaten gewählt werden; Verwenden Sie fünffaches überschüssiges Histon, wenn keine Informationen verfügbar sind. - Zentrifugieren Sie den Histon-Chaperon-H2A/H2B-Komplex bei 16.200 x g für 5 min bei 4 °C, um den Niederschlag zu entfernen. Als nächstes wird die Probe auf die mit Gleichgewichtspuffer (in Schritt 2.1 vorbereitet) voräquilibrierte Spinsäule geladen und 30 min bei 4 °C inkubiert.
- Waschen Sie die Säule mit 100 CV (4 ml) Waschpuffer [20 mM Tris-HCl (pH 7,5), 300 mM NaCl, 50 mM Imidazol, 0,2% Tween-20 und 1 mM β-ME], um überschüssiges H2A/H2B-Dimer zu entfernen. Als nächstes mischen Sie den Histon-Chaperon-H2A/H2B-Komplex mit 20-60 μM H3/H4-Tetramer und inkubieren 30 min auf Eis.
- Waschen Sie die Säule erneut mit 100 CV (4 ml) Waschpuffer (vorbereitet in Schritt 3.4), um ungebundenes H3/H4-Tetramer zu entfernen, und eluieren Sie die Probe mit Elutionspuffer (vorbereitet in Schritt 2.5). Die eluierten Proben 18% SDS-PAGE unterziehen und nach der Färbung mit Coomassie Brilliant Blue R250 visualisieren.
HINWEIS: Der Assay könnte umgekehrt werden, wobei zuerst H3 / H4-Tetramer mit dem Chaperon gemischt werden kann, der Komplex an Ni-NTA-Perlen binden darf und der Komplex dann mit unterschiedlichen Konzentrationen von H2A / H2B-Dimer inkubiert werden kann.
4. Analytische Ultrazentrifugation - Sedimentationsgeschwindigkeit (AUC-SV) Experimente zur Analyse der Bindungsstöchiometrie zwischen Histon-Chaperonen und Histonen
- Probenvorbereitung für AUC
- Dialysieren Sie das rekonstituierte Histon H2A/H2B-Dimer, H3/H4-Tetramer und das Histon-Chaperon separat durch einen 7 kDa Cut-off-Dialyseschlauch 23 gegen einen Dialysepuffer [20 mM Tris (pH 7,5), 300 mM NaCl und 1 mM β-ME] (siehe Materialtabelle). Um Hintergrundfehler aufgrund von Pufferkonflikten zu minimieren, führen Sie die Dialyse umfassend gegen den Dialysepuffer durch, vorzugsweise dreimal über einen Zeitraum von 24 Stunden.
HINWEIS: Der anfängliche OD 280 der Proteinproben sollte einen zwei- bis dreifach höheren Wert haben, um einen endgültigen OD280 von 0,3-0,5 zu erreichen. Dies geschieht im Wesentlichen, um die Auswirkungen der Verdünnung aufzuheben. - Reinigen Sie H2A/H2B-Dimer, H3/H4-Tetramer und das Histon-Chaperon einzeln mit dem Dialysepuffer unter Verwendung der analytischen Größenausschlusschromatographie (wie in Schritt 1 erwähnt). Speichern Sie den Puffer aus dem Durchlauf, um später weitere Verdünnungen vorzubereiten und als Referenz in der AUC-Zelle zu verwenden.
- Dialysieren Sie das rekonstituierte Histon H2A/H2B-Dimer, H3/H4-Tetramer und das Histon-Chaperon separat durch einen 7 kDa Cut-off-Dialyseschlauch 23 gegen einen Dialysepuffer [20 mM Tris (pH 7,5), 300 mM NaCl und 1 mM β-ME] (siehe Materialtabelle). Um Hintergrundfehler aufgrund von Pufferkonflikten zu minimieren, führen Sie die Dialyse umfassend gegen den Dialysepuffer durch, vorzugsweise dreimal über einen Zeitraum von 24 Stunden.
- Probenbeladung für AUC
- Mischen Sie die gereinigten Proteine in einem Endvolumen von 450 μL unter Verwendung eines Dialysepuffers aus Schritt 4.1.1, um einen OD280 von 0,3-0,6 zu erreichen. Mischen Sie das Histon-Chaperon mit H2A / H2B-Dimer oder H3 / H4-Tetramer für die Komplexbildung in separaten Reaktionsröhrchen. Inkubieren Sie die Proteinmischungen für 2-3 h.
HINWEIS: Alternativ können Sedimentationsdaten mit einem interferenzoptischen Scansystem in der analytischen Ultrazentrifuge erfasst werden. Separat, um gereinigte Proteine zu mischen, fixieren Sie die Histon-Chaperon-Konzentration und inkubieren Sie sie mit steigenden Konzentrationen der Histon-Oligomere, um die genaue Stöchiometrie zu erhalten. - Die Zelle wird mit einem Doppelsektor-Herzstück und Quarzfenstern für das AUC-SV-Experiment unter Verwendung eines Absorptionsdetektors der analytischen Ultrazentrifuge zusammengesetzt, wie zuvor im Detailbeschrieben 24.
- Füllen Sie 400 μL der Probenlösung und 420 μL Dialysepuffer in die beiden Sektoren (Proben- bzw. Referenzsektor) der Zelle.
HINWEIS: Im Referenzsektor wird ein größeres Puffervolumen verwendet, um den Referenzmeniskus über dem Meniskus der Probe zu halten. Wenn Sie jedoch ein optisches Interferenzsystem verwenden, füllen Sie die beiden Sektoren mit gleichem Volumen. - Wiegen und balancieren Sie die Zellen genau aus und laden Sie sie in einen viersitzigen Titanrotor (siehe Materialtabelle). Richten Sie die Zellen anhand der Markierungen an der Unterseite der Zellen und am Rotor aus. Laden Sie den Rotor in die Zentrifuge, schließen Sie den Deckel und lassen Sie ein Vakuum entwickeln, bis der Druck auf weniger als 15 Mikrometer Hg fällt und sich die Rotortemperatur auf 20 ° C stabilisiert (dauert normalerweise 2-2,5 h).
HINWEIS: Zu den AUC-Betriebsparametern gehören die experimentelle Temperatur, die Rotordrehzahl, das Intervall zwischen den Scans und die Anzahl der zu erfassenden Scans. Bei SV-Experimenten wird das Scanintervall entsprechend der Molekülmasse des Proteins angegeben; Kleinere Proteine benötigen größere Zeitintervalle zwischen den Scans. Die Rotordrehzahl wird ebenfalls entsprechend der erwarteten Molekülmasse des Proteins eingestellt und das Experiment bei 20 °C durchgeführt. Die Absorptionsdaten werden bei 280 nm überwacht. - Um die genaue Stöchiometrie zu erhalten, halten Sie die Histon-Chaperon-Konzentration konstant und inkubieren Sie mit steigenden Konzentrationen von Histon-Oligomeren, um eine Sättigung zu erreichen.
- Mischen Sie die gereinigten Proteine in einem Endvolumen von 450 μL unter Verwendung eines Dialysepuffers aus Schritt 4.1.1, um einen OD280 von 0,3-0,6 zu erreichen. Mischen Sie das Histon-Chaperon mit H2A / H2B-Dimer oder H3 / H4-Tetramer für die Komplexbildung in separaten Reaktionsröhrchen. Inkubieren Sie die Proteinmischungen für 2-3 h.
- AUC-Datenanalyse
- Führen Sie die Datenanalyse wie zuvor beschriebendurch 25. Berechnen Sie kurz die Dichte und Viskosität der Pufferkomponenten mit dem Programm SEDNTERP26 (siehe Materialtabelle). In ähnlicher Weise berechnen Sie das partielle spezifische Volumen des Proteins basierend auf seiner Aminosäurezusammensetzung, ebenfalls mit SEDNTERP.
- Laden Sie die Daten vom AUC-Gerät in das Programm SEDFIT27 und definieren Sie den Meniskus (rote Linie), den Zellenboden (blaue Linie) und die Datenanalysegrenzen (grüne Linien). Wählen Sie die kontinuierliche C(s)-Verteilung als Modell.
- Als nächstes stellen Sie die maximale Auflösung auf 100 ein. Sedimentationskoeffizient (s), s min: 0 und s max: 10-15; Setzen Sie das Reibungsverhältnis zunächst auf 1,2 und entscheiden Sie sich für Floating, um das Verhältnis aus den Daten abzuleiten. Konfidenzniveau (F-Verhältnis; bestimmt die Größe der Regularisierung) auf 0,68 für die 1-Sigma-Regularisierung einstellen; partielle spezifische Volumen-, Pufferdichte- und Pufferviskositätswerte einstellen, wie sie von SEDNTERP erhalten werden.
- Drücken Sie RUN, damit die Software die Lamm-Gleichung27 lösen kann. Passen Sie die Parameter an, wenn die Daten signifikant nicht übereinstimmen. Nachdem Sie die Parameter angepasst haben, drücken Sie FIT, um alle Parameter zu verfeinern. Bewerten Sie die Qualität der Anpassung basierend auf dem RMSD-Wert (Root-Mean Square Deviation), der weniger als 0,01 Signaleinheiten betragen sollte.
- Schätzen Sie die Molekülmassen der Peaks, indem Sie die Option Peak "Mw in c(s)" in der Anzeigefunktion der Hauptsymbolleiste anzeigen, die Informationen über das "s" des Moleküls / Komplexes liefert.
5. Plasmid-Supercoiling-Assay zur Bestätigung der Histon-Chaperoning-Funktion
- Nukleosomen-Assemblierungsreaktion
- 2 μM H3/H4-Tetramer und 4 μM H2A/H2B-Dimer mit steigenden Konzentrationen des Histon-Chaperons (1-6 μM) in einem Montagepuffer [20 mM Tris HCl (pH 7,5), 1 mM DTT, 1 mMMgCl2, 0,1 mg/ml BSA und 100 mM NaCl] zu einem Endvolumen von 50 μL mischen. Die Mischung bei 4 °C 30 min inkubieren.
- Gleichzeitig werden in einer separaten Reaktion 500 ng des negativ supercoiled pUC19-Plasmids mit 1 μg Topoisomerase I-Enzym (siehe Materialtabelle) im Montagepuffer in einem Endvolumen von 50 μL vorbehandelt und bei 30 °C für 30 min inkubiert.
HINWEIS: Topoisomerase I entspannt die supercoiled doppelsträngige Plasmid-DNA, indem sie einen einzelsträngigen Nick erzeugt. Ein Topoisomerase-I-Enzym eukaryotischen Ursprungs, wie die kommerziell erhältliche Weizenkeimtopoisomerase I oder die rekombinant exprimierte Drosophila melanogaster Topoisomerase I, könnte verwendet werden. - Als nächstes wird das H3/H4-Tetramer, H2A/H2B-Dimer, Histon-Chaperon-Gemisch (ab Schritt 5.1.1), das entspannte Plasmid-DNA-Reaktionsgemisch (aus Schritt 5.1.2) kombiniert und bei 30 °C 90 min weiter inkubiert.
HINWEIS: Richten Sie zwei Kontrollreaktionen für den Assay ein; eine mit dem Histon-Chaperon und der entspannten Plasmid-DNA (aber nicht die Histone) und die andere mit den Histon-Oligomeren und der entspannten Plasmid-DNA (aber nicht mit dem Histon-Chaperon). - Stoppen Sie die Montagereaktion durch Zugabe von 100 μL 2x Stop-Puffer (40 mM EDTA, 2% SDS und 0,4 mg/ml Proteinase K) und inkubieren Sie bei 37 °C für 30 min.
HINWEIS: Stop Buffer deproteinisiert die Plasmid-DNA durch Denaturierung und Proteolyse von gebundenen Histonen.
- Phenol-Chloroform-Extraktion und Ethanolfällung
- In das Röhrchen mit dem Reaktionsgemisch aus Schritt 5.1.4 wird eine gleiche Menge Tris-gesättigtes Phenol gegeben und gut gemischt, gefolgt von einer Zentrifugation bei 16.200 x g für 10 min bei Raumtemperatur.
- Sammeln Sie vorsichtig die obere wässrige Phase mit der Plasmid-DNA mit einer Mikropipette und mischen Sie sie mit einem gleichen Volumen Chloroform. Vortex das Gemisch und Zentrifugieren bei 16.200 x g für 10 min bei Raumtemperatur.
HINWEIS: Isoamylalkohol könnte in diesem Schritt enthalten sein, um eine unscharfe Grenzfläche zwischen der wässrigen und der organischen Phase zu vermeiden. - Als nächstes sammeln Sie die obere wässrige Phase, fügen Sie 1/10 Volumen von 3 M Natriumacetat (pH 5,5) und 2,5 Volumen eiskaltes Ethanol hinzu (siehe Materialtabelle). Mischen Sie die Lösung gut, indem Sie das Röhrchen 3-4 mal umdrehen und die Mischung in einem -20 °C Gefrierschrank für 30 min aufbewahren, um die Plasmid-DNA vollständig auszufällen.
- Die Probe aus Schritt 5.2.3 bei 16200 x g 10 min zentrifugieren und den Überstand vorsichtig verwerfen. Halten Sie die Röhrchen bei Raumtemperatur offen, bis auch nur Spuren von Ethanol verdampft sind und die gefällte Plasmid-DNA in der Röhre verbleibt.
- Führen Sie die Agarose-Gelelektrophorese durch, um den Plasmid-Supercoiling-Effekt zu beobachten.
- Die ausgefällte Plasmid-DNA aus Schritt 5.2.4 wird in sterilem doppelt destilliertem Wasser gelöst.
- Die Proben werden auf einem 1%igen Agarosegel in 1x Tris-acetat-EDTA (TAE)-Puffer (40 mM Tris, 20 mM Essigsäure und 1 mM EDTA) aufgelöst (siehe Materialtabelle).
- Färben Sie das Gel mit einer Konzentration von 0,2-0,5 μg / ml Ethidiumbromid und beobachten Sie es unter UV-Strahlung, um die DNA-Banden auf dem Gel sichtbar zu machen.
Ergebnisse
Die rekombinante N-terminale Nukleoplasmin-Domäne des Proteins FKBP53 aus Arabidopsis thaliana wurde einer analytischen SEC unterzogen. Das Elutionspeakvolumen wurde gegen die Standardkurve aufgetragen, um seinen oligomeren Zustand zu identifizieren. Die analytischen SEC-Ergebnisse zeigten, dass die Domäne als Pentamer in Lösung mit einer ungefähren Molekülmasse von 58 kDa existiert (Abbildung 1A,B). Weiterhin wurde die Nukleoplasmindomäne in Verbindung mit analytischem SEC auf thermische und chemische Stabilität analysiert. Die Nukleoplasminprobe, die einer Wärmebehandlung bis zu 90 °C unterzogen wurde, zeigte keine offensichtliche Verschiebung des Elutionsvolumens und der Peakhöhe im Vergleich zu den bei 20 °C gehaltenen Proben, was darauf hindeutet, dass die Domäne sehr thermostabil ist (Abbildung 1C). Ebenso zeigte die Nukleoplasmindomäne eine Salzstabilität von bis zu 2 M NaCl (Abbildung 1D) und eine Harnstoffstabilität von bis zu 4 M (Abbildung 1E). Das Nukleoplasminpentamer begann jedoch in höheren Harnstoffkonzentrationen zu dissoziieren.
Ein Pull-Down-Assay wurde durchgeführt, um die Art der Wechselwirkungen zu bestimmen, die zur komplexen Bildung zwischen dem Histon-Chaperon (Nukleoplasmindomäne von AtFKBP53) und den Histon-Oligomeren H2A/H2B-Dimer und H3/H4-Tetramer beitragen. Die Wechselwirkung der Nukleoplasmindomäne mit H2A/H2B-Dimer war bis zu einer Salzkonzentration von 0,4 M NaCl stabil (Abbildung 2A). Im Vergleich dazu war die Assoziation mit H3/H4 bis zu 0,7 M NaCl relativ stabil (Abbildung 2B). Die Fähigkeit der Chaperon-Histon-Komplexe, hohen Salzkonzentrationen standzuhalten, deutet auf die Rolle hydrophober Wechselwirkungen bei der Stabilisierung der Komplexe hin. Der Chaperonkomplex mit H3/H4, der auch in hohen Salzkonzentrationen stabil ist, deutet auf eine vorherrschende Rolle hydrophober Wechselwirkungen bei der Komplexbildung hin. Die geringere Stabilität des H2A/H2B-Chaperonkomplexes in hohen Salzkonzentrationen zeigt eine signifikante Rolle für elektrostatische Wechselwirkungen bei der Komplexbildung. In einem anderen Experiment wurde der Pulldown-Assay verwendet, um zu untersuchen, ob das Chaperon entweder H2A / H2B-Dimer oder H3 / H4-Tetramer bevorzugt. Die Ergebnisse zeigten, dass das Chaperon gleichzeitig und unabhängig von der Reihenfolge, in der sie dem Chaperon hinzugefügt werden, an H2A/H2B-Dimer und H3/H4-Tetramer bindet (Abbildung 2C,D). Dies deutete darauf hin, dass das Chaperon separate Stellen für seine Interaktion mit den Histon-Oligomeren besitzt.
AUC-SV-Experimente (Abbildung 3) wurden durchgeführt, um die Stöchiometrie der Interaktion zwischen Histon-Oligomeren und Chaperonen zu untersuchen. Die AUC-SV-Datenanalyse lieferte einen Sedimentationskoeffizienten (s) von 5,40 S für die AtFKBP53-Nukleoplasmindomäne im Komplex mit H2A/H2B, der einer Molekülmasse von 104 kDa entsprach. Der Komplex der Nukleoplasmindomäne mit H3/H4 ergab einen Sedimentationskoeffizienten von 7,35 S, was 129 kDa entspricht. Die geschätzte Molekülmasse der Komplexe zeigt, dass das pentamerische Nukleoplasmin in einer 1:1-Stöchiometrie einen Komplex sowohl mit H2A/H2B-Dimer als auch mit H3/H4-Tetramer bildet.
Es ist wichtig zu zeigen, dass das Protein Histon-Oligomere auf DNA ablagern kann, um zu bestätigen, dass es sich um ein Histon-Chaperon handelt. Zu diesem Zweck wurde ein Plasmid-Supercoiling-Assay verwendet (Abbildung 4). Das entspannte zirkuläre Plasmid wurde mit den Histon-Oligomeren H2A/H2B und H3/H4 mit den rekombinanten Pflanzen-Histon-Chaperonen der NAP-Familie - AtNRP1 und AtNRP228 - inkubiert. Die Anwesenheit des Chaperons erhöhte die Menge an supercoiled Plasmid, was darauf hindeutet, dass es Histone auf der DNA ablagern könnte, um Nukleosomen zu bilden, was zu DNA-Supercoiling führt.

Abbildung 1: Oligomerer Zustand und Stabilität der Nukleoplasmindomäne von AtFBP53. (A) Analytisches Größenausschlusschromatographieprofil der AtFKBP53-Nukleoplasmindomäne. (B) Kalibrierkurve unter Verwendung von Kugelsternproteinen bekannter Molekülmasse. Die blauen Punkte repräsentieren die Molekülmasse der bekannten Proteine, während der rote Punkt die AtFKBP53-Nukleoplasmindomäne darstellt. (440 kDa - Ferritin, 158 kDa-Aldolase, 75 kDa-con-Albumin, 44 kDa-Ovalbumin, 6,5 kDa-Aprotinin). (C) Analytisches Größenausschlusschromatogramm von 500 μL 0,5 mg/ml AtFKBP53-Nukleoplasmindomäne, die einer Wärmebehandlung bei verschiedenen Temperaturen unterzogen wurde: 20 °C (grün), 40 °C (orange), 60 °C (schwarz), 90 °C (hellblau). (D) Analytisches Größenausschlusschromatogramm von 500 μL 0,5 mg/ml AtFKBP53-Nukleoplasmindomäne in Puffern mit unterschiedlichen NaCl-Konzentrationen: 0,3 M (violett), 0,6 M (rot), 1,0 M (hellblau), 1,5 M (grün), 2,0 M (schwarz). (E) Analytisches Größenausschlusschromatogramm der AtFKBP53-Nukleoplasmindomäne in Puffern mit unterschiedlichen Harnstoffkonzentrationen: 0 M (Kontrolle; hellblau), 1,0 M (rosa), 2,0 M (schwarz), 3,0 M (dunkelblau), 4,0 M (grün), 5,0 M (braun). Das Nukleoplasminpentamer zeigt eine hohe Stabilität gegenüber thermischen und chemischen Stressbedingungen. Die Abbildung ist Referenz21 entnommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
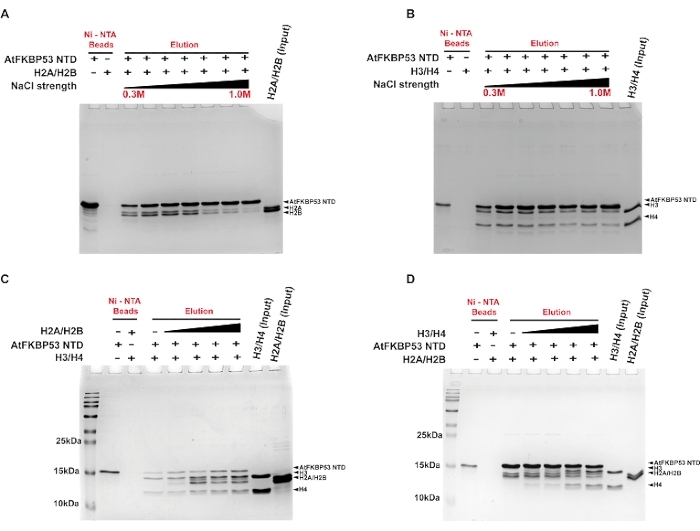
Abbildung 2: Pull-down-Assays für die Interaktion der Nukleoplasmindomäne von AtFKBP53 mit Histon-Oligomeren. 18% SDS-PAGE Bilder der Elutionsfraktionen aus den Assays sind hier dargestellt. Pull-Down-Assay für ( A ) 20 μM H2A/H2B-Dimer und (B) 20 μM H3/H4-Tetramer mit 5 μM AtFKBP53-Nukleoplasmindomäne in steigenden Konzentrationen von NaCl im Bereich von 0,3 M, 0,5 M, 0,6 M, 0,7 M, 0,8 M, 0,9 M und 1,0 M. 5 μM AtFKBP53 FKBD wurde hier als Negativkontrolle verwendet. Für das kompetitive Bindungsexperiment wurde ( C ) eine Mischung aus 5 μM AtFKBP53 Nukleoplasmindomäne und 20 μM H3/H4 Tetramer inkubiert mit einem Bereich von 20-60 μM H2A/H2B Dimer und (D) eine Mischung aus 5 μM AtFKBP53 Nukleoplasmindomäne und 20 μM H2A/H2B Dimer inkubiert mit einem Bereich von 20-60 μM H3/H4 Tetramer verwendet. Die Markierung AtFKBP53 entspricht der Nukleoplasmindomäne von AtFKBP53. Elutionenfraktionen zeigen eine gleichzeitige Bindung der beiden Histon-Oligomere an das Nukleoplasmin. Die Abbildung ist Referenz21 entnommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Analytisches Ultrazentrifugations-Sedimentationsgeschwindigkeits-Experiment (AUC-SV) von Histon-Oligomeren, der Nukleoplasmindomäne von AtFKBP53 und ihren Komplexen. Die AUC-Fernverteilung vs. Diagramm des Sedimentationskoeffizienten (S). Die erhaltenen Werte des Sedimentationskoeffizienten (s) und der Molekülmassen werden ebenfalls angegeben. Die Markierung AtFKBP53 entspricht der Nukleoplasmindomäne von AtFKBP53. Die geschätzten Molekülmassen zeigen eine 1:1-Stöchiometrie für die AtFKBP53-Nukleoplasmindomäne mit den Histon-Oligomeren H2A/H2B-Dimer und H3/H4-Tetramer. 450 μL aller Proteinproben mit einem OD280 von 0,3-0,5 wurden für die AUC-SV-Experimente verwendet. Die Abbildung ist Referenz21 entnommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Plasmid-Supercoiling-Assay. Plasmid-Supercoiling-Assay für die Histon-Chaperone AtNRP1 und AtNRP2. Für das Experiment wurden 500 ng pUC19-Plasmid-DNA mit 1 μg Topoisomerase I vorbehandelt. 4 μM AtNRP1, 4 μM AtNRP2 und eine Mischung aus 4 μM H2A/H2B-Dimer und 2 μM H3/H4-Tetramer dienten als Kontrolle, die keine Supercoiling-Aktivität zeigt, wenn sie mit der vorbehandelten pUC19-DNA inkubiert wird. Die Bahnen mit einer Mischung aus 4 μM H2A/H2B Dimer und 2 μM H3/H4 Tetramer und je 4 μM AtNRP1 und AtNRP2 zeigen die Bildung von supercoiled DNA bei Inkubation mit der vorbehandelten pUC19 DNA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Diese Arbeit demonstriert und validiert einen umfassenden Satz von Protokollen für die biochemische und biophysikalische Charakterisierung eines mutmaßlichen Histon-Chaperons. Hier wurden rekombinant exprimierte und gereinigte Proteine der NAP-Familie, AtNRP1 und AtNRP2, und die N-terminale Nukleoplasmindomäne von AtFKBP53 verwendet, um die Protokolle zu demonstrieren. Die gleiche Reihe von Experimenten könnte sehr gut verwendet werden, um die funktionellen Eigenschaften von bisher nicht charakterisierten Histon-Chaperonen von jedem Organismus abzugrenzen.
Der erste Teil des Protokollabschnitts beinhaltet die Untersuchung des oligomeren Zustands und der Stabilität eines Histon-Chaperons. Mehrere Berichte deuten darauf hin, dass Histon-Chaperone eine beträchtliche Vielfalt in ihrem oligomeren Zustand aufweisen. Zum Beispiel existiert humanes CAF-1 als Monomer29. Mitglieder der NAP-Familie existieren als Dimer oder Tetramer 29,30,31. Nukleoplasmine zeigen pentamerische und oft decamerische oligomere Zustände32,33. Ein analytisches SEC-Experiment kann den oligomeren Zustand eines Histon-Chaperons bestimmen, und AUC-SV-Experimente können dasselbe bestätigen. Es ist bekannt, dass einige der Histon-Chaperone unter verschiedenen thermischen und chemischen Stressbedingungen sehr stabil sind33,34. Die thermischen und chemischen Stabilitätsmerkmale von Histon-Chaperonen konnten auch in Verbindung mit analytischen SEC untersucht werden. Darüber hinaus könnte die Zirkulardichroismusspektroskopie effektiv zur eingehenden Analyse der Veränderungen in der Sekundärstruktur des Chaperons verwendet werden, wenn sie steigenden Temperaturen oder höheren Konzentrationen eines chemischen Wirkstoffs ausgesetzt wird.
Der zweite Teil des Protokollabschnitts behandelt Pull-Down-Assays, die die grundlegenden Wechselwirkungen untersuchen könnten, die die Assoziation von Histon-Oligomeren mit dem Chaperon unterstützen, indem sie einen Salzgradientenansatz und einen kompetitiven Pull-Down-Assay verwenden, um die Histon-Oligomer-Präferenz eines Chaperons zu identifizieren. Wenn der Komplex mit einem leichten Anstieg der Salzkonzentration auseinanderfällt, würde dies auf einen großen Beitrag elektrostatischer Wechselwirkungen zur Stabilisierung des Komplexes hindeuten. Ein intakter Komplex in hohem Salzgehalt würde auf eine signifikante Rolle der Hydrophobie bei der Stabilisierung des Komplexes hindeuten35. Der kompetitive Pulldown-Assay könnte leicht verwendet werden, um die Spezifität oder Präferenz eines Histon-Chaperons für eine bestimmte Histon-Oligomerklasse zu bestimmen. Basierend auf ihrer Präferenz gegenüber Histon-Oligomeren können Histon-Chaperone in drei Kategorien eingeteilt werden, wie H2A / H2B-Chaperone, H3 / H4-Chaperone und H2A / H2B-H3 / H4-Chaperone10,36. Darüber hinaus könnte bei Bedarf die isotherme Titrationskalorimetrie (ITC) verwendet werden, um die Histon-Oligomer-Spezifität eines gegebenen Chaperons und die thermodynamischen Eigenschaften ihrer Wechselwirkungen zu verstehen.
Der dritte Teil des Protokollabschnitts befasst sich mit der Untersuchung der Wechselwirkungsstöchiometrie zwischen einem Histon-Chaperon und den Histon-Oligomeren. Im Allgemeinen unterscheiden sich die verschiedenen Familien der Histonchaperone erheblich in der Stöchiometrie ihrer Assoziation mit H2A/H2B bzw. H3/H4 21,28,37,38. Das AUC-SV-Experiment hilft bei der Ermittlung des Sedimentationskoeffizienten (s) und der Molekülmasse eines Proteins oder seines Komplexes, was sehr nützlich ist, um die Stöchiometrie in der komplexen Formation genau abzuschätzen. Alternativ kann die ITC auch zur Untersuchung der Stöchiometrie eingesetzt werden.
Der vierte Teil des Protokollabschnitts konzentriert sich auf die Untersuchung der Nukleosomenanordnungsfunktion von Histon-Chaperonen. Histon-Chaperone spielen eine entscheidende Rolle bei der Nukleosomenanordnung, die lebenswichtige zelluläre Prozesse wie Replikation, Transkription und DNA-Reparatur reguliert39. Der Plasmid-Supercoiling-Assay, der typischerweise für die In-vitro-Bewertung der Histon-Chaperoning-Aktivität von Histon-Chaperonen verwendet wird, wird in diesem Abschnitt erläutert.
Es kann angemerkt werden, dass nicht alle Histon-Chaperone vollständig strukturiert sind. Von wenigen sind bekannt, dass sie intrinsisch ungeordnete Regionen haben40,41. Daher sind thermische und chemische Stabilitätstests für solche Proteine möglicherweise nicht geeignet. Darüber hinaus haben Histon-Chaperone aus verschiedenen Organismen unterschiedliche oligomere Zustände und differentielle Fähigkeiten, an Histone zu binden. Daher kann dieses Protokoll ein guter Ausgangspunkt sein, würde aber bei Bedarf Änderungen nach sich ziehen.
Offenlegungen
Es wurde kein Interessenkonflikt erklärt.
Danksagungen
Die außerordentlichen Zuschüsse an Dileep Vasudevan vom Science and Engineering Research Board, Government of India [CRG/2018/000695/PS] und dem Department of Biotechnology, Ministry of Science and Technology, Government of India [BT/INF/22/SP33046/2019], sowie die interne Unterstützung durch das Institute of Life Sciences, Bhubaneswar, werden sehr gewürdigt. Wir danken Frau Sudeshna Sen und Frau Annapurna Sahoo für ihre Hilfe bei der Histonvorbereitung. Die Gespräche mit unseren Kollegen Dr. Chinmayee Mohapatra, Manas Kumar Jagdev und Dr. Shaikh Nausad Hossain werden ebenfalls gewürdigt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acrylamide | MP Biomedicals | 814326 | |
| Agarose | MP Biomedicals | 193983 | |
| AKTA Pure 25M FPLC | Cytiva | 29018226 | Instrument for protein purification |
| Ammonium persulfate (APS) | Sigma | A3678 | |
| An-60Ti rotor | Beckman Coulter | 361964 | Rotor for analytical ultracentrifugation |
| Bovine serum albumin (BSA) | Sigma | A7030 | |
| Chloroform | Sigma | C2432 | |
| Coomassie brilliant blue R 250 | Sigma | 1.15444 | |
| Dialysis tubing (7 kDa cut-off) | Thermo Fisher | 68700 | For dialysing protein samples |
| Dithiothreitol (DTT) | MP Biomedicals | 100597 | |
| DNA Loading Dye | New England Biolabs | B7025S | |
| EDTA disodium salt | MP Biomedicals | 194822 | |
| Electronic balance | Shimadzu | ATX224R | |
| Ethanol | Sigma | E7023 | |
| Ethidium bromide (EtBr) | Sigma | E8751 | |
| Gel Doc System | Bio-Rad | 12009077 | For imaging gels after staining |
| Horizontal gel electrophoresis apparatus | Bio-Rad | 1704405 | Instrument for agarose gel electrophoresis |
| Hydrochloric acid (HCl) | Sigma | 320331 | |
| Imidazole | MP Biomedicals | 102033 | |
| Magnesium chloride (MgCl2) | Sigma | M8266 | |
| Micropipettes | Eppendorf | Z683779 | For pipetting of micro-volumes |
| Mini-PROTEAN electrophoresis system | Bio-Rad | 1658000 | Instrument for SDS-PAGE |
| N,N-methylene-bis-acrylamide | MP Biomedicals | 800172 | |
| Nano drop | Thermo Fisher | ND-2000 | For measurement of protein and DNA concentrations |
| Ni-NTA agarose | Invitrogen | R901-15 | Resin beads for pull-down assay |
| Optima AUC analytical ultracentrifuge | Beckman Coulter | B86437 | Instrument for analytical ultracentrifugation |
| pH Meter | Mettler Toledo | MT30130863 | |
| Phenol | Sigma | P4557 | |
| Plasmid isolation kit | Qiagen | 27104 | |
| Proteinase K | Sigma-Aldrich | 1.07393 | |
| pUC19 | Thermo Fisher | SD0061 | Plasmid for supercoiling assay |
| Refrigerated high-speed centrifuge | Thermo Fisher | 75002402 | |
| SDS-PAGE protein marker | Bio-Rad | 1610317 | |
| SEDFIT | Free software program for analytical ultracentrifugation data analysis | ||
| SEDNTERP | Free software program to estimate viscosity and density of buffer and partial specific volume of a protein | ||
| SigmaPrep Spin Columns | Sigma | SC1000 | For pull-down assay |
| Sodium acetate | Sigma | S2889 | |
| Sodium chloride (NaCl) | Merck | S9888 | |
| Sodium dodecyl sulfate (SDS) | MP Biomedicals | 102918 | |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | Column for analytical size-exclusion chromatography |
| Superdex 75 Increase 10/300 GL | Cytiva | 29148721 | Column for analytical size-exclusion chromatography |
| TEMED | Sigma | 1.10732 | |
| Topoisomerase I | Inspiralis | WGT102 | Enzyme used in plasmid supercoiling assay |
| Tris base | Merck | T1503 | |
| Tween-20 | Sigma | P1379 | |
| Urea | MP Biomedicals | 191450 | |
| Water bath | Nüve | NB 5 | For heat treatment of protein samples |
| β-mercaptoethanol (β-ME) | Sigma | M6250 |
Referenzen
- Hübner, M. R., Eckersley-Maslin, M. A., Spector, D. L. Chromatin organization and transcriptional regulation. Current Opinion in Genetics and Development. 23 (2), 89-95 (2013).
- Lai, W. K. M., Pugh, B. F. Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nature Reviews Molecular Cell Biology. 18 (9), 548-562 (2017).
- Kim, U. J., Han, M., Kayne, P., Grunstein, M. Effects of histone H4 depletion on the cell cycle and transcription of Saccharomyces cerevisiae. EMBO Journal. 7 (7), 2211-2219 (1988).
- Prado, F., Aguilera, A. Partial depletion of histone H4 increases homologous recombination-mediated genetic instability. Molecular and Cellular Biology. 25 (4), 1526-1536 (2005).
- Meeks-Wagner, D., Hartwell, L. H. Normal stoichiometry of histone dimer sets is necessary for high fidelity of mitotic chromosome transmission. Cell. 44 (1), 43-52 (1986).
- Groth, A., et al. Human Asf1 regulates the flow of S phase histones during replicational stress. Molecular Cell. 17 (2), 301-311 (2005).
- Laskey, R. A., Honda, B. M., Mills, A. D., Finch, J. T. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA. Nature. 275 (5679), 416-420 (1978).
- Das, C., Tyler, J. K., Churchill, M. E. A. The histone shuffle: histone chaperones in an energetic dance. Trends in Biochemical Sciences. 35 (9), 476-489 (2010).
- Akey, C. W., Luger, K. Histone chaperones and nucleosome assembly. Current Opinion in Structural Biology. 13 (1), 6-14 (2003).
- De Koning, L., Corpet, A., Haber, J. E., Almouzni, G. Histone chaperones: An escort network regulating histone traffic. Nature Structural and Molecular Biology. 14 (11), 997-1007 (2007).
- Eitoku, M., Sato, L., Senda, T., Horikoshi, M. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly. Cellular and Molecular Life Sciences. 65 (3), 414-444 (2008).
- Quivy, J. P., Grandi, P., Almouzni, G. Dimerization of the largest subunit of chromatin assembly factor 1: importance in vitro and during Xenopus early development. EMBO Journal. 20 (8), 2015-2027 (2001).
- Ray-Gallet, D., et al. HIRA is critical for a nucleosome assembly pathway independent of DNA synthesis. Molecular Cell. 9 (5), 1091-1100 (2002).
- Frehlick, L. J., Eirín-López, J. M., Ausió, J. New insights into the nucleophosmin/nucleoplasmin family of nuclear chaperones. Bioessays. 29 (1), 49-59 (2007).
- Ito, T., Bulger, M., Kobayashi, R., Kadonaga, J. T. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays. Molecular and Cellular Biology. 16 (6), 3112-3124 (1996).
- Elsässer, S. J., D'Arcy, S. Towards a mechanism for histone chaperones. Biochimica et Biophysica Acta. 1819 (3-4), 211-221 (2013).
- Rodríguez-Campos, A., Koop, R., Faraudo, S., Beato, M. Transcriptionally competent chromatin assembled with exogenous histones in a yeast whole cell extract. Nucleic Acids Research. 32 (13), 111(2004).
- Levenstein, M. E., Kadonaga, J. T. Biochemical analysis of chromatin containing recombinant Drosophila core histones. Journal of Biological Chemistry. 277 (10), 8749-8754 (2002).
- Huang, S., et al. Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing. Proceedings of the National Academy of Sciences of the United States of America. 102 (38), 13410-13415 (2005).
- Swaminathan, V., Kishore, A. H., Febitha, K. K., Kundu, T. K. Human histone chaperone nucleophosmin enhances acetylation-dependent chromatin transcription. Molecular and Cellular Biology. 25 (17), 7534-7545 (2005).
- Singh, A. K., Datta, A., Jobichen, C., Luan, S., Vasudevan, D. AtFKBP53: A chimeric histone chaperone with functional nucleoplasmin and PPIase domains. Nucleic Acids Research. 48 (3), 1531-1550 (2020).
- Scofield, B. T. K. H. Protein Electrophoresis. , (2012).
- Andrew, S. M., Titus, J. A., Zumstein, L. Dialysis and concentration of protein solutions. Current Protocols in Toxicology, Appendix 3. , 1-5 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. Journal of Visualized Experiments. (33), e1530(2009).
- Padavannil, A., Brautigam, C. A., Chook, Y. M. Molecular size analysis of recombinant importin-histone complexes using analytical ultracentrifugation. Bio-protocol. 10 (10), 3625(2019).
- Zhao, H., Brautigam, C. A., Ghirlando, R., Schuck, P. Overview of current methods in sedimentation velocity and sedimentation equilibrium analytical ultracentrifugation. Current Protocols in Protein Science. , Chapter 20, Unit20.12 (2013).
- Schuck, P. Size-distribution analysis of macromolecules by sedimentation velocity ultracentrifugation and Lamm equation modelling. Biophysical Journal. 78 (3), 1606-1619 (2000).
- Kumar, A., Kumar Singh, A., Chandrakant Bob de, R., Vasudevan, D. Structural characterization of Arabidopsis thaliana NAP1-related protein 2 (AtNRP2) and comparison with its homolog AtNRP1. Molecules. 24 (12), 2258(2019).
- Liu, W. H., Roemer, S. C., Port, A. M., Churchill, M. E. A. CAF-1-induced oligomerization of histones H3/H4 and mutually exclusive interactions with Asf1 guide H3/H4 transitions among histone chaperones and DNA. Nucleic Acids Research. 45 (16), 9809(2017).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Newman, E. R., et al. Large multimeric assemblies of nucleosome assembly protein and histones revealed by small-angle X-ray scattering and electron microscopy. Journal of Biological Chemistry. 287 (32), 26657-26665 (2012).
- Edlich-Muth, C., et al. The pentameric nucleoplasmin fold is present in Drosophila FKBP39 and a large number of chromatin-related proteins. Journal of Molecular Biology. 427 (10), 1949-1963 (2015).
- Franco, A., et al. Structural insights into the ability of nucleoplasmin to assemble and chaperone histone octamers for DNA deposition. Scientific Reports. 9 (1), 9487(2019).
- Xiao, H., Jackson, V., Lei, M. The FK506-binding protein, Fpr4, is an acidic histone chaperone. FEBS Letters. 580 (18), 4357-4364 (2006).
- Graziano, G. Role of hydrophobic effect in the salt-induced dimerization of bovine beta-lactoglobulin at pH 3. Biopolymers. 91 (12), 1182-1188 (2009).
- Burgess, R. J., Zhang, Z. Histone chaperones in nucleosome assembly and human disease. Nature Structural and Molecular Biology. 20 (1), 14-22 (2013).
- Donham, D. C., Scorgie, J. K., Churchill, M. E. The activity of the histone chaperone yeast Asf1 in the assembly and disassembly of histone H3/H4-DNA complexes. Nucleic Acids Research. 39 (13), 5449-5458 (2011).
- Avvakumov, N., Nourani, A., Côté, J. Histone chaperones: Modulators of chromatin marks. Molecular Cell. 41 (5), 502-514 (2011).
- Ransom, M., Dennehey, B. K., Tyler, J. K. Chaperoning histones during DNA replication and repair. Cell. 140 (2), 183-195 (2010).
- Chu, X., et al. Importance of electrostatic interactions in the association of intrinsically disordered histone chaperone Chz1 and histone H2A.Z-H2B. PLoS Computational Biology. 8 (7), 1002608(2012).
- Heidarsson, P. O., et al. Disordered proteins enable histone chaperoning on the nucleosome. bioRxiv. , (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten