Method Article
In vitro Caractérisation des chaperons de histones à l’aide d’essais analytiques, de rabattement et de chaperoning
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit une batterie de méthodes qui comprend la chromatographie analytique taille-exclusion pour étudier l’oligomérisation et la stabilité des histones chaperones, le test pull-down pour démêler les interactions hiperon-histone, l’ASC pour analyser la stœchiométrie des complexes protéiques et le test de chaperonnage des histones pour caractériser fonctionnellement un chaperon histones putatif in vitro.
Résumé
Les protéines d’histone s’associent à l’ADN pour former la chromatine eucaryote. L’unité de base de la chromatine est un nucléosome, constitué d’un octamère d’histones constitués de deux copies des histones centrales H2A, H2B, H3 et H4, enroulées par l’ADN. L’octamère est composé de deux copies d’un dimère H2A/H2B et d’une seule copie d’un tétramère H3/H4. Les histones du noyau hautement chargées sont sujettes à des interactions non spécifiques avec plusieurs protéines du cytoplasme cellulaire et du noyau. Les chaperons d’histones forment une classe diversifiée de protéines qui transportent les histones du cytoplasme dans le noyau et facilitent leur dépôt sur l’ADN, facilitant ainsi le processus d’assemblage des nucléosomes. Certains chaperons d’histones sont spécifiques pour H2A/H2B ou H3/H4, et certains fonctionnent comme chaperons pour les deux. Ce protocole décrit comment des techniques de laboratoire in vitro telles que les essais de pull-down, la chromatographie analytique d’exclusion de la taille, l’ultracentrifugation analytique et le test de chaperonnage des histones pourraient être utilisées en tandem pour confirmer si une protéine donnée est fonctionnelle en tant que chaperon d’histones.
Introduction
Les nucléosomes composés d’ADN et de protéines histones forment l’unité structurelle de la chromatine et régulent plusieurs événements cellulaires critiques. Les nucléosomes sont repositionnés et remodelés dynamiquement pour rendre l’ADN accessible à divers processus tels que la réplication, la transcription et la traduction 1,2. Les histones qui sont très basiques ont tendance à interagir avec les protéines acides dans le milieu cellulaire ou subissent une agrégation, conduisant ainsi à divers défauts cellulaires 3,4,5. Un groupe de protéines dédiées appelées chaperons d’histones aide au transport des histones du cytoplasme vers le noyau et prévient les événements aberrants d’agrégation histone-ADN 6,7. Fondamentalement, la plupart des chaperons d’histones stockent et transfèrent les histones sur l’ADN à une force ionique physiologique, facilitant ainsi la formation de nucléosomes 8,9. Certains chaperons d’histones ont une nette préférence pour les oligomères d’histones H2A/H2B ou H3/H410.
Les chaperons d’histones sont caractérisés en fonction de leur capacité à assembler des nucléosomes dépendants ou indépendants de la synthèse de l’ADN11. Par exemple, le facteur d’assemblage de la chromatine 1 (CAF-1) est dépendant tandis que le régulateur d’histones A (HIRA) est indépendant de la synthèse de l’ADN12,13. De même, la famille des nucléoplasmines des chaperons d’histones est impliquée dans la décondensation de la chromatine des spermatozoïdes et l’assemblage des nucléosomes14. Les membres de la famille des protéines d’assemblage des nucléosomes (NAP) facilitent la formation de structures de type nucléosome in vitro et sont impliqués dans la navette des histones entre le cytoplasme et le noyau15. Les nucléoplasmines et les protéines de la famille NAP sont toutes deux des chaperons fonctionnels des histones, mais ne partagent aucune caractéristique structurelle. Essentiellement, aucune caractéristique structurelle unique ne permet la classification d’une protéine comme chaperond’histones 16. L’utilisation d’essais fonctionnels et biophysiques ainsi que d’études structurelles fonctionne mieux pour caractériser les chaperons d’histones.
Ce travail décrit des méthodes biochimiques et biophysiques pour caractériser une protéine en tant que chaperon d’histones qui facilite l’assemblage des nucléosomes. Tout d’abord, une chromatographie analytique d’exclusion de taille a été réalisée pour analyser le statut oligomère et la stabilité des chaperons d’histones. Ensuite, un test de pull-down a été effectué pour déterminer les forces motrices et la nature compétitive des interactions hiperon-histone. Cependant, les paramètres hydrodynamiques de ces interactions n’ont pas pu être calculés avec précision à l’aide de la chromatographie analytique taille-exclusion en raison de la forme de la protéine et de ses complexes qui ont un impact sur leur migration à travers la colonne. Par conséquent, l’ultracentrifugation analytique a été utilisée, qui fournit des propriétés macromoléculaires en solution qui incluent un poids moléculaire précis, la stœchiométrie de l’interaction et la forme des molécules biologiques. Des études antérieures ont largement utilisé le test de chaperonnage in vitro des histones pour caractériser fonctionnellement les chaperons d’histones tels que yScS116 17, DmACF18, ScRTT106p19, HsNPM120. Le test de chaperonnage des histones a également été utilisé pour caractériser fonctionnellement les protéines en tant que chaperons d’histones.
Protocole
1. Chromatographie analytique d’exclusion de taille pour élucider le statut oligomère et la stabilité des chaperons d’histones
- Analyse du statut oligomérique des chaperons d’histones
- Équilibrer une colonne de chromatographie analytique d’exclusion de taille (SEC) de 24 mL avec 1,2 volume de colonne (CV), c.-à-d. 28,8 mL de tampon SEC dégazé [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl et 1 mM de β-mercaptoéthanol (β-ME)] à 4 °C (voir le tableau des matériaux).
REMARQUE : Le type de colonne, la composition du tampon et le pH du tampon peuvent être choisis en fonction de la protéine d’intérêt. Le volume d’injection de l’échantillon ne doit pas dépasser 500 μL pour une colonne de 24 mL. En outre, la pression de la colonne doit être maintenue en dessous de 5 MPa. - À partir d’une solution mère de protéines à concentration plus élevée, préparer 500 μL d’échantillon de protéines à 0,5 mg/mL dans un tampon SEC dégazé et l’injecter dans la colonne prééquilibrée à l’aide d’une boucle d’injection de 500 μL. Laisser procéder la chromatographie à un débit isocratique de 0,2 à 0,3 mL/min avec le tampon SEC à 4 °C.
- Surveiller le profil d’élution de la protéine en mesurant l’absorbance à une longueur d’onde de 280 nm. Lorsqu’il s’agit de protéines dépourvues de résidus aromatiques, mesurer l’absorbance à 214 nm.
- Utiliser le volume d’élution de la protéine pour calculer son poids moléculaire approximatif en kDa à l’aide de la courbe d’étalonnagestandard 21.
NOTA: La courbe d’étalonnage est préparée en traçant le volume de rétention des protéines de poids moléculaire connues par rapport au logarithme de leurs masses moléculaires respectives (log Mr), éluées à l’aide de la même colonne.
- Équilibrer une colonne de chromatographie analytique d’exclusion de taille (SEC) de 24 mL avec 1,2 volume de colonne (CV), c.-à-d. 28,8 mL de tampon SEC dégazé [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl et 1 mM de β-mercaptoéthanol (β-ME)] à 4 °C (voir le tableau des matériaux).
- Analyse de la stabilité thermique des chaperons d’histones
- Prélever 500 μL de 0,5 mg/mL de l’échantillon de protéines préparé dans un tampon SEC dégazé (identique à celui utilisé au point 1.1.1) dans des tubes de microcentrifugation individuels et chauffer chaque tube à une température particulière comprise entre 20 °C et 90 °C (20 °C, 40 °C, 60 °C et 90 °C) pendant 10 minutes au bain-marie.
- Ensuite, centrifuger les échantillons traités thermiquement à 16 200 x g pendant 10 min à 4 °C, recueillir le surnageant avec une micropipette et injecter chaque échantillon individuellement à l’aide d’une boucle d’injection de 500 μL dans la colonne d’analyse, prééquilibrée avec le tampon SEC à 4 °C.
- Laisser procéder la chromatographie à un débit isocratique de 0,2 à 0,3 mL/min avec le tampon SEC à 4 °C.
- Observez la position et la hauteur des pics d’élution et recherchez l’apparition de pics supplémentaires pour les différents échantillons.
- Analyse de la stabilité chimique des chaperons d’histones
- Pour examiner la stabilité au sel des chaperons d’histones, incuber 500 μL d’échantillon de protéines à 0,5 mg/mL préparé dans un tampon Tris [20 mM de Tris-HCl (pH 7,5) et 1 mM de β-ME] complété par des concentrations croissantes de NaCl (300 mM, 600 mM, 1 M, 1,5 M et 2 M) dans des tubes microcentrifuges séparés pendant 30 minutes à 4 °C. Centrifuger les échantillons à 16 200 x g pendant 10 min à 4 °C et retenir le surnageant.
- Ensuite, chargez les échantillons de protéines dans différentes concentrations de NaCl individuellement, en utilisant une boucle d’injection de 500 μL dans la colonne d’analyse prééquilibrée avec 1,2 CV (28,8 mL) du tampon respectif contenant des concentrations croissantes de NaCl à 4 °C.
- Laisser l’essai de chromatographie se dérouler à un débit isocratique de 0,2 à 0,3 mL/min avec 1 CV (24 mL) du tampon respectif à 4 °C.
- Observez la position et la hauteur des pics d’élution et recherchez l’apparition de pics supplémentaires pour les différents échantillons.
- De même, pour l’analyse de la stabilité de l’urée, incuber 500 μL d’échantillon de protéines à 0,5 mg/mL préparé dans un tampon Tris [20 mM de Tris-HCl (pH 7,5) et 1 mM de β-ME] complété par des concentrations croissantes d’urée (1 M, 2 M, 3 M, 4 M et 5 M) dans des tubes microcentrifuges séparés pendant 16 h à température ambiante. Centrifuger les échantillons à 16 200 x g pendant 10 min à température ambiante et retenir le surnageant.
- Ensuite, chargez les échantillons de protéines traitées à l’urée individuellement à l’aide d’une boucle d’injection de 500 μL dans la colonne d’analyse prééquilibrée avec 1,2 CV (28,8 mL) du tampon correspondant contenant différentes concentrations d’urée à température ambiante.
- Laisser la chromatographie se dérouler à un débit isocratique de 0,2 à 0,3 mL/min avec 1 CV (24 mL) du tampon respectif à température ambiante.
ATTENTION: Ne pas effectuer les expériences avec un tampon contenant de l’urée à une température plus basse car l’urée a tendance à cristalliser et à endommager la colonne. - Observez la position et la hauteur des pics d’élution et recherchez l’apparition de pics supplémentaires pour les différents échantillons.
2. Tests de réduction basés sur le gradient de sel pour comprendre le type d’interactions contribuant à la formation complexe entre les oligomères d’histones et un chaperon d’histones
- Pour chaque réaction de pull-down, pipeter 40 μL de résine Ni-NTA dans une colonne de spin et laver avec de l’eau stérile bidistillée. Par la suite, équilibrer la résine avec 100 CV (4 mL) de tampon d’équilibre [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 10 mM d’imidazole, 10 μg/mL de BSA et 1 mM de β-ME] (voir le tableau des matériaux).
REMARQUE: Le pull-down peut également être effectué dans un tube microcentrifuge de 1,5 mL. - Préparer l’échantillon en mélangeant 5 μM du chaperon d’histones marqués His avec 20 μM de dimère d’histone H2A/H2B ou de tétramère H3/H4 dans le tampon d’équilibre. Incuber l’échantillon sur de la glace pendant 1 h.
NOTE: Les dimères H2A/H2B et les tétramères H3/H4 sont préparés à partir d’histones humainesrecombinantes 21, et l’intégrité des oligomères est confirmée sur la base des masses moléculaires estimées par ultracentrifugation analytique (ASC). Les mêmes oligomères d’histones ont été utilisés pour toutes les expériences mentionnées ci-dessous. - Charger les échantillons dans des colonnes de spin pré-équilibrées séparées avec de la résine Ni-NTA de l’étape 2.1, chacune étiquetée pour une concentration particulière de sel, et conserver les colonnes pendant 30 minutes à 4 °C. Centrifuger les colonnes à 1000 x g pendant 1 min.
- Ensuite, laver les colonnes avec 100 CV (4 mL) de tampon de lavage [20 mM de Tris-HCl (pH 7,5), 50 mM d’imidazole, 0,2 % de Tween-20 et 1 mM de β-ME] contenant différentes concentrations de sel (c.-à-d. 300 mM, 500 mM, 600 mM, 700 mM, 800 mM, 900 mM et 1 M NaCl). Laver chaque colonne avec un tampon ayant une concentration particulière en sel.
- Après l’étape de lavage salin, éluer la protéine des différentes colonnes en utilisant 100 μL de tampon d’élution [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 300 mM d’imidazole et 1 mM de β-ME].
- Ensuite, soumettre les échantillons élués à 18% SDS-PAGE22 et visualiser le gel après coloration avec Coomassie Brilliant Blue R250 (voir Tableau des matériaux). Alternativement, vous pouvez charger directement la résine sur le gel SDS-PAGE au lieu d’éluer la protéine liée de la résine Ni-NTA.
NOTE: Les compositions tampons d’équilibre, de lavage et d’élution et le pH peuvent être modifiés en fonction de la protéine d’intérêt.
3. Test de pull-down compétitif pour identifier la préférence d’un chaperon d’histones pour H2A/H2B ou H3/H4
- Préparer la colonne de spin comme décrit à l’étape 2.1
- Incuber 5 μM du chaperon d’histones avec 20 μM de dimères H2A/H2B dans 300 μL de tampon d’équilibre (préparé à l’étape 2.1) pendant 30 min sur glace.
NOTE: Le rapport entre l’oligomère d’histones et le chaperon d’histones dans la réaction peut être choisi en fonction des données de stœchiométrie de liaison connues; Utilisez cinq fois l’excès d’histone si aucune information n’est disponible. - Centrifuger le complexe chaperon-H2A/H2B d’histones à 16 200 x g pendant 5 min à 4 °C pour éliminer tout précipité. Ensuite, chargez l’échantillon sur la colonne de spin pré-équilibrée avec un tampon d’équilibrage (préparé à l’étape 2.1) et incuber pendant 30 min à 4 °C.
- Laver la colonne avec 100 CV (4 mL) de tampon de lavage [20 mM de Tris-HCl (pH 7,5), 300 mM de NaCl, 50 mM d’imidazole, 0,2 % de Tween-20 et 1 mM de β-ME] pour éliminer l’excès de dimères H2A/H2B. Ensuite, mélangez le complexe chaperon-H2A/H2B d’histones avec 20-60 μM de tétramère H3/H4 et incuber pendant 30 minutes sur de la glace.
- Laver de nouveau la colonne avec 100 CV (4 mL) de tampon de lavage (préparé à l’étape 3.4) pour éliminer tout tétramère H3/H4 non lié et éluer l’échantillon à l’aide d’un tampon d’élution (préparé à l’étape 2.5). Soumettre les échantillons élués à 18% SDS-PAGE et visualiser après coloration avec Coomassie Brilliant Blue R250.
NOTE: Le test pourrait être inversé dans lequel, d’abord, le tétramère H3 / H4 peut être mélangé avec le chaperon, le complexe autorisé à se lier aux billes de Ni-NTA, et ensuite être incubé avec des concentrations variables de dimère H2A / H2B.
4. Expériences analytiques d’ultracentrifugation - vitesse de sédimentation (ASC-SV) pour analyser la stœchiométrie de liaison entre les chaperons d’histones et les histones
- Préparation des échantillons pour l’ASC
- Dialyser le dimère d’histone H2A/H2B reconstitué, le tétramère H3/H4 et le chaperon d’histones séparément à l’aide d’un tube de dialyse de coupure de 7 kDa23, contre un tampon de dialyse [20 mM de Tris (pH 7,5), 300 mM de NaCl et 1 mM de β-ME] (voir le tableau des matériaux). Pour minimiser l’erreur de fond due à l’incompatibilité du tampon, effectuez une dialyse intensive contre le tampon de dialyse, de préférence trois fois sur une période de 24 heures.
NOTE: La DO 280 initiale des échantillons de protéines doit avoir une valeur deux à trois fois plus élevée pour atteindre une DO280 finale de 0,3-0,5. Ceci est essentiellement fait pour annuler les effets de la dilution. - Purifier le dimère H2A/H2B, le tétramère H3/H4 et le chaperon d’histones individuellement avec le tampon de dialyse, en utilisant la chromatographie analytique d’exclusion de taille (comme mentionné à l’étape 1). Conserver le tampon de l’essai pour préparer d’autres dilutions ultérieurement et l’utiliser comme référence dans la cellule ASC.
- Dialyser le dimère d’histone H2A/H2B reconstitué, le tétramère H3/H4 et le chaperon d’histones séparément à l’aide d’un tube de dialyse de coupure de 7 kDa23, contre un tampon de dialyse [20 mM de Tris (pH 7,5), 300 mM de NaCl et 1 mM de β-ME] (voir le tableau des matériaux). Pour minimiser l’erreur de fond due à l’incompatibilité du tampon, effectuez une dialyse intensive contre le tampon de dialyse, de préférence trois fois sur une période de 24 heures.
- Chargement d’échantillons pour l’AUC
- Mélanger les protéines purifiées dans un volume final de 450 μL à l’aide d’un tampon de dialyse de l’étape 4.1.1 pour atteindre une DO280 de 0,3-0,6. Mélanger le chaperon d’histones avec un dimère H2A/H2B ou un tétramère H3/H4 pour la formation de complexes dans des tubes de réaction séparés. Incuber les mélanges de protéines pendant 2-3 h.
NOTE: Alternativement, les données de sédimentation peuvent être acquises avec un système de balayage optique d’interférence dans l’ultracentrifugeuse analytique. Séparément, pour mélanger des protéines purifiées, fixer la concentration d’hiperon d’histones et l’incuber avec des concentrations croissantes d’oligomères d’histones pour obtenir la stœchiométrie exacte. - Assembler la cellule avec une pièce maîtresse à double secteur et des fenêtres en quartz pour l’expérience ASC-SV à l’aide d’un détecteur d’absorbance de l’ultracentrifugeuse analytique comme décrit précédemment en détail24.
- Remplir 400 μL de la solution échantillon et 420 μL de tampon de dialyse dans les deux secteurs (échantillon et secteur de référence, respectivement) de la cellule.
NOTA : Un plus grand volume de tampon est utilisé dans le secteur de référence pour maintenir le ménisque de référence au-dessus du ménisque de l’échantillon. Cependant, tout en utilisant un système d’interférences optiques, remplissez les deux secteurs avec un volume égal. - Pesez et équilibrez avec précision les cellules et chargez-les dans un rotor en titane à quatre places (voir le tableau des matériaux). Alignez les cellules à l’aide des marques fournies au bas des cellules et du rotor. Chargez le rotor dans la centrifugeuse, fermez le couvercle et laissez développer un vide jusqu’à ce que la pression chute à moins de 15 microns de Hg et que la température du rotor se stabilise à 20 °C (prend généralement 2-2,5 h).
REMARQUE: Les paramètres de fonctionnement de la SSC comprennent la température expérimentale, la vitesse du rotor, l’intervalle entre les balayages et le nombre de balayages à collecter. Dans le cas des expériences SV, l’intervalle de balayage est donné en fonction de la masse moléculaire de la protéine; Les protéines plus petites nécessitent des intervalles de temps plus longs entre les scans. La vitesse du rotor est également réglée en fonction de la masse moléculaire attendue de la protéine, et l’expérience est menée à 20 °C. Les données d’absorbance sont surveillées à 280 nm. - Pour obtenir la stœchiométrie exacte, maintenir la concentration d’hiperon d’hiperon constante et incuber avec des concentrations croissantes d’oligomères d’histones pour atteindre la saturation.
- Mélanger les protéines purifiées dans un volume final de 450 μL à l’aide d’un tampon de dialyse de l’étape 4.1.1 pour atteindre une DO280 de 0,3-0,6. Mélanger le chaperon d’histones avec un dimère H2A/H2B ou un tétramère H3/H4 pour la formation de complexes dans des tubes de réaction séparés. Incuber les mélanges de protéines pendant 2-3 h.
- Analyse des données de la CUA
- Effectuer l’analyse des données comme décrit précédemment25. En bref, calculez la densité et la viscosité des composants tampons à l’aide du programme SEDNTERP26 (voir tableau des matériaux). De même, calculez le volume spécifique partiel de la protéine en fonction de sa composition en acides aminés, également en utilisant SEDNTERP.
- Chargez les données de la machine AUC dans le programme SEDFIT27 et définissez les limites du ménisque (ligne rouge), du fond de la cellule (ligne bleue) et de l’analyse des données (lignes vertes). Choisissez la distribution continue C(s) comme modèle.
- Ensuite, définissez la résolution maximale jusqu’à 100; Définir les coefficients de sédimentation (s), s min: 0 et s max: 10-15; régler le rapport de frottement à 1,2 initialement et choisir de flotter pour dériver le rapport à partir des données; fixer le niveau de confiance (rapport F; qui détermine l’ampleur de la régularisation) à 0,68 pour la régularisation 1 sigma; définir les valeurs de volume spécifique partiel, de densité tampon et de viscosité du tampon obtenues à partir de SEDNTERP.
- Appuyez sur RUN pour permettre au logiciel de résoudre l’équation de Lamm27. Ajustez les paramètres en cas de non-concordance significative des données. Après avoir ajusté les paramètres, appuyez sur FIT pour affiner tous les paramètres. Évaluer la qualité de l’ajustement en fonction de la valeur de l’écart quadratique moyen (RMSD), qui doit être inférieure à 0,01 unité de signal.
- Estimez les masses moléculaires des pics en choisissant l’option: afficher le pic « Mw en c(s) » dans la fonction d’affichage de la barre d’outils principale, qui fournira des informations sur le 's' de la molécule / complexe.
5. Dosage de superenroulement plasmidique pour confirmer la fonction chaperonnante des histones
- Réaction d’assemblage des nucléosomes
- Mélanger 2 μM de tétramère H3/H4 et 4 μM de dimères H2A/H2B avec des concentrations croissantes du chaperon d’histones (1-6 μM) dans un tampon d’assemblage [20 mM de Tris HCl (pH 7,5), 1 mM de DTT, 1 mM de MgCl2,0,1 mg/mL de BSA et 100 mM de NaCl] jusqu’à obtenir un volume final de 50 μL. Incuber le mélange à 4 °C pendant 30 min.
- Simultanément, dans une réaction séparée, prétraiter 500 ng du plasmide pUC19 superenroulé négativement avec 1 μg d’enzyme topoisomérase I (voir tableau des matériaux) dans le tampon d’assemblage dans un volume final de 50 μL et incuber à 30 °C pendant 30 min.
NOTE: La topoisomérase I détend l’ADN plasmidique double brin superenroulé en générant une entaille simple brin. Une enzyme topoisomérase I d’origine eucaryote, telle que la topoisomérase I du germe de blé disponible dans le commerce ou la topisomérase I de Drosophila melanogaster exprimée par recombinaison, pourrait être utilisée. - Ensuite, combinez le tétramère H3/H4, le dimère H2A/H2B, le mélange chaperon d’histones (à partir de l’étape 5.1.1), le mélange de réaction d’ADN plasmidique détendu (à partir de l’étape 5.1.2) et incuber à 30 °C pendant 90 min.
REMARQUE : Configurer deux réactions de contrôle pour le test; l’un ayant le chaperon d’histones et l’ADN plasmidique détendu (mais pas les histones) et l’autre ayant les oligomères d’histones et l’ADN plasmidique détendu (mais pas le chaperon d’histones). - Arrêter la réaction d’assemblage en ajoutant 100 μL de tampon stop 2x (40 mM d’EDTA, 2 % de SDS et 0,4 mg/mL de protéinase K) et incuber à 37 °C pendant 30 min.
REMARQUE: Stop buffer déprotéinise l’ADN plasmidique par dénaturation et protéolyse des histones liées.
- Extraction phénol-chloroforme et précipitation à l’éthanol
- Ajouter un volume égal de phénol saturé en tris dans le tube contenant le mélange réactionnel de l’étape 5.1.4 et bien mélanger, puis centrifuger à 16 200 x g pendant 10 min à température ambiante.
- Prélever délicatement la phase aqueuse supérieure contenant l’ADN plasmidique à l’aide d’une micropipette et mélanger avec un volume égal de chloroforme. Vortex le mélange et centrifuger à 16 200 x g pendant 10 min à température ambiante.
NOTE: L’alcool isoamylique pourrait être inclus à cette étape pour éviter une interface floue entre les phases aqueuse et organique. - Ensuite, prélevez la phase aqueuse supérieure, ajoutez 1/10e de volume d’acétate de sodium 3 M (pH 5,5) et 2,5 volumes d’éthanol glacé (voir le tableau des matériaux). Bien mélanger la solution en retournant le tube 3-4 fois et garder le mélange dans un congélateur à -20 °C pendant 30 min pour une précipitation complète de l’ADN plasmidique.
- Centrifuger l’échantillon de l’étape 5.2.3 à 16200 x g pendant 10 min et éliminer délicatement le surnageant. Gardez les tubes ouverts à température ambiante jusqu’à ce que même des traces d’éthanol s’évaporent, laissant l’ADN plasmidique précipité dans le tube.
- Effectuer l’électrophorèse sur gel d’agarose pour observer l’effet plasmidique superenroulant.
- Dissoudre l’ADN plasmidique précipité de l’étape 5.2.4 dans de l’eau stérile bidistillée.
- Résoudre les échantillons sur un gel d’agarose à 1 % dans 1x tampon Tris-acétate-EDTA (TAE) (40 mM de Tris, 20 mM d’acide acétique et 1 mM d’EDTA) (voir le tableau des matériaux).
- Colorer le gel avec une concentration de bromure d’éthidium de 0,2 à 0,5 μg/mL et observer sous UV pour visualiser les bandes d’ADN sur le gel.
Résultats
Le domaine nucléoplasmine N-terminal recombinant de la protéine FKBP53 d’Arabidopsis thaliana a été soumis à une SEC analytique. Le volume du pic d’élution a été tracé par rapport à la courbe standard pour identifier son état oligomère. Les résultats analytiques de la SEC ont révélé que le domaine existe en tant que pentamer en solution, avec une masse moléculaire approximative de 58 kDa (Figure 1A,B). De plus, la stabilité thermique et chimique du domaine nucléoplasmine a été analysée en conjonction avec la SEC analytique. L’échantillon de nucléoplasmine soumis à un traitement thermique jusqu’à 90 °C n’a montré aucun changement apparent du volume d’élution et de la hauteur du pic par rapport aux échantillons maintenus à 20 °C, ce qui suggère que le domaine est très thermostable (figure 1C). De même, le domaine nucléoplasmine présentait une stabilité en sel jusqu’à 2 M de NaCl (Figure 1D) et une stabilité à l’urée jusqu’à 4 M (Figure 1E). Cependant, le pentamer de nucléoplasmine a commencé à se dissocier à des concentrations d’urée plus élevées.
Un essai de pull-down a été effectué pour déterminer le type d’interactions contribuant à la formation complexe entre le chaperon d’histones (domaine nucléoplasmine d’AtFKBP53) et les oligomères d’histones H2A/H2B dimères et H3/H4 tétramère à l’aide d’un gradient de lavage au sel. L’interaction du domaine nucléoplasmine avec le dimère H2A/H2B était stable jusqu’à une concentration en sel de 0,4 M NaCl (Figure 2A). En comparaison, l’association avec H3/H4 était raisonnablement stable jusqu’à 0,7 M NaCl (Figure 2B). La capacité des complexes chaperon-histones à résister à une concentration élevée de sel suggère le rôle des interactions hydrophobes dans la stabilisation des complexes. Le complexe chaperon avec H3/H4 étant stable même à des concentrations élevées de sel suggère un rôle prédominant des interactions hydrophobes dans la formation du complexe. La stabilité plus faible du complexe H2A/H2B-chaperon à des concentrations élevées de sel révèle un rôle significatif pour les interactions électrostatiques dans la formation du complexe. Dans une autre expérience, le test pull-down a été utilisé pour examiner si le chaperon préfère le dimère H2A/H2B ou le tétramère H3/H4. Les résultats ont révélé que le chaperon se lie au dimère H2A/H2B et au tétramère H3/H4 simultanément et quel que soit l’ordre dans lequel ils sont ajoutés au chaperon (Figure 2C,D). Cela indique que le chaperon possède des sites distincts pour son interaction avec les oligomères d’histones.
Des expériences ASC-SV (Figure 3) ont été réalisées pour étudier la stœchiométrie de l’interaction entre les oligomères d’histones et les chaperons L’analyse des données de l’ASC-SV a fourni une valeur de coefficient de sédimentation (s) de 5,40 S pour le domaine nucléoplasmine AtFKBP53 en complexe avec H2A/H2B qui correspondait à une masse moléculaire de 104 kDa. Le complexe du domaine nucléoplasmine avec H3/H4 a donné une valeur de coefficient de sédimentation de 7,35 S, correspondant à 129 kDa. La masse moléculaire estimée des complexes révèle que la nucléoplasmine pentamère forme un complexe avec à la fois le dimère H2A/H2B et le tétramère H3/H4 dans une stœchiométrie 1:1.
Il est essentiel de montrer que la protéine peut déposer des oligomères d’histones sur l’ADN pour confirmer qu’il s’agit d’un chaperon d’histones. À cette fin, un essai de superenroulement plasmidique a été adopté (Figure 4). Le plasmide circulaire détendu a été incubé avec les oligomères d’histones H2A/H2B et H3/H4 avec les chaperons d’histones végétaux recombinants de la famille NAP - AtNRP1 et AtNRP228. La présence du chaperon a augmenté la quantité de plasmide superenroulé, suggérant qu’il pourrait déposer des histones sur l’ADN pour former des nucléosomes, provoquant un surenroulement de l’ADN.

Figure 1 : État oligomérique et stabilité du domaine nucléoplasmine d’AtFBP53. (A) Profil analytique de chromatographie d’exclusion de taille du domaine nucléoplasmine AtFKBP53. (B) Courbe d’étalonnage obtenue à l’aide de protéines globulaires de masse moléculaire connue. Les points bleus représentent la masse moléculaire des protéines connues, tandis que le point rouge représente le domaine nucléoplasmine AtFKBP53. (440 kDa - ferritine, 158 kDa-aldolase, 75 kDa-con albumine, 44 kDa-ovalbumine, 6,5 kDa-aprotinine). (C) Chromatogramme analytique d’exclusion de taille de 500 μL de 0,5 mg/mL du domaine nucléoplasmine AtFKBP53 soumis à un traitement thermique à différentes températures: 20 °C (vert), 40 °C (orange), 60 °C (noir), 90 °C (bleu clair). (D) Chromatogramme analytique d’exclusion de taille de 500 μL de 0,5 mg/mL du domaine nucléoplasmine AtFKBP53 dans des tampons contenant différentes concentrations de NaCl : 0,3 M (violet), 0,6 M (rouge), 1,0 M (bleu clair), 1,5 M (vert), 2,0 M (noir). (E) Chromatogramme analytique taille-exclusion du domaine nucléoplasmine AtFKBP53 dans des tampons présentant différentes concentrations d’urée : 0 M (témoin; bleu clair), 1,0 M (rose), 2,0 M (noir), 3,0 M (bleu foncé), 4,0 M (vert), 5,0 M (brun). Le pentamer de nucléoplasmine montre une grande stabilité aux conditions de stress thermique et chimique. La figure est adaptée de la référence21. Veuillez cliquer ici pour voir une version agrandie de cette figure.
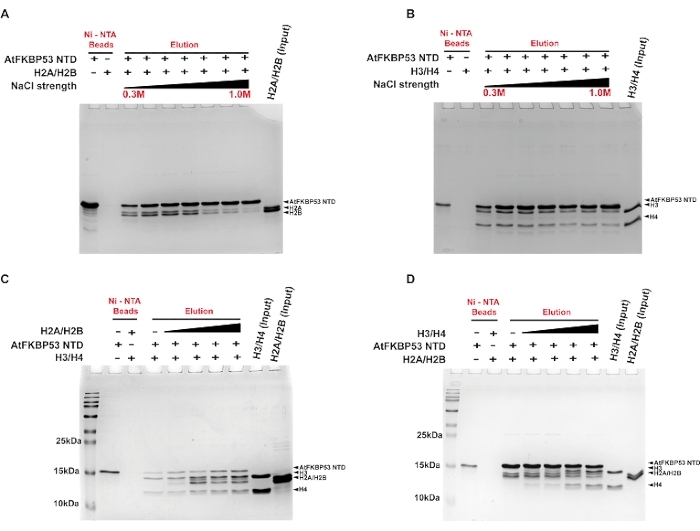
Figure 2 : Tests déroulants pour l’interaction du domaine nucléoplasmine de l’AtFKBP53 avec les oligomères d’histones. 18% des images SDS-PAGE des fractions d’élution des tests sont présentées ici. Le dosage de (A) 20 μM de dimères H2A/H2B et (B) de 20 μM de tétramère H3/H4 avec un domaine nucléoplasmine AtFKBP53 de 5 μM à des concentrations croissantes de NaCl comprises entre 0,3 M, 0,5 M, 0,6 M, 0,7 M, 0,8 M, 0,9 M et 1,0 M. 5 μM AtFKBP53 FKBD a été utilisé comme témoin négatif ici. Pour l’expérience de liaison compétitive, (C) un mélange de 5 μM de domaine nucléoplasmine AtFKBP53 et de 20 μM de tétramère H3/H4 incubé avec une gamme de 20-60 μM de dimères H2A/H2B et (D) un mélange de 5 μM de domaine nucléoplasmine AtFKBP53 et de 20 μM de dimères H2A/H2B incubés avec une gamme de 20-60 μM de tétramère H3/H4 a été utilisé. Le label AtFKBP53 correspond au domaine nucléoplasmine d’AtFKBP53. Les fractions d’élution montrent une liaison simultanée des deux oligomères d’histones à la nucléoplasmine. La figure est adaptée de la référence21. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Expérience analytique d’ultracentrifugation - vitesse de sédimentation (ASC-SV) des oligomères d’histones, du domaine nucléoplasmine d’AtFKBP53 et de leurs complexes. La distribution de la distance de l’AUC par rapport à la placette du coefficient de sédimentation (S). Les valeurs des coefficient(s) de sédimentation obtenus et les masses moléculaires sont également fournies. Le label AtFKBP53 correspond au domaine nucléoplasmine d’AtFKBP53. Les masses moléculaires estimées révèlent une stœchiométrie 1:1 pour le domaine nucléoplasmine AtFKBP53 avec les oligomères d’histones H2A/H2B dimères et H3/H4 tétramères. 450 μL de tous les échantillons de protéines ayant une DO280 de 0,3-0,5 ont été utilisés pour les expériences ASC-SV. La figure est adaptée de la référence21. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Essai de superenroulement plasmidique. Dosage plasmidique superenroulé pour les chaperons d’histones AtNRP1 et AtNRP2. 500 ng d’ADN plasmidique pUC19 ont été prétraités avec 1 μg de topoisomérase I pour l’expérience. 4 μM AtNRP1, 4 μM AtNRP2 et un mélange de 4 μM de dimères H2A/H2B et de 2 μM de tétramère H3/H4 ont été comme témoins qui ne montrent aucune activité de surenroulement lorsqu’ils sont incubés avec l’ADN pUC19 prétraité. Les voies avec un mélange de 4 μM H2A/H2B de dimère et 2 μM H3/H4 de tétramère et 4 μM chacune d’AtNRP1 et AtNRP2 montrent la formation d’ADN superenroulé lors de l’incubation avec l’ADN pUC19 prétraité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Ce travail démontre et valide un ensemble complet de protocoles pour la caractérisation biochimique et biophysique d’un chaperon putatif d’histones. Ici, des protéines de la famille NAP exprimées et purifiées par recombinaison, AtNRP1 et AtNRP2, et le domaine nucléoplasmine N-terminal d’AtFKBP53 ont été utilisés pour démontrer les protocoles. Le même ensemble d’expériences pourrait très bien être utilisé pour délimiter les attributs fonctionnels des chaperons d’histones non caractérisés auparavant de n’importe quel organisme.
La première partie de la section du protocole consiste à étudier l’état oligomère et la stabilité d’un chaperon d’histone. Plusieurs rapports indiquent que les chaperons d’histones présentent une diversité considérable dans leur état oligomère. Par exemple, CAF-1 humain existe en tant que monomère29. Les membres de la famille NAP existent sous forme de dimère ou de tétramère 29,30,31. Les nucléoplasmines révèlent des états oligomères pentamères et souvent décamoriques32,33. Une expérience analytique SEC peut déterminer l’état oligomère d’un chaperon d’histones, et les expériences ASC-SV peuvent confirmer la même chose. Plusieurs des chaperons d’histones sont connus pour être très stables dans diverses conditions de stress thermique et chimique33,34. Les caractéristiques de stabilité thermique et chimique des chaperons d’histones pourraient également être explorées conjointement avec l’analyse SEC. En outre, la spectroscopie de dichroïsme circulaire pourrait être utilisée efficacement pour une analyse approfondie des changements dans la structure secondaire du chaperon lorsqu’il est soumis à des températures croissantes ou à des concentrations plus élevées d’un agent chimique.
La deuxième partie de la section du protocole couvre les essais de pull-down qui pourraient examiner les interactions fondamentales qui facilitent l’association des oligomères d’histones avec le chaperon en utilisant une approche de gradient de sel et un test de pull-down compétitif pour identifier la préférence d’oligomère d’histones d’un chaperon. Si le complexe se désagrège avec une légère augmentation de la concentration en sel, cela suggérerait une contribution majeure des interactions électrostatiques dans la stabilisation du complexe. Un complexe intact à haute teneur en sel suggérerait un rôle significatif pour l’hydrophobicité dans la stabilisation du complexe35. Le test de pull-down compétitif pourrait être facilement utilisé pour déterminer la spécificité ou la préférence d’un chaperon d’histones par rapport à une classe spécifique d’oligomères d’histones. En fonction de leur préférence pour les oligomères d’histones, les chaperons d’histones peuvent être classés en trois catégories telles que les chaperons H2A/H2B, les chaperons H3/H4 et les chaperons H2A/H2B-H3/H410,36. De plus, si nécessaire, la calorimétrie de titrage isotherme (ITC) pourrait être utilisée pour comprendre la spécificité des oligomères d’histones d’un chaperon donné et les caractéristiques thermodynamiques de leurs interactions.
La troisième partie de la section du protocole couvre l’étude de la stœchiométrie d’interaction entre un chaperon d’histones et les oligomères d’histones. En général, les différentes familles d’histones chaperones diffèrent considérablement par la stœchiométrie de leur association avec H2A/H2B ou/et H3/H4 21,28,37,38. L’expérience ASC-SV aide à obtenir les coefficients de sédimentation (s) et la masse moléculaire d’une protéine ou de son complexe, ce qui devient très utile pour estimer avec précision la stœchiométrie dans la formation complexe. Alternativement, ITC peut également être utilisé pour examiner la stœchiométrie.
La quatrième partie de la section du protocole se concentre sur l’étude de la fonction d’assemblage des nucléosomes des chaperons d’histones. Les chaperons d’histones jouent un rôle crucial dans l’assemblage des nucléosomes, qui régule les processus cellulaires vitaux tels que la réplication, la transcription et la réparation de l’ADN39. Le test de superenroulement plasmidique qui est généralement utilisé pour l’évaluation in vitro de l’activité chaperonnante des histones chaperones est développé dans cette section.
On peut noter que tous les chaperons d’histones ne sont pas entièrement structurés. Peu sont connus pour avoir des régions intrinsèquement désordonnées40,41. Par conséquent, les tests de stabilité thermique et chimique peuvent ne pas convenir à ces protéines. De plus, les chaperons d’histones de différents organismes ont des états oligomères différents et des capacités différentielles pour se lier aux histones. Par conséquent, ce protocole peut être un bon point de départ, mais impliquerait des modifications si nécessaire.
Déclarations de divulgation
Aucun conflit d’intérêts n’a été déclaré.
Remerciements
Les subventions extra-muros accordées à Dileep Vasudevan par le Science and Engineering Research Board, gouvernement indien [CRG/2018/000695/PS] et le Département de la biotechnologie, ministère de la Science et de la Technologie, gouvernement de l’Inde [BT/INF/22/SP33046/2019], ainsi que le soutien intra-muros de l’Institut des sciences de la vie, Bhubaneswar, sont grandement appréciés. Nous remercions Mme Sudeshna Sen et Mme Annapurna Sahoo pour leur aide dans la préparation des histones. Les discussions avec nos collègues, le Dr Chinmayee Mohapatra, M. Manas Kumar Jagdev et le Dr Shaikh Nausad Hossain, sont également reconnues.
matériels
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acrylamide | MP Biomedicals | 814326 | |
| Agarose | MP Biomedicals | 193983 | |
| AKTA Pure 25M FPLC | Cytiva | 29018226 | Instrument for protein purification |
| Ammonium persulfate (APS) | Sigma | A3678 | |
| An-60Ti rotor | Beckman Coulter | 361964 | Rotor for analytical ultracentrifugation |
| Bovine serum albumin (BSA) | Sigma | A7030 | |
| Chloroform | Sigma | C2432 | |
| Coomassie brilliant blue R 250 | Sigma | 1.15444 | |
| Dialysis tubing (7 kDa cut-off) | Thermo Fisher | 68700 | For dialysing protein samples |
| Dithiothreitol (DTT) | MP Biomedicals | 100597 | |
| DNA Loading Dye | New England Biolabs | B7025S | |
| EDTA disodium salt | MP Biomedicals | 194822 | |
| Electronic balance | Shimadzu | ATX224R | |
| Ethanol | Sigma | E7023 | |
| Ethidium bromide (EtBr) | Sigma | E8751 | |
| Gel Doc System | Bio-Rad | 12009077 | For imaging gels after staining |
| Horizontal gel electrophoresis apparatus | Bio-Rad | 1704405 | Instrument for agarose gel electrophoresis |
| Hydrochloric acid (HCl) | Sigma | 320331 | |
| Imidazole | MP Biomedicals | 102033 | |
| Magnesium chloride (MgCl2) | Sigma | M8266 | |
| Micropipettes | Eppendorf | Z683779 | For pipetting of micro-volumes |
| Mini-PROTEAN electrophoresis system | Bio-Rad | 1658000 | Instrument for SDS-PAGE |
| N,N-methylene-bis-acrylamide | MP Biomedicals | 800172 | |
| Nano drop | Thermo Fisher | ND-2000 | For measurement of protein and DNA concentrations |
| Ni-NTA agarose | Invitrogen | R901-15 | Resin beads for pull-down assay |
| Optima AUC analytical ultracentrifuge | Beckman Coulter | B86437 | Instrument for analytical ultracentrifugation |
| pH Meter | Mettler Toledo | MT30130863 | |
| Phenol | Sigma | P4557 | |
| Plasmid isolation kit | Qiagen | 27104 | |
| Proteinase K | Sigma-Aldrich | 1.07393 | |
| pUC19 | Thermo Fisher | SD0061 | Plasmid for supercoiling assay |
| Refrigerated high-speed centrifuge | Thermo Fisher | 75002402 | |
| SDS-PAGE protein marker | Bio-Rad | 1610317 | |
| SEDFIT | Free software program for analytical ultracentrifugation data analysis | ||
| SEDNTERP | Free software program to estimate viscosity and density of buffer and partial specific volume of a protein | ||
| SigmaPrep Spin Columns | Sigma | SC1000 | For pull-down assay |
| Sodium acetate | Sigma | S2889 | |
| Sodium chloride (NaCl) | Merck | S9888 | |
| Sodium dodecyl sulfate (SDS) | MP Biomedicals | 102918 | |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | Column for analytical size-exclusion chromatography |
| Superdex 75 Increase 10/300 GL | Cytiva | 29148721 | Column for analytical size-exclusion chromatography |
| TEMED | Sigma | 1.10732 | |
| Topoisomerase I | Inspiralis | WGT102 | Enzyme used in plasmid supercoiling assay |
| Tris base | Merck | T1503 | |
| Tween-20 | Sigma | P1379 | |
| Urea | MP Biomedicals | 191450 | |
| Water bath | Nüve | NB 5 | For heat treatment of protein samples |
| β-mercaptoethanol (β-ME) | Sigma | M6250 |
Références
- Hübner, M. R., Eckersley-Maslin, M. A., Spector, D. L. Chromatin organization and transcriptional regulation. Current Opinion in Genetics and Development. 23 (2), 89-95 (2013).
- Lai, W. K. M., Pugh, B. F. Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nature Reviews Molecular Cell Biology. 18 (9), 548-562 (2017).
- Kim, U. J., Han, M., Kayne, P., Grunstein, M. Effects of histone H4 depletion on the cell cycle and transcription of Saccharomyces cerevisiae. EMBO Journal. 7 (7), 2211-2219 (1988).
- Prado, F., Aguilera, A. Partial depletion of histone H4 increases homologous recombination-mediated genetic instability. Molecular and Cellular Biology. 25 (4), 1526-1536 (2005).
- Meeks-Wagner, D., Hartwell, L. H. Normal stoichiometry of histone dimer sets is necessary for high fidelity of mitotic chromosome transmission. Cell. 44 (1), 43-52 (1986).
- Groth, A., et al. Human Asf1 regulates the flow of S phase histones during replicational stress. Molecular Cell. 17 (2), 301-311 (2005).
- Laskey, R. A., Honda, B. M., Mills, A. D., Finch, J. T. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA. Nature. 275 (5679), 416-420 (1978).
- Das, C., Tyler, J. K., Churchill, M. E. A. The histone shuffle: histone chaperones in an energetic dance. Trends in Biochemical Sciences. 35 (9), 476-489 (2010).
- Akey, C. W., Luger, K. Histone chaperones and nucleosome assembly. Current Opinion in Structural Biology. 13 (1), 6-14 (2003).
- De Koning, L., Corpet, A., Haber, J. E., Almouzni, G. Histone chaperones: An escort network regulating histone traffic. Nature Structural and Molecular Biology. 14 (11), 997-1007 (2007).
- Eitoku, M., Sato, L., Senda, T., Horikoshi, M. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly. Cellular and Molecular Life Sciences. 65 (3), 414-444 (2008).
- Quivy, J. P., Grandi, P., Almouzni, G. Dimerization of the largest subunit of chromatin assembly factor 1: importance in vitro and during Xenopus early development. EMBO Journal. 20 (8), 2015-2027 (2001).
- Ray-Gallet, D., et al. HIRA is critical for a nucleosome assembly pathway independent of DNA synthesis. Molecular Cell. 9 (5), 1091-1100 (2002).
- Frehlick, L. J., Eirín-López, J. M., Ausió, J. New insights into the nucleophosmin/nucleoplasmin family of nuclear chaperones. Bioessays. 29 (1), 49-59 (2007).
- Ito, T., Bulger, M., Kobayashi, R., Kadonaga, J. T. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays. Molecular and Cellular Biology. 16 (6), 3112-3124 (1996).
- Elsässer, S. J., D'Arcy, S. Towards a mechanism for histone chaperones. Biochimica et Biophysica Acta. 1819 (3-4), 211-221 (2013).
- Rodríguez-Campos, A., Koop, R., Faraudo, S., Beato, M. Transcriptionally competent chromatin assembled with exogenous histones in a yeast whole cell extract. Nucleic Acids Research. 32 (13), 111(2004).
- Levenstein, M. E., Kadonaga, J. T. Biochemical analysis of chromatin containing recombinant Drosophila core histones. Journal of Biological Chemistry. 277 (10), 8749-8754 (2002).
- Huang, S., et al. Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing. Proceedings of the National Academy of Sciences of the United States of America. 102 (38), 13410-13415 (2005).
- Swaminathan, V., Kishore, A. H., Febitha, K. K., Kundu, T. K. Human histone chaperone nucleophosmin enhances acetylation-dependent chromatin transcription. Molecular and Cellular Biology. 25 (17), 7534-7545 (2005).
- Singh, A. K., Datta, A., Jobichen, C., Luan, S., Vasudevan, D. AtFKBP53: A chimeric histone chaperone with functional nucleoplasmin and PPIase domains. Nucleic Acids Research. 48 (3), 1531-1550 (2020).
- Scofield, B. T. K. H. Protein Electrophoresis. , (2012).
- Andrew, S. M., Titus, J. A., Zumstein, L. Dialysis and concentration of protein solutions. Current Protocols in Toxicology, Appendix 3. , 1-5 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. Journal of Visualized Experiments. (33), e1530(2009).
- Padavannil, A., Brautigam, C. A., Chook, Y. M. Molecular size analysis of recombinant importin-histone complexes using analytical ultracentrifugation. Bio-protocol. 10 (10), 3625(2019).
- Zhao, H., Brautigam, C. A., Ghirlando, R., Schuck, P. Overview of current methods in sedimentation velocity and sedimentation equilibrium analytical ultracentrifugation. Current Protocols in Protein Science. , Chapter 20, Unit20.12 (2013).
- Schuck, P. Size-distribution analysis of macromolecules by sedimentation velocity ultracentrifugation and Lamm equation modelling. Biophysical Journal. 78 (3), 1606-1619 (2000).
- Kumar, A., Kumar Singh, A., Chandrakant Bob de, R., Vasudevan, D. Structural characterization of Arabidopsis thaliana NAP1-related protein 2 (AtNRP2) and comparison with its homolog AtNRP1. Molecules. 24 (12), 2258(2019).
- Liu, W. H., Roemer, S. C., Port, A. M., Churchill, M. E. A. CAF-1-induced oligomerization of histones H3/H4 and mutually exclusive interactions with Asf1 guide H3/H4 transitions among histone chaperones and DNA. Nucleic Acids Research. 45 (16), 9809(2017).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Newman, E. R., et al. Large multimeric assemblies of nucleosome assembly protein and histones revealed by small-angle X-ray scattering and electron microscopy. Journal of Biological Chemistry. 287 (32), 26657-26665 (2012).
- Edlich-Muth, C., et al. The pentameric nucleoplasmin fold is present in Drosophila FKBP39 and a large number of chromatin-related proteins. Journal of Molecular Biology. 427 (10), 1949-1963 (2015).
- Franco, A., et al. Structural insights into the ability of nucleoplasmin to assemble and chaperone histone octamers for DNA deposition. Scientific Reports. 9 (1), 9487(2019).
- Xiao, H., Jackson, V., Lei, M. The FK506-binding protein, Fpr4, is an acidic histone chaperone. FEBS Letters. 580 (18), 4357-4364 (2006).
- Graziano, G. Role of hydrophobic effect in the salt-induced dimerization of bovine beta-lactoglobulin at pH 3. Biopolymers. 91 (12), 1182-1188 (2009).
- Burgess, R. J., Zhang, Z. Histone chaperones in nucleosome assembly and human disease. Nature Structural and Molecular Biology. 20 (1), 14-22 (2013).
- Donham, D. C., Scorgie, J. K., Churchill, M. E. The activity of the histone chaperone yeast Asf1 in the assembly and disassembly of histone H3/H4-DNA complexes. Nucleic Acids Research. 39 (13), 5449-5458 (2011).
- Avvakumov, N., Nourani, A., Côté, J. Histone chaperones: Modulators of chromatin marks. Molecular Cell. 41 (5), 502-514 (2011).
- Ransom, M., Dennehey, B. K., Tyler, J. K. Chaperoning histones during DNA replication and repair. Cell. 140 (2), 183-195 (2010).
- Chu, X., et al. Importance of electrostatic interactions in the association of intrinsically disordered histone chaperone Chz1 and histone H2A.Z-H2B. PLoS Computational Biology. 8 (7), 1002608(2012).
- Heidarsson, P. O., et al. Disordered proteins enable histone chaperoning on the nucleosome. bioRxiv. , (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon