Method Article
In vitro Caratterizzazione degli Chaperoni degli istoni mediante saggi analitici, pull-down e chaperoning
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive una batteria di metodi che include la cromatografia analitica di esclusione dimensionale per studiare l'oligomerizzazione e la stabilità dell'istone chaperone, il saggio pull-down per svelare le interazioni chaperone-istone degli istoni, l'AUC per analizzare la stechiometria dei complessi proteici e il saggio di chaperoning degli istoni per caratterizzare funzionalmente un presunto chaperone istonico in vitro.
Abstract
Le proteine istoniche si associano al DNA per formare la cromatina eucariotica. L'unità di base della cromatina è un nucleosoma, costituito da un ottamero istonico costituito da due copie degli istoni H2A, H2B, H3 e H4, avvolti dal DNA. L'ottamero è composto da due copie di un dimero H2A/H2B e una singola copia di un tetramero H3/H4. Gli istoni del nucleo altamente carichi sono soggetti a interazioni non specifiche con diverse proteine nel citoplasma cellulare e nel nucleo. Gli chaperoni istonici formano una classe diversificata di proteine che trasportano gli istoni dal citoplasma al nucleo e aiutano la loro deposizione sul DNA, assistendo così il processo di assemblaggio del nucleosoma. Alcuni chaperoni istonici sono specifici per H2A / H2B o H3 / H4, e alcuni funzionano come chaperoni per entrambi. Questo protocollo descrive come le tecniche di laboratorio in vitro come i saggi pull-down, la cromatografia analitica ad esclusione dimensionale, l'ultra-centrifugazione analitica e il test di chaperoning degli istoni potrebbero essere utilizzate in tandem per confermare se una data proteina è funzionale come chaperone istonico.
Introduzione
I nucleosomi composti da DNA e proteine istoniche formano l'unità strutturale della cromatina e regolano diversi eventi cellulari critici. I nucleosomi vengono riposizionati e rimodellati dinamicamente per rendere il DNA accessibile a vari processi come la replicazione, la trascrizione e la traduzione 1,2. Gli istoni altamente basici tendono ad interagire con le proteine acide nell'ambiente cellulare o subiscono aggregazione, portando così a vari difetti cellulari 3,4,5. Un gruppo di proteine dedicate chiamate chaperoni degli istoni aiuta il trasporto degli istoni dal citoplasma al nucleo e previene eventi aberranti di aggregazione istone-DNA 6,7. Fondamentalmente, la maggior parte degli chaperoni degli istoni immagazzinano e trasferiscono gli istoni sul DNA a forza ionica fisiologica, favorendo così la formazione dei nucleosomi 8,9. Alcuni chaperoni istonici hanno una netta preferenza per gli oligomeri istonici H2A/H2B o H3/H410.
Gli chaperoni istonici sono caratterizzati in base alla loro capacità di assemblare nucleosomi dipendenti o indipendenti dalla sintesi del DNA11. Ad esempio, il fattore di assemblaggio della cromatina-1 (CAF-1) è dipendente mentre il regolatore degli istoni A (HIRA) è indipendente dalla sintesi del DNA12,13. Allo stesso modo, la famiglia nucleoplasmina degli chaperoni istonici è coinvolta nella decondensazione della cromatina spermatica e nell'assemblaggio dei nucleosomi14. I membri della famiglia delle proteine di assemblaggio dei nucleosomi (NAP) facilitano la formazione di strutture simili ai nucleosomi in vitro e sono coinvolti nello spostamento degli istoni tra citoplasma e nucleo15. Le nucleoplasmine e le proteine della famiglia NAP sono entrambe chaperoni funzionali degli istoni ma non condividono alcuna caratteristica strutturale. Essenzialmente, nessuna singola caratteristica strutturale consente la classificazione di una proteina come chaperone istonico16. L'uso di saggi funzionali e biofisici insieme a studi strutturali funzionano meglio nella caratterizzazione degli accompagnatori degli istoni.
Questo lavoro descrive metodi biochimici e biofisici per caratterizzare una proteina come un chaperone istonico che aiuta l'assemblaggio del nucleosoma. In primo luogo, è stata effettuata una cromatografia analitica di esclusione dimensionale per analizzare lo stato oligomerico e la stabilità degli chaperoni istonici. Successivamente, è stato eseguito un test pull-down per determinare le forze motrici e la natura competitiva delle interazioni istone chaperone-istone. Tuttavia, i parametri idrodinamici di queste interazioni non hanno potuto essere calcolati con precisione utilizzando la cromatografia analitica di esclusione dimensionale a causa della forma della proteina e dei suoi complessi che influenzano la loro migrazione attraverso la colonna. Pertanto, è stata utilizzata l'ultracentrifugazione analitica, che fornisce proprietà macromolecolari in soluzione che includono il peso molecolare accurato, la stechiometria di interazione e la forma delle molecole biologiche. Studi precedenti hanno ampiamente utilizzato il test di chaperoning degli istoni in vitro per caratterizzare funzionalmente gli chaperoni istonici come yScS116 17, DmACF18, ScRTT106p19, HsNPM120. Il saggio di chaperoning degli istoni è stato utilizzato anche per caratterizzare funzionalmente le proteine come chaperoni istoniche.
Protocollo
1. Cromatografia analitica di esclusione dimensionale per chiarire lo stato oligomerico e la stabilità degli chaperoni istonici
- Analisi dello stato oligomerico degli chaperoni istonici
- Equilibrare una colonna analitica di cromatografia ad esclusione dimensionale (SEC) da 24 mL con un volume (CV) di 1,2 colonne, cioè 28,8 mL di tampone SEC degassato [20 mM di Tris-HCl (pH 7,5), 300 di mM NaCl e 1 mM di β-mercaptoetanolo (β-ME)] a 4 °C (vedere Tabella dei materiali).
NOTA: il tipo di colonna, la composizione del tampone e il pH del tampone possono essere selezionati in base alla proteina di interesse. Il volume di iniezione del campione non deve superare 500 μL per una colonna da 24 ml. Inoltre, la pressione della colonna deve essere mantenuta al di sotto di 5 MPa. - Da una soluzione madre proteica a concentrazione più elevata, preparare 500 μL di campione proteico 0,5 mg/ml in tampone SEC degassato e iniettarlo nella colonna pre-equilibrata utilizzando un ciclo di iniezione da 500 μL. Lasciare procedere la cromatografia ad una portata isocratica di 0,2-0,3 ml/min con il tampone SEC a 4 °C.
- Monitorare il profilo di eluizione della proteina misurando l'assorbanza a una lunghezza d'onda di 280 nm. Quando si tratta di proteine prive di residui aromatici, misurare l'assorbanza a 214 nm.
- Utilizzare il volume di eluizione della proteina per calcolare il suo peso molecolare approssimativo in kDa utilizzando la curva di calibrazione standard21.
NOTA: La curva di calibrazione viene preparata tracciando il volume di ritenzione delle proteine a peso molecolare noto rispetto al log dei rispettivi pesi molecolari (log Mr), eluito utilizzando la stessa colonna.
- Equilibrare una colonna analitica di cromatografia ad esclusione dimensionale (SEC) da 24 mL con un volume (CV) di 1,2 colonne, cioè 28,8 mL di tampone SEC degassato [20 mM di Tris-HCl (pH 7,5), 300 di mM NaCl e 1 mM di β-mercaptoetanolo (β-ME)] a 4 °C (vedere Tabella dei materiali).
- Analisi della stabilità termica degli chaperoni istonici
- Prelevare 500 μL di 0,5 mg/ml del campione proteico preparato in tampone SEC degassato (lo stesso utilizzato al punto 1.1.1) in singole provette per microcentrifuga e riscaldare ciascuna provetta ad una temperatura particolare compresa tra 20 °C e 90 °C (20 °C, 40 °C, 60 °C e 90 °C) per 10 minuti a bagnomaria.
- Successivamente, centrifugare i campioni trattati termicamente a 16.200 x g per 10 minuti a 4 °C, raccogliere il surnatante con una micropipetta e iniettare ciascun campione singolarmente utilizzando un circuito di iniezione da 500 μL nella colonna analitica, pre-equilibrato con il tampone SEC a 4 °C.
- Lasciare procedere la cromatografia ad una portata isocratica di 0,2-0,3 ml/min con il tampone SEC a 4 °C.
- Osservare la posizione e l'altezza dei picchi di eluizione e cercare la comparsa di picchi aggiuntivi per i diversi campioni.
- Analisi della stabilità chimica degli chaperoni istonici
- Per esaminare la stabilità salina degli chaperoni istoniche, incubare 500 μL di 0,5 mg/ml di campione proteico preparato in un tampone Tris [20 mM di Tris-HCl (pH 7,5) e 1 mM di β-ME] integrato con concentrazioni crescenti di NaCl (300 mM, 600 mM, 1 M, 1,5 M e 2 M) in provette di microcentrifuga separate per 30 minuti a 4 °C. Centrifugare i campioni a 16.200 x g per 10 minuti a 4 °C e conservare il surnatante.
- Successivamente, caricare i campioni proteici in diverse concentrazioni di NaCl singolarmente, utilizzando un ciclo di iniezione di 500 μL nella colonna analitica pre-equilibrato con 1,2 CV (28,8 ml) del rispettivo tampone contenente concentrazioni crescenti di NaCl a 4 °C.
- Lasciare che la cromatografia proceda ad una velocità di flusso isocratico di 0,2-0,3 mL/min con 1 CV (24 mL) del rispettivo tampone a 4 °C.
- Osservare la posizione e l'altezza dei picchi di eluizione e cercare la comparsa di picchi aggiuntivi per i diversi campioni.
- Allo stesso modo, per l'analisi di stabilità dell'urea, incubare 500 μL di 0,5 mg/ml di campione proteico preparato in un tampone Tris [20 mM di Tris-HCl (pH 7,5) e 1 mM di β-ME] integrato con concentrazioni crescenti di urea (1 M, 2 M, 3 M, 4 M e 5 M) in provette di microcentrifuga separate per 16 ore a temperatura ambiente. Centrifugare i campioni a 16.200 x g per 10 minuti a temperatura ambiente e conservare il surnatante.
- Quindi, caricare i campioni di proteine trattate con urea singolarmente utilizzando un circuito di iniezione da 500 μL nella colonna analitica pre-equilibrata con 1,2 CV (28,8 ml) del tampone corrispondente contenente diverse concentrazioni di urea a temperatura ambiente.
- Lasciare che la cromatografia proceda ad una velocità di flusso isocratico di 0,2-0,3 mL/min con 1 CV (24 mL) del rispettivo tampone a temperatura ambiente.
ATTENZIONE: Non eseguire gli esperimenti con tampone contenente urea a temperatura inferiore poiché l'urea tende a cristallizzare e danneggiare la colonna. - Osservare la posizione e l'altezza dei picchi di eluizione e cercare la comparsa di picchi aggiuntivi per i diversi campioni.
2. Saggi di pull-down basati sul gradiente salino per comprendere il tipo di interazioni che contribuiscono alla complessa formazione tra oligomeri istonici e chaperone istonico
- Per ogni reazione di pull-down assay, pipettare 40 μL di resina Ni-NTA in una colonna di spin e lavare con acqua sterile bidistillata. Successivamente, equilibrare la resina con 100 CV (4 mL) di tampone di equilibrio [20 mM di Tris-HCl (pH 7,5), 300 mM di NaCl, 10 mM di imidazolo, 10 μg/mL di BSA e 1 mM di β-ME] (vedere Tabella dei materiali).
NOTA: Il pull-down può essere eseguito anche in una provetta da microcentrifuga da 1,5 ml. - Preparare il campione mescolando 5 μM dello chaperone istonico marcato con His con 20 μM di dimero H2A/H2B o tetramero H3/H4 nel tampone di equilibrio. Incubare il campione su ghiaccio per 1 ora.
NOTA: Il dimero H2A/H2B e il tetramero H3/H4 sono preparati da istoni umani ricombinanti21 e l'integrità degli oligomeri è confermata sulla base delle masse molecolari stimate mediante ultracentrifugazione analitica (AUC). Gli stessi oligomeri istonici sono stati utilizzati per tutti gli esperimenti menzionati di seguito. - Caricare i campioni in colonne di spin pre-equilibrate separate con resina Ni-NTA dal punto 2.1, ciascuna etichettata per una particolare concentrazione di sale, e mantenere le colonne per 30 minuti a 4 °C. Centrifugare le colonne a 1000 x g per 1 min.
- Quindi, lavare le colonne con 100 CV (4 ml) di tampone di lavaggio [20 mM di Tris-HCl (pH 7,5), 50 mM di imidazolo, 0,2% di Tween-20 e 1 mM di β-ME] contenenti diverse concentrazioni di sale (cioè 300 mM, 500 mM, 600 mM, 700 mM, 800 mM, 900 mM e 1 M NaCl). Lavare ogni colonna con un tampone avente una particolare concentrazione salina.
- Dopo la fase di lavaggio del sale, eluire la proteina dalle diverse colonne utilizzando 100 μL di tampone di eluizione [20 mM di Tris-HCl (pH 7,5), 300 mM di NaCl, 300 mM di imidazolo e 1 mM di β-ME].
- Successivamente, sottoporre i campioni eluiti al 18% SDS-PAGE22 e visualizzare il gel dopo la colorazione con Coomassie Brilliant Blue R250 (vedere Tabella dei materiali). In alternativa, è possibile caricare direttamente la resina sul gel SDS-PAGE invece di eluire la proteina legata dalla resina Ni-NTA.
NOTA: Le composizioni e il pH dei tamponi di equilibrazione, lavaggio ed eluizione possono essere modificati a seconda della proteina di interesse.
3. Saggio di pull-down competitivo per identificare la preferenza di un accompagnatore istonico per H2A/H2B o H3/H4
- Preparare la colonna di spin come descritto nel passaggio 2.1
- Incubare 5 μM dello chaperone istonico con 20 μM di dimero H2A/H2B in 300 μL di tampone di equilibrio (preparato nella fase 2.1) per 30 minuti su ghiaccio.
NOTA: Il rapporto tra oligomero istonico e chaperone istonico nella reazione può essere scelto in base ai dati noti di stechiometria di legame; Utilizzare l'istone in eccesso cinque volte se non sono disponibili informazioni. - Centrifugare il complesso chaperone-H2A/H2B degli istoni a 16.200 x g per 5 minuti a 4 °C per rimuovere qualsiasi precipitato. Quindi, caricare il campione sulla colonna di spin pre-bilanciata con buffer di equilibrio (preparato al punto 2.1) e incubare per 30 minuti a 4 °C.
- Lavare la colonna con 100 CV (4 ml) di tampone di lavaggio [20 mM di Tris-HCl (pH 7,5), 300 mM di NaCl, 50 mM di imidazolo, 0,2% di Tween-20 e 1 mM di β-ME] per rimuovere il dimero H2A/H2B in eccesso. Quindi, mescolare il complesso chaperone-H2A/H2B dell'istone con 20-60 μM di tetramero H3/H4 e incubare per 30 minuti su ghiaccio.
- Rilavare la colonna con 100 CV (4 ml) di tampone di lavaggio (preparato al punto 3.4) per rimuovere qualsiasi tetramero H3/H4 non legato ed eluire il campione utilizzando tampone di eluizione (preparato al punto 2.5). Sottoporre i campioni eluiti al 18% SDS-PAGE e visualizzarli dopo la colorazione con Coomassie Brilliant Blue R250.
NOTA: Il test potrebbe essere invertito in cui, in primo luogo, il tetramero H3 / H4 può essere miscelato con lo chaperone, il complesso può essere lasciato legare alle perle Ni-NTA e il complesso quindi essere incubato con concentrazioni variabili di dimero H2A / H2B.
4. Esperimenti analitici di ultracentrifugazione - velocità di sedimentazione (AUC-SV) per analizzare la stechiometria di legame tra chaperoni istonici e istoni
- Preparazione del campione per AUC
- Dializzare separatamente il dimero H2A/H2B dell'istone ricostituito, il tetramero H3/H4 e l'istone chaperone attraverso un tubo di dialisi cut-off 7 kDa 23, contro un tampone di dialisi [20 mM di Tris (pH 7,5), 300 mM di NaCl e 1 mM di β-ME] (vedere Tabella dei materiali). Per ridurre al minimo l'errore di fondo dovuto alla mancata corrispondenza del buffer, eseguire la dialisi estensivamente contro il buffer di dialisi, preferibilmente tre volte in un periodo di 24 ore.
NOTA: L'OD 280 iniziale dei campioni proteici dovrebbe avere un valore da due a tre volte superiore per ottenere un OD280 finale di 0,3-0,5. Questo viene essenzialmente fatto per annullare gli effetti della diluizione. - Purificare il dimero H2A/H2B, il tetramero H3/H4 e l'accompagnatore istonico singolarmente con il tampone di dialisi, utilizzando la cromatografia analitica di esclusione dimensionale (come menzionato nella fase 1). Salvare il buffer dall'esecuzione per preparare ulteriori diluizioni in seguito e utilizzarlo come riferimento nella cella AUC.
- Dializzare separatamente il dimero H2A/H2B dell'istone ricostituito, il tetramero H3/H4 e l'istone chaperone attraverso un tubo di dialisi cut-off 7 kDa 23, contro un tampone di dialisi [20 mM di Tris (pH 7,5), 300 mM di NaCl e 1 mM di β-ME] (vedere Tabella dei materiali). Per ridurre al minimo l'errore di fondo dovuto alla mancata corrispondenza del buffer, eseguire la dialisi estensivamente contro il buffer di dialisi, preferibilmente tre volte in un periodo di 24 ore.
- Caricamento del campione per AUC
- Mescolare le proteine purificate in un volume finale di 450 μL utilizzando tampone di dialisi dal punto 4.1.1 per raggiungere un OD280 di 0,3-0,6. Mescolare lo chaperone istonico con dimero H2A/H2B o tetramero H3/H4 per la formazione di complessi in provette di reazione separate. Incubare le miscele proteiche per 2-3 ore.
NOTA: In alternativa, i dati di sedimentazione possono essere acquisiti con un sistema di scansione ottica di interferenza nell'ultracentrifuga analitica. Separatamente, per mescolare proteine purificate, fissare la concentrazione di chaperone istonico e incubarla con concentrazioni crescenti degli oligomeri istonici per ottenere l'esatta stechiometria. - Assemblare la cella con un centrotavola a doppio settore e finestre di quarzo per l'esperimento AUC-SV utilizzando un rivelatore di assorbanza dell'ultracentrifuga analitica come descritto in precedenza nel dettaglio24.
- Riempire 400 μL della soluzione del campione e 420 μL di tampone di dialisi nei due settori (rispettivamente campione e settore di riferimento) della cellula.
NOTA: Nel settore di riferimento viene utilizzato un volume maggiore di tampone per mantenere il menisco di riferimento al di sopra del menisco del campione. Tuttavia, durante l'utilizzo di un sistema di interferenza ottica, riempire i due settori con lo stesso volume. - Pesare e bilanciare accuratamente le celle e caricarle in un rotore in titanio a quattro posti (vedere la tabella dei materiali). Allineare le celle utilizzando i segni forniti nella parte inferiore delle celle e del rotore. Caricare il rotore nella centrifuga, chiudere il coperchio e lasciare sviluppare un vuoto fino a quando la pressione scende a meno di 15 micron di Hg e la temperatura del rotore si stabilizza a 20 °C (di solito richiede 2-2,5 h).
NOTA: i parametri operativi AUC includono la temperatura sperimentale, la velocità del rotore, l'intervallo tra le scansioni e il numero di scansioni da raccogliere. Nel caso di esperimenti SV, l'intervallo di scansione è dato in base alla massa molecolare della proteina; Le proteine più piccole richiedono intervalli di tempo più ampi tra le scansioni. La velocità del rotore viene inoltre impostata in base alla massa molecolare attesa della proteina e l'esperimento viene condotto a 20 °C. I dati di assorbanza sono monitorati a 280 nm. - Per ottenere l'esatta stechiometria, mantenere costante la concentrazione di chaperone istonico e incubare con concentrazioni crescenti di oligomeri istonici per raggiungere la saturazione.
- Mescolare le proteine purificate in un volume finale di 450 μL utilizzando tampone di dialisi dal punto 4.1.1 per raggiungere un OD280 di 0,3-0,6. Mescolare lo chaperone istonico con dimero H2A/H2B o tetramero H3/H4 per la formazione di complessi in provette di reazione separate. Incubare le miscele proteiche per 2-3 ore.
- Analisi dei dati AUC
- Eseguire l'analisi dei dati come descritto in precedenza25. In breve, calcolare la densità e la viscosità per i componenti tampone utilizzando il programma SEDNTERP26 (vedi Tabella dei materiali). Allo stesso modo, calcolare il volume specifico parziale della proteina in base alla sua composizione aminoacidica, utilizzando anche SEDNTERP.
- Caricare i dati dalla macchina AUC nel programma SEDFIT27 e definire il menisco (linea rossa), il fondo della cella (linea blu) e i limiti di analisi dei dati (linee verdi). Scegliere la distribuzione continua C(s) come modello.
- Quindi, impostare la risoluzione massima fino a 100; coefficiente di sedimentazione (s), s min: 0 e s max: 10-15; impostare inizialmente il rapporto di attrito su 1,2 e scegliere di galleggiare per ricavare il rapporto dai dati; impostare il livello di confidenza (rapporto F; che determina l'entità della regolarizzazione) a 0,68 per la regolarizzazione 1 sigma; impostare i valori di volume specifico parziale, densità del tampone e viscosità del tampone ottenuti da SEDNTERP.
- Premere RUN per consentire al software di risolvere l'equazione di Lamm27. Regolare i parametri in caso di mancata corrispondenza dei dati in caso di significativa discrepanza. Dopo aver regolato i parametri, premere FIT per perfezionare tutti i parametri. Valutare la qualità dell'adattamento in base al valore della deviazione quadratica media (RMSD), che deve essere inferiore a 0,01 unità di segnale.
- Stimare le masse molecolari dei picchi scegliendo l'opzione: mostra il picco "Mw in c(s)" nella funzione di visualizzazione della barra degli strumenti principale, che fornirà informazioni sulla 's' della molecola / complesso.
5. Saggio di superavvolgimento plasmidico per confermare la funzione di chaperoning degli istoni
- Reazione di assemblaggio dei nucleosomi
- Mescolare 2 μM di tetramero H3/H4 e 4 μM di dimero H2A/H2B con concentrazioni crescenti dello chaperone istonico (1-6 μM) in un tampone di assemblaggio [20 mM di Tris HCl (pH 7,5), 1 mM di DTT, 1 mM di MgCl2, 0,1 mg/ml di BSA e 100 mM di NaCl] fino ad un volume finale di 50 μL. Incubare la miscela a 4 °C per 30 minuti.
- Contemporaneamente, in una reazione separata, pretrattare 500 ng del plasmide pUC19 sovraarrotolato negativamente con 1 μg di enzima topoisomerasi I (vedere Tabella dei materiali) nel tampone di assemblaggio in un volume finale di 50 μL e incubare a 30 °C per 30 minuti.
NOTA: La topoisomerasi I rilassa il DNA plasmidico a doppio filamento superarrotolato generando un nick a singolo filamento. Potrebbe essere utilizzato un enzima topoisomerasi I di origine eucariotica, come la topoisomerasi I del germe di grano disponibile in commercio o la Drosophila melanogaster topoisomerasi I espressa in modo ricombinante. - Quindi, combinare il tetramero H3/H4, il dimero H2A/H2B, la miscela di chaperone istonico (dal punto 5.1.1), la miscela di reazione del DNA plasmidico rilassato (dal punto 5.1.2) e incubare ulteriormente a 30 °C per 90 minuti.
NOTA: impostare due reazioni di controllo per il test; uno con l'istone chaperone e il DNA plasmidico rilassato (ma non gli istoni) e l'altro con gli oligomeri istonici e il DNA plasmidico rilassato (ma non l'istone chaperone). - Interrompere la reazione di assemblaggio aggiungendo 100 μL di tampone di arresto 2x (40 mM di EDTA, 2% di SDS e 0,4 mg/ml di proteinasi K) e incubare a 37 °C per 30 minuti.
NOTA: Stop buffer deproteinizza il DNA plasmidico mediante denaturazione e proteolisi degli istoni legati.
- Estrazione fenolo-cloroformio e precipitazione di etanolo
- Aggiungere un volume uguale di fenolo saturo di Tris nella provetta contenente la miscela di reazione del punto 5.1.4 e mescolare bene, seguita da centrifugazione a 16.200 x g per 10 minuti a temperatura ambiente.
- Raccogliere delicatamente la fase acquosa superiore avendo il DNA plasmidico con una micropipetta e mescolare con un volume uguale di cloroformio. Vortice la miscela e centrifugare a 16.200 x g per 10 minuti a temperatura ambiente.
NOTA: l'alcol isoamilico potrebbe essere incluso in questa fase per evitare un'interfaccia sfocata tra le fasi acquosa e organica. - Quindi, raccogliere la fase acquosa superiore, aggiungere 1/10 del volume di acetato di sodio 3 M (pH 5,5) e 2,5 volumi di etanolo ghiacciato (vedi Tabella dei materiali). Mescolare bene la soluzione capovolgendo il tubo 3-4 volte e conservare la miscela in un congelatore a -20 °C per 30 minuti per la completa precipitazione del DNA plasmide.
- Centrifugare il campione dal punto 5.2.3 a 16200 x g per 10 minuti ed eliminare delicatamente il surnatante. Tenere i tubi aperti a temperatura ambiente fino a quando anche tracce di etanolo evaporano, lasciando il DNA plasmidico precipitato nel tubo.
- Eseguire l'elettroforesi su gel di agarosio per osservare l'effetto superavvolgente plasmide.
- Sciogliere il DNA plasmidico precipitato dal punto 5.2.4 in acqua sterile bidistillata.
- Risolvere i campioni su un gel di agarosio all'1% in 1x tampone Tris-acetato-EDTA (TAE) (40 mM di Tris, 20 mM di acido acetico e 1 mM di EDTA) (vedere Tabella dei materiali).
- Colorare il gel con una concentrazione di 0,2-0,5 μg/mL di bromuro di etidio e osservare sotto UV per visualizzare le bande di DNA sul gel.
Risultati
Il dominio nucleoplasmina N-terminale ricombinante della proteina FKBP53 da Arabidopsis thaliana è stato sottoposto a SEC analitico. Il volume di picco dell'eluizione è stato tracciato rispetto alla curva standard per identificare il suo stato oligomerico. I risultati analitici del SEC hanno rivelato che il dominio esiste come pentamero in soluzione, con una massa molecolare approssimativa di 58 kDa (Figura 1A,B). Inoltre, il dominio della nucleoplasmina è stato analizzato per la stabilità termica e chimica in combinazione con SEC analitico. Il campione di nucleoplasmina sottoposto a trattamento termico fino a 90 °C non ha mostrato alcuno spostamento apparente nel volume di eluizione e nell'altezza di picco rispetto ai campioni mantenuti a 20 °C, suggerendo che il dominio è altamente termostabile (Figura 1C). Allo stesso modo, il dominio della nucleoplasmina mostrava stabilità salina fino a 2 M di NaCl (Figura 1D) e stabilità dell'urea fino a 4 M (Figura 1E). Tuttavia, il pentamero nucleoplasmina ha iniziato a dissociarsi in concentrazioni di urea più elevate.
È stato eseguito un test pull-down per determinare il tipo di interazioni che contribuiscono alla complessa formazione tra lo chaperone istonico (dominio nucleoplasmina di AtFKBP53) e gli oligomeri istonici H2A/H2B dimero e H3/H4 tetramero utilizzando un gradiente di lavaggio a sale. L'interazione del dominio della nucleoplasmina con il dimero H2A/H2B era stabile fino a una concentrazione salina di 0,4 M NaCl (Figura 2A). In confronto, l'associazione con H3/H4 era ragionevolmente stabile fino a 0,7 M NaCl (Figura 2B). La capacità dei complessi chaperone-istone di resistere ad un'alta concentrazione salina suggerisce il ruolo delle interazioni idrofobiche nella stabilizzazione dei complessi. Il complesso chaperone con H3/H4 stabile anche in alte concentrazioni di sale suggerisce un ruolo predominante delle interazioni idrofobiche nella formazione del complesso. La minore stabilità del complesso H2A/H2B-chaperone in alte concentrazioni di sale rivela un ruolo significativo per le interazioni elettrostatiche nella formazione del complesso. In un altro esperimento, il test pull-down è stato utilizzato per esaminare se l'accompagnatore preferisce il dimero H2A / H2B o il tetramero H3 / H4. I risultati hanno rivelato che lo chaperone si lega al dimero H2A/H2B e al tetramero H3/H4 simultaneamente e indipendentemente dall'ordine in cui vengono aggiunti al chaperone (Figura 2C,D). Ciò indica che lo chaperone possiede siti separati per la sua interazione con gli oligomeri istoniche.
Sono stati condotti esperimenti AUC-SV (Figura 3) per studiare la stechiometria di interazione tra oligomeri istonici e chaperoni. L'analisi dei dati AUC-SV ha fornito un valore del coefficiente di sedimentazione (s) di 5,40 S per il dominio della nucleoplasmina AtFKBP53 in complesso con H2A/H2B che corrispondeva a una massa molecolare di 104 kDa. Il complesso del dominio nucleoplasmina con H3/H4 ha dato un valore del coefficiente di sedimentazione di 7,35 S, corrispondente a 129 kDa. La massa molecolare stimata dei complessi rivela che la nucleoplasmina pentamerica forma un complesso con dimero H2A/H2B e tetramero H3/H4 in una stechiometria 1:1.
È essenziale dimostrare che la proteina può depositare oligomeri istonici sul DNA per confermare che si tratta di un chaperone istonico. A tal fine, è stato adottato un saggio di superavvolgimento plasmidico (Figura 4). Il plasmide circolare rilassato è stato incubato con gli oligomeri istonici H2A/H2B e H3/H4 con gli chaperoni istonici vegetali ricombinanti della famiglia NAP - AtNRP1 e AtNRP228. La presenza dello chaperone ha aumentato la quantità di plasmide superarrotolato, suggerendo che potrebbe depositare istoni sul DNA per formare nucleosomi, causando il superavvolgimento del DNA.

Figura 1: Stato oligomerico e stabilità del dominio nucleoplasmina di AtFBP53. (A) Profilo cromatografico analitico di esclusione dimensionale del dominio nucleoplasmina AtFKBP53. (B) Curva di taratura ottenuta utilizzando proteine globulari di massa molecolare nota. I punti blu rappresentano la massa molecolare delle proteine note, mentre il punto rosso rappresenta il dominio della nucleoplasmina AtFKBP53. (440 kDa - ferritina, 158 kDa-aldolasi, 75 kDa-con albumina, 44 kDa-ovalbumina, 6,5 kDa-aprotinina). (C) Cromatogramma analitico di esclusione dimensionale di 500 μL di dominio nucleoplasmina AtFKBP53 0,5 mg/mL sottoposto a trattamento termico a diverse temperature: 20 °C (verde), 40 °C (arancione), 60 °C (nero), 90 °C (azzurro). (D) Cromatogramma analitico di esclusione dimensionale di 500 μL di 0,5 mg/mL di dominio nucleoplasmina AtFKBP53 in tamponi contenenti diverse concentrazioni di NaCl: 0,3 M (viola), 0,6 M (rosso), 1,0 M (azzurro), 1,5 M (verde), 2,0 M (nero). (E) Cromatogramma analitico di esclusione dimensionale del dominio nucleoplasmina AtFKBP53 in tamponi con diverse concentrazioni di urea: 0 M (controllo; azzurro), 1,0 M (rosa), 2,0 M (nero), 3,0 M (blu scuro), 4,0 M (verde), 5,0 M (marrone). Il pentamero della nucleoplasmina mostra un'elevata stabilità alle condizioni di stress termico e chimico. La figura è adattata dal riferimento21. Fare clic qui per visualizzare una versione ingrandita di questa figura.
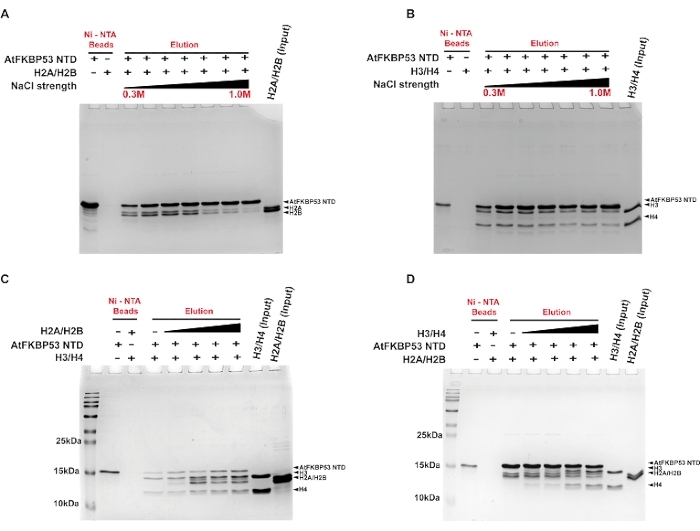
Figura 2: Saggi pull-down per l'interazione del dominio nucleoplasmina di AtFKBP53 con oligomeri istonici. Il 18% delle immagini SDS-PAGE delle frazioni di eluizione dei saggi sono presentate qui. Saggio pull-down per (A) dimero H2A/H2B da 20 μM e tetramero H3/ H4 da 20 μM con dominio nucleoplasmina AtFKBP53 da 5 μM in concentrazioni crescenti di NaCl nell'intervallo 0,3 M, 0,5 M, 0,6 M, 0,7 M, 0,8 M, 0,9 M e 1,0 M. 5 μM AtFKBP53 FKBD è stato utilizzato come controllo negativo qui. Per l'esperimento di legame competitivo, ( C ) è stata utilizzata una miscela di 5 μM di dominio nucleoplasmina AtFKBP53 e tetramero H3/H4 da 20 μM incubato con un intervallo di dimero H2A/H2B da 20-60 μM e (D) una miscela di 5 μM di dominio nucleoplasmina AtFKBP53 e dimero H2A/H2B da 20 μM incubato con un intervallo di 20-60 μM H3/H4 tetramero. L'etichetta AtFKBP53 corrisponde al dominio nucleoplasmina di AtFKBP53. Le frazioni di eluizione mostrano il legame simultaneo di entrambi gli oligomeri istonici alla nucleoplasmina. La figura è adattata dal riferimento21. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 3: Esperimento analitico ultracentrifugazione - velocità di sedimentazione (AUC-SV) di oligomeri istonici, il dominio nucleoplasmina di AtFKBP53 e i loro complessi. La distribuzione della distanza AUC vs. coefficiente di sedimentazione (S) grafico. Vengono inoltre forniti i valori del coefficiente o dei coefficienti di sedimentazione ottenuti e le masse molecolari. L'etichetta AtFKBP53 corrisponde al dominio nucleoplasmina di AtFKBP53. Le masse molecolari stimate rivelano una stechiometria 1:1 per il dominio della nucleoplasmina AtFKBP53 con gli oligomeri istonici H2A/H2B dimero e H3/H4 tetramero. 450 μL di tutti i campioni proteici con un OD280 di 0,3-0,5 sono stati utilizzati per gli esperimenti AUC-SV. La figura è adattata dal riferimento21. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Saggio di superavvolgimento plasmide. Saggio di superavvolgimento plasmidico per gli accompagnatori istonici AtNRP1 e AtNRP2. 500 ng di DNA plasmidico pUC19 sono stati pretrattati con 1 μg di topoisomerasi I per l'esperimento. 4 μM AtNRP1, 4 μM AtNRP2 e una miscela di dimero H2A/H2B da 4 μM e tetramero H3/H4 da 2 μM non mostrano attività di superavvolgimento quando incubati con il DNA pUC19 pretrattato. Le corsie con una miscela di 4 μM H2A/H2B di dimero e 2 μM H3/H4 di tetramero e 4 μM ciascuna di AtNRP1 e AtNRP2 mostrano la formazione di DNA superarrotolato dopo l'incubazione con il DNA pUC19 pretrattato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Questo lavoro dimostra e convalida una serie completa di protocolli per la caratterizzazione biochimica e biofisica di un presunto chaperone istonico. Qui, le proteine della famiglia NAP espresse e purificate in modo ricombinante, AtNRP1 e AtNRP2, e il dominio nucleoplasmina N-terminale di AtFKBP53 sono stati utilizzati per dimostrare i protocolli. Lo stesso insieme di esperimenti potrebbe benissimo essere usato per delineare gli attributi funzionali di chaperoni istonici precedentemente non caratterizzati da qualsiasi organismo.
La prima parte della sezione del protocollo riguarda lo studio dello stato oligomerico e della stabilità di un chaperone istonico. Diversi rapporti indicano che gli istone chaperones mostrano una notevole diversità nel loro stato oligomerico. Ad esempio, il CAF-1 umano esiste come monomero29. I membri della famiglia NAP esistono come dimero o tetramero 29,30,31. Le nucleoplasmine rivelano stati oligomerici pentamerici e spesso decamerici32,33. Un esperimento analitico SEC può determinare lo stato oligomerico di un chaperone istonico e gli esperimenti AUC-SV possono confermare lo stesso. Molti degli accompagnatori istonici sono noti per essere altamente stabili in varie condizioni di stress termico e chimico33,34. Le caratteristiche di stabilità termica e chimica degli chaperoni istonici potrebbero anche essere esplorate in combinazione con il SEC analitico. Inoltre, la spettroscopia di dicroismo circolare potrebbe essere efficacemente utilizzata per un'analisi approfondita dei cambiamenti nella struttura secondaria dello chaperone quando sottoposto a temperature crescenti o concentrazioni più elevate di un agente chimico.
La seconda parte della sezione del protocollo copre saggi pull-down che potrebbero esaminare le interazioni fondamentali che aiutano l'associazione degli oligomeri istonici con lo chaperone utilizzando un approccio a gradiente salino e un saggio di pull-down competitivo per identificare la preferenza oligomerica istonica di un chaperone. Se il complesso cade a pezzi con un leggero aumento della concentrazione di sale, ciò suggerirebbe un importante contributo delle interazioni elettrostatiche nella stabilizzazione del complesso. Un complesso intatto in alto sale suggerirebbe un ruolo significativo per l'idrofobicità nella stabilizzazione del complesso35. Il saggio di pull-down competitivo potrebbe essere facilmente impiegato per determinare la specificità o la preferenza di un chaperone istonico rispetto a una specifica classe di oligomeri istoniche. In base alla loro preferenza verso gli oligomeri istonici, gli chaperoni istonici possono essere classificati in tre categorie come chaperoni H2A/H2B, chaperoni H3/H4 e chaperoni H2A/H2B-H3/H4 10,36. Inoltre, se necessario, la calorimetria isotermica a titolazione (ITC) potrebbe essere utilizzata per comprendere la specificità degli oligomeri istonici di un dato chaperone e le caratteristiche termodinamiche delle loro interazioni.
La terza parte della sezione protocollo riguarda lo studio della stechiometria di interazione tra un chaperone istonico e gli oligomeri istoniche. In generale, le diverse famiglie di chaperoni istonici differiscono considerevolmente per la stechiometria della loro associazione con H2A/H2B e/e H3/H4 21,28,37,38. L'esperimento AUC-SV aiuta ad ottenere il coefficiente (s) di sedimentazione e la massa molecolare di una proteina o del suo complesso, che diventa molto utile per stimare accuratamente la stechiometria nella formazione del complesso. In alternativa, ITC può anche essere utilizzato per esaminare la stechiometria.
La quarta parte della sezione del protocollo si concentra sullo studio della funzione di assemblaggio dei nucleosomi degli accompagnatori degli istoni. Gli chaperoni istonici svolgono un ruolo cruciale nell'assemblaggio dei nucleosomi, che regola i processi cellulari vitali come la replicazione, la trascrizione e la riparazione del DNA39. In questa sezione viene elaborato il saggio di superavvolgimento plasmidico che viene tipicamente impiegato per la valutazione in vitro dell'attività di chaperoning degli istoni degli chaperoni istoniche.
Si può notare che non tutti gli accompagnatori degli istoni sono completamente strutturati. Pochi sono noti per avere regioni intrinsecamente disordinate40,41. Pertanto, i saggi di stabilità termica e chimica potrebbero non essere adatti per tali proteine. Inoltre, gli chaperoni istonici di diversi organismi hanno diversi stati oligomerici e capacità differenziali di legarsi agli istoni. Pertanto, questo protocollo può essere un buon punto di partenza, ma comporterebbe modifiche necessarie.
Divulgazioni
Non è stato dichiarato alcun conflitto di interessi.
Riconoscimenti
Le sovvenzioni extramurali a Dileep Vasudevan dal Science and Engineering Research Board, Government of India [CRG/2018/000695/PS] e dal Department of Biotechnology, Ministry of Science and Technology, Government of India [BT/INF/22/SP33046/2019], nonché il supporto intramurale dell'Institute of Life Sciences, Bhubaneswar sono ampiamente riconosciuti. Ringraziamo la signora Sudeshna Sen e la signora Annapurna Sahoo per il loro aiuto nella preparazione degli istoni. Sono state riconosciute anche le discussioni con i nostri colleghi Dr. Chinmayee Mohapatra, Mr. Manas Kumar Jagdev e Dr. Shaikh Nausad Hossain.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acrylamide | MP Biomedicals | 814326 | |
| Agarose | MP Biomedicals | 193983 | |
| AKTA Pure 25M FPLC | Cytiva | 29018226 | Instrument for protein purification |
| Ammonium persulfate (APS) | Sigma | A3678 | |
| An-60Ti rotor | Beckman Coulter | 361964 | Rotor for analytical ultracentrifugation |
| Bovine serum albumin (BSA) | Sigma | A7030 | |
| Chloroform | Sigma | C2432 | |
| Coomassie brilliant blue R 250 | Sigma | 1.15444 | |
| Dialysis tubing (7 kDa cut-off) | Thermo Fisher | 68700 | For dialysing protein samples |
| Dithiothreitol (DTT) | MP Biomedicals | 100597 | |
| DNA Loading Dye | New England Biolabs | B7025S | |
| EDTA disodium salt | MP Biomedicals | 194822 | |
| Electronic balance | Shimadzu | ATX224R | |
| Ethanol | Sigma | E7023 | |
| Ethidium bromide (EtBr) | Sigma | E8751 | |
| Gel Doc System | Bio-Rad | 12009077 | For imaging gels after staining |
| Horizontal gel electrophoresis apparatus | Bio-Rad | 1704405 | Instrument for agarose gel electrophoresis |
| Hydrochloric acid (HCl) | Sigma | 320331 | |
| Imidazole | MP Biomedicals | 102033 | |
| Magnesium chloride (MgCl2) | Sigma | M8266 | |
| Micropipettes | Eppendorf | Z683779 | For pipetting of micro-volumes |
| Mini-PROTEAN electrophoresis system | Bio-Rad | 1658000 | Instrument for SDS-PAGE |
| N,N-methylene-bis-acrylamide | MP Biomedicals | 800172 | |
| Nano drop | Thermo Fisher | ND-2000 | For measurement of protein and DNA concentrations |
| Ni-NTA agarose | Invitrogen | R901-15 | Resin beads for pull-down assay |
| Optima AUC analytical ultracentrifuge | Beckman Coulter | B86437 | Instrument for analytical ultracentrifugation |
| pH Meter | Mettler Toledo | MT30130863 | |
| Phenol | Sigma | P4557 | |
| Plasmid isolation kit | Qiagen | 27104 | |
| Proteinase K | Sigma-Aldrich | 1.07393 | |
| pUC19 | Thermo Fisher | SD0061 | Plasmid for supercoiling assay |
| Refrigerated high-speed centrifuge | Thermo Fisher | 75002402 | |
| SDS-PAGE protein marker | Bio-Rad | 1610317 | |
| SEDFIT | Free software program for analytical ultracentrifugation data analysis | ||
| SEDNTERP | Free software program to estimate viscosity and density of buffer and partial specific volume of a protein | ||
| SigmaPrep Spin Columns | Sigma | SC1000 | For pull-down assay |
| Sodium acetate | Sigma | S2889 | |
| Sodium chloride (NaCl) | Merck | S9888 | |
| Sodium dodecyl sulfate (SDS) | MP Biomedicals | 102918 | |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | Column for analytical size-exclusion chromatography |
| Superdex 75 Increase 10/300 GL | Cytiva | 29148721 | Column for analytical size-exclusion chromatography |
| TEMED | Sigma | 1.10732 | |
| Topoisomerase I | Inspiralis | WGT102 | Enzyme used in plasmid supercoiling assay |
| Tris base | Merck | T1503 | |
| Tween-20 | Sigma | P1379 | |
| Urea | MP Biomedicals | 191450 | |
| Water bath | Nüve | NB 5 | For heat treatment of protein samples |
| β-mercaptoethanol (β-ME) | Sigma | M6250 |
Riferimenti
- Hübner, M. R., Eckersley-Maslin, M. A., Spector, D. L. Chromatin organization and transcriptional regulation. Current Opinion in Genetics and Development. 23 (2), 89-95 (2013).
- Lai, W. K. M., Pugh, B. F. Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nature Reviews Molecular Cell Biology. 18 (9), 548-562 (2017).
- Kim, U. J., Han, M., Kayne, P., Grunstein, M. Effects of histone H4 depletion on the cell cycle and transcription of Saccharomyces cerevisiae. EMBO Journal. 7 (7), 2211-2219 (1988).
- Prado, F., Aguilera, A. Partial depletion of histone H4 increases homologous recombination-mediated genetic instability. Molecular and Cellular Biology. 25 (4), 1526-1536 (2005).
- Meeks-Wagner, D., Hartwell, L. H. Normal stoichiometry of histone dimer sets is necessary for high fidelity of mitotic chromosome transmission. Cell. 44 (1), 43-52 (1986).
- Groth, A., et al. Human Asf1 regulates the flow of S phase histones during replicational stress. Molecular Cell. 17 (2), 301-311 (2005).
- Laskey, R. A., Honda, B. M., Mills, A. D., Finch, J. T. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA. Nature. 275 (5679), 416-420 (1978).
- Das, C., Tyler, J. K., Churchill, M. E. A. The histone shuffle: histone chaperones in an energetic dance. Trends in Biochemical Sciences. 35 (9), 476-489 (2010).
- Akey, C. W., Luger, K. Histone chaperones and nucleosome assembly. Current Opinion in Structural Biology. 13 (1), 6-14 (2003).
- De Koning, L., Corpet, A., Haber, J. E., Almouzni, G. Histone chaperones: An escort network regulating histone traffic. Nature Structural and Molecular Biology. 14 (11), 997-1007 (2007).
- Eitoku, M., Sato, L., Senda, T., Horikoshi, M. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly. Cellular and Molecular Life Sciences. 65 (3), 414-444 (2008).
- Quivy, J. P., Grandi, P., Almouzni, G. Dimerization of the largest subunit of chromatin assembly factor 1: importance in vitro and during Xenopus early development. EMBO Journal. 20 (8), 2015-2027 (2001).
- Ray-Gallet, D., et al. HIRA is critical for a nucleosome assembly pathway independent of DNA synthesis. Molecular Cell. 9 (5), 1091-1100 (2002).
- Frehlick, L. J., Eirín-López, J. M., Ausió, J. New insights into the nucleophosmin/nucleoplasmin family of nuclear chaperones. Bioessays. 29 (1), 49-59 (2007).
- Ito, T., Bulger, M., Kobayashi, R., Kadonaga, J. T. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays. Molecular and Cellular Biology. 16 (6), 3112-3124 (1996).
- Elsässer, S. J., D'Arcy, S. Towards a mechanism for histone chaperones. Biochimica et Biophysica Acta. 1819 (3-4), 211-221 (2013).
- Rodríguez-Campos, A., Koop, R., Faraudo, S., Beato, M. Transcriptionally competent chromatin assembled with exogenous histones in a yeast whole cell extract. Nucleic Acids Research. 32 (13), 111(2004).
- Levenstein, M. E., Kadonaga, J. T. Biochemical analysis of chromatin containing recombinant Drosophila core histones. Journal of Biological Chemistry. 277 (10), 8749-8754 (2002).
- Huang, S., et al. Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing. Proceedings of the National Academy of Sciences of the United States of America. 102 (38), 13410-13415 (2005).
- Swaminathan, V., Kishore, A. H., Febitha, K. K., Kundu, T. K. Human histone chaperone nucleophosmin enhances acetylation-dependent chromatin transcription. Molecular and Cellular Biology. 25 (17), 7534-7545 (2005).
- Singh, A. K., Datta, A., Jobichen, C., Luan, S., Vasudevan, D. AtFKBP53: A chimeric histone chaperone with functional nucleoplasmin and PPIase domains. Nucleic Acids Research. 48 (3), 1531-1550 (2020).
- Scofield, B. T. K. H. Protein Electrophoresis. , (2012).
- Andrew, S. M., Titus, J. A., Zumstein, L. Dialysis and concentration of protein solutions. Current Protocols in Toxicology, Appendix 3. , 1-5 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. Journal of Visualized Experiments. (33), e1530(2009).
- Padavannil, A., Brautigam, C. A., Chook, Y. M. Molecular size analysis of recombinant importin-histone complexes using analytical ultracentrifugation. Bio-protocol. 10 (10), 3625(2019).
- Zhao, H., Brautigam, C. A., Ghirlando, R., Schuck, P. Overview of current methods in sedimentation velocity and sedimentation equilibrium analytical ultracentrifugation. Current Protocols in Protein Science. , Chapter 20, Unit20.12 (2013).
- Schuck, P. Size-distribution analysis of macromolecules by sedimentation velocity ultracentrifugation and Lamm equation modelling. Biophysical Journal. 78 (3), 1606-1619 (2000).
- Kumar, A., Kumar Singh, A., Chandrakant Bob de, R., Vasudevan, D. Structural characterization of Arabidopsis thaliana NAP1-related protein 2 (AtNRP2) and comparison with its homolog AtNRP1. Molecules. 24 (12), 2258(2019).
- Liu, W. H., Roemer, S. C., Port, A. M., Churchill, M. E. A. CAF-1-induced oligomerization of histones H3/H4 and mutually exclusive interactions with Asf1 guide H3/H4 transitions among histone chaperones and DNA. Nucleic Acids Research. 45 (16), 9809(2017).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Newman, E. R., et al. Large multimeric assemblies of nucleosome assembly protein and histones revealed by small-angle X-ray scattering and electron microscopy. Journal of Biological Chemistry. 287 (32), 26657-26665 (2012).
- Edlich-Muth, C., et al. The pentameric nucleoplasmin fold is present in Drosophila FKBP39 and a large number of chromatin-related proteins. Journal of Molecular Biology. 427 (10), 1949-1963 (2015).
- Franco, A., et al. Structural insights into the ability of nucleoplasmin to assemble and chaperone histone octamers for DNA deposition. Scientific Reports. 9 (1), 9487(2019).
- Xiao, H., Jackson, V., Lei, M. The FK506-binding protein, Fpr4, is an acidic histone chaperone. FEBS Letters. 580 (18), 4357-4364 (2006).
- Graziano, G. Role of hydrophobic effect in the salt-induced dimerization of bovine beta-lactoglobulin at pH 3. Biopolymers. 91 (12), 1182-1188 (2009).
- Burgess, R. J., Zhang, Z. Histone chaperones in nucleosome assembly and human disease. Nature Structural and Molecular Biology. 20 (1), 14-22 (2013).
- Donham, D. C., Scorgie, J. K., Churchill, M. E. The activity of the histone chaperone yeast Asf1 in the assembly and disassembly of histone H3/H4-DNA complexes. Nucleic Acids Research. 39 (13), 5449-5458 (2011).
- Avvakumov, N., Nourani, A., Côté, J. Histone chaperones: Modulators of chromatin marks. Molecular Cell. 41 (5), 502-514 (2011).
- Ransom, M., Dennehey, B. K., Tyler, J. K. Chaperoning histones during DNA replication and repair. Cell. 140 (2), 183-195 (2010).
- Chu, X., et al. Importance of electrostatic interactions in the association of intrinsically disordered histone chaperone Chz1 and histone H2A.Z-H2B. PLoS Computational Biology. 8 (7), 1002608(2012).
- Heidarsson, P. O., et al. Disordered proteins enable histone chaperoning on the nucleosome. bioRxiv. , (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon