Method Article
In vitro Caracterización de chaperonas de histonas mediante ensayos analíticos, pull-down y acompañantes
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe una batería de métodos que incluye cromatografía analítica de exclusión de tamaño para estudiar la oligomerización y estabilidad de la chaperona de histonas, ensayo de extracción hacia abajo para desentrañar las interacciones chaperona-histona de histonas, AUC para analizar la estequiometría de los complejos de proteínas y ensayo de acompañamiento de histonas para caracterizar funcionalmente una chaperona de histonas putativa in vitro.
Resumen
Las proteínas histonas se asocian con el ADN para formar la cromatina eucariota. La unidad básica de la cromatina es un nucleosoma, formado por un octámero de histonas que consiste en dos copias de las histonas centrales H2A, H2B, H3 y H4, envueltas por el ADN. El octámero está compuesto por dos copias de un dímero H2A/H2B y una sola copia de un tetrámero H3/H4. Las histonas centrales altamente cargadas son propensas a interacciones no específicas con varias proteínas en el citoplasma celular y el núcleo. Las chaperonas de histonas forman una clase diversa de proteínas que transportan histonas desde el citoplasma al núcleo y ayudan a su deposición en el ADN, ayudando así al proceso de ensamblaje de nucleosomas. Algunas chaperonas de histonas son específicas para H2A/H2B o H3/H4, y algunas funcionan como chaperonas para ambas. Este protocolo describe cómo las técnicas de laboratorio in vitro , como los ensayos pull-down, la cromatografía analítica de exclusión de tamaño, la ultracentrifugación analítica y el ensayo de acompañamiento de histonas, podrían usarse en conjunto para confirmar si una proteína dada es funcional como una chaperona de histonas.
Introducción
Los nucleosomas compuestos de ADN y proteínas histonas forman la unidad estructural de la cromatina y regulan varios eventos celulares críticos. Los nucleosomas se reposicionan y remodelan dinámicamente para hacer que el ADN sea accesible a diversos procesos como la replicación, la transcripción y la traducción 1,2. Las histonas que son altamente básicas tienden a interactuar con proteínas ácidas en el medio celular o sufren agregación, lo que lleva a varios defectos celulares 3,4,5. Un grupo de proteínas dedicadas llamadas chaperonas histonas ayudan al transporte de histonas desde el citoplasma hasta el núcleo y previenen eventos aberrantes de agregación histona-ADN 6,7. Fundamentalmente, la mayoría de las chaperonas de histonas almacenan y transfieren histonas al ADN con fuerza iónica fisiológica, ayudando así a la formación de nucleosomas 8,9. Algunas chaperonas de histonas tienen una preferencia definida por los oligómeros de histonas H2A/H2B o H3/H410.
Las chaperonas de histonas se caracterizan por su capacidad para ensamblar nucleosomas dependientes o independientes de la síntesis de ADN11. Por ejemplo, el factor de ensamblaje de la cromatina-1 (CAF-1) es dependiente, mientras que el regulador de histonas A (HIRA) es independiente de la síntesis de ADN12,13. Del mismo modo, la familia de nucleoplasminas de las chaperonas histonas está involucrada en la descondensación de la cromatina espermática y el ensamblaje de nucleosomas14. Los miembros de la familia de proteínas de ensamblaje de nucleosomas (NAP) facilitan la formación de estructuras similares a nucleosomas in vitro y están involucrados en el transporte de histonas entre el citoplasma y el núcleo15. Las nucleoplasminas y las proteínas de la familia NAP son chaperonas de histonas funcionales, pero no comparten ninguna característica estructural. Esencialmente, ninguna característica estructural única permite la clasificación de una proteína como una chaperona de histonas16. El uso de ensayos funcionales y biofísicos junto con estudios estructurales funcionan mejor para caracterizar las chaperonas de histonas.
Este trabajo describe métodos bioquímicos y biofísicos para caracterizar una proteína como una chaperona de histonas que ayuda al ensamblaje de nucleosomas. En primer lugar, se realizó cromatografía analítica de exclusión de tamaño para analizar el estado oligomérico y la estabilidad de las chaperonas de histonas. A continuación, se realizó un ensayo pull-down para determinar las fuerzas impulsoras y la naturaleza competitiva de las interacciones chaperona-histona de histonas. Sin embargo, los parámetros hidrodinámicos de estas interacciones no pudieron calcularse con precisión utilizando cromatografía analítica de exclusión de tamaño debido a la forma de la proteína y sus complejos que afectan su migración a través de la columna. Por lo tanto, se utilizó la ultracentrifugación analítica, que proporciona propiedades macromoleculares en solución que incluyen el peso molecular preciso, la estequiometría de interacción y la forma de las moléculas biológicas. Estudios anteriores han utilizado ampliamente el ensayo de acompañamiento de histonas in vitro para caracterizar funcionalmente las chaperonas de histonas como yScS116 17, DmACF18, ScRTT106p19, HsNPM120. El ensayo de acompañamiento de histonas también se utilizó para caracterizar funcionalmente las proteínas como chaperonas de histonas.
Protocolo
1. Cromatografía analítica de exclusión de tamaño para dilucidar el estado oligomérico y la estabilidad de las chaperonas de histonas
- Análisis del estado oligomérico de las chaperonas de histonas
- Equilibrar una columna de cromatografía analítica de exclusión de tamaño (SEC) de 24 ml con 1,2 ml de volumen de columna (CV), es decir, 28,8 ml de tampón SEC desgasificado [20 mM de Tris-HCl (pH 7,5), 300 de NaCl mM y 1 mM de β-mercaptoetanol (β-ME)] a 4 °C (ver Tabla de materiales).
NOTA: El tipo de columna, la composición del tampón y el pH del tampón se pueden seleccionar en función de la proteína de interés. El volumen de inyección de la muestra no debe exceder los 500 μL para una columna de 24 ml. Además, la presión de la columna debe mantenerse por debajo de 5 MPa. - A partir de una solución madre de proteínas de mayor concentración, preparar 500 μL de muestra de proteína de 0,5 mg/ml en tampón SEC desgasificado e inyectarla en la columna preequilibrada utilizando un bucle de inyección de 500 μL. Permitir que la ejecución cromatográfica proceda a un caudal isocrático de 0,2-0,3 ml/min con el tampón SEC a 4 °C.
- Monitoree el perfil de elución de la proteína midiendo la absorbancia a una longitud de onda de 280 nm. Cuando se trata de proteínas que carecen de residuos aromáticos, medir la absorbancia a 214 nm.
- Utilice el volumen de elución de la proteína para calcular su peso molecular aproximado en kDa utilizando la curva de calibración estándar21.
NOTA: La curva de calibración se prepara trazando el volumen de retención de proteínas de peso molecular conocidas contra el logaritmo de sus respectivos pesos moleculares (log Mr), eluidos utilizando la misma columna.
- Equilibrar una columna de cromatografía analítica de exclusión de tamaño (SEC) de 24 ml con 1,2 ml de volumen de columna (CV), es decir, 28,8 ml de tampón SEC desgasificado [20 mM de Tris-HCl (pH 7,5), 300 de NaCl mM y 1 mM de β-mercaptoetanol (β-ME)] a 4 °C (ver Tabla de materiales).
- Análisis de la estabilidad térmica de las chaperonas de histonas
- Tomar 500 μL de 0,5 mg/ml de la muestra de proteína preparada en tampón SEC desgasificado (igual que el utilizado en 1.1.1) en tubos de microcentrífuga individuales y calentar cada tubo a una temperatura particular que oscile entre 20 °C y 90 °C (20 °C, 40 °C, 60 °C y 90 °C) durante 10 min en un baño maría.
- Posteriormente, centrifugar las muestras tratadas térmicamente a 16.200 x g durante 10 min a 4 °C, recoger el sobrenadante con una micropipeta e inyectar cada muestra individualmente utilizando un bucle de inyección de 500 μL en la columna analítica, preequilibrado con el tampón SEC a 4 °C.
- Permitir que la ejecución cromatográfica proceda a un caudal isocrático de 0,2-0,3 ml/min con el tampón SEC a 4 °C.
- Observe la posición y la altura de los picos de elución y busque la aparición de picos adicionales para las diferentes muestras.
- Análisis de la estabilidad química de las chaperonas de histonas
- Para examinar la estabilidad salina de las chaperonas de histonas, incubar 500 μL de 0,5 mg/ml de muestra de proteína preparada en un tampón Tris [20 mM de Tris-HCl (pH 7,5) y 1 mM de β-ME] suplementado con concentraciones crecientes de NaCl (300 mM, 600 mM, 1 M, 1,5 M y 2 M) en tubos de microcentrífuga separados durante 30 min a 4 °C. Centrifugar las muestras a 16.200 x g durante 10 min a 4 °C y retener el sobrenadante.
- A continuación, cargue las muestras de proteínas en diferentes concentraciones de NaCl individualmente, utilizando un bucle de inyección de 500 μL en la columna analítica preequilibrada con 1,2 CV (28,8 ml) del tampón respectivo que contiene concentraciones crecientes de NaCl a 4 °C.
- Permitir que la ejecución cromatográfica proceda a un caudal isocrático de 0,2-0,3 ml/min con 1 CV (24 ml) del tampón respectivo a 4 °C.
- Observe la posición y la altura de los picos de elución y busque la aparición de picos adicionales para las diferentes muestras.
- Del mismo modo, para el análisis de estabilidad de la urea, incubar 500 μL de 0,5 mg/ml de muestra de proteína preparada en un tampón Tris [20 mM de Tris-HCl (pH 7,5) y 1 mM de β-ME] suplementado con concentraciones crecientes de urea (1 M, 2 M, 3 M, 4 M y 5 M) en tubos de microcentrífuga separados durante 16 h a temperatura ambiente. Centrifugar las muestras a 16.200 x g durante 10 min a temperatura ambiente y retener el sobrenadante.
- A continuación, cargue las muestras de proteínas tratadas con urea individualmente utilizando un bucle de inyección de 500 μL en la columna analítica preequilibrada con 1,2 CV (28,8 ml) del tampón correspondiente que contiene diferentes concentraciones de urea a temperatura ambiente.
- Permita que la ejecución cromatográfica proceda a un caudal isocrático de 0,2-0,3 ml/min con 1 CV (24 ml) del tampón respectivo a temperatura ambiente.
PRECAUCIÓN: No realice los experimentos con tampón que contenga urea a una temperatura más baja ya que la urea tiende a cristalizar y dañar la columna. - Observe la posición y la altura de los picos de elución y busque la aparición de picos adicionales para las diferentes muestras.
2. Ensayos pull-down basados en gradiente de sal para comprender el tipo de interacciones que contribuyen a la formación compleja entre oligómeros de histonas y una chaperona de histonas
- Para cada reacción del ensayo pull-down, pipetear 40 μL de resina Ni-NTA en una columna de centrifugado y lavar con agua estéril de doble destilación. Posteriormente, equilibre la resina con 100 CV (4 mL) de tampón de equilibrio [20 mM de Tris-HCl (pH 7.5), 300 mM de NaCl, 10 mM de imidazol, 10 μg/mL de BSA y 1 mM de β-ME] (ver Tabla de materiales).
NOTA: La extracción también se puede realizar en un tubo de microcentrífuga de 1,5 ml. - Preparar la muestra mezclando 5 μM de la chaperona de histonas marcada con His con 20 μM de dímero H2A/H2B de histona o tetrámero H3/H4 en el tampón de equilibrio. Incubar la muestra en hielo durante 1 h.
NOTA: El dímero H2A/H2B y el tetrámero H3/H4 se preparan a partir de histonas humanas recombinantes21, y la integridad de los oligómeros se confirma sobre la base de las masas moleculares estimadas por ultracentrifugación analítica (AUC). Los mismos oligómeros de histonas se han utilizado para todos los experimentos mencionados a continuación. - Cargue las muestras en columnas de centrifugado preequilibradas separadas con resina Ni-NTA del paso 2.1, cada una etiquetada para una concentración de sal particular, y mantenga las columnas durante 30 minutos a 4 °C. Centrifugar las columnas a 1000 x g durante 1 min.
- A continuación, lave las columnas con 100 CV (4 ml) de tampón de lavado [20 mM de Tris-HCl (pH 7.5), 50 mM de imidazol, 0.2% de Tween-20 y 1 mM de β-ME] que contengan diferentes concentraciones de sal (es decir, 300 mM, 500 mM, 600 mM, 700 mM, 800 mM, 900 mM y 1 M NaCl). Lave cada columna con un tampón que tenga una concentración particular de sal.
- Después de la etapa de lavado con sal, eluya la proteína de las diferentes columnas utilizando 100 μL de tampón de elución [20 mM de Tris-HCl (pH 7.5), 300 mM de NaCl, 300 mM de imidazol y 1 mM de β-ME].
- Posteriormente, someta las muestras eluyidas al 18% SDS-PAGE22 y visualice el gel después de la tinción con Coomassie Brilliant Blue R250 (ver Tabla de materiales). Alternativamente, puede cargar directamente la resina en el gel SDS-PAGE en lugar de liberar la proteína unida de la resina Ni-NTA.
NOTA: Las composiciones del tampón de equilibrio, lavado y elución y el pH pueden modificarse dependiendo de la proteína de interés.
3. Ensayo pull-down competitivo para identificar la preferencia de una chaperona de histonas por H2A/H2B o H3/H4
- Preparar la columna de giro como se describe en el paso 2.1
- Incubar 5 μM de la chaperona de histonas con 20 μM de dímero H2A/H2B en 300 μL de tampón de equilibrio (preparado en el paso 2.1) durante 30 min sobre hielo.
NOTA: La proporción de oligómero de histonas a chaperona de histonas en la reacción se puede elegir en función de los datos conocidos de estequiometría de unión; Use un exceso de histona cinco veces mayor si no hay información disponible. - Centrifugar el complejo chaperona de histonas-H2A/H2B a 16.200 x g durante 5 min a 4 °C para eliminar cualquier precipitado. A continuación, cargar la muestra en la columna de centrifugado preequilibrada con tampón de equilibrio (preparado en el paso 2.1) e incubar durante 30 min a 4 °C.
- Lave la columna con 100 CV (4 ml) de tampón de lavado [20 mM de Tris-HCl (pH 7.5), 300 mM de NaCl, 50 mM de imidazol, 0.2% de Tween-20 y 1 mM de β-ME] para eliminar el exceso de dímero H2A/H2B. A continuación, mezcle el complejo chaperona histona-H2A / H2B con 20-60 μM de tetrámero H3 / H4 e incube durante 30 minutos en hielo.
- Vuelva a lavar la columna con 100 CV (4 ml) de tampón de lavado (preparado en el paso 3.4) para eliminar cualquier tetrámero H3/H4 no unido y eluya la muestra utilizando un tampón de elución (preparado en el paso 2.5). Sujete las muestras eluyidas al 18% SDS-PAGE y visualice después de la tinción con Coomassie Brilliant Blue R250.
NOTA: El ensayo podría invertirse en el que, primero, el tetrámero H3 / H4 se puede mezclar con la chaperona, se permite que el complejo se una a las perlas de Ni-NTA y luego se incuba el complejo con concentraciones variables de dímero H2A / H2B.
4. Experimentos analíticos de ultracentrifugación - velocidad de sedimentación (AUC-SV) para analizar la estequiometría de unión entre chaperonas de histonas e histonas
- Preparación de muestras para AUC
- Dializar el dímero H2A/H2B de histona reconstituido, el tetrámero H3/H4 y la chaperona de histonas por separado a través de un tubo de diálisis de corte de 7 kDa23, contra un tampón de diálisis [20 mM de Tris (pH 7.5), 300 mM de NaCl y 1 mM de β-ME] (ver Tabla de materiales). Para minimizar el error de fondo debido a la falta de coincidencia del búfer, realice diálisis extensivamente contra el tampón de diálisis, preferiblemente tres veces durante un período de 24 h.
NOTA: El ODinicial 280 de las muestras de proteína debe tener un valor de dos a tres veces mayor para lograr un OD280 final de 0.3-0.5. Esto se hace esencialmente para anular los efectos de la dilución. - Purificar el dímero H2A/H2B, el tetrámero H3/H4 y la chaperona de histonas individualmente con el tampón de diálisis, utilizando cromatografía analítica de exclusión de tamaño (como se mencionó en el paso 1). Guarde el tampón de la ejecución para preparar diluciones adicionales más adelante y para usarlo como referencia en la celda AUC.
- Dializar el dímero H2A/H2B de histona reconstituido, el tetrámero H3/H4 y la chaperona de histonas por separado a través de un tubo de diálisis de corte de 7 kDa23, contra un tampón de diálisis [20 mM de Tris (pH 7.5), 300 mM de NaCl y 1 mM de β-ME] (ver Tabla de materiales). Para minimizar el error de fondo debido a la falta de coincidencia del búfer, realice diálisis extensivamente contra el tampón de diálisis, preferiblemente tres veces durante un período de 24 h.
- Carga de muestras para AUC
- Mezclar las proteínas purificadas en un volumen final de 450 μL utilizando tampón de diálisis a partir del paso 4.1.1 para alcanzar un OD280 de 0,3-0,6. Mezcle la chaperona de histonas con dímero H2A/H2B o tetrámero H3/H4 para la formación de complejos en tubos de reacción separados. Incubar las mezclas de proteínas durante 2-3 h.
NOTA: Alternativamente, los datos de sedimentación se pueden adquirir con un sistema de escaneo óptico de interferencia en la ultracentrífuga analítica. Por separado, para mezclar proteínas purificadas, fijar la concentración de chaperonas de histonas e incubarla con concentraciones crecientes de oligómeros de histonas para obtener la estequiometría exacta. - Ensamble la celda con una pieza central de doble sector y ventanas de cuarzo para el experimento AUC-SV utilizando un detector de absorbancia de la ultracentrífuga analítica como se describió anteriormente en detalle24.
- Llene 400 μL de la solución de muestra y 420 μL de tampón de diálisis en los dos sectores (muestra y sectores de referencia, respectivamente) de la célula.
NOTA: Se utiliza un mayor volumen de tampón en el sector de referencia para mantener el menisco de referencia por encima del menisco de la muestra. Sin embargo, mientras usa un sistema de interferencia óptica, llene los dos sectores con el mismo volumen. - Pese y equilibre con precisión las celdas y cárguelas en un rotor de titanio de cuatro lugares (consulte la Tabla de materiales). Alinee las celdas utilizando las marcas proporcionadas en la parte inferior de las celdas y el rotor. Cargue el rotor en la centrífuga, cierre la tapa y permita desarrollar un vacío hasta que la presión descienda a menos de 15 micras de Hg y la temperatura del rotor se estabilice a 20 ° C (generalmente toma 2-2.5 h).
NOTA: Los parámetros de funcionamiento del AUC incluyen la temperatura experimental, la velocidad del rotor, el intervalo entre escaneos y el número de escaneos que se recopilarán. En el caso de los experimentos SV, el intervalo de exploración se da de acuerdo con la masa molecular de la proteína; Las proteínas más pequeñas requieren intervalos de tiempo más grandes entre las exploraciones. La velocidad del rotor también se establece de acuerdo con la masa molecular esperada de la proteína, y el experimento se lleva a cabo a 20 ° C. Los datos de absorbancia se monitorizan a 280 nm. - Para obtener la estequiometría exacta, mantenga constante la concentración de chaperonas de histonas e incube con concentraciones crecientes de oligómeros de histonas para lograr la saturación.
- Mezclar las proteínas purificadas en un volumen final de 450 μL utilizando tampón de diálisis a partir del paso 4.1.1 para alcanzar un OD280 de 0,3-0,6. Mezcle la chaperona de histonas con dímero H2A/H2B o tetrámero H3/H4 para la formación de complejos en tubos de reacción separados. Incubar las mezclas de proteínas durante 2-3 h.
- Análisis de datos de AUC
- Realizar el análisis de datos como se describió anteriormente25. Brevemente, calcule la densidad y viscosidad de los componentes del tampón utilizando el programa SEDNTERP26 (consulte la Tabla de materiales). Del mismo modo, calcule el volumen específico parcial de la proteína en función de su composición de aminoácidos, utilizando también SEDNTERP.
- Cargue los datos de la máquina AUC en el programa SEDFIT27 y defina el menisco (línea roja), la parte inferior de la celda (línea azul) y los límites del análisis de datos (líneas verdes). Elija la distribución continua de C(s) como modelo.
- A continuación, establezca la resolución máxima en 100; establecer coeficiente de sedimentación (s), s min: 0 y s max: 10-15; establecer la relación de fricción en 1,2 inicialmente y optar por flotar para derivar la relación a partir de los datos; establecer el nivel de confianza (relación F; que determina la magnitud de la regularización) en 0,68 para la regularización 1 sigma; establecer valores parciales específicos de volumen, densidad tampón y viscosidad tampón obtenidos de SEDNTERP.
- Presione EJECUTAR para permitir que el software resuelva la ecuación de Lamm27. Ajuste los parámetros si hay una discrepancia significativa de datos. Después de ajustar los parámetros, presione FIT para refinar todos los parámetros. Evalúe la calidad del ajuste en función del valor de desviación cuadrática media (RMSD), que debe ser inferior a 0,01 unidades de señal.
- Estime las masas moleculares de los picos eligiendo la opción: mostrar pico "Mw en c (s)" en la función de visualización de la barra de herramientas principal, que proporcionará información sobre la 's' de la molécula / complejo.
5. Ensayo de superenrollamiento de plásmidos para confirmar la función de acompañamiento de histonas
- Reacción de ensamblaje de nucleosomas
- Mezclar 2 μM de tetrámero H3/H4 y 4 μM de dímero H2A/H2B con concentraciones crecientes de la chaperona de histonas (1-6 μM) en un tampón de ensamblaje [20 mM de Tris HCl (pH 7,5), 1 mM de DTT, 1 mM de MgCl2, 0,1 mg/ml de BSA y 100 mM de NaCl] hasta un volumen final de 50 μL. Incubar la mezcla a 4 °C durante 30 min.
- Simultáneamente, en una reacción separada, pretratar 500 ng del plásmido pUC19 en espiral negativamente con 1 μg de enzima topoisomerasa I (ver Tabla de materiales) en el tampón de ensamblaje en un volumen final de 50 μL e incubar a 30 °C durante 30 min.
NOTA: La topoisomerasa I relaja el ADN plásmido bicatenario superenrollado generando un nick monocatenario. Se podría utilizar una enzima topoisomerasa I de origen eucariota, como la topoisomerasa I del germen de trigo disponible comercialmente o la topoisomerasa I de Drosophila melanogaster expresada recombinantemente. - A continuación, combinar el tetrámero H3/H4, el dímero H2A/H2B, la mezcla de chaperonas de histonas (del paso 5.1.1), la mezcla de reacción de ADN plásmido relajado (del paso 5.1.2) e incubar aún más a 30 °C durante 90 min.
NOTA: Configure dos reacciones de control para el ensayo; uno con la chaperona de histonas y el ADN plásmido relajado (pero no las histonas) y el otro con los oligómeros de histonas y el ADN plásmido relajado (pero no la chaperona de histonas). - Detenga la reacción de ensamblaje agregando 100 μL de tampón de parada 2x (40 mM de EDTA, 2% de SDS y 0.4 mg / ml de proteinasa K) e incubar a 37 °C durante 30 min.
NOTA: Stop buffer desproteiniza el ADN plásmido mediante desnaturalización y proteólisis de histonas unidas.
- Extracción de fenol-cloroformo y precipitación de etanol
- Añadir un volumen igual de fenol saturado de Tris en el tubo que contiene la mezcla de reacción del paso 5.1.4 y mezclar bien, seguido de centrifugación a 16,200 x g durante 10 min a temperatura ambiente.
- Recoger suavemente la fase acuosa superior que tiene el ADN plásmido con una micropipeta y mezclar con un volumen igual de cloroformo. Vaporizar la mezcla y centrifugar a 16.200 x g durante 10 min a temperatura ambiente.
NOTA: El alcohol isoamílico podría incluirse en este paso para evitar una interfaz difusa entre las fases acuosa y orgánica. - A continuación, recoja la fase acuosa superior, agregue 1/10 de volumen de acetato de sodio 3 M (pH 5.5) y 2.5 volúmenes de etanol helado (consulte la Tabla de materiales). Mezclar bien la solución invirtiendo el tubo 3-4 veces y mantener la mezcla en un congelador de -20 °C durante 30 minutos para la precipitación completa del ADN plásmido.
- Centrifugar la muestra del paso 5.2.3 a 16200 x g durante 10 min y desechar suavemente el sobrenadante. Mantenga los tubos abiertos a temperatura ambiente hasta que incluso pequeñas cantidades de etanol se evaporen, dejando el ADN plásmido precipitado en el tubo.
- Realice la electroforesis en gel de agarosa para observar el efecto de superenrollamiento del plásmido.
- Disolver el ADN plásmido precipitado del paso 5.2.4 en agua estéril de doble destilación.
- Resuelva las muestras en un gel de agarosa al 1% en 1x tampón Tris-acetato-EDTA (TAE) (40 mM de Tris, 20 mM de ácido acético y 1 mM de EDTA) (ver Tabla de materiales).
- Tiñe el gel con una concentración de 0.2-0.5 μg / ml de bromuro de etidio y observe bajo UV para visualizar las bandas de ADN en el gel.
Resultados
El dominio nucleoplasmina N-terminal recombinante de la proteína FKBP53 de Arabidopsis thaliana se sometió a SEC analítico. El volumen pico de elución se trazó contra la curva estándar para identificar su estado oligomérico. Los resultados analíticos de la SEC revelaron que el dominio existe como un pentámero en solución, con una masa molecular aproximada de 58 kDa (Figura 1A, B). Además, el dominio de nucleoplasmina se analizó para determinar la estabilidad térmica y química junto con la SEC analítica. La muestra de nucleoplasmina sometida a tratamiento térmico hasta 90 °C no mostró ningún cambio aparente en el volumen de elución y la altura máxima en comparación con las muestras mantenidas a 20 °C, lo que sugiere que el dominio es altamente termoestable (Figura 1C). Asimismo, el dominio nucleoplasmina mostró estabilidad salina hasta 2 M de NaCl (Figura 1D) y estabilidad de urea hasta 4 M (Figura 1E). Sin embargo, el pentámero de nucleoplasmina comenzó a disociarse en concentraciones más altas de urea.
Se realizó un ensayo pull-down para determinar el tipo de interacciones que contribuyen a la formación de complejos entre la chaperona de histonas (dominio nucleoplasmina de AtFKBP53) y los oligómeros de histonas H2A/H2B dímero y tetrámero H3/H4 utilizando un gradiente de lavado con sal. La interacción del dominio nucleoplasmina con el dímero H2A/H2B fue estable hasta una concentración salina de 0,4 M NaCl (Figura 2A). En comparación, la asociación con H3/H4 fue razonablemente estable hasta 0,7 M NaCl (Figura 2B). La capacidad de los complejos chaperona-histona para soportar altas concentraciones de sal sugiere el papel de las interacciones hidrofóbicas en la estabilización de los complejos. El complejo chaperona con H3/H4 estable incluso en altas concentraciones de sal sugiere un papel predominante de las interacciones hidrofóbicas en la formación del complejo. La menor estabilidad del complejo H2A/H2B-chaperona en altas concentraciones de sal revela un papel significativo para las interacciones electrostáticas en la formación del complejo. En otro experimento, el ensayo pull-down se utilizó para examinar si la chaperona prefiere el dímero H2A/H2B o el tetrámero H3/H4. Los resultados revelaron que la chaperona se une al dímero H2A/H2B y al tetrámero H3/H4 simultáneamente e independientemente del orden en que se añadan a la chaperona (Figura 2C,D). Esto indicó que la chaperona posee sitios separados para su interacción con los oligómeros de histonas.
Se realizaron experimentos AUC-SV (Figura 3) para estudiar la estequiometría de la interacción entre oligómeros de histonas y chaperonas. El análisis de datos de AUC-SV proporcionó un valor de coeficiente de sedimentación (s) de 5.40 S para el dominio de nucleoplasmina AtFKBP53 en complejo con H2A / H2B que correspondía a una masa molecular de 104 kDa. El complejo del dominio nucleoplasmina con H3/H4 dio un valor de coeficiente de sedimentación de 7,35 S, correspondiente a 129 kDa. La masa molecular estimada de los complejos revela que la nucleoplasmina pentamérica forma complejos con dímero H2A/H2B y tetrámero H3/H4 en una estequiometría 1:1.
Es esencial demostrar que la proteína puede depositar oligómeros de histonas en el ADN para confirmar que es una chaperona de histonas. Con este fin, se adoptó un ensayo de superenrollamiento de plásmidos (Figura 4). El plásmido circular relajado se incubó con los oligómeros de histonas H2A/H2B y H3/H4 con las chaperonas de histonas de plantas recombinantes de la familia NAP - AtNRP1 y AtNRP228. La presencia de la chaperona aumentó la cantidad de plásmido superenrollado, lo que sugiere que podría depositar histonas en el ADN para formar nucleosomas, causando superenrollamiento del ADN.

Figura 1: Estado oligomérico y estabilidad del dominio nucleoplasmina de AtFBP53. (A) Perfil de cromatografía analítica de exclusión de tamaño del dominio de nucleoplasmina AtFKBP53. (B) Curva de calibración obtenida utilizando proteínas globulares de masa molecular conocida. Los puntos azules representan la masa molecular de las proteínas conocidas, mientras que el punto rojo representa el dominio de nucleoplasmina AtFKBP53. (440 kDa - ferritina, 158 kDa-aldolasa, 75 kDa-con albúmina, 44 kDa-ovoalbúmina, 6,5 kDa-aprotinina). (C) Cromatograma analítico de exclusión de tamaño de 500 μL de 0,5 mg/ml del dominio de nucleoplasmina AtFKBP53 sometido a tratamiento térmico a diferentes temperaturas: 20 °C (verde), 40 °C (naranja), 60 °C (negro), 90 °C (azul claro). (D) Cromatograma analítico de exclusión de tamaño de 500 μL de 0,5 mg/ml del dominio de nucleoplasmina AtFKBP53 en tampones que contienen diferentes concentraciones de NaCl: 0,3 M (púrpura), 0,6 M (rojo), 1,0 M (azul claro), 1,5 M (verde), 2,0 M (negro). (E) Cromatograma analítico de exclusión de tamaño del dominio de nucleoplasmina AtFKBP53 en tampones con diferentes concentraciones de urea: 0 M (control; azul claro), 1,0 M (rosa), 2,0 M (negro), 3,0 M (azul oscuro), 4,0 M (verde), 5,0 M (marrón). El pentámero de nucleoplasmina muestra una alta estabilidad a las condiciones de estrés térmico y químico. La figura está adaptada de la Referencia21. Haga clic aquí para ver una versión más grande de esta figura.
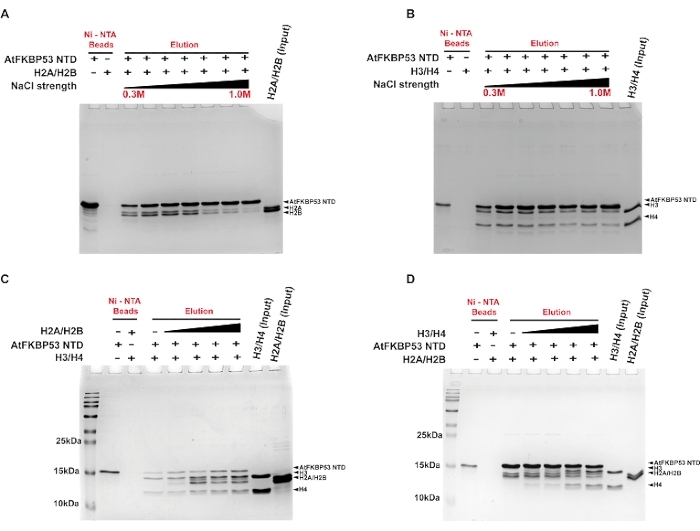
Figura 2: Ensayos pull-down para la interacción del dominio nucleoplasmina de AtFKBP53 con oligómeros de histonas. El 18% de las imágenes SDS-PAGE de las fracciones de elución de los ensayos se presentan aquí. Ensayo de extracción para ( A ) dímero H2A/H2B de 20 μM y (B) tetrámero H3/H4 de 20 μM con dominio de nucleoplasmina AtFKBP53 de 5 μM en concentraciones crecientes de NaCl en el rango de 0,3 M, 0,5 M, 0,6 M, 0,7 M, 0,8 M, 0,9 M y 1,0 M. 5 μM AtFKBP53 FKBD se utilizó como control negativo aquí. Para el experimento de unión competitiva, ( C ) se ha utilizado una mezcla de 5 μM del dominio nucleoplasmina AtFKBP53 y 20 μM H3/H4 tetrámero incubado con un rango de dímero H2A/H2B de 20-60 μM y (D) una mezcla de 5 μM del dominio nucleoplasmina AtFKBP53 y dímero H2A/H2B de 20 μM incubado con un rango de tetrámero H3/H4 de 20-60 μM. La etiqueta AtFKBP53 corresponde al dominio nucleoplasmina de AtFKBP53. Las fracciones de elución muestran la unión simultánea de ambos oligómeros de histonas a la nucleoplasmina. La figura está adaptada de la Referencia21. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Experimento analítico de ultracentrifugación - velocidad de sedimentación (AUC-SV) de oligómeros de histonas, el dominio de nucleoplasmina de AtFKBP53 y sus complejos. La distribución de distancia AUC vs. diagrama del coeficiente de sedimentación (S). También se proporcionan los valores de coeficiente de sedimentación (s) obtenidos y las masas moleculares. La etiqueta AtFKBP53 corresponde al dominio nucleoplasmina de AtFKBP53. Las masas moleculares estimadas revelan una estequiometría 1:1 para el dominio de nucleoplasmina AtFKBP53 con los oligómeros de histonas H2A/H2B dímero y tetrámero H3/H4. 450 μL de todas las muestras de proteínas con un OD280 de 0,3-0,5 se utilizaron para los experimentos AUC-SV. La figura está adaptada de la Referencia21. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Ensayo de superenrollamiento de plásmidos. Ensayo de superenrollamiento de plásmidos para las chaperonas de histonas AtNRP1 y AtNRP2. Se trataron previamente 500 ng de ADN plásmido pUC19 con 1 μg de topoisomerasa I para el experimento. 4 μM AtNRP1, 4 μM AtNRP2 y una mezcla de 4 μM H2A/H2B dímero y 2 μM H3/H4 tetramer fueron como control que no muestra actividad de superbobinado cuando se incubó con el ADN pUC19 pretratado. Los carriles con una mezcla de 4 μM H2A/H2B de dímero y 2 μM H3/H4 de tetrámero y 4 μM cada uno de AtNRP1 y AtNRP2 muestran la formación de ADN superenrollado tras la incubación con el ADN pUC19 pretratado. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este trabajo demuestra y valida un conjunto completo de protocolos para la caracterización bioquímica y biofísica de una chaperona de histonas putativa. En esta invención, se utilizaron proteínas de la familia NAP expresadas y purificadas recombinantemente, AtNRP1 y AtNRP2, y el dominio de nucleoplasmina N-terminal de AtFKBP53 para demostrar los protocolos. El mismo conjunto de experimentos podría muy bien usarse para delinear los atributos funcionales de las chaperonas de histonas previamente no caracterizadas de cualquier organismo.
La primera parte de la sección de protocolo implica investigar el estado oligomérico y la estabilidad de una chaperona de histonas. Varios informes indican que las chaperonas de histonas exhiben una diversidad considerable en su estado oligomérico. Por ejemplo, el CAF-1 humano existe como un monómero29. Los miembros de la familia NAP existen como dímero o tetrámero 29,30,31. Las nucleoplasminas revelan estados oligoméricos pentaméricos y a menudo decamericos32,33. Un experimento analítico SEC puede determinar el estado oligomérico de una chaperona de histonas, y los experimentos AUC-SV pueden confirmar lo mismo. Se sabe que varias de las chaperonas de histonas son altamente estables bajo diversas condiciones de estrés térmico y químico33,34. Las características de estabilidad térmica y química de las chaperonas de histonas también podrían explorarse junto con SEC analítica. Además, la espectroscopia de dicroísmo circular podría utilizarse eficazmente para el análisis en profundidad de los cambios en la estructura secundaria de la chaperona cuando se somete a temperaturas crecientes o concentraciones más altas de un agente químico.
La segunda parte de la sección del protocolo cubre ensayos desplegables que podrían examinar las interacciones fundamentales que ayudan a la asociación de oligómeros de histonas con la chaperona mediante el uso de un enfoque de gradiente de sal y un ensayo de extracción competitiva para identificar la preferencia de oligómero de histonas de un chaperón. Si el complejo se desmorona con un ligero aumento en la concentración de sal, eso sugeriría una contribución importante de las interacciones electrostáticas en la estabilización del complejo. Un complejo intacto con alto contenido de sal sugeriría un papel significativo para la hidrofobicidad en la estabilización del complejo35. El ensayo pull-down competitivo podría emplearse fácilmente para determinar la especificidad o preferencia de una chaperona de histonas a una clase específica de oligómeros de histonas. Según su preferencia por los oligómeros de histonas, las chaperonas de histonas se pueden clasificar en tres categorías, como chaperonas H2A / H2B, chaperonas H3 / H4 y chaperonas H2A / H2B-H3 / H410,36. Además, si es necesario, la calorimetría de titulación isotérmica (ITC) podría usarse para comprender la especificidad del oligómero de histonas de una chaperona dada y las características termodinámicas de sus interacciones.
La tercera parte de la sección de protocolo cubre la investigación de la estequiometría de interacción entre una chaperona de histonas y los oligómeros de histonas. En general, las diferentes familias de chaperonas histonas difieren considerablemente por la estequiometría de su asociación con H2A/H2B y/o H3/H4 21,28,37,38. El experimento AUC-SV ayuda a obtener el coeficiente de sedimentación (s) y la masa molecular de una proteína o su complejo, lo que se vuelve muy útil para estimar con precisión la estequiometría en la formación compleja. Alternativamente, ITC también se puede utilizar para examinar la estequiometría.
La cuarta parte de la sección de protocolo se centra en investigar la función de ensamblaje de nucleosomas de las chaperonas de histonas. Las chaperonas de histonas juegan un papel crucial en el ensamblaje de nucleosomas, que regula procesos celulares vitales como la replicación, la transcripción y la reparación del ADN39. En esta sección se elabora el ensayo de superenrollamiento de plásmidos que se emplea típicamente para la evaluación in vitro de la actividad de acompañamiento de histonas de chaperonas de histonas.
Cabe señalar que no todas las chaperonas de histonas están completamente estructuradas. Se sabe que pocos tienen regiones intrínsecamente desordenadas40,41. Por lo tanto, los ensayos de estabilidad térmica y química pueden no ser adecuados para tales proteínas. Además, las chaperonas de histonas de diferentes organismos tienen diferentes estados oligoméricos y capacidades diferenciales para unirse a las histonas. Por lo tanto, este protocolo puede ser un buen punto de partida, pero implicaría modificaciones según sea necesario.
Divulgaciones
No se declaró ningún conflicto de intereses.
Agradecimientos
Las subvenciones extramuros a Dileep Vasudevan de la Junta de Investigación de Ciencia e Ingeniería del Gobierno de la India [CRG/2018/000695/PS] y el Departamento de Biotecnología, Ministerio de Ciencia y Tecnología del Gobierno de la India [BT/INF/22/SP33046/2019], así como el apoyo intramuros del Instituto de Ciencias de la Vida, Bhubaneswar, son muy reconocidos. Agradecemos a la Sra. Sudeshna Sen y a la Sra. Annapurna Sahoo por su ayuda con la preparación de histonas. También se reconocen las conversaciones con nuestros colegas el Dr. Chinmayee Mohapatra, el Sr. Manas Kumar Jagdev y el Dr. Shaikh Nausad Hossain.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | Sigma | A6283 | |
| Acrylamide | MP Biomedicals | 814326 | |
| Agarose | MP Biomedicals | 193983 | |
| AKTA Pure 25M FPLC | Cytiva | 29018226 | Instrument for protein purification |
| Ammonium persulfate (APS) | Sigma | A3678 | |
| An-60Ti rotor | Beckman Coulter | 361964 | Rotor for analytical ultracentrifugation |
| Bovine serum albumin (BSA) | Sigma | A7030 | |
| Chloroform | Sigma | C2432 | |
| Coomassie brilliant blue R 250 | Sigma | 1.15444 | |
| Dialysis tubing (7 kDa cut-off) | Thermo Fisher | 68700 | For dialysing protein samples |
| Dithiothreitol (DTT) | MP Biomedicals | 100597 | |
| DNA Loading Dye | New England Biolabs | B7025S | |
| EDTA disodium salt | MP Biomedicals | 194822 | |
| Electronic balance | Shimadzu | ATX224R | |
| Ethanol | Sigma | E7023 | |
| Ethidium bromide (EtBr) | Sigma | E8751 | |
| Gel Doc System | Bio-Rad | 12009077 | For imaging gels after staining |
| Horizontal gel electrophoresis apparatus | Bio-Rad | 1704405 | Instrument for agarose gel electrophoresis |
| Hydrochloric acid (HCl) | Sigma | 320331 | |
| Imidazole | MP Biomedicals | 102033 | |
| Magnesium chloride (MgCl2) | Sigma | M8266 | |
| Micropipettes | Eppendorf | Z683779 | For pipetting of micro-volumes |
| Mini-PROTEAN electrophoresis system | Bio-Rad | 1658000 | Instrument for SDS-PAGE |
| N,N-methylene-bis-acrylamide | MP Biomedicals | 800172 | |
| Nano drop | Thermo Fisher | ND-2000 | For measurement of protein and DNA concentrations |
| Ni-NTA agarose | Invitrogen | R901-15 | Resin beads for pull-down assay |
| Optima AUC analytical ultracentrifuge | Beckman Coulter | B86437 | Instrument for analytical ultracentrifugation |
| pH Meter | Mettler Toledo | MT30130863 | |
| Phenol | Sigma | P4557 | |
| Plasmid isolation kit | Qiagen | 27104 | |
| Proteinase K | Sigma-Aldrich | 1.07393 | |
| pUC19 | Thermo Fisher | SD0061 | Plasmid for supercoiling assay |
| Refrigerated high-speed centrifuge | Thermo Fisher | 75002402 | |
| SDS-PAGE protein marker | Bio-Rad | 1610317 | |
| SEDFIT | Free software program for analytical ultracentrifugation data analysis | ||
| SEDNTERP | Free software program to estimate viscosity and density of buffer and partial specific volume of a protein | ||
| SigmaPrep Spin Columns | Sigma | SC1000 | For pull-down assay |
| Sodium acetate | Sigma | S2889 | |
| Sodium chloride (NaCl) | Merck | S9888 | |
| Sodium dodecyl sulfate (SDS) | MP Biomedicals | 102918 | |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | Column for analytical size-exclusion chromatography |
| Superdex 75 Increase 10/300 GL | Cytiva | 29148721 | Column for analytical size-exclusion chromatography |
| TEMED | Sigma | 1.10732 | |
| Topoisomerase I | Inspiralis | WGT102 | Enzyme used in plasmid supercoiling assay |
| Tris base | Merck | T1503 | |
| Tween-20 | Sigma | P1379 | |
| Urea | MP Biomedicals | 191450 | |
| Water bath | Nüve | NB 5 | For heat treatment of protein samples |
| β-mercaptoethanol (β-ME) | Sigma | M6250 |
Referencias
- Hübner, M. R., Eckersley-Maslin, M. A., Spector, D. L. Chromatin organization and transcriptional regulation. Current Opinion in Genetics and Development. 23 (2), 89-95 (2013).
- Lai, W. K. M., Pugh, B. F. Understanding nucleosome dynamics and their links to gene expression and DNA replication. Nature Reviews Molecular Cell Biology. 18 (9), 548-562 (2017).
- Kim, U. J., Han, M., Kayne, P., Grunstein, M. Effects of histone H4 depletion on the cell cycle and transcription of Saccharomyces cerevisiae. EMBO Journal. 7 (7), 2211-2219 (1988).
- Prado, F., Aguilera, A. Partial depletion of histone H4 increases homologous recombination-mediated genetic instability. Molecular and Cellular Biology. 25 (4), 1526-1536 (2005).
- Meeks-Wagner, D., Hartwell, L. H. Normal stoichiometry of histone dimer sets is necessary for high fidelity of mitotic chromosome transmission. Cell. 44 (1), 43-52 (1986).
- Groth, A., et al. Human Asf1 regulates the flow of S phase histones during replicational stress. Molecular Cell. 17 (2), 301-311 (2005).
- Laskey, R. A., Honda, B. M., Mills, A. D., Finch, J. T. Nucleosomes are assembled by an acidic protein which binds histones and transfers them to DNA. Nature. 275 (5679), 416-420 (1978).
- Das, C., Tyler, J. K., Churchill, M. E. A. The histone shuffle: histone chaperones in an energetic dance. Trends in Biochemical Sciences. 35 (9), 476-489 (2010).
- Akey, C. W., Luger, K. Histone chaperones and nucleosome assembly. Current Opinion in Structural Biology. 13 (1), 6-14 (2003).
- De Koning, L., Corpet, A., Haber, J. E., Almouzni, G. Histone chaperones: An escort network regulating histone traffic. Nature Structural and Molecular Biology. 14 (11), 997-1007 (2007).
- Eitoku, M., Sato, L., Senda, T., Horikoshi, M. Histone chaperones: 30 years from isolation to elucidation of the mechanisms of nucleosome assembly and disassembly. Cellular and Molecular Life Sciences. 65 (3), 414-444 (2008).
- Quivy, J. P., Grandi, P., Almouzni, G. Dimerization of the largest subunit of chromatin assembly factor 1: importance in vitro and during Xenopus early development. EMBO Journal. 20 (8), 2015-2027 (2001).
- Ray-Gallet, D., et al. HIRA is critical for a nucleosome assembly pathway independent of DNA synthesis. Molecular Cell. 9 (5), 1091-1100 (2002).
- Frehlick, L. J., Eirín-López, J. M., Ausió, J. New insights into the nucleophosmin/nucleoplasmin family of nuclear chaperones. Bioessays. 29 (1), 49-59 (2007).
- Ito, T., Bulger, M., Kobayashi, R., Kadonaga, J. T. Drosophila NAP-1 is a core histone chaperone that functions in ATP-facilitated assembly of regularly spaced nucleosomal arrays. Molecular and Cellular Biology. 16 (6), 3112-3124 (1996).
- Elsässer, S. J., D'Arcy, S. Towards a mechanism for histone chaperones. Biochimica et Biophysica Acta. 1819 (3-4), 211-221 (2013).
- Rodríguez-Campos, A., Koop, R., Faraudo, S., Beato, M. Transcriptionally competent chromatin assembled with exogenous histones in a yeast whole cell extract. Nucleic Acids Research. 32 (13), 111(2004).
- Levenstein, M. E., Kadonaga, J. T. Biochemical analysis of chromatin containing recombinant Drosophila core histones. Journal of Biological Chemistry. 277 (10), 8749-8754 (2002).
- Huang, S., et al. Rtt106p is a histone chaperone involved in heterochromatin-mediated silencing. Proceedings of the National Academy of Sciences of the United States of America. 102 (38), 13410-13415 (2005).
- Swaminathan, V., Kishore, A. H., Febitha, K. K., Kundu, T. K. Human histone chaperone nucleophosmin enhances acetylation-dependent chromatin transcription. Molecular and Cellular Biology. 25 (17), 7534-7545 (2005).
- Singh, A. K., Datta, A., Jobichen, C., Luan, S., Vasudevan, D. AtFKBP53: A chimeric histone chaperone with functional nucleoplasmin and PPIase domains. Nucleic Acids Research. 48 (3), 1531-1550 (2020).
- Scofield, B. T. K. H. Protein Electrophoresis. , (2012).
- Andrew, S. M., Titus, J. A., Zumstein, L. Dialysis and concentration of protein solutions. Current Protocols in Toxicology, Appendix 3. , 1-5 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. Assembly, loading, and alignment of an analytical ultracentrifuge sample cell. Journal of Visualized Experiments. (33), e1530(2009).
- Padavannil, A., Brautigam, C. A., Chook, Y. M. Molecular size analysis of recombinant importin-histone complexes using analytical ultracentrifugation. Bio-protocol. 10 (10), 3625(2019).
- Zhao, H., Brautigam, C. A., Ghirlando, R., Schuck, P. Overview of current methods in sedimentation velocity and sedimentation equilibrium analytical ultracentrifugation. Current Protocols in Protein Science. , Chapter 20, Unit20.12 (2013).
- Schuck, P. Size-distribution analysis of macromolecules by sedimentation velocity ultracentrifugation and Lamm equation modelling. Biophysical Journal. 78 (3), 1606-1619 (2000).
- Kumar, A., Kumar Singh, A., Chandrakant Bob de, R., Vasudevan, D. Structural characterization of Arabidopsis thaliana NAP1-related protein 2 (AtNRP2) and comparison with its homolog AtNRP1. Molecules. 24 (12), 2258(2019).
- Liu, W. H., Roemer, S. C., Port, A. M., Churchill, M. E. A. CAF-1-induced oligomerization of histones H3/H4 and mutually exclusive interactions with Asf1 guide H3/H4 transitions among histone chaperones and DNA. Nucleic Acids Research. 45 (16), 9809(2017).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Newman, E. R., et al. Large multimeric assemblies of nucleosome assembly protein and histones revealed by small-angle X-ray scattering and electron microscopy. Journal of Biological Chemistry. 287 (32), 26657-26665 (2012).
- Edlich-Muth, C., et al. The pentameric nucleoplasmin fold is present in Drosophila FKBP39 and a large number of chromatin-related proteins. Journal of Molecular Biology. 427 (10), 1949-1963 (2015).
- Franco, A., et al. Structural insights into the ability of nucleoplasmin to assemble and chaperone histone octamers for DNA deposition. Scientific Reports. 9 (1), 9487(2019).
- Xiao, H., Jackson, V., Lei, M. The FK506-binding protein, Fpr4, is an acidic histone chaperone. FEBS Letters. 580 (18), 4357-4364 (2006).
- Graziano, G. Role of hydrophobic effect in the salt-induced dimerization of bovine beta-lactoglobulin at pH 3. Biopolymers. 91 (12), 1182-1188 (2009).
- Burgess, R. J., Zhang, Z. Histone chaperones in nucleosome assembly and human disease. Nature Structural and Molecular Biology. 20 (1), 14-22 (2013).
- Donham, D. C., Scorgie, J. K., Churchill, M. E. The activity of the histone chaperone yeast Asf1 in the assembly and disassembly of histone H3/H4-DNA complexes. Nucleic Acids Research. 39 (13), 5449-5458 (2011).
- Avvakumov, N., Nourani, A., Côté, J. Histone chaperones: Modulators of chromatin marks. Molecular Cell. 41 (5), 502-514 (2011).
- Ransom, M., Dennehey, B. K., Tyler, J. K. Chaperoning histones during DNA replication and repair. Cell. 140 (2), 183-195 (2010).
- Chu, X., et al. Importance of electrostatic interactions in the association of intrinsically disordered histone chaperone Chz1 and histone H2A.Z-H2B. PLoS Computational Biology. 8 (7), 1002608(2012).
- Heidarsson, P. O., et al. Disordered proteins enable histone chaperoning on the nucleosome. bioRxiv. , (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados