Method Article
Um kit de ferramentas de transcrição de transcrição de estreptomíceos de alto rendimento para biologia sintética e aplicações de produtos naturais
Neste Artigo
Resumo
Este protocolo detalha um método aprimorado para sintetizar altos rendimentos de proteínas recombinantes a partir de um sistema de tradução de transcrição livre de células (TX-TL) livre de células .
Resumo
Estreptomias spp. são uma das principais fontes de antibióticos clínicos e produtos químicos industriais. Estreptomyces venezuelae ATCC 10712 é uma cepa de rápido crescimento e um produtor natural de clororamfenicol, jadomicina e espikromicina, o que o torna um candidato atraente como um chassi de biologia sintética de última geração. Portanto, ferramentas genéticas que aceleram o desenvolvimento da S. venezuelae ATCC 10712, bem como outros modelos de Estreptomia spp. são altamente desejáveis para a engenharia e descoberta de produtos naturais. Para isso, um sistema dedicado de isento de células S. venezuelae ATCC 10712 é fornecido neste protocolo para permitir a expressão heteróloga de alto rendimento de genes G+C (%) elevados. Este protocolo é adequado para reações em lote de pequena escala (10-100 μL) em formato de placa de 96 ou 384 poços, enquanto as reações são potencialmente escaláveis. O sistema livre de células é robusto e pode alcançar altos rendimentos (~5-10 μ M) para uma gama de proteínas recombinantes em uma configuração mínima. Este trabalho também incorpora um amplo instrumento de plasmídeo para medição em tempo real de mRNA e síntese proteica, bem como a coloração de fluorescência em gel de proteínas marcadas. Este protocolo também pode ser integrado com fluxos de trabalho de caracterização de expressão genética de alto throughput ou o estudo de vias enzimóricas de genes G+C (%) altos presentes nos genomas de Actinomycetes.
Introdução
Os sistemas de tradução de transcrição sem células (TX-TL) fornecem uma plataforma ideal de prototipagem para biologia sintética para implementar ciclos rápidos de projeto-construção-teste-aprender, a estrutura de engenharia conceitual para biologia sintética1. Além disso, há um crescente interesse em sistemas TX-TL para a produção de proteína recombinante de alto valor em um ambiente de reação aberta22, por exemplo, para incorporar aminoácidos não-padrão em conjugados de anticorpos 3. Especificamente, tX-TL requer um extrato celular, DNA plasmídeo ou linear, e uma solução energética para catalisar a síntese proteica em reações em lote ou semicontinuos. Enquanto o Escherichia coli TX-TL é o sistema dominante sem células, vários sistemas TX-TL não-modelo emergentes têm atraído atenção para diferentes aplicações4,5,6,7,8. As principais vantagens do TX-TL incluem escala flexível (nanoliter para escala de litro)9,10, reprodutibilidade forte e fluxos de trabalho automatizados8,11,12. Em particular, a automação do TX-TL permite a caracterização acelerada de partes genéticas e elementos regulatórios8,12,13.
Em termos de configuração de reação, o TX-TL requer fontes de energia primárias e secundárias, bem como aminoácidos, cofatores, aditivos e uma sequência de DNA de modelo. Os triptofatos nucleotídeos (NTPs) fornecem a principal fonte de energia para conduzir mRNA inicial (ATP, GTP, CTP e UTP) e síntese proteica (apenas ATP e GTP). Para aumentar os rendimentos de TX-TL, os NTPs são regenerados através do catabolismo de uma fonte de energia secundária, como maltose14, maltodextrina15, glicose14, 3-fosfogliceto (3-PGA)16, fosfoenolpyruvate17 e L-glutamate18. Esta atividade metabólica inerente é surpreendentemente versátil, mas mal estudada, especialmente em sistemas TX-TL emergentes. Cada fonte de energia tem propriedades e vantagens distintas em termos de rendimento ATP, estabilidade química e custo, o que é uma consideração importante para reações TX-TL dimensionadas. Até agora, os protocolos atuais para E. coli TX-TL atingiram até 4,0 mg/mL (~157 μM) para o modelo de proteína fluorescente verde (GFP), usando uma mistura de 3-PGA (30 mM), maltodextrina (60 mM) e costela D-ose (30 mM) como fonte de energia secundária19.
Recentemente, houve um crescente interesse em estudar vias biossintéticas metabólicas secundárias nos sistemas TX-TL20,21,22. Especificamente, a Actinobacteria é uma das principais fontes de metabólitos secundários, incluindo antibióticos e produtos químicos agrícolas23,24. Seus genomas são enriquecidos com os chamados aglomerados genéticos biossintéticos (BGCs), que codificam caminhos enzimáticos para a biossíntese metabólica secundária. Para o estudo de partes genéticas actinobactérias e vias biossintéticas, uma série de sistemas TX-TL baseados em Estreptomíces foram recentemente desenvolvidos5,6,25,26. Estes sistemas especializados de Estreptomia TX-TL são potencialmente benéficos pelas seguintes razões: [1] provisão de um ambiente de dobramento de proteína nativa para enzimas de Estreptomia spp.26; [2] acesso a um pool de tRNA ideal para alta expressão genética G+C (%) ; [3] metabolismo primário ativo, que potencialmente pode ser sequestrado para o fornecimento de precursores biossintéticos; e [4] provisão de enzimas, precursores ou cofatores do metabolismo secundário presentes no extrato celular nativo. Assim, um kit de ferramentas S.venezuelae TX-TL de alto rendimento foi recentemente estabelecido para aproveitar essas capacidades únicas5.
Streptomyces venezuelae é um emergente hospedeiro para biologia sintética com uma rica história em biotecnologia industrial5,27,28,29 e como um sistema modelo para estudar a divisão celular e regulação genética em Actinobacteria30,31,32. A cepa principal, S. venezuelae ATCC 10712, possui um genoma relativamente grande de 8,22 Mb com 72,5% de teor de G+C (%) (número de adesão: CP029197), que codifica 7377 sequências de codificação, 21 rRNAs, 67 tRNAs e 30 clusters genéticos biossintéticos27. Em biologia sintética, s. venezuelae ATCC 10712 é um chassi atraente para a expressão heteróloga de vias biossintéticas. Ao contrário da maioria das outras manchas de Estreptomia, ela fornece várias vantagens fundamentais, incluindo um tempo de duplicação rápida (~40 min), uma extensa gama de ferramentas genéticas e experimentais5,28, falta de agrupamento micelial e esporulação em mídia líquida28,33. Vários estudos também demonstraram o uso de S. venezuelae para produção heteróloga de uma variedade diversificada de metabólitos secundários, incluindo poliketídeos, peptídeos ribossômicos e nãoribosomais34,35,36,37,38. Essas características combinadas fazem dessa cepa um hospedeiro microbiano atraente para aplicações de biologia sintética e engenharia metabólica. Embora s. venezuelae não seja o modelo dominante de Estreptomia para expressão genética heteróloga, com desenvolvimentos adicionais, ele é preparado para uso mais amplo dentro da descoberta de produtos naturais.
Este manuscrito apresenta um protocolo detalhado (Figura 1) para um sistema S. venezuelae TX-TL de alto rendimento, que foi atualizado do protocolo original publicado anteriormente26. Neste trabalho, a solução energética e as condições de reação foram otimizadas para aumentar o rendimento da proteína até 260 μg/mL para a proteína repórter mScarlet-I em uma reação em lote de 4h, 10 μL, utilizando um plasmídeo padrão, pTU1-A-SP44-mScarlet-I. Este plasmídeo foi especificamente projetado para permitir vários métodos de detecção de expressão proteica. O protocolo também é simplificado, enquanto o sistema de energia foi otimizado para reduzir a complexidade e o custo de configurar reações livres de células sem comprometer o rendimento. Juntamente com o sistema TX-TL otimizado, uma biblioteca de partes genéticas foi desenvolvida para a expressão genética de ajuste fino e como ferramentas fluorescentes para monitorar tx-tl em tempo real, criando assim uma plataforma versátil para prototipagem de expressão genética e caminhos biossintéticos de produtos naturais de Estreptomyces spp. e Actinobacteria relacionada.
Neste trabalho, o plasmídeo padrão recomendado (pTU1-A-SP44-mScarlet-I) pode ser usado para estabelecer o fluxo de trabalho S. venezuelae TX-TL em um novo laboratório e está disponível no AddGene (ver Tabela Suplementar S1). pTU1-A-SP44-mScarlet-I oferece ao usuário a flexibilidade para estudar outros quadros de leitura aberta (ORFs). O ORF mScarlet-I é otimizado para a expressão genética S. venezuelae. O promotor SP44 é um forte promotor constitutivo que é altamente ativo tanto em E. coli quanto em Streptomyces spp.39. O plasmídeo tem dois locais de enzima de restrição única (NdeI, BamHI) para permitir a subs clonagem de novos ORFs no quadro com um sistema de etiqueta de aglutinante arsádico de coresceína (FlAsH) do terminal C. Alternativamente, ambas as tags podem ser removidas com a inclusão de um códon stop após a subs clonagem de um novo gene. Com este vetor base, a expressão de alto rendimento de uma gama de proteínas tem sido demonstrada, ou seja, proteínas da via de biossíntese de oxitetracíclina e um síntese de peptídeo não cansarático não característico (NRPS) de Estreptomíces rimosus (Figura 2). Em termos de detecção de mRNA, o plasmídeo padrão pTU1-A-SP44-mScarlet-I contém um aptamer dBroccoli (na região 3'-não traduzida) para detecção com a sonda 3,5-difluoro-4-hydroxibenzylidene imidazolinone (DFHBI). Para maior flexibilidade, um porta-ferramentas de peças MoClo compatíveis com EcoFlex40 também foi disponibilizado no AddGene, incluindo um vetor de visão de visor de estreptomíceos compatível com EcoFlex (pSF1C-A-RFP/pSF2C-A-RFP) e uma gama de plasmídeos de variante pTU1-A-SP44 expressando proteína de fluorescência verde superpatosa (sfGFP), mScarlet-I, mVenus-I e β-glucurdasonie (GUS). Em particular, o plasmídeo pSF1C-A é derivado do pAV-gapdh28 e é curado de locais BsaI/BsmBI para montagem moClo. pSF1C-A-RFP/pSF2C-A-RFP é equivalente ao pTU1-A-RFP/pTU2-A-RFP do EcoFlex40, mas contém funcionalidade adicional para conjugação e integração cromossômica em Estreptomíces spp. usando o sistema de integração phiC3128.
A primeira etapa do protocolo envolve o crescimento do S. venezuelae ATCC 10712 ou uma cepa intimamente relacionada, colheita celular em fase de médio exponencial, etapas de lavagem celular e equilíbrio em buffers S30A e S30B. Esta etapa requer três dias, e o tempo para o crescimento celular pode ser usado para preparar os componentes restantes conforme descrito abaixo. As células colhidas são então líficas por sônica, esclarecidas e submetidas a uma reação de escoado. Nesta fase final de preparação, os extratos celulares podem ser preparados para armazenamento a longo prazo a -80 °C para minimizar a perda de atividade. Para a montagem de reações TX-TL usando este protocolo, é apresentado um Streptomyces Master Mix (SMM), com a opção de um formato de Solução de Energia Mínima (MES) que dá rendimentos comparáveis. Além disso, recomenda-se a estria de uma nova cultura de S. venezuelae ATCC 10712 a partir de um estoque de glicerol de -80 °C em uma placa de ágar gym e incubar a 28 °C por pelo menos 48-72 h até que colônias únicas sejam visíveis. Apenas culturas frescas devem ser usadas para as seguintes etapas.
Protocolo
NOTA: Veja a Tabela 1 e a Tabela 2 para receitas para placa de ágar e tampão de lavagem S30A e S30B.
1. Elaboração de soluções e orientação geral
- Mantenha todas as soluções, células (pós-crescimento) e extratos celulares no gelo após a preparação, a menos que uma exceção seja declarada.
- Estoques de armazenamento para 1 M Mg-glutamato, 4 M K-glutamato, 40% (w/v) PEG 6000, 1 g/mL de ácido polivinylsulfônico à temperatura ambiente e todos os outros estoques a -80 °C. Minimizar o número de ciclos de congelamento para evitar a degradação química.
- Para a elaboração de estoques de solução energética (ver Tabela 3) como 3-PGA (requer ajuste de pH), siga as orientações fornecidas no protocolo E. coli TX-TL41.
NOTA: Todos os componentes são totalmente solúveis em ddH2O e armazenados como alíquotas no congelador -80 °C. - Descongelar estoques individuais ou soluções energéticas (descritas posteriormente) no gelo. Aqueça o estoque de aminoácidos a 42 °C com vórtice por ~15-30 min para solubilizar todos os aminoácidos.
- À medida que alguns aminoácidos (L-Cys, L-Tyr, L-Leu) precipitam no gelo, minimizando o tempo de descanso, deixe esta solução à temperatura ambiente e use um vórtice para dissolver.
- Adicione os volumes calculados (Tabela 3) de soluções de estoque e água e misture bem usando um vórtice.
- Alíquotar a solução de energia como alíquotas de 20-100 μL por tubo, ou conforme desejado, no gelo e armazenar a -80 °C até usar mais.
2. Preparação de células S. venezuelae ATCC 10712
- Dia 1-Preparação de mídia/buffer e pré-cultura durante a noite
- Prepare 1 L de meio líquido gym estéril em um frasco de 2 L perplexo, conforme descrito na Tabela 1. Consulte a Tabela de Materiais para obter fontes de equipamentos/químicos/reagentes.
- Prepare 1 x 50 mL de meio líquido GYM estéril em um frasco de Erlenmeyer de 250 mL, conforme descrito na Tabela 1.
- Prepare 100 mL de 1 M HEPES-KOH pH 7.5, 100 mL de 1 M MgCl2 e 500 mL de soluções de 4 M NH4Cl para fazer 1 L de S30A e 1 L de tampões de lavagem S30B. Veja a Tabela 2 para as receitas.
- Prepare a pré-cultura da noite para o dia. Pré-aqueça os estéreis 50 mL de meio líquido GYM em um frasco de Erlenmeyer de 250 mL a 28 °C por 30 min.
- Inocular uma única colônia de S. venezuelae ATCC 10712 (ou tensão relacionada) a partir de uma placa de ágar gym em pré-reboque 50 mL de gym médio líquido e incubar a 28 °C, 200 rpm por 16 h (pré-cultura durante a noite).
- Dia 2- Prepare a pré-cultura diurna e a principal cultura de crescimento.
- Pré-quente 50 mL de meio líquido GYM estéril em um frasco de 250 mL Erlenmeyer a 28 °C por 30 min.
- Transfira 1 mL de pré-cultura durante a noite para 50 mL pré-aquecidos de meio líquido GYM e incubar a 28 °C, 200 rpm por 8h (pré-cultura diurna).
- Após este período de crescimento, verifique o OD600 em um espectotúmetro usando uma diluição de 1:10 com meio GYM estéril em um cuvette plástico de 1 mL (comprimento do caminho) de 1 cm.
NOTA: O OD600 deveria ter atingido pelo menos 3-4. Se houver um crescimento ruim, é aconselhável repetir as etapas 2.2.1-2.2.2. - Subcultura 0,25 mL de pré-cultura diurna em 1 L de meio gym líquido em frascos de 2 L perplexos.
- Agite durante a noite a 28 °C, 200 rpm por 14 h.
- Células do 3º dia
- Após o período anterior de incubação (14h), registo o OD600 da cultura principal. Diluir a cultura noturna 1:10 com meio gym fresco para medição OD600 .
NOTA: O OD600 deveria ter atingido 3.0-4.0 nesta fase. - Se OD600<3.0, aumente a velocidade de agitação para 250-300 rpm e cresça até que um OD600 de 3.0 seja atingido. Cresça por não mais do que 2 h adicionais (16 h no total).
- Se OD600>3.0, transfira as culturas para recipientes de centrifugação e esfrie rapidamente no gelo molhado por 30 minutos.
- Enquanto espera que a cultura celular esfrie no gelo, prepare 4 mL de tampões frescos de 1 M dithiothreitol (DTT), S30A e S30B, como descrito na Tabela 1, e mantenha-os no gelo. Consulte a Tabela de Materiais para a fonte química/reagente.
- Pré-pesar um tubo vazio de centrífugas de 50 mL e pré-frio a -20 °C.
- Adicione 2 mL de 1 M DTT a 1 L de tampão S30A no gelo e misture bem.
NOTA: Adicione DTT aos buffers de lavagem S30A e S30B somente antes de usá-los. - Centrífugas a 6.000 × g, 4 °C, 10 min, e descartam cuidadosamente o sobrenante em um movimento rápido e único.
NOTA: Se a pelota estiver perturbada, maximize a retenção celular com meio GYM residual e continue o protocolo. - Adicione 500 mL de tampão S30A e resuspenque as células sacudindo as garrafas de centrifugação vigorosamente até que as células sejam dispersas homogêneas.
- Centrifugar as células a 6.000 × g, 4 °C, 6 min, e descartar cuidadosamente o sobrenante.
NOTA: Embora a pelota de célula seja mais firme neste momento, algumas células permanecerão em suspensão (ver Figura 1). Trate como descrito em 2.3.7 e retenha o maior número possível de células. - Repetição de passos 2.3.8-2.3.9.
- Adicione 2 mL de 1 M DTT a 1 L de tampão S30B no gelo e misture bem. Adicione 500 mL de tampão S30B às células. Repita o passo 2.3.9.
- Resuspenda a pelota de célula em 10 mL de tampão S30B e transfira para o tubo de centrífuga pré-pesado de 50 mL. Se necessário, transfira as células residuais com um buffer adicional de 5-10 mL de S30B. Encha a 50 mL com S30B.
- Centrífugas a 6.000 × g, 4 °C, 10 min e descartam cuidadosamente o sobrenante.
- Repita o passo 2.3.13.
- Aspire cuidadosamente o supernatante S30B restante com uma pipeta de 100-200 μL.
- Pesar a pelota de célula molhada.
NOTA: O peso típico da pelota de célula molhada para 1 L de cultura de GINÁSTICA durante a noite (OD600 = 3,0) é ~4,5 g. - Para cada 1 g de células molhadas, adicione 0,9 mL de tampão S30B. Resuspend as células usando uma pipeta Pasteur ou vórtice.
- Centrifugar brevemente (~10 s) até 500 × g para sedimentar as células.
NOTA: O protocolo pode ser pausado neste momento, e as células podem ser congeladas em nitrogênio líquido ou gelo seco e armazenadas a -80°C. Por segurança, use equipamentos de proteção individual (EPIs) apropriados ao manusear nitrogênio líquido, incluindo escudos faciais e luvas.
- Após o período anterior de incubação (14h), registo o OD600 da cultura principal. Diluir a cultura noturna 1:10 com meio gym fresco para medição OD600 .
3. Lise celular por sônica para obter o extrato celular bruto
NOTA: Nesta fase, o usuário pode optar por interromper as células por sônicação em frações de 1 mL (opção 1) ou como uma suspensão celular maior (5 mL) em um tubo de 50 mL (opção 2). Ambas as opções foram detalhadas abaixo para garantir a reprodutibilidade, já que o volume final da suspensão celular pode mudar devido à perda de células durante as etapas anteriores de colheita e lavagem. Um novo usuário deve tentar a opção 2.1 primeiro para estabelecer o protocolo.
- Lysis celular por sonicação em frações de 1 mL
- Usando uma ponta de pipeta de 1 mL (corte a extremidade da ponta para aumentar o tamanho do furo), transfira 1 mL da suspensão celular para tubos de microcentrifuuge de 2 mL.
NOTA: Se as células estiverem congeladas, descongele rapidamente o tubo de 50 mL contendo a pelota em água morna antes da lise celular. Transfira o tubo para o gelo molhado assim que a pelota começar a descongelar, e esfrie por 10 minutos. - Coloque cada tubo de microcentrifuuge em um béquer de água gelada, usando um rack de tubo de plástico para segurar o tubo para sônicação.
NOTA: Devido à sensibilidade do extrato celular ao superaquecimento, é fundamental garantir que os tubos não aqueçam para evitar a precipitação proteica e reduzam a atividade enzimática. - Use uma sonda sonicator com uma ponta de 3 mm de diâmetro e limpe-a com 70% (v/v) de etanol e água duplamente destilada (ddH2O). Abaixe a ponta do sonicador na suspensão celular até ficar ~1 cm abaixo da superfície líquida.
- Insira as seguintes configurações no sonicator: frequência de 20 kHz, 65% de amplitude, pulso de 10 s TEMPO ON, 10 s pulsos OFF time, 1 min tempo total de sônicação.
- Execute o protocolo de sônica. Mova o tubo para cima/para baixo e para os lados durante os dois primeiros ciclos de descanso para garantir que as células sejam uniformemente sônicas. Regisso da entrada de energia.
NOTA: Para segurança, use proteção auditiva adequada durante a sônica. A viscosidade diminuirá à medida que as células são interrompidas, e a pelota de célula molhada creme pálido deve se transformar em um fluido marrom homogêneo. A entrada de energia recomendada é de 240 J por mL de células molhadas. Se as células forem apenas parcialmente lisecidas, a suspensão ainda aparecerá cor de creme com aglomerados viscosos de células, particularmente nas laterais do tubo. - Inverta o tubo 2-3 vezes e repita a sônica para um ou dois ciclos adicionais de 10 s, misturando-se com frequência até que as células sejam totalmente interrompidas.
- Usando uma ponta de pipeta de 1 mL (corte a extremidade da ponta para aumentar o tamanho do furo), transfira 1 mL da suspensão celular para tubos de microcentrifuuge de 2 mL.
- Lysis celular por sonicação de uma suspensão celular de 5 mL
- Se as células estiverem congeladas, descongele rapidamente o tubo de 50 mL contendo a pelota em água morna com agitação antes da lise celular. Transfira o tubo para o gelo molhado assim que a pelota começar a descongelar, e esfrie por 10 minutos.
- Gire brevemente o tubo a 500 x g para sedimentar as células.
- Coloque o tubo de 50 mL em um béquer de água gelada para sônicação.
NOTA: Devido à sensibilidade do extrato celular ao superaquecimento, é fundamental garantir que os tubos não aqueçam para evitar a precipitação proteica e reduzam a atividade enzimática. - Use uma sonda sonicator com uma ponta de 6 mm de diâmetro e limpe-a com 70% (v/v) de etanol e ddH2O (veja o esquema visual da sonda de 6 mm na Figura 1). Abaixe a ponta do sonicador na suspensão celular (~5 mL) até ficar ~1 cm abaixo da superfície líquida.
- Insira as seguintes configurações no sonicator: frequência de 20 kHz, 65% de amplitude, 10 s pulso ON time, 10 s pulsos OFF time, 1 min tempo total de sonicação por mL de células molhadas (5 min no total).
- Execute o protocolo de sônica. Mova o tubo para cima/para baixo e para os lados durante os dois primeiros ciclos de descanso para garantir que as células sejam uniformemente sônicas.
NOTA: Para segurança, use proteção auditiva adequada durante a sônica. A viscosidade diminuirá à medida que as células são interrompidas, e a pelota de célula molhada creme pálido deve se transformar em um fluido marrom homogêneo. Regisso da entrada de energia. Recomenda-se uma entrada de energia ideal de 240 J por mL de células molhadas (~1200 J no total a partir de 5 min de sonicação). - Se algumas células permanecerem intactas, siga a orientação do passo 3.1.5.
- Transfira os extratos celulares para tubos de microcentrifuuge de 2 mL.
4. Esclarecimento de extrato celular e reação de escoado
- Centrifugar as células líficas a 16.000 × g por 10 min a 4 °C para remover os detritos celulares. Transfira o supernatante para tubos de microcentrifuus de 1,5 mL como alíquotas de 1 mL.
- Realize a reação de escorrida para os extratos celulares. Incubar os tubos de 1,5 mL contendo os extratos celulares a 30 °C por 60 min em um bloco de calor ou incubadora sem tremer.
- Centrifugar os extratos celulares a 16.000 × g por 10 min a 4 °C. Acumule os supernantes em um tubo de centrífuga de 15 mL. Misture o supernatante invertendo o tubo cinco vezes até ficar homogêneo, em seguida, mantenha-o no gelo. Inverta suavemente para evitar a formação de bolhas de ar.
- Diluir 10 μL do extrato celular 100 vezes com tampão S30B e medir a concentração total de proteína usando um ensaio de Bradford com três repetições técnicas (ver Material Suplementar S2 para orientação de ensaio de Bradford).
- Se a concentração proteica for de 20-25 mg/mL, transfira os extratos celulares como alíquotas de 100 μL em novos tubos de 1,5 mL, congele em nitrogênio líquido e armazene a -80 °C.
NOTA: Para segurança, use EPI apropriado ao manusear nitrogênio líquido, incluindo escudos faciais e luvas. - Se a concentração de proteína for <20 mg/mL, repita as etapas brutas de preparação do extrato bruto para garantir que o extrato celular de alta qualidade e os rendimentos de TX-TL sejam comparáveis ao trabalho publicado anteriormente5.
5. Preparação do modelo de DNA plasmídeo
- Purifique o plasmídeo pTU1-A-SP44-mScarlet-I (origem pUC19) de uma cepa de plasmídeo E. coli recém-transformada (DH10β, JM109) cultivada em 50 mL de cultura LB (com carbenicilina de 100 mg/mL) usando um kit apropriado de purificação de DNA plasmídeo conforme as instruções do fabricante.
- Elute o plasmídeo em 2 x 300 μL de água sem nuclease e combine as frações.
- Adicione 0,1 volumes (66 μL) de acetato de sódio de 3 M (pH 5.2).
- Adicione 0,7 volumes (462 μL) de isopropanol.
- Incubar o DNA a -20 °C por 30 min.
- Centrifugar a 16.000 × g por 30 min a 4 °C e descartar o supernaspe.
- Adicione 2 mL de 70% (v/v) de etanol à pelota de DNA.
- Inverta o tubo 3-4 vezes para resuspensar a pelota de DNA plasmida.
- Centrifugar a 16.000 × g por 5 min a 4 °C e descartar o supernaspe.
- Repita as etapas 5.7-5.9 e remova todo o líquido visível.
- Seque a pelota de DNA por 10-30 min ou seque por 5 minutos com uma centrífuga de vácuo.
- Resuspenque a pelota seca com 600 μL de ddH2O sem nuclease.
- Meça a concentração e pureza do DNA usando um espectrofotômetro.
- Prepare 50-100 μL aliquots e armazene a -20 °C.
NOTA: Recomenda-se uma alta concentração de DNA na faixa de 500-1000 ng/μL devido às restrições de volume apertadas das reações livres de células. Diluir o estoque de DNA plasmídeo para 80 nM; 168 ng/μL pTU1-A-SP44-mScarlet-I plasmid é equivalente a 80 nM.
6. Preparação da solução Streptomyces Master Mix (SMM)
- Solução de aminoácido
- Use o kit amostrador de aminoácidos para evitar erros manuais e reduzir o tempo de preparação, seguindo as instruções fornecidas on-line pelo fabricante.
- Diluir a solução de estoque de aminoácidos de 20x usando ddH2O para uma concentração final de 6 mM (5 mM L-Leu).
- Diluir ainda mais para 2,4 mM (2 mM L-Leu) dentro da solução SMM de 2,4x (ver Tabela 3).
NOTA: A concentração final na reação TX-TL é de 1 mM 19x aminoácidos e 0,83 mM L-Leu.
- Solução energética e aditivos
- Prepare os outros componentes da solução 2.4x SMM seguindo a receita descrita na Tabela 3.
- Alternativamente, prepare uma Solução de Energia Mínima 2,4x (MES) seguindo a receita descrita na Tabela 3.
7. Configuração de uma reação padrão S. venezuelae TX-TL
- Descongele o extrato celular, solução SMM (ou MES) e DNA plasmídeo no gelo. Pré-chill uma placa de 384-bem a -20 °C.
- Configure reações TX-TL onde 25% do volume é DNA plasmídeo, 33,33% é extrato celular e 41,67% é solução SMM ; mantê-los no gelo para evitar viés de tempo de início.
NOTA: Foi fornecido um modelo TX-TL padrão (Tabela 4) para calcular o volume de reagentes necessários com base no número de reações. O volume padrão para uma reação de 33 μL é o seguinte: 11 μL de extrato celular, 13,75 μL de SMM e 8,25 μL de DNA plasmídeo. - Vórtice suavemente a mistura para ~5 s em uma configuração de baixa velocidade para garantir que a solução seja homogênea. Evite a formação de espuma/bolha.
- Transfira 10 alíquotas de μL em três poços de uma placa de 384 poços como um triplicado técnico sem introduzir bolhas de ar. Sele a placa com uma tampa transparente e gire a 400 × g para 5 s.
- Incubar a reação a 28 °C, seja em uma incubadora (para leituras de ponto final) ou em um leitor de placas sem tremer.
NOTA: As reações normalmente requerem 3-4 h para atingir a conclusão. Consulte material suplementar S2 para obter orientação sobre um leitor de placas e medidas padrão mScarlet-I.
Resultados
Este protocolo detalhado é fornecido como um exemplo para ajudar o usuário a estabelecer um sistema TX-TL de Estreptomíces baseado na cepa do modelo S. venezuelae ATCC 10712 (Figura 1). O usuário pode procurar estudar outras cepas de Estreptomia; no entanto, os estágios de crescimento/colheita de outras cepas com tempos de duplicação mais longos ou preferências distintas de crescimento precisarão ser otimizados sob medida para alcançar resultados de pico. Para o resultado representativo, a proteína fluorescente mScarlet-I do plasmídeo padrão pTU1-A-SP44-mScarlet-I (Figura 2 e Figura 3) foi otimizada para fornecer expressão de alto rendimento no S. venezuelae TX-TL com uma gama de métodos de detecção (SDS-PAGE, fluorescência). Além disso, este plasmídeo padrão foi modificado para demonstrar a síntese de uma gama de enzimas metabólicas secundárias de S. rimosus (Figura 2)5. Finalmente, um fluxo de trabalho potencial para a biosíntese de produtos naturais dimensionados é mostrado como um esquema para o uso de um caminho modelo a partir dos estágios iniciais da biossíntese de heme. O fluxo de trabalho é potencialmente adaptável a outras vias biossintéticas metabólicas secundárias. Como diretriz, este protocolo deve fornecer um rendimento mínimo de 2,8 μM para sfGFP e 3,5 μM para mScarlet-I/mVenus a partir da expressão plasmids fornecidos em AddGene. Esses números permitem a variação típica do lote (até 28%) observada em dados anteriores5, embora os rendimentos superiores a 10 μM mScarlet-I tenham sido alcançados com lotes ótimos (dados não publicados).
Medindo S. venezuelae TX-TL do gene mScarlet-I usando cinco métodos distintos
A expressão do plasmídeo padrão pTU1-A-SP44-mScarlet-I é mostrada, com a medição da expressão mScarlet-I utilizando cinco métodos diferentes: 1. medição de fluorescência em tempo real do mRNA usando o aptamer dBroccoli, 2. medição de fluorescência em tempo real da proteína mScarlet-I imatura usando o sistema de tag FlAsH, 3. medição de fluorescência em tempo real da proteína mScarlet-I madura, 4. fluorescência em gel de mScarlet-I usando tag FlAsH, tag FlAsH, tag e 5. Coloração azul coomassie de proteínas totais livres de células. Para esses dados, as reações foram configurados em tubos de microcentrifuus de 2 mL como reações de 33 μL (para amostras de ponto final) ou como um triplicado técnico de 10 μL em placas de 384 poços em um leitor de placas. Uma proteína mScarlet-I de marca tripla (N-terminal His6, C-terminal Flag e C-terminal FlAsH) foi separadamente purificada para criar um padrão de calibração para medições, usando o plasmídeo pET15b-mScarlet-I, que é descrito ainda mais no Material Suplementar S2. Os dados para esses experimentos são mostrados na Figura 3. Mais detalhes sobre o método de coloração da fluorescência em gel estão disponíveis no Material Suplementar S3.
S. venezuelae TX-TL de biosíntese de heme em estágio inicial
Para servir como modelo de via biossintética de produto natural, a biossíntese 'one-pot' do uroporphyrinogen III (uro'gen III) foi realizada utilizando-se a expressão pTU1-A-SP44-hemC-hemD/cysGA-hemid5. Este modelo de via biossintética foi escolhido como uro'gen III é altamente sensível ao oxigênio e rapidamente oxida (perda de seis elétrons) para uroporphyrina III, que exibe forte fluorescência vermelha. Isso permite a fácil detecção da reação em tempo real usando medidas de fluorescência e/ou HPLC-MS (Figura 4), como descrito anteriormente5. Além disso, essas reações foram estudadas utilizando-se um lote ou método semicontinuoso. Uma reação semicontinuosa é uma estratégia, que usa um dispositivo de microdiálise42,43 que fornece energia adicional (NTPs, fonte de energia secundária) e aminoácidos para prolongar o tempo de reação e aumentar os rendimentos da síntese proteica. Aqui, o método semicontinuoso é usado para aumentar a reação do modelo de heme e separar as proteínas TX-TL do produto de reação para facilitar a purificação e análise pelo HPLC-MS. Mais detalhes dos métodos estão disponíveis no Material Suplementar S4 ou para dados, consulte trabalhos anteriores5. Reações semicontinuosas sem células também são descritas em trabalhos anteriores42,43. O fluxo de trabalho esquemático de exemplo demonstrado aqui (Figura 4) é potencialmente adaptável a outras vias biossintéticas de produtos naturais.
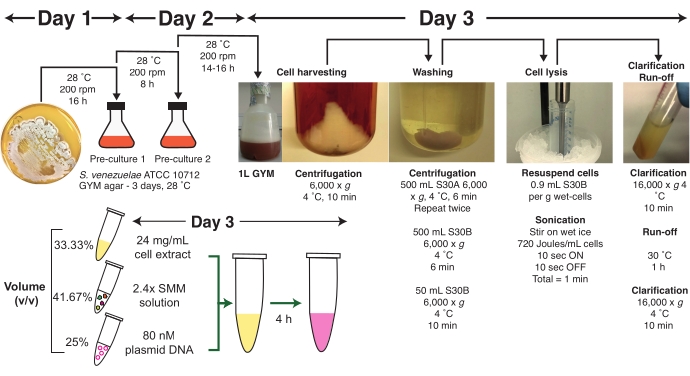
Figura 1: Visão geral do protocolo Streptomyces venezuelae TX-TL. Um resumo do protocolo é ilustrado, incluindo um prazo recomendado de três dias. O protocolo é dividido em estágios distintos de crescimento celular, colheita celular, lavagem celular, lise celular por sônica, esclarecimento, reação de escoamento, preparação de mistura mestre (SMM), preparação de DNA plasmídeo e o conjunto de reação TX-TL. O protocolo completo é descrito em detalhes dentro do texto, juntamente com orientações e dicas práticas. Abreviaturas: SMM = Streptomyces Master Mix; TX-TL = transcrição-tradução. Clique aqui para ver uma versão maior desta figura.

Figura 2: Síntese de proteínas de alto rendimento de genes G+C (%) (%) síntese de proteínas fluorescentes sfGFP, mVenus-I e mScarlet-I. (B) Síntese de enzimas biossintéticas de Estreptomia rimosus. Abreviação: EV = Vetor Vazio; NRPS = sintetização de peptídeo nãoribosomal. O valor é modificado a partir de 5. Consulte o protocolo e os arquivos suplementares para configuração e metodologia de reação. Clique aqui para ver uma versão maior desta figura.

Figura 3: Medição de TX-TL de cinco vias com o plasmídeo pTU1-A-SP44-mScarlet-I. (A) Design plasmídeo, incluindo as seguintes características: SP44 é um forte promotor constitutivo ativo em Streptomyces spp. e E. coli; pET-RBS é derivado dos plasmídeos de expressão pET e é altamente ativo tanto em Streptomyces spp. quanto em E. coli5,40; Gene mScarlet-I otimizado para codon, que codifica um derivado de proteína vermelho-fluorescente44; Tag FLAG terminal C para purificação de cromatografia de afinidade ou detecção de manchas ocidentais; Etiqueta FLAsH terminal C para rotulagem fluorescente para coloração em gel ou medição em tempo real da síntese de proteínas nascentes; aptamer dBroccoli para medição de mRNA em tempo real usando a sonda DFHBI; Bba_B0015 exterminador de transcrição, que são altamente eficientes em S. venezuelae ATCC 107125; marcador de resistência à anicilina; e pUC19 origem da replicação. (B) Expressão mRNA em tempo real, detectada com o aptamer dBroccoli e a sonda DFHBI (excitação 483-14 nm, emissão 530-30 nm). (C) Detecção de síntese de proteínas nascentes em tempo real com sonda fluorescente FLAsH-EDT2 (excitação 500-10 nm, emissão 535-10 nm). (D) Medição da fluorescência em tempo real da síntese de mScarlet-I (excitação 565-10 nm, emissão 600-10 nm). (E) Coloração em gel com a sonda fluorescente FLAsH-EDT2. (F) Coloração azul coomassie de proteínas TX-TL totais com padrão his6-mScarlet-I purificado para comparação. As reações foram executadas sob as condições descritas no protocolo com 40 nM de modelo de DNA plasmídeo. Todos os dados de fluorescência são representados como RFU, e as barras de erro (desvio padrão de três repetições técnicas) são representadas dentro de uma área sombreada cinza. Abreviaturas: TX-TL = transcrição-tradução; FlAsH = fluorescein arsenical hairpin; DFHBI = 3,5-difluoro-4-hidroxibenzylidene imidazolinone; RFU = unidades de fluorescência relativa. Clique aqui para ver uma versão maior desta figura.

Figura 4: Fluxo de trabalho esquemático para a reação semicontinuosa S. venezuelae TX-TL. Um exemplo de fluxo de trabalho para o produto natural TX-TL, usando o operon biossintético heme em estágio inicial e a análise a jusante pelo HPLC-MS. Reações e análises são detalhadas no material suplementar. O valor é modificado a partir de 5. Abreviaturas: SMM = Streptomyces Master Mix; TX-TL = transcrição-tradução; ALA = ácido 5-aminolevulinico; SPE = extração de fase sólida; ESI-MS = espectrometria de massa de pulverização eletrônica; HPLC-MS = espectrometria de massa líquida de alto desempenho. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Receita para GYM de crescimento bacteriano médio e placa de ágar GYM. Clique aqui para baixar esta Tabela.
Tabela 2: Reagentes para preparar tampões de lavagem S30A e S30B. Esta informação foi adaptada de Kieser et al. 45 Abreviação: DTT = dithiothreitol. Clique aqui para baixar esta Tabela.
Tabela 3: Receita para fazer as soluções S. venezuelae MES e SMM. Abreviaturas: MES = Solução de Energia Mínima; SMM = Streptomyces Master Mix; NTP = triphosfato nucleosídeo; PEG 6000 = polietileno glicol 6000; 3-PGA = 3-fosfoglicerate; G6P = glicose-6-fosfato; PVSA = ácido polivinylsulfônico. Clique aqui para baixar esta Tabela.
Tabela 4: Receita para reação S. venezuelae TX-TL. Abreviaturas: MES = Solução de Energia Mínima; SMM = Streptomyces Master Mix; TX-TL = transcrição-tradução. Clique aqui para baixar esta Tabela.
Tabela Suplementar S1: Plasmids para S. venezuelae TX-TL. Abreviação: TX-TL = transcrição-tradução. Clique aqui para baixar esta Tabela.
Material Suplementar S2: mScarlet-I calibração padrão preparação e medidas do leitor de placas. Clique aqui para baixar este Arquivo.
Material Suplementar S3: Métodos de tag FLAsH. Abreviação: FlAsH = fluorescein arsenical hairpin. Clique aqui para baixar este Arquivo.
Material Suplementar S4: Reação semicontinuosa, purificação e HPLC-MS. Clique aqui para baixar este Arquivo.
Discussão
Neste manuscrito, um protocolo S. venezuelae TX-TL de alto rendimento foi descrito com etapas detalhadas que são simples de conduzir tanto para usuários experientes quanto para novos usuários de sistemas TX-TL. Vários recursos dos protocolos Streptomyces45 e E. coli TX-TL41 foram removidos para estabelecer um protocolo mínimo, mas de alto rendimento para o S. venezuelae TX-TL5,26. O fluxo de trabalho recomendado aqui é garantir que s. venezuelae está crescendo rapidamente no meio rico escolhido, para ser capaz de inocular a cultura final à noite. Isso permite a colheita celular no pico de crescimento na manhã seguinte e permite que o usuário colmeia e prepara o extrato celular ativo no mesmo dia. Seguindo este protocolo simplificado, espera-se que um único pesquisador possa completar o protocolo convenientemente em uma estrutura de três dias. Um kit de ferramentas plasmídeos complementares também foi fornecido para o sistema S. venezuelae TX-TL, incluindo um sistema de plasmídeo de expressão forte (pTU1-A-SP44-mScarlet-I), que fornece ampla funcionalidade para análise de mRNA/proteína. Este plasmídeo padrão é alimentado pelo promotor sp44 constitutivo que é altamente ativo em uma gama de Estreptomyces spp. e em E. coli39. Para demonstrar o potencial inicial do kit de ferramentas S. venezuelae TX-TL, os resultados representativos mostram a síntese de alto rendimento de uma gama de proteínas fluorescentes, enzimas metabolitas secundárias e a biossíntese de uma via de produto natural modelo (da biosíntese heme).
No geral, o protocolo contém uma descrição detalhada do sistema S. venezuelae TX-TL, bem como dicas práticas para preparar os três componentes essenciais da reação TX-TL: (1) extrato celular, (2) solução Streptomyces Master Mix (SMM) e (3) DNA plasmídeo. Este protocolo não requer equipamentos especializados e requer apenas habilidades rotineiras de microbiologia e bioquímica; portanto, é acessível à maioria dos laboratórios. O protocolo é adequado para reações de pequena escala (10-100 μL) e de maior escala (~2,5 mL), embora alguma otimização do tamanho da reação/aeração possa influenciar o rendimento da proteína. O volume de reação recomendado é de 33 μL em um tubo de 2 mL ou 10 μL em uma placa de 384 poços. O extrato bruto leva cinco dias para ser preparado por uma única pessoa a partir de um estoque de glicerol. Cada litro (L) de cultura produz pelo menos 5 mL de extrato celular (equivalente a ~1500 x 10 μL TX-TL reações)-esta é uma estimativa conservadora e explica a perda de amostras durante as etapas de lavagem e esclarecimento do extrato celular. Cada etapa do protocolo é independente e pode ser otimizada pelo usuário para atender às suas necessidades. Uma grande limitação para todos os sistemas livres de células é a variação de lote46,47. Fatores genéricos incluem erro de pipetação, experiência do usuário, variação do lote de mídia e diferenças no equipamento. Introduzimos especificamente uma mistura mestre para minimizar o erro de pipetação e fornecemos instruções detalhadas que cobrem o uso de mídia e equipamentos. Até o momento, o protocolo é reproduzível por uma gama de usuários em pelo menos cinco grupos de pesquisa no Reino Unido. No entanto, não se sabe qual papel a variação biológica contribui para a variabilidade do lote livre de células. Ao lado das diferenças globais de regulação da expressão genética, a plasticidade do genoma em Streptomyces spp. é amplamente relatada e é um potencial contribuinte48. Para investigar a variação do lote, recomenda-se crescer até quatro culturas 1 L separadas derivadas de quatro colônias únicas cultivadas da noite para o dia. Anteriormente, observou-se variação de até 28% (em termos de desvio padrão) entre quatro lotes biológicos (4 L por lote fornecidos ~20 mL de extrato celular)5. Com base nesses dados, uma meta mínima razoável para um novo usuário é de 2,8 μM para sfGFP e 3,5 μM mScarlet-I/mVenus-I usando os plasmídeos disponíveis no AddGene - essas metas são 30% menores do que a média observada em dados anteriores. Se a análise hplc-ms a jusante for desejada, o PEG 6000 pode ser removido das misturas principais, embora uma diminuição no rendimento global do TX-TL possa ser esperada em até 50%.
Em termos do potencial de sistemas especializados de isenção de células de Estreptomíces5,6, há um desejo crescente de desenvolver novas ferramentas de laboratório molhado para aplicações bioprospectantes, como produtos naturais. O gênero Streptomyces está mergulhado na história da descoberta de produtos naturais, incluindo antibióticos, herbicidas e medicamentos farmacêuticos49. O crescente conhecimento adquirido a partir de projetos de sequenciamento de genomas inteiros e as mais recentes ferramentas bioinformáticas50,51,52 revelou um nível sem precedentes de produtos naturais codificados pelos BGCs dentro dos genomas microbianos53. Desbloquear essas informações genéticas - que se prevê manter novas drogas/produtos químicos e enzimas úteis à biotecnologia - exigirá o desenvolvimento de novas estratégias de biologia sintética, incluindo novos sistemas de expressão e uma série de ferramentas de engenharia metabólica54. Sistemas TX-TL especializados baseados em Estreptomia são vantajosos para estudar genes e elementos regulatórios de Actinobacterias e genomas relacionados pelas seguintes razões: [1] disponibilidade de um ambiente dobrável de proteína nativa26, [2] acesso a um pool de tRNA ideal para alta expressão genética G+C (%) e [3] um metabolismo primário ativo para o fornecimento potencial de precursores biossintéticos. Além disso, uma vantagem fundamental dos sistemas livres de células é a caracterização de alto rendimento de partes genéticas e expressão genética, usando sequenciamento de última geração13 e robótica de manuseio líquido acústico8,11,12. Em resumo, o kit de ferramentas S. venezuelae TX-TL5 fornece uma ferramenta complementar no campo da biologia sintética para produtos naturais. O kit de ferramentas S. venezuelae TX-TL apoiará o desenvolvimento do S. venezuelae como um sistema modelo e fornecerá um método para projetar novas peças/ferramentas de biologia sintética e explorar caminhos biossintéticos metabólicos secundários e enzimas.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Os autores gostariam de reconhecer o seguinte suporte à pesquisa: EPSRC [EP/K038648/1] para SJM como PDRA com PSF; Wellcome Trust patrocinou bolsa issf para SJM com PSF no Imperial College London; Bolsa de pesquisa da Royal Society [RGS\R1\191186]; Prêmio Wellcome Trust SEED [217528/Z/19/Z] para SJM na Universidade de Kent; e Global Challenges Research Fund (GCRF) Bolsa de doutorado para KC na Universidade de Kent.
Materiais
| Name | Company | Catalog Number | Comments |
| 2.5 L UltraYield Flask | Thomson | 931136-B | |
| 3-PGA (>93%) | Sigma | P8877 | |
| 384 Well Black/Clear Bottom Plate | ThermoFisher | 10692202 | |
| Ammonium chloride (98%) | Fluorochem | 44722 | |
| ATP, CTP, UTP, GTP (100 mM solution, >99%) | ThermoFisher | R0481 | |
| Carbenicillin (contact supplier for purity) | Melford | C46000-25.0 | |
| D-(+)-glucose (contact supplier for purity) | Melford | G32040 | |
| DFHBI (≥98% - HPLC) | Sigma | SML1627 | |
| DTT (contact supplier for purity) | Melford | MB1015 | |
| FlAsH-EDT2 (contact supplier for purity) | Santa Cruz Biotech | sc-363644 | |
| Glucose-6-phosphate (>98%) | Sigma | G7879 | |
| HEPES Free Acid (contact supplier for purity) | Melford | B2001 | |
| L-glutamic acid hemimagnesium salt tetrahydrate (>98%) | Sigma | 49605 | |
| Magnesium chloride (98%) | Fluorochem | 494356 | |
| Malt extract | Sigma | 70167-500G | |
| PEG-6000 | Sigma | 807491 | |
| Pierce 96-well Microdialysis Plate, 10K MWCO | ThermoFisher | 88260 | |
| Poly(vinyl sulfate) potassium salt | Sigma | 271969 | |
| Potassium glutamate (>99%) | Sigma | G1149 | |
| RTS amino acid sampler | 5 Prime | 2401530 | |
| Sodium chloride (99%) | Fluorochem | 94554 | |
| Supelclean LC-18 SPE C-18 SPE column (1 g) | Sigma | 505471 | |
| Yeast Extract | Melford | Y1333 | |
| Equipment | |||
| Platereader | BMG | Omega |
Referências
- Carbonell, P., et al. An automated Design-Build-Test-Learn pipeline for enhanced microbial production of fine chemicals. Communications Biology. 1, 66 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods Protocols. 2 (1), 24 (2019).
- Zimmerman, E. S., et al. Production of site-specific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system. Bioconjugate Chemistry. 25 (2), 351-361 (2014).
- Wiegand, D. J., Lee, H. H., Ostrov, N., Church, G. M. Cell-free protein expression using the rapidly growing bacterium Vibrio natriegens. Journal of Visualized Experiments: JoVE. (145), e59495 (2019).
- Moore, S. J., et al. A Streptomyces venezuelae cell-free toolkit for synthetic biology. ACS Synthetic Biology. 10 (2), 402-411 (2021).
- Xu, H., Liu, W. -. Q., Li, J. Translation related factors improve the productivity of a Streptomyces-based cell-free protein synthesis system. ACS Synthetic Biology. 9 (5), 1221-1224 (2020).
- Yim, S. S., et al. Multiplex transcriptional characterizations across diverse bacterial species using cell-free systems. Molecular Systems Biology. 15 (8), 8875 (2019).
- Moore, S. J., et al. Rapid acquisition and model-based analysis of cell-free transcription-translation reactions from nonmodel bacteria. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4340-4349 (2018).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production--a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Geertz, M., Shore, D., Maerkl, S. J. Massively parallel measurements of molecular interaction kinetics on a microfluidic platform. Proceedings of the National Academy of Sciences of the United States of America. 109 (41), 16540-16545 (2012).
- McManus, J. B., Emanuel, P. A., Murray, R. M., Lux, M. W. A method for cost-effective and rapid characterization of engineered T7-based transcription factors by cell-free protein synthesis reveals insights into the regulation of T7 RNA polymerase-driven expression. Archives of Biochemistry and Biophysics. 674, 108045 (2019).
- McManus, J. B., et al. A method for cost-effective and rapid characterization of genetic parts. bioRxiv. , (2021).
- Park, J., Yim, S. S., Wang, H. H. High-throughput transcriptional characterization of regulatory sequences from bacterial Biosynthetic Gene Clusters. ACS Synthetic Biology. , (2021).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, 8 (2010).
- Karim, A. S., Heggestad, J. T., Crowe, S. A., Jewett, M. C. Controlling cell-free metabolism through physiochemical perturbations. Metabolic Engineering. 45, 86-94 (2018).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Garenne, D., Thompson, S., Brisson, A., Khakimzhan, A., Noireaux, V. The all-E. coli TXTL toolbox 3.0: New capabilities of a cell-free synthetic biology platform. Synthetic Biology. , (2021).
- Goering, A. W., et al. In vitro reconstruction of nonribosomal peptide biosynthesis directly from DNA using cell-free protein synthesis. ACS Synthetic Biology. 6 (1), 39-44 (2017).
- Khatri, Y., et al. Multicomponent microscale biosynthesis of unnatural cyanobacterial indole alkaloids. ACS Synthetic Biology. 9 (6), 1349-1360 (2020).
- Zhuang, L., et al. Total in vitro biosynthesis of the nonribosomal macrolactone peptide valinomycin. Metabolic Engineering. 60, 37-44 (2020).
- Hoskisson, P. A., Seipke, R. F. Cryptic or silent? The known unknowns, unknown knowns, and unknown unknowns of secondary metabolism. mBio. 11 (5), 02642 (2020).
- Bentley, S. D., et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature. 417 (6885), 141-147 (2002).
- Li, J., Wang, H., Kwon, Y. -. C., Jewett, M. C. Establishing a high yielding Streptomyces-based cell-free protein synthesis system. Biotechnology and Bioengineering. 114 (6), 1343-1353 (2017).
- Moore, S. J., Lai, H. -. E., Needham, H., Polizzi, K. M., Freemont, P. S. Streptomyces venezuelae TX-TL - a next generation cell-free synthetic biology tool. Biotechnology Journal. 12 (4), (2017).
- Kim, W., et al. Comparative genomics determines strain-dependent secondary metabolite production in Streptomyces venezuelae strains. Biomolecules. 10 (6), 864 (2020).
- Phelan, R. M., et al. Development of next generation synthetic biology tools for use in Streptomyces venezuelae. ACS Synthetic Biology. 6 (1), 159-166 (2017).
- Song, J. Y., et al. Complete genome sequence of Streptomyces venezuelae ATCC 15439, a promising cell factory for production of secondary metabolites. Journal of Biotechnology. 219, 57-58 (2016).
- Bush, M. J., Bibb, M. J., Chandra, G., Findlay, K. C., Buttner, M. J. Genes required for aerial growth, cell division, and chromosome segregation are targets of WhiA before sporulation in Streptomyces venezuelae. mBio. 4 (5), 00684 (2013).
- Schumacher, M. A., et al. The crystal structure of the RsbN-σBldN complex from Streptomyces venezuelae defines a new structural class of anti-σ factor. Nucleic Acids Research. 46 (14), 7405-7417 (2018).
- Ramos-León, F., et al. A conserved cell division protein directly regulates FtsZ dynamics in filamentous and unicellular actinobacteria. Elife. 10, 63387 (2021).
- Bush, M. J., Tschowri, N., Schlimpert, S., Flärdh, K., Buttner, M. J. c-di-GMP signalling and the regulation of developmental transitions in Streptomycetes. Nature Reviews. Microbiology. 13 (12), 749-760 (2015).
- Ehrlich, J., Gottlieb, D., Burkholder, P. R., Anderson, L. E., Pridham, T. G. Streptomyces venezuelae, n. sp., the source of chloromycetin. Journal of Bacteriology. 56 (4), 467-477 (1948).
- Inahashi, Y., et al. Watasemycin biosynthesis in Streptomyces venezuelae: thiazoline C-methylation by a type B radical-SAM methylase homologue. Chemical Science. 8 (4), 2823-2831 (2017).
- Jakeman, D. L., et al. Antimicrobial activities of jadomycin B and structurally related analogues. Antimicrobial Agents and Chemotherapy. 53 (3), 1245-1247 (2009).
- Kodani, S., Sato, K., Hemmi, H., Ohnish-Kameyama, M. Isolation and structural determination of a new hydrophobic peptide venepeptide from Streptomyces venezuelae. Journal of Antibiotics. 67 (12), 839-842 (2014).
- Akey, D. L., et al. Structural basis for macrolactonization by the pikromycin thioesterase. Nature Chemical Biology. 2 (10), 537-542 (2006).
- Bai, C., et al. Exploiting a precise design of universal synthetic modular regulatory elements to unlock the microbial natural products in Streptomyces. Proceedings of the National Academy of Sciences of the United States of America. 112 (39), 12181-12186 (2015).
- Moore, S. J., et al. EcoFlex: A multifunctional MoClo kit for E. coli synthetic biology. ACS Synthetic Biology. 5 (10), 1059-1069 (2016).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. Journal of Visualized Experiments: JoVE. (79), e50762 (2013).
- Kim, D. M., Choi, C. Y. A semicontinuous prokaryotic coupled transcription/translation system using a dialysis membrane. Biotechnology Progress. 12 (5), 645-649 (1996).
- Liu, Y., Fritz, B. R., Anderson, M. J., Schoborg, J. A., Jewett, M. C. Characterizing and alleviating substrate limitations for improved in vitro ribosome construction. ACS Synthetic Biology. 4 (4), 454-462 (2015).
- Bindels, D. S., et al. mScarlet: a bright monomeric red fluorescent protein for cellular imaging. Nature Methods. 14, 53-56 (2017).
- Hopword, D. A., Kieser, T., Bibb, M. J., Buttner, M. J., Chater, K. Practical Streptomyces genetics. John Innes Foundation. , (2000).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnology and Bioengineering. 115 (8), 1904-1914 (2018).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Hoff, G., Bertrand, C., Piotrowski, E., Thibessard, A., Leblond, P. Genome plasticity is governed by double strand break DNA repair in Streptomyces. Scientific Reports. 8, 5272 (2018).
- Bibb, M. J. Regulation of secondary metabolism in streptomycetes. Current Opinion in Microbiology. 8 (2), 208-215 (2005).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43 (1), 237-243 (2015).
- Navarro-Muñoz, J. C., et al. A computational framework to explore large-scale biosynthetic diversity. Nature Chemical Biology. 16, 60-68 (2020).
- Alanjary, M., et al. The Antibiotic Resistant Target Seeker (ARTS), an exploration engine for antibiotic cluster prioritization and novel drug target discovery. Nucleic Acids Research. 45 (1), 42-48 (2017).
- Medema, M. H., Fischbach, M. A. Computational approaches to natural product discovery. Nature Chemical Biology. 11 (9), 639-648 (2015).
- Whitford, C. M., Cruz-Morales, P., Keasling, J. D., Weber, T. The Design-Build-Test-Learn cycle for metabolic engineering of Streptomycetes. Essays in Biochemistry. , (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados