Method Article
Un toolkit di trascrizione-traduzione di streptomyces ad alto rendimento per la biologia sintetica e le applicazioni dei prodotti naturali
In questo articolo
Riepilogo
Questo protocollo descrive in dettaglio un metodo avanzato per sintetizzare alte rese di proteine ricombinanti da un sistema di trascrizione senza cellule streptomyces venezuelae (TX-TL).
Abstract
Streptomyces spp. sono una delle principali fonti di antibiotici clinici e prodotti chimici industriali. Streptomyces venezuelae ATCC 10712 è una varietà in rapida crescita e un produttore naturale di cloramfenicolo, jadomicina e pikromicina, il che lo rende un candidato attraente come telaio di biologia sintetica di prossima generazione. Pertanto, gli strumenti genetici che accelerano lo sviluppo di S. venezuelae ATCC 10712, così come altri modelli di Streptomyces spp., sono altamente desiderabili per l'ingegneria e la scoperta di prodotti naturali. A tal fine, in questo protocollo viene fornito un sistema S . venezuelae ATCC 10712 privo di cellule dedicato per consentire l'espressione eterologa ad alto rendimento di geni ad alto G + C (%). Questo protocollo è adatto per reazioni batch su piccola scala (10-100 μL) in formato piastra a 96 pozzetti o 384 pozzetti, mentre le reazioni sono potenzialmente scalabili. Il sistema privo di cellule è robusto e può raggiungere rese elevate (~ 5-10 μ M) per una gamma di proteine ricombinanti in una configurazione minima. Questo lavoro incorpora anche un ampio set di strumenti plasmidici per la misurazione in tempo reale dell'mRNA e della sintesi proteica, nonché la colorazione a fluorescenza in gel delle proteine marcate. Questo protocollo può anche essere integrato con flussi di lavoro di caratterizzazione dell'espressione genica ad alto rendimento o con lo studio di percorsi enzimatici da geni ad alto G + C (%) presenti nei genomi di Actinomiceti.
Introduzione
I sistemi di trascrizione-traduzione senza cellule (TX-TL) forniscono una piattaforma di prototipazione ideale per la biologia sintetica per implementare cicli rapidi di progettazione-costruzione-test-apprendimento, il framework di ingegneria concettuale per la biologia sintetica1. Inoltre, vi è un crescente interesse per i sistemi TX-TL per la produzione di proteine ricombinanti di alto valore in un ambiente a reazione aperta2, ad esempio, per incorporare amminoacidi non standard nei coniugati anticorpo-farmaco3. In particolare, TX-TL richiede un estratto cellulare, plasmide o DNA lineare e una soluzione energetica per catalizzare la sintesi proteica in reazioni batch o semicontinue. Mentre Escherichia coli TX-TL è il sistema dominante senza cellule, un certo numero di sistemi TX-TL emergenti non modello hanno attirato l'attenzione per diverse applicazioni4,5,6,7,8. I principali vantaggi di TX-TL includono scalabilità flessibile (scala da nanolitri a litro)9,10, forte riproducibilità e flussi di lavoro automatizzati8,11,12. In particolare, l'automazione di TX-TL consente la caratterizzazione accelerata di parti genetiche ed elementi regolatori8,12,13.
In termini di configurazione della reazione, TX-TL richiede sia fonti di energia primaria che secondaria, nonché amminoacidi, cofattori, additivi e una sequenza di DNA modello. I nucleotidi trifosfati (NTP) forniscono la fonte di energia primaria per guidare l'mRNA iniziale (ATP, GTP, CTP e UTP) e la sintesi proteica (solo ATP e GTP). Per aumentare i rendimenti di TX-TL, gli NTP vengono rigenerati attraverso il catabolismo di una fonte di energia secondaria, come il maltosio14, la maltodestrina15, il glucosio14, il 3-fosfoglicerato (3-PGA)16, il fosfoenolpiruvato17 e il L-glutammato18. Questa attività metabolica intrinseca è sorprendentemente versatile, ma scarsamente studiata, specialmente nei sistemi TX-TL emergenti. Ogni fonte di energia ha proprietà e vantaggi distinti in termini di resa di ATP, stabilità chimica e costo, che è una considerazione importante per le reazioni TX-TL su larga scala. Finora, gli attuali protocolli per E. coli TX-TL hanno raggiunto fino a 4,0 mg / mL (~ 157 μM) per la proteina fluorescente verde modello (GFP), utilizzando una miscela di 3-PGA (30 mM), maltodestrina (60 mM) e D-ribosio (30 mM) come fonte di energia secondaria19.
Recentemente, c'è stato un crescente interesse nello studio delle vie biosintetiche dei metaboliti secondari nei sistemi TX-TL20,21,22. In particolare, gli Actinobatteri sono una delle principali fonti di metaboliti secondari, compresi antibiotici e prodotti chimici agricoli23,24. I loro genomi sono arricchiti con i cosiddetti cluster di geni biosintetici (BGC), che codificano percorsi enzimatici per la biosintesi del metabolita secondario. Per lo studio delle parti genetiche di Actinobacteria e delle vie biosintetiche, è stata recentemente sviluppata una serie di sistemi TX-TL basati su Streptomyces5,6,25,26. Questi sistemi specializzati Streptomyces TX-TL sono potenzialmente utili per i seguenti motivi: [1] fornitura di un ambiente di ripiegamento proteico nativo per gli enzimi di Streptomyces spp.26; [2] accesso a un pool di tRNA ottimale per un'elevata espressione genica G+C (%); [3] metabolismo primario attivo, che potenzialmente può essere dirottato per la fornitura di precursori biosintetici; e [4] fornitura di enzimi, precursori o cofattori dal metabolismo secondario presenti nell'estratto cellulare nativo. Pertanto, un toolkit S.venezuelae TX-TL ad alto rendimento è stato recentemente istituito per sfruttare queste capacità uniche5.
Streptomyces venezuelae è un ospite emergente per la biologia sintetica con una ricca storia nella biotecnologia industriale5,27,28,29 e come sistema modello per lo studio della divisione cellulare e della regolazione genetica in Actinobacteria30,31,32. Il ceppo di tipo principale, S. venezuelae ATCC 10712, ha un genoma relativamente grande di 8,22 Mb con il 72,5% di contenuto G + C (%) (numero di adesione: CP029197), che codifica 7377 sequenze codificanti, 21 rRNA, 67 tRNA e 30 cluster di geni biosintetici27. In biologia sintetica, S. venezuelae ATCC 10712 è un telaio attraente per l'espressione eterologa di vie biosintetiche. A differenza della maggior parte delle altre macchie di Streptomyces, offre diversi vantaggi chiave, tra cui un rapido tempo di raddoppio (~ 40 min), una vasta gamma di strumenti genetici e sperimentali5,28, mancanza di aggregazione miceliale e sporulazione in mezzi liquidi28,33. Diversi studi hanno anche dimostrato l'uso di S. venezuelae per la produzione eterologa di una vasta gamma di metaboliti secondari, tra cui polichetidi, peptidi ribosomiali e non ribostomali34,35,36,37,38. Queste caratteristiche combinate rendono questo ceppo un ospite microbico attraente per la biologia sintetica e le applicazioni di ingegneria metabolica. Mentre S. venezuelae non è il modello dominante di Streptomyces per l'espressione genica eterologa, con ulteriori sviluppi, è pronto per un uso più ampio all'interno della scoperta di prodotti naturali.
Questo manoscritto presenta un protocollo dettagliato (Figura 1) per un sistema S. venezuelae TX-TL ad alto rendimento, che è stato aggiornato dal protocollo originale precedentemente pubblicato26. In questo lavoro, la soluzione energetica e le condizioni di reazione sono state ottimizzate per aumentare la resa proteica fino a 260 μg/mL per la proteina reporter mScarlet-I in una reazione batch di 4 ore, 10 μL, utilizzando un plasmide standard, pTU1-A-SP44-mScarlet-I. Questo plasmide è stato specificamente progettato per consentire vari metodi di rilevamento dell'espressione proteica. Anche il protocollo è semplificato, mentre il sistema energetico è stato ottimizzato per ridurre la complessità e il costo di impostare reazioni senza cellule senza compromettere la resa. Insieme al sistema TX-TL ottimizzato, è stata sviluppata una libreria di parti genetiche per la messa a punto dell'espressione genica e come strumenti fluorescenti per il monitoraggio di TX-TL in tempo reale, creando così una piattaforma versatile per la prototipazione dell'espressione genica e delle vie biosintetiche del prodotto naturale da Streptomyces spp. e relativi Actinobacteria.
In questo lavoro, il plasmide standard raccomandato (pTU1-A-SP44-mScarlet-I) può essere utilizzato per stabilire il flusso di lavoro S. venezuelae TX-TL in un nuovo laboratorio ed è disponibile su AddGene (vedere Tabella supplementare S1). pTU1-A-SP44-mScarlet-I offre all'utente la flessibilità di studiare altri frame a lettura aperta (ORF). L'ORF mScarlet-I è ottimizzato per il codone per l'espressione genica di S. venezuelae. Il promotore SP44 è un forte promotore costitutivo che è molto attivo sia in E. coli che in Streptomyces spp.39. Il plasmide ha due siti enzimatici di restrizione unici (NdeI, BamHI) per consentire la sub-clonazione di nuovi ORF in-frame con un sistema congiunto C-terminale FLAG-tag e fluorescein arsenical hairpin (FlAsH) binder tag. In alternativa, entrambi i tag possono essere rimossi con l'inclusione di un codone di arresto dopo la sub-clonazione di un nuovo gene. Con questo vettore di base, è stata dimostrata l'espressione ad alto rendimento di una gamma di proteine, vale a dire proteine della via di biosintesi dell'ossitetraciclina e una peptide sintetasi non ribosomiale non caratterizzata (NRPS) da Streptomyces rimosus (Figura 2). In termini di rilevamento dell'mRNA, il plasmide standard pTU1-A-SP44-mScarlet-I contiene un aptamero dBroccoli (nella regione 3'-non tradotto) per il rilevamento con la sonda 3,5-difluoro-4-idrossibenzilidene imidazolinone (DFHBI). Per una maggiore flessibilità, è stato reso disponibile anche un set di strumenti di parti MoClo compatibili con EcoFlex40 su AddGene, tra cui un vettore navetta Streptomyces compatibile con EcoFlex (pSF1C-A-RFP/pSF2C-A-RFP) e una gamma di plasmidi varianti pTU1-A-SP44 che esprimono la proteina di fluorescenza verde superfolder (sfGFP), mScarlet-I, mVenus-I e β-glucuronidasi (GUS). In particolare, il plasmide pSF1C-A è derivato da pAV-gapdh28 ed è polimerizzato di siti BsaI/BsmBI per l'assemblaggio MoClo. pSF1C-A-RFP/pSF2C-A-RFP è equivalente a pTU1-A-RFP/pTU2-A-RFP di EcoFlex40 ma contiene funzionalità aggiuntive per la coniugazione e l'integrazione cromosomica in Streptomyces spp. utilizzando il sistema di integrasi phiC3128.
La prima fase del protocollo prevede la crescita del S. venezuelae ATCC 10712 o di un ceppo strettamente correlato, la raccolta delle cellule in fase media-esponenziale, le fasi di lavaggio cellulare e l'equilibrio nei tamponi S30A e S30B. Questa fase richiede tre giorni e il tempo per la crescita cellulare può essere utilizzato per preparare i componenti rimanenti come descritto di seguito. Le cellule raccolte vengono quindi lisate mediante sonicazione, chiarificate e sottoposte a una reazione di deflusso. In questa fase finale della preparazione, gli estratti cellulari possono essere preparati per la conservazione a lungo termine a -80 °C per ridurre al minimo la perdita di attività. Per l'assemblaggio di reazioni TX-TL utilizzando questo protocollo, viene presentato uno Streptomyces Master Mix (SMM), con l'opzione di un formato MES (Minimal Energy Solution) che offre rese comparabili. Inoltre, si raccomanda di strisciare una coltura fresca di S. venezuelae ATCC 10712 da un brodo di glicerolo a -80 ° C su una piastra di agar GYM e incubare a 28 ° C per almeno 48-72 ore fino a quando non sono visibili singole colonie. Solo le colture fresche dovrebbero essere utilizzate per i seguenti passaggi.
Protocollo
NOTA: vedere la Tabella 1 e la Tabella 2 per le ricette per GYM medium e piastra di agar e tamponi di lavaggio S30A e S30B.
1. Preparazione di soluzioni e linee guida generali
- Mantenere tutte le soluzioni, le cellule (post-crescita) e gli estratti cellulari sul ghiaccio dopo la preparazione, a meno che non venga indicata un'eccezione.
- Conservare le scorte per 1 M Mg-glutammato, 4 M K-glutammato, 40% (p/v) PEG 6000, 1 g/mL di acido polivinilsolfonico a temperatura ambiente e tutti gli altri stock a -80 °C. Ridurre al minimo il numero di cicli di congelamento-scongelamento per evitare la degradazione chimica.
- Per la preparazione di scorte di soluzioni energetiche (vedere Tabella 3) come 3-PGA (richiede un aggiustamento del pH), seguire le indicazioni fornite nel protocollo E. coli TX-TL41.
NOTA: Tutti i componenti sono completamente solubili in ddH2O e conservati come aliquote nel congelatore a -80 °C. - Scongelare singoli stock o soluzioni energetiche (descritte più avanti) sul ghiaccio. Riscaldare lo stock di amminoacidi a 42 °C con vortice per ~ 15-30 minuti per solubilizzare tutti gli amminoacidi.
- Poiché alcuni amminoacidi (L-Cys, L-Tyr, L-Leu) precipitano sul ghiaccio, riducendo al minimo il tempo di riposo, lasciare questa soluzione a temperatura ambiente e utilizzare un vortice per dissolversi.
- Aggiungere i volumi calcolati (Tabella 3) di soluzioni stock e acqua e mescolare bene usando un vortice.
- Aliquotare la soluzione energetica come aliquote da 20-100 μL per tubo, o a piacere, su ghiaccio e conservare a -80 °C fino a nuovo utilizzo.
2. Preparazione delle celle S. venezuelae ATCC 10712
- Giorno 1-Preparazione media/tampone e pre-coltura notturna
- Preparare 1 L di mezzo liquido gym sterile in un matraccio sconcertato da 2 L, come descritto nella Tabella 1. Vedere la tabella dei materiali per le fonti di apparecchiature/sostanze chimiche/reagenti.
- Preparare 1 x 50 mL di mezzo liquido gym sterile in un matraccio Erlenmeyer da 250 mL, come descritto nella Tabella 1.
- Preparare 100 mL di 1 M HEPES-KOH pH 7,5, 100 mL di 1 M MgCl2 e 500 mL di 4 M NH4Cl soluzioni per produrre 1 L di S30A e 1 L di tamponi di lavaggio S30B. Vedi Tabella 2 per le ricette.
- Prepara la pre-cultura durante la notte. Preriscaldare i 50 mL sterili di mezzo liquido GYM in un matraccio Erlenmeyer da 250 mL a 28 °C per 30 min.
- Inoculare una singola colonia di S. venezuelae ATCC 10712 (o ceppo correlato) da una piastra di agar GYM in 50 mL preriscaldati di mezzo liquido GYM e incubare a 28 °C, 200 rpm per 16 ore (pre-coltura notturna).
- Giorno 2 - Preparare la pre-cultura diurna e la cultura della crescita principale.
- Preriscaldare 50 mL di liquido sterile GYM in un matraccio Erlenmeyer da 250 mL a 28 °C per 30 min.
- Trasferire 1 mL di pre-coltura notturna in 50 mL di terreno liquido GYM preriscaldato e incubare a 28 °C, 200 giri/min per 8 ore (pre-coltura diurna).
- Dopo questo periodo di crescita, controllare l'OD600 in uno spettrofotometro utilizzando una diluizione 1:10 con mezzo GYM sterile in una cuvetta di plastica da 1 mL (1 cm di lunghezza del percorso).
NOTA: l'OD600 dovrebbe aver raggiunto almeno 3-4. Se c'è una scarsa crescita, è consigliabile ripetere i passaggi 2.2.1-2.2.2. - Sottocoltura 0,25 mL di precoltura diurna in 1 L di liquido GYM medium in flaconi sconcertati da 2 L.
- Agitare la notte a 28 °C, 200 giri/min per 14 ore.
- Giorno 3-Cellule di raccolta
- Dopo il precedente periodo di incubazione (14 h), registrare l'OD600 della coltura principale. Diluire la coltura notturna 1:10 con un mezzo GYM fresco per la misurazione di OD600 .
NOTA: l'OD600 dovrebbe aver raggiunto 3.0-4.0 in questa fase. - Se OD600<3.0, aumentare la velocità di scuotimento a 250-300 rpm e crescere fino a raggiungere un OD600 di 3.0. Crescere per non più di 2 ore aggiuntive (16 ore in totale).
- Se OD600>3.0, trasferire le colture in contenitori di centrifugazione e raffreddare rapidamente su ghiaccio bagnato per 30 min.
- In attesa che la coltura cellulare si raffreddi sul ghiaccio, preparare 4 ml di tamponi freschi da 1 M ditiotreitolo (DTT), S30A e S30B, come descritto nella Tabella 1, e tenerli sul ghiaccio. Vedere la tabella dei materiali per la fonte chimica/reagente.
- Pre-pesare un tubo di centrifuga vuoto da 50 ml e pre-raffreddare a -20 °C.
- Aggiungere 2 mL di 1 M DTT a 1 L di tampone S30A sul ghiaccio e mescolare bene.
NOTA: aggiungere DTT ai tamponi di lavaggio S30A e S30B solo prima di utilizzarli. - Centrifugare le celle a 6.000 × g, 4 °C, 10 min, e scartare con cura il surnatante con un movimento rapido e unico.
NOTA: Se il pellet è disturbato, massimizzare la ritenzione cellulare con il mezzo GYM residuo e continuare il protocollo. - Aggiungere 500 ml di tampone S30A e risospese le cellule agitando vigorosamente i flaconi di centrifugazione fino a quando i grumi cellulari sono dispersi in modo omogeneo.
- Centrifugare le cellule a 6.000 × g, 4 °C, 6 min, e scartare con cura il surnatante.
NOTA: anche se il pellet cellulare sarà più solido a questo punto, alcune celle rimarranno in sospensione (vedere Figura 1). Trattare come descritto al punto 2.3.7 e trattenere il maggior numero possibile di cellule. - Ripetere i passaggi 2.3.8-2.3.9.
- Aggiungere 2 mL di 1 M DTT a 1 L di tampone S30B sul ghiaccio e mescolare bene. Aggiungere 500 mL di buffer S30B alle celle. Ripetere il passaggio 2.3.9.
- Risospesare il pellet cellulare in 10 mL di tampone S30B e trasferirlo nel tubo centrifugo da 50 mL pre-pesato e pre-refrigerato. Se necessario, trasferire le celle residue con ulteriori 5-10 mL di buffer S30B. Riempire fino a 50 ml con S30B.
- Centrifugare le celle a 6.000 × g, 4 °C, 10 min, e scartare con cura il surnatante.
- Ripetere il passaggio 2.3.13.
- Aspirare accuratamente il surnatante S30B rimanente con una pipetta da 100-200 μL.
- Pesare il pellet a celle umide.
NOTA: il peso tipico del pellet a celle umide per 1 L di coltura GYM durante la notte (OD600 = 3,0) è di ~ 4,5 g. - Per ogni 1 g di cellule umide, aggiungere 0,9 mL di tampone S30B. Risospesce le celle usando una pipetta Pasteur o un vortice.
- Centrifugare brevemente (~10 s) fino a 500 × g per sedimentare le cellule.
NOTA: il protocollo può essere sospeso a questo punto e le cellule possono essere congelate su azoto liquido o ghiaccio secco e conservate a -80 ° C. Per sicurezza, indossare adeguati dispositivi di protezione individuale (DPI) quando si maneggia azoto liquido, comprese protezioni facciali e guanti.
- Dopo il precedente periodo di incubazione (14 h), registrare l'OD600 della coltura principale. Diluire la coltura notturna 1:10 con un mezzo GYM fresco per la misurazione di OD600 .
3. Lisi cellulare mediante sonicazione per ottenere l'estratto di cellule grezze
NOTA: in questa fase, l'utente può scegliere di interrompere le celle mediante sonicazione in frazioni da 1 mL (opzione 1) o come sospensione cellulare più grande (5 mL) in un tubo da 50 mL (opzione 2). Entrambe le opzioni sono state dettagliate di seguito per garantire la riproducibilità, poiché il volume finale della sospensione cellulare può cambiare a causa della perdita di cellule durante le precedenti fasi di raccolta e lavaggio. Un nuovo utente deve prima tentare l'opzione 2.1 per stabilire il protocollo.
- Lisi cellulare mediante sonicazione in frazioni da 1 mL
- Utilizzando una punta della pipetta da 1 mL (tagliare l'estremità della punta per aumentare la dimensione del foro), trasferire 1 mL della sospensione cellulare in tubi microcentrifuga da 2 mL.
NOTA: Se le cellule sono congelate, scongelare rapidamente il tubo da 50 ml contenente il pellet in acqua tiepida prima della lisi cellulare. Trasferire il tubo sul ghiaccio bagnato non appena il pellet ha iniziato a scongelarsi e raffreddare per 10 minuti. - Posizionare ogni tubo di microcentrifuga in un becher di acqua ghiacciata, utilizzando un rack di tubi di plastica per contenere il tubo per la sonicazione.
NOTA: A causa della sensibilità dell'estratto cellulare al surriscaldamento, è fondamentale assicurarsi che i tubi non si riscaldino per prevenire la precipitazione proteica e la ridotta attività enzimatica. - Utilizzare una sonda sonicator con una punta di 3 mm di diametro e pulirla con etanolo al 70% (v/v) e acqua a doppia distillazione (ddH2O). Abbassare la punta del sonicatore nella sospensione cellulare fino a quando non si trova a ~ 1 cm sotto la superficie del liquido.
- Immettere le seguenti impostazioni nel sonicatore: frequenza 20 kHz, ampiezza del 65%, tempo di accensione dell'impulso di 10 s, tempo di spegnimento degli impulsi di 10 s, tempo di sonicazione totale di 1 minuto.
- Eseguire il protocollo di sonicazione. Spostare il tubo su/ giù e lateralmente durante i primi due cicli di riposo per garantire che le celle siano uniformemente sonicate. Registrare l'input di energia.
NOTA: per sicurezza, indossare un'adeguata protezione dell'udito durante la sonicazione. La viscosità diminuirà man mano che le cellule vengono interrotte e il pellet di cellule umide di crema pallida dovrebbe trasformarsi in un fluido marrone omogeneo. L'apporto energetico raccomandato è di 240 J per mL di celle umide. Se le cellule sono solo parzialmente lisate, la sospensione apparirà ancora color crema con ciuffi viscosi di cellule, in particolare sui lati del tubo. - Invertire il tubo 2-3 volte e ripetere la sonicazione per un ulteriore uno o due cicli di 10 s, mescolando frequentemente fino a quando le cellule sono completamente interrotte.
- Utilizzando una punta della pipetta da 1 mL (tagliare l'estremità della punta per aumentare la dimensione del foro), trasferire 1 mL della sospensione cellulare in tubi microcentrifuga da 2 mL.
- Lisi cellulare mediante sonicazione di una sospensione cellulare da 5 ml
- Se le cellule sono congelate, scongelare rapidamente il tubo da 50 ml contenente il pellet in acqua tiepida con agitazione prima della lisi cellulare. Trasferire il tubo sul ghiaccio bagnato non appena il pellet ha iniziato a scongelarsi e raffreddare per 10 minuti.
- Ruotare brevemente il tubo a 500 x g per sedimentare le cellule.
- Posizionare il tubo da 50 ml in un becher di acqua ghiacciata per la sonicazione.
NOTA: A causa della sensibilità dell'estratto cellulare al surriscaldamento, è fondamentale assicurarsi che i tubi non si riscaldino per prevenire la precipitazione proteica e la ridotta attività enzimatica. - Utilizzare una sonda sonicator con una punta di 6 mm di diametro e pulirla con etanolo al 70% (v/v) e ddH2O (vedere lo schema visivo della sonda da 6 mm nella Figura 1). Abbassare la punta del sonicatore nella sospensione cellulare (~ 5 mL) fino a quando non si trova a ~ 1 cm sotto la superficie del liquido.
- Immettere le seguenti impostazioni nel sonicatore: frequenza 20 kHz, ampiezza del 65%, tempo di accensione dell'impulso di 10 s, tempo di spegnimento degli impulsi di 10 s, tempo di sonicazione totale di 1 minuto per mL di celle umide (5 minuti in totale).
- Eseguire il protocollo di sonicazione. Spostare il tubo su/ giù e lateralmente durante i primi due cicli di riposo per garantire che le celle siano uniformemente sonicate.
NOTA: per sicurezza, indossare un'adeguata protezione dell'udito durante la sonicazione. La viscosità diminuirà man mano che le cellule vengono interrotte e il pellet di cellule umide di crema pallida dovrebbe trasformarsi in un fluido marrone omogeneo. Registrare l'input di energia. Si consiglia un apporto energetico ottimale di 240 J per mL di celle umide (~ 1200 J in totale da 5 minuti di sonicazione). - Se alcune celle rimangono intatte, seguire le indicazioni del passaggio 3.1.5.
- Trasferire gli estratti cellulari in 2 mL di tubi microcentrifuga.
4. Chiarificazione dell'estratto cellulare e reazione di deflusso
- Centrifugare le celle lisate a 16.000 × g per 10 minuti a 4 °C per rimuovere i detriti cellulari. Trasferire il surnatante in tubi microcentrifuga da 1,5 mL come aliquote da 1 mL.
- Eseguire la reazione di deflusso per gli estratti cellulari. Incubare i tubi da 1,5 ml contenenti gli estratti cellulari a 30 °C per 60 minuti su un blocco di calore o un'incubatrice senza agitare.
- Centrifugare gli estratti cellulari a 16.000 × g per 10 min a 4 °C. Raggruppare i supernatanti in un tubo di centrifuga da 15 ml. Mescolare il surnatante invertendo il tubo cinque volte fino a renderlo omogeneo, quindi tenerlo sul ghiaccio. Invertire delicatamente per evitare la formazione di bolle d'aria.
- Diluire 10 μL dell'estratto cellulare 100 volte con tampone S30B e misurare la concentrazione proteica totale utilizzando un test Bradford con tre ripetizioni tecniche (vedere Materiale supplementare S2 per la guida al test Bradford).
- Se la concentrazione proteica è di 20-25 mg/mL, trasferire gli estratti cellulari come aliquote da 100 μL in nuovi tubi da 1,5 mL, congelare in azoto liquido e conservare a -80 °C.
NOTA: per motivi di sicurezza, indossare DPI appropriati quando si maneggia azoto liquido, comprese protezioni facciali e guanti. - Se la concentrazione proteica è <20 mg/mL, ripetere le fasi di preparazione dell'estratto grezzo per garantire che l'estratto cellulare di alta qualità e le rese di TX-TL siano paragonabili al lavoro pubblicato in precedenza5.
5. Preparazione del modello di DNA plasmidico
- Purificare il plasmide pTU1-A-SP44-mScarlet-I (origine pUC19) da un ceppo plasmidico di E. coli appena trasformato (DH10β, JM109) coltivato in 50 mL di coltura LB (con 100 mg/ mL di carbenicillina) utilizzando un kit di purificazione del DNA plasmidico appropriato secondo le istruzioni del produttore.
- Eluire il plasmide in 2 x 300 μL di acqua priva di nucleasi e combinare le frazioni.
- Aggiungere 0,1 volumi (66 μL) di acetato di sodio 3 M (pH 5,2).
- Aggiungere 0,7 volumi (462 μL) di isopropanolo.
- Incubare il DNA a -20 °C per 30 min.
- Centrifugare a 16.000 × g per 30 minuti a 4 °C ed eliminare il surnatante.
- Aggiungere 2 ml di etanolo al 70% (v/v) al pellet di DNA.
- Invertire il tubo 3-4 volte per risospese il pellet di DNA plasmidico.
- Centrifugare a 16.000 × g per 5 minuti a 4 °C ed eliminare il surnatante.
- Ripetere i passaggi 5.7-5.9 e rimuovere tutto il liquido visibile.
- Asciugare all'aria il pellet di DNA per 10-30 minuti o asciugare per 5 minuti con una centrifuga sottovuoto.
- Sospendere il pellet essiccato con 600 μL di ddH2O senza nucleasi.
- Misurare la concentrazione e la purezza del DNA utilizzando uno spettrofotometro.
- Preparare aliquote da 50-100 μL e conservare a -20 °C.
NOTA: Si raccomanda un'alta concentrazione di DNA nell'intervallo 500-1000 ng/μL a causa dei vincoli di volume ristretti delle reazioni prive di cellule. Diluire lo stock di DNA plasmidico a 80 nM; 168 ng/μL pTU1-A-SP44-mIl plasmide di Scarlet-I equivale a 80 nM.
6. Preparazione della soluzione Streptomyces Master Mix (SMM)
- Soluzione di aminoacidi
- Utilizzare il kit di campionatori di aminoacidi per evitare errori manuali e ridurre i tempi di preparazione, seguendo le istruzioni del produttore fornite online.
- Diluire la soluzione madre di amminoacidi 20x utilizzando ddH2O fino a una concentrazione finale di 6 mM (5 mM L-Leu).
- Diluire ulteriormente a 2,4 mM (2 mM L-Leu) all'interno della soluzione SMM 2,4x (vedere Tabella 3).
NOTA: La concentrazione finale nella reazione TX-TL è di 1 mM 19x amminoacidi e 0,83 mM L-Leu.
- Soluzioni energetiche e additivi
- Preparare gli altri componenti nella soluzione SMM 2,4x seguendo la ricetta descritta nella Tabella 3.
- In alternativa, preparare una soluzione meS (Minimal Energy Solution) 2,4x, seguendo la ricetta descritta nella Tabella 3.
7. Impostazione di una reazione standard S. venezuelae TX-TL
- Scongelare l'estratto cellulare, la soluzione SMM (o MES) e il DNA plasmidico sul ghiaccio. Pre-raffreddare una piastra da 384 pozzetti a -20 °C.
- Impostare reazioni TX-TL in cui il 25% del volume è DNA plasmidico, il 33,33% è estratto cellulare e il 41,67% è soluzione SMM ; tenerli sul ghiaccio per evitare distorsioni dell'ora di inizio.
NOTA: è stato fornito un modello TX-TL standard (Tabella 4) per calcolare il volume di reagenti necessari in base al numero di reazioni. Il volume standard per una reazione di 33 μL è il seguente: 11 μL di estratto cellulare, 13,75 μL di SMM e 8,25 μL di DNA plasmidico. - Ruotare delicatamente la miscela per ~ 5 s a bassa velocità per garantire che la soluzione sia omogenea. Evitare la formazione di schiuma/bolle.
- Trasferire aliquote da 10 μL in tre pozzetti di una piastra a 384 pozzetti come tripliceto tecnico senza introdurre bolle d'aria. Sigillare la piastra con un coperchio trasparente e ruotare a 400 × g per 5 s.
- Incubare la reazione a 28 °C in un incubatore (per letture end-point) o in un lettore di piastre senza agitare.
NOTA: le reazioni richiedono in genere 3-4 ore per raggiungere il completamento. Vedere Materiale supplementare S2 per indicazioni su un lettore di piastre e misurazioni standard mScarlet-I.
Risultati
Questo protocollo dettagliato viene fornito come esempio per aiutare l'utente a stabilire un sistema Streptomyces TX-TL basato sul ceppo modello S. venezuelae ATCC 10712 (Figura 1). L'utente può cercare di studiare altri ceppi di Streptomyces; tuttavia, le fasi di crescita/raccolta di altri ceppi con tempi di raddoppio più lunghi o preferenze di crescita distinte dovranno essere ottimizzate su misura per ottenere risultati ottimali. Per il risultato rappresentativo, la proteina fluorescente mScarlet-I del plasmide standard pTU1-A-SP44-mScarlet-I (Figura 2 e Figura 3) è stata ottimizzata per fornire espressione ad alto rendimento in S. venezuelae TX-TL con una gamma di metodi di rilevamento (SDS-PAGE, fluorescenza). Inoltre, questo plasmide standard è stato modificato per dimostrare la sintesi di una serie di enzimi metaboliti secondari da S. rimosus (Figura 2)5. Infine, un potenziale flusso di lavoro per la biosintesi di prodotti naturali su larga scala viene mostrato come uno schema per l'utilizzo di un percorso modello dalle prime fasi della biosintesi dell'eme. Il flusso di lavoro è potenzialmente adattabile ad altre vie biosintetiche del metabolita secondario. Come linea guida, questo protocollo dovrebbe fornire una resa minima di 2,8 μM per sfGFP e 3,5 μM per mScarlet-I/mVenus dai plasmidi di espressione forniti su AddGene. Queste cifre consentono una variazione tipica dei lotti (fino al 28%) osservata nei dati precedenti5, sebbene rendimenti superiori a 10 μM mScarlet-I siano stati raggiunti con lotti ottimali (dati non pubblicati).
Misurazione di S. venezuelae TX-TL del gene mScarlet-I utilizzando cinque metodi distinti
Viene mostrata l'espressione del plasmide standard pTU1-A-SP44-mScarlet-I, con la misurazione dell'espressione di mScarlet-I utilizzando cinque diversi metodi: 1. misurazione della fluorescenza in tempo reale dell'mRNA utilizzando l'aptamero dBroccoli, 2. misurazione della fluorescenza in tempo reale della proteina mScarlet-I immatura utilizzando il sistema di tag FlAsH, 3. misurazione della fluorescenza in tempo reale della proteina mScarlet-I matura, 4. colorazione fluorescente in gel di mScarlet-I utilizzando il tag FlAsH, e 5. Colorazione blu Coomassie di proteine totali prive di cellule. Per questi dati, le reazioni sono state impostate in tubi microcentrifuga da 2 ml come reazioni da 33 μL (per campioni di punto finale) o come tripliceto tecnico da 10 μL in piastre da 384 pozzetti in un lettore di piastre. Una proteina mScarlet-I triplo marcata (N-terminale His6, C-terminal Flag e C-terminale FlAsH) è stata purificata separatamente per creare uno standard di calibrazione per le misurazioni, utilizzando il plasmide pET15b-mScarlet-I, che è descritto ulteriormente nel materiale supplementare S2. I dati per questi esperimenti sono mostrati nella Figura 3. Ulteriori dettagli sul metodo di colorazione a fluorescenza in gel sono disponibili nel materiale supplementare S3.
S. venezuelae TX-TL della biosintesi dell'eme in fase iniziale
Per fungere da modello di via biosintetica del prodotto naturale, la biosintesi "one-pot" dell'uroporfirinogeno III (uro'gen III) è stata eseguita utilizzando il plasmide di espressione pTU1-A-SP44-hemC-hemD/cysGA-hemB5. Questo modello di via biosintetica è stato scelto in quanto l'uro'gen III è altamente sensibile all'ossigeno e si ossida rapidamente (perdita di sei elettroni) in uroporfirina III, che mostra una forte fluorescenza rossa. Ciò consente un facile rilevamento della reazione in tempo reale utilizzando misurazioni di fluorescenza e/o HPLC-MS (Figura 4), come descritto in precedenza5. Inoltre, queste reazioni sono state studiate utilizzando un metodo batch o semicontinuo. Una reazione semicontinua è una strategia, che utilizza un dispositivo di microdialisi42,43 che fornisce energia aggiuntiva (NTP, fonte di energia secondaria) e amminoacidi per prolungare il tempo di reazione e aumentare le rese di sintesi proteica. Qui, il metodo semicontinuo viene utilizzato per aumentare la reazione del modello eme e separare le proteine TX-TL dal prodotto di reazione per facilitare la purificazione e l'analisi da parte di HPLC-MS. Ulteriori dettagli sui metodi sono disponibili nel materiale supplementare S4 o per i dati, vedere il lavoro precedente5. Reazioni semicontinue prive di cellule sono descritte anche in lavori precedenti42,43. Il flusso di lavoro schematico di esempio qui dimostrato (Figura 4) è potenzialmente adattabile ad altri percorsi biosintetici naturali del prodotto.
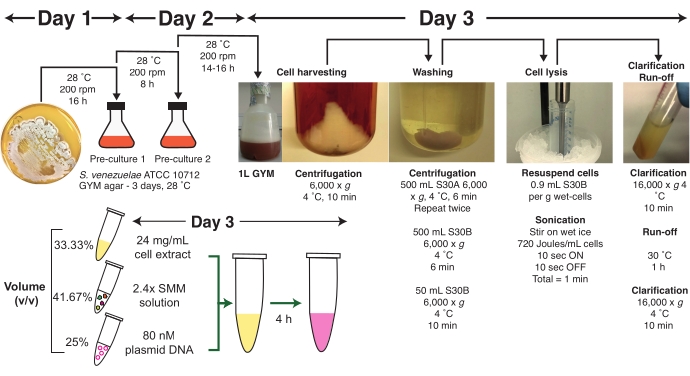
Figura 1: Panoramica del protocollo Streptomyces venezuelae TX-TL. Viene illustrato un riepilogo del protocollo, incluso un intervallo di tempo consigliato di tre giorni. Il protocollo è suddiviso in fasi distinte di crescita cellulare, raccolta cellulare, lavaggio cellulare, lisi cellulare mediante sonicazione, chiarificazione, reazione di deflusso, preparazione del master mix (SMM), preparazione del DNA plasmidico e assemblaggio della reazione TX-TL. Il protocollo completo è descritto in dettaglio all'interno del testo, insieme a linee guida e suggerimenti pratici. Abbreviazioni: SMM = Streptomyces Master Mix; TX-TL = trascrizione-traduzione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Sintesi proteica ad alto rendimento da geni G+C (%) elevati. (A) Sintesi di proteine fluorescenti sfGFP, mVenus-I e mScarlet-I. (B) Sintesi di enzimi biosintetici da Streptomyces rimosus. Abbreviazione: EV = Empty Vector; NRPS = peptide sintetasi non ribosomiale. La figura è modificata da 5. Si prega di consultare il protocollo e i file supplementari per la configurazione e la metodologia della reazione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Misurazione di TX-TL a cinque vie con il plasmide pTU1-A-SP44-mScarlet-I. (A) Progettazione del plasmide comprendente le seguenti caratteristiche: SP44 è un forte promotore costitutivo attivo in Streptomyces spp. ed E. coli; pET-RBS è derivato dai plasmidi di espressione pET ed è altamente attivo sia in Streptomyces spp. che in E. coli5,40; Gene mScarlet-I ottimizzato per Streptomyces codon, che codifica per un derivato proteico rosso-fluorescente44; C-terminal FLAG-tag per la purificazione della cromatografia di affinità o il rilevamento di western blotting; C-terminale FlAsH tag per l'etichettatura fluorescente per la colorazione in-gel o la misurazione in tempo reale della sintesi proteica nascente; aptamero dBroccoli per la misura in tempo reale dell'mRNA utilizzando la sonda DFHBI; Bba_B0015 terminatore di trascrizione, che sono altamente efficienti in S. venezuelae ATCC 107125; marcatore di resistenza all'ampicillina; e pUC19 origine della replica. (B) Espressione di mRNA in tempo reale, rilevata con l'aptamero dBroccoli e la sonda DFHBI (eccitazione 483-14 nm, emissione 530-30 nm). (C) Rilevazione della sintesi proteica nascente in tempo reale con sonda fluorescente FlAsH-EDT2 (eccitazione 500-10 nm, emissione 535-10 nm). (D) Misurazione della fluorescenza in tempo reale della sintesi di mScarlet-I (eccitazione 565-10 nm, emissione 600-10 nm). (E) Colorazione in gel con la sonda fluorescente FlAsH-EDT2. (F) Colorazione blu coomassie di proteine TX-TL totali con standard His6-mScarlet-I purificato per il confronto. Le reazioni sono state eseguite nelle condizioni descritte nel protocollo con 40 nM di modello di DNA plasmidico. Tutti i dati di fluorescenza sono rappresentati come RFU e le barre di errore (deviazione standard di tre ripetizioni tecniche) sono rappresentate all'interno di un'area grigia ombreggiata. Abbreviazioni: TX-TL = trascrizione-traduzione; FlAsH = fluoresceina hairpin arsenical; DFHBI = 3,5-difluoro-4-idrossibenzilidene imidazolinone; RFU = unità di fluorescenza relative. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Flusso di lavoro schematico per la reazione semicontinua S. venezuelae TX-TL. Un flusso di lavoro di esempio per il prodotto naturale TX-TL, utilizzando l'operone biosintetico eme in fase iniziale e l'analisi a valle di HPLC-MS. Le reazioni e le analisi sono dettagliate nel materiale supplementare. La figura è modificata da 5. Abbreviazioni: SMM = Streptomyces Master Mix; TX-TL = trascrizione-traduzione; ALA = acido 5-aminolevulinico; SPE = estrazione in fase solida; ESI-MS = spettrometria di massa a spruzzo di elettroni; HPLC-MS = cromatografia liquida ad alte prestazioni-spettrometria di massa. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Ricetta per il mezzo di crescita batterica GYM e la piastra di agar GYM. Fare clic qui per scaricare questa tabella.
Tabella 2: Reagenti per la preparazione dei tamponi di lavaggio S30A e S30B. Queste informazioni sono state adattate da Kieser et al. 45 Abbreviazione: DTT = ditiotreitolo. Fare clic qui per scaricare questa tabella.
Tabella 3: Ricetta per realizzare le soluzioni S. venezuelae MES e SMM. Abbreviazioni: MES = Minimal Energy Solution; SMM = Streptomyces Master Mix; NTP = nucleoside trifosfato; PEG 6000 = polietilenglicole 6000; 3-PGA = 3-fosfoglicerato; G6P = glucosio-6-fosfato; PVSA = acido polivinilsolfonico. Fare clic qui per scaricare questa tabella.
Tabella 4: Ricetta per la reazione di S. venezuelae TX-TL. Abbreviazioni: MES = Minimal Energy Solution; SMM = Streptomyces Master Mix; TX-TL = trascrizione-traduzione. Fare clic qui per scaricare questa tabella.
Tabella supplementare S1: Plasmidi per il flusso di lavoro S. venezuelae TX-TL. Abbreviazione: TX-TL = trascrizione-traduzione. Fare clic qui per scaricare questa tabella.
Materiale supplementare S2: preparazione standard di calibrazione mScarlet-I e misure del lettore di piastre. Fare clic qui per scaricare questo file.
Materiale supplementare S3: metodi FlAsH-tag. Abbreviazione: FlAsH = fluorescein arsenical hairpin. Fare clic qui per scaricare questo file.
Materiale supplementare S4: reazione semicontinua, purificazione e HPLC-MS. Fare clic qui per scaricare questo file.
Discussione
In questo manoscritto, è stato descritto un protocollo S. venezuelae TX-TL ad alto rendimento con passaggi dettagliati che sono semplici da condurre sia per gli utenti esperti che per quelli nuovi dei sistemi TX-TL. Diverse caratteristiche dei protocolli Streptomyces45 ed E. coli TX-TL41 esistenti sono state rimosse per stabilire un protocollo minimo, ma ad alto rendimento per S. venezuelae TX-TL5,26. Il flusso di lavoro qui raccomandato è quello di garantire che S. venezuelae stia crescendo rapidamente nel mezzo ricco scelto, per essere in grado di inoculare la cultura finale la sera. Ciò consente la raccolta cellulare al picco di crescita la mattina seguente e consente all'utente di raccogliere e preparare l'estratto cellulare attivo lo stesso giorno. Seguendo questo protocollo semplificato, ci si aspetta che un singolo ricercatore possa completare il protocollo comodamente in un framework di tre giorni. Un toolkit plasmidico complementare è stato inoltre fornito per il sistema S. venezuelae TX-TL, incluso un sistema plasmidico a forte espressione (pTU1-A-SP44-mScarlet-I), che fornisce un'ampia funzionalità per l'analisi di mRNA / proteine. Questo plasmide standard è alimentato dal promotore costitutivo SP44 che è altamente attivo in una gamma di Streptomyces spp. e in E. coli39. Per dimostrare il potenziale iniziale del toolkit S. venezuelae TX-TL, i risultati rappresentativi mostrano la sintesi ad alto rendimento di una gamma di proteine fluorescenti, enzimi metaboliti secondari e la biosintesi di un percorso di prodotto naturale modello (dalla biosintesi dell'eme).
Nel complesso, il protocollo contiene una descrizione dettagliata del sistema S. venezuelae TX-TL, nonché consigli pratici per preparare i tre componenti essenziali della reazione TX-TL: (1) estratto cellulare, (2) soluzione streptomyces Master Mix (SMM) e (3) DNA plasmidico. Questo protocollo non richiede attrezzature specializzate e richiede solo competenze di microbiologia e biochimica di routine; quindi, è accessibile alla maggior parte dei laboratori. Il protocollo è adatto per reazioni su piccola scala (10-100 μL) e su larga scala (~ 2,5 ml), sebbene una certa ottimizzazione della dimensione / aerazione della reazione possa influenzare la resa proteica. Il volume di reazione raccomandato è di 33 μL in un tubo da 2 mL o di 10 μL in una piastra da 384 pozzetti. L'estratto grezzo richiede cinque giorni per essere preparato da una sola persona a partire da una scorta di glicerolo. Ogni litro (L) di coltura produce almeno 5 ml di estratto cellulare (equivalente a ~ 1500 x 10 μL TX-TL reazioni)-questa è una stima prudente e tiene conto della perdita del campione durante le fasi di lavaggio e la chiarificazione dell'estratto cellulare. Ogni fase del protocollo è indipendente e può essere ottimizzata dall'utente per soddisfare le sue esigenze. Una delle principali limitazioni per tutti i sistemi senza celle è la variazione dei lotti46,47. I fattori generici includono l'errore di pipettaggio, l'esperienza utente, la variazione dei batch multimediali e le differenze tra le apparecchiature. Introduciamo specificamente un mix principale per ridurre al minimo gli errori di pipettaggio e forniamo istruzioni dettagliate che coprono l'uso di supporti e apparecchiature. Ad oggi, il protocollo è riproducibile da una serie di utenti in almeno cinque gruppi di ricerca del Regno Unito. Tuttavia, non è noto quale ruolo la variazione biologica contribuisca alla variabilità dei lotti senza cellule. Oltre alle differenze globali di regolazione dell'espressione genica, la plasticità del genoma in Streptomyces spp. è ampiamente riportata ed è un potenziale contributore48. Per studiare la variazione del lotto, si consiglia di coltivare fino a quattro colture separate da 1 L derivate da quattro singole colonie coltivate durante la notte. In precedenza, è stata osservata una variazione fino al 28% (in termini di deviazione standard) tra quattro lotti biologici (4 L per lotto fornito ~ 20 ml di estratto cellulare)5. Sulla base di questi dati, un obiettivo minimo ragionevole per un nuovo utente è di 2,8 μM per sfGFP e 3,5 μM mScarlet-I/mVenus-I utilizzando i plasmidi disponibili su AddGene: questi target sono inferiori del 30% rispetto alla media osservata nei dati precedenti. Se si desidera l'analisi HPLC-MS a valle, il PEG 6000 può essere rimosso dalle miscele master, sebbene ci si possa aspettare una diminuzione della resa complessiva tx-TL fino al 50%.
In termini di potenziale dei sistemi specializzati senza cellule di Streptomyces5,6, vi è un crescente desiderio di sviluppare nuovi strumenti di laboratorio bagnati per applicazioni bioprospettative come i prodotti naturali. Il genere Streptomyces è immerso nella storia della scoperta di prodotti naturali, tra cui antibiotici, erbicidi e farmaci farmaceutici49. Le crescenti conoscenze acquisite dai progetti di sequenziamento dell'intero genoma e dai più recenti strumenti bioinformatici50,51,52 hanno rivelato un livello senza precedenti di prodotti naturali codificati dai BGC all'interno dei genomi microbici53. Lo sblocco di queste informazioni genetiche, che si prevede contengano nuovi farmaci/sostanze chimiche ed enzimi utili alla biotecnologia, richiederà lo sviluppo di nuove strategie di biologia sintetica, compresi nuovi sistemi di espressione e una serie di strumenti di ingegneria metabolica54. I sistemi TX-TL specializzati basati su Streptomyces sono vantaggiosi per studiare geni ed elementi regolatori da Actinobacteria e genomi correlati per i seguenti motivi: [1] disponibilità di un ambiente di ripiegamento proteico nativo26, [2] accesso a un pool di tRNA ottimale per un'elevata espressione genica G + C (%) e [3] un metabolismo primario attivo per il potenziale apporto di precursori biosintetici. Inoltre, un vantaggio chiave dei sistemi privi di cellule è la caratterizzazione ad alto rendimento delle parti genetiche e dell'espressione genica, utilizzando il sequenziamento di nuova generazione13 e la robotica acustica per la gestione dei liquidi8,11,12. In sintesi, il toolkit S. venezuelae TX-TL5 fornisce uno strumento complementare nel campo della biologia sintetica per prodotti naturali. Il toolkit S. venezuelae TX-TL sosterrà l'ulteriore sviluppo di S. venezuelae come sistema modello e fornirà un metodo per progettare nuove parti / strumenti di biologia sintetica ed esplorare percorsi biosintetici del metabolita secondario ed enzimi.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano riconoscere il seguente supporto alla ricerca: EPSRC [EP/K038648/1] per SJM come PDRA con PSF; Wellcome Trust ha sponsorizzato la borsa di studio ISSF per SJM con PSF presso l'Imperial College di Londra; Borsa di ricerca della Royal Society [RGS\R1\191186]; Premio Wellcome Trust SEED [217528/Z/19/Z] per SJM presso l'Università del Kent; e Global Challenges Research Fund (GCRF) Ph.D. borsa di studio per KC presso l'Università del Kent.
Materiali
| Name | Company | Catalog Number | Comments |
| 2.5 L UltraYield Flask | Thomson | 931136-B | |
| 3-PGA (>93%) | Sigma | P8877 | |
| 384 Well Black/Clear Bottom Plate | ThermoFisher | 10692202 | |
| Ammonium chloride (98%) | Fluorochem | 44722 | |
| ATP, CTP, UTP, GTP (100 mM solution, >99%) | ThermoFisher | R0481 | |
| Carbenicillin (contact supplier for purity) | Melford | C46000-25.0 | |
| D-(+)-glucose (contact supplier for purity) | Melford | G32040 | |
| DFHBI (≥98% - HPLC) | Sigma | SML1627 | |
| DTT (contact supplier for purity) | Melford | MB1015 | |
| FlAsH-EDT2 (contact supplier for purity) | Santa Cruz Biotech | sc-363644 | |
| Glucose-6-phosphate (>98%) | Sigma | G7879 | |
| HEPES Free Acid (contact supplier for purity) | Melford | B2001 | |
| L-glutamic acid hemimagnesium salt tetrahydrate (>98%) | Sigma | 49605 | |
| Magnesium chloride (98%) | Fluorochem | 494356 | |
| Malt extract | Sigma | 70167-500G | |
| PEG-6000 | Sigma | 807491 | |
| Pierce 96-well Microdialysis Plate, 10K MWCO | ThermoFisher | 88260 | |
| Poly(vinyl sulfate) potassium salt | Sigma | 271969 | |
| Potassium glutamate (>99%) | Sigma | G1149 | |
| RTS amino acid sampler | 5 Prime | 2401530 | |
| Sodium chloride (99%) | Fluorochem | 94554 | |
| Supelclean LC-18 SPE C-18 SPE column (1 g) | Sigma | 505471 | |
| Yeast Extract | Melford | Y1333 | |
| Equipment | |||
| Platereader | BMG | Omega |
Riferimenti
- Carbonell, P., et al. An automated Design-Build-Test-Learn pipeline for enhanced microbial production of fine chemicals. Communications Biology. 1, 66 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods Protocols. 2 (1), 24 (2019).
- Zimmerman, E. S., et al. Production of site-specific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system. Bioconjugate Chemistry. 25 (2), 351-361 (2014).
- Wiegand, D. J., Lee, H. H., Ostrov, N., Church, G. M. Cell-free protein expression using the rapidly growing bacterium Vibrio natriegens. Journal of Visualized Experiments: JoVE. (145), e59495 (2019).
- Moore, S. J., et al. A Streptomyces venezuelae cell-free toolkit for synthetic biology. ACS Synthetic Biology. 10 (2), 402-411 (2021).
- Xu, H., Liu, W. -. Q., Li, J. Translation related factors improve the productivity of a Streptomyces-based cell-free protein synthesis system. ACS Synthetic Biology. 9 (5), 1221-1224 (2020).
- Yim, S. S., et al. Multiplex transcriptional characterizations across diverse bacterial species using cell-free systems. Molecular Systems Biology. 15 (8), 8875 (2019).
- Moore, S. J., et al. Rapid acquisition and model-based analysis of cell-free transcription-translation reactions from nonmodel bacteria. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4340-4349 (2018).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production--a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Geertz, M., Shore, D., Maerkl, S. J. Massively parallel measurements of molecular interaction kinetics on a microfluidic platform. Proceedings of the National Academy of Sciences of the United States of America. 109 (41), 16540-16545 (2012).
- McManus, J. B., Emanuel, P. A., Murray, R. M., Lux, M. W. A method for cost-effective and rapid characterization of engineered T7-based transcription factors by cell-free protein synthesis reveals insights into the regulation of T7 RNA polymerase-driven expression. Archives of Biochemistry and Biophysics. 674, 108045 (2019).
- McManus, J. B., et al. A method for cost-effective and rapid characterization of genetic parts. bioRxiv. , (2021).
- Park, J., Yim, S. S., Wang, H. H. High-throughput transcriptional characterization of regulatory sequences from bacterial Biosynthetic Gene Clusters. ACS Synthetic Biology. , (2021).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, 8 (2010).
- Karim, A. S., Heggestad, J. T., Crowe, S. A., Jewett, M. C. Controlling cell-free metabolism through physiochemical perturbations. Metabolic Engineering. 45, 86-94 (2018).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Garenne, D., Thompson, S., Brisson, A., Khakimzhan, A., Noireaux, V. The all-E. coli TXTL toolbox 3.0: New capabilities of a cell-free synthetic biology platform. Synthetic Biology. , (2021).
- Goering, A. W., et al. In vitro reconstruction of nonribosomal peptide biosynthesis directly from DNA using cell-free protein synthesis. ACS Synthetic Biology. 6 (1), 39-44 (2017).
- Khatri, Y., et al. Multicomponent microscale biosynthesis of unnatural cyanobacterial indole alkaloids. ACS Synthetic Biology. 9 (6), 1349-1360 (2020).
- Zhuang, L., et al. Total in vitro biosynthesis of the nonribosomal macrolactone peptide valinomycin. Metabolic Engineering. 60, 37-44 (2020).
- Hoskisson, P. A., Seipke, R. F. Cryptic or silent? The known unknowns, unknown knowns, and unknown unknowns of secondary metabolism. mBio. 11 (5), 02642 (2020).
- Bentley, S. D., et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature. 417 (6885), 141-147 (2002).
- Li, J., Wang, H., Kwon, Y. -. C., Jewett, M. C. Establishing a high yielding Streptomyces-based cell-free protein synthesis system. Biotechnology and Bioengineering. 114 (6), 1343-1353 (2017).
- Moore, S. J., Lai, H. -. E., Needham, H., Polizzi, K. M., Freemont, P. S. Streptomyces venezuelae TX-TL - a next generation cell-free synthetic biology tool. Biotechnology Journal. 12 (4), (2017).
- Kim, W., et al. Comparative genomics determines strain-dependent secondary metabolite production in Streptomyces venezuelae strains. Biomolecules. 10 (6), 864 (2020).
- Phelan, R. M., et al. Development of next generation synthetic biology tools for use in Streptomyces venezuelae. ACS Synthetic Biology. 6 (1), 159-166 (2017).
- Song, J. Y., et al. Complete genome sequence of Streptomyces venezuelae ATCC 15439, a promising cell factory for production of secondary metabolites. Journal of Biotechnology. 219, 57-58 (2016).
- Bush, M. J., Bibb, M. J., Chandra, G., Findlay, K. C., Buttner, M. J. Genes required for aerial growth, cell division, and chromosome segregation are targets of WhiA before sporulation in Streptomyces venezuelae. mBio. 4 (5), 00684 (2013).
- Schumacher, M. A., et al. The crystal structure of the RsbN-σBldN complex from Streptomyces venezuelae defines a new structural class of anti-σ factor. Nucleic Acids Research. 46 (14), 7405-7417 (2018).
- Ramos-León, F., et al. A conserved cell division protein directly regulates FtsZ dynamics in filamentous and unicellular actinobacteria. Elife. 10, 63387 (2021).
- Bush, M. J., Tschowri, N., Schlimpert, S., Flärdh, K., Buttner, M. J. c-di-GMP signalling and the regulation of developmental transitions in Streptomycetes. Nature Reviews. Microbiology. 13 (12), 749-760 (2015).
- Ehrlich, J., Gottlieb, D., Burkholder, P. R., Anderson, L. E., Pridham, T. G. Streptomyces venezuelae, n. sp., the source of chloromycetin. Journal of Bacteriology. 56 (4), 467-477 (1948).
- Inahashi, Y., et al. Watasemycin biosynthesis in Streptomyces venezuelae: thiazoline C-methylation by a type B radical-SAM methylase homologue. Chemical Science. 8 (4), 2823-2831 (2017).
- Jakeman, D. L., et al. Antimicrobial activities of jadomycin B and structurally related analogues. Antimicrobial Agents and Chemotherapy. 53 (3), 1245-1247 (2009).
- Kodani, S., Sato, K., Hemmi, H., Ohnish-Kameyama, M. Isolation and structural determination of a new hydrophobic peptide venepeptide from Streptomyces venezuelae. Journal of Antibiotics. 67 (12), 839-842 (2014).
- Akey, D. L., et al. Structural basis for macrolactonization by the pikromycin thioesterase. Nature Chemical Biology. 2 (10), 537-542 (2006).
- Bai, C., et al. Exploiting a precise design of universal synthetic modular regulatory elements to unlock the microbial natural products in Streptomyces. Proceedings of the National Academy of Sciences of the United States of America. 112 (39), 12181-12186 (2015).
- Moore, S. J., et al. EcoFlex: A multifunctional MoClo kit for E. coli synthetic biology. ACS Synthetic Biology. 5 (10), 1059-1069 (2016).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. Journal of Visualized Experiments: JoVE. (79), e50762 (2013).
- Kim, D. M., Choi, C. Y. A semicontinuous prokaryotic coupled transcription/translation system using a dialysis membrane. Biotechnology Progress. 12 (5), 645-649 (1996).
- Liu, Y., Fritz, B. R., Anderson, M. J., Schoborg, J. A., Jewett, M. C. Characterizing and alleviating substrate limitations for improved in vitro ribosome construction. ACS Synthetic Biology. 4 (4), 454-462 (2015).
- Bindels, D. S., et al. mScarlet: a bright monomeric red fluorescent protein for cellular imaging. Nature Methods. 14, 53-56 (2017).
- Hopword, D. A., Kieser, T., Bibb, M. J., Buttner, M. J., Chater, K. Practical Streptomyces genetics. John Innes Foundation. , (2000).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnology and Bioengineering. 115 (8), 1904-1914 (2018).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Hoff, G., Bertrand, C., Piotrowski, E., Thibessard, A., Leblond, P. Genome plasticity is governed by double strand break DNA repair in Streptomyces. Scientific Reports. 8, 5272 (2018).
- Bibb, M. J. Regulation of secondary metabolism in streptomycetes. Current Opinion in Microbiology. 8 (2), 208-215 (2005).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43 (1), 237-243 (2015).
- Navarro-Muñoz, J. C., et al. A computational framework to explore large-scale biosynthetic diversity. Nature Chemical Biology. 16, 60-68 (2020).
- Alanjary, M., et al. The Antibiotic Resistant Target Seeker (ARTS), an exploration engine for antibiotic cluster prioritization and novel drug target discovery. Nucleic Acids Research. 45 (1), 42-48 (2017).
- Medema, M. H., Fischbach, M. A. Computational approaches to natural product discovery. Nature Chemical Biology. 11 (9), 639-648 (2015).
- Whitford, C. M., Cruz-Morales, P., Keasling, J. D., Weber, T. The Design-Build-Test-Learn cycle for metabolic engineering of Streptomycetes. Essays in Biochemistry. , (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon