Method Article
Fluxo de trabalho abrangente da proteômica de espingarda baseada em espectrometria de massa de amostras de tecido
Neste Artigo
Resumo
O protocolo descrito fornece uma análise de proteômica quantitativa otimizada das amostras de tecido usando duas abordagens: quantitação baseada em rótulos e sem rótulos. As abordagens baseadas em rótulos têm a vantagem de uma quantitação mais precisa de proteínas, enquanto uma abordagem sem rótulos é mais econômica e usada para analisar centenas de amostras de uma coorte.
Resumo
Os recentes avanços na espectrometria de massa resultaram em análises proteômicas profundas, juntamente com a geração de conjuntos de dados robustos e reprodutíveis. No entanto, apesar dos avanços técnicos consideráveis, a preparação amostral de bioespecímenos como sangue do paciente, CSF e tecido ainda representa desafios consideráveis. Para identificar biomarcadores, a proteômica tecidual geralmente fornece uma fonte amostral atraente para traduzir os achados da pesquisa do banco para a clínica. Pode revelar potenciais biomarcadores candidatos para o diagnóstico precoce de câncer e doenças neurodegenerativas, como doença de Alzheimer, doença de Parkinson, etc. A proteômica tecidual também produz uma riqueza de informações sistêmicas baseadas na abundância de proteínas e ajuda a abordar questões biológicas interessantes.
A análise quantitativa da proteômica pode ser agrupada em duas categorias amplas: uma abordagem baseada em rótulos e uma abordagem sem rótulos. Na abordagem baseada em rótulos, proteínas ou peptídeos são rotulados usando isótopos estáveis como SILAC (rotulagem estável de isótopos com aminoácidos na cultura celular) ou por marcas químicas como ICAT (tags de afinidade codificadas por isótopos), TMT (marca de massa tandem) ou iTRAQ (tag isobárica para quantitação relativa e absoluta). Abordagens baseadas em rótulos têm a vantagem de uma quantificação mais precisa de proteínas e usando rótulos isobáricos, várias amostras podem ser analisadas em um único experimento. A abordagem sem rótulos fornece uma alternativa econômica às abordagens baseadas em rótulos. Centenas de amostras de pacientes pertencentes a uma coorte em particular podem ser analisadas e comparadas com outras coortes baseadas em características clínicas. Aqui, descrevemos um fluxo de trabalho de proteômica quantitativa otimizado para amostras de tecido usando métodos de perfil proteome sem rótulos e baseados em rótulos, que é crucial para aplicações em ciências da vida, especialmente projetos baseados em descobertas de biomarcadores.
Introdução
As tecnologias de proteômica têm o potencial de permitir a identificação e quantificação de potenciais marcadores candidatos que possam auxiliar na detecção e prognóstico da doença1. Os recentes avanços no campo da espectrometria de massa aceleraram a pesquisa clínica no nível da proteína. Pesquisadores estão tentando enfrentar o desafio da complicada civilologia de várias doenças usando proteômica baseada em espectrometria de massa, que agora oferece maior sensibilidade para identificação e quantificação de proteínas2. A medição quantitativa precisa das proteínas é crucial para compreender a cooperação dinâmica e espacial entre proteínas em indivíduos saudáveis e doentes3; no entanto, essa análise em larga escala não é fácil.
Uma das principais limitações do perfil proteômico dos espécimes clínicos é a complexidade das amostras biológicas. Muitos tipos diferentes de amostras têm sido investigados para estudar a doença proteome, como linhas celulares, plasma e tecidos4,5. As linhas celulares são amplamente utilizadas como modelos em experimentos in vitro para imitar diferentes estágios de progressão da doença. No entanto, uma grande limitação com as linhas celulares é que elas facilmente adquirem alterações genotípicas e fenotípicas durante o processo de cultura celular6. Fluidos corporais como o plasma podem ser uma fonte atraente para a descoberta de biomarcadores; no entanto, devido às proteínas altamente abundantes e à amplitude dinâmica da concentração de proteínas, a proteômica plasmática é um pouco mais desafiadora7. Aqui, os peptídeos originados das proteínas mais abundantes podem suprimir aqueles derivados das proteínas de baixa abundância, mesmo que a relação massa/carga seja a mesma6. Embora tenha havido avanços nas tecnologias de esgotamento e fracionamento nos últimos anos, obter uma boa cobertura ainda continua sendo uma grande limitação da proteômica plasmática8,9. O uso de tecidos para investigação proteômica da biologia da doença é preferido, pois as amostras de tecido são mais proximais aos locais da doença e oferecem altas informações fisiológicas e patológicas para fornecer melhores insights sobre a biologia da doença10,11.
Neste manuscrito, fornecemos um protocolo simplificado para a proteômica quantitativa das amostras de tecido. Usamos um tampão contendo 8 M de ureia para a preparação do tecido, pois este buffer é compatível com investigações baseadas em espectrometria de massa. No entanto, é obrigatório limpar os peptídeos para remover sais antes de injetá-los no espectrômetro de massa. Um ponto importante a ser lembrado é reduzir a concentração de ureia para menos de 1 M antes de adicionar trippsina para digestão proteica, pois a trippsina exibe baixa atividade na concentração de 8 M de ureia. Explicamos duas abordagens de proteômica global quantitativa: quantificação baseada em rótulos usando iTRAQ (tags isobáricas para quantificação relativa e absoluta) e quantificação sem rótulos (LFQ). A proteômica quantitativa baseada em iTRAQ é usada principalmente para comparar múltiplas amostras variando em sua condição biológica (por exemplo, normal versus doença ou amostras tratadas). A abordagem utiliza reagentes isobáricos para rotular as aminas primárias n-terminal de peptídeos12. Os reagentes iTRAQ contêm um grupo de repórteres de n-metil piperazina, um grupo balanceador, e um grupo de éster de succinimida N-hidroxy que reage com aminas primárias N-terminal de peptídeos13. Peptídeos digeridos de cada condição são rotulados com um reagente iTRAQ específico. Após a rotulagem, a reação é interrompida e peptídeos rotulados de diferentes condições são agrupados em um único tubo. Esta mistura de amostra combinada é analisada por espectrômetro de massa para identificação e quantificação. Após a análise de MS/MS, fragmentos de íons repórteres com baixas massas moleculares são gerados e as intensidades de íons desses repórteres são usadas para a quantificação das proteínas.
Outra abordagem, quantificação sem rótulos é usada para determinar o número relativo de proteínas em amostras complexas sem rotular peptídeos com isótopos estáveis.
Protocolo
Este estudo foi revisado e aprovado pelos conselhos de revisão institucional e pelo comitê de ética do Instituto Indiano de Tecnologia Bombaim (IITB-IEC/2016/026). Os pacientes/participantes forneceram seu consentimento por escrito para participar deste estudo.
1. Preparação de tecido lysate
NOTA: Realize todas as etapas a seguir no gelo para manter os proteases inativos. Certifique-se de que os bisturis e os tubos utilizados são estéreis para evitar qualquer contaminação cruzada.
- Tome ~30 mg de tecido em um tubo de batida de contas, adicione 200 μL de 1x phosphate tampão salino (PBS) e vórtice dele.
NOTA: Neste estudo, foram levados tecidos tumorais humanos congelados frescos para a preparação do liseto. O protocolo pode ser usado para qualquer tecido congelado fresco com algumas alterações dependendo do tipo de tecidos (tecidos moles ou duros) e da complexidade celular dos tecidos. - Depois disso, gire o tubo para assentar o tecido e remova cuidadosamente o PBS usando uma pipeta. Faça outra lavagem pbs se ainda há vestígios de sangue no tecido.
- Adicione 300 μL de tampão de ureia (8 M ure, 50 mM Tris pH 8.0, 75 mM NaCl, 1 mM MgCl2) e coquetel inibidor de protease (PIC) conforme o protocolo do fabricante.
NOTA: O volume do tampão de lise deve ser suficiente para moer o tecido durante o processo de sônica e para suspender o que é extraído. Tampão de lise muito pouco pode resultar em lise tecidual ineficiente, enquanto o tampão de lise demais diluirá o lise proteico. - Coloque o tubo no gelo e sonicar o tecido em uma amplitude de 40% por 2,5 min com ciclos de pulso de 5 s (ON/OFF, respectivamente).
- Adicione contas de zircônio aos tubos e homogeneize o tecido usando um batedor de contas para 90 s com incubação de 5 minutos no gelo. Repita este passo duas vezes.
- Uma vez que o tecido esteja adequadamente homogeneizado, incubar o tubo no gelo por 10 minutos.
- Após a incubação, centrifugar a amostra a 6.018 x g por 15 min a 4 °C para separar os detritos celulares do sobrenatante.
- Colete o supernatante no tubo fresco rotulado e armazene a -80 °C como alíquotas até que use mais.
2. Quantificação de proteínas e verificação de qualidade de tecidos lysates
- Quantifique a concentração de proteína no tecido lysate utilizando o reagente de Bradford conforme descrito no Arquivo Suplementar 1.
- Seguindo a quantificação da proteína, execute 10 μg de tecido lysate em um gel SDS-PAGE de 12% para verificar a qualidade do lysate.
NOTA: O processamento mais a jusante deve ser realizado apenas para os lysates que limpam as verificações de qualidade.
3. Digestão enzimática de proteínas
NOTA: Os passos para digestão enzimática são mostrados na Figura 1a.
- Para digestão, tome 50 μg de proteínas e adicione ddH2O para compensar o volume a 20 μL.
- Agora, prepare 20 mM Tris (2-carboxyethyl) fosfina (TCEP) do estoque (0,5 M TCEP) adicionando 0,8 μL de estoque ao liseto proteico para reduzir as ligações de dissulfeto nas proteínas e incubar a amostra a 37 °C por 60 min.
- Prepare 40 mM iodoacetamida (IAA) em ddH2O e adicione 1,6 μL para aquilar os resíduos de cisteína reduzidos. Incubar no escuro por 10 minutos à temperatura ambiente.
- Adicione tampão de diluição contendo 25 mM Tris pH 8.0 e 1 mM CaCl2 em uma razão de 1:8 para diluir a concentração de ureia para menos de 1 M na amostra. Neste momento, verifique o pH.
NOTA: Se usar trippsina como enzima de digestão, certifique-se de que a concentração de ureia seja inferior a 1 M. - Para realizar a digestão, adicione trippsina em uma relação enzima/substrato de 1:50. Incubar os tubos a 37 °C em um banho seco tremendo por 16 h para digestão durante a noite.
NOTA: A enzima trypsin é uma protease altamente reativa que é propensa à auto-digestão. Tome cuidado extra e realize a adição de trippsina rapidamente sobre o gelo. - Após 16 h de incubação, seque os peptídeos digeridos em um concentrador de vácuo.
4. Dessacionando peptídeos digeridos
NOTA: Para realizar a dessação dos peptídeos, use as pontas do palco C18.
- Ative a ponta do estágio C18 adicionando 50 μL de metanol. Centrifugar a ponta a 1.000 x g por 2 min na RT. Descarte o filtrado coletado na parte inferior do tubo. Repita duas vezes.
- Adicione 50 μL de acetonitrila em ácido fórmico de 0,1% para lavar a ponta do palco. Centrifugar o tubo a 1.000 x g por 2 min na RT. Descarte o filtrado coletado na parte inferior do tubo. Repita este passo duas vezes.
- Adicione 50 μL de 0,1% (v/v) FA para equilibrar a coluna. Novamente, realize a centrifugação a 1.000 x g por 2 min no RT e descarte o filtrado.
- Reconstituir os peptídeos digeridos secos em 50 μL de ácido fórmico de 0,1%.
NOTA: Evite a formação de bolhas de ar dentro das pontas do estágio ao passar a amostra. As pontas do palco não devem ser completamente secas durante a etapa de centrifugação, pois a secagem pode levar à perda de peptídeos. - Adicione os peptídeos reconstituídos na ponta do palco ativado e passe a amostra através da ponta do palco por centrifugação a 1.000 x g por 2 min. Repita este passo pelo menos quatro vezes. Armazene o fluxo a 4 °C.
- Para lavar a amostra, adicione 50 μL de ácido fórmico de 0,1% (v/v). Repita a etapa de centrifugação e descarte o filtrado.
- Para a eluição de peptídeos, adicione 50 μL de 40% (v/v) ACN em 0,1% de ácido fórmico (v/v) e passe-o pela ponta do palco por centrifugação. Colete o filtrado em um tubo fresco. Repita a etapa com 50% e 60% de ACN em ácido fórmico de 0,1% e colete o filtrado no mesmo tubo fresco.
- Seque os peptídeos dessarados coletados no tubo fresco usando um concentrador de vácuo.
NOTA: Os peptídeos secos dessacados estão prontos para serem injetados, ou podem ser armazenados a -20 °C por 6 meses. Para armazenamento a longo prazo (>6 meses), armazene os peptídeos a -80 °C.
5. Quantificação de peptídeos desselted
- Reconstitua os peptídeos secos dessacados em 0,1% fa.
- Limpe a placa de medição fotométrica com tecido livre de fiapos usando 70% de etanol.
- Use 2 μL de 0,1% fa para definir o em branco.
- Adicione 2 μL de amostras reconstituídas na placa em réplicas.
- Coloque a placa no espectrômetro e meça a absorvância em 205 nm e 280 nm.
- Calcular absortividade molar (ε) usando a seguinte fórmula:
ε = 27 / [1 - 3,85 * A280 / A205]
NOTA: A absortividade molar (ε) é uma medida da probabilidade da transição eletrônica ou quão bem uma espécie absorve o comprimento de onda particular da radiação que está sendo incidente sobre ela. O valor de ε deve estar na faixa de 31 mL mg-1cm-1 a 33 mL mg-1cm-1. Se o valor não cair na faixa, isso indica que as amostras não são digeridas corretamente. - Calcule a concentração de peptídeos em μg/μL usando a seguinte fórmula:
Concentração de peptídeo = Net OD (205) / 0,051 * ε
6. Quantitação sem rótulos (LFQ) dos peptídeos digeridos
NOTA: Para a quantitação sem rótulos, utilize os parâmetros LC e MS mencionados no Arquivo Suplementar 2. Um dado de alta cobertura foi obtido quando três réplicas biológicas do mesmo tipo da amostra foram executadas no espectrômetro de massa.
- Configuração de cromatografia líquida
- Após a quantificação de peptídeos desselted, tome 2 μg de peptídeos em um frasco e compor o volume até 10 μL usando FA 0,1%. A concentração de peptídeos dessalted será de 200 ng/μL.
- Abra o amostrador automático do sistema de cromatografia líquida (ver Tabela de Materiais) e coloque o frasco dentro do autosampler.
- Use 0,1% (v/v) FA para equilibrar a coluna pré-coluna e a coluna analítica.
- Pegue 1 μg de peptídeo digerido dessel do frasco e carregue-o na coluna.
- Defina o gradiente lc de acordo com a complexidade da amostra. Neste experimento, foi utilizado gradiente LC para 120 min para quantitação sem rótulos das amostras de tecido.
- Configuração em MS: Antes de otimizar quaisquer ensaios de proteômica, realize uma verificação de controle de qualidade do instrumento monitorando alguns peptídeos do Bovine Serum Albumin (BSA) utilizando qualquer software para adequação do sistema e analisando a cobertura do BSA (Figura 2A,B). Os parâmetros de aquisição foram definidos no instrumento utilizando o software de aquisição de dados MS (ver Tabela de Materiais).
- Abra o software, clique duas vezes em Configuração de instrumentos e selecione o modelo de peptídeos-ID com parâmetros padrão.
- Defina os parâmetros de MS usando o arquivo suplementar 2 e salve-o como um novo método.
- Agora, abra o software para preencher os detalhes da amostra; clique duas vezes na Configuração de sequênciae preencha os detalhes como tipo de amostra, nome da amostra, localização de salvamento de arquivos, arquivo do método de instrumento, volume de injeção e posição da amostra.
- Uma vez que todas as informações sejam preenchidas, selecione a linha e inicie a Execução.
7. Quantitação baseada em rótulos (iTRAQ) de peptídeos digeridos
NOTA: A quantificação baseada em rótulos pode ser realizada usando diferentes rótulos isobáricos, como reagentes iTRAQ ou TMT, etc. Aqui, o iTRAQ 4-plex foi utilizado para a rotulagem de peptídeos digeridos a partir de três amostras de tecido. O procedimento de rotulagem iTRAQ 4-plex é mencionado abaixo.
- Rotulagem de peptídeos digeridos usando reagentes iTRAQ.
NOTA: Neste experimento, são utilizados peptídeos de três amostras de tecido. De cada amostra de tecido, 80 μg de peptídeos digeridos são tomados em quatro tubos para rotulagem com reagentes iTRAQ (114, 115, 116 e 117) (ver Arquivo Suplementar 3 para os parâmetros experimentais detalhados).- Antes de usar o reagente iTRAQ, leve cada frasco do reagente à temperatura ambiente (aproximadamente 5 min). Dê uma breve rotação de aproximadamente 30 s para trazer a solução na parte inferior de cada frasco.
NOTA: Certifique-se de que em cada frasco, a solução de 10-15 μL deve estar presente. - Para rotulagem iTRAQ, reconstitua os peptídeos secos em 20 μL de tampão de dissolução fornecido no kit de rotulagem iTRAQ.
- Reconstitua os rótulos adicionando 70 μL de etanol do frasco fornecido no kit e misture a solução para 30 s e gire-a por 10 s.
NOTA: É aconselhável que todas as etapas sejam executadas de acordo com as instruções do fabricante. - Adicione os rótulos iTRAQ homogêneos (114, 115, 116 e 117) aos seus respectivos tubos contendo amostras de peptídeos e permita que ocorra a reação de rotulagem.
- Misture os componentes de cada tubo vórtice do tubo por 30 s e, em seguida, gire o tubo por 10 s para trazer a mistura de volta para o fundo do tubo.
NOTA: Verifique o pH da solução usando papel pH. pH deve ser maior que 8; se não, adicione até 10 μL do tampão de dissolução para ajustar o pH. - Incubar cada tubo em temperatura ambiente por 90 minutos. No final da reação, sacie qualquer rótulo desvinculado do excesso no tubo adicionando água de grau MS.
- Incubar os tubos em temperatura ambiente por 30 min a 1 h.
- Uma vez que a incubação acabou, transfira todo o conteúdo rotulado para um único tubo e seque os peptídeos rotulados em um concentrador de vácuo.
NOTA: Um procedimento de rotulagem semelhante pode ser seguido para rotulagem TMT.
- Antes de usar o reagente iTRAQ, leve cada frasco do reagente à temperatura ambiente (aproximadamente 5 min). Dê uma breve rotação de aproximadamente 30 s para trazer a solução na parte inferior de cada frasco.
- Configuração de cromatografia líquida
- Reconstitua as amostras em ácido fórmico de 0,1%, abra o autosampler de nano LC e coloque as amostras dentro do autosampler. Use os parâmetros mencionados no Arquivo Suplementar 2 para a configuração LC.
- Defina o gradiente lc de acordo com a complexidade da amostra. O gradiente LC de 180 min foi utilizado neste experimento para a quantitação baseada em rótulos (iTRAQ) das amostras de tecido.
NOTA: Para amostras menos complexas, o gradiente curto pode separar eficientemente a maioria dos peptídeos. No entanto, se a amostra for muito complexa, use um gradiente mais longo para uma melhor separação dos peptídeos.
- Configuração de MS para técnica iTRAQ
- Configure todos os parâmetros de MS para a quantitação baseada em rótulos da mesma forma usada para a quantitação sem rótulo, exceto para a energia de colisão, que foi definida para 35% para fragmentação de MS/MS na quantitação baseada em rótulo.
8. Análise de dados
- Analise os arquivos brutos (espectro MS/MS) obtidos a partir do espectrômetro de massa LC usando um software de análise comercialmente disponível (ver Tabela de Materiais).
NOTA: O banco de dados Human Reference Proteome da Uniprot (UP00005640) composto por 71.785 sequências de proteínas foi utilizado para obter identidades proteicas utilizando motores de busca Sequest HT e Mascot (v2.6.0). Os parâmetros para quantitação sem rótulos e quantitação baseada em rótulos estão descritos no Arquivo Suplementar 4.
Resultados
Usamos duas abordagens diferentes para a descoberta de proteômicas: abordagens proteômicas sem rótulos e baseadas em rótulos. O perfil proteico das amostras de tecido em SDS-PAGE mostrou as proteínas intactas e poderia ser considerado para análise proteômica(Figura 2A). A verificação de controle de qualidade do instrumento foi monitorada via software de adequação do sistema e mostrou a variação diária no desempenho do instrumento (Figura 2B). Observou-se cobertura sequencial de 91% da amostra BSA em 30 min de gradiente de LC(Figura 2C). O gradiente LC foi otimizado utilizando 500 ng de digestão celular HeLa comercial e observamos 2425 proteínas em um gradiente de 2 h em comparação com 1488 proteínas em um gradiente de 1 h(Figura 2D). Conseguimos identificar, em média, 2.428 proteínas nas três réplicas técnicas de uma amostra de tecido de piscina(Figura 2E).
Os parâmetros otimizados de LC e MS foram aplicados em três amostras de tecido biológico diferentes (Figura 3 e Arquivo Suplementar 2). O cromatógrama mostrou boa reprodutibilidade entre três diferentes amostras de tecido biológico. Identificamos 2725, 2748 e 2718 proteínas quantificáveis das amostras de tecido 1, 2 e 3, respectivamente, utilizando uma abordagem baseada sem rótulos. Observou-se que 151 proteínas foram comuns no primeiro e segundo experimentos do LFQ, 163 proteínas foram compartilhadas entre o segundo e o terceiro experimentos de LFQ, e 187 proteínas foram compartilhadas entre o primeiro e o terceiro experimentos, enquanto 2190 proteínas foram comuns em todas as três amostras de tecido(Figura 4A).
Inspecionamos o cromatógrafo e verificamos os rótulos iTRAQ e descobrimos que ele estava presente em quase todos os espectros de MS/MS. Os três conjuntos foram executados para o experimento iTRAQ. As proteínas obtidas de cada conjunto foram 2455, 2285 e 2307, respectivamente. Foram encontradas 287 proteínas comuns na amostra 1 e na amostra 2, 183 proteínas foram comuns na amostra 2 e na amostra 3, e 195 proteínas foram comuns na amostra 1 e na amostra 3. O número total de proteínas comuns nas três amostras foi de 1557 (Figura 4B).
Comparamos correspondências espectrais totais de peptídeos (PSMs), grupos de peptídeos, proteínas totais, grupos proteicos e o número de proteínas obtidas após 1% de FDR do experimento LFQ e iTRAQ(Figura 4C).

Figura 1: Fluxo de trabalho para proteômica tecidual. (A) As etapas de processamento da amostra para preparar amostras de tecido lysate para a análise de MS. (B) Etapas para a quantitação sem rótulos. (C) Etapas para a quantitação baseada em rótulos. (D) Etapas para análise de dados usando um descobridor de proteome. Clique aqui para ver uma versão maior desta figura.

Figura 2: Controle de verificação de qualidade das amostras de tecido e reprodutibilidade do instrumento. (A) Verificação de qualidade dos tecidos lysates em 12% SDS-PAGE(B) Monitoramento de alguns peptídeos da BSA utilizando Panorama para verificar a variabilidade do instrumento nos diferentes dias. (C) A cobertura sequencial da BSA em três réplicas técnicas. (D) Otimização dos parâmetros de LC para amostras de tecido. (E) O número de espectrais de peptídeos, peptídeos e proteínas em três amostras biológicas diferentes. Clique aqui para ver uma versão maior desta figura.

Figura 3: Parâmetros de LC e MS para análise de proteômica da amostra de tecido. (A,B) O gradiente de cromatografia líquida utilizado para separar os peptídeos para quantitação sem rótulo (A) e quantitação à base de rótulos(B)da amostra de tecido. (C) Os parâmetros de MS para quantitação sem rótulos e quantitação baseada em rótulos. Clique aqui para ver uma versão maior desta figura.
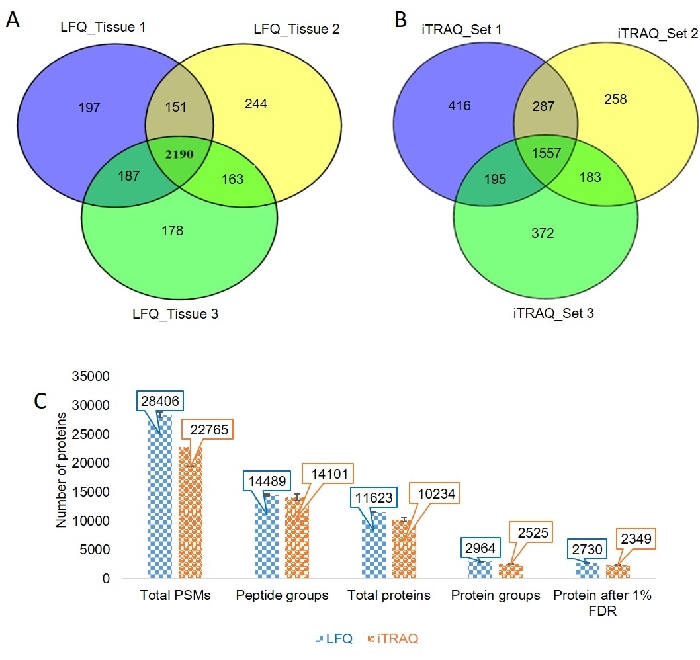
Figura 4: Quantitação sem rótulos e baseada em rótulos da amostra de tecido. (A) O diagrama de Venn representa as proteínas comuns e exclusivas nas amostras de tecido 1, 2 e 3 do experimento sem rótulos. (B) O diagrama de venn representa as proteínas comuns e exclusivas nas amostras de tecido 1, 2 e 3 do experimento baseado em rótulos. (C) A análise comparativa do número de fósforos espectrais de peptídeos (PSMs), grupos de peptídeos, proteínas totais, grupos proteicos e número de proteínas após 1% de FDR em quantitação sem rótulo (LFQ) e experimento à base de rótulos (iTRAQ). Clique aqui para ver uma versão maior desta figura.
Arquivos Complementares. Clique aqui para baixar este Arquivo.
Discussão
A proteômica tecidual de amostras biológicas nos permite explorar novos biomarcadores potenciais associados a diferentes estágios de progressão da doença. Também explica o mecanismo de sinalização e caminhos associados à progressão da doença. O protocolo descrito para análise de proteômica quantitativa tecidual fornece dados de boa cobertura reprodutíveis. A maioria das etapas foram adaptadas a partir das instruções do fabricante. Para obter dados de alta qualidade, as etapas a seguir são mais cruciais. Portanto, deve-se dar um cuidado extra durante a realização dessas etapas.
A digestão incompleta de proteínas e a contaminação da queratina podem fornecer menos cobertura de proteínas (n < 1000), afetando assim o experimento global. O pH das amostras (pH 8) e a concentração de ureia nas amostras (menos de 1 M) garantirão a digestão eficiente das proteínas. O uso de tampão fresco e manuseio de amostras com cuidado reduzirá as chances de contaminação por queratina. Os reagentes iTRAQ são extremamente caros e requer uma plataforma ms sofisticada para executar o MS/MS e o software para analisar os dados. Os experimentos de proteômica são sensíveis à contaminação de sais, quantificação de peptídeos e eficiência de rotulagem de reagentes iTRAQ/TMT. Antes da análise de MS/MS, certifique-se de que os peptídeos digeridos sejam devidamente dessacidos para reduzir o ruído de fundo nos dados. No caso da técnica iTRAQ, a fragmentação da etiqueta anexada gera um íon repórter de baixa massa molecular que pode ser usado para quantificar relativamente os peptídeos e as proteínas das quais se originaram, enquanto para abordagem livre de rótulos, a área sob a curva é considerada para a quantitação. Para aumentar a confiança na quantificação de proteínas, especialmente devem ser realizados experimentos independentes de validação, mrm/PRM.
A análise de amostras de tecido utilizando dois métodos de quantitação (proteômica sem rótulos e à base de rótulos) foi descrita para obter uma boa cobertura de proteínas. A abordagem proteômica quantitativa sem rótulos oferece diversas vantagens para seu uso em estudos clínicos. As amostras são executadas de forma independente, e isso é particularmente importante para estudos que são realizados para uma coorte de pacientes, pois há um grande número de amostras a serem analisadas via espectrômetro de massa. Usando uma réplica técnica, como um pool de peptídeos otimizados, pode-se garantir uma boa reprodutibilidade mesmo que as amostras sejam executadas em diferentes pontos de tempo. Essa abordagem tem sido utilizada em grandes estudos de coorte, como o CPTAC, que é um esforço de muitas comunidades internacionais14.
Os potenciais alvos emergindo do estudo poderiam ser considerados para validação por meio de abordagens de proteômica direcionada. Concluímos que os projetos baseados na análise de amostras de tecidos poderiam ser fortemente beneficiados pelos fluxos de trabalho detalhados da proteômica quantitativa fornecidos neste estudo. As etapas mencionadas ajudarão a otimizar o método e mapear o proteome das amostras de tecido. A seleção de técnicas proteômicas quantitativas pode depender do número de amostras, disponibilidade de plataformas de MS e da questão biológica a ser abordada.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Reconhecemos o Projeto MHRD-UAY (UCHHATAR AVISHKAR YOJANA), projeto #34_IITB para ss e massfiitb facility na IIT Bombay apoiado pelo Departamento de Biotecnologia (BT/PR13114/INF/22/206/2015) para realizar todos os experimentos relacionados ao MS.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acetonitrile (MS grade) | Fisher Scientific | A/0620/21 | |
| Bovine Serum Albumin | HiMedia | TC194-25G | |
| Calcium chloride | Fischer Scienific | BP510-500 | |
| Formic acid (MS grade) | Fisher Scientific | 147930250 | |
| Iodoacetamide | Sigma | 1149-25G | |
| Isopropanol (MS grade) | Fisher Scientific | Q13827 | |
| Magnesium Chloride | Fischer Scienific | BP214-500 | |
| Methanol (MS grade) | Fisher Scientific | A456-4 | |
| MS grade water | Pierce | 51140 | |
| Phosphate Buffer Saline | HiMedia | TL1006-500ML | |
| Protease inhibitor cocktail | Roche Diagnostics | 11873580001 | |
| Sodium Chloride | Merck | DF6D661300 | |
| TCEP | Sigma | 646547 | |
| Tris Base | Merck | 648310 | |
| Trypsin (MS grade) | Pierce | 90058 | |
| Bradford Reagent | Bio-Rad | 5000205 | |
| Urea | Merck | MB1D691237 | |
| Supplies | |||
| Hypersil Gold C18 column | Thermo | 25002-102130 | |
| Micropipettes | Gilson | F167380 | |
| Stage tips | MilliPore | ZTC18M008 | |
| Zirconia/Silica beads | BioSpec products | 11079110z | |
| Equipment | |||
| Bead beater (Homogeniser) | Bertin Minilys | P000673-MLYS0-A | |
| Microplate reader (spectrophotometer) | Thermo | MultiSkan Go | |
| pH meter | Eutech | CyberScan pH 510 | |
| Probe Sonicator | Sonics Materials, Inc | VCX 130 | |
| Shaking Drybath | Thermo | 88880028 | |
| Orbitrap Fusion mass spectrometer | Thermo | FSN 10452 | |
| Nano LC | Thermo | EASY-nLC1200 | |
| Vacuum concentrator | Thermo | Savant ISS 110 | |
| Software | |||
| Proteome Discoverer | Thrermo | Proteome Discoverer 2.2.0.388 |
Referências
- Petricoin, E., Wulfkuhle, J., Espina, V., Liotta, L. A. Clinical proteomics: revolutionizing disease detection and patient tailoring therapy. Journal of Proteome Research. 3 (2), 209-217 (2004).
- Geho, D. H., Petricoin, E. F., Liotta, L. A. Blasting into the microworld of tissue proteomics: a new window on cancer. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 10 (3), 825-827 (2004).
- Hashimoto, Y., Greco, T. M., Cristea, I. M. Contribution of mass spectrometry-based proteomics to discoveries in developmental biology. Advances in Experimental Medicine and Biology. 1140, 143-154 (2019).
- Faria, S. S., et al. A timely shift from shotgun to targeted proteomics and how it can be groundbreaking for cancer research. Frontiers in Oncology. 7, 13 (2017).
- Ray, S., et al. Proteomic technologies for the identification of disease biomarkers in serum: advances and challenges ahead. Proteomics. 11 (11), 2139-2161 (2011).
- Chen, E. I., Yates, J. R. Cancer proteomics by quantitative shotgun proteomics. Molecular Oncology. 1 (2), 144-159 (2007).
- Geyer, P. E., Holdt, L. M., Teupser, D., Mann, M. Revisiting biomarker discovery by plasma proteomics. Molecular Systems Biology. 13 (9), 942 (2017).
- Ray, S., et al. Proteomic analysis of Plasmodium falciparum induced alterations in humans from different endemic regions of India to decipher malaria pathogenesis and identify surrogate markers of severity. Journal of Proteomics. 127, 103-113 (2015).
- Ray, S., et al. Clinicopathological analysis and multipronged quantitative proteomics reveal oxidative stress and cytoskeletal proteins as possible markers for severe vivax malaria. Scientific Reports. 6, 24557 (2016).
- Sharma, S., et al. Multipronged quantitative proteomic analyses indicate modulation of various signal transduction pathways in human meningiomas. Proteomics. 15 (2-3), 394-407 (2015).
- Sharma, S., Ray, S., Moiyadi, A., Sridhar, E., Srivastava, S. Quantitative proteomic analysis of meningiomas for the identification of surrogate protein markers. Scientific Reports. 4, 7140 (2014).
- Aslam, B., Basit, M., Nisar, M. A., Khurshid, M., Rasool, M. H. Proteomics: Technologies and their applications. Journal of Chromatographic Science. 55 (2), 182-196 (2017).
- Wiese, S., Reidegeld, K. A., Meyer, H. E., Warscheid, B. Protein labeling by iTRAQ: A new tool for quantitative mass spectrometry in proteome research. Proteomics. 7 (3), 340-350 (2007).
- Rudnick, P. A., et al. A description of the Clinical Proteomic Tumor Analysis Consortium (CPTAC) common data analysis pipeline. Journal of Proteome Research. 15 (3), 1023-1032 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados