Method Article
Flux de travail complet de protéomique des échantillons de tissus basée sur la spectrométrie de masse
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le protocole décrit fournit une analyse protéomique quantitative optimisée des échantillons de tissus à l’aide de deux approches : la quantification basée sur l’étiquette et la quantification sans étiquette. Les approches basées sur l’étiquette ont l’avantage d’une quantification plus précise des protéines, tandis qu’une approche sans étiquette est plus rentable et utilisée pour analyser des centaines d’échantillons d’une cohorte.
Résumé
Les progrès récents de la spectrométrie de masse ont abouti à une analyse protéomique approfondie ainsi qu’à la génération d’ensembles de données robustes et reproductibles. Cependant, malgré les progrès techniques considérables, la préparation d’échantillons à partir d’échantillons biologiques tels que le sang des patients, le LCR et les tissus pose encore des défis considérables. Pour identifier les biomarqueurs, la protéomique tissulaire fournit souvent une source d’échantillon attrayante pour traduire les résultats de la recherche du laboratoire à la clinique. Il peut révéler des biomarqueurs potentiels candidats pour le diagnostic précoce du cancer et des maladies neurodégénératives telles que la maladie d’Alzheimer, la maladie de Parkinson, etc. La protéomique tissulaire produit également une mine d’informations systémiques basées sur l’abondance des protéines et aide à répondre à des questions biologiques intéressantes.
L’analyse protéomique quantitative peut être regroupée en deux grandes catégories : une approche basée sur l’étiquette et une approche sans étiquette. Dans l’approche basée sur l’étiquette, les protéines ou les peptides sont marqués à l’aide d’isotopes stables tels que SILAC (marquage isotopique stable avec des acides aminés en culture cellulaire) ou par des marqueurs chimiques tels que ICAT (marqueurs d’affinité codés isotopiques), TMT (marqueur de masse tandem) ou iTRAQ (marqueur isobare pour la quantification relative et absolue). Les approches basées sur l’étiquette ont l’avantage d’une quantification plus précise des protéines et en utilisant des étiquettes isobares, plusieurs échantillons peuvent être analysés en une seule expérience. L’approche sans étiquette offre une alternative rentable aux approches basées sur l’étiquette. Des centaines d’échantillons de patients appartenant à une cohorte particulière peuvent être analysés et comparés à d’autres cohortes en fonction des caractéristiques cliniques. Ici, nous avons décrit un flux de travail protéomique quantitatif optimisé pour les échantillons de tissus à l’aide de méthodes de profilage du protéome sans étiquette et basées sur l’étiquette, ce qui est crucial pour les applications dans les sciences de la vie, en particulier les projets basés sur la découverte de biomarqueurs.
Introduction
Les technologies protéomiques ont le potentiel de permettre l’identification et la quantification de marqueurs candidats potentiels qui peuvent aider à la détection et au pronostic de la maladie1. Les progrès récents dans le domaine de la spectrométrie de masse ont accéléré la recherche clinique au niveau des protéines. Les chercheurs tentent de relever le défi de la pathobiologie compliquée de plusieurs maladies en utilisant la protéomique basée sur la spectrométrie de masse, qui offre maintenant une sensibilité accrue pour l’identification et la quantification des protéines2. Une mesure quantitative précise des protéines est cruciale pour comprendre la coopération dynamique et spatiale entre les protéines chez les individus sains et malades3; cependant, une telle analyse à l’échelle du protéome n’est pas facile.
L’une des principales limites du profilage protéomique des échantillons cliniques est la complexité des échantillons biologiques. De nombreux types d’échantillons différents ont été étudiés pour étudier le protéome de la maladie, tels que les lignées cellulaires, le plasma et les tissus4,5. Les lignées cellulaires sont largement utilisées comme modèles dans des expériences in vitro pour imiter différents stades de la progression de la maladie. Cependant, une limitation majeure avec les lignées cellulaires est qu’elles acquièrent facilement des changements génotypiques et phénotypiques au cours du processus de culture cellulaire6. Les fluides corporels tels que le plasma pourraient être une source attrayante pour la découverte de biomarqueurs; cependant, en raison de la forte abondance des protéines et de la plage dynamique de concentration en protéines, la protéomique plasmatique est un peu plus difficile7. Ici, les peptides issus des protéines les plus abondantes peuvent supprimer celles dérivées des protéines peu abondantes même si le rapport masse/charge est le même6. Bien qu’il y ait eu des progrès dans les technologies d’épuisement et de fractionnement au cours des dernières années, obtenir une bonne couverture reste une limitation majeure de la protéomiqueplasmatique 8,9. L’utilisation de tissus pour l’investigation protéomique de la biologie de la maladie est préférée car les échantillons de tissus sont les plus proches des sites de la maladie et offrent des informations physiologiques et pathologiques élevées pour fournir de meilleures informations sur la biologie de la maladie10,11.
Dans ce manuscrit, nous avons fourni un protocole simplifié pour la protéomique quantitative des échantillons de tissus. Nous avons utilisé un tampon contenant de l’urée de 8 M pour la préparation du lysate tissulaire car ce tampon est compatible avec les investigations basées sur la spectrométrie de masse. Cependant, il est obligatoire de nettoyer les peptides pour éliminer les sels avant de les injecter dans le spectromètre de masse. Un point important à retenir est de réduire la concentration d’urée à moins de 1 M avant d’ajouter de la trypsine pour la digestion des protéines, car la trypsine présente une faible activité à une concentration d’urée de 8 M. Nous avons expliqué deux approches de la protéomique globale quantitative : la quantification basée sur l’étiquette à l’aide d’iTRAQ (étiquettes isobares pour la quantification relative et absolue) et la quantification sans étiquette (LFQ). La protéomique quantitative basée sur l’iTRAQ est principalement utilisée pour comparer plusieurs échantillons dont l’état biologique varie (p. ex., échantillons normaux par rapport à la maladie ou traités). L’approche utilise des réactifs isobares pour étiqueter les amines primaires N-terminales des peptides12. Les réactifs iTRAQ contiennent un groupe rapporteur N-méthyl pipérazine, un groupe équilibreur et un groupe ester N-hydroxy succinimide qui réagit avec les amines primaires N-terminales des peptides13. Les peptides digérés de chaque condition sont marqués avec un réactif iTRAQ particulier. Après le profilage, la réaction est arrêtée et les peptides marqués de différentes conditions sont regroupés dans un seul tube. Ce mélange d’échantillons combinés est analysé par spectromètre de masse pour identification et quantification. Après l’analyse MS/MS, des fragments d’ions rapporteurs de faible masse moléculaire sont générés et les intensités ioniques de ces ions rapporteurs sont utilisées pour la quantification des protéines.
Une autre approche, la quantification sans étiquette, est utilisée pour déterminer le nombre relatif de protéines dans des échantillons complexes sans étiqueter les peptides avec des isotopes stables.
Protocole
Cette étude a été examinée et approuvée par les comités d’examen institutionnels et le comité d’éthique de l’Institut indien de technologie de Bombay (IITB-IEC/2016/026). Les patients/participants ont donné leur consentement écrit pour participer à cette étude.
1. Préparation du lysate tissulaire
REMARQUE: Effectuez toutes les étapes suivantes sur la glace pour garder les protéases inactives. Assurez-vous que les scalpels et les tubes utilisés sont stériles pour éviter toute contamination croisée.
- Prenez environ 30 mg de tissu dans un tube battant les billes, ajoutez 200 μL de solution saline tampon phosphate (PBS) et vortexez-la.
REMARQUE: Dans cette étude, des tissus tumoraux cérébraux humains frais congelés ont été prélevés pour la préparation du lysate. Le protocole peut être utilisé pour tout tissu frais congelé avec quelques changements en fonction du type de tissus (tissus mous ou durs) et de la complexité cellulaire des tissus. - Après cela, faites tourner le tube pour déposer le tissu et retirez soigneusement le PBS à l’aide d’une pipette. Effectuez un autre lavage PBS s’il reste encore des traces de sang dans les tissus.
- Ajouter 300 μL de tampon de lyse de l’urée (8 M d’urée, 50 mM tris pH 8,0, 75 mM naCl, 1 mM MgCl2)et un cocktail d’inhibiteurs de protéase (PIC) conformément au protocole du fabricant.
REMARQUE: Le volume du tampon de lyse doit être suffisant pour broyer le tissu pendant le processus de sonication et pour suspendre ce qui est extrait. Trop peu de tampon de lyse peut entraîner une lyse tissulaire inefficace, tandis qu’un tampon de lyse trop important diluera le lysate de protéine. - Placez le tube sur de la glace et soniquez le tissu à une amplitude de 40% pendant 2,5 min avec des cycles d’impulsion de 5 s (ON / OFF, respectivement).
- Ajouter des billes de zirconium aux tubes et homogénéiser le tissu à l’aide d’un batteur de billes pendant 90 s avec 5 min d’incubation sur glace. Répétez cette étape deux fois.
- Une fois que le tissu est correctement homogénéisé, incuber le tube sur de la glace pendant 10 min.
- Après incubation, centrifuger l’échantillon à 6 018 x g pendant 15 min à 4 °C pour séparer les débris cellulaires du surnageant.
- Recueillir le surnageant dans le tube frais étiqueté et le conserver à -80 °C sous forme d’aliquotes jusqu’à une utilisation ultérieure.
2. Quantification des protéines et contrôle de la qualité des lysates tissulaires
- Quantifier la concentration de protéines dans le lysate tissulaire à l’aide du réactif de Bradford tel que décrit dans le dossier supplémentaire 1.
- Après la quantification des protéines, faire passer 10 μg de lysate tissulaire sur un gel SDS-PAGE à 12% pour vérifier la qualité du lysate.
NOTE: Le traitement ultérieur en aval ne doit être effectué que pour les lysates qui effectuent les contrôles de qualité.
3. Digestion enzymatique des protéines
REMARQUE: Les étapes de la digestion enzymatique sont illustrées à la figure 1a.
- Pour la digestion, prendre 50 μg de protéines et ajouter ddH2O pour porter le volume à 20 μL.
- Maintenant, préparez 20 mM de tris (2-carboxyéthyl) phosphine (TCEP) à partir du stock (0,5 M TCEP) en ajoutant 0,8 μL du stock au lysate de protéine pour réduire les liaisons disulfure dans les protéines et incuber l’échantillon à 37 °C pendant 60 min.
- Préparer 40 mM d’iodoacétamide (IAA) dans du ddH2O et ajouter 1,6 μL pour alkyler les résidus de cystéine réduits. Incuber dans l’obscurité pendant 10 min à température ambiante.
- Ajouter un tampon de dilution contenant 25 mM de Tris pH 8,0 et 1 mM deCaCl2 dans un rapport de 1:8 pour diluer la concentration d’urée à moins de 1 M dans l’échantillon. À ce stade, vérifiez le pH.
REMARQUE: Si vous utilisez la trypsine comme enzyme de digestion, assurez-vous que la concentration d’urée est inférieure à 1 M. - Pour effectuer la digestion, ajoutez de la trypsine à un rapport enzyme/substrat de 1:50. Incuber les tubes à 37 °C dans un bain sec et agité pendant 16 h pour une digestion nocturne.
REMARQUE: L’enzyme trypsine est une protéase hautement réactive qui est sujette à l’auto-digestion. Prenez des précautions supplémentaires et effectuez l’ajout de trypsine rapidement sur la glace. - Après 16 h d’incubation, sécher les peptides digérés dans un concentrateur sous vide.
4. Dessalage des peptides digérés
REMARQUE: Pour effectuer le dessalage des peptides, utilisez des embouts de stade C18.
- Activez l’embout de l’étage C18 en ajoutant 50 μL de méthanol. Centrifuger la pointe à 1 000 x g pendant 2 min à RT. Jeter le filtrat recueilli au fond du tube. Répétez deux fois.
- Ajouter 50 μL d’acétonitrile dans de l’acide formique à 0,1 % pour laver la pointe de la scène. Centrifuger le tube à 1 000 x g pendant 2 min à RT. Jeter le filtrat recueilli au fond du tube. Répétez cette étape deux fois.
- Ajouter 50 μL de 0,1 % (v/v) FA pour équilibrer la colonne. Encore une fois, effectuez la centrifugation à 1 000 x g pendant 2 min à TA et jetez le filtrat.
- Reconstituer les peptides digérés séchés dans 50 μL d’acide formique à 0,1 %.
REMARQUE: Évitez la formation de bulles d’air à l’intérieur des extrémités de la scène lors du passage de l’échantillon. Les pointes de stade ne doivent pas être complètement séchées pendant l’étape de centrifugation, car le séchage peut entraîner une perte de peptides. - Ajouter les peptides reconstitués dans la pointe de l’étape activée et faire passer l’échantillon à travers la pointe de l’étape par centrifugation à 1 000 x g pendant 2 min. Répétez cette étape au moins quatre fois. Conserver le débit à 4 °C.
- Pour laver l’échantillon, ajouter 50 μL d’acide formique à 0,1 % (v/v). Répétez l’étape de centrifugation et jetez le filtrat.
- Pour l’élution des peptides, ajouter 50 μL d’ACN à 40 % (v/v) dans de l’acide formique à 0,1 % (v/v) et le faire passer à travers la pointe de l’étape par centrifugation. Recueillir le filtrat dans un tube frais. Répétez l’étape avec 50% et 60% d’ACN dans 0,1% d’acide formique et collectez le filtrat dans le même tube frais.
- Sécher les peptides dessalés recueillis dans le tube frais à l’aide d’un concentrateur sous vide.
REMARQUE: Les peptides dessalés séchés sont prêts à être injectés, ou ils peuvent être conservés à -20 ° C pendant 6 mois. Pour un stockage à long terme (>6 mois), stockez les peptides à -80 °C.
5. Quantification des peptides dessalés
- Reconstituer les peptides dessalés séchés dans 0,1% FA.
- Essuyez la plaque de mesure photométrique avec du tissu non pelucheux à l’aide d’éthanol à 70%.
- Utilisez 2 μL de 0,1 % FA pour définir le blanc.
- Ajouter 2 μL d’échantillons reconstitués sur la plaque en répétitions.
- Placez la plaque dans le spectrophotomètre et mesurez l’absorbance à 205 nm et 280 nm.
- Calculer l’absorptivité molaire (ε) à l’aide de la formule suivante :
ε = 27 / [1 - 3,85 * A280 / A205]
REMARQUE: L’absorptivité molaire (ε) est une mesure de la probabilité de la transition électronique ou de la façon dont une espèce absorbe la longueur d’onde particulière du rayonnement qui y est incident. La valeur de ε doit être comprise entre 31 mL mg-1cm-1 et 33 mL mg-1cm-1. Si la valeur ne se situe pas dans la plage, cela indique que les échantillons ne sont pas correctement digérés. - Calculer la concentration peptidique en μg/μL à l’aide de la formule suivante :
Concentration de peptide = OD nette (205) / 0,051 * ε
6. Quantification sans étiquette (LFQ) des peptides digérés
REMARQUE: Pour une quantification sans étiquette, utilisez les paramètres LC et MS mentionnés dans le fichier supplémentaire 2. Des données à couverture élevée ont été obtenues lorsque trois répliques biologiques du même type d’échantillon ont été exécutées dans le spectromètre de masse.
- Configuration de la chromatographie liquide
- Après la quantification des peptides dessalés, prendre 2 μg de peptides dans un flacon et porter le volume à 10 μL en utilisant 0,1% DE FA. La concentration de peptides dessalés sera de 200 ng/μL.
- Ouvrez l’auto-échantillonneur du système de chromatographie liquide (voir Tableau des matériaux)et placez le flacon à l’intérieur de l’échantillonneur automatique.
- Utilisez 0,1 % (v/v) d’AF pour équilibrer la pré-colonne et la colonne analytique.
- Prenez 1 μg de peptide digéré dessalé du flacon et chargez-le sur la colonne.
- Définissez le gradient LC en fonction de la complexité de l’échantillon. Dans cette expérience, le gradient LC a été utilisé pendant 120 minutes pour la quantification sans étiquette des échantillons de tissus.
- Configuration de la SEP: Avant d’optimiser les tests protéomiques, effectuez un contrôle de la qualité de l’instrument en surveillant certains peptides de l’albumine sérique bovine (BSA) à l’aide de tout logiciel pour l’adéquation du système et en analysant la couverture de BSA (Figure 2A, B). Les paramètres d’acquisition ont été définis dans l’instrument à l’aide du logiciel d’acquisition de données MS (voir Tableau des matériaux).
- Ouvrez le logiciel, double-cliquez sur Instrument Set Up et sélectionnez le modèle de peptides-ID avec les paramètres par défaut.
- Définissez les paramètres MS à l’aide du fichier supplémentaire 2 et enregistrez-le en tant que nouvelle méthode.
- Maintenant, ouvrez le logiciel pour remplir les détails de l’exemple; double-cliquez sur Configuration de la séquenceet remplissez les détails tels que le type d’échantillon, le nom de l’échantillon, l’emplacement d’enregistrement du fichier, le fichier de méthode de l’instrument, le volume d’injection et la position de l’échantillon.
- Une fois toutes les informations remplies, sélectionnez la ligne et démarrez l’exécuter.
7. Quantification par étiquette (iTRAQ) des peptides digérés
REMARQUE: La quantification basée sur l’étiquette peut être effectuée à l’aide de différentes étiquettes isobares telles que les réactifs iTRAQ ou TMT, etc. Ici, iTRAQ 4-plex a été utilisé pour le étiquetage de peptides digérés à partir de trois échantillons de tissus. La procédure d’étiquetage iTRAQ 4-plex est mentionnée ci-dessous.
- Étiquetage des peptides digérés à l’aide de réactifs iTRAQ.
REMARQUE: Dans cette expérience, des peptides provenant de trois échantillons de tissus sont utilisés. De chaque échantillon de tissu, 80 μg de peptides digérés sont prélevés dans quatre tubes pour le étiquetage avec des réactifs iTRAQ (114, 115, 116 et 117) (voir le dossier supplémentaire 3 pour les paramètres expérimentaux détaillés).- Avant d’utiliser le réactif iTRAQ, porter chaque flacon du réactif à température ambiante (environ 5 min). Donner un bref tour d’environ 30 s pour amener la solution au fond de chaque flacon.
REMARQUE: Assurez-vous que dans chaque flacon, une solution de 10 à 15 μL doit être présente. - Pour le étiquetage iTRAQ, reconstituer les peptides séchés dans 20 μL de tampon de dissolution fourni dans le kit de étiquetage iTRAQ.
- Reconstituer les étiquettes en ajoutant 70 μL d’éthanol à partir du flacon fourni dans le kit et mélanger la solution pendant 30 s et la faire tourner pendant 10 s.
REMARQUE: Il est conseillé que toutes les étapes soient effectuées conformément aux instructions du fabricant. - Ajouter les étiquettes iTRAQ mélangées de manière homogène (114, 115, 116 et 117) à leurs tubes respectifs contenant des échantillons de peptides et permettre la réaction de étiquetage.
- Mélangez les composants de chaque tube en faisant tourbillon du tube pendant 30 s, puis faites tourner le tube pendant 10 s pour ramener le mélange au fond du tube.
REMARQUE: Vérifiez le pH de la solution à l’aide de papier pH. Le pH doit être supérieur à 8; sinon, additionner jusqu’à 10 μL du tampon de dissolution pour ajuster le pH. - Incuber chaque tube à température ambiante pendant 90 min. À la fin de la réaction, éteindre tout excès d’étiquette non liée dans le tube en ajoutant de l’eau de qualité MS.
- Incuber les tubes à température ambiante pendant 30 min à 1 h.
- Une fois l’incubation terminée, transférez tout le contenu marqué dans un seul tube et séchez les peptides marqués dans un concentrateur sous vide.
REMARQUE: Une procédure d’étiquetage similaire peut être suivie pour l’étiquetage TMT.
- Avant d’utiliser le réactif iTRAQ, porter chaque flacon du réactif à température ambiante (environ 5 min). Donner un bref tour d’environ 30 s pour amener la solution au fond de chaque flacon.
- Configuration de la chromatographie liquide
- Reconstituer les échantillons en acide formique à 0,1%, ouvrir l’échantillonneur automatique de nano LC et placer les échantillons à l’intérieur de l’échantillonneur automatique. Utilisez les paramètres mentionnés dans le fichier supplémentaire 2 pour la configuration LC.
- Définissez le gradient LC en fonction de la complexité de l’échantillon. Un gradient LC de 180 min a été utilisé dans cette expérience pour la quantification basée sur l’étiquette (iTRAQ) des échantillons de tissus.
REMARQUE: Pour les échantillons moins complexes, un gradient court peut séparer efficacement la plupart des peptides. Cependant, si l’échantillon est très complexe, utilisez un gradient plus long pour une meilleure séparation des peptides.
- Configuration MS pour la technique iTRAQ
- Configurez tous les paramètres MS pour la quantification basée sur l’étiquette de la même manière que celui utilisé pour la quantification sans étiquette, à l’exception de l’énergie de collision, qui a été fixée à 35 % pour la fragmentation MS/MS dans la quantification basée sur l’étiquette.
8. Analyse des données
- Analyser les fichiers bruts (spectre MS/MS) obtenus à partir du spectromètre de masse LC à l’aide d’un logiciel d’analyse disponible dans le commerce (voir Tableau des matériaux).
REMARQUE: La base de données human reference Proteome d’Uniprot (UP000005640) comprenant 71 785 séquences de protéines a été utilisée pour obtenir des identités de protéines à l’aide des moteurs de recherche Sequest HT et Mascot (v2.6.0). Les paramètres de la quantification sans étiquette et de la quantification basée sur l’étiquette sont décrits dans le fichier supplémentaire 4.
Résultats
Nous avons utilisé deux approches différentes pour la protéomique de découverte : les approches protéomiques sans étiquette et les approches protéomiques basées sur l’étiquette. Le profil protéique des échantillons de tissus sur SDS-PAGE a montré les protéines intactes et a pu être pris en compte pour l’analyse protéomique (Figure 2A). Le contrôle de la qualité de l’instrument a été surveillé à l’intermédiaire d’un logiciel d’adéquation du système et a montré la variation journalaire des performances de l’instrument (Figure 2B). Nous avons observé une couverture séquentielle de 91 % de l’échantillon BSA en 30 min de gradient LC(Figure 2C). Le gradient LC a été optimisé à l’aide de 500 ng de digestion cellulaire HeLa commerciale et nous avons observé 2425 protéines dans un gradient de 2 h par rapport à 1488 protéines dans un gradient de 1 h(Figure 2D). Nous avons pu identifier, en moyenne, 2428 protéines dans les trois répliques techniques d’un échantillon de tissu de pool(Figure 2E).
Les paramètres optimisés de LC et de SEP ont été appliqués à trois échantillons de tissus biologiques différents(figure 3 et dossier supplémentaire 2). Le chromatogramme a montré une bonne reproductibilité entre trois échantillons de tissus biologiques différents. Nous avons identifié 2725, 2748 et 2718 protéines quantifiables à partir d’échantillons de tissus 1, 2 et 3, respectivement, en utilisant une approche sans étiquette. Nous avons observé que 151 protéines étaient communes dans les première et deuxième expériences LFQ, 163 protéines ont été partagées entre les deuxième et troisième expériences LFQ, et 187 protéines ont été partagées entre la première et la troisième expérience, tandis que 2190 protéines étaient communes dans les trois échantillons de tissus(Figure 4A).
Nous avons inspecté le chromatogramme et vérifié les étiquettes iTRAQ et constaté qu’il était présent dans presque tous les spectres MS/MS. Les trois ensembles ont été exécutés pour l’expérience iTRAQ. Le nombre de protéines obtenues à partir de chaque ensemble était de 2455, 2285 et 2307, respectivement. 287 protéines se sont avérées communes dans l’échantillon 1 et l’échantillon 2, 183 protéines étaient communes dans l’échantillon 2 et l’échantillon 3, et 195 protéines étaient communes dans les échantillons 1 et 3. Le nombre total de protéines communes dans les trois échantillons était de 1557(figure 4B).
Nous avons comparé les correspondances spectrales peptidiques totales (PSM), les groupes peptidiques, les protéines totales, les groupes protéiques et le nombre de protéines obtenues après 1% FDR de l’expérience LFQ et iTRAQ(Figure 4C).

Figure 1: Flux de travail pour la protéomique tissulaire. (A) Les étapes de traitement des échantillons pour préparer les échantillons de lysate tissulaire pour l’analyse de la SEP. (B) Étapes pour une quantification sans étiquette. (C) Étapes de quantification basée sur l’étiquette. (D) Étapes d’analyse des données à l’aide d’un découvreur de protéome. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2: Contrôle de la qualité des échantillons de tissus et de la reproductibilité de l’instrument. (A) Contrôle de la qualité des lysates tissulaires sur 12% SDS-PAGE (B) Surveillance de certains peptides de BSA à l’aide de Panorama pour vérifier la variabilité de l’instrument entre les différents jours. (C) La couverture séquentielle de BSA en trois répliques techniques. (D) Optimisation des paramètres LC pour les échantillons de tissus. (E) Nombre de correspondances spectrales peptidiques, de peptides et de protéines dans trois échantillons biologiques différents. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3: Paramètres LC et MS pour l’analyse protéomique de l’échantillon de tissu. (A,B) Gradient de chromatographie liquide utilisé pour séparer les peptides pour la quantification sans étiquette (A) et la quantification basée sur l’étiquette (B) de l’échantillon de tissu. (C) Les paramètres de l’État membre pour la quantification sans étiquette et la quantification basée sur l’étiquette. Veuillez cliquer ici pour voir une version agrandie de cette figure.
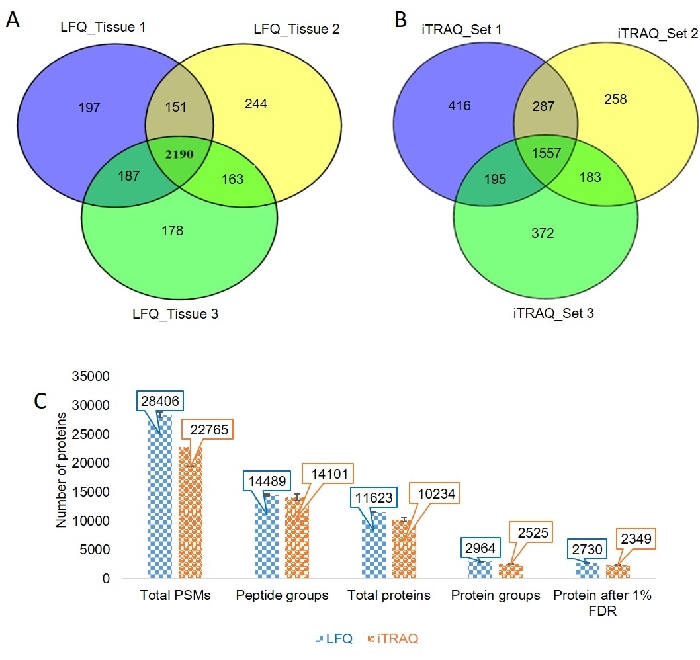
Figure 4: Quantification sans étiquette et basée sur l’étiquette de l’échantillon de tissu. (A) Le diagramme de Venn représente les protéines communes et exclusives dans les échantillons de tissus 1, 2 et 3 de l’expérience sans étiquette. (B) Le diagramme de Venn représente les protéines communes et exclusives dans les échantillons de tissus 1, 2 et 3 de l’expérience basée sur l’étiquette. (C) L’analyse comparative du nombre d’appatations spectrales peptidiques (PSM), de groupes peptidiques, de protéines totales, de groupes protéiques et d’indices protéiques après 1 % de FDR dans la quantification sans étiquette (LFQ) et l’expérience basée sur l’étiquette (iTRAQ). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dossiers supplémentaires. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La protéomique tissulaire des échantillons biologiques nous permet d’explorer de nouveaux biomarqueurs potentiels associés à différents stades de progression de la maladie. Il explique également le mécanisme de signalisation et les voies associées à la progression de la maladie. Le protocole décrit pour l’analyse protéomique quantitative des tissus fournit de bonnes données de couverture reproductibles. La plupart des étapes ont été adaptées des instructions du fabricant. Afin d’obtenir des données de haute qualité, les étapes suivantes sont les plus cruciales. Par conséquent, des précautions supplémentaires doivent être prises lors de l’exécution de ces étapes.
La digestion incomplète des protéines et la contamination de la kératine peuvent fournir une couverture moindre des protéines (n < 1000), affectant ainsi l’expérience globale. Le pH des échantillons (pH 8) et la concentration d’urée dans les échantillons (moins de 1 M) assureront la digestion efficace des protéines. L’utilisation de tampon frais et la manipulation des échantillons avec soin rédiront les risques de contamination par la kératine. Les réactifs iTRAQ sont extrêmement coûteux et nécessitent une plate-forme MS sophistiquée pour effectuer le MS / MS et un logiciel pour analyser les données. Les expériences de protéomique sont sensibles à la contamination par les sels, à la quantification peptidique et à l’efficacité de l’étiquetage des réactifs iTRAQ/TMT. Avant l’analyse MS/MS, assurez-vous que les peptides digérés sont correctement dessalés pour réduire le bruit de fond dans les données. Dans le cas de la technique iTRAQ, la fragmentation de la balise attachée génère un ion rapporteur de masse moléculaire faible qui peut être utilisé pour quantifier relativement les peptides et les protéines dont ils sont issus, tandis que pour l’approche sans étiquette, l’aire sous la courbe est considérée pour la quantification. Pour augmenter la confiance dans la quantification des protéines, des expériences de validation indépendantes, en particulier, MRM / PRM doivent être effectuées.
L’analyse d’échantillons de tissus à l’aide de deux méthodes de quantification (protéomique sans étiquette et protéomique basée sur l’étiquette) a été décrite pour obtenir une bonne couverture des protéines. L’approche protéomique quantitative sans étiquette offre plusieurs avantages pour son utilisation dans les études cliniques. Les échantillons sont exécutés indépendamment, ce qui est particulièrement important pour les études entreprises pour une cohorte de patients, car il existe un grand nombre d’échantillons à analyser par spectromètre de masse. En utilisant une réplique technique telle qu’un pool de peptides optimisés, on peut assurer une bonne reproductibilité même si les échantillons sont exécutés à des moments différents. Cette approche a été utilisée dans de grandes études de cohorte telles que le CPTAC, qui est un effort de nombreuses communautés internationales14.
Les cibles potentielles issues de l’étude pourraient être considérées pour validation à l’aide d’approches protéomiques ciblées. Nous concluons que les projets basés sur l’analyse d’échantillons de tissus pourraient être fortement bénéficiés des flux de travail détaillés de la protéomique quantitative fournis dans cette étude. Les étapes mentionnées aideront à optimiser la méthode et à cartographier le protéome des échantillons de tissus. Le choix des techniques protéomiques quantitatives peut dépendre du nombre d’échantillons, de la disponibilité des plateformes de SEP et de la question biologique à traiter.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous reconnaissons le projet MHRD-UAY (UCHHATAR AVISHKAR YOJANA), projet #34_IITB à SS et MASSFIITB Facility à IIT Bombay soutenu par le Département de biotechnologie (BT / PR13114 / INF / 22/206/ 2015) pour mener à bien toutes les expériences liées à la SEP.
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acetonitrile (MS grade) | Fisher Scientific | A/0620/21 | |
| Bovine Serum Albumin | HiMedia | TC194-25G | |
| Calcium chloride | Fischer Scienific | BP510-500 | |
| Formic acid (MS grade) | Fisher Scientific | 147930250 | |
| Iodoacetamide | Sigma | 1149-25G | |
| Isopropanol (MS grade) | Fisher Scientific | Q13827 | |
| Magnesium Chloride | Fischer Scienific | BP214-500 | |
| Methanol (MS grade) | Fisher Scientific | A456-4 | |
| MS grade water | Pierce | 51140 | |
| Phosphate Buffer Saline | HiMedia | TL1006-500ML | |
| Protease inhibitor cocktail | Roche Diagnostics | 11873580001 | |
| Sodium Chloride | Merck | DF6D661300 | |
| TCEP | Sigma | 646547 | |
| Tris Base | Merck | 648310 | |
| Trypsin (MS grade) | Pierce | 90058 | |
| Bradford Reagent | Bio-Rad | 5000205 | |
| Urea | Merck | MB1D691237 | |
| Supplies | |||
| Hypersil Gold C18 column | Thermo | 25002-102130 | |
| Micropipettes | Gilson | F167380 | |
| Stage tips | MilliPore | ZTC18M008 | |
| Zirconia/Silica beads | BioSpec products | 11079110z | |
| Equipment | |||
| Bead beater (Homogeniser) | Bertin Minilys | P000673-MLYS0-A | |
| Microplate reader (spectrophotometer) | Thermo | MultiSkan Go | |
| pH meter | Eutech | CyberScan pH 510 | |
| Probe Sonicator | Sonics Materials, Inc | VCX 130 | |
| Shaking Drybath | Thermo | 88880028 | |
| Orbitrap Fusion mass spectrometer | Thermo | FSN 10452 | |
| Nano LC | Thermo | EASY-nLC1200 | |
| Vacuum concentrator | Thermo | Savant ISS 110 | |
| Software | |||
| Proteome Discoverer | Thrermo | Proteome Discoverer 2.2.0.388 |
Références
- Petricoin, E., Wulfkuhle, J., Espina, V., Liotta, L. A. Clinical proteomics: revolutionizing disease detection and patient tailoring therapy. Journal of Proteome Research. 3 (2), 209-217 (2004).
- Geho, D. H., Petricoin, E. F., Liotta, L. A. Blasting into the microworld of tissue proteomics: a new window on cancer. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 10 (3), 825-827 (2004).
- Hashimoto, Y., Greco, T. M., Cristea, I. M. Contribution of mass spectrometry-based proteomics to discoveries in developmental biology. Advances in Experimental Medicine and Biology. 1140, 143-154 (2019).
- Faria, S. S., et al. A timely shift from shotgun to targeted proteomics and how it can be groundbreaking for cancer research. Frontiers in Oncology. 7, 13 (2017).
- Ray, S., et al. Proteomic technologies for the identification of disease biomarkers in serum: advances and challenges ahead. Proteomics. 11 (11), 2139-2161 (2011).
- Chen, E. I., Yates, J. R. Cancer proteomics by quantitative shotgun proteomics. Molecular Oncology. 1 (2), 144-159 (2007).
- Geyer, P. E., Holdt, L. M., Teupser, D., Mann, M. Revisiting biomarker discovery by plasma proteomics. Molecular Systems Biology. 13 (9), 942 (2017).
- Ray, S., et al. Proteomic analysis of Plasmodium falciparum induced alterations in humans from different endemic regions of India to decipher malaria pathogenesis and identify surrogate markers of severity. Journal of Proteomics. 127, 103-113 (2015).
- Ray, S., et al. Clinicopathological analysis and multipronged quantitative proteomics reveal oxidative stress and cytoskeletal proteins as possible markers for severe vivax malaria. Scientific Reports. 6, 24557 (2016).
- Sharma, S., et al. Multipronged quantitative proteomic analyses indicate modulation of various signal transduction pathways in human meningiomas. Proteomics. 15 (2-3), 394-407 (2015).
- Sharma, S., Ray, S., Moiyadi, A., Sridhar, E., Srivastava, S. Quantitative proteomic analysis of meningiomas for the identification of surrogate protein markers. Scientific Reports. 4, 7140 (2014).
- Aslam, B., Basit, M., Nisar, M. A., Khurshid, M., Rasool, M. H. Proteomics: Technologies and their applications. Journal of Chromatographic Science. 55 (2), 182-196 (2017).
- Wiese, S., Reidegeld, K. A., Meyer, H. E., Warscheid, B. Protein labeling by iTRAQ: A new tool for quantitative mass spectrometry in proteome research. Proteomics. 7 (3), 340-350 (2007).
- Rudnick, P. A., et al. A description of the Clinical Proteomic Tumor Analysis Consortium (CPTAC) common data analysis pipeline. Journal of Proteome Research. 15 (3), 1023-1032 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon