Method Article
Umfassender Workflow der massenspektrometrischen Schrotflintenproteomik von Gewebeproben
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Das beschriebene Protokoll bietet eine optimierte quantitative Proteomik-Analyse von Gewebeproben mit zwei Ansätzen: markierungsbasierte und markierungsfreie Quantifizierung. Markierungsbasierte Ansätze haben den Vorteil einer genaueren Quantifizierung von Proteinen, während ein markierungsfreier Ansatz kostengünstiger ist und zur Analyse von Hunderten von Proben einer Kohorte verwendet wird.
Zusammenfassung
Jüngste Fortschritte in der Massenspektrometrie haben zu einer tiefen proteomischen Analyse zusammen mit der Generierung robuster und reproduzierbarer Datensätze geführt. Trotz der erheblichen technischen Fortschritte stellt die Probenvorbereitung aus Bioproben wie Patientenblut, Liquor und Gewebe jedoch immer noch erhebliche Herausforderungen dar. Für die Identifizierung von Biomarkern bietet die Gewebeproteomik oft eine attraktive Probenquelle, um die Forschungsergebnisse von der Bank in die Klinik zu übertragen. Es kann potenzielle Biomarkerkandidaten für die Früherkennung von Krebs und neurodegenerativen Erkrankungen wie Alzheimer, Parkinson usw. aufdecken. Die Gewebeproteomik liefert auch eine Fülle von systemischen Informationen, die auf der Fülle von Proteinen basieren und helfen, interessante biologische Fragen zu beantworten.
Die quantitative Proteomik-Analyse kann in zwei große Kategorien eingeteilt werden: einen labelbasierten und einen labelfreien Ansatz. Im labelbasierten Ansatz werden Proteine oder Peptide mit stabilen Isotopen wie SILAC (stabile Isotopenmarkierung mit Aminosäuren in Zellkultur) oder durch chemische Tags wie ICAT (isotopencodierte Affinity-Tags), TMT (Tandem-Massen-Tag) oder iTRAQ (isobarer Tag für relative und absolute Quantifizierung) markiert. Markierungsbasierte Ansätze haben den Vorteil einer genaueren Quantifizierung von Proteinen und mit isobaren Markierungen können mehrere Proben in einem einzigen Experiment analysiert werden. Der etikettenfreie Ansatz bietet eine kostengünstige Alternative zu etikettenbasierten Ansätzen. Hunderte von Patientenproben, die zu einer bestimmten Kohorte gehören, können analysiert und mit anderen Kohorten basierend auf klinischen Merkmalen verglichen werden. Hier haben wir einen optimierten quantitativen Proteomik-Workflow für Gewebeproben unter Verwendung von markierungsfreien und markierungsbasierten Proteom-Profiling-Methoden beschrieben, der für Anwendungen in den Biowissenschaften, insbesondere für Projekte auf Biomarker-Entdeckung, von entscheidender Bedeutung ist.
Einleitung
Proteomik-Technologien haben das Potenzial, die Identifizierung und Quantifizierung potenzieller Kandidatenmarker zu ermöglichen, die bei der Erkennung und Prognose der Krankheit helfen können1. Jüngste Fortschritte auf dem Gebiet der Massenspektrometrie haben die klinische Forschung auf Proteinebene beschleunigt. Forscher versuchen, die Herausforderung der komplizierten Pathobiologie mehrerer Krankheiten mit Hilfe der massenspektrometrischen Proteomik anzugehen, die jetzt eine erhöhte Empfindlichkeit für die Identifizierung und Quantifizierung von Proteinen bietet2. Eine genaue quantitative Messung von Proteinen ist entscheidend, um die dynamische und räumliche Zusammenarbeit zwischen Proteinen bei gesunden und kranken Individuen zu verstehen3; Eine solche Analyse auf proteomweiter Skala ist jedoch nicht einfach.
Eine wesentliche Einschränkung der proteomischen Profilierung klinischer Proben ist die Komplexität biologischer Proben. Viele verschiedene Arten von Proben wurden untersucht, um das Krankheitsproteom zu untersuchen, wie Zelllinien, Plasma und Gewebe4,5. Zelllinien werden häufig als Modelle in In-vitro-Experimenten verwendet, um verschiedene Stadien des Krankheitsverlaufs nachzuahmen. Eine wesentliche Einschränkung bei Zelllinien besteht jedoch darin, dass sie während des Prozesses der Zellkultur leicht genotypische und phänotypische Veränderungen erwerben6. Körperflüssigkeiten wie Plasma könnten eine attraktive Quelle für die Entdeckung von Biomarkern sein; Aufgrund der sehr reichhaltigen Proteine und des dynamischen Bereichs der Proteinkonzentration ist die Plasmaproteomik jedoch etwas anspruchsvoller7. Hier können Peptide, die aus den am häufigsten vorkommenden Proteinen stammen, diejenigen unterdrücken, die von den wenig reichlich vorhandenen Proteinen abgeleitet sind, auch wenn das Masse-Ladungs-Verhältnis gleichist 6. Obwohl es in den letzten Jahren Fortschritte bei den Depletions- und Fraktionierungstechnologien gegeben hat, bleibt eine gute Abdeckung immer noch eine große Einschränkung der Plasmaproteomik8,9. Die Verwendung von Geweben zur proteomischen Untersuchung der Krankheitsbiologie wird bevorzugt, da Gewebeproben am nahesten zu den Krankheitsstellen sind und hohe physiologische und pathologische Informationen bieten, um bessere Einblicke in die Krankheitsbiologie zu geben10,11.
In diesem Manuskript haben wir ein vereinfachtes Protokoll für die quantitative Proteomik von Gewebeproben bereitgestellt. Für die Gewebelysatzubereitung haben wir einen Puffer mit 8 M Harnstoff verwendet, da dieser Puffer mit massenspektrometrischen Untersuchungen kompatibel ist. Es ist jedoch zwingend erforderlich, die Peptide zu reinigen, um Salze zu entfernen, bevor sie in das Massenspektrometer injiziert werden. Ein wichtiger Punkt, an den Sie sich erinnern sollten, ist, die Harnstoffkonzentration auf weniger als 1 M zu reduzieren, bevor Trypsin für die Proteinverdauung hinzugefügt wird, da Trypsin bei einer Harnstoffkonzentration von 8 M eine geringe Aktivität aufweist. Wir haben zwei Ansätze der quantitativen globalen Proteomik erläutert: die markierungsbasierte Quantifizierung mit iTRAQ (isobare Tags für relative und absolute Quantifizierung) und die markierungsfreie Quantifizierung (LFQ). Die iTRAQ-basierte quantitative Proteomik wird hauptsächlich zum Vergleich mehrerer Proben verwendet, die sich in ihrem biologischen Zustand (z. B. normale versus Krankheit oder behandelte Proben) auskennen. Der Ansatz verwendet isobare Reagenzien, um die N-terminalen primären Amine von Peptiden12zu kennzeichnen. Die iTRAQ-Reagenzien enthalten eine N-Methylpipepasin-Reportergruppe, eine Balancer-Gruppe und eine N-Hydroxysuccinimidester-Gruppe, die mit N-terminalen primären Aminen von Peptiden reagiert13. Verdaute Peptide aus jeder Erkrankung werden mit einem bestimmten iTRAQ-Reagenz markiert. Nach der Markierung wird die Reaktion gestoppt und markierte Peptide aus verschiedenen Bedingungen in einem einzigen Röhrchen zusammengefasst. Dieses kombinierte Probengemisch wird mit einem Massenspektrometer zur Identifizierung und Quantifizierung analysiert. Nach der MS/MS-Analyse werden Reporterionenfragmente mit niedermolekularen Massen erzeugt und die Ionendichten dieser Reporterionen für die Quantifizierung der Proteine verwendet.
Ein weiterer Ansatz, die markierungsfreie Quantifizierung, wird verwendet, um die relative Anzahl von Proteinen in komplexen Proben zu bestimmen, ohne Peptide mit stabilen Isotopen zu markieren.
Protokoll
Diese Studie wurde von institutionellen Prüfungsausschüssen und der Ethikkommission des Indian Institute of Technology Bombay (IITB-IEC/2016/026) geprüft und genehmigt. Die Patienten/Teilnehmer gaben ihre schriftliche Zustimmung zur Teilnahme an dieser Studie.
1. Gewebelysatpräparat
HINWEIS: Führen Sie alle folgenden Schritte auf dem Eis aus, um die Proteasen inaktiv zu halten. Stellen Sie sicher, dass die Skalpelle und alle verwendeten Röhrchen steril sind, um Kreuzkontaminationen zu vermeiden.
- Nehmen Sie ~ 30 mg Gewebe in einem Perlenschlagröhrchen, fügen Sie 200 μL 1x Phosphatpuffersalzlösung (PBS) hinzu und wirbeln Sie es ein.
HINWEIS: In dieser Studie wurden frische gefrorene menschliche Hirntumorgewebe für die Lysatzubereitung genommen. Das Protokoll kann für jedes frische gefrorene Gewebe mit einigen Änderungen in Abhängigkeit von der Art des Gewebes (Weich- oder Hartgewebe) und der zellulären Komplexität des Gewebes verwendet werden. - Danach drehen Sie den Schlauch, um das Gewebe abzusetzen, und entfernen Sie das PBS vorsichtig mit einer Pipette. Führen Sie eine weitere PBS-Wäsche durch, wenn noch Blutspuren im Gewebe vorhanden sind.
- Fügen Sie 300 μL Harnstofflysepuffer (8 M Harnstoff, 50 mM Tris pH 8,0, 75 mM NaCl, 1 mMMgCl2)und Protease-Inhibitor-Cocktail (PIC) gemäß dem Protokoll des Herstellers hinzu.
HINWEIS: Das Volumen des Lysepuffers sollte ausreichen, um das Gewebe während des Beschallungsprozesses zu schleifen und das extrahierte Zu aussetzen. Zu wenig Lysepuffer kann zu einer ineffizienten Gewebelyse führen, während zu viel Lysepuffer das Proteinlysat verdünnt. - Legen Sie das Röhrchen auf Eis und beschallen Sie das Gewebe mit einer Amplitude von 40% für 2,5 min mit Pulszyklen von 5 s (EIN / AUS).
- Zirkoniumperlen in die Röhrchen geben und das Gewebe mit einem Perlenschläger für 90 s mit 5 min Inkubation auf Eis homogenisieren. Wiederholen Sie diesen Schritt zweimal.
- Sobald das Gewebe ausreichend homogenisiert ist, inkubieren Sie das Röhrchen für 10 Minuten auf Eis.
- Nach der Inkubation zentrifugieren Sie die Probe bei 6.018 x g für 15 min bei 4 °C, um die Zellreste vom Überstand zu trennen.
- Den Überstand in der frisch beschrifteten Tube sammeln und bis zur weiteren Verwendung bei -80 °C aliquots lagern.
2. Proteinquantifizierung und Qualitätskontrolle von Gewebelysaten
- Quantifizieren Sie die Proteinkonzentration im Gewebelysat mit Bradfords Reagenz, wie in der Ergänzungsdatei 1beschrieben.
- Nach der Proteinquantifizierung lassen Sie 10 μg Gewebelysat auf einem 12% SDS-PAGE-Gel laufen, um die Qualität des Lysats zu überprüfen.
HINWEIS: Eine weitere Weiterverarbeitung darf nur für die Lysate durchgeführt werden, die die Qualitätsprüfungen durchführen.
3. Enzymatische Verdauung von Proteinen
HINWEIS: Die Schritte für den enzymatischen Aufschluss sind in Abbildung 1a dargestellt.
- Für die Verdauung nehmen Sie 50 μg Proteine und fügen ddH2O hinzu, um das Volumen auf 20 μL aufzugleichen.
- Bereiten Sie nun 20 mM Tris (2-Carboxyethyl) Phosphin (TCEP) aus dem Stamm (0,5 M TCEP) vor, indem Sie 0,8 μL aus dem Stamm zum Proteinlysat hinzufügen, um die Disulfidbindungen in den Proteinen zu reduzieren und die Probe bei 37 °C für 60 min zu inkubieren.
- 40 mM Iodoacetamid (IAA)in ddH2 O vorbereiten und 1,6 μL hinzufügen, um die reduzierten Cysteinreste zu alkylieren. Im Dunkeln 10 min bei Raumtemperatur inkubieren.
- Verdünnungspuffer mit 25 mM Tris pH 8,0 und 1 mM CaCl2 im Verhältnis 1:8 hinzufügen, um die Harnstoffkonzentration in der Probe auf weniger als 1 M zu verdünnen. Überprüfen Sie an dieser Stelle den pH-Wert.
HINWEIS: Wenn Sie Trypsin als Verdauungsenzym verwenden, stellen Sie sicher, dass die Harnstoffkonzentration weniger als 1 M beträgt. - Um die Verdauung durchzuführen, fügen Sie Trypsin in einem Enzym/Substrat-Verhältnis von 1:50 hinzu. Die Röhrchen bei 37 °C in einem Schütteltrockenbad für 16 h zur Verdauung über Nacht inkubieren.
HINWEIS: Das Trypsin-Enzym ist eine hochreaktive Protease, die zur Selbstverdauung neigt. Achten Sie besonders darauf und führen Sie die Zugabe von Trypsin schnell über das Eis durch. - Nach 16 h Inkubation die verdauten Peptide in einem Vakuumkonzentrator trocknen.
4. Entsalzung von verdauten Peptiden
HINWEIS: Um die Entsalzung von Peptiden durchzuführen, verwenden Sie C18-Stufenspitzen.
- Aktivieren Sie die C18-Stufenspitze durch Zugabe von 50 μL Methanol. Zentrifugieren Sie die Spitze bei 1.000 x g für 2 min bei RT. Entsorgen Sie das am Boden des Röhrchens gesammelte Filtrat. Wiederholen Sie diesen Vorgang zweimal.
- Fügen Sie 50 μL Acetonitril in 0,1% Ameisensäure hinzu, um die Bühnenspitze zu waschen. Zentrifugieren Sie das Röhrchen bei 1.000 x g für 2 min bei RT. Entsorgen Sie das am Boden des Röhrchens gesammelte Filtrat. Wiederholen Sie diesen Schritt zweimal.
- Fügen Sie 50 μL von 0,1% (v/v) FA hinzu, um die Spalte auszugleichen. Führen Sie erneut die Zentrifugation bei 1.000 x g für 2 min bei RT durch und entsorgen Sie das Filtrat.
- Rekonstituieren Sie die getrockneten verdauten Peptide in 50 μL 0,1% Ameisensäure.
HINWEIS: Vermeiden Sie luftblasenartige Bildung in den Bühnenspitzen, während Sie die Probe passieren. Die Stufenspitzen sollten während des Zentrifugationsschritts nicht vollständig getrocknet werden, da das Trocknen zu Peptidverlust führen kann. - Die rekonstituierten Peptide in die aktivierte Stufenspitze geben und die Probe durch Zentrifugation bei 1.000 x g für 2 min durch die Stufenspitze leiten. Wiederholen Sie diesen Schritt mindestens viermal. Lagern Sie den Durchfluss bei 4 °C.
- Um die Probe zu waschen, fügen Sie 50 μL 0,1% (v/v) Ameisensäure hinzu. Wiederholen Sie den Zentrifugationsschritt und entsorgen Sie das Filtrat.
- Für die Elution von Peptiden 50 μL 40% (v/v) ACN in 0,1% Ameisensäure (v/v) hinzufügen und durch Zentrifugation durch die Stufenspitze leiten. Sammeln Sie das Filtrat in einem frischen Röhrchen. Wiederholen Sie den Schritt mit 50% und 60% ACN in 0,1% Ameisensäure und sammeln Sie das Filtrat im selben frischen Röhrchen.
- Trocknen Sie die entsalzten Peptide, die in der frischen Tube gesammelt wurden, mit einem Vakuumkonzentrator.
HINWEIS: Die getrockneten entsalzten Peptide sind bereit für die Injektion, oder es kann bei -20 °C für 6 Monate gelagert werden. Zur Langzeitlagerung (>6 Monate) lagern Sie die Peptide bei -80 °C.
5. Quantifizierung entsalzter Peptide
- Rekonstituieren Sie die getrockneten entsalzten Peptide in 0,1% FA.
- Wischen Sie die photometrische Messplatte mit fusselfreiem Gewebe mit 70% Ethanol ab.
- Verwenden Sie 2 μL von 0,1% FA, um den Rohling einzustellen.
- Fügen Sie 2 μL rekonstituierte Proben in Replikaten auf die Platte hinzu.
- Legen Sie die Platte in das Spektralphotometer und messen Sie die Absorption bei 205 nm und 280 nm.
- Berechnen Sie die molare Absorptionsfähigkeit (ε) mit der folgenden Formel:
ε = 27 / [1 - 3,85 * A280 / A205]
HINWEIS: Die molare Absorptionsfähigkeit (ε) ist ein Maß für die Wahrscheinlichkeit des elektronischen Übergangs oder wie gut eine Spezies die bestimmte Wellenlänge der Strahlung absorbiert, die auf sie eintrifft. Der Wert der ε sollte im Bereich von 31 ml mg-1cm-1 bis 33 ml mg-1cm-1 liegen. Wenn der Wert nicht in den Bereich fällt, bedeutet dies, dass die Proben nicht ordnungsgemäß verdaut werden. - Berechnen Sie die Peptidkonzentration in μg/μL mit der folgenden Formel:
Peptidkonzentration = Netto-OD (205) / 0,051 * ε
6. Markierungsfreie Quantifizierung (LFQ) der verdauten Peptide
HINWEIS: Für eine etikettenfreie Quantifizierung verwenden Sie die in der Zusatzdatei 2 genanntenParameter LC und MS. Eine hohe Abdeckung wurde erhalten, wenn drei biologische Replikate des gleichen Probentyps im Massenspektrometer ausgeführt wurden.
- Aufbau der Flüssigkeitschromatographie
- Nach der Quantifizierung der entsalzten Peptide nehmen Sie 2 μg Peptide in eine Durchstechflasche und machen das Volumen mit 0,1% FA auf 10 μL. Die Konzentration der entsalzten Peptide wird 200 ng / μL sein.
- Öffnen Sie den Auto-Sampler des Flüssigkeitschromatographie-Systems (siehe Materialtabelle)und legen Sie die Durchstechflasche in den Autosampler.
- Verwenden Sie 0,1% (v/v) FA, um die Vorsäule und die Analysespalte auszugleichen.
- Nehmen Sie 1 μg entsalztes verdautes Peptid aus der Durchstechflasche und laden Sie es auf die Säule.
- Stellen Sie den LC-Gradienten entsprechend der Probenkomplexität ein. In diesem Experiment wurde der LC-Gradient für 120 min zur markierungsfreien Quantifizierung der Gewebeproben verwendet.
- MS-Setup: Bevor Sie Proteomik-Assays optimieren, führen Sie eine Qualitätskontrolle des Instruments durch, indem Sie einige Peptide von Rinderserumalbumin (BSA) mit einer beliebigen Software für die Systemeignung überwachen und die Abdeckung von BSA analysieren (Abbildung 2A, B). Die Erfassungsparameter wurden mit der MS-Datenerfassungssoftware in das Gerät eingestellt (siehe Materialtabelle).
- Öffnen Sie die Software, doppelklicken Sie auf Instrument Setup und wählen Sie die Vorlage aus peptides-ID mit Standardparametern aus.
- Legen Sie die MS-Parameter mit Supplementary File 2 fest und speichern Sie sie als neue Methode.
- Öffnen Sie nun die Software, um die Beispieldetails auszufüllen. Doppelklicken Sie auf die Sequenzeinrichtungund geben Sie die Details wie Probentyp, Probenname, Dateispeicherort, Instrumentenmethodendatei, Injektionsvolumen und Position der Probe ein.
- Sobald alle Informationen ausgefüllt sind, wählen Sie die Zeile aus und starten Sie die Option Ausführen.
7. Markierungsbasierte Quantifizierung (iTRAQ) von verdauten Peptiden
HINWEIS: Die etikettenbasierte Quantifizierung kann mit verschiedenen isobaren Labels wie iTRAQ- oder TMT-Reagenzien usw. durchgeführt werden. Hier wurde iTRAQ 4-plex zur Markierung von verdauten Peptiden aus drei Gewebeproben eingesetzt. Das Verfahren der iTRAQ 4-Plex-Kennzeichnung wird im Folgenden erwähnt.
- Markierung von verdauten Peptiden mit iTRAQ-Reagenzien.
HINWEIS: In diesem Experiment werden Peptide aus drei Gewebeproben verwendet. Von jeder Gewebeprobe werden 80 μg verdaute Peptide in vier Röhrchen zur Markierung mit iTRAQ-Reagenzien (114, 115, 116 und 117) entnommen (siehe Ergänzungsdatei 3 für die detaillierten experimentellen Parameter).- Bevor Sie das iTRAQ-Reagenz verwenden, bringen Sie jede Durchstechflasche des Reagenzes auf Raumtemperatur (ca. 5 min). Geben Sie einen kurzen Spin von ca. 30 s, um die Lösung am Boden jeder Durchstechflasche zu bringen.
HINWEIS: Stellen Sie sicher, dass in jeder Durchstechflasche 10-15 μL Lösung vorhanden sein sollte. - Für die iTRAQ-Markierung rekonstituieren Sie die getrockneten Peptide in 20 μL Auflösungspuffer, der im iTRAQ-Markierungskit enthalten ist.
- Rekonstituieren Sie die Etiketten, indem Sie 70 μL Ethanol aus der im Kit enthaltenen Durchstechflasche hinzufügen und die Lösung für 30 s mischen und für 10 s spinnen.
HINWEIS: Es ist ratsam, dass alle Schritte gemäß den Anweisungen des Herstellers ausgeführt werden. - Fügen Sie die homogen gemischten iTRAQ-Etiketten (114, 115, 116 und 117) zu ihren jeweiligen Röhrchen hinzu, die Peptidproben enthalten, und lassen Sie die Markierungsreaktion stattfinden.
- Mischen Sie die Komponenten jedes Rohrs, indem Sie das Rohr für 30 s vortexen, und drehen Sie dann das Rohr für 10 s, um die Mischung wieder auf den Boden des Rohrs zu bringen.
HINWEIS: Überprüfen Sie den pH-Wert der Lösung mit pH-Papier. Der pH-Wert sollte größer als 8 sein; Wenn nicht, fügen Sie bis zu 10 μL des Auflösungspuffers hinzu, um den pH-Wert einzustellen. - Inkubieren Sie jedes Röhrchen bei Raumtemperatur für 90 min. Am Ende der Reaktion löschen Sie überschüssiges ungebundenes Etikett im Röhrchen durch Zugabe von MS-Wasser.
- Inkubieren Sie die Röhrchen bei Raumtemperatur für 30 min bis 1 h.
- Sobald die Inkubation beendet ist, den gesamten markierten Inhalt in ein einziges Röhrchen geben und die markierten Peptide in einem Vakuumkonzentrator trocknen.
HINWEIS: Ein ähnliches Kennzeichnungsverfahren kann für die TMT-Etikettierung befolgt werden.
- Bevor Sie das iTRAQ-Reagenz verwenden, bringen Sie jede Durchstechflasche des Reagenzes auf Raumtemperatur (ca. 5 min). Geben Sie einen kurzen Spin von ca. 30 s, um die Lösung am Boden jeder Durchstechflasche zu bringen.
- Aufbau der Flüssigkeitschromatographie
- Rekonstituieren Sie die Proben in 0,1% Ameisensäure, öffnen Sie den Autosampler von Nano LC und legen Sie die Proben in den Autosampler. Verwenden Sie die in Zusatzdatei 2 für die LC-Einrichtung genannten Parameter.
- Stellen Sie den LC-Gradienten entsprechend der Komplexität der Probe ein. In diesem Experiment wurde ein LC-Gradient von 180 min für die markierungsbasierte Quantifizierung (iTRAQ) der Gewebeproben verwendet.
HINWEIS: Bei weniger komplexen Proben kann ein kurzer Gradient die meisten Peptide effizient trennen. Wenn die Probe jedoch sehr komplex ist, verwenden Sie einen längeren Gradienten für eine bessere Trennung der Peptide.
- MS-Setup für iTRAQ-Technik
- Richten Sie alle MS-Parameter für die markierungsbasierte Quantifizierung auf die gleiche Weise ein, wie sie für die markierungsfreie Quantifizierung verwendet wird, mit Ausnahme der Kollisionsenergie, die für die MS/MS-Fragmentierung in der markierungsbasierten Quantifizierung auf 35% festgelegt wurde.
8. Datenanalyse
- Analysieren Sie die Rohdateien (MS/MS-Spektrum), die sie aus dem LC-Massenspektrometer erhalten, mit einer handelsüblichen Analysesoftware (siehe Materialtabelle).
HINWEIS: Die Human Reference Proteome Datenbank von Uniprot (UP000005640), die 71.785 Proteinsequenzen umfasst, wurde verwendet, um Proteinidentitäten mit sequest HT und Mascot (v2.6.0) Suchmaschinen zu erhalten. Die Parameter für die markierungsfreie Quantifizierung und die markierungsbasierte Quantifizierung sind in Ergänzungsdatei 4beschrieben.
Ergebnisse
Wir haben zwei verschiedene Ansätze für die Entdeckungsproteomik verwendet: markierungsfreie und markierungsbasierte Proteomik-Ansätze. Das Proteinprofil von Gewebeproben auf SDS-PAGE zeigte die intakten Proteine und konnte für die proteomische Analyse in Betracht gezogen werden (Abbildung 2A). Die Qualitätskontrolle des Instruments wurde über eine Systemeignungssoftware überwacht und zeigte die tagesmäßige Variation der Geräteleistung auf (Abbildung 2B). Wir beobachteten eine Sequenzabdeckung von 91% der BSA-Probe in 30 Minuten LC-Gradient (Abbildung 2C). Der LC-Gradient wurde mit 500 ng kommerziellem HeLa-Zellverdau optimiert und wir beobachteten 2425 Proteine in einem 2-h-Gradienten im Vergleich zu 1488 Proteinen in einem 1-h-Gradienten (Abbildung 2D). Wir konnten durchschnittlich 2428 Proteine über alle drei technischen Nachbildungen einer Poolgewebeprobe identifizieren (Abbildung 2E).
Die optimierten LC- und MS-Parameter wurden auf drei verschiedene biologische Gewebeproben angewendet (Abbildung 3 und Ergänzungsdatei 2). Das Chromatogramm zeigte eine gute Reproduzierbarkeit zwischen drei verschiedenen biologischen Gewebeproben. Wir identifizierten 2725, 2748 und 2718 quantifizierbare Proteine aus gewebeproben 1, 2 und 3 mit einem markierungsfreien Ansatz. Wir beobachteten, dass 151 Proteine im ersten und zweiten LFQ-Experiment üblich waren, 163 Proteine zwischen dem zweiten und dritten LFQ-Experiment und 187 Proteine zwischen dem ersten und dritten Experiment, während 2190 Proteine in allen drei Gewebeproben üblich waren (Abbildung 4A).
Wir haben das Chromatogramm und die iTRAQ-Etiketten inspiziert und festgestellt, dass es in fast allen MS/MS-Spektren vorhanden ist. Die drei Sets wurden für das iTRAQ-Experiment ausgeführt. Die Anzahl der Proteine, die aus jedem Satz erhalten wurden, war 2455, 2285 bzw. 2307. Es wurde festgestellt, dass 287 Proteine in Probe 1 und Probe 2 häufig sind, 183 Proteine in Probe 2 und Probe 3 und 195 Proteine in Probe 1 und Probe 3. Die Gesamtzahl der Proteine, die in allen drei Proben üblich waren, betrug 1557 (Abbildung 4B).
Wir verglichen die Gesamtpeptidspektralübereinstimmungen (PSMs), Peptidgruppen, Gesamtproteine, Proteingruppen und die Anzahl der Proteine, die nach 1% FDR aus dem LFQ- und iTRAQ-Experiment erhalten wurden (Abbildung 4C).

Abbildung 1: Workflow für die Gewebeproteomik. (A) Die Schritte der Probenverarbeitung zur Vorbereitung von Proben aus Gewebelysat für die MS-Analyse. (B) Schritte zur kennzeichnungsfreien Quantifizierung. (C) Schritte für die etikettenbasierte Quantifizierung. (D) Schritte zur Datenanalyse mit einem Proteomentdecker. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Qualitätskontrolle der Gewebeproben und Reproduzierbarkeit des Instruments. (A) Qualitätskontrolle von Gewebelysaten auf 12% SDS-PAGE (B) Überwachung einiger Peptide von BSA mit Panorama zur Überprüfung der Variabilität des Instruments über die verschiedenen Tage. (C) Die Sequenzabdeckung von BSA in drei technischen Replikaten. (D) Optimierung der LC-Parameter für Gewebeproben. (E) Die Anzahl der Peptidspektral-Übereinstimmungen, Peptide und Proteine in drei verschiedenen biologischen Proben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: LC- und MS-Parameter für die proteomikische Analyse von Gewebeproben. (A,B) Der Flüssigkeitschromatographie-Gradient, der zur Trennung der Peptide für die markierungsfreie Quantifizierung (A) und die markierungsbasierte Quantifizierung (B) der Gewebeprobe verwendet wird. (C) Die MS-Parameter für die markierungsfreie Quantifizierung und die markierungsbasierte Quantifizierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
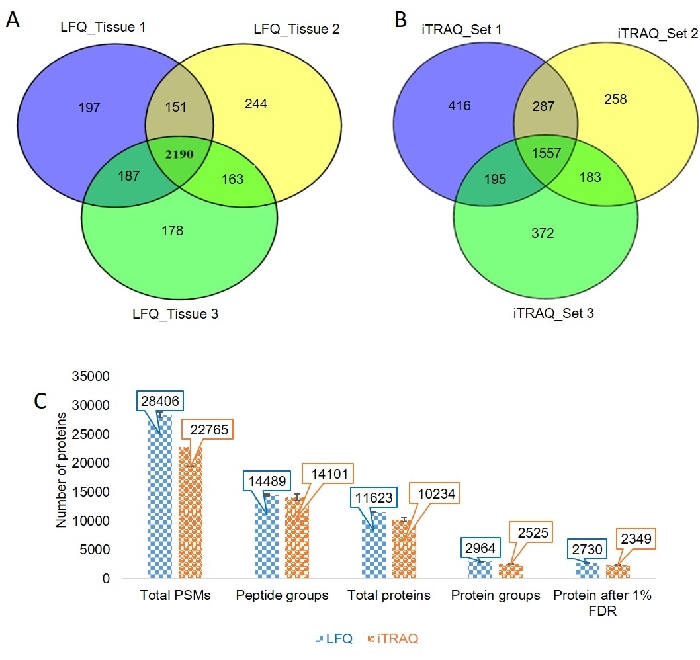
Abbildung 4: Markierungsfreie und markierungsbasierte Quantifizierung der Gewebeprobe. (A) Venn-Diagramm stellt die gemeinsamen und exklusiven Proteine in gewebeproben 1, 2 und 3 des markierungsfreien Experiments dar. (B) Das Venn-Diagramm stellt die gemeinsamen und ausschließlichen Proteine in den Gewebeproben 1, 2 und 3 des markierungsbasierten Experiments dar. (C) Die vergleichende Analyse der Anzahl der Peptidspektralübereinstimmungen (PSMs), Peptidgruppen, Gesamtproteine, Proteingruppen und Proteinzahl nach 1% FDR in markierungsfreier Quantifizierung (LFQ) und markierungsbasiertem Experiment (iTRAQ). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Dateien. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Gewebeproteomik biologischer Proben ermöglicht es uns, neue potenzielle Biomarker zu erforschen, die mit verschiedenen Stadien des Krankheitsverlaufs verbunden sind. Es erklärt auch den Mechanismus der Signalübertragung und die mit dem Fortschreiten der Krankheit verbundenen Wege. Das beschriebene Protokoll für die quantitative Gewebeproteomik-Analyse liefert reproduzierbare gute Abdeckungsdaten. Die meisten Schritte wurden aus den Anweisungen des Herstellers angepasst. Um qualitativ hochwertige Daten zu erhalten, sind die folgenden Schritte am wichtigsten. Daher sollte bei der Durchführung dieser Schritte besondere Vorsicht walten lassen.
Die unvollständige Verdauung von Proteinen und die Kontamination von Keratin können zu einer geringeren Abdeckung von Proteinen (n < 1000) führten, wodurch sich dies auf das Gesamtexperiment auswirkte. Der pH-Wert der Proben (pH 8) und die Harnstoffkonzentration in den Proben (weniger als 1 M) gewährleisten einen effizienten Aufschluss von Proteinen. Die Verwendung von frischem Puffer und der vorsichtige Umgang mit Proben verringern die Wahrscheinlichkeit einer Keratinkontamination. iTRAQ-Reagenzien sind extrem teuer und erfordern eine ausgeklügelte MS-Plattform, um die MS / MS durchzuführen, und Software zur Analyse der Daten. Die Proteomik-Experimente sind empfindlich gegenüber Verunreinigungen durch Salze, Peptidquantifizierung und Markierungseffizienz von iTRAQ / TMT-Reagenzien. Stellen Sie vor der MS/MS-Analyse sicher, dass die verdauten Peptide ordnungsgemäß entsalzt werden, um das Hintergrundrauschen in den Daten zu reduzieren. Im Falle der iTRAQ-Technik erzeugt die Fragmentierung des angehängten Tags ein niedermolekulares Massenreporterion, mit dem die Peptide und die Proteine, aus denen sie stammen, relativ quantifiziert werden können, während beim markierungsfreien Ansatz der Bereich unter der Kurve für die Quantifizierung berücksichtigt wird. Um das Vertrauen in die Quantifizierung von Proteinen zu erhöhen, sollten unabhängige Validierungsexperimente, insbesondere MRM/PRM, durchgeführt werden.
Die Analyse von Gewebeproben mit zwei Quantifizierungsmethoden (markierungsfreie und markierungsbasierte Proteomik) wurde beschrieben, um eine gute Abdeckung von Proteinen zu erhalten. Der markierungsfreie quantitative Proteomik-Ansatz bietet mehrere Vorteile für den Einsatz in klinischen Studien. Die Proben werden unabhängig voneinander durchgeführt, was besonders wichtig für Studien ist, die für eine Patientenkohorte durchgeführt werden, da eine große Anzahl von Proben über Ein massenspektrometer analysiert werden muss. Mit einem technischen Replikat wie einem Pool optimierter Peptide kann eine gute Reproduzierbarkeit auch dann sichergestellt werden, wenn die Proben zu unterschiedlichen Zeitpunkten ausgeführt werden. Dieser Ansatz wurde in großen Kohortenstudien wie dem CPTAC verwendet, was eine Anstrengung vieler internationaler Gemeinschaften ist14.
Die potenziellen Ziele, die sich aus der Studie ergeben, könnten für die Validierung mit gezielten Proteomik-Ansätzen in Betracht gezogen werden. Wir kommen zu dem Schluss, dass die Projekte, die auf der Analyse von Gewebeproben basieren, stark von den detaillierten Arbeitsabläufen der quantitativen Proteomik profitieren könnten, die in dieser Studie bereitgestellt werden. Die genannten Schritte helfen, die Methode zu optimieren und das Proteom von Gewebeproben abzubilden. Die Auswahl quantitativer proteomischer Techniken kann von der Anzahl der Proben, der Verfügbarkeit von MS-Plattformen und der zu behandelnden biologischen Frage abhängen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Wir danken dem MHRD-UAY-Projekt (UCHHATAR AVISHKAR YOJANA), dem Projekt #34_IITB SS und massfiitB Facility am IIT Bombay, das vom Department of Biotechnology (BT / PR13114 / INF / 22 / 206 / 2015) unterstützt wird, um alle MS-bezogenen Experimente durchzuführen.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acetonitrile (MS grade) | Fisher Scientific | A/0620/21 | |
| Bovine Serum Albumin | HiMedia | TC194-25G | |
| Calcium chloride | Fischer Scienific | BP510-500 | |
| Formic acid (MS grade) | Fisher Scientific | 147930250 | |
| Iodoacetamide | Sigma | 1149-25G | |
| Isopropanol (MS grade) | Fisher Scientific | Q13827 | |
| Magnesium Chloride | Fischer Scienific | BP214-500 | |
| Methanol (MS grade) | Fisher Scientific | A456-4 | |
| MS grade water | Pierce | 51140 | |
| Phosphate Buffer Saline | HiMedia | TL1006-500ML | |
| Protease inhibitor cocktail | Roche Diagnostics | 11873580001 | |
| Sodium Chloride | Merck | DF6D661300 | |
| TCEP | Sigma | 646547 | |
| Tris Base | Merck | 648310 | |
| Trypsin (MS grade) | Pierce | 90058 | |
| Bradford Reagent | Bio-Rad | 5000205 | |
| Urea | Merck | MB1D691237 | |
| Supplies | |||
| Hypersil Gold C18 column | Thermo | 25002-102130 | |
| Micropipettes | Gilson | F167380 | |
| Stage tips | MilliPore | ZTC18M008 | |
| Zirconia/Silica beads | BioSpec products | 11079110z | |
| Equipment | |||
| Bead beater (Homogeniser) | Bertin Minilys | P000673-MLYS0-A | |
| Microplate reader (spectrophotometer) | Thermo | MultiSkan Go | |
| pH meter | Eutech | CyberScan pH 510 | |
| Probe Sonicator | Sonics Materials, Inc | VCX 130 | |
| Shaking Drybath | Thermo | 88880028 | |
| Orbitrap Fusion mass spectrometer | Thermo | FSN 10452 | |
| Nano LC | Thermo | EASY-nLC1200 | |
| Vacuum concentrator | Thermo | Savant ISS 110 | |
| Software | |||
| Proteome Discoverer | Thrermo | Proteome Discoverer 2.2.0.388 |
Referenzen
- Petricoin, E., Wulfkuhle, J., Espina, V., Liotta, L. A. Clinical proteomics: revolutionizing disease detection and patient tailoring therapy. Journal of Proteome Research. 3 (2), 209-217 (2004).
- Geho, D. H., Petricoin, E. F., Liotta, L. A. Blasting into the microworld of tissue proteomics: a new window on cancer. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 10 (3), 825-827 (2004).
- Hashimoto, Y., Greco, T. M., Cristea, I. M. Contribution of mass spectrometry-based proteomics to discoveries in developmental biology. Advances in Experimental Medicine and Biology. 1140, 143-154 (2019).
- Faria, S. S., et al. A timely shift from shotgun to targeted proteomics and how it can be groundbreaking for cancer research. Frontiers in Oncology. 7, 13 (2017).
- Ray, S., et al. Proteomic technologies for the identification of disease biomarkers in serum: advances and challenges ahead. Proteomics. 11 (11), 2139-2161 (2011).
- Chen, E. I., Yates, J. R. Cancer proteomics by quantitative shotgun proteomics. Molecular Oncology. 1 (2), 144-159 (2007).
- Geyer, P. E., Holdt, L. M., Teupser, D., Mann, M. Revisiting biomarker discovery by plasma proteomics. Molecular Systems Biology. 13 (9), 942 (2017).
- Ray, S., et al. Proteomic analysis of Plasmodium falciparum induced alterations in humans from different endemic regions of India to decipher malaria pathogenesis and identify surrogate markers of severity. Journal of Proteomics. 127, 103-113 (2015).
- Ray, S., et al. Clinicopathological analysis and multipronged quantitative proteomics reveal oxidative stress and cytoskeletal proteins as possible markers for severe vivax malaria. Scientific Reports. 6, 24557 (2016).
- Sharma, S., et al. Multipronged quantitative proteomic analyses indicate modulation of various signal transduction pathways in human meningiomas. Proteomics. 15 (2-3), 394-407 (2015).
- Sharma, S., Ray, S., Moiyadi, A., Sridhar, E., Srivastava, S. Quantitative proteomic analysis of meningiomas for the identification of surrogate protein markers. Scientific Reports. 4, 7140 (2014).
- Aslam, B., Basit, M., Nisar, M. A., Khurshid, M., Rasool, M. H. Proteomics: Technologies and their applications. Journal of Chromatographic Science. 55 (2), 182-196 (2017).
- Wiese, S., Reidegeld, K. A., Meyer, H. E., Warscheid, B. Protein labeling by iTRAQ: A new tool for quantitative mass spectrometry in proteome research. Proteomics. 7 (3), 340-350 (2007).
- Rudnick, P. A., et al. A description of the Clinical Proteomic Tumor Analysis Consortium (CPTAC) common data analysis pipeline. Journal of Proteome Research. 15 (3), 1023-1032 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten