Method Article
Determinação da eficiência do inibidor químico contra o crescimento intracelular toxoplasma Gondii usando um ensaio de crescimento baseado em luciferase
Neste Artigo
Resumo
Apresentado aqui é um protocolo para avaliar a eficácia da inibição de compostos químicos contra o crescimento intracelular in vitro de Toxoplasma gondii usando um ensaio de crescimento à base de luciferase. A técnica é usada para confirmar a especificidade da inibição pela exclusão genética do gene alvo correspondente. A inibição do LHVS contra a protease TgCPL é avaliada como um exemplo.
Resumo
Toxoplasma gondii é um patógeno protozoário que afeta amplamente a população humana. Os antibióticos atuais utilizados no tratamento da toxoplasmose clínica são limitados. Além disso, apresentam efeitos colaterais adversos em certos grupos de pessoas. Portanto, a descoberta de novas terapêuticas para toxoplasmose clínica é imperativa. O primeiro passo do novo desenvolvimento de antibióticos é identificar compostos químicos que ausitem alta eficácia na inibição do crescimento de parasitas usando uma estratégia de triagem de alto desempenho. Como um patógeno intracelular obrigatório, o Toxoplasma só pode se replicar dentro das células hospedeiras, o que proíbe o uso de medidas de absorvância óptica como um indicador rápido de crescimento. Apresentado aqui é um protocolo detalhado para um ensaio de crescimento baseado em luciferase. Como exemplo, este método é usado para calcular o tempo de duplicação de parasitas toxoplasma do tipo selvagem e medir a eficácia do crescimento intracelular de morfolinurea-leucil-leucyl-vinil sulfone (LHVS, um composto de protease cisteína) em relação à inibição do crescimento intracelular parasitário. Também é descrito um protocolo de exclusão genética baseado em CRISPR-Cas9 no Toxoplasma usando regiões homólogas de 50 bp para recombinação dependente de homologia (HDR). Ao quantificar as eficácias de inibição do LHVS em tipos selvagens e tgCPL (Toxoplasma cathepsin L-like protease)-deficientes, é mostrado que o LHVS inibe o crescimento de parasitas do tipo selvagem de forma mais eficiente do que o crescimento de Δcpl, sugerindo que o TgCPL é um alvo ao que o LHVS se liga no Toxoplasma. A alta sensibilidade e o fácil funcionamento deste ensaio de crescimento à base de luciferase tornam-no adequado para monitorar a proliferação do Toxoplasma e avaliar a eficácia da droga de forma de alta produtividade.
Introdução
Toxoplasma gondii é um parasita intracelular altamente bem sucedido que infecta aproximadamente um terço da população humana. Sua alta taxa de transmissão é predominantemente devido às suas diversas rotas de transmissão, incluindo consumo de carne mal cozida, exposição a reservatórios de mamíferos e transmissão congênita durante o nascimento. T. gondii causa principalmente infecções oportunistas que podem levar à morbidade e mortalidade graves em indivíduos imunocomprometidos1,2,3,4,5,6. Os antibióticos atualmente utilizados no tratamento da toxoplasmose aguda são particularmente ineficientes no tratamento de infecções congênitas e latentes e causam reações severas em alguns indivíduos3,7,8. Assim, existe uma necessidade urgente de identificar novas terapêuticas. Entender as diferenças nos processos subcelulares dentro do Toxoplasma e seu hospedeiro ajudará a identificar potenciais alvos de drogas. Portanto, técnicas eficientes e convenientes de manipulação do genoma são necessárias para estudar os papéis dos genes individuais dentro do Toxoplasma. Além disso, o Toxoplasma pertence ao filo Apicomplexa, que inclui vários outros patógenos humanos significativos, como Plasmodium spp. e Cryptosporidium spp. Assim, o Toxoplasma pode ser usado como um organismo modelo para ajudar a estudar a biologia básica em outros parasitas apicomplexos.
Para identificar novos antibióticos contra patógenos microbianos, a triagem de alto desempenho de uma biblioteca de compostos químicos é inicialmente realizada para determinar sua eficácia na repressão do crescimento microbiano. Até agora, vários ensaios de crescimento baseados em microplacas foram desenvolvidos para medir o crescimento intracelular de T. gondii (ou seja, quantificação radioativa baseadaem 3 H-uracil9, detecção quantitativa de parasitas baseados em ELISA usando anticorpos t. gondii-específicos10,11, medição baseada em proteína de repórter usando β-galactosidase ou YFP expressante toxoplasma cepas12,13, e um ensaio de imagem de alto conteúdo recentemente desenvolvido14).
Todas essas estratégias individuais têm vantagens únicas; no entanto, certas limitações também restringem suas aplicações. Por exemplo, uma vez que o Toxoplasma só pode se replicar dentro de células animais nucleadas, a autofluorescência e a ligação não específica de anticorpos anti-T.gondii às células hospedeiras causam interferência em medidas baseadas em fluorescência. Além disso, o uso de isótopos radioativos requer conformidade especial de segurança e potenciais problemas de segurança. Alguns desses ensaios são mais adequados para avaliar o crescimento em um único ponto de tempo, em vez de um monitoramento contínuo do crescimento.
Apresentado aqui é um protocolo baseado em luciferase para a quantificação do crescimento intracelular do Toxoplasma. Em um estudo anterior, o gene da luciferase de NanoLuc foi clonado sob o promotor da tubbulina Toxoplasma, e esta construção de expressão de luciferase foi transfeccionada em parasitas do tipo selvagem (RHΔku80Δhxg) para criar um RHΔku80Δhxg::NLuc strain (referido como RHΔku80::NLuc a seguir)15. Essa cepa serviu como a cepa parental para determinação do crescimento intracelular e exclusão genética neste estudo. Utilizando o RHΔku80:: A cepade NLuc, o crescimento de parasitas em fibroblastos de prepúcio humano (HFFs) foi monitorado durante um período de 96 h pós-infecção para calcular o tempo de duplicação do parasita.
Além disso, a eficácia da inibição do LHVS contra o crescimento de parasitas pode ser determinada plotando as taxas de crescimento do Toxoplasma contra concentrações de LHVS seriais para identificar o valor ic50. A literatura anterior relatou que o TgCPL é um dos principais alvos de LHVS em parasitas e que o tratamento com LHVS diminui o desenvolvimento de infecções agudas e crônicas de Toxoplasma 16,,17,18,19. Além disso, RHΔku80::NLuc foi usado como a cepa parental para modificação do genoma para gerar uma cepa tgCPL-deficiente(RHΔku80Δcpl::NLuc), e a inibição do LHVS foi medida contra este mutante. Ao observar uma mudança de valores de IC50 para LHVS nos parasitas deficientes tgCPLem comparação com a cepa WT, foi validado que o TgCPL é alvo do LHVS in vivo.
Neste protocolo, RHΔku80::NLuc é usado como a cepa parental, que não tem um caminho de junção final não homólogo eficiente (NHEJ), facilitando assim a recombinação dupla mente dependente da homologia (HDR)20,21. Além disso, as regiões homólogas de 50 bp são flanqueadas em ambas as extremidades de um de resistência a drogas por PCR. O produto PCR serve como um modelo de reparo para remover todo o lócus genético via HDR usando ferramentas de edição de genoma baseadas em CRISPR-Cas9. Tais regiões homólogos curtas podem ser facilmente incorporadas em primers, fornecendo uma estratégia conveniente para a produção do modelo de reparo. Este protocolo pode ser modificado para realizar a exclusão genética universal e a marcação endógena genética.
Por exemplo, em nossa publicação mais recente, três genes protease, TgCPL, TgCPB (Toxoplasma cathepsin B-like protease) e TgSUB1 (Toxoplasma subtilisin-like protease 1), foram geneticamente ablados em TgCRT (Toxoplasma cloroquina-resistência transporte)-deficientes parasitas usando este método15. Além disso, tgAMN (um aminopeptidase putativo N [TgAMN, TGGT1_221310]) foi marcado endógenamente15. O laboratório de Lourido também relatou o uso de regiões homólogos curtas na faixa de 40-43 bp para a introdução de mutação genética direcionada ao local e marcação de genes endógenos no genoma de Toxoplasma usando um método semelhante22. Essas modificações de genoma bem sucedidas sugerem que uma região homóloga de 40-50 bp é suficiente para uma recombinação eficiente de DNA na cepa tgKU80-deficiente, o que simplifica muito a manipulação do genoma em Toxoplasma gondii.
Protocolo
O toxoplasma gondii é categorizado no Grupo de Risco 2 e deve ser manuseado em um Nível de Biossegurança 2 (BSL-2). O protocolo foi revisado e aprovado pelo Comitê Institucional de Biossegurança da Universidade Clemson.
1. Ensaio de crescimento de toxoplasma baseado em luciferase
- Fibroblastos de prepúcio (HFFs) 1 semana antes da inoculação do parasita para garantir que as células hospedeiras sejam totalmente confluentes. Realize um ensaio simulado em uma placa transparente para garantir que os parasitas permaneçam intracelulares durante todo o período de avaliação.
NOTA: Aqui, o ensaio é conduzido em 96 microplacas de poço. De acordo com as necessidades experimentais, pode ser dimensionado até 384 ou 1536 microplacas de poço. - Passe os parasitas do Toxoplasma em HFFs confluentes 2 dias antes do uso, transferindo ~0,3-0,4 mL de parasitas totalmente lised em um frasco T25. Incubar células hospedeiras infectadas a 37 °C com 5% de CO2 durante 2 dias.
- Seringa 5 mL de parasitas recém-lised através de uma agulha de segurança de 21 G 5x para liberar parasitas intracelulares, em seguida, passar através de um filtro de 3 μm para remover detritos celulares hospedeiros. Enxágüe os parasitas residuais do frasco usando 7 mL de fenol livre de fogo D10, depois passe pelo filtro novamente.
- Parasitas de centrífuga a 1000 x g por 10 min à temperatura ambiente (RT). Despeje o sobrenadante e resuspenda a pelota em 10 mL de mídia D10 sem fenol.
- Conte parasitas usando um hemocímetro para determinar a concentração.
- Diluir parasitas para 1 x 104 parasitas/mL para a cepa do tipo selvagem (WT). Para cepas de parasitas deficientes de crescimento, aumente a concentração em conformidade para observar um aumento significativo nos sinais de luciferase.
- Aspire a mídia cuidadosamente de 96 microplacas de poço pré-semeadas com HFFs e inocular 150 μL de ressuspensão de parasitas em poços em um formato de três colunas e cinco linhas, o que representa três réplicas técnicas e cinco pontos de tempo.
- Incubar a microplaca a 37 °C e 5% CO2 por 4 h.
- Aspire a mídia cuidadosamente dos poços para remover parasitas não invadidos, em seguida, encha os poços com mídia rt fenol vermelho-livre em cada linha (exceto para a primeira linha).
- Misture volumes iguais de PBS e 2x tampão de ensaio de luciferase e diluir o substrato da luciferase para 12,5 μM.
- Adicione 100 μL de substrato de luciferase diluída em cada poço da linha superior. Incubar as microplacas em RT por 10 min para permitir que as células se esimmentem completamente.
- Meça a atividade da luciferase usando um leitor de microplacas. As configurações do leitor de placas estão listadas na Tabela 1. Cada leitura representa o número inicial de parasitas invadidos a 4h após a infecção.
- Repita as etapas 1.9-1.12 para cada linha a cada 24 h por 4 dias sem alterar o meio. Essas leituras refletem o número total de parasitas replicados às 24h, 48h, 72h e 96h pós-infecção.
- Calcule as leituras médias em cada ponto de tempo e divida-as pelas leituras médias em 4h para determinar as mudanças de dobra no crescimento do parasita ao longo do tempo.
- Plote os dados usando o software de gráficos. Uma tabela representativa de leitura de crescimento e parcelas de RHΔku80:: Os parasitasda NLuc são mostrados na Figura 1A,B.
- Para calcular o tempo de duplicação, plote os valores de log2 de alterações dobradas nos pontos de tempo individuais ao longo do tempo de incubação. Use uma função de regressão linear para calcular a inclinação, o que representa o tempo de duplicação de cada cepa(Figura 1A,C).
2. Avaliação da eficácia da inibição de compostos químicos contra o crescimento do Toxoplasma
NOTA: Aqui, a avaliação da inibição do LHVS no crescimento do Toxoplasma é apresentada como exemplo. Oito concentrações diferentes de LHVS são testadas, e três réplicas técnicas são realizadas para cada uma das três réplicas biológicas para ambos RHΔku80::NLuc e RHΔku80Δcpl::NLuc estirpes.
- Antes da infecção por parasitas, as sementes hfFs para 96 microplacas de poço no formato de três linhas e nove colunas para uma réplica biológica por composto por cepa. As células hospedeiras poderão crescer por pelo menos 7 dias antes do uso.
- Pass RHΔku80::NLuc e RHΔku80Δcpl::Parasitas NLuc por 2 dias antes do uso. Siga as etapas 1.2-1.6 para purificação e quantificação de parasitas. Ressuspensão de parasitas em mídia fenol red-free em 1 x 104 parasitas/mL.
- Mídia aspirada de uma placa contendo HFFs confluentes e inocular cada poço com 150 μL de ressuspensão de parasitas. Incubar a microplaca a 37 °C e 5% CO2 por 4 h.
- Prepare lHVS em oito concentrações diferentes em um reservatório de 12 poços por diluição serial. Geralmente, as concentrações são diminuídas em três vezes de forma serial de diluição.
NOTA: A menor concentração é reduzida em 6.561 vezes em relação à maior concentração. A mudança dobrada da diluição pode ser ajustada de acordo com diferentes propriedades de compostos individuais. - A 4h pós-infecção, aspiram a mídia para remover parasitas não invadidos e encher cada poço das colunas 2-9 com 150 μL de mídia complementada com LHVS em diferentes concentrações. Deixe a primeira coluna cheia de meio regular para servir como um controle não tratado.
- Incubar a microplaca a 37 °C e 5% CO2 por mais 96 h.
- Realizar as etapas 1.9-1.11 e medir a atividade da luciferase de poços individuais.
- Média das atividades de luciferase de três réplicas técnicas de poços de cada concentração de LHVS individual.
- Divida a atividade média da luciferase para cada concentração de LHVS pela atividade média de luciferase derivada de parasitas não tratados para calcular a atividade normalizada da luciferase em percentagem.
- Plote as atividades normalizadas de luciferase contra as concentrações individuais de LHVS usando software de gráfico(Figura 2). A inibição da pirimetatina contra o crescimento de parasitas também é medida como um controle. Pirimetana é um antibiótico clínico usado para tratar toxoplasmose aguda inibindo o metabolismo do ácido fólico no Toxoplasma.
- Calcular os valores de IC50 para compostos individuais usando o método incorporado no software de gráfico, resposta normalizada versus [inibidor], sob o programa de regressão "dose-resposta-inibição". O IC50 é calculado utilizando a seguinte fórmula:
Y = 100/(1 + X/IC50)
Onde: Y representa atividades normalizadas de luciferase de células infectadas sob diferentes concentrações de inibidor, e X representa concentrações individuais de inibidor.
3. Exclusão genética baseada em CRISPR-Cas9 em parasitas toxoplasma
- Geração de um guia de expressão de construção plasmídea RNA (sgRNA) e Cas9 para excluir um gene de interesse
- Vá para www.ToxoDB.org e recupere toda a seqüência de codificação genética, incluindo introns e éons, juntamente com 1,5 kb 5'-UTRs e 3'-UTRs (regiões não traduzidas).
NOTA: Aqui, o TgCPL (TGGT1_321530) é alvo como um exemplo representativo. - Copie a seqüência TgCPL recuperada no software de análise de seqüência (consulte Tabela de Materiais para o nome e a versão) e rotule as regiões de 5'e 3'-UTR.
- Selecione o ícone Ferramentas na barra de menu superior e selecione Clonagem | Encontre sites CRISPR.
- Escolha 3'(Cas9)' para a localização do site pam e selecione a pasta contendo a seqüência de genoma toxoplasma na seção de pontuação de especificidade. Deixe o resto das configurações como padrão.
- Escolha um sgRNA com os seguintes dois critérios: 1) mostrando uma pontuação de alta especificidade, geralmente >98%, e 2) sem um G seguindo o NGG, uma seqüência de protospacer adjacente (PAM). O sgRNA selecionado geralmente está localizado em locais próximos ao início e param códons do gene de interesse.
- Copie a seqüência do sgRNA selecionado e cole-a no modelo de cartilha a seguir.

A parte em vermelho representa a seqüência de sgRNA TgCPL selecionada. Pode ser substituído por diferentes sgRNAs para vários genes de interesse.
NOTA: Se o sgRNA selecionado não começar com G, adicione G no início do sgRNA para ajudar a melhorar sua expressão. - Realizar uma reação pcr para modificar o sgRNA de expressão plasmid pré-existente (Figura 3A) que tem como alvo toxoplasma uracil phosphoribosyltransferase (TgUPRT) gene23 usando uma pré-mistura pcr com as configurações fornecidas na Tabela 2.
- Execute o produto PCR em um gel de agarose para confirmar a amplificação bem sucedida. Espera-se que um produto PCR de 10 kb seja amplificado(Figura 3B).
- Extrair o produto PCR usando um kit de extração de gel de DNA e circularizá-lo usando um kit de mutagênese direcionado ao local. Consulte a Tabela 3 para obter a receita. Incubar a reação por 10-20 min no RT.
- Transforme o produto PCR circularizado em E. coli e escolha 10 clones para posterior verificação da incorporação do sgRNA projetado.
- Cresça dois clones e extraia plasmídeos. Corte os plasmídeos purificados com BamHI e EcoRV. Os plasmídeos candidatos renderão duas bandas a 2,4 kb e 7,2 kb(Figura 3C).
- Envie os plasmídeos para sequenciamento de Sanger usando primers reversos M13 para confirmar a substituição bem sucedida do TgUPRT sgRNA com o sgRNA projetado(Figura 3D).
- Vá para www.ToxoDB.org e recupere toda a seqüência de codificação genética, incluindo introns e éons, juntamente com 1,5 kb 5'-UTRs e 3'-UTRs (regiões não traduzidas).
- Geração de modelo de reparo para exclusão de genes via mecanismo HDR
- De acordo com os locais de segmentação do sgRNA selecionado, localize 50 bp de 5'-UTRs ou 3'-UTRs do gene-alvo para recombinação dependente de homologia (HDR, ver seção de discussão). A seleção das regiões segue os critérios listados abaixo, dependendo da localização das metas do sgRNA.
- Se o local do decote por Cas9 estiver localizado a montante a partir do códon inicial, selecione o seguinte: uma seqüência de DNA de 50 bps a montante do local do decote como a região do HDR esquerdo, e uma seqüência de DNA de 50 bp a jusante do códon stop como a região direita do HDR.
- Se o local do decote por Cas9 estiver entre os códons de início e parada, selecione o seguinte: uma seqüência de DNA de 50 bps a montante do códon inicial como a região hdr esquerda, e uma seqüência de DNA de 50 bp a jusante do códon stop como a região direita do HDR.
- Se o local do decote por Cas9 estiver localizado a jusante do códon de parada, selecione o seguinte: uma seqüência de DNA de 50 bps a montante do códon inicial como a região do HDR esquerdo, e uma seqüência de DNA de 50 bp a jusante do local do decote como a região direita do HDR.
NOTA: Para o gene TgCPL, o local do decote está localizado entre os códons start e stop. Assim, os primers a seguir são projetados para amplificar o modelo de reparo usando pMDC64 como modelo, que codifica um de resistência à pirimetatina. As seqüências em anneal preto para o plasmídeo pMDC64 para amplificação PCR. As regiões rotuladas em vermelho são seqüências específicas de TgCPLpara recombinação homóloga.
- Realizar PCR usando um pré-mix PCR sob as condições pcr descritas na Tabela 4.
- Execute o produto PCR em um gel de agarose(Figura 3E),seguido de procedimentos de extração de gel e quantificação padrão de ácido nucleico.
NOTA: Se a banda esperada não puder ser amplificada com sucesso, otimize as condições do PCR e/ou os pares de primer do switch.
- De acordo com os locais de segmentação do sgRNA selecionado, localize 50 bp de 5'-UTRs ou 3'-UTRs do gene-alvo para recombinação dependente de homologia (HDR, ver seção de discussão). A seleção das regiões segue os critérios listados abaixo, dependendo da localização das metas do sgRNA.
- Transfecção de toxoplasma
- Pass RHΔku80::Parasitas NLuc por 2 dias em um frasco T25 contendo HFFs confluentes. Um frasco T25 de parasitas totalmente lised é suficiente para duas a três transfecções.
- Os parasitas de purificação de seringas e filtros, conforme descrito na etapa 1.2. Ressuspensa os parasitas no tampão citomix e gire para baixo a 1.000 x g por 10 min no RT.
- Lave parasitas pelleted com 10 mL de tampão citomix e gire os parasitas a 1.000 x g por 10 min em RT.
- Despeje cuidadosamente o sobrenadante e resuspenda os parasitas no mesmo tampão em uma concentração de 1 x 108 parasitas/mL.
- Misture 2 μg de DNA do modelo de reparo com 20 μg dos plasmídeos de expressão sgRNA/Cas9 (razão de massa = 1:5, equivalente a uma razão molar de 1:3). Se o rendimento de amplificação do modelo de reparo for baixo, reduza a entrada de ambas as peças de DNA em conformidade. Um mínimo de 0,5 μg de modelo de reparo pode ser usado.
- Misture 400 μL de ressuspensão de parasitas, DNA e 5 μL de 200 mM ATP/500 mM de glutationa reduzida (GSH) em um tubo de centrífuga de 1,5 mL. Leve o volume total para 500 μL com tampão citomix, se necessário.
- Transfira a mistura de parasitas e DNA para uma cuvette de eletroporação (largura de 4 mm) e realize a eletroporação (tensão de 2 kV, resistência a 50 Ω) usando um aparelho de eletroporação.
- Transfira parasitas eletroportados para um frasco T25 contendo HFFs confluentes em meio D10 fresco. Aplique antibiótico apropriado para seleção de medicamentos após 24 horas.
- Mantenha a pressão seletiva da droga até que o crescimento dos parasitas transgênicos seja estável.
- Purifique o DNA genômico da população de nocaute e verifique a integração do de resistência à pirimetana no lócus TgCPL por PCR. Após verificação, prossiga para a seção 3.4. Caso contrário, realize outra rodada de transfecção de parasitas e seleção de medicamentos. A incapacidade de detectar a integração correta do de resistência à droga geralmente sugere que o gene alvo é essencial ou que o lócus genético não é acessível.
- Clonagem de parasitas nocauteados
- Semente duas microplacas de 96 poços com células HFF e incubar a 37 °C e 5% CO2 por 1 semana antes da clonagem de parasitas.
- Passe ~0,3-0,4 mL da população de parasitas transgênicos em um frasco T25 contendo HFFs confluentes e os plante por 2 dias. Considere passar mais parasitas se o mutante mostrar defeitos de crescimento.
NOTA: Para obter o melhor rendimento e viabilidade, as células hospedeiras são fortemente infectadas pelos parasitas, e a maioria dos parasitas são mantidos no estágio intracelular. - As células hospedeiras infectadas pela seringa e os parasitas recém-purificados com filtro purificam os recém-formados, conforme mencionado na etapa 1.3. Ressuspensa os parasitas no meio D10 e gire-os a 1.000 x g por 10 min no RT.
- Resuspender os parasitas pelleted em 10 mL de meio D10.
- Contagem de parasitas usando um hemocímetro para determinar a concentração de parasitas.
- Realizar uma diluição em duas etapas para levar a concentração a 10 parasitas/mL em meio D10 suplementado com o antibiótico apropriado. Normalmente, a ressuspensão inicial do parasita é diluída em 1.000 vezes, seguida de uma segunda diluição para 10 parasitas/mL.
- Mídia aspirada de 96 microplacas de poços contendo HFFs confluentes e inocular 150 μL de parasitas diluídos em cada poço.
- Incubar placas a 37 °C com 5% de CO2 durante 7 dias sem perturbação para permitir a formação da placa. O período de incubação pode ser maior se os parasitas transgênicos apresentarem defeitos de crescimento.
- Tela as placas usando um microscópio de contraste de fase e marque apenas os poços contendo uma única placa.
- Executar pcr colônia para identificar clones corretos.
- Use as pontas da pipeta para raspar o fundo de cada poço para levantar monocamadas HFF infectadas.
- Pipet 75 μL da resuspensão celular de cada um marcado bem em tubos de microcentrífuge de 1,5 mL.
- Tubos de centrífuga por 10 min em velocidade máxima em RT. Apiratee cuidadosamente o sobrenadante e resuspenda a pelota em 10,25 μL de tampão de líxio contendo tampão de diluição e aditivo de liberação de DNA fornecido no kit(Tabela de Materiais).
- Incubar as amostras por 4 min em RT, depois 2 min a 98 °C. Posteriormente, as amostras podem ser usadas para PCR ou armazenadas a -20 °C até o uso. Três conjuntos de reações pcr são usados para testar a integração do de resistência à droga e a perda do gene de interesse(Figura 4A). Consulte a Tabela 5 para configuração de reação pcr e tabela 6 para as configurações do termocicador.
- Identifique os clones corretos e transfira quatro clones para frascos T25 contendo HFFs confluentes.
- Após clones individuais de células hospedeiras, purifique o DNA genômico para verificação posterior do PCR.
- Se um anticorpo reconhecendo a proteína de interesse estiver disponível, siga um procedimento padrão de imunoblotting para verificar a perda da proteína alvo nos nocautes corretos do Toxoplasma. Imagens representativas para a triagem de um mutante de exclusão TgCPLsão mostradas na Figura 4B,C.
Resultados
A Figura 1 representa um exemplo de uma curva de crescimento para o RHΔku80::Tensão NLuc e o cálculo derivado para o seu tempo de duplicação. Geralmente, o ensaio é realizado em três réplicas técnicas para cada uma das três réplicas biológicas para explicar as variações das leituras da atividade da luciferase. Para calcular a mudança de dobra normalizada do crescimento do parasita, cada leitura a 24-96 h pós-infecção foi dividida pela leitura inicial a 4h pós-infecção, o que reflete a quantidade inicial de parasitas vivos no ensaio(Figura 1A,B). Em termos de determinar o tempo de duplicação do parasita, os valores log2 das mudanças de dobra normalizadas do crescimento de parasitas foram plotados contra cada ponto de tempo. Em seguida, o enredo foi submetido a uma função de regressão linear para obter a inclinação, o que representa o tempo de duplicação (Figura 1C).
As eficácias de inibição do LHVS em cepas decpl tipo selvagem e Δ foram determinadas pela plotagem das atividades de luciferase contra oito concentrações inibidoras na Figura 2. É essencial incluir células infectadas sem tratamento inibidor para normalização das atividades de luciferase crua no ensaio. Além disso, um experimento simulado realizado em uma microplaca clara é necessário para o ensaio para garantir que os parasitas ainda estejam na fase intracelular no final do período de ensaio.
Na Figura 3,são mostradas a geração e validação de um construto de expressão sgRNA direcionado ao TgCPL e à produção de um modelo de reparo para exclusão de TgCPL. O sgRNA de 20 bps correspondente ao gene TgUPRT codificado no plasmídeo original foi mutado para a seqüência de DNA que teve como alvo o gene TgCPL através de mutagênese dirigida ao local baseada em PCR. Para isso, as seqüências de DNA codificadas para os sgRNAs que reconhecem diferentes genes foram projetadas para o primer dianteiro, enquanto o primer reverso foi mantido inalterado para simplificar o design do primer.
A Figura 3A mostra uma região ampliada das seqüências de DNA sgRNA direcionadas ao gene TgUPRT no modelo original plasmídeos, bem como o conjunto de primer usado para a geração do vetor de expressão sgRNA linearizado. A Figura 3B mostra uma imagem representativa em gel do plasmídeo de expressão sgRNA de alvo linearizado. A Figura 3C mostra a restrição da digestão endonuclease do plasmídeo de expressão sgRNA circularizado tgCPL. Um primer reverso M13 foi usado para sequenciar o RNA guia incorporado dentro do vetor de expressão sgRNA gerado para o gene específico. Na Figura 3D,a região de DNA sequenciada foi alinhada ao modelo plasmático para a confirmação de mutagênese bem sucedida. A Figura 3E ilustra as regiões de início e fim do de resistência à pirimetaina, mostrando onde os primers podem anneal para produção do modelo de reparo para exclusão de genes TgCPL. O modelo de reparo foi amplificado por PCR e carregado em um gel de agarose de 1% para verificação de tamanho e extração de gel.
A estratégia global para a geração e triagem de nocautes TgCPL é mostrada na Figura 4. Três conjuntos de primers mostrados na Figura 4A foram usados para tela de parasitas de exclusão TgCPLpara a integração correta de 5'- e 3'-ARMs e exclusão da seqüência de codificação TgCPL. Como mostrado na Figura 4B,geralmente, sete a oito clones são selecionados para triagem inicialmente. A triagem geralmente começa com a verificação da exclusão da seqüência de codificação para o gene de interesse. Isso é seguido pela detecção de 5' e 3'-ARMs, o que ajuda a minimizar o número total de clones a serem rastreados. Uma verificação adicional por imunoblotting exibida na Figura 4C pode ser concluída se um anticorpo reconhecendo a proteína alvo estiver disponível.

Figura 1: Quantificação do crescimento intracelular para parasitas toxoplasma usando um método baseado em luciferase. (A) Leituras de atividade sinuosa de luciferase em um software de planilha. As leituras às 24h, 48h, 72h e 96h pós-infecção foram normalizadas em relação às leituras iniciais a 4h pós-infecção para o cálculo das alterações dobradas no crescimento do parasita. (B) Os dados normalizados foram mediados e plotados. ( C) Os valores log2 das alterações da dobra também foram plotados e submetidos à regressão linear para determinação do tempo de duplicação do parasita. Clique aqui para ver uma versão maior desta figura.
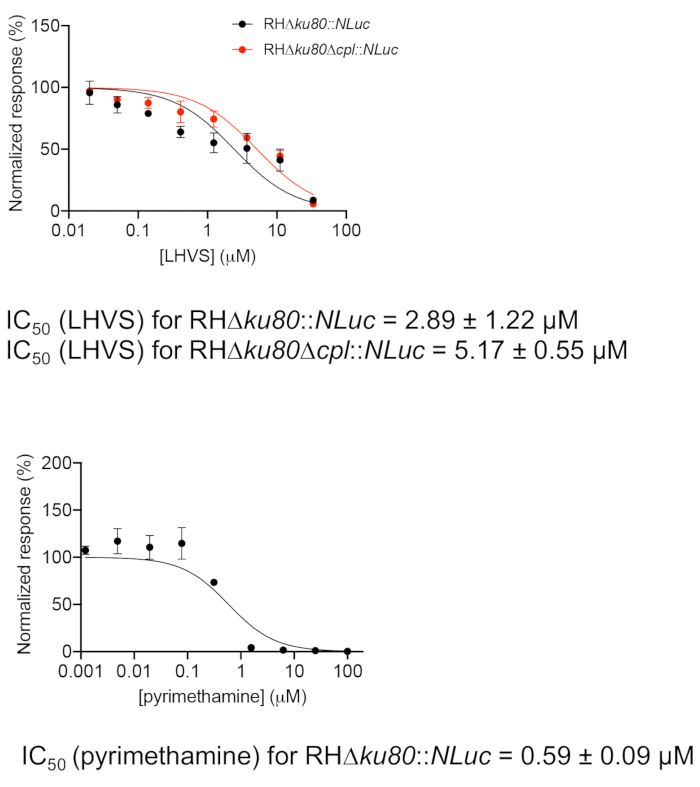
Figura 2: Avaliação da eficácia da inibição de LHVS e pirimetana utilizando o ensaio de crescimento à base de luciferase. Os parasitas foram inoculados em uma microplaca de 96 poços por 4h para permitir a invasão de células hospedeiras. Parasitas não invadidos foram lavados, e a placa foi preenchida com mídia contendo diferentes concentrações de LHVS ou pirimetana e incubada por mais 96 h antes da determinação da atividade da luciferase. As leituras de luciferase medidas para parasitas tratados com concentrações de inibidores individuais foram normalizadas contra o sinal detectado de parasitas não tratados. Os dados foram traçados em um programa de gráficos, e foi realizada uma análise de regressão para determinação do IC50. O ensaio foi repetido em três réplicas biológicas com três réplicas técnicas cada. Os dados representam a média ± SEM, n = 3 réplicas biológicas. Clique aqui para ver uma versão maior desta figura.
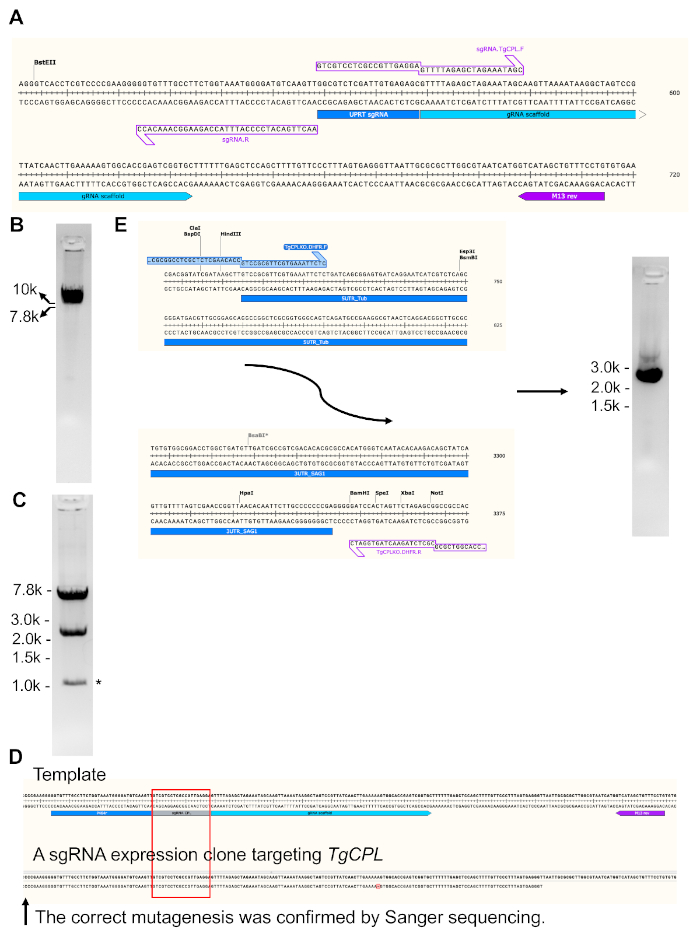
Figura 3: Geração do construto plasmídeo expressando sgRNA direcionado ao TgCPL e produção de um modelo de reparo para exclusão tgCPL. (A) O plasmídeo pSAG1-Cas9-sgRNA-UPRT23 foi modificado por meio de um kit de mutagênese direcionado ao local para substituição do sgRNA direcionado ao gene TgUPRT para TgCPL. A região de codificação sgRNA é ampliada para mostrar áreas para as quais os primers anneal. Após a PCR, o plasmídeo mutado foi linearizado e carregado em um gel de agarose de 1% para verificação de amplificação bem sucedida, seguido pela extração de gel. (B) A imagem em gel do construto de expressão sgRNA linearizado amplificado pelo PCR. ( C) Após a extração em gel, o produto PCR foi circularizado e posteriormente transformado em E. coli. Os clones contendo os plasmídeos esperados foram examinados por restrição de digestão endonuclease e sequenciamento de DNA. Os tamanhos da banda após a digestão do DNA foram de 7,2 bp e 2,4 kb. A banda gerada por decotes não específicos de endonucleases é rotulada por asterisco. (D) O primer reverso M13 rotulado na figura foi usado para sequenciar a região de RNA guia mutada dentro do vetor de expressão sgRNA gerado. A região de DNA seqüenciada foi alinhada ao modelo plasmático para confirmar a mutagênese bem sucedida. (E) Neste estudo, as regiões homólogas de 50 bp correspondentes aos 5'- e 3'-UTRs de TgCPL foram projetadas nos primers para amplificação do modelo de reparo e flanqueadas nas extremidades de 5'- e 3'-extremidades do de resistência à pirimethamina por PCR, respectivamente. A eletroforese do gel de agarose foi utilizada para verificar o tamanho correto do produto PCR antes da extração de gel. O tamanho esperado do modelo de reparo é de ~2,7 kb. Normalmente, 5-6 μg de modelo de reparo podem ser obtidos a partir de 200 μL de reação PCR. Clique aqui para ver uma versão maior desta figura.
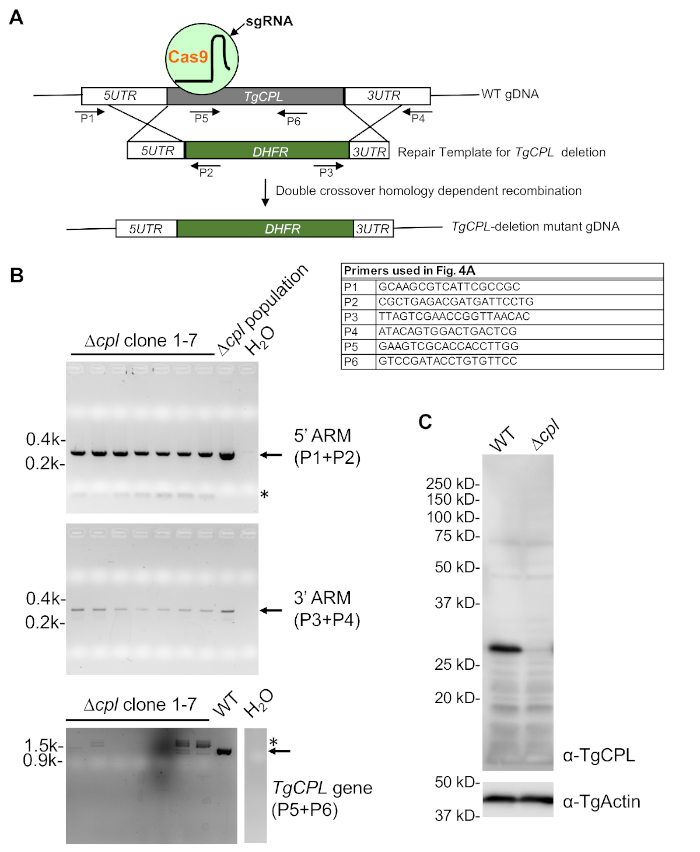
Figura 4: Confirmação de PCR e imunoblotting de parasitas deficientes tgCPL. (A) Um diagrama esquemático que retrata as estratégias gerais de exclusão de TgCPLno Toxoplasma e pcr-based triagem dos clones de nocaute TgCPL corretos. Os primers usados para a triagem são rotulados. (B) A eletroforese do gel de PCR e agarose foi utilizada para selecionar clones contendo a integração correta do de resistência à pirimetana no lócus TgCPL e perda do gene TgCPL. O DNA genômico da populaçãode Δ cpl serviu como um controle positivo para a detecção de 5'e 3'-ARM, enquanto o DNA genômico WT foi usado para a detecção do gene TgCPL como um controle positivo. A água foi usada em vez de modelo de DNA nas reações pcr para servir como um controle negativo. As bandas esperadas são denotadas por setas, enquanto amplificações PCR não específicas são rotuladas por asteriscos. (C) O clone 1 identificado pela triagem pcr foi cultivado na cultura tecidual para preparação de lysate celular e análise de imunoblotting para confirmar a perda de expressão tgCPL no nocaute. TgActin foi usado como controle de carga. Clique aqui para ver uma versão maior desta figura.
| Luciferase: | Extremidade |
| Tempo de integração: | 1 s |
| Conjunto de filtros - Emissão: | Luz total |
| Óptica: | Início |
| Ganhar: | 135 |
| Velocidade de leitura: | Normal |
| Atraso: | 100 ms |
| Altura de leitura: | 4,5 mm |
Tabela 1: Configurações do leitor de microplacas para medição da atividade da luciferase durante o ensaio de crescimento de Toxoplasma baseado em luciferase.
| Desnaturação inicial: | 98 °C por 5 min |
| 25 ciclos de | |
| Desnaturação: | 98 °C para 5 s |
| Recozimento: | 60 °C para 15 s |
| Extensão: | 72 °C por 1 min |
| Extensão final: | 72 °C por 10 min |
Tabela 2: Configurações do termociclador para geração de vetor de expressão sgRNA.
| Amostra | Volume (μl) |
| Produto PCR (10-50 ng) | 1 |
| 2X KLD (quinase, ligase, DpnI) Retol | 5 |
| Mistura de enzimas KLD 10X | 1 |
| Água sem nuclease | 3 |
| Total | 10 |
Tabela 3: Receita de reação para circularização do vetor de expressão sgRNA.
| Desnaturação inicial: | 98 °C por 5 min |
| 35 ciclos de | |
| Desnaturação: | 98 °C para 15 s |
| Recozimento: | 58 °C por 15 s |
| Extensão: | 72 °C para 30 s por kb |
| Extensão final: | 72 °C por 10 min |
Tabela 4: Configuração do termociclador para geração de modelo de reparo.
| Amostra | Volume (μl) |
| DNA genômico total de Toxoplasma | 1 |
| Primer dianteiro (25 μM) | 0.2 |
| Primer reverso (25 μM) | 0.2 |
| 2x pcr master premix | 5 |
| Água sem nuclease | 3.6 |
| Total | 10 |
Tabela 5: Receita de reação colony PCR para triagem de clones únicos de Toxoplasma.
| Desnaturação inicial: | 98 °C por 5 min |
| 35 ciclos de | |
| Desnaturação: | 98 °C para 5 s |
| Recozimento: | 55 - 62 °C para 5 s |
| Extensão: | 72 °C para 20 s por kb |
| Extensão final: | 72 °C por 1 min |
Tabela 6: Configuração do termociclizador para triagem de clones toxoplasma únicos.
| Tampão Cytomix | HEPES de 25 mM, pH 7.6, 120 mM KCl, 10 mM K2HPO4/KH2PO4, 5 mM MgCl2, 0.015 mM CaCl2, e 2 mM EGTA. |
| Meio D10 | DMEM 1X (Corning, Cat #: 10-013-CV), 10 mM HEPES, 10% (v/v) Soro de Bezerro Cósmico (Hyclone, Cat #: SH30087,03), 1 mM piruvato de sódio, 4 mM L-glutamina, 100 unidades/mL de penicilina, e 100 μg/mL de estreptomicina. |
| Fenol meio livre-vermelho | DMEM/ Altamente Modificado (Hyclone, Gato #: SH30284.02), 10 mM HEPES, 10% (v/v) Soro de Bezerro Cósmico (Hyclone, Gato #: SH30087.03), 1 mM piruvato de sódio, 4 mM L-glutamina, 100 unidades/mL de penicilina, e 100 μl de st.reptomycina. |
| 2X NLuc Buffer | 100 mM MES, pH 6.0, 1mM CDTA, 0,5% Tergitol, 0,05% Mazu DF 204, 150 mM KCl, 1 mM DTT, 35 mM Thiourea. |
Tabela Suplementar 1: Receitas para tampões.
Discussão
++Este protocolo descreve um protocolo baseado em luciferase para avaliar o crescimento do Toxoplasma intracelular e avaliar a eficácia da inibição de compostos químicos contra o crescimento de parasitas. Comparado com as estratégias existentes disponíveis para medir o crescimento do Toxoplasma intracelular, este método apresenta alta sensibilidade e especificidade. Enquanto monitora o crescimento de parasitas, recomenda-se um ensaio simulado em uma microplaca clara de 96 poços para confirmar que a cepa testada não prematuramente lise células hospedeiras antes do final do período de avaliação. Caso contrário, as leituras de luminescência não refletirão com precisão o crescimento de parasitas, uma vez que o Toxoplasma só se replica dentro das células hospedeiras.
Observou-se que o corantes vermelhos fenol sacia rapidamente a atividade da luciferase, o que pode resultar em diferenças significativas nas leituras de luciferase entre as réplicas técnicas devido ao atraso nas medições individuais do poço pelo leitor da placa. Portanto, é ideal preparar HFFs em meio fenol sem vermelho antes de semeamento nas 96 microplacas de poço. Além disso, no caso de alta atividade da luciferase, a interferência transversal pode levar a uma variação significativa entre os poços vizinhos que apresentam forte atividade de luciferase. Por isso, recomenda-se colocar uma coluna vazia entre cada cepa.
Especificamente, para o RHΔku80:: A cepaNLuc, 1.500 parasitas são inoculados em cada poço para o ensaio de crescimento. Uma vez que o tempo de duplicação para os parasitas WT Toxoplasma é ~6-8 h24, espera-se que veja um aumento na atividade de luciferase de 8-a 16 vezes a 24 h pós-infecção. No entanto, certas cepas com defeitos de crescimento significativos só produzirão um ligeiro aumento na atividade da luciferase. Portanto, se o inóculo do parasita inicial for baixo, a variação inerente na atividade da luciferase irá mascarar a observação de um aumento da luminescência ao longo do período de crescimento. Assim, recomenda-se vacinar um maior número de parasitas para obter uma mudança de dobra precisa para cepas com deficiências de crescimento.
No protocolo, o projeto de RNA do guia segue as regras gerais utilizadas para a modificação do genoma baseado em CRISPR-Cas9 em células de mamíferos25. Atualmente, muitos tipos de software e plataformas online fornecem serviços para o design de RNA guia em vários organismos, tais como CHOPCHOP26, E-CRISP27e EuPaGDT28. Aqui, o software comercial (Table of Materials) é usado para projetar sgRNA. Comparado com os programas on-line mencionados anteriormente, este software fornece um ambiente local para o design sgRNA. Ele calcula as pontuações de atividade e especificidade para cada candidato sgRNA usando um algoritmo publicado anteriormente25,29.
Tecnicamente, qualquer sgRNA localizado dentro de um gene de interesse que contenha uma pontuação de alta especificidade pode mediar eficientemente o decote do DNA genômico para recombinação dependente da homologia a jusante. Na prática, é preferível o sgRNA visando uma região próxima ao codon de partida ou parada. Genes de interesse podem ser marcados endógenamente com epítopos usando o sgRNA, o que gera uma lacuna dupla mente encalhada no final do gene. A marcação endógena de um gene antes de sua exclusão ajudará a confirmar a perda genética através da detecção de imunoblotting no caso de que um anticorpo contra a proteína de interesse não está disponível. A marcação de epítopos de um gene também pode ajudar a determinar a localização subcelular da proteína de interesse através da microscopia de imunofluorescência. Além disso, se o gene alvo é essencial, o sgRNA reconhecendo a região inicial do gene pode ser usado para a substituição de seu promotor cognato por um promotor responsivo de tetraciclina para gerar um nocaute condicional.
Além disso, o protocolo descreve uma técnica de exclusão genética no Toxoplasma, substituindo o gene TgCPL por um de resistência à pirimetaina. Usando diferentes modelos de plasmídeos codificando outros de resistência a medicamentos, os investigadores podem modificar seqüências de primer para incorporar outros genes de resistência a antibióticos no modelo de reparo via PCR. Além disso, este protocolo pode ser modificado para realizar outras modificações do genoma, como marcação genética endógena, substituição de promotores e mutagênese direcionada ao local. Vale ressaltar que as regiões homólogas utilizadas neste protocolo têm apenas 50 bp de comprimento. Um estudo separado usou com sucesso sequências de DNA homólogo de 40-43 bp para introduzir mutações de nucleotídeos únicos e marcação de epítopos genéticos em parasitas toxoplasma 22. Sequências de DNA homólogos em tão curto comprimento podem ser facilmente incorporadas em primers. Embora não tenhamos avaliado quantitativamente a eficiência do HDR para este comprimento específico da região homóloga, parece que uma região de 40-50 bp é suficiente para uma recombinação eficiente de DNA na cepa Toxoplasma deficiente TgKu80,como evidenciado pela manipulação genética bem sucedida de vários genes recentemente alcançados15,22.
Durante a determinação de eficácia de compostos químicos, se o gene alvo da droga em potencial é essencial, uma comparação de mudanças nos valores de IC50 entre as cepas de tipo selvagem e knockout não é prática. Nesse cenário, é necessário um ensaio que meça diretamente a capacidade dos compostos químicos de inibir a atividade proteica recombinante para avaliar a eficácia e a especificidade dos fármacos. A literatura recente relatou as pontuações de aptidão de genes individuais no Toxoplasma, realizando uma tela CRISPR wide genoma30, que pode servir como um guia para ajudar a avaliar a dificuldade de gerar um mutante de nocaute direto para o gene de interesse.
Em conjunto, o protocolo descreve a conclusão bem sucedida de um ensaio de crescimento intracelular de toxoplasma baseado em luciferase e uma estratégia de avaliação para inibidores químicos contra o crescimento do Toxoplasma. Também é detalhado um protocolo de edição de genoma baseado em CRISPR-Cas9 para exclusão de genes em parasitas toxoplasma, que tem sido amplamente utilizado no campo. Laboratórios individuais podem modificar o protocolo descrito de acordo com as necessidades experimentais, como marcação de genes endógenos, troca de marcadores de seleção de medicamentos e alteração do período de avaliação para o crescimento de parasitas intracelulares.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Os autores gostariam de agradecer aos Drs. Sibley e Carruthers por compartilharem os anticorpos pSAG1-Cas9-sgRNA-TgUPRT e anti-TgCPL e TgActin. Este trabalho foi apoiado pelo fundo Clemson Startup (para Z.D.), Knights Templar Eye Foundation Pediatric Oftalmology Career-Starter Research Grant (para Z.D.), uma bolsa piloto de uma bolsa NIH COBRE P20GM109094 (para Z.D.), e NIH R01AI143707 (para Z.D.). Os financiadores não tiveram papel na concepção do estudo, coleta e análise de dados, decisão de publicação ou elaboração do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
Referências
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados