Method Article
使用基于路西法抹去的生长测定,确定化学抑制剂对细胞内弓形体贡迪生长的抑制效率
摘要
本文介绍的是一种协议,用于使用基于荧光酶的生长测定来评估化合物对弓形体淋酸体内生长的抑制作用。该技术用于通过基因删除相应的靶基因来确认抑制特异性。以LHVS对TgCPL蛋白酶的抑制为例。
摘要
弓形虫是一种原生动物病原体,广泛影响人类。目前用于治疗临床弓形虫病的抗生素是有限的。此外,它们在某些人群中表现出不良的副作用。因此,发现临床弓形虫病的新药势在必行。新型抗生素开发的第一步是使用高通量筛选策略识别在抑制寄生虫生长方面表现出高疗效的化合物。作为一种义务细胞内病原体,弓形虫只能在宿主细胞内复制,这禁止使用光学吸收度测量作为生长的快速指标。此处介绍的是基于荧光酶的生长测定的详细协议。例如,该方法用于计算野生型弓形虫寄生虫的倍增时间,并测量莫诺林-利基-同苯-乙烯磺胺苯基(LHVS,一种半胱氨酸蛋白酶靶向化合物)对抑制寄生虫细胞内生长的疗效。还描述了一个基于CRISPR-Cas9的基因删除协议在弓形体使用50bp同源区域进行同源相关重组(HDR)。通过量化LHVS在野生型和TgCPL(弓形体cathein L样蛋白酶)缺乏寄生虫的抑制作用,表明LHVS抑制野生型寄生虫生长比μcpl生长更有效,这表明TgCPL是LHVS结合到弓形虫的目标。这种基于荧光酶的生长测定具有高灵敏度和易于操作,因此适用于监测弓形虫的增殖和以高通量方式评估药物疗效。
引言
弓形虫是一种非常成功的细胞内寄生虫,它感染了大约三分之一的人类。其高传播率主要得益于其不同的传播途径,包括食用未煮熟的肉类、接触哺乳动物的水库以及分娩期间的先天性传播。T. Gondii主要导致机会性感染,可导致免疫功能,低下的个人1,2,3,4,5,62,3的严重发病率和死亡率。4,5,61目前用于治疗急性弓形虫病的抗生素在治疗先天性和潜伏性感染方面效率特别低,在3、7、87,8引起严重反应。3因此,迫切需要确定新的治疗方法。了解弓形虫及其宿主中亚细胞过程的差异将有助于确定潜在的药物靶点。因此,需要高效和方便的基因组操作技术来研究弓形体中单个基因的作用。此外,弓形虫属于植物性阿皮金,其中包括其他几种重要的人类病原体,如疟原虫和隐孢子虫。因此,弓形虫可以作为模型有机体,以帮助研究其他阿皮质寄生虫的基本生物学。
为了识别针对微生物病原体的新型抗生素,最初对化合物库进行了高通量筛选,以确定其在抑制微生物生长方面的有效性。到目前为止,已经开发出几种基于微孔板的生长测定法,用于测量T的细胞内生长。 贡迪(即放射性3H-uracil基于合并的定量9,定量ELISA为基础的寄生虫检测使用T.贡迪特异性抗体10,11,记者蛋白质为基础的测量10,11使用β-加拉西西酶或YFP表达弓形虫菌株12,13,,13和最近开发的高含量成像检测14)。
这些个别策略都有独特的优势;但是,某些限制也限制了其应用。例如,由于弓形体只能在核动物细胞内复制,因此自荧光和非特异性结合抗T.贡迪抗体对宿主细胞的宿主细胞会导致荧光测量的干扰。此外,使用放射性同位素需要特殊的安全合规性和潜在的安全问题。其中一些测定更适合在单个时间点评估增长,而不是持续监测增长。
此处介绍的是一种基于荧光酶的协议,用于定量细胞内弓形虫生长。在以前的研究中,NanoLuc Luciferase基因在弓形体图布林促进剂下被克隆,这种透明酶表达结构被转染成野生型(RH_ku80+hxg菌株)寄生虫,以产生RH_ku80=hxg::hxgNLuc菌株(ku80称为RH_ku80::后NLuc) 15。ku80在这项研究中,这种菌株作为父母在细胞内生长测定和基因缺失的菌株。使用RH_ku80::NLuc菌株,在感染后96小时期间监测人类前皮纤维细胞(HFFs)中的寄生虫生长,以计算寄生虫加倍的时间。
此外,通过绘制弓形虫酸的增长率以确定IC50值,可以确定LHVS对寄生虫生长的抑制作用。此前有文献记载,TgCPL是寄生虫中LHVS的主要目标,使用LHVS治疗可减少急性和慢性,17,18,19弓形虫感染的发展。此外,RH_ku80::NLuc用作用于基因组修饰的父母菌株,以产生TgCPL缺陷菌株(RH+ku80=cpl::NLuc),并针对此突变体测量 LHVS 的抑制。通过观察TgCPL缺乏寄生虫中LHVS的IC50值与WT菌株相比的向上变化,经验证TgCPL是LHVS体内的目标。
在此协议中,RH_ku80::NLuc用作父母应变,缺乏有效的非同源端联通路(NHEJ),从而促进双交叉同源性重组(HDR)20,21。20,21此外,50 bp 同源区域在 PCR 的耐药盒的两端侧翼。PCR 产品用作修复模板,使用基于 CRISPR-Cas9 的基因组编辑工具通过 HDR 去除整个基因位点。这种短的同源区域可以很容易地集成到底漆中,为维修模板的生产提供了方便的策略。可以修改该协议,执行通用基因删除和内源基因标记。
例如,在我们最近的出版物中,三个蛋白酶基因,TgCPL,TgCPB(弓形体乙型蛋白酶)和TgSUB1(弓形体亚骨素样蛋白酶1),在TgCRT(弓形素氯奎酮抗药性运输器)中基因消解,缺乏寄生虫使用这种方法15。 TgCPB此外,TgAMN(一种假定的氨肽酶N[TgAMN,TGGT1_221310])被内质标记15。Lourido实验室还报告说,使用40-43bp范围内的短同源区域,使用类似的方法22在弓形体基因组中引入位点基因突变和内源基因标记。这些成功的基因组修改表明,40-50bp同源区域足以在TgKU80缺陷菌株中进行有效的DNA重组,这大大简化了弓形虫腺的基因组操作。
研究方案
弓形虫贡迪被分类在风险组 2 中,必须在生物安全级别 2 (BSL-2) 处理。该议定书已得到克莱姆森大学机构生物安全委员会的审查和批准。
1. 基于路西法删除的弓形虫生长测定
- 在寄生虫接种前1周种子人类前皮成纤维细胞(HFFs),以确保宿主细胞完全融合。在透明板中进行模拟测定,以确保寄生虫在整个评估过程中保持细胞内。
注:在这里,测定是在96孔微孔进行。根据实验需要,可扩展到384或1536孔微板。 - 在使用前2天将弓形虫寄生虫转移到T25烧瓶中,将±0.3-0.4 mL完全裂原的寄生虫转移到汇出的HFF。在37°C下孵育受感染的宿主细胞,2天用5%的CO2孵育。
- 注射器5 mL的新鲜磨碎的寄生虫通过21 G安全针5倍,以释放细胞内寄生虫,然后通过3μm过滤器去除宿主细胞碎片。使用7 mL无酚D10介质从烧瓶中冲洗残留的寄生虫,然后再次通过过滤器。
- 在室温 (RT) 下,在 1000 x g下离心寄生虫 10 分钟。倒入上清液,在10 mL无酚无红D10介质中重新悬浮颗粒。
- 使用血液测量仪对寄生虫进行计数以确定浓度。
- 将寄生虫稀释至1 x 104寄生虫/mL,用于野生类型 (WT) 菌株。对于生长缺乏的寄生虫菌株,相应地增加浓度,以观察到荧光酶信号的显著增加。
- 从96个孔微孔中小心吸气介质,预置氢氟碳化合物,以三列和五行的形式将150μL的寄生虫重新悬浮到井中,代表三个技术复制和五个时间点。
- 在37°C和5%CO2孵育微板4小时。
- 小心地从井中吸气介质,去除未入侵的寄生虫,然后在每排用RT酚无红介质填充井中(第一行除外)。
- 混合等量的PBS和2x荧光酶测定缓冲液,将荧光酶基板稀释至12.5μM。
- 将 100 μL 的稀释荧光酶基板添加到顶排的每个井中。在RT孵育微板10分钟,使细胞完全解解。
- 使用微孔板读取器测量荧光酶活性。板读取器设置列在表 1中。每一次读数表示感染后4小时入侵的寄生虫的初始数量。
- 每行每 24 小时重复步骤 1.9-1.12,每 24 小时重复 4 天,而不更改介质。这些读数反映在感染后24小时、48小时、72小时和96小时复制的寄生虫总数。
- 计算每个时间点的平均读数,并将它们除以 4 小时的平均读数,以确定寄生虫生长随时间的变化。
- 使用图形软件绘制数据。图1A,B显示了具有代表性的生长读数表和RH_ku80::NLuc寄生虫的地块。ku80
- 要计算加倍时间,请绘制在孵育时间各个时间点的折叠变化的 log2 值。使用线性回归函数计算斜率,该斜率表示每个应变的倍增时间(图 1A,C)。
2. 对弓形虫生长的化合物抑制效果评价
注:这里以对弓形体生长抑制性评价为例。测试了 8 种不同浓度的 LHVS,并针对 RH_ku80::NLuc和RH_ku80μcpl的NLuc菌株分别进行了三次技术复制。
- 在寄生虫感染之前,将HFF的种子以三行和9列的形式,以96个井微板的形式,每个菌株进行一个生物复制。宿主细胞在使用前将被允许生长至少7天。
- 在使用前 2 天通过RH_ku80::NLuc和 RH=ku80=cpl::NLuc寄生虫。按照步骤1.2-1.6进行寄生虫纯化和定量。在1 x 104寄生虫/mL时,在无酚红介质中重新悬浮寄生虫。
- 从含有汇液氢氟碳化合物的板中吸气介质,用150μL的寄生虫重新悬浮接种每个孔。在37°C和5%CO2孵育微板4小时。
- 通过连续稀释,在12口井储层中制备8个不同浓度的LHVS。通常,浓度以连续稀释的方式减少三倍。
注:与最高浓度相比,最低浓度降低6,561倍。稀释的褶皱变化可根据不同化合物的不同特性进行相应调整。 - 在感染后4小时,吸气介质去除未入侵的寄生虫,并从2-9栏中填充每口井,以150μL的介质补充不同浓度的LHVS。让第一列充满常规介质,作为未经处理的控制。
- 在37°C和5%CO2孵育微孔板,再延长96小时。
- 执行步骤 1.9-1.11 并测量单个井的透明酶活性。
- 平均从每个LHVS浓度的井中三个技术复制的荧光酶活性。
- 将每个LHVS浓度的平均荧光酶活性除以从未经处理的寄生虫中提取的平均透明酶活性,以百分比计算正态的透明酶活性。
- 使用图形软件根据单个 LHVS 浓度绘制标准化的荧光酶活性(图2)。对寄生虫生长抑制苯丙胺也作为对照进行测量。苯丙胺是一种临床抗生素,用于通过抑制弓形虫的叶酸代谢来治疗急性弓形虫病。
- 在"剂量-反应抑制"回归程序下,使用图形软件中的嵌入式方法计算单个化合物的IC50值,将正态响应与[抑制剂]进行规范化响应。IC50使用以下公式计算:
Y = 100/(1 = X/IC50)
地点:Y表示受感染细胞在不同浓度抑制剂下的正常透明酶活性,X表示抑制剂的个体浓度。
3.弓形虫寄生虫中基于CRISPR-Cas9的基因删除
- 生成用于删除感兴趣的基因的质粒结构,表达指南RNA(sgRNA)和Cas9
- 转到www.ToxoDB.org并检索整个基因编码序列,包括内子和外音,以及 1.5 kb 5'-UTR 和 3'-TRS(未翻译区域)。
注:此处,TgCPL(TGGT1_321530)作为具有代表性的示例。 - 将检索到的 TgCPL序列复制到序列分析软件中(有关名称和版本,请参阅材料表),并标注 5'和 3'-UTR 区域。
- 选择顶部菜单栏中的"工具"图标,然后选择"克隆 " |查找 CRISPR 网站。
- 为 PAM 站点位置选择3'(Cas9)',并在特异性评分部分选择包含弓形虫基因组序列的文件夹。将其余设置保留为默认值。
- 选择具有以下两个条件的 sgRNA:1) 显示高特异性分数,通常 >98%,2) 在 NGG(原间隔相邻图案 (PAM) 序列之后缺少 G。选定的sgRNA通常位于靠近感兴趣基因的起始和停止的部位。
- 复制所选 sgRNA 的序列并将其粘贴到以下底漆模板中。

红色部分表示选定的TgCPL sgRNA 序列。它可以被不同的sgRNA取代,用于各种感兴趣的基因。
注:如果所选 sgRNA 不以 G 开头,则在 sgRNA 的开头添加 G 以帮助增强其表达。 - 执行PCR反应,修改先前存在的质粒表示sgRNA(图3A),该基因以弓形体尿素磷酸酯转移酶(TgUPRT)基因23为目标,使用PCR预混与表2中提供的设置。
- 在琼脂胶凝胶上运行PCR产品,以确认成功放大。一个10 kb的PCR产品预计将被放大(图3B)。
- 使用DNA凝胶提取试剂盒提取PCR产品,并使用现场定向诱变试剂盒进行循环。有关配方,请参阅表 3。在RT孵化反应10-20分钟。
- 将圆形PCR产品转化为大肠杆菌,并挑选10个克隆,以进一步验证设计sgRNA的加入。
- 种植两个克隆和提取质粒。用 BamHI 和 EcoRV 切割纯化质粒。候选质粒将产生两个波段,在2.4 kb和7.2 kb(图3C)。
- 使用 M13 反向引物发送桑格测序的质粒,以确认TgUPRT sgRNA 成功更换为设计的 sgRNA(图 3D)。
- 转到www.ToxoDB.org并检索整个基因编码序列,包括内子和外音,以及 1.5 kb 5'-UTR 和 3'-TRS(未翻译区域)。
- 通过HDR机制生成基因删除修复模板
- 根据所选 sgRNA 的定位位,找到 50 bp 的 5'-UTR 或 3' -UTR 的目标基因进行同源相关重组(HDR,请参阅讨论部分)。区域的选择遵循以下标准,具体取决于 sgRNA 目标的位置。
- 如果 Cas9 的裂解位位于起始切口的上游,请选择以下内容:从裂解部位上游作为左 HDR 区域的 50 bp DNA 序列,以及从止流器 codon 下游作为右 HDR 区域的 50 bp DNA 序列。
- 如果 Cas9 的裂解位位于开始和停止协成之间,请选择以下内容:从起始 Codon 作为左侧 HDR 区域的起始切口上游的 50 bp DNA 序列,以及从停止 Codon 作为右侧 HDR 区域下游的 50 bp DNA 序列。
- 如果 Cas9 的裂解位位于止流器的下游,请选择以下内容:从起始切口上游作为左 HDR 区域的 50 bp DNA 序列,以及从裂解部位下游作为右 HDR 区域的 50 bp DNA 序列。
注:对于TgCPL基因,裂解位位于启动和止停止子体之间。因此,以下底漆设计用于使用 pMDC64 作为模板放大维修模板,该模板对苯丙胺电阻盒进行编码。黑色锥形的序列到pMDC64质粒,用于PCR扩增。以红色标记的区域是TgCPL- 用于同源重组的特定序列。
- 在表 4中描述的 PCR 条件下,使用 PCR 预混料执行 PCR。
- 在琼脂胶凝胶上运行PCR产品(图3E),然后是凝胶提取和标准核酸定量程序。
注:如果无法成功放大预期频段,请优化 PCR 条件和/或切换底漆对。
- 根据所选 sgRNA 的定位位,找到 50 bp 的 5'-UTR 或 3' -UTR 的目标基因进行同源相关重组(HDR,请参阅讨论部分)。区域的选择遵循以下标准,具体取决于 sgRNA 目标的位置。
- 弓形虫转染
- 在含有汇素氢氟碳化合物的T25烧瓶中通过RH+ku80::NLuc寄生虫2天。完全被冲冲的寄生虫的T25烧瓶足以进行两到三次转染。
- 步骤 1.2 中所述的注射器和过滤器纯化寄生虫。在细胞混合缓冲液中重新悬浮寄生虫,并在RT时以1,000 x g旋转10分钟。
- 用10mL的细胞混合缓冲液清洗颗粒化的寄生虫,并在RT时以1,000 x g旋转寄生虫10分钟。
- 小心地从上清液中倒入,以1 x 108寄生虫/mL的浓度在同一缓冲液中重新悬浮寄生虫。
- 将 2 μg 的修复模板 DNA 与 20 μg 的 sgRNA/Cas9 表达质粒(质量比 = 1:5,相当于 1:3 摩尔比)混合。如果修复模板的放大率较低,请相应地减少两个DNA片段的输入。可以使用至少 0.5 μg 的维修模板。
- 在1.5 mL离心管中混合400 μL的寄生虫再悬浮、DNA和5 μL的200 mMATP/500 mM减少谷胱甘肽(GSH)。如果需要,使用细胞混合缓冲液将总体积达到 500 μL。
- 将寄生虫和DNA的混合物转移到电镀皿(4 mm 间隙宽度),并使用电穿孔器进行电穿孔(2 kV 电压,50 Ω 电阻)。
- 将电化寄生虫转移到含有新鲜D10介质中汇有氢氟碳化合物的T25烧瓶中。在24小时后应用适当的抗生素选择药物。
- 保持药物选择性压力,直到转基因寄生虫的生长稳定。
- 从挖空种群中纯化基因组DNA,并通过PCR检查热铁矿抗性盒与TgCPL位点的整合。经核实后,继续第3.4节。如果没有,执行另一轮寄生虫转染和药物选择。无法检测抗药性盒的正确整合通常表明目标基因是必不可少的,或者基因位点不可接近。
- 克隆挖空寄生虫
- 在克隆寄生虫之前,用HFF细胞在37°C和5%CO2孵育1周。2
- 通过含有汇源氢氟碳化合物的T25烧瓶中转基因寄生虫种群的±0.3-0.4mL,并种植2天。如果突变体表现出生长缺陷,请考虑传递更多的寄生虫。
注:为了达到最佳的产生力和生存能力,宿主细胞被寄生虫严重感染,并且大多数寄生虫都保存在细胞内阶段。 - 如步骤 1.3 中提到的注射器感染的宿主细胞和过滤器纯化新鲜被水化的寄生虫。在D10介质中重新悬浮寄生虫,并在RT时以1,000 x g将其旋转10分钟。
- 在10mL的D10介质中重新悬浮颗粒化的寄生虫。
- 使用血液测量仪对寄生虫进行计数,以确定寄生虫的浓度。
- 进行两步稀释,使浓度在D10介质中浓度为10种寄生虫/mL,并辅以适当的抗生素。通常,最初的寄生虫再悬浮被稀释1,000倍,然后第二次稀释到10种寄生虫/mL。
- 96 孔微孔中的吸气介质含有汇入 HFF,并将 150 μL 稀释的寄生虫接种到每个井中。
- 在37°C下孵育板,5%CO2,7天无干扰,允许斑块形成。如果转基因寄生虫表现出生长缺陷,潜伏期可能更长。
- 使用相位对比显微镜筛选板,并仅标记包含单个斑块的孔。
- 执行聚位 PCR 以识别正确的克隆。
- 使用移液器尖端刮起每个井的底部,以提升受感染的 HFF 单层。
- 移液器 75 μL 的电池重新悬浮从每个标记到 1.5 mL 微离心管。
- 在RT时,离心管以最高速度工作10分钟。小心地吸气上清液,并在10.25μL的压解缓冲液中重新悬浮,其中含有稀释缓冲液和试剂盒中提供的脱氧核糖核酸释放添加剂(材料表)。
- 在RT孵化样品4分钟,然后在98°C孵育2分钟。之后,样品可用于PCR或储存在-20°C,直到使用。三组PCR反应用于测试耐药盒的整合和感兴趣的基因的丧失(图4A)。有关 PCR 反应设置,请参阅表 5,有关热循环器设置的表 6。
- 识别正确的克隆,并将四个克隆转移到含有汇液的 T25 瓶中。
- 在单个克隆后,将宿主细胞进行纯化,以进行进一步的PCR验证。
- 如果有识别感兴趣的蛋白质的抗体,请遵循标准免疫膨胀程序,以验证在正确的弓形蛋白敲除液中的目标蛋白的丢失。用于筛选TgCPL-删除突变体的代表性图像如图4B、C所示。
结果
图 1表示 RH_ku80::NLuc应变的生长曲线示例及其加倍时间的派生计算。通常,对三种生物复制进行三种技术复制,以解释荧光酶活性读数的变化。为了计算寄生虫生长的正化褶皱变化,每次在感染后24-96小时读数除以感染后4小时的初始读数,这反映了测定中活寄生虫的起始量(图1A,B)。在确定寄生虫加倍时间方面,针对每个时间点绘制了寄生虫生长的标准化褶皱变化的log2值。接下来,绘图受到线性回归函数的影响,以获得坡度,该斜率表示倍增时间(图 1C)。
通过绘制图2中8种抑制剂浓度的荧光酶活性,确定了LHVS在野生型和βcpl菌株中的抑制作用。在测定中,必须包括未进行抑制治疗的受感染细胞,以使原料荧光酶活性正常化。此外,在透明微孔中进行的模拟实验需要用于测定,以确保寄生虫在测定期结束时仍处于细胞内阶段。
在图 3中,显示了针对TgCPL 的 sgRNA表达式构造的生成和验证,以及TgCPL删除的修复模板的生成和验证。与原始质粒编码的TgUPRT基因匹配的20bp sgRNA通过基于PCR的站点定向诱变,被变异到针对TgCPL基因的DNA序列中。为此,对识别不同基因的sgRNA的DNA序列编码被设计成正向引漆,而反向引底器保持不变,以简化引漆设计。
图3A显示了针对原始模板质粒中的TgUPRT基因的sgRNADNA序列的放大区域,以及用于生成线性化sgRNA表达载体的底漆集。图3B显示了线性TgCPL-瞄准sgRNA表达质粒的代表性凝胶图。图3CC显示了循环TgCPL-靶向sgRNA表达质粒的限制内联酶消化。M13反向引底剂用于对特定基因生成的sgRNA表达载体中的合并引导RNA进行测序。在图3D中,序列DNA区域与质粒模板对齐,以确认成功的诱变。图 3E说明了热质胺耐带盒的开始和结束区域,显示了引漆可用于生产TgCPL基因删除修复模板的位置。修复模板被PCR放大,并加载到1%的琼脂胶凝胶大小验证和凝胶提取。
图4显示了TgCPL淘汰生成和筛选的总体策略。图4A所示的三组引漆用于筛选TgCPL-删除寄生虫,以正确集成5'和3'-ARMs并删除TgCPL编码序列。如图4B所示,通常选择 7 到 8 个克隆进行筛选。筛选通常从检查删除感兴趣的基因的编码序列开始。随后检测 5' 和 3'-ARM,这有助于最大限度地减少要筛选的克隆总数。如果能获得识别目标蛋白的抗体,则可以通过图 4C中显示的免疫印迹进行进一步验证。

图1:使用基于荧光酶的方法对弓形虫寄生虫进行细胞内生长定量。(A) 电子表格软件中的原始透明酶活动读数。感染后24小时、48小时、72小时和96小时时,对照感染后4小时的初始读数进行标准化,以计算寄生虫生长的褶皱变化。(B) 规范化数据被平均和绘制。(C) 折叠变化的 log2 值也被绘制并经过线性回归,以确定寄生虫的倍增时间。请点击此处查看此图形的较大版本。
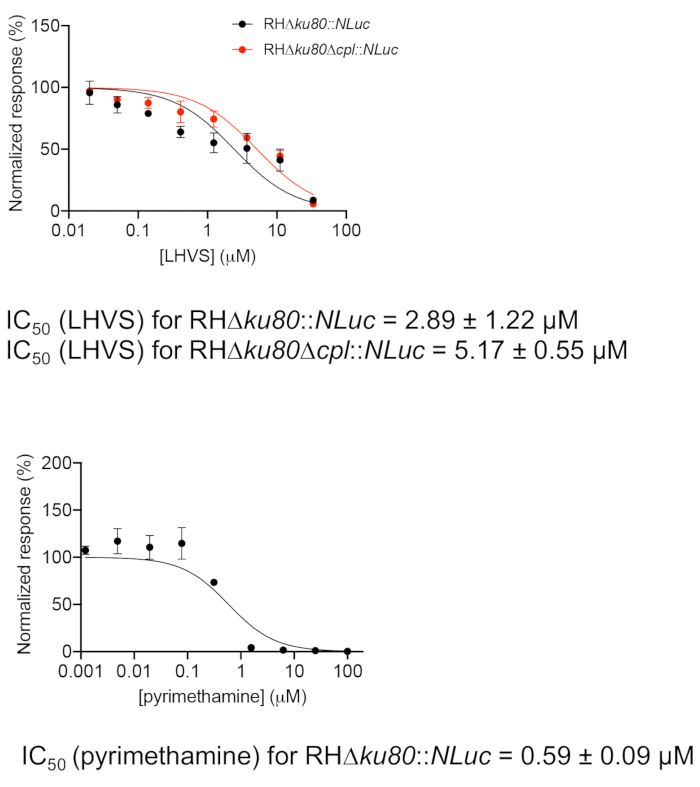
图2:使用基于荧光酶的生长测定的LHVS和苯丙胺的抑制疗效评估。寄生虫被接种到96孔微孔中4小时,以允许宿主细胞入侵。非入侵的寄生虫被冲走,盘子里装满了含有不同浓度的LHVS或苯丙胺的介质,在确定透明酶活性之前再孵育96小时。与未经处理的寄生虫检测到的信号对治疗个体抑制剂浓度的寄生虫的经测荧光酶读数进行标准化。数据在图形程序中绘制,并执行IC50测定的回归分析。该测定在三个生物复制中重复,每个进行三次技术复制。数据表示均值 = SEM,n = 3 生物复制。请点击此处查看此图形的较大版本。
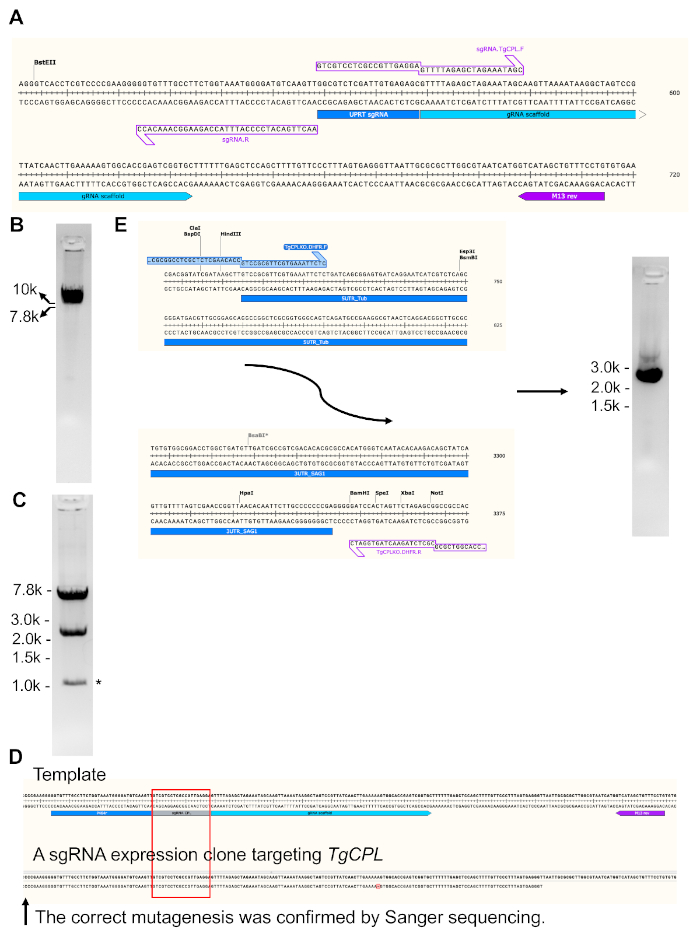
图3:生成针对TgCPL的质粒结构表示sgRNA,并生成TgCPL删除修复模板。(A) 原pSAG1-Cas9-sgRNA-UPRT质粒23通过现场定向诱变试剂盒进行改性,以替代针对TgUPRT基因的sgRNATgCPL。 TgCPLsgRNA 编码区域被放大,以显示引素对该区域。PCR后,突变的质粒被线性化,并加载到1%的琼脂胶凝胶中,以验证成功扩增,然后提取凝胶。(B) PCR放大线性sgRNA表达构造的凝胶图像。(C) 凝胶萃取后,PCR 产物被循环化,随后转化为大肠杆菌。通过限制性内丘酸酶消化和DNA测序筛选含有预期质粒的克隆。DNA消化后的带状尺寸分别为7.2bp和2.4kb。由非特异性裂解产生的带从内核酸被标记星号。(D) 图中标记的 M13 反向引底剂用于对生成的TgCPL-目标 sgRNA 表达向量内的突变引导RNA区域进行序列。序列的DNA区域与质粒模板对齐,以确认成功的诱变。(E) 在这项研究中,与TgCPL的5'和3'-TRS匹配的50bp同源区域被设计成修复模板的放大底漆,并分别在PCR的5'-和3'末端的苯丙胺电阻盒上。在凝胶提取之前,Agarose 凝胶电泳用于验证 PCR 产品的正确尺寸。修复模板的预期大小为 ±2.7 kb。通常,5-6 μg的修复模板可以从200μL的PCR反应中获得。请点击此处查看此图形的较大版本。
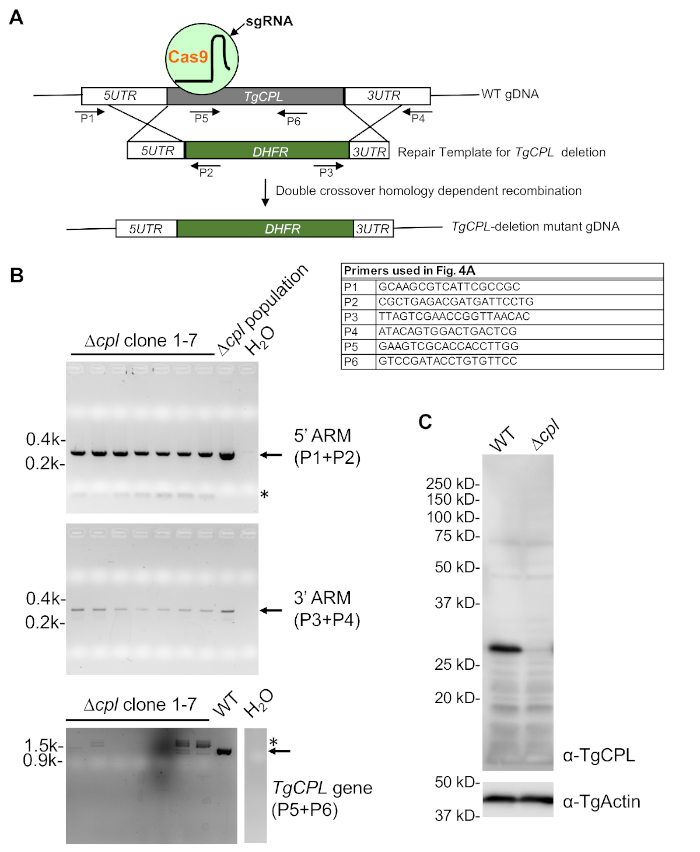
图4:TgCPL缺乏寄生虫的PCR和免疫印迹确认。(A) 描述弓形虫中TgCPL缺失的一般Toxoplasma策略的原理图,以及基于PCR的基于PCR筛选的正确TgCPL敲除克隆。用于筛选的底漆已标记。(B) PCR 和琼脂凝胶电泳用于选择含有热乙胺电阻盒正确集成TgCPL位点和TgCPL基因损失的克隆。μcpl种群的基因组DNA作为5'和3'-ARM检测的阳性控制,而WT基因组DNA用于TgCPL基因的检测作为阳性对照。水在PCR反应中使用代替DNA模板作为负控制。预期波段用箭头表示,而非特异性 PCR 放大用星号标记。(C) 通过PCR筛选鉴定的克隆1生长在细胞凝结制剂的组织培养中,并进一步免疫凝血分析,以确认在敲除中TgCPL表达的丧失。TgActin 用作装载控制。请点击此处查看此图形的较大版本。
| 路西法删除: | 端点 |
| 集成时间: | 1 s |
| 过滤器集 - 排放: | 全光 |
| 光学: | 返回页首 |
| 获得: | 135 |
| 读取速度: | 正常 |
| 延迟: | 100 毫秒 |
| 读取高度: | 4.5 毫米 |
表1:在基于荧光酶的弓形虫生长测定过程中,用于荧光酶活性测量的微板读取器设置。
| 初始变性: | 98 °C 5分钟 |
| 25 个周期 | |
| 变性: | 98 °C 5 s |
| 退火: | 60 °C 15 s |
| 扩展: | 72 °C 1 分钟 |
| 最终扩展: | 72 °C 10分钟 |
表2:用于生成 sgRNA 表达向量的热循环器设置。
| 样品 | 体积(μl) |
| PCR 产品 (10-50 ng) | 1 |
| 2X KLD (激酶, 联利塞, DpnI) 反应缓冲器 | 5 |
| 10X KLD 酶混合物 | 1 |
| 无核酸水 | 3 |
| 总 | 10 |
表3:sgRNA表达载体循环反应配方。
| 初始变性: | 98 °C 5分钟 |
| 35 个周期 | |
| 变性: | 98 °C 15 s |
| 退火: | 58 °C 15 s |
| 扩展: | 72 °C,每 kb 30 s |
| 最终扩展: | 72 °C 10分钟 |
表 4:用于生成维修模板的热循环器设置。
| 样品 | 体积(μl) |
| 总弓形虫基因组DNA | 1 |
| 正向引漆 (25 μM) | 0.2 |
| 反向底漆 (25 μM) | 0.2 |
| 2x PCR 主预混料 | 5 |
| 无核酸水 | 3.6 |
| 总 | 10 |
表5:用于筛选单弓形虫克隆的殖民地PCR反应配方。
| 初始变性: | 98 °C 5分钟 |
| 35 个周期 | |
| 变性: | 98 °C 5 s |
| 退火: | 55 - 62 °C 5 s |
| 扩展: | 72 °C,每 kb 20 s |
| 最终扩展: | 72 °C 1 分钟 |
表6:用于筛选单个弓形虫克隆的热循环器设置。
| 细胞混合缓冲器 | 25 mM HEPES、pH 7.6、120 mM KCl、10 mM K2HPO4/KH2PO4、5mM MgCl 2、0.015 mM CaCl2和 2 mM EGTA。2 |
| D10 中等 | DMEM 1X (康宁, Cat #: 10-013-CV), 10 mM HEPES, 10% (v/v) 宇宙小牛血清 (Hyclone, Cat #: SH30087.03), 1 mM 丙酮钠, 4 mM L-谷氨酰胺, 100 单位/mL青霉素, 和100 μg/mL链球菌素. |
| 酚类无红介质 | DMEM/ 高度改性(Hyclone, Cat #: SH30284.02),10 mM HEPES,10%(v/v)宇宙小牛血清(Hyclone,Cat :SH30087.03),1 mM 丙酮钠,4 mM L-谷氨酰胺,100 单位/mL青霉素,以及100微克/米耳的链球菌素。 |
| 2X NLuc 缓冲器 | 100 mM MES, pH 6.0, 1mM CDTA, 0.5% Tergitol, 0.05% 马祖 DF 204, 150 mM KCl, 1 mM DTT, 35 mM Thiourea. |
补充表 1:缓冲区的配方。
讨论
*本协议描述了一种基于荧光酶的协议,用于评估细胞内弓形虫的生长,并评估化合物对寄生虫生长的抑制作用。与现有的测量细胞内弓形虫生长的策略相比,该方法具有高灵敏度和特异性。在监测寄生虫生长时,建议在96孔微孔的模拟检测中确认测试菌株在评估期结束前不会过早压出宿主细胞。否则,发光读数将不能准确反映寄生虫的生长,因为弓形体只在宿主细胞内复制。
据观察,苯酚红染料能迅速淬火,由于板读取器对个别井的测量延迟,这可能导致技术复制的荧光酶读数存在显著差异。因此,在96井微板播种之前,最好在无酚无红介质中制备氢氟碳化合物。此外,在高荧光酶活性的情况下,跨井干扰可能导致附近油井之间表现出强烈的荧光酶活性的显著差异。因此,建议在每个应变之间放置一个空列。
具体来说,对于RH+ku80::NLuc菌株,1500种寄生虫接种到每个井中进行生长测定。由于WT弓形虫寄生虫的倍增时间是+6-8小时24,预计在感染后24小时,荧光酶活性将增加8至16倍。然而,某些具有显著生长缺陷的菌株只会使荧光酶活性略有增加。因此,如果最初的寄生虫内膜低,荧光酶活性的固有变异将掩盖在生长期发光增加的观察。因此,建议接种数量较多的寄生虫,以实现生长缺陷菌株的精确褶皱变化。
在协议中,指南RNA设计遵循了哺乳动物细胞25中基于CRISPR-Cas9的基因组修改的一般规则。目前,许多类型的软件和在线平台为各种生物体的RNA设计提供指导性RNA设计提供服务,如CHOPCHOP26、E-CRISP27和EuPaGDT28。2827在这里, 商业软件 (材料表) 用于设计 sgRNA.与前面提到的在线程序相比,该软件为sgRNA设计提供了一个局部环境。它使用先前发布的算法25,29,29计算每个候选sgRNA的活动和特异性分数。
从技术上讲,任何位于感兴趣的基因内、包含高特异性分数的sgRNA都能有效地调解基因组DNA的裂解,以便进行下游同源性重组。实际上,sgRNA 是首选,目标是接近开始或停止 codon 的区域。感兴趣的基因可以使用sgRNA进行内生标记,在基因末端产生双链间隙。在基因缺失之前,在基因缺失之前,在抗体对感兴趣的蛋白质不可用的情况下,通过免疫印迹检测有助于确认基因丢失。基因的Epitope标记也可以帮助通过免疫荧光显微镜确定感兴趣的蛋白质的亚细胞位置。此外,如果靶基因是必不可少的,sgRNA识别该基因的开始区域可用于替换其同源促进剂到四环素反应促进剂,以产生有条件的敲除。
此外,该协议还描述了一种在弓形体中删除基因的技术,用热米特哈明耐性盒取代TgCPL基因。通过使用编码其他耐药性盒的不同质粒模板,研究人员可以通过PCR修改引漆序列,将其他抗生素耐药性基因纳入修复模板。此外,可以修改该协议以执行其他基因组修改,如内源基因标记、促进剂替代和站点定向突变。值得注意的是,该协议中使用的同源区域长度仅为50个基点。另一项研究成功地使用了40-43bp同源DNA序列,在弓形虫寄生虫22中引入了单核苷酸突变和基因表位标记。如此短长度的同源DNA序列可以很容易地融入引素中。虽然我们没有定量评估这个同源区域特定长度的HDR效率,但似乎40-50bp区域足以在TgKu80缺陷弓形虫菌株中进行有效的DNA重组,最近成功对几个基因进行基因操作就证明了这一,点。
在化合物的疗效测定过程中,如果潜在的药物靶基因至关重要,则对野生型和挖空菌株之间的IC50值变化进行比较是不现实的。在这种情况下,需要直接测量化合物抑制重组蛋白活性的能力的测定,以评估药物的疗效和特异性。最近的文献报道了弓形体中单个基因的适应性评分,通过执行一个基因组宽的CRISPR屏幕30,它可以作为指南,以帮助评估为感兴趣的基因生成直敲直突变体的难度。
总之,该协议描述了成功完成基于荧光酶的细胞内弓形虫生长测定和化学抑制剂对弓形虫生长的评估策略。此外,还详细介绍了一种基于CRISPR-Cas9的基因组编辑协议,用于弓形虫寄生虫的基因删除,该协议已广泛应用于该领域。个别实验室可根据实验需要修改所述方案,如内源基因标记、切换药物选择标记、改变细胞内寄生虫生长的评估周期。
披露声明
作者没有什么可透露的。
致谢
作者要感谢Sibley博士和卡拉瑟斯分享pSAG1-Cas9-sgRNA-TgUPRT质粒和抗TgCPL和TgActin抗体。这项工作得到了克莱姆森启动基金(至Z.D.)、骑士圣殿眼基金会儿科眼科职业启动研究补助金(至Z.D.)、NIH COBRE赠款P20GM109094(至Z.D.)和NIH R01AI143707(至Z.D.)的试点赠款的支持。资助者在研究设计、数据收集和分析、决定出版或编写手稿方面没有作用。
材料
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
参考文献
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。