Method Article
Détermination de l’efficacité de l’inhibiteur chimique contre la croissance intracellulaire de Toxoplasma Gondii utilisant un essai de croissance basé sur luciferase
Dans cet article
Résumé
Présenté ici est un protocole pour évaluer l’efficacité d’inhibition des composés chimiques contre la croissance intracellulaire in vitro de Toxoplasma gondii en utilisant un essai de croissance à base de luciferase. La technique est utilisée pour confirmer la spécificité de l’inhibition par la suppression génétique du gène cible correspondant. L’inhibition de LHVS contre la protéase TgCPL est évaluée en exemple.
Résumé
Toxoplasma gondii est un agent pathogène protozoaire qui affecte largement la population humaine. Les antibiotiques actuels utilisés pour le traitement de la toxoplasmose clinique sont limités. En outre, ils présentent des effets secondaires indésirables dans certains groupes de personnes. Par conséquent, la découverte de nouvelles thérapies pour la toxoplasmose clinique est impérative. La première étape du développement d’antibiotiques de nouveau est d’identifier des composés chimiques montrant une efficacité élevée dans l’inhibition de la croissance des parasites à l’aide d’une stratégie de dépistage à haut débit. En tant qu’agent pathogène intracellulaire obligatoire, Toxoplasma ne peut se répliquer qu’à l’intérieur des cellules hôtes, ce qui interdit l’utilisation de mesures d’absorption optique comme indicateur rapide de croissance. Présenté ici est un protocole détaillé pour un essai de croissance basé sur la luciferase. À titre d’exemple, cette méthode est utilisée pour calculer le temps de doublement des parasites Toxoplasma de type sauvage et mesurer l’efficacité du phényl de sulfone morpholinurea-leucyl-homophenyl-vinyle (LHVS, un composé de protéase-ciblage de cystéine) concernant l’inhibition de la croissance intracellulaire parasite. Un protocole de suppression de gènes à base de CRISPR-Cas9 à base de CRISPR-Cas9 dans Toxoplasma utilisant 50 régions homologues de 50 bp pour la recombinaison dépendante de l’homologie (HDR). En quantifiant les efficacités d’inhibition du LHVS dans les parasites de type sauvage et TgCPL (Toxoplasma cathepsin L-like protease), il est démontré que le LHVS inhibe la croissance des parasites de type sauvage plus efficacement que la croissance de la TgCPL est une cible à laquelle LHVS se lie dans Toxoplasma. La sensibilité élevée et le fonctionnement facile de cet essai de croissance basé sur la luciferase le rendent approprié pour surveiller la prolifération de Toxoplasma et évaluer l’efficacité de drogue d’une manière élevée de débit.
Introduction
Toxoplasma gondii est un parasite intracellulaire obligatoire très réussi qui infecte environ un tiers de la population humaine. Son taux de transmission élevé est principalement dû à ses diverses voies de transmission, y compris la consommation de viande insuffisamment cuite, l’exposition aux réservoirs de mammifères et la transmission congénitale pendant la naissance. T. gondii provoque principalement des infections opportunistes qui peuvent conduire à une morbidité sévère et la mortalité chez les individus immunodéprimés1,2,3,4,5,6. Les antibiotiques actuellement utilisés pour le traitement de la toxoplasmose aigue sont particulièrement inefficaces dans le traitement des infections congénitales et latentes et provoquent des réactions graves chez certaines personnes3,7,8. Il existe donc un besoin urgent d’identifier de nouvelles thérapies. Comprendre les différences dans les processus subcellulaires au sein de Toxoplasma et de son hôte aidera à identifier les cibles potentielles de médicaments. Par conséquent, des techniques efficaces et pratiques de manipulation du génome sont nécessaires pour étudier les rôles des gènes individuels au sein de Toxoplasma. En outre, Toxoplasma appartient au phylum Apicomplexa, qui comprend plusieurs autres agents pathogènes humains importants, tels que Plasmodium spp. et Cryptosporidium spp. Par conséquent, Toxoplasma peut être utilisé comme un organisme modèle pour aider à étudier la biologie de base chez d’autres parasites apicomplexan.
Pour identifier de nouveaux antibiotiques contre les agents pathogènes microbiens, le dépistage à haut débit d’une bibliothèque de composés chimiques est initialement effectué pour déterminer leur efficacité dans la répression de la croissance microbienne. Jusqu’à présent, plusieurs essais de croissance à base de microplates ont été développés pour mesurer la croissance intracellulaire de T. gondii (c.-à-d. la quantification à base d’incorporation 3H-uracil9, la détection quantitative des parasites à base d’ELISA à l’aide de T. gondii-anticorps spécifiques10,11, la mesure protéinée journaliste à l’aide de souches Toxoplasma 12,13et une imagerie à haute teneur récemment développée assay14).
Ces stratégies individuelles ont toutes des avantages uniques; toutefois, certaines limitations limitent également leurs demandes. Par exemple, puisque Toxoplasma ne peut se répliquer qu’à l’intérieur des cellules animales nucléées, l’autofluorescence et la liaison non spécifique des anticorpsanti-T. gondii pour héberger des cellules provoquent des interférences dans les mesures à base de fluorescence. De plus, l’utilisation d’isotopes radioactifs exige une conformité particulière à la sécurité et des problèmes de sécurité potentiels. Certains de ces essais sont plus appropriés pour évaluer la croissance à un seul moment plutôt que la surveillance continue de la croissance.
Présenté ici est un protocole basé sur la luciferase pour la quantification de la croissance intracellulaire de Toxoplasma. Dans une étude précédente, le gène NanoLuc luciferase a été cloné sous le promoteur de toxoplasma tubulin, et cette construction d’expression luciferase a été transfected dans le type sauvage (RH-ku80ô hxg souche) parasites pour créer un RH'ku80hxg::NLuc souche (appelé RH'ku80::NLuc ici)15. Cette souche a servi de souche parentale pour la détermination intracellulaire de croissance et la suppression de gène dans cette étude. À l’aide duRH-ku80:: soucheNLuc, la croissance des parasites dans les fibroblastes en prépuce humain (FSF) a été surveillée sur une période de 96 h post-infection pour calculer le temps de doublement des parasites.
En outre, l’efficacité d’inhibition de LHVS contre la croissance de parasite peut être déterminée en traçant des taux de croissance de Toxoplasma contre des concentrations sérieuses de LHVS pour identifier la valeurIC 50. La littérature précédente a rapporté que TgCPL est une cible principale de LHVS dans les parasites et que le traitement avec LHVS diminue le développement des infections aigues et chroniques de Toxoplasma 16,17,18,19. En outre, RH-ku80::NLuc a été utilisé comme la souche parentale pour la modification du génome pour générer une souche TgCPLdéficiente (RH'ku80'cpl::NLuc), et l’inhibition de LHVS a été mesurée par rapport à ce mutant. En observant un décalage des valeurs IC50 pour LHVS dans les parasites TgCPL-déficientspar rapport à la souche WT, il a été validé que TgCPL est ciblé par LHVS in vivo.
Dans ce protocole,RH-ku80::NLuc est utilisé comme la souche parentale, qui n’a pas une voie efficace non-homologe de jointure (NHEJ), facilitant ainsi double multisegment homologie-dépendance recombinaison (HDR)20,21. En outre, 50 régions homologues bp sont flanqués aux deux extrémités d’une cassette de résistance aux médicaments par PCR. Le produit PCR sert de modèle de réparation pour enlever l’ensemble du locus génétique via HDR à l’aide d’outils d’édition du génome à base de CRISPR-Cas9. Ces courtes régions homologues peuvent être facilement incorporées dans les amorces, fournissant une stratégie pratique pour la production du modèle de réparation. Ce protocole peut être modifié pour effectuer la suppression universelle des gènes et le marquage des gènes endogènes.
Par exemple, dans notre publication la plus récente, trois gènes de protéase, TgCPL, TgCPB (Toxoplasma cathepsin B-like protease), et TgSUB1 (Toxoplasma subtilisin-like protease 1), ont été génétiquement ablés dans TgCRT (Toxoplasma chloroquine-resistance transporteur) -parasites déficients en utilisant cette méthode15. En outre, TgAMN (un aminopeptidase putatif N [TgAMN, TGGT1_221310]) a été étiqueté endogènement15. Le laboratoire de Lourido a également rapporté l’utilisation de régions homologues courtes dans la gamme de 40-43 bp pour l’introduction de la mutation génétique dirigée par le site et le marquage de gène endogène dans le génome de Toxoplasma utilisant une méthode similaire22. Ces modifications réussies du génome suggèrent qu’une région homologue de 40-50 bp est suffisante pour la recombinaison efficace de l’ADN dans la souche TgKU80-déficiente,qui simplifie grandement la manipulation du génome dans Toxoplasma gondii.
Protocole
Toxoplasma gondii est classé dans le groupe de risque 2 et doit être traité à un niveau de biosécurité 2 (BSL-2). Le protocole a été examiné et approuvé par le Comité de biosécurité institutionnelle de l’Université Clemson.
1. Toxoplasma croissance basée à Luciferase
- Fibroblastes de prépuce humain de graines (FFC) 1 semaine avant l’inoculation de parasite pour s’assurer que les cellules hôtes sont entièrement confluentes. Effectuez un simulacre d’essai dans une plaque transparente pour s’assurer que les parasites restent intracellulaires tout au long de la période d’évaluation.
REMARQUE: Ici, l’essai est effectué en 96 microplates de puits. Selon les besoins expérimentaux, il peut être mis à l’échelle jusqu’à 384 ou 1536 microplates de puits. - Passer les parasites Toxoplasma en FFC confluents 2 jours avant utilisation en transférant 0,3 à 0,4 ml de parasites entièrement lysed dans un flacon T25. Incuber les cellules hôtes infectées à 37 oC avec 5% de CO2 pendant 2 jours.
- Syringe 5 mL de parasites fraîchement lysed à travers une aiguille de sécurité 21 G 5x pour libérer les parasites intracellulaires, puis passer à travers un filtre de 3 m pour enlever les débris cellulaires hôtes. Rincer les parasites résiduels hors du flacon à l’aide de 7 ml de milieu D10 sans phénol, puis passer à nouveau à travers le filtre.
- Centrifuge parasites à 1000 x g pendant 10 min à température ambiante (RT). Verser le supernatant et réutiliser la pastille dans 10 ml de phénol sans phénol D10 médias.
- Comptez les parasites à l’aide d’un hémocytomètre pour déterminer la concentration.
- Diluer les parasites à 1 x 104 parasites/mL pour la souche de type sauvage (WT). Pour les souches parasites déficientes en croissance, augmentez la concentration en conséquence pour observer une augmentation significative des signaux de luciferase.
- Aspirez soigneusement les supports de 96 microplaces de puits pré-ensemencées de FFC et inoculez 150 L de réanimation de parasite dans des puits dans un format de trois colonnes et cinq rangées, ce qui représente trois répliques techniques et cinq points de temps.
- Incuber la microplaque à 37 oC et 5% de CO2 pour 4 h.
- Aspirez soigneusement les médias des puits pour éliminer les parasites non envahis, puis remplissez les puits avec des supports sans phénol RT dans chaque rangée (sauf pour la première rangée).
- Mélanger des volumes égaux de TAMPON d’analyse de 2x luciferase et diluer le substrat de luciferase à 12,5 m.
- Ajouter 100 L de substrat de luciferase dilué dans chaque puits de la rangée supérieure. Incuber les microplates à RT pendant 10 min pour permettre aux cellules de lyse complètement.
- Mesurer l’activité de luciferase à l’aide d’un lecteur de microplaque. Les paramètres du lecteur de plaques sont répertoriés dans le tableau 1. Chaque lecture représente le nombre initial de parasites envahis à 4 h post-infection.
- Répéter les étapes 1.9-1.12 pour chaque rangée toutes les 24 h pendant 4 jours sans changer le milieu. Ces lectures reflètent le nombre total de parasites répliqués à 24 h, 48 h, 72 h et 96 h post-infection.
- Calculez les lectures moyennes à chaque point de temps et divisez-les par les lectures moyennes à 4 h pour déterminer les changements de pli dans la croissance des parasites au fil du temps.
- Tracez les données à l’aide d’un logiciel graphique. Une table de lecture de croissance représentative et des parcelles de RH-ku80:: Les parasitesNLuc sont montrés dans la figure 1A,B.
- Pour calculer le temps de doublement, tracez les valeurs journal2 des changements de pli aux points de temps individuels au cours du temps d’incubation. Utilisez une fonction de régression linéaire pour calculer la pente, ce qui représente le temps de doublement de chaque souche(figure 1A,C).
2. Évaluation de l’efficacité de l’inhibition du composé chimique contre la croissance de Toxoplasma
REMARQUE : Ici, l’évaluation de l’inhibition de LHVS dans la croissance de Toxoplasma est présentée comme exemple. Huit concentrations différentes de LHVS sont testées, et trois répliques techniques sont effectuées pour chacune des trois répliques biologiques pour les deux RHku80::NLuc et RH'ku80'cpl:: souchesNLuc.
- Avant l’infection par parasite, les FSF de graine à 96 microplaques de puits dans le format de trois rangées et neuf colonnes pour une réplique biologique par composé par souche. Les cellules hôtes seront autorisées à se développer pendant au moins 7 jours avant l’utilisation.
- PassRH-ku80::NLuc et RH'ku80'cpl::NLuc parasites pendant 2 jours avant l’utilisation. Suivez les étapes 1.2-1.6 pour la purification et la quantification des parasites. Résuspendez les parasites dans les médias sans phénol à 1 x 104 parasites/mL.
- Aspirer les supports d’une plaque contenant des FFC confluents et inoculer chaque puits avec 150 l de résuspension parasite. Incuber la microplaque à 37 oC et 5% de CO2 pour 4 h.
- Préparer le LHVS à huit concentrations différentes dans un réservoir de 12 puits par dilution en série. En général, les concentrations sont multipliées par trois d’une manière de dilution sérielle.
REMARQUE : La concentration la plus faible est réduite de 6 561 fois par rapport à la concentration la plus élevée. Le changement de pli de la dilution peut être ajusté en conséquence en fonction des différentes propriétés des composés individuels. - À 4 h post-infection, les supports d’aspiration pour enlever les parasites non envahis et remplir chacun des colonnes 2-9 avec 150 L de médias complétés par LHVS à différentes concentrations. Laissez la première colonne remplie de milieu régulier pour servir de contrôle non traité.
- Incuber la microplaque à 37 oC et 5% de CO2 pour un supplément de 96 h.
- Effectuez les étapes 1.9-1.11 et mesurez l’activité de luciferase des puits individuels.
- Moyenne des activités de luciferase de trois répliques techniques à partir de puits de chaque concentration individuelle de LHVS.
- Divisez l’activité moyenne de luciferase pour chaque concentration de LHVS par l’activité moyenne de luciferase dérivée des parasites non traités pour calculer l’activité normalisée de luciferase en pourcentage.
- Tracez les activités de luciferase normalisées par rapport aux concentrations individuelles de LHVS à l’aide d’un logiciel graphique(figure 2). L’inhibition de la pyriméthamine contre la croissance des parasites est également mesurée comme un contrôle. La pyriméthamine est un antibiotique clinique utilisé pour traiter la toxoplasmose aigue en inhibant le métabolisme de l’acide folique dans Toxoplasma.
- Calculez les valeurs IC50 pour les composés individuels à l’aide de la méthode intégrée dans le logiciel graphique, réponse normalisée par rapport [inhibiteur], dans le cadre du programme de régression de « dose-réponse-inhibition ». L’IC50 est calculé à l’aide de la formule suivante :
Y 100/(1 X/IC50)
Où : Y représente les activités normalisées de luciferase des cellules infectées sous différentes concentrations d’inhibiteur, et X représente des concentrations individuelles d’inhibiteur.
3. Suppression génétique à base de CRISPR-Cas9 chez les parasites Toxoplasma
- Génération d’une construction plasmide exprimant l’ARN guide (sgRNA) et Cas9 pour la suppression d’un gène d’intérêt
- Allez à www.ToxoDB.org et récupérer toute la séquence de codage génétique, y compris les introns et les exons, ainsi que 1,5 kb 5'-UTR et 3'-UTR (régions non traduites).
REMARQUE : Ici, TgCPL (TGGT1_321530) est visé comme un exemple représentatif. - Copiez la séquence TgCPL récupérée dans le logiciel d’analyse de séquence (se référer à La Table des Matériaux pour le nom et la version) et étiquetez les régions 5'- et 3'-UTR.
- Sélectionnez l’icône Tools dans la barre de menu supérieure, puis sélectionnez Cloning . Trouver des sites CRISPR.
- Choisissez 3'(Cas9) ' pour l’emplacement du site PAM et sélectionnez le dossier contenant la séquence du génome de Toxoplasma dans la section de notation de spécificité. Laissez le reste des paramètres par défaut.
- Choisissez un sgRNA avec les deux critères suivants: 1) montrant un score de spécificité élevé, généralement -gt;98%, et 2) manquant d’un G suivant le NGG, un protospacer motif adjacent (PAM) séquence. Le sgRNA sélectionné est généralement situé à des endroits proches du début et arrêter les codons du gène d’intérêt.
- Copiez la séquence du sgRNA sélectionné et collez-le dans le modèle d’amorce suivant.

La partie en rouge représente la séquence TgCPL sgRNA sélectionnée. Il peut être remplacé par différents sgRNAs pour divers gènes d’intérêt.
REMARQUE : Si le sgRNA sélectionné ne commence pas avec G, ajoutez G au début du sgRNA pour aider à améliorer son expression. - Effectuez une réaction PCR pour modifier le plasmide préexistant exprimant le sgRNA (figure 3A) qui cible Toxoplasma uracil phosphoribosyltransferase (TgUPRT) gène23 à l’aide d’un prémélange PCR avec les paramètres fournis dans le tableau 2.
- Exécuter le produit PCR sur un gel agarose pour confirmer l’amplification réussie. On s’attend à ce qu’un produit PCR de 10 kb soit amplifié(figure 3B).
- Extraire le produit PCR à l’aide d’un kit d’extraction de gel d’ADN et circulairez-le à l’aide d’un kit de mutagenesis dirigé par le site. Consultez le tableau 3 pour la recette. Incuber la réaction pendant 10-20 min à RT.
- Transformez le produit PCR circulaire en E. coli et choisissez 10 clones pour une vérification plus poussée de l’incorporation du sgRNA conçu.
- Cultivez deux clones et extrayez des plasmides. Couper les plasmides purifiés avec BamHI et EcoRV. Les plasmides candidats donneront deux bandes à 2,4 kb et 7,2 kb(figure 3C).
- Envoyez les plasmides pour le séquençage Sanger à l’aide d’amorces inversées M13 pour confirmer le remplacement réussi de TgUPRT sgRNA avec le sgRNA conçu(figure 3D).
- Allez à www.ToxoDB.org et récupérer toute la séquence de codage génétique, y compris les introns et les exons, ainsi que 1,5 kb 5'-UTR et 3'-UTR (régions non traduites).
- Génération de modèle de réparation pour la suppression des gènes via le mécanisme HDR
- Selon les sites de ciblage du sgRNA sélectionné, localisez 50 bp de 5'-UTR ou 3'-UTRs du gène cible pour la recombinaison dépendante de l’homologie (HDR, voir section de discussion). La sélection des régions suit les critères énumérés ci-dessous, selon l’emplacement des cibles sgRNA.
- Si le site de clivage par Cas9 est situé en amont à partir du codon de départ, sélectionnez ce qui suit: une séquence d’ADN de 50 bp en amont du site de clivage comme la région HDR gauche, et une séquence d’ADN de 50 bp en aval de l’arrêt codon comme la bonne région HDR.
- Si le site de clivage par Cas9 se trouve entre le début et l’arrêt des codons, sélectionnez ce qui suit : une séquence d’ADN de 50 bp en amont du codon de départ comme région HDR gauche, et une séquence d’ADN de 50 bp en aval du codon d’arrêt comme région HDR droite.
- Si le site de clivage par Cas9 est situé en aval du codon d’arrêt, sélectionnez ce qui suit : une séquence d’ADN de 50 bp en amont du codon de départ comme région HDR gauche, et une séquence d’ADN de 50 bp en aval du site de clivage comme région HDR droite.
REMARQUE : Pour le gène TgCPL, le site de clivage est situé entre les codons de démarrage et d’arrêt. Ainsi, les amorces suivantes sont conçues pour amplifier le modèle de réparation en utilisant pMDC64 comme modèle, qui code une cassette de résistance à la pyriméthamine. Les séquences en anneal noir au plasmide pMDC64 pour l’amplification DE PCR. Les régions étiquetées en rouge sont des séquences spécifiques À TgCPLpour la recombinaison homologue.
- Effectuez le PCR à l’aide d’un prémélange PCR dans les conditions PCR décrites dans le tableau 4.
- Exécuter le produit PCR sur un gel d’agarose(figure 3E), suivi de l’extraction du gel et des procédures standard de quantification de l’acide nucléique.
REMARQUE : Si la bande attendue ne peut pas être amplifiée avec succès, optimisez les conditions pcR et/ou changez les paires d’amorce.
- Selon les sites de ciblage du sgRNA sélectionné, localisez 50 bp de 5'-UTR ou 3'-UTRs du gène cible pour la recombinaison dépendante de l’homologie (HDR, voir section de discussion). La sélection des régions suit les critères énumérés ci-dessous, selon l’emplacement des cibles sgRNA.
- Transfection Toxoplasma
- PassRH-ku80:: parasitesNLuc pendant 2 jours dans un flacon T25 contenant des FFC confluents. Un flacon T25 de parasites entièrement lysed est suffisant pour deux à trois transfections.
- Syringe et filtre-purifier les parasites comme décrit dans l’étape 1.2. Résuspendez les parasites dans le tampon cytomix et tournez vers le bas à 1.000 x g pendant 10 min à RT.
- Laver les parasites granulés avec 10 ml de tampon cytomix et faire tourner les parasites à 1000 x g pendant 10 min à RT.
- Versez soigneusement le supernatant et réutilisez les parasites dans le même tampon à une concentration de 1 x 108 parasites/mL.
- Mélanger 2 g d’ADN du modèle de réparation avec 20 g de plasmides d’expression sgRNA/Cas9 (rapport de masse de 1:5, soit l’équivalent d’un rapport de 1:3 molaire). Si le rendement d’amplification du modèle de réparation est faible, réduisez l’entrée des deux pièces d’ADN en conséquence. Un minimum de 0,5 g de modèle de réparation peut être utilisé.
- Mélanger 400 l de réanimation de parasite, d’ADN et de 5 L de glutathion réduite DE 200 mM ATP/500 mM (GSH) dans un tube de centrifugeuse de 1,5 mL. Porter le volume total à 500 L avec tampon cytomix, si nécessaire.
- Transférer le mélange de parasites et d’ADN dans une cuvette d’électroporation (largeur d’écart de 4 mm) et effectuer une électroporation (2 kV de tension, 50 résistance) à l’aide d’un appareil d’électroporation.
- Transférer les parasites électropolés dans un flacon T25 contenant des FFC confluents dans un milieu frais D10. Appliquer antibiotique approprié pour la sélection des médicaments après 24 h.
- Maintenez la pression sélective des médicaments jusqu’à ce que la croissance des parasites transgéniques soit stable.
- Purifier l’ADN génomique de la population knock-out et vérifier l’intégration de la cassette de résistance à la pyriméthamine dans le locus TgCPL par PCR. Après vérification, passez à l’article 3.4. Si ce n’est pas le cas, effectuez une autre série de transfection de parasites et de sélection de médicaments. L’incapacité de détecter l’intégration correcte de la cassette de résistance aux médicaments suggère généralement que le gène cible est essentiel ou que le locus génétique n’est pas accessible.
- Clonage de parasites knock-out
- Semences deux microplaques de puits 96 avec des cellules HFF et incuber à 37 oC et 5 % de CO2 pendant 1 semaine avant le clonage des parasites.
- Passer de 0,3 à 0,4 ml de la population de parasites transgéniques dans un flacon T25 contenant des FFC confluents et les cultiver pendant 2 jours. Envisagez de passer plus de parasites si le mutant montre des défauts de croissance.
REMARQUE : Pour obtenir le meilleur rendement et la meilleure viabilité, les cellules hôtes sont fortement infectées par les parasites, et la plupart des parasites sont conservés au stade intracellulaire. - Cellules hôtes infectées par la seringue et parasites frais lysés comme mentionné à l’étape 1.3. Resuspendez les parasites dans D10 milieu et les faire tourner vers le bas à 1.000 x g pendant 10 min à RT.
- Résuspendez les parasites granulés dans 10 ml de D10 moyen.
- Comptez les parasites à l’aide d’un hémocytomètre pour déterminer la concentration de parasites.
- Effectuer une dilution en deux étapes pour amener la concentration à 10 parasites/mL dans le milieu D10 complété par l’antibiotique approprié. Habituellement, la réanimation initiale de parasite est diluée par 1.000 fois, suivie d’une deuxième dilution à 10 parasites/mL.
- Aspirer les supports de 96 microplates de puits contenant des FFC confluents et inoculer 150 L de parasites dilués dans chaque puits.
- Plaques incubate à 37 oC avec 5% de CO2 pendant 7 jours sans perturbation pour permettre la formation de plaque. La période d’incubation peut être plus longue si les parasites transgéniques présentent des défauts de croissance.
- Filtrer les plaques à l’aide d’un microscope à contraste de phase et marquer uniquement les puits contenant une seule plaque.
- Effectuez la colonie PCR pour identifier les clones corrects.
- Utilisez des pointes pipette pour gratter le fond de chaque puits pour soulever les monocouches infectées HFF.
- Pipet 75 L de la résuspension cellulaire de chaque puits marqué dans 1,5 mL tubes de microcentrifuge.
- Tubes de centrifugeuse pendant 10 min à vitesse maximale à RT. Aspirez soigneusement le supernatant et réutilisez la granule dans 10,25 l de tampon de lyse contenant le tampon de dilution et l’additif de libération d’ADN fourni dans le kit (Tableau des matériaux).
- Incuber les échantillons pendant 4 min à RT, puis 2 min à 98 oC. Par la suite, les échantillons peuvent être utilisés pour pcR ou stockés à -20 oC jusqu’à l’utilisation. Trois séries de réactions PCR sont utilisées pour tester l’intégration de la cassette de résistance aux médicaments et la perte du gène d’intérêt(figure 4A). Consultez le tableau 5 pour la configuration de réaction PCR et le tableau 6 pour les réglages thermocycler.
- Identifiez les clones corrects et transférez quatre clones dans des flacons T25 contenant des FFC confluents.
- Après que les clones individuels lyse cellules hôtes, purifier l’ADN génomique pour la vérification plus poussée de PCR.
- Si un anticorps reconnaissant la protéine d’intérêt est disponible, suivez une procédure d’immunoblotting standard pour vérifier la perte de la protéine cible dans les KO Toxoplasma corrects. Des images représentatives pour le dépistage d’un mutant TgCPL-deletion sont montrées dans la figure 4B,C.
Résultats
La figure 1 représente un exemple de courbe de croissance pour le RHku80::NLuc strain et le calcul dérivé pour son temps de doublement. En général, l’essai est effectué en trois répliques techniques pour chacune des trois répliques biologiques pour tenir compte des variations des lectures d’activité de luciferase. Afin de calculer le changement de pli normalisé de la croissance des parasites, chaque lecture à 24-96 h post-infection a été divisée par la lecture initiale à 4 h post-infection, qui reflète la quantité de départ de parasites vivants dans l’essai (figure 1A,B). En termes de détermination du temps de doublement des parasites, les valeurs de journal2 des changements de pli normalisés de la croissance des parasites ont été tracées contre chaque point de temps. Ensuite, l’intrigue a été soumise à une fonction de régression linéaire pour obtenir la pente, ce qui représente le doublement du temps(figure 1C).
Les efficacités d’inhibition du LHVS dans les souches de type sauvage et decpl ont été déterminées en traçant des activités de luciferase contre huit concentrations d’inhibiteurs dans la figure 2. Il est essentiel d’inclure les cellules infectées sans traitement inhibiteur pour la normalisation des activités de luciferase brute dans l’essai. En outre, une expérience fictive effectuée dans une microplaque claire est nécessaire pour l’essai pour s’assurer que les parasites sont encore dans le stade intracellulaire à la fin de la période d’essai.
Dans la figure 3, la génération et la validation d’une construction d’expression sgRNA ciblant TgCPL et la production d’un modèle de réparation pour la suppression de TgCPL sont montrées. Le 20 bp sgRNA correspondant au gène TgUPRT encodé dans le plasmide original a été muté à la séquence d’ADN ciblant le gène TgCPL via la mutanèse dirigée par site PCR. Pour ce faire, les séquences d’ADN codage pour les sgRNAs qui reconnaissent différents gènes ont été conçus à l’apprêt avant, tandis que l’apprêt inverse a été maintenu inchangé pour simplifier la conception de l’amorce.
La figure 3A montre une région zoomée des séquences d’ADN sgRNA ciblant le gène TgUPRT dans le modèle original plasmide ainsi que l’ensemble d’apprêt utilisé pour la génération du vecteur d’expression sgRNA linéaire. La figure 3B montre une image représentative de gel du plasmide linéaire d’expression de sgRNA- TgCPL-ciblant. La figure 3C montre la digestion de l’endonuclease de restriction du plasmide circulaire d’expression de sgRNA- de TgCPL-ciblant. Une introduction inversée M13 a été utilisée pour séquencer l’ARN guide incorporé dans le vecteur d’expression sgRNA généré pour le gène spécifique. Dans la figure 3D, la région d’ADN séquencée a été alignée sur le modèle plasmide pour la confirmation de la mutanèse réussie. La figure 3E illustre les régions de début et de fin de la cassette de résistance à la pyriméthamine, montrant où les amorces peuvent anneal pour la production du modèle de réparation pour la suppression du gène TgCPL. Le modèle de réparation a été amplifié par PCR et chargé dans un gel d’agarose de 1 % pour la vérification de la taille et l’extraction du gel.
La stratégie globale pour la génération et le dépistage par KO de TgCPL est indiquée dans la figure 4. Trois ensembles d’amorces indiqués dans la figure 4A ont été utilisés pour filtrer les parasites de suppression TgCPLpour l’intégration correcte de 5'- et 3'-ARMs et la suppression de la séquence de codage TgCPL. Comme le montre la figure 4B, en général, sept à huit clones sont choisis pour le dépistage au départ. Le dépistage commence généralement par la vérification de la suppression de la séquence de codage pour le gène d’intérêt. Elle est suivie d’une détection de 5' et 3'-ARMs, ce qui permet de minimiser le nombre total de clones à dépister. Une vérification plus poussée par immunoblotting affichée dans la figure 4C peut être effectuée si un anticorps reconnaissant la protéine cible est disponible.

Figure 1 : Quantification de croissance intracellulaire pour les parasites de Toxoplasma utilisant une méthode basée sur la luciferase. (A) Raw luciferase activity readings in a spreadsheet software. Les lectures à 24 h, 48 h, 72 h et 96 h post-infection ont été normalisées contre les lectures initiales à 4 h post-infection pour calculer les changements de pli dans la croissance de parasite. (B) Les données normalisées ont été moyennes et tracées. (C) Les valeurs de journal2 des changements de pli ont également été tracées et soumises à la régression linéaire pour déterminer le temps de doublement du parasite. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
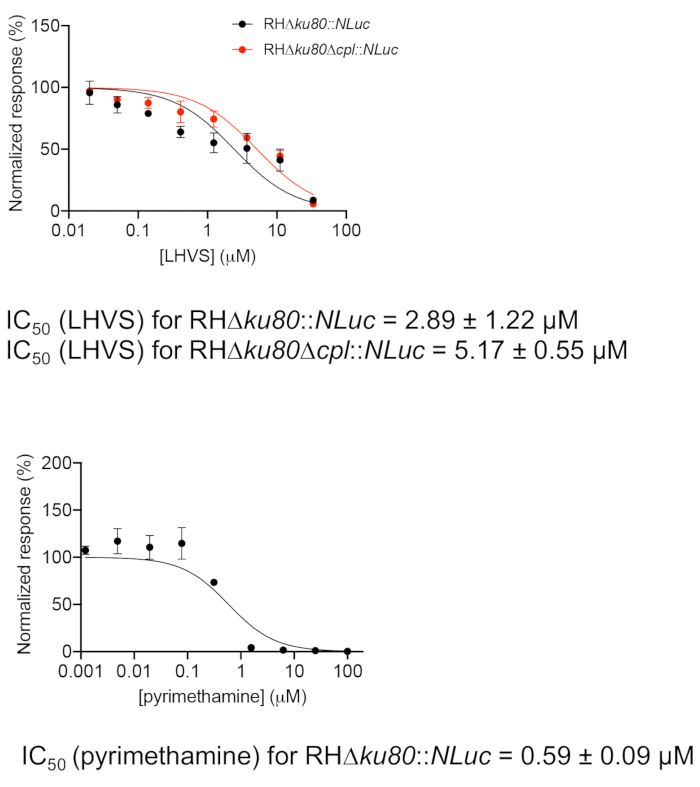
Figure 2 : Évaluation de l’efficacité de l’inhibition du LHVS et de la pyriméthamine utilisant l’essai de croissance à base de luciferase. Les parasites ont été inoculés dans une microplaque de puits de 96 pendant 4 h pour permettre l’invasion des cellules hôtes. Les parasites non envahis ont été emportés, et la plaque a été remplie de supports contenant différentes concentrations de LHVS ou de pyriméthamine et incubée pour un supplément de 96 h avant la détermination de l’activité de luciferase. Les lectures mesurées de luciferase pour des parasites traités avec des concentrations individuelles d’inhibiteur ont été normalisées contre le signal détecté des parasites non traités. Les données ont été tracées dans un programme de graphique, et une analyse de régression pour la détermination d’IC50 a été effectuée. L’essai a été répété en trois répliques biologiques avec trois répliques techniques chacune. Les données représentent la moyenne des répliques biologiques de seM, n et 3. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
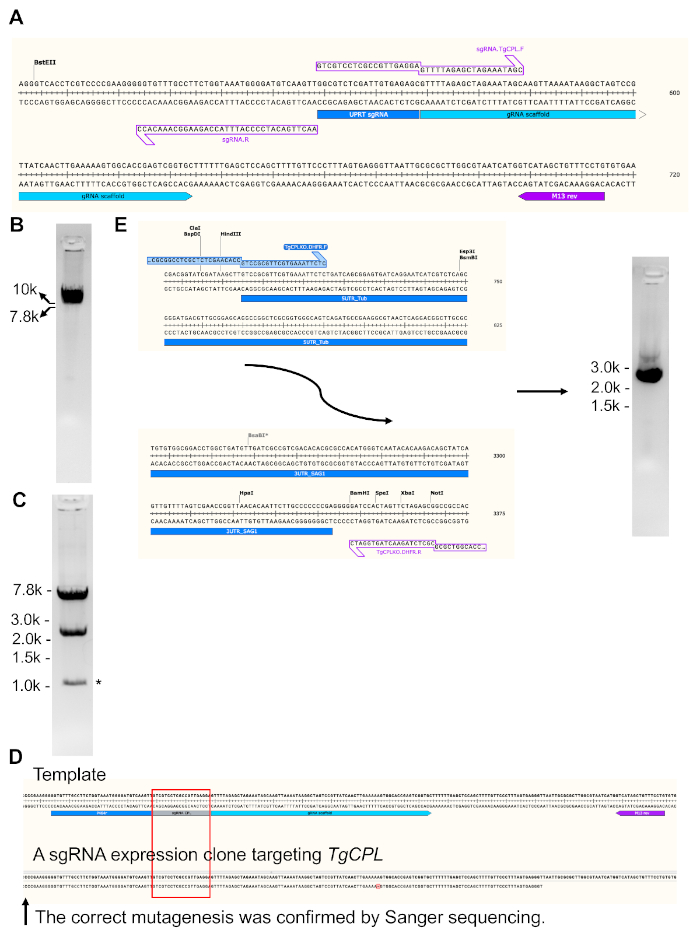
Figure 3 : Génération de la construction de plasmide exprimant le sgRNA ciblant TgCPL et la production d’un modèle de réparation pour la suppression de TgCPL. (A) Le pSAG1-Cas9-sgRNA-UPRTplasmid 23 original a été modifié via un kit de mutagenesis dirigé par le site pour le remplacement du sgRNA ciblant le gène TgUPRT à TgCPL. La région de codage sgRNA est agrandie pour montrer les zones auxquelles les amorces anneal. Après PCR, le plasmide muté a été linéaire et chargé dans un gel d’agarose de 1% pour la vérification de l’amplification réussie, suivie de l’extraction de gel. (B) L’image de gel de la construction d’expression linéaire sarisée PCR. (C) Après l’extraction de gel, le produit PCR a été circulaire et transformé par la suite en E. coli. Les clones contenant les plasmides attendus ont été examinés par la digestion d’endonuclease de restriction et le séquençage d’ADN. Les tailles de bande après la digestion d’ADN étaient 7.2 bp et 2.4 kb. Le groupe généré par le clivage non spécifique des endonucleases est étiqueté par astérisque. (D) L’amorce inversée M13 étiquetée dans la figure a été utilisée pour séquencer la région mutée d’ARN guide dans le vecteur d’expression sgRNA généré de TgCPL-ciblant. La région séquencée d’ADN a été alignée au modèle plasmide pour confirmer la mutagenesèse réussie. (E) Dans cette étude, 50 régions homologues de 50 bp correspondant aux 5'- et 3'-UTR de TgCPL ont été conçus dans les amorces pour l’amplification du modèle de réparation et flanqués aux 5'- et 3'-extrémités de la cassette de résistance à la pyriméthamine par PCR, respectivement. L’électrophoresis de gel d’agarose a été employé pour vérifier la taille correcte du produit de PCR avant l’extraction de gel. La taille prévue du modèle de réparation est de 2,7 kb. Habituellement, 5-6 g de modèle de réparation peuvent être obtenus à partir de 200 L de réaction PCR. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
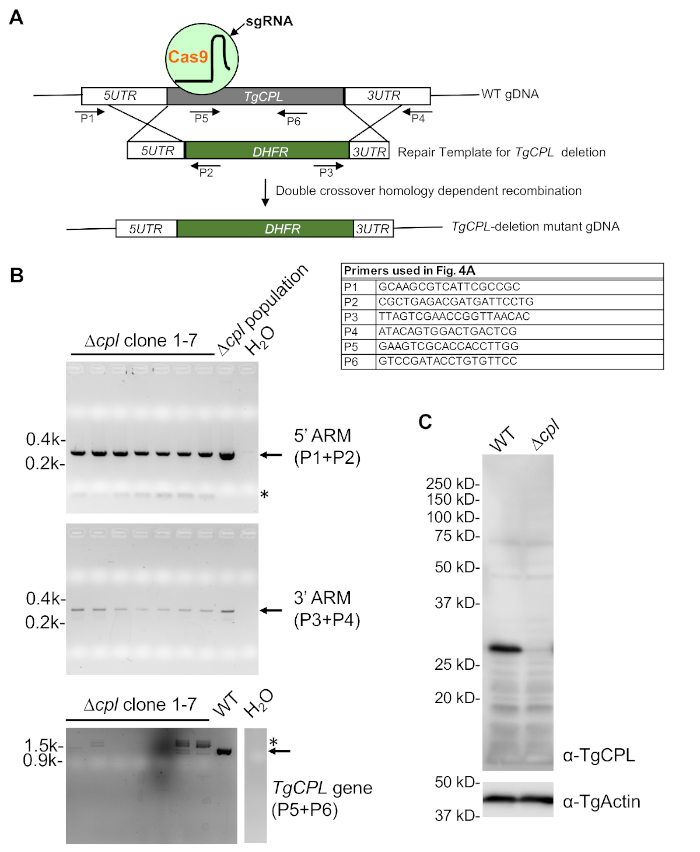
Figure 4 : Confirmation de PCR et d’immunoblotting des parasites TgCPL-déficients. (A) Un diagramme schématique illustrant les stratégies générales de TgCPL-suppression dans Toxoplasma et PCR-basé dépistage des clones tgCPL corrects. Les amorces utilisées pour le dépistage sont étiquetées. (B) PCR et agarose gel electrophoresis ont été utilisés pour sélectionner des clones contenant l’intégration correcte de la cassette de résistance à la pyriméthamine dans le locus TgCPL et la perte du gène TgCPL. L’ADN génomique de la populationde Cpl a servi de contrôle positif à la détection de 5'- et 3'-ARM, tandis que l’ADN génomique WT a été utilisé pour la détection du gène TgCPL comme un contrôle positif. L’eau a été utilisée au lieu du modèle d’ADN dans les réactions de PCR pour servir de contrôle négatif. Les bandes attendues sont dénotées par des flèches, tandis que les amplifications PCR non spécifiques sont étiquetées par des astérisques. (C) Clone 1 identifié par le criblage de PCR a été cultivé dans la culture de tissu pour la préparation de lysate de cellules et l’analyse supplémentaire d’immunoblotting pour confirmer la perte de l’expression de TgCPL dans le knock-out. TgActin a été utilisé comme contrôle de chargement. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| Luciferase: | Terminaison |
| Temps d’intégration : | 1 s |
| Ensemble de filtres - Émission: | Pleine lumière |
| Optique: | Retour au début |
| Gain: | 135 |
| Lire la vitesse: | Normal |
| Retard: | 100 ms |
| Lire la hauteur: | 4,5 mm |
Tableau 1 : Paramètres de lecteur de microplaque pour la mesure de l’activité de luciferase pendant l’essai de croissance de Toxoplasma à base de luciferase.
| Dénaturation initiale : | 98 oC pour 5 min |
| 25 cycles de | |
| Dénaturation: | 98 oC pour 5 s |
| Recuit: | 60 oC pour 15 s |
| Extension: | 72 oC pour 1 min |
| Extension finale : | 72 oC pendant 10 min |
Tableau 2 : Réglages thermocycler pour la génération du vecteur d’expression sgRNA.
| Échantillon | Volume (l) |
| Produit PCR (10-50 ng) | 1 |
| 2X KLD (kinase, ligase, DpnI) Zone tampon de réaction | 5 |
| 10X KLD Enzyme Mix | 1 |
| Eau sans nucléase | 3 |
| Total | 10 |
Tableau 3 : Recette de réaction pour la circularisation du vecteur d’expression sgRNA.
| Dénaturation initiale : | 98 oC pour 5 min |
| 35 cycles de | |
| Dénaturation: | 98 oC pour 15 s |
| Recuit: | 58 oC pour 15 s |
| Extension: | 72 oC pour 30 s par kb |
| Extension finale : | 72 oC pendant 10 min |
Tableau 4 : Réglage thermocycleur pour la génération du modèle de réparation.
| Échantillon | Volume (l) |
| TOTAL De l’ADN génomique de Toxoplasma | 1 |
| Apprêt avant (25 m) | 0.2 |
| Amorce inversée (25 m) | 0.2 |
| 2x PCR maître prémix | 5 |
| Eau sans nucléase | 3.6 |
| Total | 10 |
Tableau 5 : Recette de réaction Colony PCR pour le dépistage de clones Toxoplasma simples.
| Dénaturation initiale : | 98 oC pour 5 min |
| 35 cycles de | |
| Dénaturation: | 98 oC pour 5 s |
| Recuit: | 55 - 62 oC pour 5 s |
| Extension: | 72 oC pour 20 s par kb |
| Extension finale : | 72 oC pour 1 min |
Tableau 6 : Réglage thermocyclère pour le dépistage des clones Toxoplasma simples.
| Tampon Cytomix | 25 mM HEPES, pH 7,6, 120 mM KCl, 10 mM K2HPO4/KH2PO4, 5 mM MgCl2, 0,015 mM CaCl2, et 2 mM EGTA. |
| D10 moyen | DMEM 1X (Corning, Cat : 10-013-CV), 10 mM HEPES, 10 % (v/v) Sérum cosmique de veau (Hyclone, Chat : SH30087.03), 1 mM de pyruvate de sodium, 4 mM L-glutamine, 100 unités/mL de pénicilline, et 100 g/mL de sptocinmy. |
| Phenol milieu sans rouge | DMEM/ Hautement modifié (Hyclone, Cat : SH30284.02), 10 mM HEPES, 10% (v/v) Sérum cosmique de veau (Hyclone, Chat : SH30087.03), 1 mM de pyruvate de sodium, 4 mM L-glutamine, 100 unités/mL de pénicilline, et 100 g/mL de sptocinmy. |
| 2X NLuc Buffer | 100 mM MES, pH 6.0, 1mM CDTA, 0.5% Tergitol, 0.05% Mazu DF 204, 150 mM KCl, 1 mM DTT, 35 mM Thiourea. |
Tableau supplémentaire 1 : Recettes pour tampons.
Discussion
Ce protocole décrit un protocole basé sur la luciferase pour évaluer la croissance intracellulaire de Toxoplasma et évaluer l’efficacité de l’inhibition des composés chimiques contre la croissance des parasites. Comparée aux stratégies existantes disponibles pour mesurer la croissance intracellulaire de Toxoplasma, cette méthode présente une sensibilité et une spécificité élevées. Tout en surveillant la croissance des parasites, un essai simulé dans une microplaque claire de puits est recommandé pour confirmer que la souche testée n’analyse pas prématurément les cellules hôtes avant la fin de la période d’évaluation. Sinon, les lectures de luminescence ne refléteront pas exactement la croissance des parasites, puisque Toxoplasma ne se reproduit qu’à l’intérieur des cellules hôtes.
Il a été observé que le colorant rouge phénol étanche rapidement l’activité de luciferase, ce qui peut entraîner des différences significatives dans les lectures de luciferase parmi les répliques techniques en raison d’un retard dans les mesures individuelles de puits par le lecteur de plaque. Par conséquent, il est optimal de préparer des FFS dans un milieu sans phénol sans rouge avant l’ensemencement dans les 96 microplates de puits. En outre, dans le cas d’une forte activité de luciferase, l’interférence croisée peut conduire à des variations significatives entre les puits voisins présentant une forte activité de luciferase. Par conséquent, il est recommandé de placer une colonne vide entre chaque souche.
Plus précisément, pour le RHku80:: soucheNLuc, 1500 parasites sont inoculés dans chaque puits pour l’essai de croissance. Étant donné que le temps de doublement pour les parasites WT Toxoplasma est de 6-8 h24, on s’attend à une augmentation de l’activité de luciferase de 8 à 16 fois à 24 h post-infection. Cependant, certaines souches présentant des défauts de croissance importants ne donneront qu’une légère augmentation de l’activité de luciferase. Par conséquent, si l’inoculum parasite initial est faible, la variation inhérente à l’activité de luciferase masquera l’observation d’une augmentation de la luminescence au cours de la période de croissance. Par conséquent, il est recommandé d’inoculer un plus grand nombre de parasites pour obtenir un changement précis de pli pour les souches présentant des carences de croissance.
Dans le protocole, la conception de l’ARN guide suit les règles générales utilisées pour la modification du génome à base de CRISPR-Cas9 dans les cellules de mammifères25. Actuellement, de nombreux types de logiciels et de plates-formes en ligne fournissent des services pour la conception d’ARN guide dans divers organismes, tels que CHOPCHOP26, E-CRISP27, et EuPaGDT28. Ici, les logiciels commerciaux (Tableau des matériaux) sont utilisés pour concevoir le sgRNA. Par rapport aux programmes en ligne mentionnés précédemment, ce logiciel fournit un environnement local pour la conception de sgRNA. Il calcule les scores d’activité et de spécificité pour chaque candidat sgRNA à l’aide d’un algorithme précédemment publié25,29.
Techniquement, tout sgRNA situé dans un gène d’intérêt qui contient un score de spécificité élevé peut efficacement médiation du clivage de l’ADN génomique pour la recombinaison dépendante de l’homologie en aval. Dans la pratique, le sgRNA ciblant une région proche du codon de départ ou d’arrêt est préférable. Les gènes d’intérêt peuvent être endogènement épitopiques-marqués à l’aide du sgRNA, qui génère un écart à double brin à la fin du gène. Endogènement marquage d’un gène avant sa suppression aidera à confirmer la perte de gène par la détection d’immunoblotting dans le cas où un anticorps contre la protéine d’intérêt n’est pas disponible. L’épitope-marquage d’un gène peut également aider à déterminer l’emplacement subcellulaire de la protéine d’intérêt par microscopie d’immunofluorescence. En outre, si le gène cible est essentiel, sgRNA reconnaissant la région de départ du gène peut être utilisé pour le remplacement de son promoteur cognate à un promoteur tétracycline-sensible pour générer un KNOCK conditionnel.
En outre, le protocole décrit une technique pour la suppression des gènes dans Toxoplasma en remplaçant le gène TgCPL par une cassette de résistance à la pyriméthamine. En utilisant différents modèles de plasmides codant d’autres cassettes de résistance aux médicaments, les chercheurs peuvent modifier des séquences d’amorce pour incorporer d’autres gènes de résistance aux antibiotiques dans le modèle de réparation via PCR. En outre, ce protocole peut être modifié pour effectuer d’autres modifications du génome, telles que le marquage des gènes endogènes, le remplacement du promoteur et la mutanèse dirigée par le site. Il est à noter que les régions homologues utilisées dans ce protocole ne mesurent que 50 bpj. Une étude distincte a utilisé avec succès 40-43 séquences d’ADN homologues de 40-43 bp pour introduire des mutations mono-nucléotide et le marquage d’épitope de gène dans les parasites de Toxoplasma 22. Des séquences d’ADN homologues en si courte durée peuvent être facilement incorporées dans les amorces. Bien que nous n’ayons pas évalué quantitativement l’efficacité HDR pour cette longueur particulière de région homologue, il semble qu’une région de 40-50 bp est suffisante pour la recombinaison efficace de l’ADN dans la souche TgKu80-déficiente De Toxoplasma, comme en témoigne la manipulation génétique réussie de plusieurs gènes récemment atteint15,22.
Pendant la détermination d’efficacité des composés chimiques, si le gène cible de drogue potentiel est essentiel, une comparaison des décalages dans les valeurs d’IC50 entre les souches sauvages-type et knockout n’est pas pratique. Dans ce scénario, un essai mesurant directement la capacité des composés chimiques à inhiber l’activité des protéines recombinantes est nécessaire pour évaluer l’efficacité et la spécificité des médicaments. La littérature récente a rapporté les scores de forme physique des gènes individuels dans Toxoplasma en exécutant un écran CRISPR large de génome30, qui peut servir de guide pour aider à évaluer la difficulté de générer un mutant de KNOCK droite pour le gène d’intérêt.
Pris ensemble, le protocole décrit l’achèvement réussi d’un essai de croissance intracellulaire de Toxoplasma à base de luciferase et d’une stratégie d’évaluation pour les inhibiteurs chimiques contre la croissance de Toxoplasma. Un protocole d’édition du génome basé sur CRISPR-Cas9 pour la suppression des gènes chez les parasites toxoplasma, qui a été largement utilisé dans le domaine, est également détaillé. Les laboratoires individuels peuvent modifier le protocole décrit en fonction des besoins expérimentaux, tels que le marquage des gènes endogènes, le changement de marqueurs de sélection de médicaments, et la modification de la période d’évaluation pour la croissance intracellulaire de parasite.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs tient à remercier les Drs Sibley et Carruthers d’avoir partagé des anticorps pSAG1-Cas9-sgRNA-TgUPRT et anti-TgCPL et TgActin. Ce travail a été soutenu par le fonds Clemson Startup (à Z.D.), Knights Templar Eye Foundation Pediatric Ophthalmology Career-Starter Research Grant (à Z.D.), une subvention pilote d’une subvention COBRE NIH P20GM109094 (à Z.D.), et NIH R01AI143707 (à Z.D.). Les bailleurs de fonds n’avaient aucun rôle à jouer dans la conception, la collecte et l’analyse des données, la décision de publier ou la préparation du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
Références
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon