Method Article
Identificação de vias hospedeiras alvo de proteínas efetoras bacterianas usando toxicidade de levedura e telas supressoras
Neste Artigo
Resumo
Patógenos bacterianos secretam proteínas no hospedeiro que têm como alvo processos biológicos cruciais. Identificar as vias hospedeiras alvo de proteínas efeteroras bacterianas é fundamental para abordar a patogênese molecular. Aqui, um método usando um supressor de levedura modificado e tela de toxicidade para elucidar as vias hospedeiras alvo de proteínas efívoras tóxicas é descrito.
Resumo
Bactérias intracelulares secretam fatores de virulência chamados de proteínas efetentes no citosol hospedeiro que agem para subverter as proteínas hospedeiras e/ou suas vias biológicas associadas em benefício da bactéria. A identificação de proteínas de efetoras bacterianas putativas tornou-se mais gerenciável devido aos avanços no sequenciamento do genoma bacteriano e ao advento de algoritmos que permitem a identificação de silicos de genes que codificam candidatos de secreção e/ou eucarióticos Domínios. No entanto, a identificação desses importantes fatores de virulência é apenas um passo inicial. Naturalmente, o objetivo é determinar a função molecular das proteínas efetivas e elucidar como eles interagem com o hospedeiro. Nos últimos anos, técnicas como a tela de dois híbridos de levedura e imunoprecipitações em larga escala, juntamente com espectrometria de massa, ajudaram na identificação de interações proteína-proteína. Embora a identificação de um parceiro de ligação do hospedeiro seja o primeiro passo crucial para elucidar a função molecular de uma proteína efetora bacteriana, às vezes a proteína hospedeira tem múltiplas funções biológicas (por exemplo, actina, clathrina, tubulina) ou a proteína bacteriana não pode ligar fisicamente as proteínas hospedeiras, privando o pesquisador de informações cruciais sobre a via de hospedeiro precisa que está sendo manipulada. Uma tela modificada da toxicidade do fermento acoplada com uma tela do supressor foi adaptada para identificar os caminhos do anfitrião impactados por proteínas bacterianas do efetor. A tela da toxicidade confia em um efeito tóxico no fermento causado pela proteína do efetor que interfere com os caminhos biológicos do anfitrião, que manifesta frequentemente como um defeito do crescimento. A expressão de uma biblioteca genômica de levedura é usada para identificar fatores hospedeiros que suprimem a toxicidade da proteína efetora bacteriana e, assim, identificam proteínas na via que a proteína efetora tem como alvo. Este protocolo contém instruções detalhadas para a toxicidade e telas supressoras. Estas técnicas podem ser realizadas em qualquer laboratório capaz de clonagem molecular e cultivo de levedura e Escherichia coli.
Introdução
O primeiro relato de procedimentos semelhantes aos apresentados aqui caracterizou o efetor SidD, um deAMPylase que modifica Rab11. Técnicas comparáveis foram utilizadas para a caracterização de vários efetores de Pneumofilia L. 1,2,3. O ensaio foi adaptado para caracterizar umaproteínade efeito 4 tipo IV de Coxiella burnetii, e recentemente a utilidade desta técnica foi expandida para a caracterização das proteínas de membrana de inclusão de Clamídia trachomatis 5 .
Este protocolo pode ser dividido em duas partes principais: 1) a tela de toxicidade de levedura, em que a proteína efetora bacteriana de interesse é expressa em levedura e clones são rastreados para um fenótipo tóxico, como evidenciado por um defeito de crescimento, e 2) a tela supressor a levedura , em que o fenótipo tóxico é suprimido pela expressão de uma biblioteca genômica de levedura na cepa tóxica. Assim, a tela de toxicidade é uma tela para fenótipos tóxicos que se manifestam como defeitos de crescimento quando o efetor bacteriano de interesse é superexpresso. Clones tóxicos, transformados com sucesso e expressando o efetor bacteriano, são selecionados e guardados para o próximo passo. O segundo grande passo envolve a superexpressão de uma biblioteca genômica de levedura parcialmente digerida no clone tóxico de levedura. Plasmids que compõem a biblioteca genômica de levedura sugerida para o uso neste protocolo carregam inserções de 5 a 20 kb, geralmente correspondentes a quadros de leitura abertos de levedura de 3 a 13 (ORF) de um tamanho genético médio de ~1,5 kb em todos os plasmídeos, representando todo o genoma do fermento coberto aproximadamente 10x. Esta parte do ensaio é chamada a tela do supressor, porque o objetivo é suprimir a toxicidade da proteína bacteriana do efetor. Os plasmídeos potenciais do supressor são isolados do fermento, sequenciados, e dos ORFs de supressão identificados. A lógica subjacente à tela supressora é que a proteína efetora se liga, interage com, e /ou sobrecarrega componentes da via hospedeira que visa, e que fornecer essas proteínas hospedeiras de volta em excesso pode resgatar o efeito tóxico na via e, portanto, o defeito de crescimento. Assim, orfs identificados que suprimem a toxicidade muitas vezes representam vários participantes de uma via de acolhimento. Experimentos ortogonais são então realizados para verificar se o efetor bacteriano interage com a via implicada. Isto é especialmente necessário se um parceiro vinculativo, como clathrin ou actina foi identificado, porque essas proteínas estão envolvidas em uma infinidade de processos hospedeiros. Outras experiências podem então elucidar a função fisiológica da proteína efetiva durante a infecção. As telas de toxicidade e supressortambém são ferramentas poderosas para decifrar a função fisiológica de proteínas efetoras bacterianas que não ligam fisicamente proteínas hospedeiras com afinidades suficientes para detectar por imunoprecipitação ou que interagem com o host em interações enzimáticas hit-and-run que não podem ser detectadas por uma tela híbrida de levedura dois.
Embora a tela supressora possa ser um método poderoso para revelar potenciais interações fisiológicas entre proteínas efetoras bacterianas e vias hospedeiras, a proteína efetora bacteriana deve induzir um defeito de crescimento na levedura, caso contrário, usá-lo no tela supressor será de pouca utilidade. Além disso, o fenótipo tóxico deve resultar em pelo menos um déficit de crescimento de 2 a 3 log10 ou será difícil identificar supressores. Se um laboratório é criado para a cultura celular, proteínas efecionistas de triagem para toxicidade em linhas celulares comuns, como HeLa, muitas vezes pode dar uma visão sobre se vale a pena o esforço para prosseguir com a tela de toxicidade de levedura. Expressão ectópica da proteína efetora nas células HeLa, por vezes, resulta em toxicidade que se correlaciona muito fortemente com a toxicidade na cepa de levedura usada para estas telas4. Características observáveis de estresse nas células HeLa incluem perda de fibras de estresse, desprendimento celular da placa e condensação nuclear indicando apoptose. Qualquer indicação visual de estresse nas células HeLa tornar a proteína de interesse um bom candidato para induzir um defeito de crescimento na levedura, que se replicam muito mais rapidamente e são, portanto, mais sensíveis à perturbação de vias essenciais.
Note-se que a tela supressora nem sempre identifica parceiros de ligação do hospedeiro como supressores, mas ainda pode implicar componentes críticos da via de hospedeiro (s) direcionada, produzindo uma visão holística dos processos biológicos que estão sendo sequestrados proteína efetora bacteriana. Na superfície, isso parece contraintuitivo, porque fornecer o parceiro de ligação da proteína efetiva em excesso seria esperado para resgatar o defeito de crescimento. Nos esforços para identificar caminhos visados pela proteína c. trachomatis efetivo CT229 (CpoS), que se liga a pelo menos 10 diferentes Rab GTPases durante a infecção5, nenhum dos parceiros de ligação Rab suprimiu a toxicidade do CT229. No entanto, foram identificados numerosos supressores envolvidos no tráfico de vesículas revestidas de clathrin (CCV), o que levou a um trabalho adicional demonstrando que o CT229 subverte especificamente o tráfico de CCV dependente da Rab. Da mesma forma, ao investigar a proteína efetora C. burnetii Cbu0041 (CirA) vários Rho GTPases que resgataram o defeito de crescimento da levedura foram identificados, e mais tarde descobriu-se que a CirA funciona como uma proteína ativadora de GTPase (GAP) para RhoA4.
A utilidade da tela supressora de levedura para elucidar as vias hospedeiras alvo de proteínas efívoras não pode ser exagerada, e outros pesquisadores que tentam caracterizar proteínas efívoras intracelulares podem se beneficiar muito estas técnicas. Estes ensaios são de valor se as imunoprecipitações e/ou telas híbridas de levedura dois não conseguiram encontrar um parceiro vinculativo e podem elucidar quais as vias que são alvo da proteína efetora bacteriana. Aqui, protocolos detalhados para a toxicidade e telas supressoras para identificar as vias biológicas do hospedeiro alvo de proteínas efetoras bacterianas intracelulares são fornecidos, bem como alguns dos obstáculos comuns experimentados ao usar esses ensaios e seus soluções correspondentes.
Protocolo
1. Preparação de mídia e reagentes
NOTA: As placas devem ser preparadas antes do dia do ensaio e são boas para 1 mês. Os meios e os reagents podem ser feitos em qualquer ponto e são bons por 1 mês.
- Prepare 1 L da solução de glicose (10% w/v) dissolvendo 100 g de D-(+)-glicose em 800 mL de água destilada em um copo de 1.000 mL. Ajuste o volume para 1 L com água destilada. Filtre através de um filtro estéril de 0,2 μm em uma garrafa de armazenamento de mídia estéril 1 L.
- Prepare 1 L da solução de galactose (10% w/v) dissolvendo 100 g de D-(+)- galactose em 800 mL de água destilada em um copo de 1 L. Ajuste o volume para 1 mL usando água destilada e filtre através de um filtro estéril de 0,2 μm em uma garrafa de armazenamento de mídia estéril de 1.000 mL.
- Prepare 1 L de extrato de levedura peptone dextrose (YPD) ágar dissolvendo 10 g de extrato de levedura, 20 g de peptone, 20 g de glicose e 20 g de ágar em 1.000 mL de água destilada. Autoclave por 20 min e arrefecer em 56 °C banho de água até esfriar. Despeje em placas de 100 mm usando ~ 20 mL de mídia por prato.
- Prepare 1 L de caldo de YPD dissolvendo 10 g de extrato de levedura, 20 g de peptone e 20 g de glicose em 1.000 mL de água destilada. Autoclave por 20 min.
- Prepare o recil sintético (SD) uracil (Ura-) agar de glicose. Para 1 L, dissolva 6,7 g de base de nitrogênio levedura sem aminoácidos, 1,9 g de suplemento de abandono sem uracil e 15 g de ágar em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56 °C até que a temperatura é ~ 50-60 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro graduado estéril, adicione 200 mL da solução da glicose preparada na etapa 1.1. Misture bem rodando suavemente ou coloque no prato de agitação. Despeje em placas de 100 mm usando ~ 20 mL de mídia por prato.
- Prepare o retocil sintético (SD) uracil (Ura-) ágar de galactose. Para 1 L, dissolva 6,7 g de base de nitrogênio levedura sem aminoácidos, 1,9 g de suplemento de abandono sem uracil e 15 g de ágar em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56 °C até que a temperatura é ~ 50-60 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro estéril graduado, adicione 200 mL da solução de galactose preparada na etapa 1.2. Misture bem rodando suavemente ou coloque no prato agitar. Despeje em placas de 100 mm usando ~ 20 mL de mídia por prato.
- Prepare o dcaldo de glicose de abandono sintético (SD) (Ura-). Para 1 L, dissolva 6,7 g de base de nitrogênio levedura sem aminoácidos e 1,9 g de suplemento de abandono sem uracil em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56 °C até que a temperatura é ~ 50-60 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro estéril graduado, adicione 200 ml da solução de glicose preparada na etapa 1.1.
- Prepare o retocil sintético (SD) uracil (Ura-) leucina (Leu-) gara de glicose. Para 1 L, dissolva 6,7 g da base de nitrogênio levedura sem aminoácidos; 1,9 g de suplemento de abandono sem uracil, leucina e triptofano; 15 g de ágar; e 0,076 g de triptofano em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56 °C até que a temperatura é ~ 50-60 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro graduado estéril, adicione 200 mL da solução da glicose preparada na etapa 1.1. Despeje em placas de 100 mm usando ~ 20 mL de mídia por prato.
- Prepare abandono sintético (SD) uracil (Ura-) leucina (Leu-) ágar galactose. Para 1 L, dissolva 6,7 g de base de nitrogênio levedura sem aminoácidos; 1,9 g de suplemento de abandono sem uracil, leucina e triptofano; 15 g de ágar; e 0,076 g de triptofano em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56 °C até que a temperatura é ~ 50-60 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro estéril graduado, adicione 200 mL da solução de galactose preparada na etapa 1.2. Misture bem rodando suavemente ou colocando em uma placa de agitação. Despeje em placas de 100 mm usando ~ 20 mL de mídia por prato.
- Prepare abandono sintético (SD) uracil (Ura-) leucina (Leu-) caldo de glicose. Para 1 L, dissolva 6,7 g de base de nitrogênio levedura sem aminoácidos; 1,9 g de suplemento de abandono sem uracil, leucina, triptofano; e 0,076 g de triptofano em 800 mL de água destilada. Autoclave por 20 min e arrefecer em um banho de água de 56-60 °C até que a temperatura é ~ 50 °C. Usando uma pipeta sorológica de 50 mL ou um cilindro graduado estéril, adicione 200 mL da solução da glicose preparada na etapa 1.1.
- Prepare a solução de polietilenoglicol (PEG). Adicione 50% w/v de polietileno glicol 3350 em água destilada. Esterilizar por autoclismo.
- Prepare 1 M de acetato de lítio (LiAc) dissolvendo 10,2 g de destilação de acetato de lítio em 200 mL de água destilada. Esterilizar por autoclismo.
- Prepare o DNA do esperma de arenque. Diluir 10 mg/mL de arenque esperma to 2 mg/mL usando água destilada. Aqueça a 100 °C por 5 min e coloque imediatamente no gelo por 5 min.
2. Cloning o gene do interesse na toxicidade do fermento plasmid pYesNTA-Kan
NOTA: Atualmente, há uma variedade de vetores de toxicidade de levedura disponíveis comercialmente e academicamente. A tela supressora de levedura pode ser usada em conjunto com muitos desses vetores desde que o plasmídeo expressando a proteína efente de interesse usa uma seleção de abandono que não uracil e um marcador antibiótico que não BlaR. Este trabalho utilizou um vetor pYesNTA modificado4,5 que inclui uma fita de resistência à kanamicina para uma triagem mais fácil para potenciais supressores. O esquema de clonagem deve permitir que a proteína efetora seja in-frame com o promotor Gal e His-Tag. A chlamydia trachomatis inclusão membrana proteína CT229 foi usado como uma prova de princípio para estes ensaios.
- Use o PCR para amplificar o CT229 do DNA genômico C. trachomatis seguindo as instruções do fabricante usando cartilha CT229 +1 Kpn F (CCGGTACCAATGAGCTGTTCTAATTTAATTTCAGGT) e CT229 XhoI R (CCCTCGAGTTTTTTTTTTTTCGGGGGATGCC) primers. Use as seguintes condições de PCR: (1) 98 °C para 30 s; (2) 98 °C para 10 s, 55 °C para 30 s, 72 °C por 2 min; (3) repetir o passo 2 para um total de 25x; (4) 72 °C por 10 min; e (5) 4 °C de porão.
- Analise 5 μL do produto PCR em um gel agarose de 1% (Figura 1).
- Purifique os 45 μL restantes do ADN usando um jogo da purificação do PCR que segue as instruções do fabricante.
- Dime os 50 μLs de inserção de PCR purificada e pYesNTA-Kan (Figura 2) para 1 h a 37 °C em um banho de água usando KpnI-HF e XhoI-HF.
NOTA: Para pYesNTA-Kan, digere 5 μg de plasmídeo usando 5 μL de KpnI-HF, 5 μl de XhoI-HF e 6 μL de buffer em um volume total de 60 μL. Para a inserção, digere os 50 μL inteiros do produto purificado do PCR usando 2 μL de KpnI-HF, 2 μL de XhoI-HF, e 6 μL do amortecedor. - Executar todo o plasmídeo digerir em um gel agarose 1%. Realize a purificação do plasmídeo digerido usando um kit de extração de gel seguindo as instruções do fabricante.
- Purifique a inserção de PCR digerido usando um kit de purificação PCR seguindo as instruções do fabricante.
- Clonar a inserção em pYesNTA-Kan usando 2 μL de pYesNTA-Kan (passo 2.5), 2 μL de dna ligase buffer, 1 μL de t4 ligase, e 15 μL de inserção (passo 2.6). Incubar à temperatura ambiente por 1 h.
- Adicione toda a reação de clonagem a 50 μL de células competentes de E. coli e realize a reação de transformação.
- Placa toda a reação de transformação em uma placa LB contendo 100 μg/mL de carbenicilina.
- Inocular 10 mL do LB contendo 100 μg/mL de carbenicilina com três colônias individuais. Incubar durante a noite em 37 °C com agitação em 150 rpm.
- Isolar o plasmídeo usando um kit de minipreparação plasmid seguindo as instruções do fabricante.
- Sequenciar os plasmídeos isolados usando primers específicos do gene (passo 2.1).
3. Teste a proteína de interesse para a toxicidade em levedura
- Streak Saccharomyces cerevisiae W303 em YPD ágar (passo 1.3) para obter colônias isoladas. Incubar a 30 °C para 24 h.
- Inocule 10 mL de caldo de YPD (passo 1.4) com uma única colônia da placa ágar (passo 3.1). Incubar a 30 °C com agitação em 150 rpm durante a noite.
- Adicione 0,5 mL da cultura durante a noite para 10 mL de caldo de YPD (passo 1.4) e incubar a 30 °C com agitação em 150 rpm para 4 h.
- Pelotas os 10 mL de cultura a 3.000 x g por 10 min a 4 °C.
- Resuspenda a pelota em 1 mL de água estéril, transfira para o tubo de microcentrífuga e pelota a 3.000 x g por 1 min à temperatura ambiente.
- Resuspenda a pelota em 1 mL de 1 mm de acetato de lítio (LiAc) e pelota a 3.000 x g por 1 min à temperatura ambiente.
- Repita a lavagem LiAc 2x.
- Retire a lavagem. Resuspenda a pelota em 2,4 mL de 50% PEG 3350. Adicione 360 μL de 1M LiAc, 500 μL de 2 mg/mL de DNA de esperma de arenque e 400 μL de água estéril.
NOTA: Isso é suficiente para 20 transformações. - Adicione 180 μL da mistura de transformação do passo 3.3.5 para um tubo de microcentrífuga contendo 5 μL de pYesNTA-Kan CT229 plasmid DNA (100-500 ng) do passo 2.12.
- Incubar em um banho de água a 30 °C por 30 min.
- Incubar em um banho de água em 42 °C por 30 min.
- Pelota a 3.000 x g para 1 min à temperatura ambiente.
- Retire a mistura de transformação com a pipeta. Resuspenda a pelota em 100 μL de água estéril. Placa a transformação em SD Ura- ágar com glicose (passo 1.5).
- Incubar placas a 30 °C para 48 h.
- Inocular 5 mL de SD Ura- caldo contendo glicose (passo 1.7) com uma única colônia da placa. Incubar durante a noite a 30 °C com agitação em 150 rpm. Inclua levedura transformada apenas com o vetor como um controle negativo.
- Adicione 180 μL de água estéril a 5 poços de uma placa de 96 poços (A2-A6).
- Vortex a cultura durante a noite para misturar.
- Adicione 180 μL de levedura ao primeiro poço (A1). Dilui-se em série 1:10 (seis amostras no total, incluindo não diluídas).
- Usando uma pipeta multicanal, local 5 μL de cada diluição em SD Ura- glicose (passo 1,5) e Ura- placas de ágar do passo 1,6. Incubar a 30 °C para 48 h.
- Avalie a toxicidade comparando o crescimento do fermento expressando a proteína efetiva de interesse cultivada na mídia contendo galactose com o crescimento da levedura expressando o vetor sozinho.
- Confirme a expressão da proteína de fusão His-tag por borrão ocidental.
4. Transforme levedura tóxica com a biblioteca genômica de levedura
- Inocular 100 mL de SD Ura- caldo de glicose (passo 1,7) usando 1 mL do estoque a partir do passo 3.4. Incubar por 16-24 h a 30 °C com agitação em 150 rpm. Coloque 900 mL de SD Ura- caldo de glicose a 30 °C durante a noite para prewarm a mídia.
- Adicione os 100 mL inteiros da cultura durante a noite ao frasco prewarmed de 1 L. Incubar por 4-5 h a 30 °C com agitação em 150 rpm.
- Pelotas a cultura em 6.000 x g para 10 min em 4 °C.
- Descarte o supernatant. Resuspenda a pelota em 250 mL de água estéril. Pelotas a cultura em 6.000 x g para 5 min em 4 °C.
- Descarte o supernatant. Resuspenda a pelota em 250 mL de 1 mM LiAc. Pelotas a cultura em 6.000 x g para 5 min em 4 °C.
- Retire o LiAc e resuspenda a pelota em 9,6 mL de 50% PEG 3350. Adicione 1,44 mL de 1M LiAc, 2 mL de 2 mg/mL de dna de esperma de arenque, 50 μg da biblioteca genômica pYep13 (ATCC 37323). Ajuste o volume para 15 mL com água estéril. Misture suavemente por inversão.
- Incubar em um banho de água a 30 °C por 30 min.
- Adicione 750 μL de dimetil sulfoxida (DMSO) para aumentar a eficiência da transformação. Incubar em um banho de água a 42 °C por 30 min. Misture por inversão suave a cada 10 min.
- Pelota o fermento em 3.000 x g para 5 min à temperatura ambiente.
- Descarte o supernatant e resuspenda a pelota em 10 mL de água estéril. Pelota a 3.000 x g por 5 min à temperatura ambiente.
- Resuspenda a pelota em 8 mL de água estéril.
- Para determinar a eficiência da transformação, diluir 1:10 e placa 100 μL de cada diluição em SD Ura- Leu- gara de glicose (passo 1,8).
- Placa 200 μL da amostra em Placas sd Ura- Leu- galactose agar (passo 1.9). Use 50 placas no total.
- Incubar a 30 °C para 48-96 h ou até que as colônias aparecem.
NOTA: Colônias geralmente aparecem em placas de ágar de glicose em 48-72 h e placas de ágar de galactose em 72-96 h.
- Colônias de remendos (resgates potenciais) em SD Ura- Leu- ágar de galactose (passo 1.9) para expandir e fazer estoque. Incubar a 30 °C para 24-48h.
- Inocular 5 mL de SD Ura- Leu- caldo de glicose (passo 1.10) usando a parte do patch. Incubar durante a noite a 26 °C com agitação em 150 rpm.
- Adicione 180 μL da água estéril a 5 poços de uma placa de 96 poços (A2-A6).
- Vortex a cultura durante a noite para misturar.
- Adicione 180 μL de levedura ao primeiro poço (A1). Dilui-se em série 1:10 (seis amostras no total, incluindo não diluídas).
- Usando uma pipeta multicanal, local 5 μL de cada diluição em SD Ura- glicose e SD Ura- placas de ágar de galactose. Inclua o efetor tóxico sozinho como um controle.
- Incubar a 30 °C para 48 h.
- Compare o crescimento do fermento expressando o efetor tóxico sozinho ao fermento que contem os supressores potenciais. Apenas proceder com potenciais supressores que diminuíram a toxicidade em comparação com o fermento expressando o efetor sozinho.
5. Identificar e confirmar supressores
- Inocular 5 mL de SD Ura- Leu- caldo de glicose (passo 1,10) com 100 μL de levedura a partir do passo 4.4 e incubar durante a noite em 30 °C com agitação em 150 rpm.
- Pelota o fermento em 3.000 x g para 2 min à temperatura ambiente. Descarte suavemente o supernatant usando uma pipeta.
- Isolar o plasmídeo com um kit de minipreparação plasmid levedura seguindo as instruções do fabricante.
- Transforme o plasmid isolado nas células competentes de E. coli e repercia toda a transformação no ágar lb com 100 μg/mL de carbenicilina. Incubar a 37 °C para 24 h.
- Inocular 10 mL de caldo LB contendo 100 μg/mL de carbenicilina, com três colônias da placa e incubada a 37 °C durante a noite com agitação em 150 rpm.
- Culturas de pelotas a 3.000 x g por 10 min à temperatura ambiente. Isolar plasmid usando um kit miniprep seguindo as instruções do fabricante.
- Retransforme o fermento tóxico com os plasmídeos isolados da etapa 5.3.1 que seguem as etapas na parte 3 do protocolo.
- Inoculate 5 mL de SD Ura- Leu- caldo de glicose com uma colônia da placa de transformação. Incubar durante a noite a 30 °C com agitação em 150 rpm.
- Spot on Ura- Leu- glicose e galactose ágar para confirmar a supressão da toxicidade.
- Sequência usando cartilha pYep13 F (ACTACGCGATCATGGCGA) e pYep13 R (TGATGCGCGCGCGATGC) primers para identificar ORFs de levedura.
Resultados
Antes que a tela real do supressor do fermento possa ser executada, a proteína do efeitor do interesse deve ser testada para a toxicidade no fermento. Isto é conseguido expressando a proteína do interesse no fermento o controle de um promotor galactose-indutor. O crescimento da glicose (condições não indutoras) deve primeiro ser comparado para garantir que a toxicidade seja especificamente devido à expressão da proteína de interesse e não seja um defeito geral. Como mostrado na Figura 3, a toxicidade se manifesta como colônias menores e/ou crescimento reduzido. Uma diminuição de log 2-3 no crescimento do fermento é ideal para as telas supressoras de levedura.
Como mostrado na Figura 4,a toxicidade e a tela supressora podem ser realizadas em oito etapas usando os métodos descritos na seção de protocolo. O gene do interesse é clonado em um vetor apropriado1,e as construções resultantes são transformadas no fermento para avaliar a toxicidade. Este estudo usou ct229 como o gene do interesse e pYesNTA-Kan como o vetor. Além disso, a expressão da proteína de fusão sua marcação foi confirmada pela mancha ocidental. Idealmente, uma redução de log 2-3 em leveduras cultivadas em mídia contendo galactose deve ser observada. Se a proteína de interesse é tóxica para levedura (Figura 3 e Figura 4), a tela supressora pode ser conduzida transformando a cepa tóxica com a biblioteca genômica de levedura pYep13. Os transformantes são banhados em ágar de glicose para determinar a eficiência da transformação e a gara de galactose para identificar potenciais supressores. Usando este protocolo, uma eficiência mínima da transformação de 5 x 105 foi conseguida com um total de 10-250 colônias no ágar da galactose. Remendar essas colônias em SD Ura- Leu- galactose agar (Figura 4)auxiliado na identificação de verdadeiros supressores, porque muitos falsos positivos não vai crescer quando corrigido. Aqui, 50 colônias foram remendadas e oito clones que suprimiram a toxicidade efetividade foram obtidos(Figura 4). Detectar supressores em SD Ura- Leu- galactose ágar foi feito para confirmar a supressão da toxicidade. Como mostrado na Figura 4, pSup1 e pSup 2 suprimiram a toxicidade da proteína efetora, enquanto pSup3 não. Portanto, o pSup3 foi descartado. Plasmídeos foram posteriormente isolados dos supressores e transformados em E. coli para aumentar o rendimento plasmídeo. O plasmídeo pode então ser retransformado no fermento tóxico para confirmar que o plasmídeo isolado suprime definitivamente a toxicidade do efeto(figura 4). Quando a maioria dos plasmídeos isolados derem o salvamento a toxicidade, ocasionalmente um plasmídeo que não suprima a toxicidade quando retransformado na tensão tóxica do fermento é obtido. Esses plasmídeos são descartados, e apenas aqueles que suprimem a toxicidade após a retransformação devem ser sequenciados.
O plasmídeo isolado conterá orfs de leveduramúltipla4. Para identificar qual é o verdadeiro supressor, cada ORF pode ser clonado individualmente e expresso na cepa de levedura tóxica, como descrito anteriormente1,2,3,4.

Figura 1: Amplificação pcr do gene alvo. CT229 de Chlamydia trachomatis serovar L2 foi PCR amplificado a partir de DNA genômico. Os produtos pcr foram analisados em um gel agarose de 1% manchado com brometo de etídio. Clique aqui para ver uma versão maior deste número.
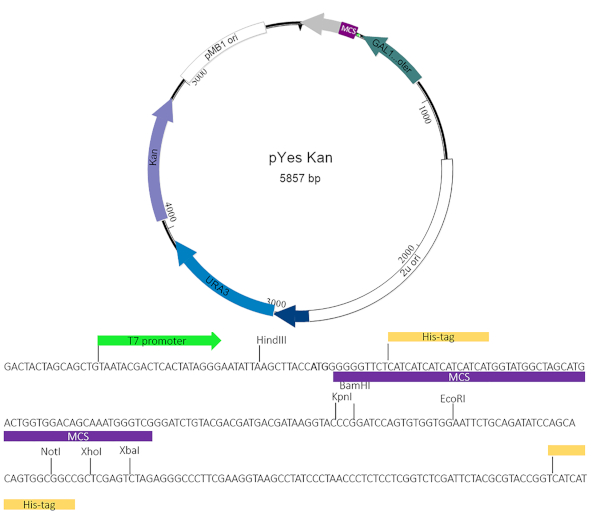
Figura 2: mapa plasmídeo pYesNTA-Kan. O gene alvo de interesse foi clonado no local de clonagem múltipla (MCS) de pYesNTA-Kan. Kanamycin foi usado para seleção em E. coli e uracil abandono foi usado para seleção em S. cerevisiae. A expressão da proteína de fusão sua-etiquetada foi verificada pela borração ocidental. Clique aqui para ver uma versão maior deste número.

Figura 3: Resultados representativos da toxicidade do fermento. As proteínas efentes de interesse foram clonadas em plasmídeos verificados em sequência syesnta-kan. Clique aqui para ver uma versão maior deste número.

Figura 4: Visão geral da tela supressora de levedura. O gene alvo de interesse foi clonado em pYesNTA-Kan e plasmídeos foram transformados em S. cerevisiae. Os transformantes foram diluídos em série e manchados em Ura- ágar da glicose e da galactose para avaliar a toxicidade. Para identificar o caminho alvejado pela proteína tóxica do efetor, a tensão do fermento que expressa a proteína tóxica foi transformada com uma biblioteca genômica do fermento. Colônias foram remendadas em Ura- Leu- ágar de galactose e avistadas para avaliar a toxicidade. Plasmídeos foram isolados daqueles que suprimem a toxicidade da proteína efetora. Plasmídeos foram retransformados na cepa de levedura tóxica para confirmar que o plasmídeo isolado é um supressor. Plasmídeos de supressores foram sequenciados para identificar os ORFs de levedura presentes. Clique aqui para ver uma versão maior deste número.
Discussão
Este protocolo descreve procedimentos passo a passo para identificar vias biológicas do hospedeiro alvo de proteínas efívoras usando uma toxicidade de levedura modificada e tela supressora. A cepa de levedura utilizada, S. cerevisiae W303, é auxotrófica tanto para uracil quanto para a leucina. A auxofia uracil da cepa é usada para selecionar levedura carregando a proteína de interesse no vetor pYesNTA-Kan, enquanto a auxofia leucine é usada para selecionar para o vetor de biblioteca genômica de levedura pYep13. Os plasmids genômicos da biblioteca do fermento carregam 3-13 ORFs, assim que cada ORF deve individualmente ser clonado no vetor6 do p415-ADH ou em um vetor similar e retransformado no fermento tóxico para identificar qual é o supressor verdadeiro. É típico identificar vários supressores representando uma via biológica hospedeira. Supressores às vezes podem ser parceiros de ligação direta da proteína bacteriana de interesse, mas nem sempre.
Gerar clones de levedura carregando a proteína bacteriana de interesse em pYesNTA-Kan é o primeiro grande passo e muito cuidado deve ser tomado para preservar a integridade desses clones o mais rapidamente possível, porque há forte seleção negativa para levedura carregando tóxico proteínas, mesmo sem indução intencional de galactose, e podem perder o plasmídeo. Ocasionalmente há uma perda do plasmídeo mesmo quando o fermento é mantido a seleção em meios do abandono. Um método foi relatado previamente para diminuir a ocorrência da perda do plasmídeo linearizando o vetor e integrando o dentro do genoma1do fermento.
Uma grande desvantagem dessas técnicas é que a tela supressora só produz dados significativos se a superexpressão da proteína efetora bacteriana desencadear um defeito de crescimento observável e consistente no fermento. A principal dificuldade neste método é quando não são identificados supressores. É difícil determinar se a falha em identificar supressores é devido a razões biológicas ou problemas técnicos. Do ponto de vista biológico, pode haver várias causas: a proteína de interesse pode ser tão tóxica que as proteínas da biblioteca genômica não são capazes de suprimir significativamente a toxicidade, a via direcionada pode ser incapaz de resgate, ou numerosos co-expressos fatores podem ser necessários para superar o efeito tóxico. Abordar a explicação biológica é um desafio, pois há muitas variáveis indefinidas para dissecar. Porque há um protocolo estabelecido, explorar explicações técnicas é uma abordagem muito mais tratável. Do ponto de vista técnico, a falha em identificar supressores é provavelmente o resultado da baixa eficiência de transformação da biblioteca genômica de levedura. Este protocolo utiliza a biblioteca genômica ATCC S. cerevisiae AB320 parcialmente digerida em pYEp13. Outras bibliotecas podem ser suficientes, bem como outros vetores, mas este protocolo usa especificamente a biblioteca ATCC e não há estudos publicados atualmente que relatam biblioteca alternativa ou fontes vetoras. Recomenda-se fortemente que a cobertura do genoma do fermento deve ser pelo menos 10x, ou a sub-representação pode impedir a identificação de supressores. A diminuição da eficiência da transformação pode trazer esses números abaixo de 1x. Assim, partes do genoma do fermento podem não estar representadas na tela do supressor. A baixa eficiência da transformação pode ser devido a muitas edições, incluindo reagents maus ou preparação pobre da biblioteca. Este método adere pela maior parte ao método da transformação de DMSO/LiAc posto primeiramente adiante pelo monte eoutros7. Recomenda-se que o novo DMSO, 50% PEG e dna de esperma de arenque fresco sejam comprados e preparados para as transformações e usados exclusivamente para esses ensaios para diminuir a contaminação ou degradação da qualidade dos reagentes. DMSO é altamente hygroscópico e absorve a água da atmosfera prontamente. Portanto, fazer muitos alíquotas de uso individuais é recomendado para evitar a abertura repetida da tampa de um recipiente e exposição à atmosfera. DNA de esperma de arenque de alta qualidade pode ser armazenado a -20 °C e permanece apto para uso por vários anos, embora uma nova preparação deva ser preparada a partir do estoque para cada transformação. Uma vez cozido e resfriado, o DNA de esperma de arenque não deve ser refervescente ou reutilizado, por isso é melhor remover apenas o que é necessário e evitar fazer mais do que o necessário para a transformação atual. A atenção aos detalhes pode melhorar muito a eficiência de transformação do fermento e melhorar as chances de identificar um supressor.
Considerando que o vetor de levedura pYep13 transporta 3 a 13 ORFs de levedura diferentes, reduzir o número de plasmídeos falso-positivos é fundamental para identificar supressores. Levedura pode pegar e abrigar várias cópias de vetores semelhantes, enquanto a sabedoria geral associada à incompatibilidade plasmídeo tradicionalmente dita que E. coli só mantém um tipo de plasmídeo com a mesma origem de replicação (ORI) de cada vez, embora este nem sempre é o caso. Isso pode se tornar um grande problema ao tentar identificar candidatos supressores. Se clones supressores de levedura forem identificados, os plasmídeos serão extraídos e retransformados em E. coli para propagação e sequenciamento subsequente para identificação. Uma vez que o candidato plasmid supressor foi isolado e retransformado em E. coli, é fortemente recomendado que pelo menos cinco colônias de E. coli são escolhidos para sequenciamento, porque é possível que clones individuais podem estar carregando plasmídeos diferentes. Desde E. coli deve somente poder propagar um plasmid de cada vez, se a isolação do fermento produz plasmids múltiplos, os clones diferentes de E. coli na placa devem carreg somente um deles de cada vez. Portanto, escolher cinco colônias com base na eficiência de transformação normal em E. coli deve mostrar se os clones estão carregando os mesmos plasmídeos ou diferentes. Se os plasmídeos múltiplos se levantam subseqüente ao sequenciamento dos plasmídeos de E. coli,a seguir cada um deve ser transformado no clone tóxico do fermento e avaliado para o salvamento do defeito do crescimento. Além disso, mesmo que apenas um único plasmúdor supressor seja identificado, para rigor e reprodutibilidade a retransformação do plasmídeo no clone tóxico é incentivado.
O ensaio do supressor do fermento é uma experiência muito grande e o tempo e os materiais apropriados devem ser planeados conformemente. Este protocolo contém muitos detalhes que devem ser respeitados estritamente e recomenda-se que o pesquisador que conduz esses ensaios leia e observe cuidadosamente o protocolo na íntegra antes de iniciar um ensaio. Este é um procedimento multipartite, incluindo bactérias e leveduras que precisam ser cultivadas em diferentes temperaturas. O isolamento de plasmídeos de levedura às vezes pode ser difícil devido à sua parede celular resistente e o uso de DMSO ajuda a alcançar maior eficiência de transformação.
Uma das maiores vantagens da toxicidade do fermento e tela supressoré que o efetor bacteriano não precisa se ligar fisicamente aos substratos hospedeiros para qualquer quantidade apreciável de tempo para detectar a via putativa alvo. Apenas um pequeno punhado de estudos relataram o uso desta ou uma técnica semelhante1-5. No entanto, existem relatos de técnicas que identificam vias hospedeiras e muitas técnicas para rastrear parceiros de ligação de proteínas efeteroras bacterianas. Kramer et al. relataram uma aproximação nova da biologia dos sistemas para identificar os caminhos alvejados por proteínas bacterianas do efetor8. A técnica usou uma coleção haploide da tensão da deleção de S. cerevisiae para selecionar para os mutantes sensíveis ao effector Osp4 de Shigella. Usando esta técnica, osp4 foi ligado à regulação da sinalização MAPK e atenuação da imunidade inata em células hospedeiras. Array-based, de alta taxa de taxa, telas automáticas de levedura dois híbridos agora pode identificar rapidamente putativo parceiros de ligação efetor bacteriana, selecionando muitos ao mesmo tempo contra milhões de proteínas hospedeiras9. Da mesma forma, imunoprecipitações de alta taxa de renda, juntamente com espectrometria de massa, estão sendo usadas para identificar em massa parceiros de ligação putativos de famílias inteiras de proteínas efetivas bacterianas10. Embora muitos desses estudos possam acelerar o calendário de caracterização de proteínas efetódicas, eles raramente fornecem informações sobre quais processos fisiológicos estão sendo manipulados e quais são o resultado holístico dessas interações. Assim, técnicas como a toxicidade do fermento e a tela supressora são essenciais para iluminar os processos celulares que estão sendo manipulados por essas proteínas efetoras. Em algum momento, observações que identificam parceiros de ligação ou vias alvo de proteínas efetentes bacterianas precisam retornar aos modelos de infecção celular para determinar se esses achados in vitro são verdadeiros em cenários fisiologicamente relevantes. A manipulação genética de patógenos intracelulares obrigatórios como C. trachomatis tem sido um grande obstáculo até recentemente11-15 e a geração de mutantes específicos do local para estudar proteínas efetóvoras não era possível. Nos últimos anos, porém, uma revolução na geração de mutantes clamídia, complementando-os, e superexpressando proteínas-alvo ocorreu. Estes avanços recentes aumentaram extremamente o valor da informação derivada dos estudos que usam a toxicidade e as telas do supressor porque é agora possível ir do fermento diretamente na infecção para sondar a validez dos resultados.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Agradecemos Shelby Andersen, Abby McCullough, e Laurel Woods por sua ajuda com essas técnicas. Este estudo foi financiado por fundos de inicialização do Departamento de Microbiologia e Imunologia da Universidade de Iowa para Mary M. Weber.
Materiais
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | BP2641500 | |
| Galactose | MilliporeSigma | G0750-1KG | |
| GeneJet Gel extraction kit | ThermoFisher Scientific | K0691 | |
| GeneJet PCR purification kit | ThermoFisher Scientific | K0701 | |
| GeneJet plasmid miniprep kit | Thermo | K0503 | |
| Glucose | MilliporeSigma | G8270-1KG | |
| Herring Sperm DNA | Promega | D1811 | |
| KpnI-HF | New England Biolabs | R3142S | |
| Lithium acetate dihydrate | MilliporeSigma | L6883-250G | |
| Peptone | Fisher Scientific | ||
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530 | |
| Poly(ethylene glycol) 3350 | MilliporeSigma | 1546547-1G | |
| pYep13 | ATCC | 37323 | |
| T4 DNA ligase | New England Biolabs | M0202S | |
| Tryptophan | MilliporeSigma | 470031-1G | |
| XhoI-HF | New England Biolabs | R0146S | |
| Yeast extract | Fisher Scientific | BP1422-500 | |
| Yeast miniprep kit | Zymo | D2001 | |
| Yeast nitrogen base without amino acids | MilliporeSigma | Y0626-250G | |
| Yeast Synthetic Drop-out Medium Supplements | MilliporeSigma | Y1501-20G | without uracil |
| Yeast Synthetic Drop-out Medium Supplements | MilliporeSigma | Y1771-20G | without uracil, leucine, tryptophan |
Referências
- Tan, Y., Luo, Z. Q. Legionella pneumophila SidD is a deAMPylase that modifies Rab1. Nature. 475 (7357), 506-509 (2011).
- Guo, Z., Stephenson, R., Qiu, J., Zheng, S., Luo, Z. A Legionella effector modulates host cytoskeletal structure by inhibiting actin polymerization. Microbes and Infection. 16 (3), 225-236 (2014).
- Tan, Y., Arnold, R. J., Luo, Z. -Q. Legionella pneumophila regulates the small GTPase Rab1 activity by reversible phosphorylcholination. Proceedings of the National Academy of Sciences of the United States of America. 108 (52), 21212-21217 (2011).
- Weber, M. M., et al. The type IV secreted effector protein CirA stimulates the GTPase activity of RhoA and is required for virulence in a mouse model of Coxiella burnetii infection. Infection and Immunity. 84, (2016).
- Faris, R., et al. Chlamydia trachomatis CT229 subverts Rab GTPase-dependent CCV trafficking pathways to promote chlamydial infection. Cell Reports. 26, 3380-3390 (2019).
- Mumberg, D., Müller, R., Funk, M. Yeast vectors for the controlled expression of heterologous proteins in different genetic backgrounds. Gene. 156, 119(1995).
- Hill, J., Donald, K. A. G., Griffiths, D. E. DMSO-enhanced whole cell yeast transformation. Nucleic Acids Research. 19 (20), 41070(1991).
- Kramer, R. W., et al. Yeast Functional Genomic Screens Lead to Identification of a Role for a Bacterial Effector in Innate Immunity Regulation. PLoS Pathogens. 3 (2), 21(2007).
- Häuser, R. T. S., Rajagopala, S. V., Uetz, P. Array-Based Yeast Two-Hybrid Screens: A Practical Guide. Two Hybrid Technologies: Methods and Protocols, Methods in Molecular Biology. , 21-38 (2012).
- Mirrashidi, K. M., et al. Global mapping of the inc-human interactome reveals that retromer restricts chlamydia infection. Cell Host and Microbe. 18 (1), 109-121 (2015).
- Wang, Y., et al. Development of a transformation system for chlamydia trachomatis: Restoration of glycogen biosynthesis by acquisition of a plasmid shuttle vector. PLoS Pathogens. 7 (9), 1002258(2011).
- Mueller, K. E., Wolf, K., Fields, K. A. Gene deletion by fluorescence-reported allelic exchange mutagenesis in Chlamydia trachomatis. mBio. 7 (1), 1-9 (2016).
- Johnson, C. M., Specific Fisher, D. J. Site-Specific, Insertional Inactivation of incA in Chlamydia trachomatis Using a Group II Intron. PLoS ONE. 8 (12), 83989(2013).
- Weber, M. M., et al. Absence of specific Chlamydia trachomatis inclusion membrane proteins triggers premature inclusion membrane lysis and host cell death. Cell Reports. 19 (7), 1406-1417 (2017).
- Weber, M. M., et al. A functional core of IncA is required for Chlamydia trachomatis inclusion fusion. Journal of Bacteriology. 198 (8), 1347-1355 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados