Method Article
酵母毒性およびサプレッサースクリーンを用いた細菌エフェクタータンパク質を標的とする宿主経路の同定
要約
細菌病原体は、重要な生物学的プロセスを標的とする宿主にタンパク質を分泌する。細菌エフェクタータンパク質を標的とする宿主経路を同定することは、分子病因に対処する鍵となる。ここで、毒性細菌エフェクタータンパク質を標的とする宿主経路を解明するために改変酵母サプレッサーおよび毒性スクリーンを用いる方法について説明する。
要約
細胞内細菌は、細菌の利益のために宿主タンパク質および/または関連する生物学的経路を覆すために作用する宿主サイトゾルにエフェクタータンパク質と呼ばれる毒性因子を分泌する。細菌ゲノムシーケンシングの進歩や、分泌候補および/または真核様をコードする遺伝子のシリコ同定を可能にするアルゴリズムの出現により、細菌エフェクタータンパク質の同定がより管理しやすくなったドメイン。しかし、これらの重要な毒性因子の同定は、最初のステップに過ぎません。当然のことながら、目的はエフェクタータンパク質の分子機能を決定し、それらが宿主とどのように相互作用するかを解明することです。近年、酵母2ハイブリッドスクリーンや大規模免疫沈降と質量分析を組み合わせた技術が、タンパク質間相互作用の同定に役立っています。宿主結合パートナーの同定は、細菌エフェクタータンパク質の分子機能を解明するための重要な第一歩であるが、宿主タンパク質が複数の生物学的機能(例えば、アクチン、クラトリン、チューブリン)を有することが判明することがある。細菌タンパク質は、ホストタンパク質を物理的に結合させないかもしれないし、操作されている正確な宿主経路に関する重要な情報を研究者から奪う。サプレッサースクリーンと組み合わせた修飾酵母毒性スクリーンは、細菌エフェクタータンパク質の影響を受ける宿主経路を同定するように適合されている。毒性スクリーンは、多くの場合、増殖欠陥として現れる宿主生物学的経路を妨害するエフェクタータンパク質によって引き起こされる酵母の毒性効果に依存する。酵母ゲノムライブラリーの発現は、細菌エフェクタータンパク質の毒性を抑制し、エフェクタータンパク質が標的とする経路内のタンパク質を同定する宿主因子を同定するために使用される。このプロトコルには、毒性画面とサプレッサー画面の両方の詳細な指示が含まれています。これらの技術は、酵母および大腸菌の分子クローニングおよび培養が可能な任意の実験室で行うことができる。
概要
ここで提示されたものと同様の手順の最初のレポートは、レジオネラ肺炎球菌型IVエフェクターSidD、Rab11を変質するdeAMPylasを特徴としました。同等の技術は、いくつかのL.肺炎球菌エフェクター1、2、3の特性評価に使用された。アッセイは、コキシエラ・ブルネチーIVエフェクタータンパク質4を特徴付けるために適応され、最近、クラミジア・トラコマティス含有膜タンパク質の特性評価のためにこの技術の有用性が拡張された5.
このプロトコルは、2つの主要な部分に分けることができる:1)目的の細菌エフェクタータンパク質が酵母およびクローンで発現され、成長欠陥によって示される有毒な表現型についてスクリーニングされ、2)酵母サプレッサースクリーン、有毒株中の酵母ゲノムライブラリーの発現により毒性表現型が抑制されるものである。したがって、毒性スクリーンは、目的の細菌エフェクターが過剰発現されたときに増殖欠陥として現れる毒性表現型の画面である。細菌エフェクターで正常に変換され、発現する有毒クローンが選択され、次のステップのために保存されます。第2の主要なステップは、有毒酵母クローン内の部分的に消化された酵母ゲノムライブラリーを過剰発現することを含む。このプロトコルで使用するために提案された酵母ゲノムライブラリを構成するプラスミドは5−20 kbのインサートを運び、通常はすべてのプラスミドにわたって〜1.5kbの平均遺伝子サイズの3−13酵母オープンリーディングフレーム(ORF)に対応し、全酵母ゲノムカバーを表す約10倍。アッセイのこの部分は、細菌エフェクタータンパク質の毒性を抑制することを目的とするように、サプレッサースクリーンと呼ばれる。潜在的なサプレッサープラスミドは、酵母から単離され、配列化され、および同定された抑制ORFである。サプレッサースクリーンの根底にある根拠は、エフェクタータンパク質が標的とする宿主経路の成分に結合し、相互作用し、および/または圧倒される成分を、それらの宿主タンパク質を過剰に戻すことができ、経路に対する毒性効果を救うことができるということです。成長欠陥。したがって、毒性を抑制するORFを同定すると、多くの場合、宿主経路の複数の参加者を表す。次に、直交実験を行い、細菌エフェクターが実際に関連経路と相互作用することを検証します。これは、これらのタンパク質が多数の宿主プロセスに関与しているため、クラトリンやアクチンなどの結合パートナーが同定された場合に特に必要です。さらなる実験は、感染中にエフェクタータンパク質の生理機能を解明することができる。毒性およびサプレッサースクリーンはまた、免疫沈降によって検出するのに十分な親和性を有する宿主タンパク質を物理的に結合しない細菌エフェクタータンパク質の生理機能を解読するための強力なツールであり、酵母2ハイブリッドスクリーンでは検出されない可能性のある酵素的ヒットアンドラン相互作用のホスト。
サプレッサースクリーンは、細菌エフェクタータンパク質と宿主経路との間の潜在的な生理学的相互作用を明らかにする強力な方法であり得るが、細菌エフェクタータンパク質は酵母の増殖欠陥を誘導しなければならない。サプレッサー画面はほとんど役に立ちません。さらに、有毒な表現型は、少なくとも2−3ログ10の成長の欠損をもたらすか、またはサプレッサーを同定することが困難であろう。細胞培養のために実験室が設置されている場合、HeLaなどの一般的な細胞株における毒性のスクリーニングエフェクタータンパク質は、酵母毒性スクリーンを進める努力に値するかどうかについての洞察を与えることができる。HeLa細胞におけるエフェクタータンパク質の湿疹発現は、これらのスクリーン4に用いられる酵母株における毒性と非常に強く相関する毒性をもたらす場合がある。HeLa細胞におけるストレスの観察可能な特徴は、ストレス繊維の喪失、プレートからの細胞剥離、およびアポトーシスを示す核縮合を含む。HeLa細胞におけるストレスの視覚的な徴候は、酵母の増殖欠陥を誘導するための良い候補となるタンパク質を作り、これははるかに迅速に複製し、したがって、本質的な経路の摂動に対してより応答性が高くなります。
サプレッサー画面は、必ずしもホスト・バインディング・パートナーをサプレッサーとして識別するとは限りませんが、ターゲットとするホスト経路の重要なコンポーネントに影響を与える可能性があり、それによってハイジャックされる生物学的プロセスの全体像が得られます。細菌エフェクタータンパク質。表面上、エフェクタータンパク質の結合パートナーを過剰に提供することは、成長欠陥を救出することが期待されるため、これは直感的ではないようです。感染5時に少なくとも10種類のRab GTPアーゼに結合するC.トラコマティスエフェクタープロテインCT229(CpoS)の標的となる経路を同定する取り組みにおいて、Rab結合パートナーのいずれもCT229の毒性を抑制しなかった。しかし、クラトリン被覆小胞(CCV)の密売に関与する多数のサプレッサーが同定され、CT229がRab依存型CCVの密売を特異的に転覆していることを示すさらなる作業につながった。同様に、酵母増殖欠陥を救出したC.ブルネティエフェクタープロテインCbu0041(CirA)を調べたところ、その後、RhoA4に対するGTPアーゼ活性化タンパク質(GAP)としてCirAが機能することが判明した。
細菌エフェクタータンパク質を標的とする宿主経路を解明するための酵母サプレッサースクリーンの有用性はいくら強調してもしすぎることはできず、細胞内細菌エフェクタータンパク質を特徴付けようとする他の研究者は、大きな利益を得ることができます。これらの技術。これらのアッセイは、免疫沈降および/または酵母2つのハイブリッドスクリーンが結合パートナーを見つけることができず、どの経路が細菌エフェクタータンパク質によって標的にされているかを解明できる場合に価値があります。ここでは、細胞内細菌エフェクタータンパク質を標的とする宿主生物学的経路を同定するための毒性およびサプレッサースクリーンの詳細なプロトコルと、これらのアッセイを使用する際に経験した一般的な障害のいくつかおよび対応するソリューション。
プロトコル
1. 媒体および試薬の調製
注:プレートは、アッセイの日の前に準備する必要があり、1ヶ月間良いです。媒体および試薬はいつでも作ることができ、1ヶ月間良いです。
- 1,000mLビーカーに800mLの蒸留水にD-(+)-グルコース100gを溶解して、グルコース溶液の1L(10%w/v)を調製する。蒸留水で体積を1Lに調整します。0.2μmの滅菌フィルターを通して滅菌1 Lメディア貯蔵ボトルにフィルターを入れます。
- 1Lビーカーに800mLの蒸留水に100gのD-(+)-ガラクトースを溶解してガラクトース溶液(10%w/v)を1L調製します。蒸留水を使用して体積を1mLに調整し、0.2μmの無菌フィルターを通して滅菌1,000 mLの媒体貯蔵ボトルにフィルターを入れます。
- 酵母エキスペプトンデキストロース(YPD)寒天の1Lを調製し、酵母エキス10g、ペプトン20g、グルコース20g、寒天20gを蒸留水1,000mLに溶解して調製する。オートクレーブを20分間、冷却するまで56°水浴で冷却する。1枚あたり約20mLの媒体を使用して100mmプレートに注ぎます。
- 酵母エキス10g、ペプトン20g、ブドウ糖20gを蒸留水1,000mLに溶解してYPDブロス1Lを調製する。オートクレーブ 20分間
- 合成ドロップアウト(SD)ウラシル(ウラ-)グルコース寒天を準備します。1Lの場合、アミノ酸を含まない酵母窒素塩基6.7g、ウラシルを含まないドロップアウトサプリメント1.9g、蒸留水800mLで15gの寒天を溶解する。20分間オートクレーブし、温度が〜50−60°Cになるまで56°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.1で調製したグルコース溶液の200 mLを追加します。軽く渦巻くか、かき混ぜ板の上に置いてよく混ぜます。1枚あたり約20mLの媒体を使用して100mmプレートに注ぎます。
- 合成ドロップアウト(SD)ウラシル(ウラ-)ガラクトース寒天を準備します。1Lの場合、アミノ酸を含まない酵母窒素塩基6.7g、ウラシルを含まないドロップアウトサプリメント1.9g、蒸留水800mLで15gの寒天を溶解する。20分間オートクレーブし、温度が〜50−60°Cになるまで56°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.2で調製したガラクトース溶液の200 mLを追加します。軽く渦巻くか、かき混ぜ板の上に置いてよく混ぜます。1枚あたり約20mLの媒体を使用して100mmプレートに注ぎます。
- 合成ドロップアウト(SD)ウラシル(ウラ-)グルコーススープを準備します。1Lの場合、アミノ酸を含まない酵母窒素塩基6.7gと蒸留水800mLでウラシルを含まないドロップアウトサプリメントを1.9g溶解する。20分間オートクレーブし、温度が〜50−60°Cになるまで56°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.1で調製したグルコース溶液を200ml添加します。
- 合成ドロップアウト(SD)ウラシル(ウラ-)ロイシン(Leu-)グルコース寒天を準備します。1Lの場合、アミノ酸を含まない酵母窒素塩基の6.7gを溶解する。ウラシル、ロイシン、トリプトファンを含まないドロップアウトサプリメントの1.9 g;寒天の15グラム;蒸留水の800 mLでトリプトファンの0.076 g.20分間オートクレーブし、温度が〜50−60°Cになるまで56°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.1で調製したグルコース溶液の200 mLを追加します。1枚あたり約20mLの媒体を使用して100mmプレートに注ぎます。
- 合成ドロップアウト(SD)ウラシル(ウラ-)ロイシン(Leu-)ガラクトース寒天を準備します。1 Lの場合、アミノ酸を含まない酵母窒素塩基の6.7gを溶解する。ウラシル、ロイシン、トリプトファンを含まないドロップアウトサプリメントの1.9 g;寒天の15グラム;蒸留水の800 mLでトリプトファンの0.076 g.20分間オートクレーブし、温度が〜50−60°Cになるまで56°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.2で調製したガラクトース溶液の200 mLを追加します。軽く旋回するか、かき混ぜ板の上に置いてよく混ぜます。1枚あたり約20mLの媒体を使用して100mmプレートに注ぎます。
- 合成ドロップアウト(SD)ウラシル(ウラ-)ロイシン(Leu-)グルコーススープを準備します。1 Lの場合、アミノ酸を含まない酵母窒素塩基の6.7gを溶解する。ウラシルなしドロップアウトサプリメントの1.9 g, ロイシン, トリプトファン;蒸留水の800 mLでトリプトファンの0.076 g.20分間オートクレーブと温度が〜50°Cになるまで56−60°水浴で冷却する。50 mL血清学的ピペットまたは滅菌等級シリンダーを使用して、ステップ1.1で調製したグルコース溶液の200 mLを追加します。
- ポリエチレングリコール(PEG)溶液を調製します。蒸留水にポリエチレングリコール3350の50%w/vを加えます。オートクレーブで殺菌します。
- 1M酢酸リチウム(LiAc)を200mLの蒸留水に脱水して1M酢酸リチウム(LiAc)を調製する。オートクレーブで殺菌します。
- ニシン精子DNAを準備します。蒸留水を用いて10mg/mLニシン精子DNAを2mg/mLに希釈する。100°Cで5分間加熱し、直ちに氷の上に5分間置きます。
2. 目的の遺伝子を酵母毒性プラスミドプラスミドpYesNTA-Kanにクローニングする
注:現在、商業的にも学術的にも様々な酵母毒性ベクターが利用可能です。酵母サプレッサースクリーンは、目的のエフェクタータンパク質を発現するプラスミドを提供するこれらのベクターの多くと併用することができ、ウラシル以外のドロップアウト選択およびBlaR以外の抗生物質マーカーを使用する。本研究では、カナマイシン耐性カセットを含む修正pYesNTAベクター4、5を用いて、潜在的なサプレッサーのスクリーニングを容易にした。クローニングスキームは、エフェクタータンパク質がGalプロモーターおよびHis-Tagとフレーム内になければならないことを可能にする必要があります。クラミジアトラコマティス封入膜タンパク質CT229は、これらのアッセイの原理の証明として使用された。
- PCR を使用して、CT229 +1 Kpn F (CCGGTACCAATGTCTCTCTCTCTTtaTTATtaTTATtaTTATTATTAGTT) および CT229 XhoI R (CCCTCGAGTTTTTTTCTGGGATCC) プライマーを使用して、製造元の指示に従ってC. トラコマティスゲノム DNA から CT229 を増幅します。次の PCR 条件を使用します: (1) 98 °C 30 s;(2) 98 °C 10 s, 55 °C 30 s, 72 °C 2 分;(3) 合計 25 倍の手順 2 を繰り返します。(4) 72 °C 10分間;および(5)4°Cホールド。
- 1%アガロースゲル上のPCR産物の5°Lを分析する(図1)。
- 製造元の指示に従って PCR 精製キットを使用して、残りの 45 μL の DNA を精製します。
- KpnI-HFおよびXhoI-HFを使用した水浴中で、精製されたPCRインサートとpYesNTA-Kan(図2)の50μLを37°Cで1時間消化します。
注:pYesNTA-Kanの場合、KpnI-HFの5°L、XhoI-HFの5°L、および60μLの総体積で6μLのバッファを使用してプラスミドの5μgを消化します。インサートの場合は、2μLのKpnI-HF、XhoI-HFの2μL、および6μLのバッファを使用して、精製されたPCR製品の50°L全体を消化します。 - 1%アガロースゲルでプラスミドダイジェスト全体を実行します。製造業者の指示に従ってゲル抽出キットを使用して消化されたプラスミドの精製を行う。
- 製造元の指示に従って、PCR 精製キットを使用して消化された PCR インサートを精製します。
- 2μLのpYesNTA-Kan(ステップ2.5)、DNAリガーゼ緩衝液2μL、T4リガーゼの1μL、および15μLのインサートを使用して、インサートをpYesNTA-Kanにクローニングします(ステップ2.6)。室温で1時間インキュベートする。
- 大腸菌能細胞の50°Lにクローニング反応全体を加え、形質転換反応を行う。
- カルベニシリンの100μg/mLを含むLBプレート上の変換反応全体をプレートします。
- 3つの個々のコロニーを有するカルベニシリンの100μg/mLを含むLBの10 mLを接種する。150 rpmで振とう37°Cで一晩インキュベートする。
- メーカーの指示に従ってプラスミドミニプレップキットを使用してプラスミドを分離します。
- 遺伝子特異的プライマーを用いて単離されたプラスミドを配列する(ステップ2.1)。
3. 酵母の毒性に関する目的のタンパク質を試験する
- YPD寒天上のストリークサッカロミセスセレビシエW303(ステップ1.3)を単離したコロニーを得た。24時間30°Cでインキュベートする。
- 寒天プレートから単一のコロニーで10mLのYPDスープ(ステップ1.4)を接種する(ステップ3.1)。一晩で150rpmで振とう30°Cでインキュベートする。
- 10 mLのYPDスープ(ステップ1.4)に一晩培養物の0.5 mLを加え、4時間150rpmで振とう30°Cでインキュベートします。
- 10mLの培養物を3,000 x gで4°Cで10分間ペレット化する。
- ペレットを滅菌水1mLで再サスペンドし、マイクロ遠心管に移し、室温で3,000 x gでペレットを1分間行う。
- ペレットを1mMの酢酸リチウム(LiAc)の1mLで再スマードし、室温で1分間3,000 x gのペレットで再スマー。
- LiAc洗浄2xを繰り返します。
- 洗浄を取り外します。50% PEG 3350 の 2.4 mL でペレットを再サスペンドします。1M LiAcの360 μL、ニシン精子DNAの2mg/mLの500 μL、滅菌水の400°Lを加えます。
注: これは 20 の変換に対して十分です。 - ステップ3.3.5から180μLの変換ミックスを、ステップ2.12から5μLのpYesNTA-Kan CT229プラスミドDNA(100−500ng)を含むマイクロ遠心管に追加します。
- 30°Cの水浴で30分間インキュベートする。
- 42°Cの水浴で30分間インキュベートする。
- 室温で1分間3,000 x gのペレット。
- ピペットで変換ミックスを削除します。ペレットを滅菌水100°Lで再サスペンドします。SDウラに変換をプレート-グルコースで寒天(ステップ1.5)。
- 48時間30°Cでプレートをインキュベートします。
- SDウラの5 mLを接種する -プレートから単一のコロニーでグルコース(ステップ1.7)を含むスープ。150 rpmで振とうで30°Cで一晩インキュベートする。ベクター単独で形質転換した酵母を陰性対照として含める。
- 96ウェルプレート(A2-A6)の5ウェルに180μLの滅菌水を加えます。
- 混ぜる一晩培養物をボルテックス。
- 最初のウェル(A1)に酵母を180μL加えます。連続希釈1:10(未希釈を含む合計6サンプル)。
- マルチチャンネルピペットを使用して、SDウラ上の各希釈のスポット5μL-グルコース(ステップ1.5)とウラ-ガラクトース(ステップ1.6)寒天プレート。48時間30°Cでインキュベートする。
- ガラクトース含有媒体上で増殖した目的のエフェクタータンパク質を発現する酵母の増殖を、ベクターのみを発現する酵母の増殖と比較して毒性を評価する。
- ウェスタンブロッティングによるHisタグ融合タンパク質の発現を確認する。
4. 酵母ゲノムライブラリーで有毒酵母を変換する
- SDウラの100 mLを接種する -グルコーススープ(ステップ1.7)からステップ3.4から1 mLのストックを使用する。150 rpmで振とう30°Cで16−24時間インキュベートする。SDウラの900 mLを置く-スラウススープを一晩で30°Cで、メディアを前温します。
- 一晩培養物の100mL全体を前温1Lフラスコに加える。150 rpmで振とう30°Cで4−5時間インキュベートします。
- 4°Cで10分間6,000 x gで培養物をペレット。
- 上清を捨てる。滅菌水の250 mLでペレットを再サスペンドします。4°Cで5分間6,000 x gで培養をペレット。
- 上清を捨てる。1 mM LiAcの250 mLでペレットを再サスペンドします。4°Cで5分間6,000 x gで培養をペレット。
- LiAcを取り外し、50%PEG 3350の9.6 mLでペレットを再サスペンドします。1M LiAc の 1.44 mL、2 mg/mL ニシン精子 DNA の 2 mL、pYep13 ゲノム ライブラリ (ATCC 37323) の 50 μg を追加します。滅菌水で体積を15mLに調整します。反転によって穏やかに混ぜる。
- 30°Cの水浴で30分間インキュベートする。
- 750 μL のジメチルスルホキシド (DMSO) を加えるため、変換効率を高めます。42°Cの水浴で30分間インキュベートし、10分ごとにやさしく反転して混ぜます。
- 室温で5分間3,000 x gで酵母をペレット。
- 上清を捨て、滅菌水10mLでペレットを再サスペンドします。室温で5分間3,000 x gのペレット。
- 滅菌水の8 mLでペレットを再サスペンドします。
- 変換効率を決定するために、SDウラ-Leu-グルコース寒天上の各希釈の1:10とプレート100μLを希釈する(ステップ1.8)。
- SDウラ上のサンプルのプレート200μL- Leu-ガラクトース寒天(ステップ1.9)プレート。全部で50枚のプレートを使用してください。
- 48-96時間またはコロニーが現れるまで30°Cでインキュベートする。
注:コロニーは一般に48-72 hのグルコース寒天プレートと72-96時間のガラクトース寒天プレートに現れます。
- SDウラのパッチコロニー(潜在的な救助)- Leu-ガラクトース寒天(ステップ1.9)を拡大し、在庫を作ります。24−48hのための30 °Cでインキュベートする。
- パッチの一部を使用して、SDウラ-Leu-グルコーススープ(ステップ1.10)の5 mLを接種します。 150 rpmで振とうで26°Cで一晩インキュベートする。
- 滅菌水の180°Lを96ウェルプレート(A2-A6)の5ウェルに加えます。
- 混ぜる一晩培養物をボルテックス。
- 最初のウェル(A1)に酵母を180μL加えます。連続希釈1:10(未希釈を含む合計6サンプル)。
- マルチチャンネルピペットを使用して、SDウラ上の各希釈のスポット5 μL-グルコースとSDウラ-ガラクトース寒天プレート。コントロールとして単独で毒性エフェクターを含む。
- 48時間30°Cでインキュベートする。
- 有毒エフェクターを単独で発現する酵母の成長を、潜在的なサプレッサーを含む酵母と比較する。エフェクターを単独で発現する酵母と比較して毒性を低下させた潜在的なサプレッサーのみ進む。
5. サプレッサーの特定と確認
- SDウラの5 mLを接種- Leu-グルコーススープ(ステップ1.10)をステップ4.4から酵母の100μLで、150rpmで振とうして30°Cで一晩インキュベートする。
- 室温で2分間3,000 x gで酵母をペレット。ピペットを使って上清をそっと捨てます。
- メーカーの指示に従って、酵母プラスミドミニプレップキットでプラスミドを分離します。
- 単離されたプラスミドを大腸菌の有能な細胞に変換し、カルベニシリンの100 μg/mLでLB寒天上の全体の形質転換をプレートします。24時間37°Cでインキュベートする。
- 100 μg/mLのカルベニシリンを含むLBブロスの10 mLを接種し、プレートから3つのコロニーを含み、150rpmで振とうして一晩37°Cでインキュベートします。
- 室温で10分間3,000 x gでペレット培養。製造元の指示に従ってミニプレップ キットを使用してプラスミドを分離します。
- プロトコルの第 3 部のステップに従って、ステップ 5.3.1 から単離されたプラスミドを使用して有毒酵母を再変換します。
- SDウラの5 mLを接種 - Leu-変換プレートからコロニーを有するグルコーススープ。150 rpmで振とうで30°Cで一晩インキュベートする。
- ウラ上のスポット- Leu-グルコースとガラクトース寒天は、毒性の抑制を確認します。
- pyep13 F (ACTACGCGATCATGGCGA) および pyep13 R (TGATGCCGGCCACGATGC) プライマーを使用して酵母 ORF を識別するシーケンス。
結果
実際の酵母サプレッサースクリーンを実行する前に、目的のエフェクタータンパク質が酵母の毒性をテストする必要があります。これは、ガラクトース誘導性プロモーターの制御下で酵母に目的とするタンパク質を発現させることによって達成される。グルコースの成長(非誘導条件)は、まず、目的のタンパク質の発現に起因する毒性が特異的であり、一般的な欠陥ではないことを保証するために比較されるべきである。図3に示すように、毒性は小さなコロニーおよび/または減少した成長として現れる。酵母の成長の2-3の対数の減少は、酵母サプレッサースクリーンに最適です。
図4に示すように、毒性およびサプレッサー画面は、プロトコルセクションで説明されている方法を使用して8つのステップで達成することができる。目的の遺伝子は適切なベクター1にクローニングされ、得られた構成要素は酵母に変換され、毒性を評価する。本研究では、目的の遺伝子としてCT229を用い、ベクターとしてpYesNTA-Kanを用いった。また、Hisタグ付き融合タンパク質の発現をウェスタンブロッティングにより確認した。理想的には、ガラクトース含有媒体上で成長した酵母の2−3対数減少を観察すべきである。目的のタンパク質が酵母に対して毒性がある場合(図3および図4)、サプレッサースクリーンは、酵母ゲノムライブラリーpYep13で有毒株を変換することによって行うことができる。形質転換体は、グルコース寒天上でめっきされ、形質転換効率とガラクトース寒天を決定し、潜在的なサプレッサーを同定します。このプロトコルを使用して、銀河寒天上の合計10-250コロニーで5 x 10 5の最小変換効率を達成しました。SD Ura- Leu-ガラクトース寒天(図4)にこれらのコロニーをパッチすると、多くの偽陽性がパッチを当てたときに成長しないので、真のサプレッサーの同定に役立つ。ここで、50個のコロニーにパッチを適用し、エフェクター毒性を抑制した8つのクローンが得られた(図4)。SDウラ上のスポッティングサプレッサー- Leu-ガラクトース寒天は、毒性の抑制を確認するために行われました。図4に示すように、pSup1およびpSup2はエフェクタータンパク質の毒性を抑制し、一方、pSup3は抑制しなかった。したがって、pSup3は廃棄された。プラスミドは、その後、サプレッサーから単離され、プラスミド収率を増加させるために大腸菌に変換された。その後、プラスミドを毒性酵母に再変換して、単離されたプラスミドがエフェクター毒性を確実に抑制することを確認することができる(図4)。単離されたプラスミドのほとんどは毒性を助長すべきであるが、時折有毒酵母株に再変換されたときに毒性を抑制しないプラスミドが得られる。これらのプラスミドは廃棄され、再変換後に毒性を抑制するもののみを配列する必要があります。
単離されたプラスミドは、複数の酵母ORF4を含む。どちらが真のサプレッサーであるかを特定するために、各ORFを個別にクローン化し、前述の1、2、3、4に記載の有毒酵母株で発現させることができる。

図1:標的遺伝子のPCR増幅クラミジア・トラコマティス・セロバーL2からのCT229は、ゲノムDNAからPCR増幅した。PCR産物を臭化エチジウムで染色した1%アガロースゲル上で分析した。この図の大きなバージョンを表示するには、ここをクリックしてください。
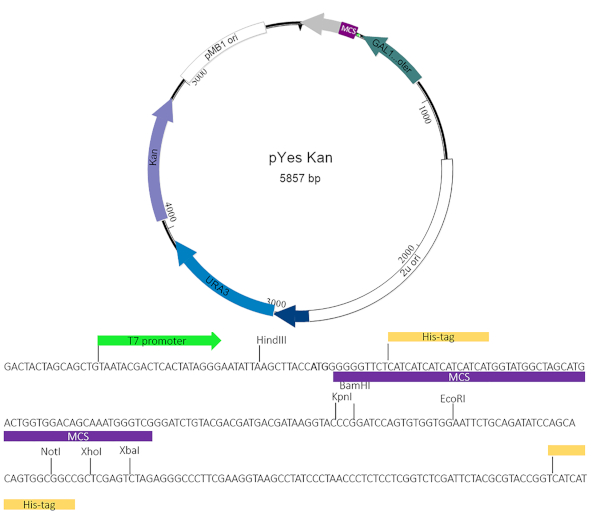
図2:pYesNTA-Kanプラスミドマップ目的の標的遺伝子をpYesNTA-Kanの多重クローニング部位(MCS)にクローニングし、大腸菌での選択にカナマイシンを用い、S.セレビシエでの選択にウラシルドロップアウトを用いた。Hisタグ付き融合タンパク質の発現をウェスタンブロッティングにより検証した。この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:代表的な酵母毒性結果目的のエフェクタータンパク質をpYesNTA-Kanにクローニングした配列検証済みプラスミドをS.セレビシエに変換し、形質転換体を連続的に希釈し、ウラに発見した-グルコースおよびガラクトース寒天は毒性を評価した。この図の大きなバージョンを表示するには、ここをクリックしてください。

図4:酵母サプレッサー画面の概要目的の標的遺伝子をpYesNTA-Kanにクローニングし、プラスミドをS.セレビシエに変換した。形質転換体を連続的に希釈し、ウラに発見した -グルコースとガラクトース寒天は、毒性を評価するために。毒性エフェクタータンパク質を標的とする経路を同定するために、毒性タンパク質を発現する酵母株を酵ゲノムライブラリーで形質転換した。コロニーはウラ- Leu-ガラクトース寒天にパッチを当て、毒性を評価するために発見されました。プラスミドは、エフェクタータンパク質の毒性を抑制するものから単離した。プラスミドを有毒酵母株に再変換し、単離されたプラスミドがサプレッサーであることを確認した。サプレッサーからのプラスミドを配列し、存在する酵母OrFを同定した。この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
このプロトコルは、修飾酵母毒性およびサプレッサースクリーンを用いて細菌エフェクタータンパク質を標的とする宿主生物学的経路を同定するためのステップバイステップの手順を概説する。使用される酵母株としては、S.セレビシエW303が、ウラシルおよびロイシンの両方に対する栄養補助である。株のウラシル栄養萎縮は、pYesNTA-Kanベクター上で目的のタンパク質を運ぶ酵母を選択するために使用され、ロイシン栄養失調は酵母ゲノムライブラリーベクターpYep13の選択に使用されます。酵母ゲノムライブラリープラスミドは3-13 ORFを運ぶので、各ORFはp415-ADHベクター6または同様のベクターに個別にクローン化され、真のサプレッサーであるものを識別するために毒性酵母に再変換する必要があります。宿主の生物学的経路を表す複数のサプレッサーを同定するのが典型的である。サプレッサーは、目的とする細菌タンパク質の直接結合パートナーになることがありますが、必ずしもそうであるとは限りません。
pYesNTA-Kanに目的の細菌タンパク質を運ぶ酵母クローンを生成することは、最初の主要なステップであり、毒性を運ぶ酵母のための強い負の選択があるので、できるだけ早くこれらのクローンの完全性を維持するために細心の注意を払う必要がありますタンパク質は、意図的なガラクトース誘導がなくても、プラスミドを失う可能性があります。時折、酵母がドロップアウト媒体上の選択の下で維持されている場合でも、プラスミド損失があります。ベクターを線形化し、酵母ゲノム1内に統合することによりプラスミド損失の発生を減少させる方法が以前に報告されている。
これらの技術の主な欠点は、細菌エフェクタータンパク質の過剰発現が酵母の観察可能で一貫した成長欠陥を引き起こす場合にのみ、サプレッサースクリーンが有意義なデータを生成することである。この方法の主な難点は、サプレッサが特定されていない場合です。サプレッサーの特定に失敗した場合、生物学的な理由や技術的な問題が原因かどうかを判断するのは困難です。生物学的観点からは、目的のタンパク質が毒性を持つ可能性があり、ゲノムライブラリーのタンパク質が毒性を有意に抑制できない、標的経路が救助できない、または多数の共発現が可能である可能性がある。有毒な効果を克服するために要因が必要な場合があります。.解剖する未定義の変数が多く存在するため、生物学的な説明に近づくことは困難です。確立されたプロトコルがあるため、技術的な説明を調べることは、はるかに難しいアプローチです。技術的な観点から、サプレッサーの同定に失敗すると、酵母ゲノムライブラリーの変換効率が低い結果である可能性が高い。このプロトコルは、pYEp13のATCC S.セレビシエAB320部分的に消化されたゲノムライブラリーを使用する。他のライブラリは他のベクターと同様に十分かもしれませんが、このプロトコルは特にATCCライブラリを使用しており、代替ライブラリまたはベクターソースを報告する研究は現在公開されていない。酵母ゲノムのカバレッジは、少なくとも10倍、または過小表現がサプレッサーの同定を妨げる可能性があることを強くお勧めします。変換効率の低下により、これらの数値が 1 倍以下になることができます。したがって、酵母ゲノムの一部は、サプレッサースクリーンに表されない場合がある。低い変換効率は、悪い試薬や不十分なライブラリの準備を含む多くの問題に起因する可能性があります。この方法は、主にヒルら7によって最初に出されたDMSO/LiAc変換方法に準拠しています。新しいDMSO、50%PEG、および新鮮なニシン精子DNAを購入して変換のために調製し、試薬の品質の汚染または劣化を減少させるためにこれらのアッセイのために排他的に使用することをお勧めします。DMSOは高吸湿性が高く、大気からの水を容易に吸収します。したがって、多くの個々のアリコートを使用することは、容器の蓋の繰り返し開口部と大気への暴露を避けるためにお勧めします。高品質のニシン精子DNAは-20°Cで保存することができ、数年間使用するのに適していますが、各変換のために新しい調製物をストックから調製する必要があります。一度沸騰して冷却すると、ニシン精子のDNAは再煮込まれたり再利用されたりしてはならないので、必要なものを取り除き、現在の変換に必要以上のものを作らないようにするのが最善です。細部への注意は酵母の形質転換効率を大幅に改善し、サプレッサーを識別する可能性を向上させることができます。
酵母pYep13ベクターが3−13種類の酵母ORFを運ぶことを考えると、偽陽性プラスミドの数を減らすことは、サプレッサーを同定する上で最も重要である。酵母は、同様のベクターの複数のコピーを取り上げ、収容することができますが、プラスミドの非互換性に関連する一般的な知恵は、伝統的に大腸菌は一度に同じ複製の起源(ORI)を持つプラスミドの1種類のみを維持することを指示しています。しかし、これは常にそうではありません。これは、サプレッサー候補を特定しようとする際に大きな問題になる可能性があります。サプレッサー酵母クローンが同定された場合、プラスミドが抽出され、同定のための伝播とその後のシーケンシングのために大腸菌に再変換されます。候補サプレッサープラスミドが分離され、大腸菌に再変換されたら、個々のクローンが運んでいる可能性があるため、少なくとも5つの大腸菌コロニーをシーケンシング用に選択することを強くお勧めします。異なるプラスミド。大腸菌は一度に1つのプラスミドしか伝播できないはずなので、酵母分離が複数のプラスミドを生み出す場合、プレート上の異なる大腸菌クローンは一度に1つだけを運ぶべきである。したがって、大腸菌における正常な形質転換効率に基づいて5つのコロニーを摘み取るには、クローンが同じまたは異なるプラスミドを運んでいるかどうかを示す必要があります。大腸菌からのプラスミドのシーケンシングの後に複数のプラスミドが生じた場合、それぞれが有毒酵母クローンに変換され、成長欠陥の救済を評価する必要があります。さらに、単一のサプレッサープラスミドのみが同定されたとしても、プラスミドを毒性クローンに対する溶解性再変換が推奨される。
酵母サプレッサーアッセイは非常に大規模な実験であり、適切な時間と材料はそれに応じて計画されるべきである。このプロトコルには、厳密に遵守しなければならない多くの詳細が含まれており、これらのアッセイを行う研究者は、アッセイを開始する前にプロトコルを注意深く読み、完全に見ることをお勧めします。これは、異なる温度で栽培する必要がある細菌と酵母の両方を含む多partite手順です。酵母からのプラスミドの分離は、そのタフな細胞壁のために困難な場合があり、DMSOの使用は、より高い形質転換効率を達成するのに役立ちます。
酵母毒性およびサプレッサースクリーンの最大の利点の1つは、細菌エフェクターが標的とされる可逆経路を検出するのに任意の時間のためにホスト基質に物理的に結合する必要がなされないことである。このまたは同様の手法1-5を使用して報告されている研究のほんの一握り.しかし、宿主経路を同定する技術の報告と、細菌エフェクタータンパク質の結合パートナーをスクリーニングする多くの技術がある。Kramer et al. は、細菌エフェクタータンパク質8によって標的とされる経路を同定するための新しいシステム生物学的アプローチを報告した。この技術は、S.セレビシエのハプロイド欠失株集を用いて、シゲラエフェクターOsp4に敏感な変異体をスクリーニングした。この技術を用いて、Osp4は、宿主細胞におけるMAPKシグナル伝達および自然免疫の減衰の調節に関連した。アレイベースの、高スループット、自動化された酵母2ハイブリッドスクリーンは、数百万個の宿主タンパク質9に対して同時に多数のスクリーニングを行うことで、可逆的な細菌エフェクター結合パートナーを迅速に識別できるようになりました。同様に、質量分析法と組み合わせた高スループット免疫沈降は、細菌エフェクタータンパク質10のファミリー全体の可換結合パートナーを一括同定するために使用されている。これらの研究の多くは、エフェクタータンパク質特性評価のタイムテーブルを加速することができますが、生理学的プロセスが操作されているもの、およびこれらの相互作用の全体的な結果が何であるかについての洞察を提供することはめったにありません。したがって、酵母毒性やサプレッサースクリーンなどの技術は、これらのエフェクタータンパク質によって操作される細胞プロセスを照らするために不可欠です。ある時点で、細菌エフェクタータンパク質を標的とする結合パートナーまたは経路を同定する観察は、これらのインビトロ所見が生理学的に関連するシナリオで真であるかどうかを判断するために細胞感染モデルに戻る必要があります。C.トラコマティスのような細胞内病原体の遺伝子操作は、最近まで11~15年まで大きなハードルとなっており、エフェクタータンパク質を研究する部位特異的変異体の生成は不可能であった。しかし、ここ数年、クラミジア突然変異体を生成し、それらを補完し、標的タンパク質を過剰発現する革命が起こりました。これらの最近の進歩は、結果の妥当性を調査するために酵母から直接感染に行くことを可能にするので、毒性およびサプレッサースクリーンを使用した研究から得られた情報の価値を大幅に高めました。
開示事項
著者らは、競合する財政的利益がないことを宣言する。
謝辞
シェルビー・アンダーセン、アビー・マッカロー、ローレル・ウッズの支援に感謝します。この研究は、アイオワ大学微生物学・免疫学科からメアリー・M・ウェーバーに資金を提供しました。
資料
| Name | Company | Catalog Number | Comments |
| Agar | Fisher Scientific | BP2641500 | |
| Galactose | MilliporeSigma | G0750-1KG | |
| GeneJet Gel extraction kit | ThermoFisher Scientific | K0691 | |
| GeneJet PCR purification kit | ThermoFisher Scientific | K0701 | |
| GeneJet plasmid miniprep kit | Thermo | K0503 | |
| Glucose | MilliporeSigma | G8270-1KG | |
| Herring Sperm DNA | Promega | D1811 | |
| KpnI-HF | New England Biolabs | R3142S | |
| Lithium acetate dihydrate | MilliporeSigma | L6883-250G | |
| Peptone | Fisher Scientific | ||
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530 | |
| Poly(ethylene glycol) 3350 | MilliporeSigma | 1546547-1G | |
| pYep13 | ATCC | 37323 | |
| T4 DNA ligase | New England Biolabs | M0202S | |
| Tryptophan | MilliporeSigma | 470031-1G | |
| XhoI-HF | New England Biolabs | R0146S | |
| Yeast extract | Fisher Scientific | BP1422-500 | |
| Yeast miniprep kit | Zymo | D2001 | |
| Yeast nitrogen base without amino acids | MilliporeSigma | Y0626-250G | |
| Yeast Synthetic Drop-out Medium Supplements | MilliporeSigma | Y1501-20G | without uracil |
| Yeast Synthetic Drop-out Medium Supplements | MilliporeSigma | Y1771-20G | without uracil, leucine, tryptophan |
参考文献
- Tan, Y., Luo, Z. Q. Legionella pneumophila SidD is a deAMPylase that modifies Rab1. Nature. 475 (7357), 506-509 (2011).
- Guo, Z., Stephenson, R., Qiu, J., Zheng, S., Luo, Z. A Legionella effector modulates host cytoskeletal structure by inhibiting actin polymerization. Microbes and Infection. 16 (3), 225-236 (2014).
- Tan, Y., Arnold, R. J., Luo, Z. -Q. Legionella pneumophila regulates the small GTPase Rab1 activity by reversible phosphorylcholination. Proceedings of the National Academy of Sciences of the United States of America. 108 (52), 21212-21217 (2011).
- Weber, M. M., et al. The type IV secreted effector protein CirA stimulates the GTPase activity of RhoA and is required for virulence in a mouse model of Coxiella burnetii infection. Infection and Immunity. 84, (2016).
- Faris, R., et al. Chlamydia trachomatis CT229 subverts Rab GTPase-dependent CCV trafficking pathways to promote chlamydial infection. Cell Reports. 26, 3380-3390 (2019).
- Mumberg, D., Müller, R., Funk, M. Yeast vectors for the controlled expression of heterologous proteins in different genetic backgrounds. Gene. 156, 119(1995).
- Hill, J., Donald, K. A. G., Griffiths, D. E. DMSO-enhanced whole cell yeast transformation. Nucleic Acids Research. 19 (20), 41070(1991).
- Kramer, R. W., et al. Yeast Functional Genomic Screens Lead to Identification of a Role for a Bacterial Effector in Innate Immunity Regulation. PLoS Pathogens. 3 (2), 21(2007).
- Häuser, R. T. S., Rajagopala, S. V., Uetz, P. Array-Based Yeast Two-Hybrid Screens: A Practical Guide. Two Hybrid Technologies: Methods and Protocols, Methods in Molecular Biology. , 21-38 (2012).
- Mirrashidi, K. M., et al. Global mapping of the inc-human interactome reveals that retromer restricts chlamydia infection. Cell Host and Microbe. 18 (1), 109-121 (2015).
- Wang, Y., et al. Development of a transformation system for chlamydia trachomatis: Restoration of glycogen biosynthesis by acquisition of a plasmid shuttle vector. PLoS Pathogens. 7 (9), 1002258(2011).
- Mueller, K. E., Wolf, K., Fields, K. A. Gene deletion by fluorescence-reported allelic exchange mutagenesis in Chlamydia trachomatis. mBio. 7 (1), 1-9 (2016).
- Johnson, C. M., Specific Fisher, D. J. Site-Specific, Insertional Inactivation of incA in Chlamydia trachomatis Using a Group II Intron. PLoS ONE. 8 (12), 83989(2013).
- Weber, M. M., et al. Absence of specific Chlamydia trachomatis inclusion membrane proteins triggers premature inclusion membrane lysis and host cell death. Cell Reports. 19 (7), 1406-1417 (2017).
- Weber, M. M., et al. A functional core of IncA is required for Chlamydia trachomatis inclusion fusion. Journal of Bacteriology. 198 (8), 1347-1355 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved