Method Article
Rotulagem específica de nucleóides mitocondriais para microscopia estruturada de iluminação com lapso de tempo
Neste Artigo
Resumo
O protocolo descreve rotulagem específica de nucleóides mitocondriais com uma mancha de gel de DNA comercialmente disponível, aquisição de série de lapso de tempo de células rotuladas ao vivo por microscopia de iluminação estruturada de super resolução (SR-SIM) e rastreamento automático do movimento nucleóide.
Resumo
Nucleóides mitocondriais são partículas compactas formadas por moléculas de DNA mitocondrial revestidas de proteínas. O DNA mitocondrial codifica tRNAs, rRNAs e vários polipeptídeos mitocondriais essenciais. Os nucleóides mitocondriais dividem e distribuem dentro da dinâmica rede mitocondrial que sofre fissão/fusão e outras alterações morfológicas. Microscopia de fluorescência viva de alta resolução é uma técnica simples para caracterizar a posição e o movimento de um nucleóide. Para esta técnica, os nucleóides são comumente rotulados através de marcas fluorescentes de seus componentes proteicos, ou seja, fator de transcrição a (TFAM). No entanto, essa estratégia precisa de superexpressão de uma construção com etiqueta de proteína fluorescente, que pode causar artefatos (relatados para tfam), e não é viável em muitos casos. Corantes orgânicos de ligação de DNA não têm essas desvantagens. No entanto, eles sempre mostram a coloração de DNAs nucleares e mitocondriais, sem especificidade para os nucleóides mitocondriais. Levando em conta as propriedades físico-químicas desses corantes, selecionamos uma mancha de gel de ácido nucleico (SYBR Gold) e alcançamos rotulagem preferencial de nucleóides mitocondriais em células vivas. Propriedades do corante, particularmente seu alto brilho ao vincular ao DNA, permitem quantificação subsequente do movimento nucleóide mitocondrial usando séries temporais de imagens de iluminação estruturadas de super-resolução.
Introdução
Moléculas circulares de DNA de 16,5 kbp constituem o material genético das mitocôndrias, codificando 22 tRNAs, 2 rRNAs e 13 polipeptídeos necessários para complexos mitocondriais oxidativos de fosforilação. DNA mitocondrial ligado ao fator de transcrição mitocondrial a (TFAM) e várias outras proteínas formam os nucleóides mitocondriais1,,2,,3,,4. Os nucleóides mitocondriais movem-se e redistribuem entre os componentes da rede mitocondrial5,6 durante sua remodelagem morfológica, fissão ou fusão, dependendo da fase do ciclo celular, estresse e outros fatores (revisados em Pernas et al.7). Além disso, o movimento dos nucleóides mitocondriais está implicado na doença de lúpus eritematoso sistêmico8 e pode desempenhar um papel em outras doenças. A microscopia de fluorescência é uma técnica simples para estudos de células vivas de organelas, mas a técnica tem uma resolução de >200 nm, que é maior que o tamanho dos nucleóides mitocondriais (~100 nm9,,10,,11,12). Esse limite tem sido contornado pelas chamadas técnicas de "super-resolução", como esgotamento estimulado de emissões (STED) e microscopia de localização de moléculas únicas (SMLM)13,14. Até agora, nucleóides mitocondriais e outros DNAs foram imagens em células vivas por microscopia direta de reconstrução óptica estocástica (dSTORM)15. Estruturas submitocínias finas com posições correlacionadas com o dNH foram observadas por DST em células vivas16. No entanto, essas técnicas de super-resolução requerem alta intensidade de iluminação, o que causa efeitos fototóxicos nas células vivas17. Portanto, a imagem de lapso de tempo de nucleóides mitocondriais com resolução além do limite de difração é desafiadora. Para lidar com isso, utilizamos microscopia de iluminação estruturada de super resolução (SR-SIM)18. O SIM requer uma dose de energia de iluminação muito menor do que o STED e o SMLM19. Além disso, ao contrário das técnicas STED e SMLM, o SIM permite imagens tridimensionais multicoloridas simples (3D) e não requer propriedades fotofísicas particulares dos fluoroforos ou composição de tampão de imagem19.
A estratégia convencional para rotular nucleóides mitocondriais em células vivas é a marcação fluorescente de uma proteína nucleóide mitocondrial, como a TFAM20. No entanto, em muitos casos, essa estratégia não é adequada. Além disso, a superexpressão do TFAM com proteína fluorescente produz um artefato sério21. A rotulagem de DNA com corantes orgânicos tem vantagens sobre uma estratégia baseada em proteína fluorescente (FP). Os corantes orgânicos são livres de restrições relacionadas à marcação FP: podem ser usados para qualquer tipo de células ou tecidos e podem ser aplicados a qualquer momento de um experimento. Imagens de células vivas de nucleóides mitocondriais foram relatadas com vários corantes de ligação de DNA: DAPI22, SYBR Green23, Vybrant DyeCycle24e picoGreen15,,25,26. Uma desvantagem substancial da maioria dos corantes de ligação de DNA para rotulagem nucleóide é que eles mancham todo o DNA dentro da célula. Direcionar um corante apenas para DNA mitocondrial é altamente desejável. Para isso, é necessária uma seleção cuidadosa de um corante que possua propriedades físico-químicas adequadas. Corantes lipofílicos que possuem carga positiva deslocalizada, como a rhodamina 123, são conhecidos por se acumularem em mitocôndrias vivas, que preservam seu potencial de membrana negativa. Além disso, um corante ideal para rotulagem específica de nucleóides mitocondriais deve ligar o DNA com alta afinidade e emitir fluorescência brilhante na ligação de DNA. Considerando esses requisitos, certas cianolinas são promissoras (por exemplo, picoGreen), mas o DNA nuclear é abundantemente manchado por esses corantes simultaneamente com DNA mitocondrial15,,25,26. O presente protocolo descreve a rotulagem específica de nucleóides mitocondriais em células vivas com outro corante de cianina, SYBR Gold (SG), e rastreamento dos nucleóides em vídeos SIM de super resolução de lapso de tempo. Além disso, as células vivas manchadas de SG podem ser imagens por qualquer tipo de microscópio fluorescente invertido (fusocal, disco giratório, epifluorescência, etc.) adequado para células vivas e equipado com uma fonte de luz de 488 nm.
Protocolo
NOTA: Todas as linhas celulares aqui mencionadas foram cultivadas em alta glicose Dulbecco's Modified Eagle's medium (DMEM) suplementado com 10% de soro bovino fetal (FBS), glutamina, penicilina/estreptomicina e piruvato. Equilibre todos os meios de comunicação e suplementos a serem usados no dia da rotulagem e imagem aquecendo-os até 37 °C em uma incubadora definida para 5% de CO2. Todo o trabalho de cultura celular, incluindo rotulagem, ocorre em condições estéreis sob uma coifa de fluxo laminar.
1. Rotulagem de células ao vivo
- Um dia antes do procedimento de rotulagem, a cultura 4 x 105 células HeLa em 2 mL de meio em uma placa de Petri de 35 mm com fundo de vidro #1,5. Slides de câmara multi-bem podem ser usados, mas os volumes devem ser ajustados proporcionalmente ao volume do poço. Diluir a suspensão celular para ter aproximadamente 50.000 células/mL e sementes de 2 mL da suspensão celular em uma placa de Petri de 35 mm.
- Prepare as seguintes diluições da solução de estoque comercial SYBR Gold (SG): 1:5.000. Prepare Mitotracker Deep Red (mancha vermelha distante) ou Mitotracker CMXRos Vermelho (mancha vermelha) (Tabela de Materiais; estoque comercial diluído 1:2.000) em meio de cultura sem fenol vermelho. Prepare as mesmas diluições do picoGreen como para SG.
NOTA: As soluções descritas na etapa 1.2 são as soluções de rotulagem 2x. Proteja todas as soluções que contenham corantes fluorescentes da luz o máximo possível. - Lave as células aderentes com 2 mL PBS uma vez. Adicione 1 ml de cultura sem fenol ao prato petri de 35 mm. Para um slide de câmara de 8 poços, use um volume igual a 1/2 da capacidade total do poço (por exemplo, adicione 125 μL se a capacidade total do poço for de 250 μL).
- Adicione 1 mL da solução de rotulagem 2x preparada na placa de 1,2 a 35 mm de Petri. Para um slide de câmara de 8 poços, use um volume igual a 1/2 da capacidade total do poço. Incubar as células por 30 min a 37 °C abaixo de 5% de CO2.
NOTA: Não incubar as células com solução de rotulagem SG por mais de 1h, pois a incubação mais longa causará rotulagem de DNA nuclear. - Aspire cuidadosamente o meio contendo os corantes fluorescentes e lave as células uma vez com PBS.
- Adicione o meio de cultura celular sem fenol e mantenha as células no escuro em uma incubadora de CO2 a 37 °C até a imagem.
- Células vivas de imagem em um microscópio SR-SIM equipado com uma unidade de incubação (veja abaixo).
2. Aquisição de Imagem SR-SIM
- Instale uma incubadora de topo de palco no estágio do microscópio. Ajuste a temperatura desejada e a concentração de CO2 (por exemplo, 37 °C e 5% para células de mamíferos) e mantenha-se aquecido por pelo menos 1h antes de iniciar a aquisição da imagem.
- Ligue todos os componentes do microscópio SR-SIM, incluindo os lasers, e deixe-os aquecer por pelo menos 1 h.
- Escolha um objetivo de imersão de alta ampliação, alta abertura numérica (NA) (por exemplo, 100x 1,46 na oil) recomendado para SR-SIM pelo fabricante do microscópio.
- Instale um slide de câmara ou um prato de 35 mm com células rotuladas (a partir da etapa 1.6) no estágio do microscópio incubado.
- Localize a área de interesse da amostra, de preferência com oculares. Para obter a melhor qualidade de imagem SR-SIM, escolha as células que estão bem ligadas ao vidro.
- Use uma câmera de dispositivo emcomento de elétrons de alta extremidade emarada de fundo (EM-CCD) para adquirir imagens SR-SIM.
- Usando o software de aquisição de imagens, defina um alto ganho EM recomendado para a câmera usada (por exemplo, 300).
- Antes de adquirir a série de lapso de tempo para rastreamento nucleóide, adquira uma imagem SR-SIM de duas cores do mesmo campo de visão: um canal para coloração mitocondrial e outro para SG. Defina o primeiro canal de cores apropriado para a mancha mitocondrial utilizada (por exemplo, para uma mancha mitocondrial muito vermelha, coloque a excitação em 633 nm ou mais e um filtro de emissão de longopass de 650 nm). Para o sinal SG, defina a excitação em 488 nm e um filtro de emissão de bandpass de 500-550 nm.
- Usando o software, defina a menor potência laser possível para linhas de manchas de 488 nm e muito vermelhas.
NOTA: Uma configuração típica é 1% de potência de saída a laser ajustada por um filtro tunable tunable acousto-óptico (AOTF). - Se o microscópio SIM adquirir canais apenas sequencialmente (configuração de câmera única), desligue o canal de detecção de manchas muito vermelho.
- Defina a aquisição de um único plano focal desligando a aquisição da pilha Z desfaçando a caixa Z-stack no software.
- Defina o menor tempo de exposição da câmera EM-CCD possível.
- Defina três rotações da grade em vez de cinco rotações.
- Para aumentar a taxa de quadros, na guia Aquisição do software, defina a câmera para ler os dados apenas a partir da área central do sensor de câmera em vez de "Full Chip".
NOTA: Por exemplo, mudar de ler uma área de 1.000 x 1.000 pixels de sensor completo para ler apenas 256 x 256 pixels permite uma redução no tempo de exposição da câmera de 50 ms para 13,4 ms e o tempo total de período de 1,8 s para 1,2 s. Se apenas uma parte central do sensor de câmera for lida, então o campo de visão será muito pequeno para um objetivo de 63x ou 100x normalmente usado para SR-SIM. Um tempo de exposição mais curto reduzirá a relação sinal-ruído das imagens. - Otimizar o tempo de exposição do laser e da câmera: adquira imagens bidimensionais SR-SIM (2D) de células rotuladas a vários valores de potência laser (por exemplo, 0,5%, 1%, 1,5%, 2%) e vários tempos de exposição (por exemplo, 13,4 ms, 25 ms, 50 ms).
- Processe os conjuntos de dados SIM brutos (ver etapa 3.1).
- Escolha os tempos de exposição de energia a laser e câmera que produzam imagens SIM com pontos brilhantes nas mitocôndrias (ou seja, os nucleóides) com pouco ou nenhum artefato gerado pelo processamento de SIM. Inspecione as áreas fora das mitocôndrias (por exemplo, 1% de potência do laser de estado sólido de 488 nm e exposição de 25 ms da câmera EM-CCD).
- Inicie a aquisição da série time lapse usando as configurações otimizadas nas etapas 2.15-2.17.
3. Processamento e Análise de Dados
- Processe conjuntos de dados SIM brutos com o módulo de iluminação estruturado de um software adequado (Tabela de Materiais): escolha a caixa de seleção automática para parâmetros de processamento SIM. Para processamento automático de vários arquivos, use a ferramenta de processamento em lote, desmarque Use Corrente para Lote,clique em Executar batch e selecione vários arquivos para processamento SIM.
- Converta os conjuntos de dados da série temporal processados por SIM em formato ims com o software conversor de arquivos Imaris (ou seja, a versão correspondente à versão do software Imaris).
- Abra um arquivo convertido no software (versão 8.4.1 ou posterior) que tenha uma licença para o módulo Lineage (ou Track).
- Inicie o assistente de criação "Spots", clicando em adicionar novos pontos. Um mago para a criação será lançado.
- Escolha a guia Criar do assistente.
- Na primeira etapa do assistente, clique em Track Spots (ao longo do tempo) e prossiga para a segunda etapa do assistente.
- Defina O Diâmetro XY Estimado para 0,1-0,15 μm, clique em Subtração de Fundo e prossiga para a terceira etapa.
- Ajuste o limiar no filtro "Qualidade" arrastando a linha vertical no histograma para que a maioria dos nucleóides sejam detectados como "manchas", enquanto os artefatos não são detectados (ou seja, os pontos detectados são marcados como bolas sobrepostas na imagem). Verifique se esse é o caso em cada quadro e reajuste o limite, se necessário. Vá para o quarto e, em seguida, para o quinto passo do mago.
- Escolha o algoritmo Autoregressive Motion. Defina distância máxima para 0,5 μm. Ajuste o tamanho da lacuna máxima para 0.
- Olhe para cada quadro da série de tempo e verifique se alguma faixa falsa é desenhada e se alguma lacuna entre as faixas é introduzida (faixas construídas pelo assistente com configurações atuais são marcadas instantaneamente como linhas sobrepostas na imagem). Ajuste "Max Distance", se necessário. Vá para o sexto passo do mago. Escolha o filtro Duração da faixa e defina um limite de 3 a 5 s.
- Se os pontos ou faixas detectados atualmente não forem ideais, volte para as etapas anteriores do assistente usando botões de navegação (na parte inferior da janela do assistente) necessários para ajustar qualquer parâmetro para a criação de pontos ou faixas.
- Quando os parâmetros afinados permitirem que o software detecte todos os pontos e construa faixas corretamente, clique no botão de navegação do Arqueiro Verde no assistente para confirmar a criação das faixas.
- Extraia as estatísticas das faixas clicando na janela ícone Estatísticas. Escolha os parâmetros estatísticos necessários (guias ValoresDetalhados e Médios em Estatísticas) e exporte os valores como arquivos csv clicando no ícone Disquete para quantificação e visualização.
NOTA: Velocidade máxima da pista, velocidade média da pista, comprimento da pista e deslocamento da faixa são exemplos de parâmetros que são importantes. - Se o conjunto de dados de imagem precisar ser apresentado ou publicado, então crie instantâneos e/ou arquivos de vídeo representando a série de lapso de tempo com faixas sobrepostas opcionalmente usando as ferramentas Snapshot ou Animation.
4. Aquisição de Imagem Confocal
- Instale uma incubadora de topo de palco no estágio do microscópio, ajuste a temperatura desejada e a concentração de CO2 (ou seja, 37 °C e 5% para células de mamíferos) e mantenha-se aquecido por pelo menos 1h antes de iniciar a aquisição da imagem.
- Ligue todos os componentes de um microscópio de varredura a laser confocal, incluindo os lasers, e deixe-os aquecer por pelo menos 1 h.
- Escolha um objetivo de ampliação desejado para alta ampliação objetiva e amostragem espacial de acordo com os critérios de Nyquist.
- Para o canal SG, defina a excitação em 488 nm e uma faixa de detecção de 500-550 nm. Para o canal de manchas mitocondriais, definido para 561 nm e 570-630 nm de emissão para um corante vermelho, ou 633 nm excitação e >650 nm emissão para um corante vermelho distante.
- Defina a menor potência laser possível (tipicamente 1% ou menos) que permite a aquisição dos sinais com ganhos de detector PMT de 700 mV.
- Adquira imagens confocal de duas cores das células, onde os nucleóides mitocondriais são detectados no "canal SG" e mitocôndrias são detectadas no canal "vermelho" ou "vermelho distante".
Resultados
Caracterização da rotulagem de células vivas com SG
Primeiro, a distribuição de SG nas células após incubação com o corante em várias diluições foi caracterizada por microscopia confocal. Após a incubação com altas concentrações de SG ou picoGreen, ambos os corantes rotularam principalmente os núcleos e apresentaram uma coloração pontual no citoplasma (Figura 1),da mesma forma aos dados publicados para outro corante de cianeto carregado positivamente (ou seja, picoGreen)15. No entanto, após a incubação com SG às 1:10.000 diluições, manchas fracas apareceram nos núcleos, enquanto no citoplasma, observamos um padrão de pontos brilhantes(Figura 1). Por outro lado, a incubação com corante picoGreen diluído 1:10.000 rendeu principalmente manchas nucleares. O sinal SG era muito mais brilhante do que o do picoGreen na mesma concentração. Os dados mostraram que a SG é mais adequada para o DNA mitocondrial de imagem do que outros corantes similares de ligação de DNA.
Para confirmar que os pontos brilhantes estão localizados nas mitocôndrias, manchamos células vivas simultaneamente com SG e mancha mitocondrial muito vermelha. Este último é um corante orgânico permeável celular com células carregadas positivamente que se acumula nas mitocôndrias das células vivas. Após a incubação com SG às 1:10.000 e 1:50.000 diluições, quase todas as manchas de SG ocorreram em mitocôndrias, enquanto ao rotular em 1:500 e 1:1.000 diluições, ocorreu uma mancha significativa dos núcleos e citoplasma(Figura 2).
Além disso, caracterizamos o curso de tempo de coloração de células vivas com SG por microscopia de lapso de tempo(Figura 3). Os gráficos de intensidade de fluorescência SG em mitocôndrias vs. tempo(Figura 3B) sugeriram que após 45 minutos, a coloração nucleóide estava perto da saturação. Assim, recomendamos tempos de incubação de ~30-60 min.
Testamos como a distribuição intracelular SG muda após a fixação e/ou permeabilização das células. A fixação (2% de paraformaldeído [PFA]) das células manchadas vivas causou uma leve redistribuição do corante ao núcleo(Figura 4A,B). A permeabilização das células fixas (0,1% Triton X100) eliminou o padrão pontilhado SG nas mitocôndrias, e a coloração dos núcleos foi dominante(Figura 4C). Se o SG foi adicionado às células após fixação e permeabilização, ele se distribuiu uniformemente através do citoplasma e núcleos(Figura 4D). Assim, as células rotuladas por SG podem ser fixadas se necessário, mas o corante não é adequado para protocolos que requerem permeabilização.
Rastreamento de nucleóides sr-SIM e mitocondrial de células vivas
As células vivas costas com SG e uma mancha mitocondrial muito vermelha(Tabela de Materiais) foram imagens 3D por uma técnica sim de super resolução. Como nas imagens confocal(Figura 2),os nucleóides mitocondriais apareceram como pontos brilhantes dentro das mitocôndrias(Figura 5A). Além disso, adquirimos uma série temporal de imagens SIM 2D e rastreamos as posições dos nucleóides em uma resolução além do limite de difração. Imediatamente antes de iniciar a série de tempo adquirimos pilhas SIM 3D no SG e nos canais de manchas mitocondriais. Então adquirimos uma série temporal apenas no canal SG e rastreamos os nucleóides mitocondriais a fim de quantificar seus movimentos. A velocidade média da pista foi de 0,042 μm/s, e a velocidade máxima instantânea da pista foi de 0,078 ± 0,012 μm/s. A maioria dos nucleóides não se deslocou longe de suas posições originais, mas mostrou movimentos aleatórios de curta distância que provavelmente estavam confinados à rede mitocondrial, como sugere uma sobreposição de faixas nas imagens mitocondriais(Figura 5B). Poucos deslocamentos direcionais rápidos ocorreram durante uma série temporal típica(Filme 1).
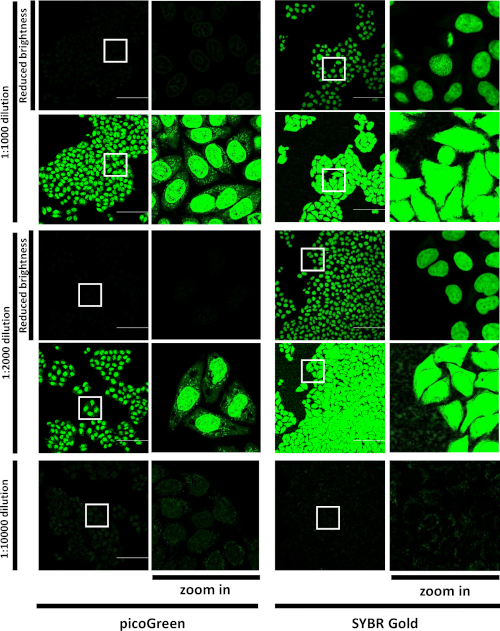
Figura 1: Comparação da rotulagem de células vivas picoGreen e SG. As células A549 foram incubadas com picoGreen ou SG em diluições indicadas e imagens. Campos representativos de visão das células rotuladas no canal verde. LSM880 Airyscan FAST, 20x/air objective, Barra de escala = 50 μm. Seções ópticas únicas. Os quadrados brancos marcam as áreas mostradas em uma ampliação mais elevada à direita dos campos de visão de 423 x 423 μm. Para diluições de 1:1.000 e 1:2.000, as mesmas imagens são mostradas em duas configurações de brilho: as configurações de brilho "padrão" otimizadas para diluição de 1:10.000 e as configurações de "brilho reduzido" ajustadas para evitar a saturação do detector. Este número foi modificado a partir de Jevtic et al.27. Clique aqui para ver uma versão maior desta figura.
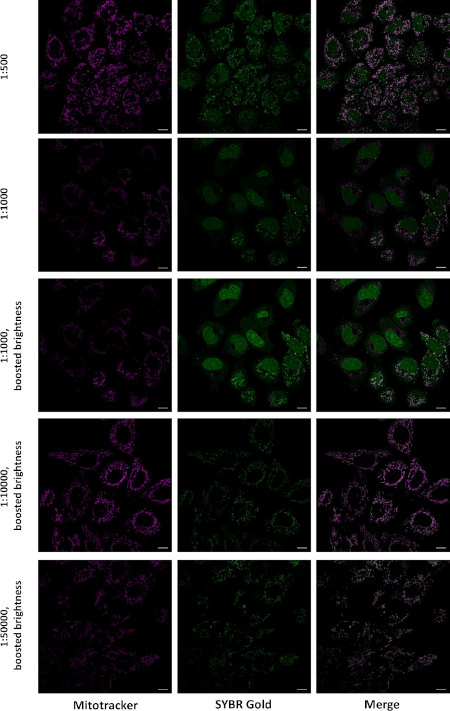
Figura 2: Localização de SG em células vivas após rotulagem em diferentes concentrações. As células HeLa foram incubadas por 30 min com uma mistura de 0,25 μM Mitotracker CMXRos Red e a diluição indicada de SG. A solução foi substituída pelo DMEM, e as imagens foram adquiridas em um microscópio Airyscan LSM880, objetivo de óleo 63x 1.4, com aquisição sequencial de canais coloridos. Únicas fatias ópticas são mostradas Barra de escala = 10 μm). Este número foi modificado a partir de Jevtic et al.27. Clique aqui para ver uma versão maior desta figura.

Figura 3: Células HeLa durante a rotulagem com SYBR Gold (SG). Primeiro, as células HeLa vivas foram rotuladas com Mitotracker CMXRos Red e lavadas. SG (diluição final 1:10.000 em DMEM) foi adicionado às células e uma série de lapso de tempo foi adquirida. Microscópio LSM880, 63x 1.4 Óleo objetivo, aquisição sequencial. Pilhas de Z foram adquiridas em cada ponto de tempo. As projeções de intensidade máxima são mostradas. (A) Intensidades médias de fluorescência SG ao longo do tempo em várias regiões de interesse. (B) Campos de visão representativos mostrando as regiões de interesse onde a fluorescência SG foi medida (retângulos coloridos). (C) Um campo de visão em vários pontos de tempo durante a incubação com SG. Uma região quadrada marcada com linha branca é mostrada em uma ampliação mais elevada na coluna direita. Este número foi modificado a partir de Jevtic et al.27. Clique aqui para ver uma versão maior desta figura.

Figura 4: Efeito da fixação e permeabilização na localização de SG nas células. As células HeLa foram manchadas com SG (estoque diluído 1:10.000) e Mitotracker CMXRos Red (0,25 μM) por 30 min. Fatias ópticas únicas foram adquiridas em um microscópio de disco giratório; aquisição sequencial; barra de escala = 10 μm. (A) Células HeLa vivas manchadas com SG. (B) Células HeLa vivas manchadas com SG e depois fixadas com 2% de PFA em PBS por 30 min. (C) Células HeLa vivas manchadas com SG, depois fixadas com 2% pfa em PBS por 30 min e permeabilizadas com 0,1% Triton X100 por 15 min. No painel inferior, a mesma imagem é mostrada, mas o brilho no canal verde é definido mais alto. (D) Células HeLa fixadas com 2% pfa, permeabilizadas com Triton X100 de 0,1% por 15 min, e depois manchadas com SG e Mitotracker CMXRos Red. Para adquirir as imagens mostradas em(D),o ganho emCCD da câmera para o canal verde foi reduzido por um fator de 12 em comparação com A-C para evitar a superexposição. Este número foi modificado a partir de Jevtic et al.27. Clique aqui para ver uma versão maior desta figura.

Figura 5: Rastreamento nucleóide em imagens SIM ao vivo. Imagens representativas de um campo de visão de células manchadas de SG. Inferior: a imagem SIM de duas cores tirada antes da aquisição da série time lapse. Verde = canal SG; magenta = canal mitotracker. Top: faixas nucleóides de uma série de tempo SIM de 50 quadros no canal SG(Filme 1); tempo de quadro = 1,8 s; rastreamento pelo software Imaris 8.4.1. As faixas são codificadas por cores por velocidade máxima da faixa (μm/s); Elyra PS.1, modo SIM, objetivo de 100x/1.46 Oil; Barra de escala = 10 μm. Este número foi modificado a partir de Jevtic et al.27. Clique aqui para ver uma versão maior desta figura.

Filme 1: Série de lapso de tempo SIM mostrando células representativas manchadas de SG. Barra de escala = 5 μm. Os nucleóides mitocondriais detectados são marcados como esferas brancas. As faixas são visualizadas como "Caudas de Dragão" (8 quadros de comprimento) e codificadas por cores de acordo com suas velocidades instantâneas máximas (a barra de cores no canto inferior direito mostra velocidades em μm/s). Nucleóides mitocondriais rastreando e visualizando pelo software Imaris 8.4.1. Este vídeo foi publicado em Jevtic et al.27. Clique aqui para ver este vídeo (Clique com o botão direito do mouse para baixar).
Discussão
Existem vários componentes críticos no protocolo: Para alcançar a rotulagem preferencial do DNA mitocondrial, a concentração do corante de ligação de DNA durante a incubação deve ser mantida muito baixa (por exemplo, uma diluição de 1:10.000 de um estoque comercial típico), e o tempo de incubação deve ser de 30 minutos. O tempo de incubação nunca deve exceder 1 h. Deve-se usar o corante SYBR Gold; outros corantes de ligação de DNA não são brilhantes o suficiente para gerar um sinal forte ao rotular em baixa concentração.
A limitação do nosso protocolo é que o corante é retirado dos nucleóides mitocondriais durante uma etapa de permeabilização. Portanto, o procedimento descrito não é adequado para protocolos convencionais de imunofluorescência. Neste caso, os nucleóides em células fixas podem ser rotulados eficientemente com outras técnicas, como anticorpos contra o DNA ou fatores de transcrição mitocondrial (TFAM).
A rotulagem direta de nucleóides mitocondriais com o corante orgânico de ligação de DNA tem vantagens sobre a rotulagem de proteínas fluorescentes: qualquer tipo de célula pode ser rotulada dentro de <1 h, sem restrições temporais ou outras restrições de expressão transitória ou estável de construtos fluorescentes marcados por proteínas. Além disso, nos protocolos atuais, foi relatada a marcação convencional de proteínas fluorescentes de TFAM para causar artefatos. Além disso, a SG mancha eficientemente os nucleóides somente se o potencial da membrana mitocondrial estiver intacto. Isso impede a aquisição de imagens de células "doentes" e mortas biologicamente irrelevantes, que não podem ser evitadas com coloração baseada em FP. Finalmente, protocolos publicados anteriormente baseados em corantes de ligação de DNA não alcançaram a coloração preferencial do DNA mitocondrial.
O protocolo proposto é rápido e simples, por isso assumimos que será amplamente utilizado para imagens vivas de nucleóides mitocondriais por várias técnicas de fluorescência, tanto a difração limitada (por exemplo, confocagem a laser, TIRF, etc.) quanto sim de super-resolução.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores reconhecem Asifa Akhtar e Angelika Rambold (ambos do Instituto Max Planck de Imunobiologia e Epigenética) por fornecer células HeLa.
Materiais
| Name | Company | Catalog Number | Comments |
| Elyra PS1 | Carl Zeiss | multi-modal super-resolution microscope containing module for super-resolution structured illumination microscopy (SR-SIM) | |
| High glucose DMEM | GIBCO/ThermoFisher | 31966021 | |
| ibidi 35 mm dish, glass bottom | Ibidi Gmbh | 81158 | |
| ibidi 8-well microSlide, glass bottom | Ibidi Gmbh | 80827 | |
| Imaris 8.4.1 | Bitplane/Oxford Instruments | image porcessing and visualisation software package | |
| iXon 885 | Andor Technologies | EMCCD camera with back-illuminated sensor | |
| LSM880 Airyscan | Carl Zeiss | laser scanning confocal microscope with array detector | |
| Mitotracker CMXRos Red | ThermoFischer | M7512 | red live cell mitochondrial stain |
| Mitotracker Deep Red FM | ThermoFischer | M22426 | far red live cell mitochondrial stain |
| picoGreen | ThermoFischer | P7581 | cell permeant DNA stain |
| Plan Apochromat 100x/1.46 Oil objective | Carl Zeiss | ||
| SYBR Gold | ThermoFischer | S11494 | cell permeant DNA stain |
| Zen Black 2012 software | Carl Zeiss | image acquisition and processing software |
Referências
- Kaufman, B. A., et al. The mitochondrial transcription factor TFAM coordinates the assembly of multiple DNA molecules into nucleoid-like structures. Molecular Biology of the Cell. 18 (9), 3225-3236 (2007).
- Farge, G., et al. Protein sliding and DNA denaturation are essential for DNA organization by human mitochondrial transcription factor A. Nature Communications. 3, 1013(2012).
- Bogenhagen, D. F. Mitochondrial DNA nucleoid structure. Biochimica et Biophysica Acta. 1819 (9-10), 914-920 (2012).
- Kang, D., Kim, S. H., Hamasaki, N. Mitochondrial transcription factor A (TFAM): roles in maintenance of mtDNA and cellular functions. Mitochondrion. 7 (1-2), 39-44 (2007).
- Tauber, J., et al. Distribution of mitochondrial nucleoids upon mitochondrial network fragmentation and network reintegration in HEPG2 cells. International Journal of Biochemistry & Cell Biology. 45 (3), 593-603 (2013).
- Garrido, N., et al. Composition and dynamics of human mitochondrial nucleoids. Molecular Biology of the Cell. 14 (4), 1583-1596 (2003).
- Pernas, L., Scorrano, L. Mito-Morphosis: Mitochondrial Fusion, Fission, and Cristae Remodeling as Key Mediators of Cellular Function. Annual Review of Physiology. 78, 505-531 (2016).
- Caielli, S., et al. Oxidized mitochondrial nucleoids released by neutrophils drive type I interferon production in human lupus. Journal of Experimental Medicine. 213 (5), 697-713 (2016).
- Okayama, S., Ohta, K., Higashi, R., Nakamura, K. Correlative light and electron microscopic observation of mitochondrial DNA in mammalian cells by using focused-ion beam scanning electron microscopy. Microscopy (Oxf). 63 (Suppl 1), i35(2014).
- Alan, L., Spacek, T., Jezek, P. Delaunay algorithm and principal component analysis for 3D visualization of mitochondrial DNA nucleoids by Biplane FPALM/dSTORM. European Biophysics Journal. 45 (5), 443-461 (2016).
- Kukat, C., et al. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Kukat, C., Larsson, N. G. mtDNA makes a U-turn for the mitochondrial nucleoid. Trends in Cell Biology. 23 (9), 457-463 (2013).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Benke, A., Manley, S. Live-cell dSTORM of cellular DNA based on direct DNA labeling. ChemBioChem. 13 (2), 298-301 (2012).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79-87 (2016).
- Waldchen, S., Lehmann, J., Klein, T., van de Linde, S., Sauer, M. Light-induced cell damage in live-cell super-resolution microscopy. Scientific Reports. 5, 15348(2015).
- Gustafsson, M. G., et al. Three-dimensional resolution doubling in wide-field fluorescence microscopy by structured illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Hirano, Y., Matsuda, A., Hiraoka, Y. Recent advancements in structured-illumination microscopy toward live-cell imaging. Microscopy (Oxf). 64 (4), 237-249 (2015).
- Brown, T. A., et al. Superresolution fluorescence imaging of mitochondrial nucleoids reveals their spatial range, limits, and membrane interaction. Molecular and Cellular Biology. 31 (24), 4994-5010 (2011).
- Lewis, S. C., Uchiyama, L. F., Nunnari, J. ER-mitochondria contacts couple mtDNA synthesis with mitochondrial division in human cells. Science. 353 (6296), aaf5549(2016).
- Dellinger, M., Geze, M. Detection of mitochondrial DNA in living animal cells with fluorescence microscopy. Journal of Microscopy. 204 (Pt 3), 196-202 (2001).
- Ban-Ishihara, R., Ishihara, T., Sasaki, N., Mihara, K., Ishihara, N. Dynamics of nucleoid structure regulated by mitochondrial fission contributes to cristae reformation and release of cytochrome c. Proceedings of the National Academy of Sciences of the United States of America. 110 (29), 11863-11868 (2013).
- Zurek-Biesiada, D., et al. Localization microscopy of DNA in situ using Vybrant((R)) DyeCycle Violet fluorescent probe: A new approach to study nuclear nanostructure at single molecule resolution. Experimental Cell Research. 343 (2), 97-106 (2016).
- He, J. Y., et al. The AAA(+) protein ATAD3 has displacement loop binding properties and is involved in mitochondrial nucleoid organization. Journal of Cell Biology. 176 (2), 141-146 (2007).
- Ashley, N., Harris, D., Poulton, J. Detection of mitochondrial DNA depletion in living human cells using PicoGreen staining. Experimental Cell Research. 303 (2), 432-446 (2005).
- Jevtic, V., Kindle, P., Avilov, S. V. SYBR Gold dye enables preferential labeling of mitochondrial nucleoids and their time-lapse imaging by structured illumination microscopy. PLoS One. 13 (9), e0203956(2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados