Method Article
Spezifische Kennzeichnung von mitochondrialen Nukleoiden für die Zeitraffer-Struktur-Beleuchtungsmikroskopie
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt die spezifische Kennzeichnung von mitochondrialen Nukleoiden mit einem handelsüblichen DNA-Gelfleck, die Erfassung von Zeitrafferserien von Live-beschrifteten Zellen durch superauflösungsstrukturierte Beleuchtungsmikroskopie (SR-SIM) und die automatische Verfolgung von Nukleoidbewegungen.
Zusammenfassung
Mitochondriale Nukleoide sind kompakte Teilchen, die aus mitochondrialen DNA-Molekülen gebildet werden, die mit Proteinen beschichtet sind. Mitochondriale DNA kodiert tRNAs, rRNAs und mehrere wesentliche mitochondriale Polypeptide. Mitochondriale Nukleoide teilen und verteilen sich innerhalb des dynamischen mitochondrialen Netzwerks, das Spalt/Fusion und andere morphologische Veränderungen durchläuft. Die hochauflösende Live-Fluoreszenzmikroskopie ist eine einfache Technik, um die Position und Bewegung eines Nukleoids zu charakterisieren. Für diese Technik werden Nukleoide häufig durch fluoreszierende Tags ihrer Proteinkomponenten, nämlich Transkriptionsfaktor a (TFAM), gekennzeichnet. Diese Strategie erfordert jedoch eine Überexpression eines fluoreszierenden Protein-tagged-Konstrukts, das Artefakte verursachen kann (für TFAM gemeldet), und in vielen Fällen nicht durchführbar ist. Organische DNA-bindende Farbstoffe haben diese Nachteile nicht. Sie zeigen jedoch immer eine Färbung sowohl der nuklearen als auch der mitochondrialen DNAs, so dass es an Spezifität für mitochondriale Nukleoide fehlt. Unter Berücksichtigung der physikalisch-chemischen Eigenschaften solcher Farbstoffe wählten wir einen Nukleinsäure-Gelfleck (SYBR Gold) aus und erreichten eine bevorzugte Kennzeichnung von mitochondrialen Nukleoiden in lebenden Zellen. Die Eigenschaften des Farbstoffs, insbesondere seine hohe Helligkeit bei Bindung an die DNA, ermöglichen eine nachträgliche Quantifizierung der mitochondrialen Nukleoidbewegung mittels Zeitreihen von superauflösenden strukturierten Beleuchtungsbildern.
Einleitung
Zirkuläre 16,5 kbp DNA-Moleküle bilden das genetische Material von Mitochondrien, die 22 tRNAs, 2 rRNAs und 13 Polypeptide kodieren, die für mitochondriale oxidative Phosphorylierungskomplexe benötigt werden. Mitochondriale DNA gebunden an mitochondrialen Transkriptionsfaktor a (TFAM) und mehrere andere Proteine bilden die mitochondrialen Nukleoide1,2,3,4. Mitochondriale Nukleoide bewegen und verteilen sich zwischen den Komponenten des mitochondrialen Netzwerks5,6 während seiner morphologischen Umgestaltung, Spaltung oder Fusion je nach Zellzyklusphase, Stress und anderen Faktoren (in Pernas et al.7). Darüber hinaus ist die Bewegung von mitochondrialen Nukleoiden an der systemischen Lupus erythematodes-Krankheit8 beteiligt und kann bei anderen Krankheiten eine Rolle spielen. Fluoreszenzmikroskopie ist eine einfache Technik für Live-Zell-Studien von Organellen, aber die Technik hat eine Auflösung von >200 nm, die größer ist als die Größe der mitochondrialen Nukleoide (ca. 100 nm9,10,11,12). Diese Grenze wurde durch sogenannte "Super-Resolution"-Techniken wie stimulierte Emissionserschöpfung (STED) und Einzelmolekül-Lokalisierungsmikroskopie (SMLM)13,14umgangen. Bisher wurden mitochondriale Nukleoide und andere DNAs in lebenden Zellen durch direkte stochastische optische Rekonstruktionsmikroskopie (dSTORM)15abgebildet. Feine submitochondriale Strukturen mit Positionen, die mit mtDNA korrelierten, wurden von STED in lebenden Zellen16beobachtet. Diese Super-Resolution-Techniken erfordern jedoch eine hohe Beleuchtungsintensität, die phototoxische Effekte auf lebende Zellen verursacht17. Daher ist die Zeitraffer-Bildgebung von mitochondrialen Nukleoiden mit einer Auflösung jenseits der Beugungsgrenze eine Herausforderung. Um diesem Problem zu begegnen, haben wir eine superhochauflösende strukturierte Beleuchtungsmikroskopie (SR-SIM)18verwendet. SIM erfordert eine viel geringere Beleuchtungsleistung als STED und SMLM19. Darüber hinaus ermöglicht SIM im Gegensatz zu STED- und SMLM-Techniken eine einfache mehrfarbige dreidimensionale (3D) Bildgebung und erfordert keine besonderen photophysikalischen Eigenschaften der Fluorophore oder der bildgebenden Pufferzusammensetzung19.
Die herkömmliche Strategie zur Kennzeichnung mitochondrialer Nukleoide in lebenden Zellen ist die fluoreszierende Kennzeichnung eines mitochondrialen Nukleoidproteins wie TFAM20. In vielen Fällen ist diese Strategie jedoch nicht geeignet. Darüber hinaus erzeugt die Überexpression von fluoreszierendem, proteingetaggten TFAM ein ernstes Artefakt21. Die Kennzeichnung von DNA mit organischen Farbstoffen hat Vorteile gegenüber einer auf Fluoreszenzprotein (FP) basierenden Strategie. Organische Farbstoffe sind frei von Einschränkungen im Zusammenhang mit FP-Tagging: Sie können für jede Art von Zellen oder Gewebe verwendet werden und können zu jedem Zeitpunkt eines Experiments angewendet werden. Live-Zell-Bildgebung von mitochondrialen Nukleoiden wurde mit mehreren DNA-bindenden Farbstoffen berichtet: DAPI22, SYBR Green23, Vybrant DyeCycle24und picoGreen15,25,26. Ein wesentlicher Nachteil der meisten DNA-bindenden Farbstoffe für die Nukleoid-Etikettierung ist, dass sie die gesamte DNA innerhalb der Zelle färben. Es ist sehr wünschenswert, einen Farbstoff ausschließlich auf mitochondriale DNA auszurichten. Um dies zu erreichen, ist eine sorgfältige Auswahl eines Farbstoffs mit geeigneten physikalisch-chemischen Eigenschaften erforderlich. Lipophile Farbstoffe, die eine delokalisierte positive Ladung besitzen, wie Rhodamine 123, sind dafür bekannt, sich in lebenden Mitochondrien anzusammeln, die ihr negatives Membranpotenzial erhalten. Darüber hinaus sollte ein idealer Farbstoff für die spezifische Etikettierung von mitochondrialen Nukleoiden DNA mit hoher Affinität binden und helle Fluoreszenz bei DNA-Bindung emittieren. Unter Berücksichtigung dieser Anforderungen sind bestimmte Cyanine vielversprechend (z.B. picoGreen), aber Kern-DNA wird reichlich durch diese Farbstoffe gleichzeitig mit mitochondrialer DNA15,25,26gefärbt. Das vorliegende Protokoll beschreibt die spezifische Kennzeichnung von mitochondrialen Nukleoiden in lebenden Zellen mit einem anderen Cyaninfarbstoff, SYBR Gold (SG), und die Verfolgung der Nukleoide in Zeitraffer-SIM-Videos mit superauflösung. Darüber hinaus können SG-befleckte lebende Zellen mit jeder Art von invertiertem Fluoreszenzmikroskop (konfokale, Spinnscheibe, Epifluoreszenz usw.) abgebildet werden, das für lebende Zellen geeignet ist und mit einer 488 nm Lichtquelle ausgestattet ist.
Protokoll
HINWEIS: Alle hier erwähnten Zelllinien wurden in dem Medium Modified Eagle (DMEM) mit hohem Glukose-Dulbecco-Wert (DMEM) kultiviert, ergänzt durch 10% fetales Rinderserum (FBS), Glutamin, Penicillin/Streptomycin und Pyruvat. Gleichgewicht aller Medien und Nahrungsergänzungsmittel, die am Tag der Etikettierung und Bildgebung verwendet werden sollen, durch Erwärmung auf bis zu 37 °C in einem Inkubator, der auf 5%CO2eingestellt ist. Alle Zellkulturarbeiten einschließlich der Etikettierung finden unter sterilen Bedingungen unter einer laminaren Strömungshaube statt.
1. Live Cell Labeling
- Einen Tag vor dem Etikettierungsverfahren 4 x 105 HeLa-Zellen in 2 ml Medium in einer 35 mm Petrischale mit #1,5 Glasboden kulturieren. Es können mehrwellige Kammerschlitten verwendet werden, aber die Volumina sollten proportional zum Volumen des Brunnens angepasst werden. Verdünnen Sie die Zellsuspension, um ca. 50.000 Zellen/ml und Samen 2 ml der Zellsuspension in einer 35 mm Petrischale zu haben.
- Bereiten Sie die folgenden Verdünnungen der kommerziellen Aktienlösung SYBR Gold (SG) vor: 1:5.000. Bereiten Sie Mitotracker Deep Red (far red stain) oder Mitotracker CMXRos Red (roter Fleck)(Materialtabelle; kommerzieller Bestand verdünnt 1:2.000) in phenolrot-freiem Kulturmedium vor. Bereiten Sie die gleichen festgelegten Verdünnungen von picoGreen wie für SG vor.
HINWEIS: Die in Schritt 1.2 beschriebenen Lösungen sind die 2x Etikettierungslösungen. Schützen Sie alle Lösungen, die Fluoreszenzfarbstoffe enthalten, so weit wie möglich vor Licht. - Waschen Sie die anhaftenden Zellen einmal mit 2 ml PBS. 1 ml phenolrot-freies Kulturmedium in die 35 mm Petrischale geben. Verwenden Sie für einen 8-Well-Kammerschlitten ein Volumen, das 1/2 der gesamten Brunnenkapazität entspricht (z. B. 125 l hinzufügen, wenn die Gesamtbrunnenkapazität 250 l beträgt).
- Fügen Sie 1 ml der 2x Etikettierlösung in Schritt 1,2 bis 35 mm Petrischale vorbereitet. Verwenden Sie für eine 8-Well-Kammerrutsche ein Volumen, das 1/2 der gesamten Brunnenkapazität entspricht. Inkubieren Sie die Zellen für 30 min bei 37 °C unter 5%CO2.
HINWEIS: Inkubieren Sie die Zellen nicht länger als 1 h mit SG-Etikettierlösung, da eine längere Inkubation zur Kennzeichnung der Kern-DNA führt. - Das Medium, das die fluoreszierenden Farbstoffe enthält, vorsichtig ansaugen und die Zellen einmal mit PBS waschen.
- Phenolrotfreie Zellkulturmedium hinzufügen und die Zellen in2 einem CO2-Inkubator bei 37 °C bis zur Bildgebung im Dunkeln halten.
- Bild lebende Zellen auf einem SR-SIM-Mikroskop, das mit einer Inkubationseinheit ausgestattet ist (siehe unten).
2. SR-SIM Bildaufnahme
- Installieren Sie einen Bühnen-Top-Inkubator auf der Mikroskop-Bühne. Stellen Sie die gewünschte Temperatur undCO2-Konzentration (z. B. 37 °C und 5 % für Säugetierzellen) ein und halten Sie sich mindestens 1 h warm, bevor Sie mit der Bildaufnahme beginnen.
- Schalten Sie alle Komponenten des SR-SIM-Mikroskops, einschließlich der Laser, ein und lassen Sie sie mindestens 1 H aufwärmen.
- Wählen Sie ein vom Mikroskophersteller für SR-SIM empfohlenes Eintauchobjektiv mit hoher Vergrößerung und hoher numerischer Blende (NA) (z. B. 100x 1,46 NA Öl).
- Installieren Sie eine kammerierte Rutsche oder 35-mm-Schale mit beschrifteten Zellen (ab Schritt 1.6) auf der inkubierten Mikroskopstufe.
- Suchen Sie den Interessenbereich auf der Probe, vorzugsweise mit einem Okular. Um die beste SR-SIM-Bildqualität zu erzielen, wählen Sie die Zellen aus, die gut am Glas befestigt sind.
- Verwenden Sie eine hinterverzinnte High-End-Elektronenmultiplikations-Charge-Coupled Device (EM-CCD) Kamera, um SR-SIM-Bilder zu erfassen.
- Legen Sie mit der Bildaufnahmesoftware eine hohe EM-Verstärkung für die verwendete Kamera (z.B. 300) fest.
- Vor dem Erwerb der Zeitrafferserie für die Nukleoidverfolgung, erwerben Sie ein zweifarbiges SR-SIM-Bild des gleichen Sichtfeldes: einen Kanal für mitochondriale Färbung und einen weiteren für SG. Stellen Sie den ersten Farbkanal entsprechend dem verwendeten mitochondrialen Fleck ein (z. B. für einen weit roten mitochondrialen Fleck, stellen Sie die Anregung auf 633 nm oder länger und einen 650 nm Langpass-Emissionsfilter). Für das SG-Signal die Anregung auf 488 nm und einen Bandpass-Emissionsfilter von 500–550 nm einstellen.
- Mit der Software, stellen Sie die niedrigste Laserleistung möglich für sowohl 488 nm und weit rote Fleckenlinien.
HINWEIS: Eine typische Einstellung ist die 1% Laserausgangsleistung, die durch einen akusto-optischen Abstimmfilter (AOTF) eingestellt wird. - Wenn das SIM-Mikroskop Kanäle nur sequenziell erfasst (Einrichtung mit einer Kamera), schalten Sie den weitroten Fleckenerkennungskanal aus.
- Legen Sie die Anschaffung einer einzelnen Fokusebene fest, indem Sie die Z-Stack-Erfassung abschalten, indem Sie die Z-Stack-Box in der Software auskreuzen.
- Stellen Sie die kürzest mögliche EM-CCD-Kamerabelichtungszeit ein.
- Legen Sie drei Drehungen des Rasters anstelle von fünf Rotationen fest.
- Um die Bildrate zu erhöhen, stellen Sie in der Registerkarte "Akquise" der Software die Kamera so ein, dass die Daten nur aus dem zentralen Bereich des Kamerasensors statt "Full Chip" gelesen werden.
HINWEIS: Beispielsweise ermöglicht der Wechsel vom Lesen eines Bereichs von 1.000 x 1.000 Pixeln Vollsensor auf nur 256 x 256 Pixel eine Reduzierung der Kamerabelichtungszeit von 50 ms auf 13,4 ms und der gesamten Framezeit von 1,8 s auf 1,2 s. Wenn nur ein zentraler Teil des Kamerasensors gelesen wird, ist das Sichtfeld für ein 63-faches oder 100-faches Objektiv, das typischerweise für SR-SIM verwendet wird, sehr klein. Eine kürzere Belichtungszeit reduziert das Signal-Rausch-Verhältnis der Bilder. - Optimieren Sie die Laserleistung und die Belichtungszeit der Kamera: Erfassen Sie zweidimensionale SR-SIM-Bilder von beschrifteten Zellen mit mehreren Laserleistungswerten (z. B. 0,5 %, 1 %, 1,5 %, 2 %) und mehrere Belichtungszeiten (z. B. 13,4 ms, 25 ms, 50 ms).
- Verarbeiten Sie die unformatierten SIM-Datasets (siehe Schritt 3.1).
- Wählen Sie Laserleistungs- und Kamerabelichtungszeiten, die SIM-Bilder mit hellen Flecken in den Mitochondrien (d. h. den Nukleoiden) mit geringen oder gar keinen Artefakten ergeben, die durch SIM-Verarbeitung erzeugt werden. Inspizieren Sie die Bereiche außerhalb der Mitochondrien (z. B. 1% Leistung des 488 nm Festkörperlasers und 25 ms Belichtung der EM-CCD-Kamera).
- Beginnen Sie mit der Erfassung der Zeitrafferserie mit den in den Schritten 2.15–2.17 optimierten Einstellungen.
3. Datenverarbeitung und -analyse
- Verarbeiten Sie Roh-SIM-Datensätze mit dem strukturierten Beleuchtungsmodul einer geeigneten Software (Tabelle der Materialien): Wählen Sie das Kontrollkästchen Auto für SIM-Verarbeitungsparameter. Für die automatische Verarbeitung mehrerer Dateien verwenden Sie das Stapelverarbeitungstool, deaktivieren Sie Die Option Aktuelles für Batch verwenden, klicken Sie auf Batch ausführen, und wählen Sie mehrere Dateien für die SIM-Verarbeitung aus.
- Konvertieren Sie SIM-verarbeitete Zeitreihen-Datensätze mit imaris Dateikonverter-Software in das ims-Format (d. h. die Version, die der Version der Imaris-Software entspricht).
- Öffnen Sie eine konvertierte Datei in der Software (Version 8.4.1 oder höher), die über eine Lizenz für das Lineage-Modul (oder Track)-Modul verfügt.
- Starten Sie den Erstellungs-Assistenten "Spots", indem Sie auf das Symbol Neue Flecken hinzufügen klicken. Ein Assistent für die Erstellung wird gestartet.
- Wählen Sie die Registerkarte Erstellen des Assistenten aus.
- Klicken Sie im ersten Schritt des Assistenten auf Punkte verfolgen (im Laufe der Zeit), und fahren Sie mit dem zweiten Schritt des Assistenten fort.
- Legen Sie den geschätzten XY-Durchmesser auf 0,1-0,15 m fest, klicken Sie auf Hintergrundsubtraktion, und fahren Sie mit dem dritten Schritt fort.
- Passen Sie den Schwellenwert im Filter"Qualität" an, indem Sie die vertikale Linie im Histogramm ziehen, sodass die Mehrheit der Nukleoide als "Spots" erkannt werden, während die Artefakte nicht erkannt werden (d. h. die erkannten Flecken werden als Kugeln markiert, die auf dem Bild überlagert sind). Überprüfen Sie, ob dies auf jedem Frame der Fall ist, und justisieren Sie den Schwellenwert bei Bedarf. Fahren Sie mit der vierten und dann mit der fünften Stufe des Assistenten fort.
- Wählen Sie den Autoregressive Motion-Algorithmus aus. Stellen Sie max. Abstand auf 0,5 m ein. Stellen Sie die maximale Lückengröße auf 0 ein.
- Schauen Sie sich jeden Frame in der Zeitreihe an und überprüfen Sie, ob falsche Spuren gezeichnet werden und ob Lücken zwischen den Spuren eingeführt werden (Vom Assistenten erstellte Spuren mit aktuellen Einstellungen werden sofort als Linien markiert, die auf dem Bild überlagert sind). Passen Sie bei Bedarf "Max Distance" an. Fahren Sie mit dem sechsten Schritt des Assistenten fort. Wählen Sie den Filter Dauer verfolgen aus, und legen Sie einen Schwellenwert von 3–5 s fest.
- Wenn die aktuell erkannten Spots oder Tracks nicht optimal sind, kehren Sie zu den vorherigen Schritten des Assistenten zurück, indem Sie Navigationsschaltflächen (unten im Assistentenfenster) verwenden, die zum Optimieren aller Parameter für die Erstellung von Spots oder Spuren erforderlich sind.
- Wenn die fein abgestimmten Parameter es der Software ermöglichen, alle Spots zu erkennen und Tracks korrekt zu erstellen, klicken Sie auf die grüne Pfeil-Navigationsschaltfläche im Assistenten, um die Erstellung der Spuren zu bestätigen.
- Extrahieren Sie die Statistiken der Spuren, indem Sie auf das Symbolfenster Statistik klicken. Wählen Sie die erforderlichen Statistikparameter ( Registerkarten. Detail und Durchschnittswerte unter Statistik) aus und exportieren Sie die Werte als csv-Dateien, indem Sie auf das Diskettenlaufwerk-Symbol für die Quantifizierung und Visualisierung klicken.
HINWEIS: Maximale Spurgeschwindigkeit, mittlere Spurgeschwindigkeit, Streckenlänge und Gleisverschiebung sind Beispiele für wichtige Parameter. - Wenn das Bild-Dataset angezeigt oder veröffentlicht werden muss, erstellen Sie Snapshots und/oder Videodateien, die die Zeitrafferserie darstellen, mit optional überlagerten Spuren mithilfe der Snapshot- oder Animationswerkzeuge.
4. Konfokale Bildaufnahme
- Installieren Sie einen Bühnen-Inkubator auf der Mikroskopstufe, stellen Sie die gewünschte Temperatur undCO2-Konzentration ein (d. h. 37 °C und 5 % für Säugetierzellen) und halten Sie sich mindestens 1 h warm, bevor Sie mit der Bildaufnahme beginnen.
- Schalten Sie alle Komponenten eines konfokalen Laserscanmikroskops ein, einschließlich der Laser, und lassen Sie sie mindestens 1 H aufwärmen.
- Wählen Sie ein gewünschtes mittleres bis hohes Vergrößerungsziel und räumliche Stichproben nach Nyquist-Kriterien.
- Stellen Sie für den SG-Kanal die Anregung auf 488 nm und einen Erfassungsbereich von 500–550 nm ein. Für den mitochondrialen Fleckenkanal, eingestellt auf 561 nm und 570–630 nm Emission für einen roten Farbstoff, oder 633 nm Anregung und >650 nm Emission für einen weit roten Farbstoff.
- Stellen Sie die geringstmögliche Laserleistung (in der Regel 1 % oder weniger) ein, die die Erfassung der Signale mit PMT-Detektorgewinnen von 700 mV ermöglicht.
- Erfassen Sie zweifarbige konfokale Bilder der Zellen, bei denen die mitochondrialen Nukleoide im "SG-Kanal" und Mitochondrien im "roten" oder "far red" Kanal detektiert werden.
Ergebnisse
Charakterisierung der Live-Zellbeschriftung mit SG
Zunächst war die Verteilung von SG in den Zellen bei der Inkubation mit dem Farbstoff bei verschiedenen Verdünnungen durch konfokale Mikroskopie gekennzeichnet. Nach der Inkubation mit hohen Konzentrationen von SG oder PicoGreen markierten beide Farbstoffe meist die Kerne und zeigten eine Punktiatfärbung im Zytoplasma (Abbildung 1), ähnlich wie veröffentlichte Daten für einen anderen positiv geladenen Cyaninfarbstoff (d.h. picoGreen)15. Bei der Inkubation mit SG bei 1:10.000 Verdünnung traten jedoch schwache Färbungen in den Kernen auf, während wir im Zytoplasma ein Muster heller Flecken beobachteten(Abbildung 1). Auf der anderen Seite führte die Inkubation mit picoGreen Farbstoff verdünnt 1:10.000 meist nukleare Färbung. Das SG-Signal war viel heller als das von picoGreen bei gleicher Konzentration. Die Daten zeigten, dass SG besser für die Abbildung mitochondrialer DNA geeignet ist als andere ähnliche DNA-bindende Farbstoffe.
Um zu bestätigen, dass die hellen Punkte in den Mitochondrien lokalisiert sind, haben wir lebende Zellen gleichzeitig mit SG und weit roten mitochondrialen Flecken befleckt. Letzteres ist ein positiv geladener zelldurchlässiger organischer Farbstoff, der sich in den Mitochondrien lebender Zellen ansammelt. Bei der Inkubation mit SG bei 1:10.000 und 1:50.000 Verdünnungen traten fast alle SG-Färbungen in Mitochondrien auf, während bei der Kennzeichnung bei 1:500 und 1:1.000 Verdünnungen eine signifikante Färbung der Kerne und des Zytoplasmas auftrat(Abbildung 2).
Darüber hinaus charakterisierten wir den zeitlichen Verlauf der Färbung von lebenden Zellen mit SG durch Zeitraffermikroskopie (Abbildung 3). Die Plots der SG-Fluoreszenzintensität in Mitochondrien vs. Zeit (Abbildung 3B) legten nahe, dass die Nukleoidfärbung nach 45 min nahe an der Sättigung war. Daher empfehlen wir Inkubationszeiten von 30 bis 60 min.
Wir haben getestet, wie sich die intrazelluläre Verteilung der SG bei der Fixierung und/oder Permeabilisierung der Zellen ändert. Die Fixierung (2% Paraformaldehyd [PFA]) der lebend gefärbten Zellen verursachte eine leichte Umverteilung des Farbstoffs an den Kern (Abbildung 4A,B). Die Permeabilisierung der festen Zellen (0,1% Triton X100) eliminierte das SG-gepunktete Muster in Mitochondrien, und die Färbung der Kerne war dominant (Abbildung 4C). Wenn die SG den Zellen nach Fixierung und Permeabilisierung zugesetzt wurde, verteilte sie sich gleichmäßig über das Zytoplasma und die Kerne (Abbildung 4D). Somit können SG-markierte Zellen bei Bedarf fixiert werden, aber der Farbstoff ist nicht für Protokolle geeignet, die eine Permeabilisierung erfordern.
Live-Zelle SR-SIM und mitochondriale Nukleoide Tracking
Lebende Zellen, die mit SG und einem weit roten mitochondrialen Fleck(Tabelle der Materialien) gecostainiert wurden, wurden mit einer superhochauflösenden SIM-Technik in 3D dargestellt. Wie in den konfokalen Bildern (Abbildung 2) erschienen mitochondriale Nukleoide als helle Flecken innerhalb der Mitochondrien (Abbildung 5A). Darüber hinaus haben wir eine Zeitreihe von 2D-SIM-Bildern erworben und die Positionen der Nukleoide mit einer Auflösung jenseits der Beugungsgrenze nachverfolgt. Unmittelbar vor Beginn der Zeitreihe erwarben wir SIM 3D-Stacks in der SG und die mitochondrialen Fleckenkanäle. Dann haben wir nur eine Zeitreihe im SG-Kanal erworben und die mitochondrialen Nukleoide verfolgt, um ihre Bewegungen zu quantifizieren. Die mittlere Geschwindigkeit der Spur betrug 0,042 m/s, und die maximale Sofortgeschwindigkeit der Strecke betrug 0,078 x 0,012 m/s. Die Mehrheit der Nukleoide verdrängte nicht weit von ihren ursprünglichen Positionen entfernt, sondern zeigte zufällige Kurzstreckenbewegungen, die wahrscheinlich auf das mitochondriale Netzwerk beschränkt waren, wie eine Überlagerung von Spuren auf den mitochondrialen Bildern nahelegt (Abbildung 5B). Während einer typischen Zeitreihe (Film 1) traten nur wenige schnelle Richtungsverschiebungen auf.
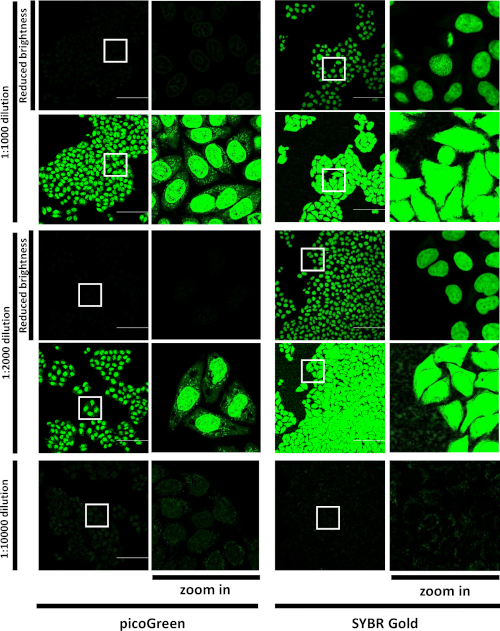
Abbildung 1: Vergleich von picoGreen und SG Live-Zellbeschriftung. A549-Zellen wurden mit PicoGreen oder SG bei indizierten Verdünnungen inkubiert und abgebildet. Repräsentative Sichtfelder der beschrifteten Zellen im grünen Kanal. LSM880 Airyscan FAST, 20x/Luftobjektiv, Skalenstange = 50 m. Einzelne optische Abschnitte. Weiße Quadrate markieren die Flächen, die mit einer höheren Vergrößerung rechts von den gesamten 423 x 423 'm Sichtfeldern dargestellt werden. Für 1:1.000 und 1:2.000 Verdünnungen werden die gleichen Bilder bei zwei Helligkeitseinstellungen angezeigt: die "Standard"-Helligkeitseinstellungen, die für 1:10.000 Verdünnung optimiert sind, und die "reduzierten Helligkeitseinstellungen", die angepasst wurden, um eine Sättigung des Detektors zu vermeiden. Diese Zahl wurde von Jevtic et al.27geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
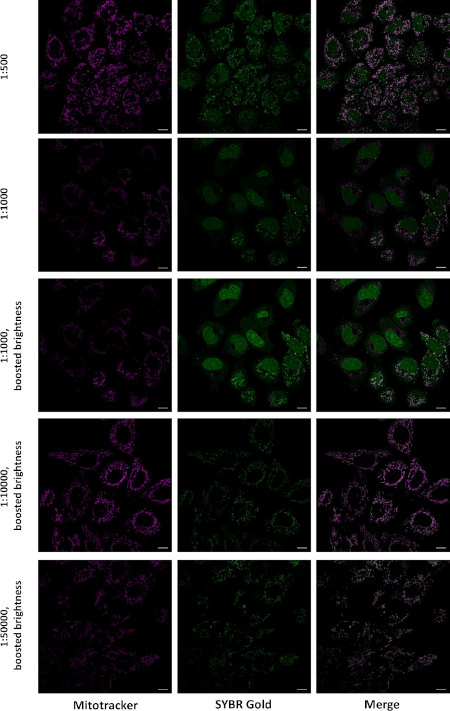
Abbildung 2: SG-Lokalisierung in lebenden Zellen bei Deretikettierung in unterschiedlichen Konzentrationen. HeLa-Zellen wurden 30 min lang mit einer Mischung aus 0,25 M Mitotracker CMXRos Red und der angegebenen SG-Verdünnung inkubiert. Die Lösung wurde durch DMEM ersetzt, und die Bilder wurden auf einem LSM880 Airyscan Mikroskop, 63x 1.4 Ölobjektiv, mit einer sequenziellen Erfassung von Farbkanälen aufgenommen. Einzelne optische Slices werden Scale bar = 10 m angezeigt. Diese Zahl wurde von Jevtic et al.27geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: HeLa-Zellen während der Etikettierung mit SYBR Gold (SG). Zuerst wurden lebende HeLa-Zellen mit Mitotracker CMXRos Red beschriftet und gewaschen. SG (Endverdünnung 1:10.000 in DMEM) wurde den Zellen hinzugefügt und eine Zeitrafferserie erworben. LSM880 Mikroskop, 63x 1.4 Ölobjektiv, sequentielle Erfassung. Z-Stacks wurden zu jedem Zeitpunkt erworben. Die maximalen Intensitätsprojektionen werden angezeigt. (A) Mittlere Intensitäten der SG-Fluoreszenz im Laufe der Zeit in mehreren Voninteressegebieten. (B) Repräsentative Sichtfelder, die die Interessengebiete anzeigen, in denen die SG-Fluoreszenz gemessen wurde (farbige Rechtecke). (C) Ein Sichtfeld an mehreren Zeitpunkten während der Inkubation mit SG. Ein quadratischer Bereich, der mit weißer Linie markiert ist, wird in der rechten Spalte mit einer höheren Vergrößerung angezeigt. Diese Zahl wurde von Jevtic et al.27geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Auswirkungen der Fixierung und Permeabilisierung auf die SG-Lokalisierung in den Zellen. HeLa-Zellen wurden 30 min lang mit SG (Stock verdünnt 1:10.000) und Mitotracker CMXRos Red (0,25 m) gefärbt. Einzelne optische Scheiben wurden auf einem Spinnscheibenmikroskop erworben; sequenzielle Akquisition; Skala bar = 10 m. (A) Live HeLa Zellen mit SG gebeizt. (B) Lebende HeLa-Zellen mit SG gefärbt und dann mit 2% PFA in PBS für 30 Min. fixiert. (C) Live HeLa Zellen mit SG gebeizt, dann mit 2% PFA in PBS für 30 min fixiert und mit 0,1% Triton X100 für 15 min permeabilisiert. Auf dem unteren Panel wird das gleiche Bild angezeigt, aber die Helligkeit im grünen Kanal ist höher eingestellt. (D) HeLa-Zellen mit 2% PFA fixiert, mit 0,1% Triton X100 für 15 min permeabilisiert und dann mit SG und Mitotracker CMXRos Red gefärbt. Um die in (D )gezeigten Bilder zu erfassen, wurde die EMCCD-Verstärkung der Kamera für den grünen Kanal im Vergleich zu A-C um den Faktor 12 reduziert, um eine Überbelichtung zu vermeiden. Diese Zahl wurde von Jevtic et al.27geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Nucleoid-Tracking auf Live-SIM-Bildern. Repräsentative Bilder eines Sichtfeldes von SG-Gefärbtenzellen. Unten: das zweifarbige SIM-Bild, das vor der Erfassung der Zeitrafferserie aufgenommen wurde. Grün = SG-Kanal; magenta = Mitotracker-Kanal. Oben: Nukleoidspuren aus einer 50-Frame-SIM-Zeitreihe im SG-Kanal (Film 1); Zeitrahmen = 1,8 s; Tracking von Imaris 8.4.1 Software. Die Spuren sind farbkodiert durch die maximale Spurgeschwindigkeit (m/s); Elyra PS.1, SIM-Modus, 100x/1.46 Ölobjektiv; Skalenbalken = 10 m. Diese Zahl wurde von Jevtic et al.27geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Film 1: SIM-Zeitrafferserie mit repräsentativen SG-befleckten Zellen. Skala bar = 5 m. Erkannte mitochondriale Nukleoide sind als weiße Kugeln markiert. Tracks werden als "Drachenschwänze" (8 Frames Länge) visualisiert und farbkodiert nach ihren maximalen Sofortgeschwindigkeiten (Farbbalken unten rechts zeigt Geschwindigkeiten in m/s). Mitochondriale Nukleoide Tracking und Visualisierung von Imaris 8.4.1 Software. Dieses Video wurde in Jevtic et al.27veröffentlicht. Bitte klicken Sie hier, um dieses Video anzusehen (Rechtsklick zum Download).
Diskussion
Es gibt mehrere kritische Komponenten des Protokolls: Um eine bevorzugte Kennzeichnung der mitochondrialen DNA zu erreichen, sollte die Konzentration des DNA-Bindungsfarbstoffs während der Inkubation sehr gering gehalten werden (z. B. eine Verdünnung eines typischen handelsüblichen Bestands von 1:10.000), und die Inkubationszeit sollte 30 min betragen. Die Inkubationszeit sollte niemals 1 h überschreiten. SYBR Goldfarbstoff sollte verwendet werden; andere DNA-bindende Farbstoffe sind nicht hell genug, um bei der Etikettierung bei geringer Konzentration ein starkes Signal zu erzeugen.
Die Einschränkung unseres Protokolls ist, dass der Farbstoff während eines Permeabilisierungsschritts aus den mitochondrialen Nukleoiden ausgewaschen wird. Daher ist das beschriebene Verfahren nicht für herkömmliche Immunfluoreszenzprotokolle geeignet. In diesem Fall können Nukleoide in festen Zellen effizient mit anderen Techniken wie Antikörpern gegen DNA oder mitochondriale Transkriptionsfaktoren (TFAM) gekennzeichnet werden.
Die direkte Etikettierung von mitochondrialen Nukleoiden mit dem DNA-bindenden organischen Farbstoff hat Vorteile gegenüber der fluoreszierenden Proteinkennzeichnung: Jede Art von Zelle kann innerhalb von <1 h gekennzeichnet werden, ohne zeitliche oder andere Beschränkungen der transienten oder stabilen Expression fluoreszierender Protein-getaggter Konstrukte. Auch in aktuellen Protokollen wurde berichtet, dass herkömmliche fluoreszierende Protein-Tagging von TFAM Artefakte verursachen. Darüber hinaus färbt SG Nukleoide nur dann effizient, wenn das mitochondriale Membranpotenzial intakt ist. Dies verhindert die Bildaufnahme von biologisch irrelevanten "kranken" und abgestorbenen Zellen, die mit FP-basierter Färbung nicht vermieden werden können. Schließlich erreichten zuvor veröffentlichte Protokolle, die auf DNA-Bindenden basierten, keine bevorzugte Färbung der mitochondrialen DNA.
Das vorgeschlagene Protokoll ist schnell und einfach, daher gehen wir davon aus, dass es weit verbreitet für die Live-Bildgebung von mitochondrialen Nukleoiden durch verschiedene Fluoreszenztechniken verwendet wird, sowohl Beugung begrenzt (z. B. Laserscanning konfokale, TIRF, etc.) als auch SuperauflösungSIM.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren würdigen Asifa Akhtar und Angelika Rambold (beide Max-Planck-Institut für Immunbiologie und Epigenetik) für die Bereitstellung von HeLa-Zellen.
Materialien
| Name | Company | Catalog Number | Comments |
| Elyra PS1 | Carl Zeiss | multi-modal super-resolution microscope containing module for super-resolution structured illumination microscopy (SR-SIM) | |
| High glucose DMEM | GIBCO/ThermoFisher | 31966021 | |
| ibidi 35 mm dish, glass bottom | Ibidi Gmbh | 81158 | |
| ibidi 8-well microSlide, glass bottom | Ibidi Gmbh | 80827 | |
| Imaris 8.4.1 | Bitplane/Oxford Instruments | image porcessing and visualisation software package | |
| iXon 885 | Andor Technologies | EMCCD camera with back-illuminated sensor | |
| LSM880 Airyscan | Carl Zeiss | laser scanning confocal microscope with array detector | |
| Mitotracker CMXRos Red | ThermoFischer | M7512 | red live cell mitochondrial stain |
| Mitotracker Deep Red FM | ThermoFischer | M22426 | far red live cell mitochondrial stain |
| picoGreen | ThermoFischer | P7581 | cell permeant DNA stain |
| Plan Apochromat 100x/1.46 Oil objective | Carl Zeiss | ||
| SYBR Gold | ThermoFischer | S11494 | cell permeant DNA stain |
| Zen Black 2012 software | Carl Zeiss | image acquisition and processing software |
Referenzen
- Kaufman, B. A., et al. The mitochondrial transcription factor TFAM coordinates the assembly of multiple DNA molecules into nucleoid-like structures. Molecular Biology of the Cell. 18 (9), 3225-3236 (2007).
- Farge, G., et al. Protein sliding and DNA denaturation are essential for DNA organization by human mitochondrial transcription factor A. Nature Communications. 3, 1013(2012).
- Bogenhagen, D. F. Mitochondrial DNA nucleoid structure. Biochimica et Biophysica Acta. 1819 (9-10), 914-920 (2012).
- Kang, D., Kim, S. H., Hamasaki, N. Mitochondrial transcription factor A (TFAM): roles in maintenance of mtDNA and cellular functions. Mitochondrion. 7 (1-2), 39-44 (2007).
- Tauber, J., et al. Distribution of mitochondrial nucleoids upon mitochondrial network fragmentation and network reintegration in HEPG2 cells. International Journal of Biochemistry & Cell Biology. 45 (3), 593-603 (2013).
- Garrido, N., et al. Composition and dynamics of human mitochondrial nucleoids. Molecular Biology of the Cell. 14 (4), 1583-1596 (2003).
- Pernas, L., Scorrano, L. Mito-Morphosis: Mitochondrial Fusion, Fission, and Cristae Remodeling as Key Mediators of Cellular Function. Annual Review of Physiology. 78, 505-531 (2016).
- Caielli, S., et al. Oxidized mitochondrial nucleoids released by neutrophils drive type I interferon production in human lupus. Journal of Experimental Medicine. 213 (5), 697-713 (2016).
- Okayama, S., Ohta, K., Higashi, R., Nakamura, K. Correlative light and electron microscopic observation of mitochondrial DNA in mammalian cells by using focused-ion beam scanning electron microscopy. Microscopy (Oxf). 63 (Suppl 1), i35(2014).
- Alan, L., Spacek, T., Jezek, P. Delaunay algorithm and principal component analysis for 3D visualization of mitochondrial DNA nucleoids by Biplane FPALM/dSTORM. European Biophysics Journal. 45 (5), 443-461 (2016).
- Kukat, C., et al. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Kukat, C., Larsson, N. G. mtDNA makes a U-turn for the mitochondrial nucleoid. Trends in Cell Biology. 23 (9), 457-463 (2013).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Benke, A., Manley, S. Live-cell dSTORM of cellular DNA based on direct DNA labeling. ChemBioChem. 13 (2), 298-301 (2012).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79-87 (2016).
- Waldchen, S., Lehmann, J., Klein, T., van de Linde, S., Sauer, M. Light-induced cell damage in live-cell super-resolution microscopy. Scientific Reports. 5, 15348(2015).
- Gustafsson, M. G., et al. Three-dimensional resolution doubling in wide-field fluorescence microscopy by structured illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Hirano, Y., Matsuda, A., Hiraoka, Y. Recent advancements in structured-illumination microscopy toward live-cell imaging. Microscopy (Oxf). 64 (4), 237-249 (2015).
- Brown, T. A., et al. Superresolution fluorescence imaging of mitochondrial nucleoids reveals their spatial range, limits, and membrane interaction. Molecular and Cellular Biology. 31 (24), 4994-5010 (2011).
- Lewis, S. C., Uchiyama, L. F., Nunnari, J. ER-mitochondria contacts couple mtDNA synthesis with mitochondrial division in human cells. Science. 353 (6296), aaf5549(2016).
- Dellinger, M., Geze, M. Detection of mitochondrial DNA in living animal cells with fluorescence microscopy. Journal of Microscopy. 204 (Pt 3), 196-202 (2001).
- Ban-Ishihara, R., Ishihara, T., Sasaki, N., Mihara, K., Ishihara, N. Dynamics of nucleoid structure regulated by mitochondrial fission contributes to cristae reformation and release of cytochrome c. Proceedings of the National Academy of Sciences of the United States of America. 110 (29), 11863-11868 (2013).
- Zurek-Biesiada, D., et al. Localization microscopy of DNA in situ using Vybrant((R)) DyeCycle Violet fluorescent probe: A new approach to study nuclear nanostructure at single molecule resolution. Experimental Cell Research. 343 (2), 97-106 (2016).
- He, J. Y., et al. The AAA(+) protein ATAD3 has displacement loop binding properties and is involved in mitochondrial nucleoid organization. Journal of Cell Biology. 176 (2), 141-146 (2007).
- Ashley, N., Harris, D., Poulton, J. Detection of mitochondrial DNA depletion in living human cells using PicoGreen staining. Experimental Cell Research. 303 (2), 432-446 (2005).
- Jevtic, V., Kindle, P., Avilov, S. V. SYBR Gold dye enables preferential labeling of mitochondrial nucleoids and their time-lapse imaging by structured illumination microscopy. PLoS One. 13 (9), e0203956(2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten