Method Article
시간 경과 구조화 조명 현미경 검사법에 대한 미토콘드리아 핵의 특정 라벨링
요약
이 프로토콜은 시판되는 DNA 젤 얼룩이 있는 미토콘드리아 핵종의 특정 라벨링, 초고해상도 구조조명 현미경(SR-SIM)에 의한 라이브 라벨링 세포의 타임랩스 시리즈 획득, 핵운동의 자동 추적을 설명합니다.
초록
미토콘드리아 핵형성은 단백질로 코팅된 미토콘드리아 DNA 분자에 의해 형성된 소형 입자입니다. 미토콘드리아 DNA는 tRNA, rRNAs 및 몇몇 필수 미토콘드리아 폴리펩티드를 인코딩합니다. 미토콘드리아 핵형성은 핵분열/융합 및 기타 형태학적 변화를 겪는 동적 미토콘드리아 네트워크 내에서 분열및 분배됩니다. 고해상도 라이브 형광 현미경 검사법은 핵신의 위치와 움직임을 특성화하는 간단한 기술입니다. 이 기술을 위해, 핵형성은 그들의 단백질 분대, 즉 전사 인자 a (TFAM)의 형광 태그를 통해 일반적으로 표지됩니다. 그러나 이 전략은 아티팩트(TFAM에 대해 보고됨)를 유발할 수 있는 형광 단백질 태그가 지정된 구성물의 과발현이 필요하며 많은 경우에 실현 가능하지 않습니다. 유기 DNA 결합 염료는 이러한 단점을 가지고 있지 않습니다. 그러나, 그(것)들은 항상 핵과 미토콘드리아 DNA 둘 다의 얼룩을 보여주기, 따라서 미토콘드리아 핵형성에 특이성이 결여됩니다. 이러한 염료의 물리 화학적 특성을 고려하여 핵산 젤 얼룩(SYBR Gold)을 선택하고 살아있는 세포에서 미토콘드리아 핵세포의 우대 라벨링을 달성했습니다. 염료의 특성, 특히 DNA에 결합시 높은 밝기는 초고해상도 구조 조명 이미지의 타임 시리즈를 사용하여 미토콘드리아 핵운동의 후속 정량화를 허용합니다.
서문
원형 16.5 kbp DNA 분자는 미토콘드리아의 유전 물질을 구성하며, 미토콘드리아 산화 인산화 복합체에 필요한 22 tRNAs, 2 rRNAs 및 13개의 폴리펩티드를 인코딩합니다. 미토콘드리아 DNA는 미토콘드리아 전사 인자 a(TFAM) 및 여러 다른 단백질에 결합하여 미토콘드리아 핵형성1,,2,,3,,4. 미토콘드리아 핵형성은 세포 주기 상, 스트레스 및 기타 요인에 따라 형태학적리모델링,핵분열 또는 융합동안 미토콘드리아네트워크의성분 들 간에 이동 및 재분배(Pernas et al.7). 또한, 미토콘드리아 핵소자의 움직임은, 전신 성 홍반성 루푸스 질환8에 연루되어 다른 질병에 역할을 할 수 있다. 형광 현미경 검사법은 세포기관의 살아있는 세포 연구를 위한 간단한 기술입니다, 그러나 기술은 미토콘드리아 핵형성의 크기보다는 더 큰 >200 nm의 해결책을 가지고 있습니다 (~100,nm9,,10,,12).12 이러한 한계는 자극방출고갈(STED) 및 단일 분자 국소화 현미경(SMLM)13,,14와같은 소위 "초고해상도" 기술에 의해 회피되었다. 지금까지, 미토콘드리아 핵소자 및 기타 DNA는 직접 적인 현미경(dSTORM)15에의해 살아있는 세포에서 영상화되었다. mtDNA와 상관관계가 있는 위치를 가진 미세한 서브-미토콘드리아 구조는 살아있는세포(16)에서STED에 의해 관찰되었다. 그러나, 이러한 초고해상도 기술은 살아있는 세포에 광독성 효과를 일으키는 높은 발광 강도를 필요로17. 따라서 회절 한계를 초과하는 해상도를 가진 미토콘드리아 핵종의 시간 경과 이미징은 어렵습니다. 이 문제를 해결하기 위해 초해상도 구조조명 현미경(SR-SIM)18을사용했습니다. SIM은 STED 및 SMLM19보다훨씬 낮은 조명 전력 용량을 필요로합니다. 더욱이, STED 및 SMLM 기술과는 달리, SIM은 간단한 다색 3차원(3D) 이미징을 허용하며, 형광단 또는 이미징 버퍼조성물(19)의특정 광물리적 특성을 필요로 하지 않는다.
살아있는 세포에서 미토콘드리아 핵을 표지하기 위한 종래의 전략은 TFAM20과같은 미토콘드리아 핵형 단백질의 형광 태깅이다. 그러나 대부분의 경우 이 전략은 적합하지 않습니다. 더욱이, 형광 단백질 태그가 붙은 TFAM의 과발현은 심각한아티팩트(21)를생성한다. 유기 염료로 DNA를 표지하는 것은 형광 단백질 (FP) 기반 전략에 비해 장점이 있습니다. 유기 염료는 FP 태깅과 관련된 제한이 없습니다 : 그들은 세포 또는 조직의 모든 유형에 사용할 수 있으며 실험의 모든 시점에서 적용 할 수 있습니다. 미토콘드리아 핵소자의 살아있는 세포 이미징은 여러 DNA 결합 염료로 보고되었다: DAPI22,SYBR 그린23,비브란트 염료사이클24,및 피코그린15,,25,,26. 핵염균 표지를 위한 대부분의 DNA 결합 염료의 실질적인 결점은 세포 내의 모든 DNA를 얼룩지게 한다는 것입니다. 미토콘드리아 DNA에전적으로 염료를 표적으로 하는 것은 매우 바람직합니다. 이를 달성하기 위해서는 적절한 물리 화학 적 특성을 지닌 염료를 신중하게 선택해야합니다. 로다민(123)과 같은 국소화된 양성전하를 보유한 친유성 염료는 살아있는 미토콘드리아에 축적되어 부정적인 막 전위를 보존하는 것으로 알려져 있다. 또한, 미토콘드리아 핵소자의 특정 라벨링을 위한 이상적인 염료는 높은 친화력으로 DNA를 결합하고 DNA 결합시 밝은 형광을 방출해야 한다. 이러한 요구 사항을 고려할 때, 특정 시아닌은 유망하지만(예를 들어, 피코그린), 핵 DNA는 미토콘드리아 DNA15,,25,,26과동시에 이들 염료에 의해 풍부하게 염색된다. 본 프로토콜은 다른 시아닌 염료, SYBR 골드 (SG) 및 시간 경과 슈퍼 해상도 SIM 비디오에서 핵의 추적과 라이브 세포에서 미토콘드리아 핵의 특정 라벨을 설명합니다. 더욱이, SG 염색된 살아있는 세포는 살아있는 세포에 적합한 모든 유형의 반전형광 현미경(공초점, 회전 디스크, 에피네시 등)에 의해 이미지화될 수 있으며 488 nm 광원을 갖추고 있다.
프로토콜
참고: 여기에서 언급된 모든 세포주들은 10% 태아 소 혈청(FBS), 글루타민, 페니실린/스트렙토마이신 및 피루바테로 보충된 고당포도도당 덜베코의 변형 된 독수리 배지 (DMEM)에서 배양되었습니다. 인큐베이터에서 최대 37°C까지 예열하여 라벨링 및 이미징 당일에 사용되는 모든 매체 및 보충제를 5%CO2로평형화한다. 라벨링을 포함한 모든 세포 배양 작업은 층류 후드 하에서 멸균 조건에서 일어난다.
1. 라이브 셀 라벨링
- 라벨링 절차 하루 전에, 배양 4 x 105 HeLa 세포를 35 mm 페트리 접시에 배지의 2 mL에서 #1.5 유리 바닥. 멀티 웰 챔버 슬라이드를 사용할 수 있지만, 볼륨은 웰의 볼륨에 비례하여 조정되어야한다. 세포 현탁액을 희석하여 약 50,000 개의 세포/mL및 종자 2 mL의 세포 현탁액을 35 mm 페트리 접시에 갖는다.
- SYBR 골드(SG) 상용 재고 솔루션의 다음 희석을 준비합니다: 1:5,000. 페놀 무첨가 배양 배지에서 미토트래커 딥 레드(원붉은 얼룩) 또는 미토트래커 CMXRos레드(적색 얼룩)(재료표;상업용 재고 희석 1:2,000)를 준비한다. SG와 동일한 피코그린 세트 희석을 준비합니다.
참고: 1.2단계에서 설명한 솔루션은 2x 라벨링 솔루션입니다. 형광 염료를 함유한 모든 용액을 가능한 한 빛으로부터 보호하십시오. - 부착 세포를 2 mL PBS로 한 번 씻으하십시오. 페놀 무방배 배지 1 ml를 35mm 페트리 접시에 넣습니다. 8웰 챔버 드 슬라이드의 경우 총 웰 용량의 1/2에 해당하는 부피를 사용합니다(예: 총 웰 용량이 250 μL인 경우 125 μL추가).
- 1.2 ~ 35mm 페트리 접시에서 제조 된 2x 라벨링 용액 1 mLof를 추가하십시오. 8웰 챔버 슬라이드의 경우 총 웰 용량의 1/2에 해당하는 볼륨을 사용합니다. 5%CO2하에서 37°C에서 30분 동안 세포를 배양한다.
참고: 더 긴 배양이 핵 DNA의 표지를 일으키는 원인이 되기 때문에, 1 시간 이상 SG 표지 해결책으로 세포를 배양하지 마십시오. - 형광 염료를 함유하는 배지를 조심스럽게 흡인하고 PBS로 세포를 한 번 세척합니다.
- 페놀 적색 무세포 배양 배지를 첨가하고CO2 인큐베이터에서 37°C에서 이미징까지 암흑에 세포를 유지한다.
- 인큐베이션 유닛이 장착된 SR-SIM 현미경상 상빙 세포(아래 참조).
2. SR-SIM 이미지 수집
- 현미경 단계에 스테이지 탑 인큐베이터를 설치합니다. 원하는 온도 및CO2 농도(예: 포유류 세포의 경우 37°C 및 5%)를 설정하고 이미지 수집을 시작하기 전에 적어도 1시간 동안 따뜻하게 유지합니다.
- 레이저를 포함한 SR-SIM 현미경의 모든 구성 요소를 켜고 적어도 1 시간 동안 따뜻하게 하십시오.
- 현미경 제조업체에서 SR-SIM에 권장하는 고배율, 높은 수치 조리개(NA) 침지 목표(예: 100x 1.46 NA 오일)를 선택하십시오.
- 배양 된 현미경 단계에 라벨이 부착 된 세포 (단계 1.6에서)로 챔버 슬라이드 또는 35mm 접시를 설치하십시오.
- 가급적 안구와 함께 샘플에 관심 영역을 찾습니다. 최상의 SR-SIM 이미징 품질을 얻으려면 유리에 잘 부착된 셀을 선택합니다.
- SR-SIM 이미지를 획득하기 위해 백 틴트 하이 엔드 전자 증식 충전 장치 (EM-CCD) 카메라를 사용합니다.
- 이미지 수집 소프트웨어를 사용하여 사용되는 카메라(예: 300)에 권장되는 높은 EM 게인을 설정합니다.
- 핵 추적을 위한 타임랩스 시리즈를 획득하기 전에 동일한 시야의 2색 SR-SIM 이미지를 획득합니다: 미토콘드리아 염색을 위한 채널 1개, SG용 채널 1개. 사용된 미토콘드리아 얼룩에 적합한 제1 색 채널을 설정합니다(예를 들어, 멀리 붉은 미토콘드리아 얼룩의 경우, 633 nm 이상및 650 nm 롱패스 방출 필터)에서 여기를 설정합니다. SG 신호의 경우 여기를 488 nm및 500-550 nm 대역 통과 방출 필터로 설정합니다.
- 이 소프트웨어를 사용하여 488 nm 및 원거리 적색 얼룩 선 모두에서 가능한 가장 낮은 레이저 전력을 설정합니다.
참고: 일반적인 설정은 acousto 광학 튜닝 필터(AOTF)에 의해 조정된 1% 레이저 출력 전력입니다. - SIM 현미경이 순차적으로(단일 카메라 설정) 채널만 획득하는 경우 원붉은 얼룩 감지 채널을 끕니다.
- 소프트웨어의 Z 스택 상자를 선택 해제하여 Z 스택 수집을 끄고 단일 초점 평면의 획득을 설정합니다.
- 가능한 한 짧은 EM-CCD 카메라 노출 시간을 설정합니다.
- 다섯 번의 회전이 아닌 그리드의 세 회전을 설정합니다.
- 프레임 속도를 높이려면 소프트웨어의 수집 탭에서"Full Chip"대신 카메라 센서의 중앙 영역에서만 데이터를 읽도록 카메라를 설정합니다.
참고: 예를 들어 1,000 x 1,000픽셀 전체 센서의 영역을 읽기에서 256 x 256 픽셀만 읽는 것으로 전환하면 카메라 노출 시간이 50ms에서 13.4ms로 감소하고 총 프레임 시간이 1.8초에서 1.2초로 줄어듭니다. 카메라 센서의 중앙 부분만 읽는 경우 일반적으로 SR-SIM에 사용되는 63x 또는 100x 대물렌즈의 시야는 매우 작습니다. 노출 시간이 짧을수록 이미지의 신호 대 잡음 비율이 줄어듭니다. - 레이저 전력 및 카메라 노출 시간 최적화: 여러 레이저 전력 값(예: 0.5%, 1%, 1.5%, 2%)에서 레이블이 부착된 셀의 SR-SIM 2차원(2D) 이미지 획득 및 여러 노출 시간 (예를 들어, 13.4 ms, 25 ms, 50 ms).
- 원시 SIM 데이터 집합을 처리합니다(3.1 단계 참조).
- SIM 처리에 의해 생성된 아티팩트가 거의 또는 전혀 없는 미토콘드리아(즉, 핵형성)에서 밝은 반점이 있는 SIM 이미지를 생성하는 레이저 전력 및 카메라 노출 시간을 선택합니다. 미토콘드리아 외부 영역을 검사합니다(예: 488 nm 솔리드 스테이트 레이저의 1% 전력 및 EM-CCD 카메라의 25ms 노출).
- 단계 2.15-2.17에 최적화된 설정을 사용하여 타임랩스 계열을 수집하기 시작합니다.
3. 데이터 처리 및 분석
- 적합한 소프트웨어의 구조화 된 조명 모듈로 원시 SIM 데이터 세트를 처리하십시오(재료 표):SIM 처리 매개 변수에 대한 자동 확인란을 선택합니다. 여러 파일을 자동으로 처리하려면 일괄 처리 도구를 사용하고 일괄 처리에 대해 현재 사용을선택 취소하고 일괄 처리를 클릭하고 SIM 처리를 위해 여러 파일을 선택합니다.
- SIM 처리된 열계 데이터 세트를 Imaris 파일 변환기 소프트웨어(즉, Imaris 소프트웨어 버전에 해당하는 버전)를 사용하여 ims 형식으로 변환합니다.
- 리니지(또는 트랙) 모듈에 대한 라이센스가 있는 소프트웨어(버전 8.4.1 이상)에서 변환된 파일을 엽니다.
- "스팟Spots만들기 마법사"를 시작하여 새 관광명소 추가 아이콘을 클릭합니다. 생성을 위한 마법사가 시작됩니다.
- 마법사의 만들기 탭을 선택합니다.
- 마법사의 첫 번째 단계에서 시간이 지남에 따라 지점 추적을 클릭하고 마법사의 두 번째 단계로 진행합니다.
- 예상 XY 지름을 0.1-0.15 μm으로 설정하고 배경 빼기를 클릭하고 세 번째 단계로 진행합니다.
- 히스토그램에서 수직 선을 드래그하여필터의임계값을 조정하여 대부분의 핵소식이 "반점"으로 감지되도록 하고 아티팩트는 감지되지 않습니다(즉, 감지된 반점은 이미지에 오버레이된 공으로 표시됩니다). 각 프레임의 경우인지 확인하고 필요한 경우 임계값을 다시 지정합니다. 네 번째 단계로 이동한 다음 마법사의 다섯 번째 단계로 이동합니다.
- 자동 회귀 모션 알고리즘을 선택합니다. 최대 거리를 0.5μm로 설정합니다. 최대 간격 크기를 0으로 설정합니다.
- 타임 시리즈의 각 프레임을 보고 잘못된 트랙이 그려져 있는지, 트랙 사이의 간격이 있는지 확인합니다(현재 설정이 있는 마법사가 빌드한 트랙은 이미지에 선으로 즉시 표시됩니다). 필요한 경우 "최대 거리"를 조정합니다. 마법사의 여섯 번째 단계로 진행합니다. 트랙 지속 시간 필터를 선택하고 3-5s의 임계값을 설정합니다.
- 현재 검색된 스팟이나 트랙이 최적이 아닌 경우 스팟 또는 트랙 생성을 위한 매개 변수를 미세 조정하는 데 필요한 탐색 단추(마법사 창 아래)를 사용하여 마법사의 이전 단계로 돌아갑니다.
- 미세 조정된 매개 변수를 통해 소프트웨어가 모든 스팟을 감지하고 트랙을 올바르게 빌드할 수 있는 경우 마법사의 녹색 화살표 탐색 단추를 클릭하여 트랙 생성을 확인합니다.
- 통계 아이콘 창을 클릭하여 트랙의 통계를 추출합니다. 필요한 통계 매개 변수(통계아래의상세 값 및 평균 값 탭)를 선택하고 정량화 및 시각화를 위해 플로피 드라이브 아이콘을 클릭하여 값을 csv 파일로 내보냅니다.
참고: 최대 트랙 속도, 평균 트랙 속도, 트랙 길이 및 트랙 변위는 중요한 매개변수의 예입니다. - 이미지 데이터 집합을 표시하거나 게시해야 하는 경우 스냅샷 또는 애니메이션 도구를 사용하여 선택적으로 오버레이된 트랙이 있는 타임랩스 시리즈를 나타내는 스냅샷 및/또는 비디오 파일을 만듭니다.
4. 공초점 이미지 획득
- 현미경 단계에 스테이지 탑 인큐베이터를 설치하고, 원하는 온도 및CO2 농도(즉, 포유류 세포의 경우 37°C 및 5%)를 설정하고 이미지 수집을 시작하기 전에 적어도 1시간 동안 따뜻하게 유지합니다.
- 레이저를 포함한 공초점 레이저 스캐닝 현미경의 모든 구성 요소를 켜고 적어도 1 시간 동안 따뜻하게 하십시오.
- 나이퀴스트 기준에 따라 원하는 중간에서 높은 배율 대및 공간 샘플링을 선택합니다.
- SG 채널의 경우 여기를 488 nm및 500-550 nm의 검출 범위를 설정합니다. 미토콘드리아 얼룩 채널의 경우, 적색 염료에 대해 561 nm 및 570-630 nm 방출, 또는 633 nm 여기 및 >650 nm 방출을 멀리 적색 염료에 대해 설정한다.
- PMT 검출기 이득이 700mV인 신호를 수집할 수 있는 가능한 최저 레이저 전력(일반적으로 1% 이하)을 설정합니다.
- "SG 채널"에서 미토콘드리아 핵이 검출되고 미토콘드리아가 "적색" 또는 "원적" 채널에서 검출되는 세포의 2색 공초점 이미지를 획득합니다.
결과
SG를 통해 라이브 셀 라벨링의 특성화
먼저, 다양한 희석제에서 염료를 배양시 세포내SG의 분포는 공초점 현미경으로 특징지어졌다. SG 또는 피코그린의 고농도로 배양한 후, 둘 다 염료는 대부분 핵에 표지하고 세포질에서 반점 염색을 나타내었다(도1),유사하게 다른 양전하파된 사이아닌 염료(즉, 피코그린)에 대한 출판된 데이터와 유사하게15. 그러나, 1:10,000 희석에서 SG로 배양시, 희미한 염색이 핵에 나타났고, 세포질에서는 밝은 반점의 패턴을관찰하였다(도 1). 한편, 피코그린 염료를 1:10,000으로 배양하여 대부분 핵 염색을 산출했다. SG 신호는 동일한 농도에서 피코그린보다 훨씬 밝습니다. 데이터는 SG가 그밖 유사한 DNA 결합 염료 보다는 화상 진찰 미토콘드리아 DNA를 위해 더 적당하다는 것을 보여주었습니다.
밝은 점이 미토콘드리아에 국한되어 있는지 확인하기 위해 SG및 멀리 붉은 미토콘드리아 얼룩과 동시에 살아있는 세포를 염색했습니다. 후자는 살아있는 세포의 미토콘드리아에 축적되는 양전하 세포 투과성 유기 염료입니다. 1:10,000 및 1:50,000 희석에서 SG를 배양한 후, 거의 모든 SG 염색이 미토콘드리아에서 발생하였고, 1:500 및 1:1,000 희석에 라벨링시, 핵 및 세포질의 유의한 염색이 발생하였다(그림2).
또한, SG로 살아있는 세포를 시간 경과 현미경으로 염색하는 시간 과정을 특징으로하였다(도 3). 미토콘드리아 대시간(그림 3B)에서SG 형광 강도의 플롯은 45분 후, 핵염색이 포화에 가까웠다는 것을 시사한다. 따라서 ~ 30-60 분의 인큐베이션 시간을 권장합니다.
우리는 세포의 고정 및 / 또는 투과에 따라 SG 세포 내 분포가 어떻게 변하는지 테스트했습니다. 살아있는 염색된 세포의 고정(2% 파라포름알데히드[PFA]])은 염료를 핵으로 약간 재분배시켰다(도4A, B). 고정된 세포의 투과율(0.1% 트리톤 X100)은 미토콘드리아에서 SG 점선 패턴을 제거하고, 핵의 염색이 지배적이었다(도4C). SG를 고정 및 투과 후 세포에 첨가한 경우, 세포질 및 핵에 균일하게분포하였다(도 4D). 따라서, SG 표지된 세포는 필요한 경우 고정될 수 있지만, 염료는 투과화가 필요한 프로토콜에 적합하지 않다.
살아있는 세포 SR-SIM 및 미토콘드리아 핵세포 추적
SG와 멀리 붉은 미토콘드리아 얼룩(표 의물질)으로코스타인 된 살아있는 세포는 초고해상도 SIM 기술에 의해 3D 를 이미지화하였다. 공초점 이미지(그림2)에서와같이, 미토콘드리아 핵형성은 미토콘드리아 내에서 밝은 반점으로 나타났다(도5A). 또한, 우리는 2D SIM 이미지의 타임 시리즈를 획득하고 회절 한계를 넘어 해상도로 핵소드의 위치를 추적. 타임 시리즈를 시작하기 직전에 우리는 SG와 미토콘드리아 얼룩 채널에서 SIM 3D 스택을 획득했습니다. 그런 다음 SG 채널에서만 타임시리즈를 획득하고 미토콘드리아 핵형성을 추적하여 움직임을 정량화했습니다. 트랙 평균 속도는 0.042 μm/s이고, 트랙의 최대 순간 속도는 0.078 ±0.012 μm/s였습니다. 핵종의 대다수는 그들의 본래 위치에서 멀리 치각하지 않았지만 미토콘드리아 심상에 있는 궤도의 오버레이로 아마 미토콘드리아 네트워크에 국한된 단거리 무작위 운동을 보여주었습니다(그림 5B). 일반적인 연수(Movie1)에서는빠른 방향 변위가 거의 발생하지 않았습니다.
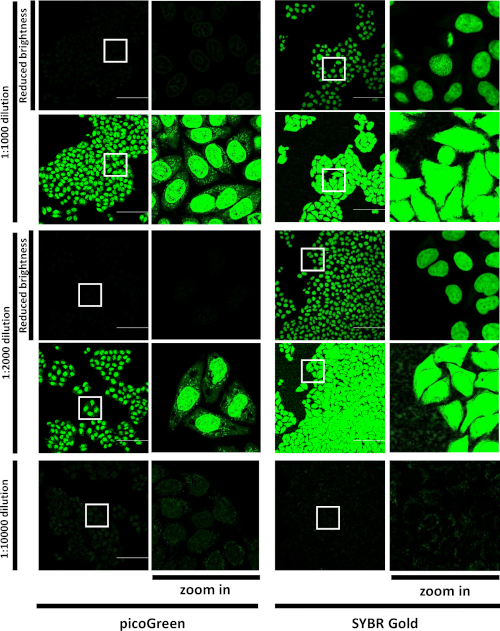
그림 1: 피코그린 및 SG 라이브 셀 라벨링의 비교. A549 세포를 피코그린 또는 SG로 배양하고 지시된 희석물에서 이미지화하였다. 녹색 채널에서 레이블이 지정된 셀의 대표 보기 필드입니다. LSM880 Airyscan FAST, 20x/공기 목표, 스케일 바 = 50 μm. 단일 광학 섹션. 흰색 사각형은 전체 423 x 423 μm 시야의 오른쪽에 더 높은 배율로 표시된 영역을 표시합니다. 1:1,000 및 1:2,000 희석의 경우 1:10,000 희석에 최적화된 "기본" 밝기 설정과 검출기 채도를 피하기 위해 조정된 "감소된 밝기" 설정의 두 가지 밝기 설정에서 동일한 이미지가 표시됩니다. 이 그림은 Jevtic 외27에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
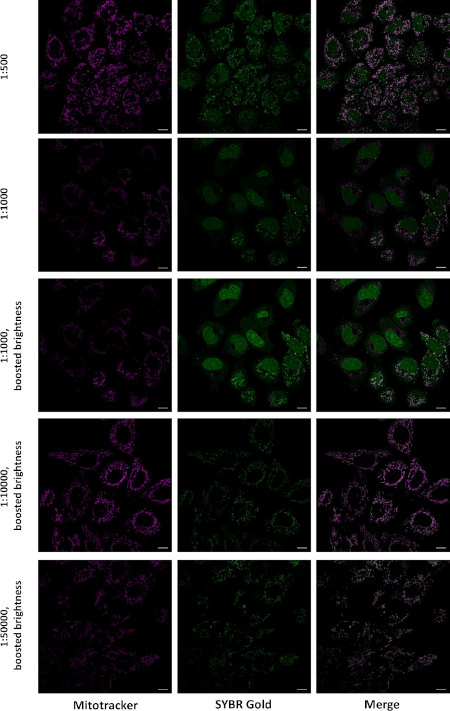
그림 2: 상이한 농도로 라벨링시 살아있는 세포에서 SG 국소화. HeLa 세포를 0.25 μM 미토트래커 CMXRos Red 및 지시된 SG 희석의 혼합물로 30분 동안 배양하였다. 이 용액은 DMEM으로 대체되었으며, 이미지는 LSM880 Airyscan 현미경, 63x 1.4 오일 목표에 획득되었으며 순차적으로 컬러 채널을 획득했습니다. 단일 광학 슬라이스는 스케일 바 = 10 μm)를 나타내었다. 이 그림은 Jevtic 외27에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: SYBR 골드(SG)로 라벨링하는 동안 헬라 세포. 먼저, 살아있는 헬라 세포를 미토트래커 CMXRos 레드로 표지하고 세척하였다. SG(DMEM에서 최종 희석 1:10,000)를 세포에 첨가하고 타임랩스 시리즈를 획득하였다. LSM880 현미경, 63x 1.4 오일 목표, 순차적 인수. Z-스택은 각 시점에서 획득되었습니다. 최대 강도 투영이 표시됩니다. (A)관심의 여러 지역에서 시간이 지남에 따라 SG 형광의 평균 강도. (B)SG 형광이 측정된 관심 영역을 나타내는 대표적인 시야(컬러 사각형). (C)SG를 가진 인큐베이션 도중 여러 시점의 시야. 흰색 선으로 표시된 사각형 영역은 오른쪽 열에 더 높은 배율로 표시됩니다. 이 그림은 Jevtic 외27에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 세포의 SG 국소화에 대한 고정 및 투과효과. HeLa 세포는 SG(재고 희석 1:10,000)와 미토트래커 CMXRos Red(0.25 μM)로 30분 동안 염색하였다. 순차적 인수; 스케일 바 = 10 μm.(A)SG로 염색 된 라이브 헬라 세포. (B)살아있는 헬라 세포를 SG로 염색한 다음 30분 동안 PBS에서 2% PFA로 고정한다음,SG로 염색한 다음, PBS에서 2% PFA로 고정한 다음 30분 동안 2%의 PFA로 고정하고 15분 동안 0.1% 트리톤 X100으로 투과하였다. 아래쪽 패널에서는 동일한 이미지가 표시되지만 녹색 채널의 밝기가 더 높게 설정됩니다. (D)2% PFA로 고정된 헬라 세포를 0.1% 트리톤 X100으로 15분 동안 투과한 다음 SG 및 미토트래커 CMXRos Red로 염색하였다. (D)에 도시된D이미지를 획득하기 위해, 과다 노출을 피하기 위해 A-C에 비해 녹색 채널용 카메라의 EMCCD 이득이 12배 감소되었다. 이 그림은 Jevtic 외27에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: 라이브 SIM 이미지에서 핵추적. SG 염색 된 세포의 시야의 대표적인 이미지. 하단: 타임랩스 시리즈를 수집하기 전에 촬영한 2색 SIM 이미지입니다. 녹색 = SG 채널; 마젠타 = 미토 트래커 채널. 위: SG 채널의 50프레임 SIM 타임시리즈에서 핵수선트랙(무비 1); 프레임 시간 = 1.8s; Imaris 8.4.1 소프트웨어에 의해 추적. 트랙은 최대 트랙 속도(μm/s)로 색상으로 구분됩니다. 엘리라 PS.1, SIM 모드, 100x/1.46 오일 목표; 배율 막대 = 10 μm. 이 그림은 Jevtic 외27에서수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

영화 1 : 대표적인 SG 염색 세포를 보여주는 SIM 타임 랩스 시리즈. 스케일 바 = 5 μm. 검출된 미토콘드리아 핵형성은 백색 구체로 표시된다. 트랙은 "드래곤 테일"(8 프레임 길이)으로 시각화되고 최대 인스턴트 속도에 따라 색상으로 구분됩니다(오른쪽 하단의 색상 막대는 μm/s의 속도를 보여줍니다). Imaris 8.4.1 소프트웨어에 의한 미토콘드리아 핵형성 추적 및 시각화. 이 비디오는 Jevtic 외27에게시되었습니다. 이 비디오를 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).
토론
프로토콜에는 몇 가지 중요한 구성 요소가 있습니다 : 미토콘드리아 DNA의 우선 라벨링을 얻으려면 배양 중에 DNA 결합 염료의 농도가 매우 낮게 유지되어야합니다 (예 : 일반적인 상용 재고의 1 :10,000 희석), 인큐베이션 시간은 30 분이어야합니다. 인큐베이션 시간은 1 h를 초과해서는 안됩니다. 다른 DNA 결합 염료는 낮은 농도에서 라벨링시 강한 신호를 생성할 만큼 충분히 밝지 않습니다.
우리의 프로토콜의 한계는 염료가 투과 단계 동안 미토콘드리아 핵형성으로부터 세척된다는 것입니다. 따라서, 기재된 절차는 통상적인 면역형광 프로토콜에 적합하지 않다. 이 경우 고정 된 세포의 핵형성은 DNA 또는 미토콘드리아 전사 인자 (TFAM)에 대한 항체와 같은 다른 기술로 효율적으로 표지 될 수 있습니다.
DNA 결합 유기 염료를 가진 미토콘드리아 핵세포의 직접 표지는 형광 성 단백질 표지에 비해 장점이 있습니다: 모든 유형의 세포는 형광 단백질 태그가 부착된 구성물의 일시적 또는 기타 일시적인 또는 안정적인 발현 제한 없이 <1 h 내에 표지될 수 있습니다. 또한, 현재 프로토콜에서, TFAM의 종래의 형광 단백질 태깅은 아티팩트를 유발하는 것으로 보고되었다. 더욱이, SG는 미토콘드리아 막 전위가 손상되지 않은 경우에만 핵을 효율적으로 염색한다. 이것은 FP 기지를 둔 염색으로 피할 수 없는 생물학적으로 무관한 "아픈" 및 죽은 세포의 심상 취득을 방지합니다. 마지막으로, DNA 결합 염료에 기초한 이전에 발표된 프로토콜은 미토콘드리아 DNA의 우대 염색을 달성하지 못했다.
제안된 프로토콜은 빠르고 간단하기 때문에 다양한 형광 기술에 의해 미토콘드리아 핵소자의 실시간 이미징에 널리 사용될 것이라고 가정하며, 회절 제한(예: 레이저 스캐닝 공초점, TIRF 등)과 초고해상도 SIM모두에 의해 널리 사용될 것입니다.
공개
저자는 공개 할 것이 없다.
감사의 말
저자는 아시파 아크타르와 안젤리카 람볼드 (면역 생물학 및 후성 유전학에 대한 막스 플랑크 연구소 모두) HeLa 세포를 제공하기위한 인정.
자료
| Name | Company | Catalog Number | Comments |
| Elyra PS1 | Carl Zeiss | multi-modal super-resolution microscope containing module for super-resolution structured illumination microscopy (SR-SIM) | |
| High glucose DMEM | GIBCO/ThermoFisher | 31966021 | |
| ibidi 35 mm dish, glass bottom | Ibidi Gmbh | 81158 | |
| ibidi 8-well microSlide, glass bottom | Ibidi Gmbh | 80827 | |
| Imaris 8.4.1 | Bitplane/Oxford Instruments | image porcessing and visualisation software package | |
| iXon 885 | Andor Technologies | EMCCD camera with back-illuminated sensor | |
| LSM880 Airyscan | Carl Zeiss | laser scanning confocal microscope with array detector | |
| Mitotracker CMXRos Red | ThermoFischer | M7512 | red live cell mitochondrial stain |
| Mitotracker Deep Red FM | ThermoFischer | M22426 | far red live cell mitochondrial stain |
| picoGreen | ThermoFischer | P7581 | cell permeant DNA stain |
| Plan Apochromat 100x/1.46 Oil objective | Carl Zeiss | ||
| SYBR Gold | ThermoFischer | S11494 | cell permeant DNA stain |
| Zen Black 2012 software | Carl Zeiss | image acquisition and processing software |
참고문헌
- Kaufman, B. A., et al. The mitochondrial transcription factor TFAM coordinates the assembly of multiple DNA molecules into nucleoid-like structures. Molecular Biology of the Cell. 18 (9), 3225-3236 (2007).
- Farge, G., et al. Protein sliding and DNA denaturation are essential for DNA organization by human mitochondrial transcription factor A. Nature Communications. 3, 1013(2012).
- Bogenhagen, D. F. Mitochondrial DNA nucleoid structure. Biochimica et Biophysica Acta. 1819 (9-10), 914-920 (2012).
- Kang, D., Kim, S. H., Hamasaki, N. Mitochondrial transcription factor A (TFAM): roles in maintenance of mtDNA and cellular functions. Mitochondrion. 7 (1-2), 39-44 (2007).
- Tauber, J., et al. Distribution of mitochondrial nucleoids upon mitochondrial network fragmentation and network reintegration in HEPG2 cells. International Journal of Biochemistry & Cell Biology. 45 (3), 593-603 (2013).
- Garrido, N., et al. Composition and dynamics of human mitochondrial nucleoids. Molecular Biology of the Cell. 14 (4), 1583-1596 (2003).
- Pernas, L., Scorrano, L. Mito-Morphosis: Mitochondrial Fusion, Fission, and Cristae Remodeling as Key Mediators of Cellular Function. Annual Review of Physiology. 78, 505-531 (2016).
- Caielli, S., et al. Oxidized mitochondrial nucleoids released by neutrophils drive type I interferon production in human lupus. Journal of Experimental Medicine. 213 (5), 697-713 (2016).
- Okayama, S., Ohta, K., Higashi, R., Nakamura, K. Correlative light and electron microscopic observation of mitochondrial DNA in mammalian cells by using focused-ion beam scanning electron microscopy. Microscopy (Oxf). 63 (Suppl 1), i35(2014).
- Alan, L., Spacek, T., Jezek, P. Delaunay algorithm and principal component analysis for 3D visualization of mitochondrial DNA nucleoids by Biplane FPALM/dSTORM. European Biophysics Journal. 45 (5), 443-461 (2016).
- Kukat, C., et al. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Kukat, C., Larsson, N. G. mtDNA makes a U-turn for the mitochondrial nucleoid. Trends in Cell Biology. 23 (9), 457-463 (2013).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Benke, A., Manley, S. Live-cell dSTORM of cellular DNA based on direct DNA labeling. ChemBioChem. 13 (2), 298-301 (2012).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79-87 (2016).
- Waldchen, S., Lehmann, J., Klein, T., van de Linde, S., Sauer, M. Light-induced cell damage in live-cell super-resolution microscopy. Scientific Reports. 5, 15348(2015).
- Gustafsson, M. G., et al. Three-dimensional resolution doubling in wide-field fluorescence microscopy by structured illumination. Biophysical Journal. 94 (12), 4957-4970 (2008).
- Hirano, Y., Matsuda, A., Hiraoka, Y. Recent advancements in structured-illumination microscopy toward live-cell imaging. Microscopy (Oxf). 64 (4), 237-249 (2015).
- Brown, T. A., et al. Superresolution fluorescence imaging of mitochondrial nucleoids reveals their spatial range, limits, and membrane interaction. Molecular and Cellular Biology. 31 (24), 4994-5010 (2011).
- Lewis, S. C., Uchiyama, L. F., Nunnari, J. ER-mitochondria contacts couple mtDNA synthesis with mitochondrial division in human cells. Science. 353 (6296), aaf5549(2016).
- Dellinger, M., Geze, M. Detection of mitochondrial DNA in living animal cells with fluorescence microscopy. Journal of Microscopy. 204 (Pt 3), 196-202 (2001).
- Ban-Ishihara, R., Ishihara, T., Sasaki, N., Mihara, K., Ishihara, N. Dynamics of nucleoid structure regulated by mitochondrial fission contributes to cristae reformation and release of cytochrome c. Proceedings of the National Academy of Sciences of the United States of America. 110 (29), 11863-11868 (2013).
- Zurek-Biesiada, D., et al. Localization microscopy of DNA in situ using Vybrant((R)) DyeCycle Violet fluorescent probe: A new approach to study nuclear nanostructure at single molecule resolution. Experimental Cell Research. 343 (2), 97-106 (2016).
- He, J. Y., et al. The AAA(+) protein ATAD3 has displacement loop binding properties and is involved in mitochondrial nucleoid organization. Journal of Cell Biology. 176 (2), 141-146 (2007).
- Ashley, N., Harris, D., Poulton, J. Detection of mitochondrial DNA depletion in living human cells using PicoGreen staining. Experimental Cell Research. 303 (2), 432-446 (2005).
- Jevtic, V., Kindle, P., Avilov, S. V. SYBR Gold dye enables preferential labeling of mitochondrial nucleoids and their time-lapse imaging by structured illumination microscopy. PLoS One. 13 (9), e0203956(2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유