Method Article
Identificação de fosfato de inositol ou proteínas Fosfoinositídeo interagindo por cromatografia de afinidade acoplada a Western blot ou espectrometria de massas
Neste Artigo
Resumo
Este protocolo centra-se na identificação de proteínas que se ligam a fosfatos de inositol ou fosfoinositídeos. Ele usa a cromatografia de afinidade com fosfatos de inositol biotinylated ou fosfoinositídeos que são imobilizados via estreptavidina para agarose ou grânulos magnéticos. Fosfato de inositol ou proteínas de ligação Fosfoinositídeo são identificados por western blotting ou espectrometria de massa.
Resumo
Fosfatos de inositol e fosfoinositídeos regulam vários processos celulares em eucariontes, incluindo expressão gênica, tráfico de vesículas, transdução de sinal, metabolismo e desenvolvimento. Estes metabolitos realizam esta atividade regulamentar ligando-se a proteínas, alterando assim a conformação proteica, a atividade catalítica e/ou as interações. O método descrito aqui utiliza cromatografia de afinidade acoplada à espectrometria de massas ou western blotting para identificar proteínas que interagem com fosfatos de inositol ou fosfoinositídeos. Fosfatos de inositol ou fosfoinositídeos são quimicamente marcados com biotina, que é então capturado via estreptavidina conjugados para agarose ou grânulos magnéticos. As proteínas são isoladas por sua afinidade de ligação ao metabolito, então eluída e identificada por espectrometria de massas ou western blotting. O método tem um fluxo de trabalho simples que é sensível, não-radioativo, Liposome-livre, e customizável, suportando a análise da interação da proteína e do metabolito com precisão. Esta abordagem pode ser usada em métodos de espectrometria de massas quantitativas rotuladas com rótulo livre ou em aminoácidos para identificar interações proteína-metabólito em amostras biológicas complexas ou usando proteínas purificadas. Este protocolo é otimizado para a análise de proteínas do Trypanosoma brucei, mas pode ser adaptado a parasitas relacionados com protozoários, leveduras ou células de mamíferos.
Introdução
Fosfatos de inositol (IPS) e fosfoinositídeos (PIS) desempenham um papel central na biologia eucariote através da regulação de processos celulares como o controle da expressão gênica1,2,3, tráfico de vesículas 4, transdução de sinal5,6, metabolismo7,8,9e desenvolvimento8,10. A função reguladora destes metabolitos resulta da sua capacidade de interagir com proteínas e, assim, regular a função proteica. Após a ligação por proteínas, IPs e PIs podem alterar a conformação de proteínas11, atividade catalítica12, ou interações13 e, portanto, afetam a função celular. IPS e PIS são distribuídos em múltiplos compartimentos subcelulares, como o núcleo2,3,14,15, retículo endoplasmático16,17, plasma membrana1 e citosol18, ambas associadas com proteínas3,19 ou com RNAs20.
A clivagem do PI associado à membrana (4, 5) P2 pela fosfolipase C resulta na liberação de ins (1, 4, 5) P3, que podem ser fosforilados ou deposforilados por IP quinases e fosfatases, respectivamente. IPs são moléculas solúveis que podem se vincular a proteínas e exercer funções regulatórias. Por exemplo, ins (1, 4, 5) P3 no metazoano podem atuar como um segundo mensageiro ligando-se aos receptores IP3, o que induz alterações conformacionais do receptor e, assim, liberação de CA2 + de lojas intracelulares11. Ins (1, 3, 4, 5) P4 liga-se ao complexo histona deacetilase e regula o conjunto complexo proteico e a atividade13. Outros exemplos de função regulatória de IPS incluem o controle da organização decromatina21, transporte de RNA22,23, edição de RNA24e transcrição1,2,3 . Em contrapartida, os pis estão frequentemente associados ao recrutamento de proteínas para a membrana plasmática ou membranas Organela25. No entanto, uma propriedade emergente do PIS é a capacidade de associar-se a proteínas em um ambiente não membranoso3,15,19,26. Este é o caso do fator esteroidogênicas do receptor nuclear, que a função de controle transcricional é regulada pelo PI (3, 4, 5) P319, e pela polimerase poli-A que a atividade enzimática é regulada pelo PI nuclear (4, 5) P226. Um papel regulatório para IPS e PIS foi demonstrado em muitos organismos, incluindolevedura 22,27, células demamíferos 19,23, Drosophila10 e worms28. De significância é o papel desses metabólitos em tripanossomas, que divergiram precocemente da linhagem eucariótica. Estes metabolitos desempenham um papel essencial no controlo transcripcional de Trypanosoma brucei 1,3, desenvolvimento8, biogênese organela e tráfego proteico29,30 , 31 de dezembro , 32, e também estão envolvidos no controle do desenvolvimento e infecção nos patógenos T. cruzi33,34,35, Toxoplasma36 e Plasmodium 5. º , 37. portanto, compreender o papel dos IPS e PIS em tripanossomas pode ajudar a elucidar a nova função biológica para essas moléculas e identificar novos alvos de drogas.
A especificidade da ligação proteína e IP ou PI depende dos domínios de interação protéica e do estado de fosforilação do inositol13,38, embora as interações com a parte lipídica do PIS também ocorra19. A variedade de IPs e PIs e suas cinases modificadoras e fosfatases fornece um mecanismo celular flexível para controlar a função protéica que é influenciada pela disponibilidade e abundância do metabolito, o estado de fosforilação do inositol e proteínas afinidade da interação1,3,13,38. Embora alguns domínios proteicos sejam bem caracterizados39,40,41, por exemplo, domínio de homologia de homologia42 e SPX (SYG1/Pho81/XPR1) domínios43 ,44,45, algumas proteínas interagem com IPS ou PIS por mecanismos que permanecem desconhecidos. Por exemplo, a proteína 1 (RAP1) do repressor-ativador de T. brucei carece de domínios de vinculação de PI canônicos, mas interage com pi (3, 4, 5) P3 e controle de transcrição de genes envolvidos na variação antigênica3. A cromatografia de afinidade e a análise de espectrometria de massas de proteínas IP ou PI que interagem de trypanosome, levedura ou células de mamíferos identificaram várias proteínas sem domínios de ligação IP ou PI conhecidos8,46, 47. os dados sugerem domínios proteicos não caracterizados adicionais que se ligam a estes metabolitos. Assim, a identificação de proteínas que interagem com IPs ou PIs pode revelar novos mecanismos de interação proteína-metabólito e novas funções reguladoras celulares para essas pequenas moléculas.
O método descrito aqui emprega a cromatografia de afinidade acoplada à mancha ocidental ou à espectrometria maciça para identificar as proteínas que ligam ao IPs ou ao PIs. Utiliza IPs biotinilados ou PIs que estão ligados a streptavidina conjugados a grânulos de agarose ou, alternativamente, capturados através de grânulos magnéticos conjugados com streptavidina (Figura 1). O método fornece um fluxo de trabalho simples que é sensível, não radioativo, Liposome-livre e é adequado para detectar a ligação de proteínas de lisados celulares ou proteínas purificadas3 (Figura 2). O método pode ser usado em Label-Free8,46 ou acoplado a espectrometria de massas quantitativa com rótulo de aminoácido47 para identificar proteínas de ligação IP ou PI de amostras biológicas complexas. Assim, este método é uma alternativa aos poucos métodos disponíveis para estudar a interação de IPs ou PIs com proteínas celulares e ajudará na compreensão da função regulatória desses metabólitos em tripanossomas e talvez outros eucariontes.
Protocolo
1. análise de proteínas de ligação IP ou PI por cromatografia de afinidade e western blotting
- Crescimento celular, Lise e cromatografia de afinidade
- Cresça as células T. brucei para a fase do mid-log e monitore a viabilidade e a densidade das células. Um total de 5,0 x 107 células é suficiente para um ensaio de ligação.
- Para as formas de corrente sanguínea, cultivar células em meios HMI-9 suplementados com soro bovino fetal a 10% (FBS) a 37 ° c e 5% CO2. Manter a densidade celular entre 8,0 x 105 a 1,6 x 106 células/ml.
Nota: a densidade superior a 1,8 x 106 células/ml pode afetar a viabilidade celular. O tempo de duplicação da cepa T. brucei 427 cultivada in vitro é entre 5,5 e 6,5 h. - Para formulários procíclica, cresça pilhas no meio SDM-79 suplementado com 10% FBS em 27 ° c e mantenha a densidade da pilha entre 1,0 x 107 e 3,0 x 107 Cells/ml.
- Para proteínas purificadas (por exemplo, proteínas recombinantes), tomar 0,5 a 1 μg de proteína e diluir em 450 μL de tampão de ligação (25 mM HEPES, 150 mM NaCl, 0,2% 4-Nonyl fenil-polietileno glicol, pH 7,4). Mantenha 5% da proteína diluída (entrada) para a análise de Western Blot. Prossiga para o passo 1.1.7.
- Para as formas de corrente sanguínea, cultivar células em meios HMI-9 suplementados com soro bovino fetal a 10% (FBS) a 37 ° c e 5% CO2. Manter a densidade celular entre 8,0 x 105 a 1,6 x 106 células/ml.
- Células centrífugas a 1.600 x g por 10 min à temperatura ambiente (RT). Descarte o sobrenadante.
Observação: consulte a etapa 2.1.2 para obter informações adicionais sobre a centrifugação de grandes volumes de cultura. - Ressuspender delicadamente o pellet em 10 mL do pH salino tamponado fosfato 7,4 suplementado com glicose de 6 milímetros (PBS-G) e pré-aquecido em 37 ° c para lavar as pilhas. Em seguida, Centrifugue as células a 1.600 x g durante 5 min em RT. Repita o procedimento duas vezes.
- Ressuscitem o pellet em 1 mL de PBS-G. Em seguida, transfira o volume para um tubo de 1,5 mL e centrifugue a 1.600 x g durante 5 min. descarte o sobrenadante.
Nota: as pelotas da pilha podem ser flash congeladas no nitrogênio líquido e ser armazenadas em-80 ° c ou nitrogênio líquido. - Ressuscitar o pellet em 0,5 mL de tampão de lise (25 mM HEPES, 150 mM NaCl, 1% t-octylphenoxypolyethoxyethanol, pH 7,4) suplementado com 1.5 x cocktail inibidor de protease e 1x inibidor da fosfatase cocktail (tabela de materiais) pré-refrigerados em gelo para Lise as células. Incubar lisado durante 10 min girando a 50 rpm a 4 ° c.
Nota: esta é uma etapa crítica porque as proteínas podem degradar se não segurado como indicado. Verificar a integridade da proteína lisado por sódio Dodecil sulfato-poliacrilamida gel electroforese (SDS/PAGE), se necessário.
Cuidado: T-octylphenoxypolyethoxyethanol é tóxico e pode causar irritação da pele e dos olhos. Use luvas, Eyeshield e proteção do acasalados. - Centrifugue o lisado a 14.000 x g durante 10 min a 4 ° c. Colete o sobrenadante em um novo tubo de 1,5 mL para ensaios de ligação. Mantenha 5% do lisado total (entrada) para a análise de mancha ocidental. O sobrenadante contém proteínas parasitas extraídas com tampão de Lise.
- Colete 50 μL de IPs ou PIs conjugados a grânulos de agarose (i.e., 50 μL de chorume) ou 50 μL de grânulos de agarose e centrifugue por 1 min a 1.000 x g. Descarte o sobrenadante e ressuscitem em 50 μL de tampão de ligação para equilibrar os grânulos. Use grânulos não conjugados como um controle. Use IP/PI-Beads com diferentes configurações de fosfato, incluindo formas não fosforiladas para controlar as interações inespecíficas.
- Adicionar 50 μL de IP-ou PI-Beads para o lisado de células ou proteínas purificadas (cada 1 mL de grânulos contém 10 nmol de IPs conjugados ou PIs). Mantenha o volume de IP-ou PI-Beads dentro de 10% do lisado total e, se necessário, ajuste o volume de reação de ligação com o tampão de ligação.
- Para ensaios de competição, adicione à reação de ligação várias concentrações de IPs não conjugados ou PIs (por exemplo, excesso de molar de 1, 10, 100 vezes comparado a IP-ou PI-Beads).
- Incubar a reacção durante 1 h, ou durante a noite, a 4 ° c e girando a 50 rpm.
Nota: as reações de ligação com proteínas purificadas podem ser feitas em RT, dependendo da estabilidade da proteína. Se usar IPs ou PIs conjugados à biotina, apenas prossiga para a etapa 1.1.9.1, caso contrário, prossiga para a etapa 1.1.10.- Adicionar 50 μL de streptavidina-conjugados a grânulos magnéticos à reacção de ligação e incubar durante 1 h a 4 ° c girando a 50 rpm.
- Centrifugue a mistura durante 1 min a 1.000 x g a 4 ° c. Retire o sobrenadante (fluxo-através) e manter o pellet. Mantenha 5% do sobrenadante para análise de Western Blot.
Nota: se usar grânulos magnéticos, remova sobrenadantes e realize lavagens subsequentes usando um suporte magnético (as centrifugações não são necessárias). - Adicionar 1 mL de tampão de lavagem (25 mM HEPES, 300 mM NaCl, 0,2% 4-Nonyl fenil-polietileno glicol, pH 7,4) e ressuscita a resina tocando ou rodando o tubo (não use uma pipeta porque os grânulos podem anexar a pontas de pipeta). Centrifugue a reacção durante 1 min a 1.000 x g a 4 ° c e elimine o sobrenadante. Repita o procedimento para um total de cinco laves.
Cuidado: o glicol de 4-Nonyl Phenyl-polietileno é tóxico e pode causar a irritação da pele e do olho. Use luvas, Eyeshield e proteção do acasalados. - Adicionar 50 μL de 2x tampão de Laemmli suplementado com 710 mm 2-Mercaptoetanol aos grânulos e misturar tocando ou Vortex para eluir as proteínas. Aqueça em 95 ° c por 5 minutos, a seguir Centrifugue para 10.000 x g por 1 minuto e colete o sobrenadante (contenha proteínas eluída). Alternativamente, proteínas eluir com 8 M de ureia/100 mm de glicina pH 2,9 para evitar o uso de SDS. Congelar o eluato em-80 ° c, caso contrário, proceder à análise Western Blot.
PRECAUÇÃO: 2-Mercaptoetanol é tóxico e pode causar irritações cutâneas, oculares e respiratórias. Use luvas e trabalhe na capa química.
- Cresça as células T. brucei para a fase do mid-log e monitore a viabilidade e a densidade das células. Um total de 5,0 x 107 células é suficiente para um ensaio de ligação.
- Western blotting análise
- Misture 15 μL de entrada (da etapa 1.1.6) ou fluxo-através de amostras (da etapa 1.1.10) com 5 μL do amortecedor de 4x Laemmli. A entrada de calor e fluxo-através de amostras para 5 min em 95 ° c. Para amostras eluidas em 8 M de ureia/100 mM de glicina pH 2,9, misture 15 μL de eluato com 5 μL de tampão de 4x Laemmli e aqueça por 5 min a 95 ° c.
Nota: esta etapa não é necessária para amostras eluída no amortecedor de 2x Laemmli. - Coloque os poços de 4-20% de gel SDS/PAGE com 2,5 μL de entrada, 2,5 μL de vazão e 20 μL de amostras eluidas e a escada de proteínas de carga de acordo com a recomendação do fabricante.
Nota: escolha o gel% de acordo com o peso molecular da proteína de interesse. - Execute SDS/PAGE em 150 V por 30-45 min no buffer de corrida, ou até que o corante azul do tampão Laemmli esteja no final do gel.
Nota: o tempo de funcionamento pode variar consoante o equipamento de laboratório. - Retire o gel das placas de vidro (ou plástico) e mergulhe no tampão de transferência durante 15 min.
- Transfira as proteínas para a membrana de difluoreto de polivinildeno (PVDF) ou membrana de nitrocelulose. Mergulhe as membranas e o papel de filtro de 3 mm no tampão de transferência. Montar um sanduíche com três folhas de papel de filtro, nitrocelulose ou membrana de PVDF, gel, e um adicional de três folhas de papel de filtro. Certifique-se de que nenhuma bolha de ar esteja presa no sanduíche. Use um rolo para remover bolhas, se necessário. Ajuste a membrana no cátodos e o gel no lado do ânodo da gaveta.
Nota: Verifique as instruções do fabricante da membrana para obter informações sobre a activação da membrana ou a preparação do borrão. - Coloque a gaveta que contém o sanduíche no tanque de transferência com tampão de transferência. Coloque o reservatório num balde de gelo ou a 4 ° c (por exemplo, na sala fria). Transferir proteínas a 100 V durante 1 h (a corrente varia entre 200-400 mA). Alternativamente, transfira durante a noite em uma corrente constante de 15 miliampères em 4 ° c.
- Retire a membrana da gaveta. Incubar a membrana em 6% de leite seco não gordo diluído em PBS com 0, 5% de polissorbato 20 (PBS-T), ou solução de bloqueio compatível, por 1 h em RT para bloquear a membrana.
Nota: antes de bloquear a membrana, a qualidade da transferência pode ser verificada utilizando a mancha de PONCEAU S. Incubar a membrana durante 1 min em 15 mL de PONCEAU S, enxague em água e visualize bandas. - Retire a solução de bloqueio e incubar a membrana por 1 h em RT com rotação de 50 rpm em anticorpos primários diluídos em 6% de leite seco não gordo diluído em PBS-T. Alternativamente, incubar a membrana durante a noite a 4 ° c com rotação de 50 rpm.
Nota: o tempo de incubação pode variar de acordo com a qualidade dos anticorpos; no entanto, a maioria dos anticorpos funcionará com incubações de 1-3 h em RT. Siga a recomendação do fabricante para a concentração ou diluição de anticorpos. - Lave a mancha incubando a membrana em PBS-T por 5 min com rotação de 50 rpm em RT. Repita o procedimento 3-5 vezes. Mais lava pode ser necessário dependendo da qualidade dos anticorpos.
- Incubar a membrana na peroxidase de rábano (HRP)-anticorpos secundários conjugados por 1 h em RT em 6% de leite seco não gordo diluído em PBS-T com rotação de 50 rpm.
Nota: siga a recomendação do fabricante para a concentração ou diluição de anticorpos. - Lave o Borrão como indicado no passo 1.2.9.
- Adicione o substrato quimioluminescente para cobrir a membrana. Retire o excesso de substrato e incubar por 5 min na RT no escuro.
Nota: consulte as instruções do fabricante para obter recomendações sobre os reagentes de quimioluminescência. - Capture o sinal quimioluminescente usando um Imager baseado em câmera. Alternativamente, use um filme de raios-X para capturar o sinal quimioluminescente.
- Misture 15 μL de entrada (da etapa 1.1.6) ou fluxo-através de amostras (da etapa 1.1.10) com 5 μL do amortecedor de 4x Laemmli. A entrada de calor e fluxo-através de amostras para 5 min em 95 ° c. Para amostras eluidas em 8 M de ureia/100 mM de glicina pH 2,9, misture 15 μL de eluato com 5 μL de tampão de 4x Laemmli e aqueça por 5 min a 95 ° c.
2. análise de proteínas IP/PI-Binding por cromatografia de afinidade e espectrometria de massas
- Crescimento celular, Lise e cromatografia de afinidade
- Cresça as células T. brucei para a fase do mid-log e monitore a viabilidade e a densidade das células.
Nota: um total de 1,0 x 1010 células é suficiente para dois ensaios de ligação. O uso de menos células do que o indicado aqui pode afetar a detecção de proteínas de baixa abundância por espectrometria de massas.- Para os formulários da corrente sanguínea de T. brucei , cresça pilhas na fase do mid-log (8,0 x 105-1,6 x 106 Cells/ml) em meios HMI-9 suplementados com 10% FBS em 37 ° c e com 5% co2. Para 427 estirpe, 5 L de cultura renderá 0,5-1,0 x 1010 células. Monitore o crescimento celular para evitar a densidade superior a 1,8 x 106 células/ml que podem afetar a viabilidade celular. Manter o volume de cultura celular para 1/10 do volume do balão; caso contrário, a taxa de crescimento das células será afetada devido à má aeração.
Nota: a estirpe 427 tem um tempo de duplicação de 5,5-6,5 h. - Para formulários procíclica, cresça pilhas no meio SDM-79 suplementado com 10% FBS em 27 ° c e mantenha a densidade da pilha entre 1,0 x 107 e 3,0 x 107 Cells/ml. 500 mL de cultura renderá 0,5-1,5 x 1010 células.
- Para os formulários da corrente sanguínea de T. brucei , cresça pilhas na fase do mid-log (8,0 x 105-1,6 x 106 Cells/ml) em meios HMI-9 suplementados com 10% FBS em 37 ° c e com 5% co2. Para 427 estirpe, 5 L de cultura renderá 0,5-1,0 x 1010 células. Monitore o crescimento celular para evitar a densidade superior a 1,8 x 106 células/ml que podem afetar a viabilidade celular. Manter o volume de cultura celular para 1/10 do volume do balão; caso contrário, a taxa de crescimento das células será afetada devido à má aeração.
- Centrifugue as células a 1.600 x g durante 15 min em RT. descarte o sobrenadante e resuma a pelota em 200 ml de PBS-g pré-aquecido a 37 ° c. Use tubos de centrifugação de fundo redondo (tabela de materiais) porque as pelotas do formulário da corrente sanguínea de T. brucei são perturbadas facilmente ao usar rotores do centrifugador do fixo-ângulo. Centrifugue as células novamente a 1.600 x g por 5 min em RT.
- Retire o sobrenadante e ressuscitem o pellet em 10 mL de PBS-G. Centrifugue as células a 1.600 x g durante 5 min em RT. Repita o procedimento e após a lavagem final, descarte o sobrenadante.
Nota: as pelotas podem ser flash congeladas em nitrogênio líquido e armazenadas em-80 ° c ou nitrogênio líquido. - Ressuscitem o pellet celular em 5 mL de tampão de Lise pré-resfriado no gelo e suplementado com coquetel de inibidor de protease de 1,5 x e coquetel de inibidor de fosfatase 1x (tabela de materiais). Incubar o lisado por 10 min a 4 ° c girando a 50 rpm.
- Centrifugue o lisado a 10.000 x g durante 10 min a 4 ° c. Recolher o sobrenadante (proteínas solubilizadas) e diluir-lo em 20 mL de tampão de ligação.
- Colete 400 μL de IPs ou PIs conjugados a grânulos de agarose (i.e., 400 μL de chorume) ou 400 μL de grânulos de controle, e centrifugue por 1 min a 1.000 x g em RT. descarte o sobrenadante e ressuscitem em 400 μL de tampão de ligação para equilibrar os grânulos. Use grânulos de agarose como um controle negativo para determinar o enriquecimento específico de interações proteína-metabolito em comparação com interações inespecíficas (por exemplo, proteínas que ligam não especificamente para os grânulos). Use IPs ou PIs com diferentes configurações de fosfato, incluindo formas não fosforiladas para controle de interações inespecíficas devido a cargas de fosfato ou ligação à biotina.
- Adicionar 400 μL de IP/PI-Beads ou grânulos de controle a 10 mL de lisado e incubados por 1 h, ou durante a noite, a 4 ° c girando a 50 rpm. Se usar IPs ou PIs conjugados com biotina somente (sem grânulos) prossiga para a etapa 2.1.7.1, caso contrário, prossiga para a etapa 2.1.8.
- Adicionar 100 μL de streptavidina-conjugados a grânulos magnéticos e incubar durante 1 h a 4 ° c girando a 50 rpm.
- Centrifugue a reacção de ligação durante 1 min a 1.000 x g a 4 ° c. Retire o sobrenadante (fluxo-através) e manter o pellet. Mantenha 5% do sobrenadante para análise de Western Blot.
- Adicione 5 mL de tampão de lavagem à pelota, misture delicadamente girando o tubo, e centrifugue então por 1 minuto em 1.000 x g em 4 ° c. Descarte o sobrenadante. Repita a lavagem cinco vezes. Se estiver usando grânulos magnéticos, colete sobrenadantes e realize lavagens usando um suporte magnético (as centrifugações não são necessárias).
- Adicionar 50 μL de 2x tampão de Laemmli (ou 8 M de ureia/100 mM de pH glicina 2,9, para evitar o uso de SDS) para os grânulos e misturar tocando ou Vortex (Evite pipetagem porque os grânulos podem anexar a pontas de pipeta) e, em seguida, aqueça a 95 ° c por 5 min. centrífuga para 10.000 x g por 1 min e recolher o sobrenadante (proteínas eluted). Repita o procedimento duas vezes para coletar um total de três frações.
- Congelar o eluato em-80 ° c, de outra forma separar as proteínas em SDS/PAGE ou manter em solução para a análise de tripsinização e espectrometria de massas.
- Cresça as células T. brucei para a fase do mid-log e monitore a viabilidade e a densidade das células.
- Tripsina digestão de proteínas para espectrometria de massas
Nota: duas variações deste procedimento são mostradas para a seção 2.2.1 (em gel) ou seção 2.2.2 (em solução) digestão de proteínas. Os tubos de ligação de baixa proteína são recomendados para evitar perdas de amostras. Consulte um químico analítico na instalação de proteômica a adequação do protocolo para amostras e instrumentos de espectrômetro de massas disponíveis.- Tripsinização in-gel das proteínas
- Após a separação da proteína em SDS/PAGE, enxague brevemente o gel na água da elevado-pureza. Transfira o gel para uma placa de vidro limpa. As bandas de proteína de consumo com uma lâmina limpa e evitar o corte extra gel fora bandas. Cortar as peças de gel em pedaços pequenos (ou seja, aproximadamente 1 mm quadrados) e transferi-los para um tubo de 1 mL. Use uma ponta de pipeta, se necessário, mas certifique-se de que a ponta da pipeta é enxaguada em etanol antes de usar.
Nota: use luvas para evitar a contaminação do gel. As peças de gel podem ser armazenadas a-20 ° c. - Adicione 100 μL de água de grau de cromatografia líquida de alta pureza ou alto desempenho a tubos para enxaguar peças de gel. Descarte a água.
- Para peças de gel com coloração fluorescente à base de Coomassie ou rutênio, incubar as peças de gel na solução de destaining (25 mM NH4HCO3 em 50% acetonitrila) por 1 h e, em seguida, descarte a solução. Repita o procedimento até que a coloração não esteja visível.
Nota: Prepare soluções contendo NH4HCO3 por diluição de uma solução de estoque em 100 mm NH4HCO3, pH 7,8.
Cuidado: o acetonitrila é um solvente volátil, inflamável e tóxico. NH4HCO3 pode causar irritação cutânea ou ocular. Use luvas e trabalhe um capuz químico. - Para peças de gel manchada de prata, incubar peças em gel em 50 μL de solução de destaining para mancha de prata (15 mM K3[Fe (CN)6], 50 mm na2S2o3) em água por 30 min. descarte a solução e lave peças de gel com 200 μl de água. Repita a lavagem cinco vezes ou até que a cor amarela do gel não esteja visível.
PRECAUÇÃO: K3[Fe (CN)6] pode provocar irritação cutânea ou ocular. Use luvas.
- Para peças de gel com coloração fluorescente à base de Coomassie ou rutênio, incubar as peças de gel na solução de destaining (25 mM NH4HCO3 em 50% acetonitrila) por 1 h e, em seguida, descarte a solução. Repita o procedimento até que a coloração não esteja visível.
- Desidratar as peças de gel utilizando 200 μL de acetonitrila durante 10 min em RT. Em seguida, descarte a solução.
Nota: as peças de gel desidratadas são menores em volume, opaco e pegajoso. Se vários pedaços de gel são combinados em um tubo, repita o procedimento para a desidratação eficiente de peças de gel. - Adicionar 50 μL (ou volume suficiente para cobrir as peças de gel) de solução de redução (10 mm ditiotreitol [DTT] em 100 mm NH4HCO3) e incubar a 56 ° c por 1 h. depois, resfriar os tubos para RT e descartar o excesso de solução de redução.
- Adicionar 50 μL (ou volume suficiente para cobrir as peças de gel) de solução de alquilação (50 mM iodoacetamida em 100 mM NH4HCO3) e incubar por 30 min na RT no escuro. Depois, descarte o excesso de solução de alquilação.
- Desidratar as peças de gel com 200 μL de acetonitrila durante 10 min em RT. Retire o acetonitrila e hidratar as peças de gel com 100 mM NH4HCO3 por 10 min em RT.
- Deshidratar as peças de gel novamente com 200 μL de acetonitrila durante 10 min em RT e descartar o excesso de solução.
- Adicionar 15 μL de tripsina de classe de espectrometria de massas diluída em 50 mM NH4HCO3 tampão, ou volume suficiente para cobrir peças de gel hidratado e incubar por 4 h, ou durante a noite, a 37 ° c. Mantenha quantidades totais de tripsina entre 100 a 500 ng (ou 20 ng tripsina/μg de proteína).
- Resfrie a amostra para RT, e centrifugue por 1 min a 2.000 x g em uma microcentrífuga. Adicionar 10-20 μL de ácido fórmico a 5% diluído em água e incubar durante 10 min na RT.
Cuidado: o ácido fórmico é inflamável, corrosivo e tóxico. Use luvas e trabalhe um capuz químico. - Centrifugador como indicado na etapa 2.2.1.9, a seguir colete o sobrenadante (contenha peptídeos extraídos) a um tubo diferente. Adicionar 20 μL de ácido fórmico a 5% diluído em 50-60% acetonitrila ao tubo e incubar por 10 min em RT. coletar frações peptídicas extraídas no mesmo tubo.
- Seque a amostra em um concentrador de vácuo e reconstitua em 10 μL de ácido acético a 0,5% e 2% de acetonitrila para análise de espectrometria de massas. Centrifugue e colete a solução na parte inferior do tubo; solução de armazenamento a-20 ° c ou-80 ° c.
- Após a separação da proteína em SDS/PAGE, enxague brevemente o gel na água da elevado-pureza. Transfira o gel para uma placa de vidro limpa. As bandas de proteína de consumo com uma lâmina limpa e evitar o corte extra gel fora bandas. Cortar as peças de gel em pedaços pequenos (ou seja, aproximadamente 1 mm quadrados) e transferi-los para um tubo de 1 mL. Use uma ponta de pipeta, se necessário, mas certifique-se de que a ponta da pipeta é enxaguada em etanol antes de usar.
- Em solução de tripsinização de proteínas
- Precipitate proteínas para reduzir o volume da amostra, dessalinagem, e troca de buffer. Adicione seis volumes de acetona refrigerada (-20 ° c) à amostra, por exemplo, 600 μL de acetona a 100 μL de amostra. Vortex e incubar a-20 ° c durante 15 min a 1 h. A solução vai ficar turva ou formar um precipitado.
Cuidado: a acetona é tóxica e inflamável. Use luvas e trabalhe um capuz químico. - Centrifugar amostras a 4 ° c durante 30 min. decantar a acetona e secar o pellet durante 15 min.
- Adicionar 10 μL de 6-8 M de ureia ou 1% de SDS em 50 mM NH4HCO3 para dissolver o pellet e o vórtice para misturar. Para quantidades maiores de proteínas (> 10 μg), utilize até 20 μL de tampão de dissolução.
- Adicionar 5 μL de solução de redução e Vortex. Gire o volume para baixo usando uma microcentrífuga. Se as amostras forem diluídas em ureia, então incubar por 1 h na RT. Se as amostras forem diluídas em SDS, então incubadas por 1 h a 56 ° c.
- Gire o volume para baixo usando uma microcentrífuga. Adicionar 3 μL de solução de alquilação e Vortex. Em seguida, gire o volume para baixo e incubar no escuro por 30 min em RT.
- Adicionar 3 μL de solução redutora para neutralizar a reacção. Diluir lentamente a amostra para 1 M de ureia ou 0, 5% SDS usando 50 mM NH4HCO3.
Nota: o tampão de digestão de tripsina deve ter detergente ou desnaturante. Os limites de concentração para desnaturantes são: 0, 5% SDS; 0,1% octil B-D-glucopirranosídeo; 0,1% 4-nonylphenyl-polietileno glicol; 0,1% t-octylphenoxypolyethoxyethanol; 0,1% polissorbato 20; 0,1% 3-[(3-cholamidopropyl) dimethylammonio]-1-propanesulfonato; < 1 M de ureia ou tiourea. - Adicionar 5 μL de tripsina de classe de espectrometria de massas diluída em 50 mM NH4HCO3 tampão e incubar por 4 h, ou durante a noite, a 37 ° c. Mantenha quantidades totais de tripsina entre 100 e 500 ng (ou 20 ng tripsina/μg de proteína).
- Esfrie a amostra para RT, e gire o volume para baixo usando uma microcentrífuga. Adicionar 5% de ácido acético (ou 5% de ácido fórmico em 50% acetonitrila) para saciar a reação.
- Seque as amostras em um concentrador do vácuo como indicado na etapa 2.2.1.11. Armazene amostras em-80 ° c. Peptídeos de desalt e concentrado usando uma coluna de fase invertida, como o zip-Tip C18 e, em seguida, analisar por espectrometria de massa.
- Precipitate proteínas para reduzir o volume da amostra, dessalinagem, e troca de buffer. Adicione seis volumes de acetona refrigerada (-20 ° c) à amostra, por exemplo, 600 μL de acetona a 100 μL de amostra. Vortex e incubar a-20 ° c durante 15 min a 1 h. A solução vai ficar turva ou formar um precipitado.
- Tripsinização in-gel das proteínas
Resultados
Análise da interação RAP1 e PI (3, 4, 5) P3 por cromatografia de afinidade e western blotting
Este exemplo ilustra a aplicação deste método para analisar a ligação de PIS por RAP1 de t. brucei lisado ou por t. brucei recombinante proteína RAP1. Lisados de T. brucei formas de corrente sanguínea que expressam hemaglutinina (ha)-Tagged RAP1 foram utilizados em ensaios de ligação. RAP1 é uma proteína envolvida no controle transcricional de genes de glicoproteína de superfície variante (VSG)3,48, que codificam para proteínas de superfície envolvidas na evasão imune parasitária por variação antigênica49. RAP1 interage dentro de um complexo proteico telomérico com a enzima fosfatidilinositol 5-fosfatase (PIP5Pase)3, que também funciona no controle da transcrição do gene VSG1,3. RAP1 tem um N-terminal de câncer de mama 1 carboxilo-terminal (brct) domínio que é seguido por um Mieloblastose (MyB) DNA-ligação de domínio e um C-terminal MyB-como o domínio3,48. No entanto, ele carece de domínios de vinculação de PI canônicos. Os ensaios de ligação foram realizados com PIs não fosforilados ou fosforilados em diferentes posições do anel de inositol e com grânulos de agarose não conjugados. A análise ocidental mostra que RAP1 se liga preferencialmente a PI (3, 4, 5) P3-Beads (Figura 3a), mas também se liga a um menor grau de PI (4, 5) P2-Beads. No entanto, não se vinculou a quaisquer outros PIs ou grânulos de agarose. Como o RAP1 faz parte de um complexo multiprotéico3, sua interação com alguns PIS pode não ser direta e, portanto, resulta da interação RAP1-ha com outras proteínas celulares que se ligam ao PIS.
Assim, para testar se RAP1 vincula diretamente ao PIs, uma C-terminally marcou 6x-sua proteína RAP1 recombinante (rRAP1) foi expressa e purificada para homogeneidade de e. coli3. A proteína foi utilizada em ensaios de ligação com PI (3, 4, 5) P3-grânulos na presença de concentrações concorrentes de PI (3, 4, 5) P3 ou PI (4, 5) P2. Western blotting mostra que o aumento das concentrações de PI (3, 4, 5) P3, mas não PI (4, 5) P2 inibe a interação de rRAP1 com PI (3, 4, 5) P3 (Figura 3B). Além disso, a adição de T. brucei purificou a enzima PIP5Pase à reação restaurada PI (3, 4, 5) P3-ligação por rRAP1, que é devido a PIP5Pase desfosforilação de livre PI (3, 4, 5) P33 e, portanto, indica que o padrão de fosforilação deste metabolito é essencial para a ligação rRAP1. Portanto, rRAP1 interage com PI (3, 4, 5) P3 como faz RAP1-HA de T. brucei lysates. Além disso, os dados mostram que a ligação de RAP1-HA de lisado a PI (4, 5) P2 provavelmente resulta da interação RAP1 com outras proteínas no complexo (por exemplo, PIP5Pase)3. Os dados ilustram a complementaridade de ensaios de ligação com lisados celulares e proteínas recombinantes. Também mostra a utilidade de ensaios de vinculação competitiva para determinar a especificidade das interações entre proteínas e PIs.
Identificação de proteínas de ligação P3 de ins (1, 4, 5) por cromatografia de afinidade e espectrometria de massas
Neste exemplo, utilizou-se a cromatografia de afinidade seguida de espectrometria de massas para identificar as proteínas T. brucei que se ligam a ins (1, 4, 5) P3; Portanto, o experimento examina as proteínas de ligação P3 potenciais ins (1, 4, 5) de formas de corrente sanguínea de T. brucei . T. brucei lisado foi incubada com ins (1, 4, 5) P3 conjugados a grânulos de agarose ou com grânulos não conjugados (usados como controle), e proteínas ligadas foram eluidas com tampão de amostra de Laemmli. A análise de SDS/PAGE mostra o enriquecimento em proteínas eluidas de ins (1, 4, 5) P3-grânulos comparados às proteínas eluidas dos grânulos de agarose controle (Figura 4a). A análise de espectrometria de massas de proteínas eluidas identificou mais de 250 proteínas, das quais 84 foram enriquecidas com grânulos P3 de ins (1, 4, 5) comparados aos grânulos de controle (Figura 4B, mudança de dobra [FC] ≥ 2, p < 0, 5). O enriquecimento de proteínas ligadas a ins (1, 4, 5) P3 em comparação com os grânulos de controle correlaciona-se com o sinal de proteína detectado por SDS/PAGE. Os dados incluem proteínas que foram validadas para vincular ins (1, 4, 5) P3 e proteínas que mecanismos de ligação a ins (1, 4, 5) P3 são desconhecidos8. Além disso, os ins (1, 4, 5) o proteoma de ligação P3 diferiu muito do de ins (1, 3, 4, 5) P4 e outros PIS8, o que sugere que algumas dessas proteínas reconhecem a configuração específica de fosfato de ins (1, 4, 5) P3. Assim, os ins marcados com biotina (1, 4, 5) P3 podem ser utilizados para cromatografia de afinidade acoplada à espectrometria de massas para identificar proteínas que se ligam a ins (1, 4, 5) P3. A abordagem pode ser explorada para identificar proteínas que se ligam a outros IPs ou PIS3,8,46,47.

Figura 1 : Reagentes de afinidade para ensaios de ligação. (A) PI (3, 4, 5) P3 (parte superior) e ins (1, 4, 5) P3 (parte inferior) conjugados à biotina na posição SN1 do inositol. Em PI (3, 4, 5) P3, a biotina é conjugada à cadeia lipídica na posição SN1 do inositol, enquanto em ins (1, 4, 5) P3 a biotina é conjugada ao fosfato na posição SN1. (B) ins (1, 4, 5) P3 é conjugado com biotina e capturado através de ligação à streptavidina conjugada a grânulos (por exemplo, agarose ou grânulos magnéticos). Variações desses reagentes usando linkers sintetizados personalizados que substituem a Biotina também são possíveis46. Por favor clique aqui para ver uma versão maior desta figura.
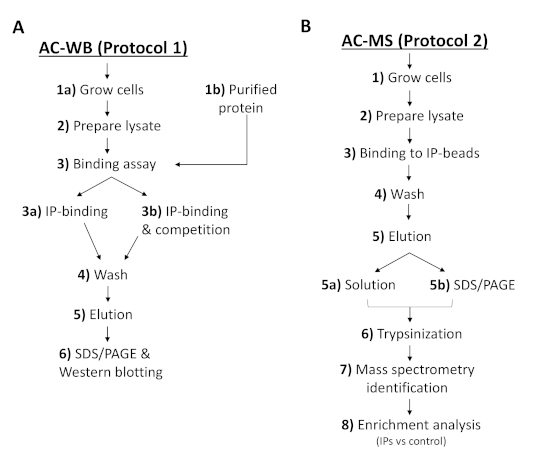
Figura 2 : O fluxo de trabalho dos protocolos descreve as etapas para a análise da interação de afinidade IP ou PI com proteínas de T. brucei e detecção por (a) western blotting ou (B) espectrometria de massas. AC-WB, cromatografia de afinidade e western blotting; AC-MS, cromatografia de afinidade e espectrometria de massas. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3 : Ligação de T. brucei RAP1 a fosfoinositídeos. (A) lisados de T. brucei (5,0 x 107 parasitas) que expressam ha-Tagged RAP1 foram incubados por 2 h a 4 ° c com 50 ΜL de PIS (cada 1 ml de grânulos de agarose contendo 10 nmol de PIS conjugados) ou grânulos de agarose (AG). A reação de ligação foi lavada e eluída com 2 x tampão da amostra de Laemmli e aquecida a 95 ° c por 5 min. as proteínas foram separadas em 4-20% SDS/Page, transferidas para uma membrana de PVDF, e sondado com anticorpos monoclonais anti-ha (1:5000, diluído em 6% PBS-leite) seguido por anti-mouse IgG-HRP (1:5000, diluído em 6% de PBS-leite), e detectado por quimioluminescência. (B) um μg de rRAP1 foi incubado por 1 h em RT com 50 ΜL de PI (3, 4, 5) P3-grânulos de agarose na presença ou ausência de 5 a 50 μm de dioctanoylglicerol (DIC8) PI (3, 4, 5) P3, 20 a 50 ΜM diC8 PI (4, 5) P2, ou 50 ΜM diC8 PI (3, 4, 5) p3 e 250 ng PIP5Pase purificados dos formulários da corrente sanguínea de T. brucei 3. A associação foi analisada por western blotting com anticorpos monoclonais anti-his HRP (1:2000, diluído em 6% de PBS-Milk) e desenvolvido por quimioluminescência. Este número foi modificado de Cestari et al.3. Por favor clique aqui para ver uma versão maior desta figura.

Figura 4 : Cromatografia de afinidade e análise de espectrometria de massas de proteínas T. brucei que se ligam a ins (1, 4, 5) P3. (A) análise de 10% SDS/PAGE de proteínas T. brucei que se ligam a ins (1, 4, 5) P3-grânulos ou grânulos de agarose. Os lisados de 5,0 x 109 parasitos foram incubados a 4 ° c por 2 h com 400 ΜL de ins (1, 4, 5) P3 conjugados a grânulos de agarose ou com grânulos de agarose sem ins (1, 4, 5) P3 [1 ml de grânulos contêm 10 nmol de ins conjugados (1, 4, 5) P3]. A reação de ligação foi lavada, eluída em 2 x tampão de amostra de Laemmli e fervida por 5 min a 95 ° c. As proteínas foram separadas em 10% SDS/PAGE e coradas com coloração de Coomassie (tabela de materiais). Os ARROWHEADS mostram as proteínas que são enriquecidas em ins (1, 4, 5) P3-grânulos comparados aos grânulos do agarose; os círculos indicam proteínas presentes em ambos os ins (1, 4, 5) P3-grânulos e grânulos do agarose, e o suporte indica as proteínas que estão atuais em ambos mas são enriquecidas em ins (1, 4, 5) P3-grânulos comparados aos grânulos do agarose. (B) dot-Plot mostra proteínas identificadas por espectrometria de massas que são enriquecidas em ins (1, 4, 5) P3-grânulos em comparação com grânulos de agarose. Enriquecimento definido pelo FC > 2 e valor de p< 0,05. Quatro repetições biológicas foram utilizadas para os agarose-Beads AC-MS, e três repetições biológicas para IP3-Beads AC-MS. a troca de proteínas identificadas em IP3-Beads versus agarose-Beads foi calculada utilizando-se a intensidade dos espectros peptídicos utilizando o MSstat50. Resultados detalhados e lista de peptídeos estão disponíveis8. Os dados brutos de espectrometria de massa também estão disponíveis com o identificador PXD005907 através do consórcio ProteomeXchange através do repositório de parceiros PRIDE. Este número foi modificado de Cestari et al.8. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
A identificação de proteínas que se ligam a IPs ou PIs é fundamental para compreender a função celular desses metabólitos. A cromatografia de afinidade acoplada ao borrão ocidental ou à espectrometria maciça oferece uma oportunidade de identificar IP ou PI que interagem proteínas e daqui obtêm introspecções em sua função regulamentar. IPs ou PIs quimicamente marcados [por exemplo, ins (1, 4, 5) P3 quimicamente ligados à biotina] e reticulados a grânulos de agarose via estreptavidina ou capturados por grânulos magnéticos estreptavidina permite o isolamento de proteínas interagindo que podem então ser identificadas por massa espectrometria ou Western Blot. Os protocolos aqui descritos foram utilizados para identificar proteínas de T. brucei3,8 e células de mamíferos47 que se ligam a estes metabolitos. As variações da aproximação que usa etiquetas personalizadas (à excepção da biotina) foram usadas igualmente no fermento46. Uma consideração importante a esta aproximação é o uso dos controles para discriminar específico das interações não específicas. Os grânulos não conjugados são um controle essencial, mas os controles adicionais podem incluir PIs não fosforilados ou inositol conjugados a grânulos3. IPS ou PIS com diferentes combinações de fosfato3,8 podem também ser utilizados porque a ligação de proteínas a estes metabolitos pode envolver domínios que discriminam a configuração de fosfato do inositol38, 39,40,41. Adicionalmente, a complexidade da amostra pode afetar a sensibilidade da abordagem e, consequentemente, diminuir a complexidade da amostra pelo fracionamento amostral pode ajudar na detecção de proteínas de baixa abundância na célula. Existem protocolos bem estabelecidos para o fracionamento celular e isolamento de mitocenon51, núcleo52, glicosoma53e flagelo54,55 de trypanosomes. Observe que os buffers e reagentes usados em fraccionamento subcelular podem precisar ser ajustados para compatibilidade com buffers e reagentes usados neste protocolo. Notavelmente, a identificação de proteínas que se ligam a IPs ou PIs por cromatografia de afinidade, conforme indicado aqui, depende da afinidade de proteínas e metabólito da interação e, portanto, as proteínas que têm uma fraca afinidade por IPs ou PIs podem não ser prontamente detectadas.
A análise da interação IP ou PI com proteínas do lisado celular também pode resultar na identificação de proteínas que não se ligam diretamente a esses metabólitos, mas que a interação resulta da Associação protéica em complexo com outras proteínas que se ligam a IPs ou PIs. Este recurso é exemplificado na Figura 3a, em que RAP1-ha de T. brucei lisado parece vincular a PI (3, 4, 5) P3-Beads e PI (4, 5) P2-Beads. No entanto, os ensaios vinculativos com o rRAP1 mostram que esta proteína se liga a PI (3, 4, 5) P3 e não a PI (4, 5) P23. Isto é ilustrado na Figura 3B, em que os ensaios competitivos mostram que PI livre (3, 4, 5) P3 mas não livre PI (4, 5) P2 compete para rRAP1-sua interação com pi (3, 4, 5) P3-Beads. A interação RAP1 aparente com PI (4, 5) P2-Beads é devida à associação RAP1 dentro de um complexo com proteínas que ligam PI (4, 5) P2 (por exemplo, PIP5Pase)3. Assim, os ensaios de ligação de lisados celulares podem identificar proteínas que se ligam direta ou indiretamente a IPs ou PIs. Notavelmente, as interações indiretas são distintas das interações inespecíficas, uma vez que a primeira pode ter uma função biológica (no contexto do complexo proteico) que afeta ou é afetada pela ligação do metabolito. Por exemplo, ins (1, 4, 5, 6) P4 vincula-se ao complexo de desacetilase de corepressor multisubunidade e controla o conjunto complexo e a atividade13. Portanto, é essencial validar interações IP/PI com proteínas. A validação da interação pode envolver ensaios de competição com um excesso de IPS ou pis (como na Figura 3B)3,8, mutações de potenciais domínios proteicos56, ou o uso de proteínas purificadas para determinar interações diretas (Figura 3A, B)3.
Outros métodos para o estudo de proteínas e interações IP ou PI incluem a ligação de proteínas a IPs ou PIs radiomarcado, o uso de IPs ou PIs vinculados a membranas hidrofóbicas como matrizes para captura de proteínas, ou a ligação de proteínas a PIs incorporados em lipossomas 42 , 57 , 58. importante, se a interação protéica com PIS necessitar de estruturas de membrana38, os ensaios baseados em lipoalguns podem ser usados como uma abordagem complementar. Limitações dessas abordagens incluem baixa taxa de transferência, baixa sensibilidade, a orientação química desconhecida da Associação IPs ou PIs para matrizes58, ou o uso de materiais radioativos42. O método descrito aqui é sensível, Liposome-livre, não-radioativo, e IP/PI-contas estão disponíveis comercialmente, e, portanto, eles não necessitam de síntese química personalizada. Além disso, a posição da biotina ligada a IPS ou PIS é bem definida e também pode ser modificada46,58, o que permite uma análise precisa da interação proteína e metabólito. O método descrito aqui também pode ser combinado com abordagens quantitativas de espectrometria de massas, como a rotulagem isotópica estável de aminoácidos na cultura celular (SILAC)47, que pode ser usada para identificar interações dinâmicas diferentes tratamentos ou condições. A cromatografia de afinidade acoplada ao borrão ocidental ou à espectrometria maciça ajudou a identificar proteínas IP-ou PI-obrigatórias numerosas de T. brucei, de pilhas de mamífero, e de fermento3,8,46, 47, incluindo proteínas que não caracterizaram domínios de ligação IP ou PI, e também ajudaram na identificação de novos domínios vinculativos, por exemplo, PI (3, 4, 5) P3 Binding Domain47.
No geral, os protocolos descritos aqui podem ser usados para examinar potenciais proteínas interagindo IP ou PI de T. brucei, e para estudar a interação molecular de proteínas com esses metabólitos. O protocolo pode ser facilmente adaptado para identificar proteínas de ligação IP ou PI de outros parasitas unicelulares ou de outros organismos, tais como células de mamíferos47 e levedura46, e ajudará a compreender ainda mais a função biológica de IPS e PIs em eucariontes.
Divulgações
O autor não tem nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelo Conselho de pesquisa de ciências naturais e engenharia do Canadá (NSERC, RGPIN-2019-04658); Suplemento de lançamento de descoberta NSERC para pesquisadores de carreira precoce (DGECR-2019-00081) e pela Universidade McGill.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetone | Sigma-Aldrich | 650501 | Ketone |
| Acetonitrile | Sigma-Aldrich | 271004 | Solvent |
| Ammonium bicarbonate | Sigma-Aldrich | A6141 | Inorganic salt |
| Centrifuge Avanti J6-MI | Beckman Coulter | Avanti J6-MI | Centrifuge for large volumes (e.g., 1L) |
| Centrifuge botles | Sigma-Aldrich | B1408 | Bottles for centrifugation of 1L of culture |
| Control Beads | Echelon | P-B000-1ml | Affinity chromatography reagent - control |
| D-(+)-Glucose | Sigma-Aldrich | G8270 | Sugar, Added in PBS to keep cells viable |
| Dithiothreitol (DTT) | Bio-Rad | 1610610 | Reducing agent |
| Dynabeads M-270 Streptavidin | ThermoFisher Scientific | 65305 | Streptavidin beads for binding to biotin ligands |
| EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitors |
| Electrophoresis running buffer | Bio-Rad | 1610732 | 25 mM Tris, 192 mM glycine, 0.1% SDS, pH 8.3 |
| Falcon 15 mL Conical Centrifuge Tubes | Corning Life Sciences | 430052 | To centrifuge 10 mL cultures |
| Formic acid | Sigma-Aldrich | 106526 | Acid |
| Glycine | Sigma-Aldrich | G7126 | Amino acid |
| HMI-9 cell culture medium | ThermoFisher Scientific | ME110145P1 | Cell culture medium for T. brucei bloodstream forms |
| Imperial Protein Stain | ThermoFisher Scientific | 24615 | Coomassie staining for protein detection in SDS/PAGE |
| Ins(1,4,5)P3 Beads | Echelon | Q-B0145-1ml | Affinity chromatography reagent |
| Instant Nonfat Dry Milk | Thomas Scientific | C837M64 | Blocking reagent for Western blotting |
| Iodoacetamide | Sigma-Aldrich | I6125 | Alkylating reagent for cysteine proteins or peptides |
| Lab Rotator | Thomas Scientific | 1159Z92 | For binding assays |
| LoBind Microcentrifuge Tubes | ThermoFisher Scientific | 13-698-793 | Low protein binding tubes for mass spectrometry |
| Nonidet P-40 (Igepal CA-630) | Sigma-Aldrich | 21-3277 | Detergent |
| PBS, pH 7.4 | ThermoFisher Scientific | 10010031 | Physiological buffer |
| Peroxidase substrate for chemiluminescence | ThermoFisher Scientific | 32106 | Substrate for Western bloting detection of proteins |
| PhosSTOP Phosphatase Inhibitor Cocktail Tablets | Roche | 4906845001 | Phosphatase inhibitors |
| PI(3)P PIP Beads | Echelon | P-B003a-1ml | Affinity chromatography reagent |
| PI(3,4)P2 PIP Beads | Echelon | P-B034a-1ml | Affinity chromatography reagent |
| PI(3,4,5)P3 diC8 | Echelon | P-3908-1mg | Affinity chromatography reagent |
| PI(3,4,5)P3 PIP Beads | Echelon | P-B345a-1ml | Affinity chromatography reagent |
| PI(3,5)P2 PIP Beads | Echelon | P-B035a-1ml | Affinity chromatography reagent |
| PI(4)P PIP Beads | Echelon | P-B004a-1ml | Affinity chromatography reagent |
| PI(4,5)P2 diC8 | Echelon | P-4508-1mg | Affinity chromatography reagent |
| PI(4,5)P2 PIP Beads | Echelon | P-B045a-1ml | Affinity chromatography reagent |
| PI(5)P PIP Beads | Echelon | P-B005a-1ml | Affinity chromatography reagent |
| Ponceau S solution | Sigma-Aldrich | P7170 | Protein staining (0.1% [w/v] in 5% acetic acid) |
| Potassium hexacyanoferrate(III) | Sigma-Aldrich | 702587 | Potassium salt |
| PtdIns PIP Beads | Echelon | P-B001-1ml | Affinity chromatography reagent |
| PVDF Membrane | Bio-Rad | 1620177 | For Western blotting |
| Refrigerated centrifuge | Eppendorf | 5910 R | Microcentrifuge for small volumes (e.g., 1.5 mL) |
| Sodium dodecyl sulfate | Sigma-Aldrich | 862010 | Detergent |
| Sodium thiosulfate | Sigma-Aldrich | 72049 | Chemical |
| SpeedVac Vacuum Concentrators | ThermoFisher Scientific | SPD120-115 | Sample concentration (e.g., for mass spectrometry) |
| T175 flasks for cell culture | ThermoFisher Scientific | 159910 | To grow 50 mL T. brucei culture |
| Trypsin, Mass Spectrometry Grade | Promega | V5280 | Trypsin for protein digestion |
| Urea | Sigma-Aldrich | U5128 | Denaturing reagent |
| Vortex | Fisher Scientific | 02-215-418 | For mixing reactions |
| Western blotting transfer buffer | Bio-Rad | 1610734 | 25 mM Tris, 192 mM glycine, pH 8.3 with 20% methanol |
| Whatman 3 mm paper | Sigma-Aldrich | WHA3030861 | Paper for Wester transfer |
| 2-mercaptoethanol (14.2 M) | Bio-Rad | 1610710 | Reducing agent |
| 2x Laemmli Sample Buffer | Bio-Rad | 161-0737 | Protein loading buffer |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels | Bio-Rad | 4561094 | Gel for protein electrophoresis |
| 4x Laemmli Sample Buffer | Bio-Rad | 161-0747 | Protein loading buffer |
Referências
- Cestari, I., Stuart, K. Inositol phosphate pathway controls transcription of telomeric expression sites in trypanosomes. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2803-2812 (2015).
- Odom, A. R., Stahlberg, A., Wente, S. R., York, J. D. A role for nuclear inositol 1,4,5-trisphosphate kinase in transcriptional control. Science. 287 (5460), 2026-2029 (2000).
- Cestari, I., McLeland-Wieser, H., Stuart, K. Nuclear Phosphatidylinositol 5-Phosphatase Is Essential for Allelic Exclusion of Variant Surface Glycoprotein Genes in Trypanosomes. Molecular and Cellular Biology. 39 (3), (2019).
- Billcliff, P. G., et al. OCRL1 engages with the F-BAR protein pacsin 2 to promote biogenesis of membrane-trafficking intermediates. Molecular Biology of the Cell. 27 (1), 90-107 (2016).
- Brochet, M., et al. Phosphoinositide metabolism links cGMP-dependent protein kinase G to essential Ca(2)(+) signals at key decision points in the life cycle of malaria parasites. PLoS Biology. 12 (3), 1001806(2014).
- Azevedo, C., Szijgyarto, Z., Saiardi, A. The signaling role of inositol hexakisphosphate kinases (IP6Ks). Advances in Enzyme Regulation. 51 (1), 74-82 (2011).
- Szijgyarto, Z., Garedew, A., Azevedo, C., Saiardi, A. Influence of inositol pyrophosphates on cellular energy dynamics. Science. 334 (6057), 802-805 (2011).
- Cestari, I., Anupama, A., Stuart, K. Inositol polyphosphate multikinase regulation of Trypanosoma brucei life stage development. Molecular Biology of the Cell. 29 (9), 1137-1152 (2018).
- Bang, S., et al. AMP-activated protein kinase is physiologically regulated by inositol polyphosphate multikinase. Proceedings of the National Academy of Sciences of the United States of America. 109 (2), 616-620 (2012).
- Seeds, A. M., Tsui, M. M., Sunu, C., Spana, E. P., York, J. D. Inositol phosphate kinase 2 is required for imaginal disc development in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 112 (51), 15660-15665 (2015).
- Hamada, K., Miyatake, H., Terauchi, A., Mikoshiba, K. IP3-mediated gating mechanism of the IP3 receptor revealed by mutagenesis and X-ray crystallography. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), 4661-4666 (2017).
- Watson, P. J., et al. Insights into the activation mechanism of class I HDAC complexes by inositol phosphates. Nature Communications. 7, 11262(2016).
- Watson, P. J., Fairall, L., Santos, G. M., Schwabe, J. W. Structure of HDAC3 bound to co-repressor and inositol tetraphosphate. Nature. 481 (7381), 335-340 (2012).
- Irvine, R. F. Nuclear lipid signalling. Nature Reviews: Molecular Cell Biology. 4 (5), 349-360 (2003).
- Sobol, M., et al. UBF complexes with phosphatidylinositol 4,5-bisphosphate in nucleolar organizer regions regardless of ongoing RNA polymerase I activity. Nucleus. 4 (6), 478-486 (2013).
- Nascimbeni, A. C., et al. ER-plasma membrane contact sites contribute to autophagosome biogenesis by regulation of local PI3P synthesis. EMBO Journal. 36 (14), 2018-2033 (2017).
- Martin, K. L., Smith, T. K. The myo-inositol-1-phosphate synthase gene is essential in Trypanosoma brucei. Biochemical Society Transactions. 33, Pt 5 983-985 (2005).
- Matsu-ura, T., et al. Cytosolic inositol 1,4,5-trisphosphate dynamics during intracellular calcium oscillations in living cells. Journal of Cell Biology. 173 (5), 755-765 (2006).
- Blind, R. D., et al. The signaling phospholipid PIP3 creates a new interaction surface on the nuclear receptor SF-1. Proceedings of the National Academy of Sciences of the United States of America. 111 (42), 15054-15059 (2014).
- Lin, A., et al. The LINK-A lncRNA interacts with PtdIns(3,4,5)P3 to hyperactivate AKT and confer resistance to AKT inhibitors. Nature Cell Biology. 19 (3), 238-251 (2017).
- Steger, D. J., Haswell, E. S., Miller, A. L., Wente, S. R., O'Shea, E. K. Regulation of chromatin remodeling by inositol polyphosphates. Science. 299 (5603), 114-116 (2003).
- York, J. D., Odom, A. R., Murphy, R., Ives, E. B., Wente, S. R. A phospholipase C-dependent inositol polyphosphate kinase pathway required for efficient messenger RNA export. Science. 285 (5424), 96-100 (1999).
- Adams, R. L., Mason, A. C., Glass, L., Aditi,, Wente, S. R. Nup42 and IP6 coordinate Gle1 stimulation of Dbp5/DDX19B for mRNA export in yeast and human cells. Traffic. 18 (12), 776-790 (2017).
- Macbeth, M. R., et al. Inositol hexakisphosphate is bound in the ADAR2 core and required for RNA editing. Science. 309 (5740), 1534-1539 (2005).
- Dickson, E. J., Hille, B. Understanding phosphoinositides: rare, dynamic, and essential membrane phospholipids. Biochemical Journal. 476 (1), 1-23 (2019).
- Mellman, D. L., et al. A PtdIns4,5P2-regulated nuclear poly(A) polymerase controls expression of select mRNAs. Nature. 451 (7181), 1013-1017 (2008).
- Lee, Y. S., Mulugu, S., York, J. D., O'Shea, E. K. Regulation of a cyclin-CDK-CDK inhibitor complex by inositol pyrophosphates. Science. 316 (5821), 109-112 (2007).
- Nagy, A. I., et al. IP3 signalling regulates exogenous RNAi in Caenorhabditis elegans. EMBO Reports. 16 (3), 341-350 (2015).
- Hall, B. S., et al. TbVps34, the trypanosome orthologue of Vps34, is required for Golgi complex segregation. Journal of Biological Chemistry. 281 (37), 27600-27612 (2006).
- Rodgers, M. J., Albanesi, J. P., Phillips, M. A. Phosphatidylinositol 4-kinase III-beta is required for Golgi maintenance and cytokinesis in Trypanosoma brucei. Eukaryotic Cell. 6 (7), 1108-1118 (2007).
- Gilden, J. K., et al. The role of the PI(3,5)P2 kinase TbFab1 in endo/lysosomal trafficking in Trypanosoma brucei. Molecular and Biochemical Parasitology. 214, 52-61 (2017).
- Demmel, L., et al. The endocytic activity of the flagellar pocket in Trypanosoma brucei is regulated by an adjacent phosphatidylinositol phosphate kinase. Journal of Cell Science. 129 (11), 2285(2016).
- Gimenez, A. M., et al. Phosphatidylinositol kinase activities in Trypanosoma cruzi epimastigotes. Molecular and Biochemical Parasitology. 203 (1-2), 14-24 (2015).
- Hashimoto, M., et al. Inositol 1,4,5-trisphosphate receptor regulates replication, differentiation, infectivity and virulence of the parasitic protist Trypanosoma cruzi. Molecular Microbiology. 87 (6), 1133-1150 (2013).
- Cestari, I., Haas, P., Moretti, N. S., Schenkman, S., Stuart, K. Chemogenetic Characterization of Inositol Phosphate Metabolic Pathway Reveals Druggable Enzymes for Targeting Kinetoplastid Parasites. Cell Chemical Biology. 23 (5), 608-617 (2016).
- Lovett, J. L., Marchesini, N., Moreno, S. N., Sibley, L. D. Toxoplasma gondii microneme secretion involves intracellular Ca(2+) release from inositol 1,4,5-triphosphate (IP(3))/ryanodine-sensitive stores. Journal of Biological Chemistry. 277 (29), 25870-25876 (2002).
- McNamara, C. W., et al. Targeting Plasmodium PI(4)K to eliminate malaria. Nature. 504 (7479), 248-253 (2013).
- Tresaugues, L., et al. Structural basis for phosphoinositide substrate recognition, catalysis, and membrane interactions in human inositol polyphosphate 5-phosphatases. Structure. 22 (5), 744-755 (2014).
- Varnai, P., et al. Quantifying lipid changes in various membrane compartments using lipid binding protein domains. Cell Calcium. 64, 72-82 (2017).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Letters. 590 (15), 2454-2468 (2016).
- Cullen, P. J., Cozier, G. E., Banting, G., Mellor, H. Modular phosphoinositide-binding domains--their role in signalling and membrane trafficking. Current Biology. 11 (21), 882-893 (2001).
- Klarlund, J. K., et al. Signaling by phosphoinositide-3,4,5-trisphosphate through proteins containing pleckstrin and Sec7 homology domains. Science. 275 (5308), 1927-1930 (1997).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Gerasimaite, R., et al. Inositol Pyrophosphate Specificity of the SPX-Dependent Polyphosphate Polymerase VTC. ACS Chemical Biology. 12 (3), 648-653 (2017).
- Potapenko, E., et al. 5-Diphosphoinositol pentakisphosphate (5-IP7) regulates phosphate release from acidocalcisomes and yeast vacuoles. Journal of Biological Chemistry. 293 (49), 19101-19112 (2018).
- Wu, M., Chong, L. S., Perlman, D. H., Resnick, A. C., Fiedler, D. Inositol polyphosphates intersect with signaling and metabolic networks via two distinct mechanisms. Proceedings of the National Academy of Sciences of the United States of America. 113 (44), 6757-6765 (2016).
- Jungmichel, S., et al. Specificity and commonality of the phosphoinositide-binding proteome analyzed by quantitative mass spectrometry. Cell Reports. 6 (3), 578-591 (2014).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137 (1), 99-109 (2009).
- Cestari, I., Stuart, K. Transcriptional Regulation of Telomeric Expression Sites and Antigenic Variation in Trypanosomes. Current Genomics. 19 (2), 119-132 (2018).
- Clough, T., Thaminy, S., Ragg, S., Aebersold, R., Vitek, O. Statistical protein quantification and significance analysis in label-free LC-MS experiments with complex designs. BMC Bioinformatics. 13, Suppl 16 6(2012).
- Schneider, A., Charriere, F., Pusnik, M., Horn, E. K. Isolation of mitochondria from procyclic Trypanosoma brucei. Methods in Molecular Biology. 372, 67-80 (2007).
- DeGrasse, J. A., Chait, B. T., Field, M. C., Rout, M. P. High-yield isolation and subcellular proteomic characterization of nuclear and subnuclear structures from trypanosomes. Methods in Molecular Biology. 463, 77-92 (2008).
- Opperdoes, F. R. A rapid method for the isolation of intact glycosomes from Trypanosoma brucei by Percoll -gradient centrifugation in a vertical rotor. Molecular and Biochemical Parasitology. 3 (3), 181-186 (1981).
- Pereira, N. M., de Souza, W., Machado, R. D., de Castro, F. T. Isolation and properties of flagella of trypanosomatids. The Journal of protozoology. 24 (4), 511-514 (1977).
- Subota, I., et al. Proteomic analysis of intact flagella of procyclic Trypanosoma brucei cells identifies novel flagellar proteins with unique sub-localization and dynamics. Molecular and Cellular Proteomics. 13 (7), 1769-1786 (2014).
- Fukuda, M., Kojima, T., Kabayama, H., Mikoshiba, K. Mutation of the pleckstrin homology domain of Bruton's tyrosine kinase in immunodeficiency impaired inositol 1,3,4,5-tetrakisphosphate binding capacity. Journal of Biological Chemistry. 271 (48), 30303-30306 (1996).
- Knodler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. BioTechniques. 38 (6), (2005).
- Best, M. D. Global approaches for the elucidation of phosphoinositide-binding proteins. Chemistry and Physics of Lipids. 182, 19-28 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados