Method Article
Identifizierung von Inositolphosphat oder Phosphoinositid-Wechselwirkungsproteinen durch Affinitätschromatographie gekoppelt an Western Blot oder Massenspektrometrie
In diesem Artikel
Zusammenfassung
Dieses Protokoll konzentriert sich auf die Identifizierung von Proteinen, die an Inositolphosphate oder Phosphoinositide binden. Es verwendet Affinitätschromatographie mit biotinylierten Inositolphosphaten oder Phosphoinositiden, die über Streptavidin zu Agarose oder magnetischen Perlen immobilisiert werden. Inositolphosphat oder Phosphoinositid-bindende Proteine werden durch western blotting oder Massenspektrometrie identifiziert.
Zusammenfassung
Inositolphosphate und Phosphoinositide regulieren mehrere zelluläre Prozesse in Eukaryoten, einschließlich Genexpression, Vesikelhandel, Signaltransduktion, Stoffwechsel und Entwicklung. Diese Metaboliten führen diese regulatorische Aktivität durch Bindung an Proteine durch und verändern dadurch die Proteinkonformation, die katalytische Aktivität und/oder die Wechselwirkungen. Die hier beschriebene Methode verwendet Affinitätschromatographie gekoppelt mit Massenspektrometrie oder Western Blotting, um Proteine zu identifizieren, die mit Inositolphosphaten oder Phosphoinositiden interagieren. Inositolphosphate oder Phosphoinositide werden chemisch mit Biotin markiert, das dann über Streptavidin konjugiert zu Agarose oder magnetischen Perlen gefangen wird. Proteine werden durch ihre Affinität zur Bindung an den Metaboliten isoliert, dann eluiert und durch Massenspektrometrie oder Western Blotting identifiziert. Die Methode verfügt über einen einfachen Workflow, der empfindlich, nicht radioaktiv, liposomenfrei und anpassbar ist und die Analyse der Protein- und Metaboliteninteraktion präzise unterstützt. Dieser Ansatz kann in etikettenfreien oder in aminosäuremarkierten quantitativen Massenspektrometriemethoden verwendet werden, um Protein-Metabolit-Wechselwirkungen in komplexen biologischen Proben oder mit gereinigten Proteinen zu identifizieren. Dieses Protokoll ist für die Analyse von Proteinen aus Trypanosoma bruceioptimiert, kann aber an verwandte protozoische Parasiten, Hefe- oder Säugetierzellen angepasst werden.
Einleitung
Inositolphosphate (IPs) und Phosphoinositide (PIs) spielen eine zentrale Rolle in der Eukaryotenbiologie durch die Regulierung zellulärer Prozesse wie die Kontrolle der Genexpression1,2,3, Vesikelhandel 4, Signaltransduktion5,6, Stoffwechsel7,8,9, und Entwicklung8,10. Die regulatorische Funktion dieser Metaboliten ergibt sich aus ihrer Fähigkeit, mit Proteinen zu interagieren und damit die Proteinfunktion zu regulieren. Bei Bindung durch Proteine können IPs und PIs die Proteinkonformation11, die katalytische Aktivität12oder Wechselwirkungen13 verändern und somit die zelluläre Funktion beeinflussen. IPs und PIs sind in mehreren subzellulären Kompartimenten verteilt, wie z. B. Kern2,3,14,15, endoplasmatisches Retikulum16,17, Plasma Membran1 und Cytosol18, entweder verbunden mit Proteinen3,19 oder mit RNAs20.
Die Spaltung des membranassoziierten PI(4,5)P2 durch Phospholipase C führt zur Freisetzung von Ins(1,4,5)P3, das durch IP-Kinäsen bzw. Phosphatasen phosphoryliert oder dephosphoryliert werden kann. IPs sind lösliche Moleküle, die an Proteine binden und regulatorische Funktionen ausüben können. Zum Beispiel kann Ins(1,4,5)P3 in Metazoan als zweiter Botenstoff durch Bindung an IP3-Rezeptoren fungieren, was Rezeptor-Konformationsänderungen induziert und somit Ca2+ aus intrazellulären Speichern freigibt11. Ins(1,3,4,5)P4 bindet an den Histon-Deacetylase-Komplex und reguliert die Proteinkomplex-Montage und -Aktivität13. Weitere Beispiele für iPs Regulatorische Funktion sind die Kontrolle der Chromatin-Organisation21, RNA-Transport22,23, RNA-Editing24und Transkription1,2,3 . Im Gegensatz dazu werden PIs oft mit der Rekrutierung von Proteinen an die Plasmamembran oder Organelle Membranen25assoziiert. Eine neu entstehende Eigenschaft von PIs ist jedoch die Fähigkeit, mit Proteinen in einer nicht-membranösen Umgebungzuassoziieren 3,15,19,26. Dies ist der Fall des steroidogenen Kernrezeptors, dessen Transkriptionskontrollfunktion durch PI(3,4,5)P319reguliert wird, und Poly-A-Polymerase, deren enzymatische Aktivität durch kerntechnische PI(4,5)P226reguliert wird. Eine regulierende Rolle für IPs und PIs wurde in vielen Organismen gezeigt, einschließlich Hefe22,27, Säugetierzellen19,23, Drosophila10 und Würmer28. Von Bedeutung ist die Rolle dieser Metaboliten bei Trypanosomen, die früh von der eukaryotischen Abstammung abwichen. Diese Metaboliten spielen eine wesentliche Rolle bei Trypanosoma brucei Transkriptionskontrolle1,3, Entwicklung8, Organelle Biogenese und Proteinverkehr29,30 , 31 , 32, und sind auch an der Kontrolle der Entwicklung und Infektion in den Erregern T. cruzi33,34,35,Toxoplasma36 und Plasmodium beteiligt 5 , 37. Daher kann das Verständnis der Rolle von IPs und PIs bei Trypanosomen dazu beitragen, neue biologische Funktionen für diese Moleküle aufzuklären und neue Wirkstoffziele zu identifizieren.
Die Spezifität von Protein- und IP- oder PI-Bindung hängt von Protein-Interagierenden Domänen und dem Phosphorylierungszustand des Inositols13,38ab, obwohl Wechselwirkungen mit dem Lipidteil von PIs ebenfalls 19 auftreten. Die Vielfalt der IPs und PIs und ihre modifizierenden Kinoseen und Phosphatasen bieten einen flexiblen zellulären Mechanismus zur Kontrolle der Proteinfunktion, der durch die Verfügbarkeit und Häufigkeit von Metaboliten, den Phosphorylierungszustand des Inositols und das Protein beeinflusst wird. Affinität der Interaktion1,3,13,38. Obwohl einige Proteindomänen gut charakterisiert sind39,40,41, z.B. Pleckstrin-Homologie-Domäne42 und SPX (SYG1/Pho81/XPR1) Domains43 ,44,45, einige Proteine interagieren mit IPs oder PIs durch Mechanismen, die unbekannt bleiben. Zum Beispiel fehlt dem Repressor-Aktivator-Protein 1 (RAP1) von T. brucei kanonische PI-bindende Domänen, sondern interagiert mit PI(3,4,5)P3 und kontrolliert die Transkription von Genen, die an antigener Variation beteiligt sind3. Affinitätschromatographie und Massenspektrometrieanalyse von IP- oder PI-interagierenden Proteinen aus Trypanosom-, Hefe- oder Säugetierzellen identifizierten mehrere Proteine ohne bekannte IP- oder PI-bindende Domänen8,46, 47. Die Daten deuten auf zusätzliche nicht charakterisierte Proteindomänen hin, die an diese Metaboliten binden. Daher kann die Identifizierung von Proteinen, die mit IPs oder PIs interagieren, neue Mechanismen der Protein-Metabolit-Interaktion und neue zelluläre Regulierungsfunktionen für diese kleinen Moleküle offenbaren.
Die hier beschriebene Methode verwendet Affinitätschromatographie gekoppelt mit western blotting oder Massenspektrometrie, um Proteine zu identifizieren, die an IPs oder PIs binden. Es verwendet biotinylierte IPs oder PIs, die entweder mit Streptavidin verbunden sind, konjugiert mit Agaroseperlen oder alternativ über streptavidin-konjugierte Magnetperlen gefangen werden (Abbildung 1). Die Methode bietet einen einfachen Workflow, der empfindlich, nicht radioaktiv, liposomenfrei ist und sich zum Nachweis der Bindung von Proteinen aus Zelllysaten oder gereinigten Proteinen eignet3 (Abbildung 2). Das Verfahren kann in etikettenfreien8,46 oder gekoppelt an Aminosäure-markierte quantitative Massenspektrometrie47 verwendet werden, um IP- oder PI-bindende Proteine aus komplexen biologischen Proben zu identifizieren. Daher ist diese Methode eine Alternative zu den wenigen verfügbaren Methoden, um die Wechselwirkung von IPs oder PIs mit zellulären Proteinen zu untersuchen und wird dazu beitragen, die regulatorische Funktion dieser Metaboliten bei Trypanosomen und vielleicht anderen Eukaryoten zu verstehen.
Protokoll
1. Analyse von IP- oder PI-bindenden Proteinen durch Affinitätschromatographie und Western Blotting
- Zellwachstum, Lyse und Affinitätschromatographie
- Wachsen Sie T. brucei-Zellen bis zur Mid-Log-Phase und überwachen Sie die Zelllebensfähigkeit und -dichte. Insgesamt reichen 5,0 x 107 Zellen für einen Bindungstest aus.
- Bei Blutkreislaufformen wachsen Zellen in HMI-9-Medien, ergänzt mit 10% fetalem Rinderserum (FBS) bei 37 °C und 5%CO2. Halten Sie die Zelldichte zwischen 8,0 x 105 bis 1,6 x 106 Zellen/ml.
HINWEIS: Eine Dichte von mehr als 1,8 x 106 Zellen/ml kann die Zelllebensfähigkeit beeinträchtigen. Die Verdoppelungszeit des in vitro angebauten T. brucei 427-Stamms liegt zwischen 5,5 und 6,5 h. - Bei prozyklischen Formen wachsen Zellen in SDM-79 Medium, ergänzt mit 10% FBS bei 27 °C, und halten die Zelldichte zwischen 1,0 x 107 und 3,0 x 107 Zellen/ml.
- Bei gereinigten Proteinen (z. B. rekombinanten Proteinen) 0,5 bis 1 g Protein einnehmen und in 450 l Bindungspuffer (25 mM HEPES, 150 mM NaCl, 0,2% 4-Nonylphenyl-Polyethylenglycol, pH 7,4) verdünnen. Halten Sie 5% des verdünnten Proteins (Input) für die Western Blot Analyse. Fahren Sie mit Schritt 1.1.7 fort.
- Bei Blutkreislaufformen wachsen Zellen in HMI-9-Medien, ergänzt mit 10% fetalem Rinderserum (FBS) bei 37 °C und 5%CO2. Halten Sie die Zelldichte zwischen 8,0 x 105 bis 1,6 x 106 Zellen/ml.
- Zentrifugenzellen bei 1.600 x g für 10 min bei Raumtemperatur (RT). Entsorgen Sie den Überstand.
HINWEIS: Weitere Informationen zur Zentrifugierung großer Kulturvolumina finden Sie in Schritt 2.1.2. - Das Pellet in 10 ml Phosphat gepufferter saline pH 7,4, ergänzt mit 6 mM Glukose (PBS-G) und vorgewärmt bei 37 °C, um die Zellen zu waschen, wird vorsichtig wieder aufgeheizt. Zentrifugieren Sie dann die Zellen bei 1.600 x g für 5 min bei RT. Wiederholen Sie den Vorgang zweimal.
- Das Pellet in 1 ml PBS-G wieder aufsetzen. Dann übertragen Sie das Volumen auf ein 1,5 ml Rohr und Zentrifuge bei 1.600 x g für 5 min. Entsorgen Sie den Überstand.
HINWEIS: Zellpellets können in flüssigem Stickstoff eingefroren und bei -80 °C oder flüssigem Stickstoff gelagert werden. - Das Pellet in 0,5 ml Lysepuffer (25 mM HEPES, 150 mM NaCl, 1% t-Octylphenoxypolyethoxyethanol, pH 7,4) mit 1,5-fachem Protease-Inhibitor-Cocktail und 1x Phosphatase-Inhibitor-Cocktail(Materialtabelle) vorgekühlt in Eis lyse die Zellen. 10 min rotieren bei 50 Umdrehungen bei 4 °C.
HINWEIS: Dies ist ein kritischer Schritt, da Proteine abgebaut werden können, wenn sie nicht wie angegeben behandelt werden. Überprüfen Sie bei Bedarf die Integrität von Proteinlysat durch Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS/PAGE).
VORSICHT: T-Octylphenoxypolyethoxyethanol ist giftig und kann Haut- und Augenreizungen verursachen. Verwenden Sie Handschuhe, Augenschutz und Gesichtsschutz. - Zentrifugieren Sie das Lysat bei 14.000 x g für 10 min bei 4 °C. Sammeln Sie den Überstand in einem neuen 1,5 ml Rohr für die Bindung von Assays. Halten Sie 5 % des gesamten Lysats (Input) für die Western Blotting-Analyse. Der Überstand enthält Parasitenproteine, die mit Lysepuffer extrahiert wurden.
- Sammeln Sie 50 L IPs oder PIs, die zu Agaroseperlen konjugiert sind (d. h. 50 l Gülle) oder 50 l Agaroseperlen und Zentrifuge für 1 min bei 1.000 x g. Entsorgen Sie den Überstand und setzen Sie ihn in 50 l Bindungspuffer aus, um die Perlen auszulagern. Verwenden Sie nicht konjugierte Perlen als Kontrolle. Verwenden Sie IP/PI-Perlen mit unterschiedlicher Phosphatkonfiguration, einschließlich nicht phosphorylierter Formen, um unspezifische Wechselwirkungen zu kontrollieren.
- Fügen Sie dem Zelllysat oder gereinigten Proteinen 50 l IP- oder PI-Perlen hinzu (jeweils 1 ml Perlen enthalten 10 Nmol konjugierter IPs oder PIs). Halten Sie das Volumen der IP- oder PI-Perlen innerhalb von 10% des Gesamtlysats und passen Sie bei Bedarf das Bindungsreaktionsvolumen mit Bindungspuffer an.
- Für Wettbewerbstests verschiedene Konzentrationen von nicht konjugierten IPs oder PIs (z. B. 1-, 10-, 100-facher Molüberschuss im Vergleich zu IP- oder PI-Perlen) zur Bindungsreaktion hinzufügen.
- Inkubieren Sie die Reaktion für 1 h oder über Nacht bei 4 °C und rotieren bei 50 Umdrehungen von 1. Minute.
HINWEIS: Bindungsreaktionen mit gereinigten Proteinen können bei RT je nach Stabilität des Proteins durchgeführt werden. Wenn Sie IPs oder PIs, die mit Biotin konjugiert sind, nur mit Schritt 1.1.9.1 fortfahren, fahren Sie andernfalls mit Schritt 1.1.10 fort.- Fügen Sie der Bindungsreaktion 50 l Streptavidin-konjugiert zu magnetischen Perlen hinzu und brüten sie 1 h bei 4 °C bei 50 Umdrehungen pro Minute.
- Zentrifugieren Sie die Mischung für 1 min bei 1.000 x g bei 4 °C. Entfernen Sie den Überstand (Durchfluss) und halten Sie das Pellet. Halten Sie 5% des Überstandes für Western Blot Analyse.
HINWEIS: Wenn Sie magnetische Perlen verwenden, entfernen Sie Überstand und führen Sie nachfolgende Wälwass mit einem magnetischen Ständer durch (Zentrifugationen sind nicht erforderlich). - Fügen Sie 1 ml Waschpuffer (25 mM HEPES, 300 mM NaCl, 0,2% 4-Nonylphenyl-Polyethylenglycol, pH 7,4) hinzu und setzen Sie das Harz durch Antippen oder Wirbeln des Rohres wieder auf (verwenden Sie keine Pipette, da Perlen an Pipettenspitzen befestigt werden können). Zentrifugieren Sie die Reaktion für 1 min bei 1.000 x g bei 4 °C und entsorgen Sie den Überstand. Wiederholen Sie den Vorgang für insgesamt fünf Wähbe.
VORSICHT: 4-Nonylphenyl-Polyethylenglycol ist giftig und kann Haut- und Augenreizungen verursachen. Verwenden Sie Handschuhe, Augenschutz und Gesichtsschutz. - Fügen Sie den Perlen 50 L 2x Laemmli-Puffer, ergänzt mit 710 mM 2-Mercaptoethanol, zu und mischen Sie sie durch Tippen oder Wirbel, um die Proteine zu vereiteln. Bei 95 °C 5 min erhitzen, dann 10.000 x g für 1 min zentrifugieren und den Überstand sammeln (eluierte Proteine enthalten). Alternativ können Elute-Proteine mit 8 M Harnstoff/100 mM Glycin-pH 2,9 verwendet werden, um die Verwendung von SDS zu vermeiden. Einfrieren Sie das Eluat bei -80 °C, andernfalls fahren Sie mit der Western Blot-Analyse fort.
VORSICHT: 2-Mercaptoethanol ist giftig und kann Haut-, Augen- und Atemwegsreizungen verursachen. Verwenden Sie Handschuhe und arbeiten Sie in der chemischen Kapuze.
- Wachsen Sie T. brucei-Zellen bis zur Mid-Log-Phase und überwachen Sie die Zelllebensfähigkeit und -dichte. Insgesamt reichen 5,0 x 107 Zellen für einen Bindungstest aus.
- Western Blotting-Analyse
- Mischen Sie 15 L-Eingangsproben (ab Schritt 1.1.6) oder Durchflussproben (ab Schritt 1.1.10) mit 5 l 4x Laemmli-Puffer. Wärmeeingangs- und Durchflussproben für 5 min bei 95 °C. Bei Proben, die in 8 M Harnstoff/100 mM Glycin-pH 2,9 eluiert werden, 15 l Eluat mit 5 l 4x Laemmli-Puffer mischen und 5 min bei 95 °C erhitzen.
HINWEIS: Dieser Schritt ist nicht für Proben erforderlich, die im 2x Laemmli Puffer eluiert werden. - Laden Sie Brunnen von 4-20% SDS/PAGE-Gel mit 2,5 l Input, 2,5 l Durchfluss und 20 l eluierten Proben und laden Sie proteinleiter gemäß der Empfehlung des Herstellers.
HINWEIS: Wählen Sie Gel% nach dem Molekulargewicht des proteins von Interesse. - Führen Sie SDS/PAGE bei 150 V für 30-45 min im Laufpuffer aus, oder bis sich der blaue Farbstoff des Laemmli-Puffers am Ende des Gels befindet.
HINWEIS: Die Laufzeit kann je nach Laborausstattung variieren. - Entfernen Sie das Gel von den Glasplatten (oder Kunststoffplatten) und tränken Sie 15 min im Transferpuffer.
- Übertragen Sie die Proteine auf polyvinylidendifluorid (PVDF) Membran oder Nitrocellulosemembran. Einweichen Membranen und 3 mm Filterpapier im Transferpuffer. Montieren Sie ein Sandwich mit drei Blatt Filterpapier, Nitrocellulose oder PVDF-Membran, Gel und weiteren drei Blatt Filterpapier. Stellen Sie sicher, dass keine Luftblasen im Sandwich gefangen sind. Verwenden Sie eine Rolle, um Blasen zu entfernen, wenn nötig. Stellen Sie die Membran auf die Kathode und das Gel auf die Anodenseite der Kassette.
HINWEIS: In den Anweisungen des Membranherstellers finden Sie Informationen zur Membranaktivierung oder zur Blot-Vorbereitung. - Legen Sie die Kassette mit dem Sandwich in den Transfertank mit Transferpuffer. Legen Sie den Tank in einen Eiskübel oder bei 4 °C (z.B. im Kühlraum). Transferproteine bei 100 V für 1 h (Strom variiert zwischen 200-400 mA). Alternativ können Sie über Nacht bei einem konstanten Strom von 15 mA bei 4 °C übertragen.
- Entfernen Sie die Membran aus der Kassette. Inkubieren Sie die Membran in 6% fettfreie Trockenmilch, die in PBS mit 0,05% Polysorbat 20 (PBS-T) oder einer kompatiblen Blockierlösung verdünnt wird, für 1 h bei RT, um die Membran zu blockieren.
HINWEIS: Vor dem Blockieren der Membran kann die Qualität des Transfers mit Demonstchen Ponceau S überprüft werden. Inkubieren Sie die Membran für 1 min in 15 ml Ponceau S, spülen Sie sie in Wasser und visualisieren Sie Bänder. - Entfernen Sie die blockierende Lösung und inkubieren Sie die Membran für 1 h bei RT mit 50 Umdrehung in primären Antikörpern, die in 6% fettfreier Trockenmilch verdünnt in PBS-T verdünnt sind. Alternativ kann die Membran über Nacht bei 4 °C mit 50 Umdrehung entumt werden.
HINWEIS: Der Zeitpunkt der Inkubation kann je nach Qualität der Antikörper variieren; Die meisten Antikörper wirken jedoch mit Inkubationen von 1-3 h bei RT. Folgen Sie der Empfehlung des Herstellers für die Konzentration oder Verdünnung von Antikörpern. - Waschen Sie den Blot, indem Sie die Membran in PBS-T für 5 min mit 50 Umdrehung bei RT inkubieren. Wiederholen Sie den Vorgang 3-5 Mal. Je nach Qualität der Antikörper können weitere Wassen erforderlich sein.
- Inkubieren Sie die Membran in Meerrettichperoxidase (HRP)-konjugierten Sekundärantikörpern für 1 h bei RT in 6% fettfreier Trockenmilch, die in PBS-T mit 50 Umdrehungen pro Minute verdünnt wird.
HINWEIS: Befolgen Sie die Empfehlung des Herstellers für Antikörperkonzentration oder Verdünnung. - Waschen Sie den Blot wie in Schritt 1.2.9 angegeben.
- Fügen Sie das chemilumineszierende Substrat hinzu, um die Membran zu bedecken. Entfernen Sie den Überschuss an Substrat und brüten für 5 min bei RT im Dunkeln.
HINWEIS: Überprüfen Sie die Anweisungen des Herstellers für Empfehlungen zu den Chemilumineszenzreagenzien. - Erfassen Sie das chemilumineszierende Signal mit einem kamerabasierten Imager. Alternativ können Sie einen Röntgenfilm verwenden, um das chemilumineszierende Signal zu erfassen.
- Mischen Sie 15 L-Eingangsproben (ab Schritt 1.1.6) oder Durchflussproben (ab Schritt 1.1.10) mit 5 l 4x Laemmli-Puffer. Wärmeeingangs- und Durchflussproben für 5 min bei 95 °C. Bei Proben, die in 8 M Harnstoff/100 mM Glycin-pH 2,9 eluiert werden, 15 l Eluat mit 5 l 4x Laemmli-Puffer mischen und 5 min bei 95 °C erhitzen.
2. Analyse von IP/PI-bindenden Proteinen durch Affinitätschromatographie und Massenspektrometrie
- Zellwachstum, Lyse und Affinitätschromatographie
- Wachsen Sie T. brucei-Zellen bis zur Mid-Log-Phase und überwachen Sie die Zelllebensfähigkeit und -dichte.
HINWEIS: Insgesamt 1,0 x 1010 Zellen reichen für zwei Bindungstests. Die Verwendung von weniger Zellen als hier angegeben kann den Nachweis von Proteinen mit geringer Häufigkeit durch Massenspektrometrie beeinflussen.- Bei T. brucei Blutkreislaufformen wachsen Zellen in der Mid-Log-Phase (8,0 x 105-1,6 x 106 Zellen/ml) in HMI-9-Medien, ergänzt mit 10% FBS bei 37 °C und mit 5% CO2 . Für 427 Stamm, 5 L Kultur ergibt 0,5-1,0 x 1010 Zellen. Überwachen Sie das Zellwachstum, um eine Dichte von mehr als 1,8 x 106 Zellen/ml zu vermeiden, die die Zelllebensfähigkeit beeinträchtigen kann. Halten Sie das Volumen der Zellkultur auf 1/10 des Kolbenvolumens; Andernfalls wird die Wachstumsrate der Zellen aufgrund der schlechten Belüftung beeinflusst.
HINWEIS: Die 427-Sorte hat eine Verdoppelungszeit von 5,5-6,5 h. - Bei prozyklischen Formen wachsen Zellen in SDM-79 Medium, ergänzt mit 10% FBS bei 27 °C, und halten die Zelldichte zwischen 1,0 x 107 und 3,0 x 107 Zellen/ml. 500 ml Kultur ergeben 0,5-1,5 x 1010 Zellen.
- Bei T. brucei Blutkreislaufformen wachsen Zellen in der Mid-Log-Phase (8,0 x 105-1,6 x 106 Zellen/ml) in HMI-9-Medien, ergänzt mit 10% FBS bei 37 °C und mit 5% CO2 . Für 427 Stamm, 5 L Kultur ergibt 0,5-1,0 x 1010 Zellen. Überwachen Sie das Zellwachstum, um eine Dichte von mehr als 1,8 x 106 Zellen/ml zu vermeiden, die die Zelllebensfähigkeit beeinträchtigen kann. Halten Sie das Volumen der Zellkultur auf 1/10 des Kolbenvolumens; Andernfalls wird die Wachstumsrate der Zellen aufgrund der schlechten Belüftung beeinflusst.
- Zentrifugieren Sie die Zellen bei 1.600 x g für 15 min bei RT. Entsorgen Sie den Überstand und setzen Sie das Pellet in 200 ml PBS-G vorgeheizt bei 37 °C wieder auf. Verwenden Sie runde Bodenzentrifugationsrohre (Materialtabelle), da T. brucei Blutstrom-Formpellets leicht gestört werden, wenn sie zentrifugierte Rotoren mit festem Winkel verwenden. Zentrifugieren Sie die Zellen bei RT wieder bei 1.600 x g für 5 min.
- Entfernen Sie den Überstand und setzen Sie das Pellet in 10 ml PBS-G wieder auf. Zentrifugieren Sie die Zellen bei 1.600 x g für 5 min bei RT. Wiederholen Sie den Vorgang und nach der letzten Wäsche, entsorgen Sie den Überstand.
HINWEIS: Pellets können in flüssigem Stickstoff eingefroren und bei -80 °C oder flüssigem Stickstoff gelagert werden. - Das Zellpellet in 5 ml Lysepuffer vorgekühlt in Eis wieder aufhängen und mit 1,5-fachem Proteasehemmer-Cocktail und 1x Phosphatase-Inhibitor-Cocktail (TabellederMaterialien) ergänzen. Das Lysat 10 min bei 4 °C bei 50 Umdrehungen bei 50 Umdrehungen bei 50 Umdrehungen inkubieren.
- Zentrifugieren Sie das Lysat bei 10.000 x g für 10 min bei 4 °C. Sammeln Sie den Überstand (solubilisierte Proteine) und verdünnen Sie es in 20 ml Bindungspuffer.
- Sammeln Sie 400 L IPs oder PIs, die zu Agaroseperlen konjugiert sind (d. h. 400 l Gülle) oder 400 l Kontrollperlen und Zentrifugen für 1 min bei 1.000 x g bei RT. Entsorgen Sie den Überstand und setzen Sie ihn in 400 l Bindungspuffer wieder auf, um die Perlen zu auslagern. Verwenden Sie Agaroseperlen als Negativkontrolle, um eine spezifische Anreicherung von Protein-Metabolit-Wechselwirkungen im Vergleich zu unspezifischen Wechselwirkungen (z. B. Proteine, die nicht spezifisch an die Perlen binden) zu bestimmen. Verwenden Sie IPs oder PIs mit unterschiedlichen Phosphatkonfigurationen, einschließlich nicht phosphorylierter Formen, um unspezifische Wechselwirkungen aufgrund von Phosphatladungen oder Bindung an Biotin zu kontrollieren.
- Fügen Sie 400 l IP/PI-Perlen hinzu oder steuern Sie Perlen zu 10 ml Lysat und inkubiert für 1 h oder über Nacht bei 4 °C rotierend bei 50 Umdrehungen pro Minute. Wenn Sie IPs oder PIs verwenden, die nur mit Biotin (ohne Perlen) konjugiert sind, fahren Sie mit Schritt 2.1.7.1 fort, andernfalls fahren Sie mit Schritt 2.1.8 fort.
- 100 L Streptavidin-konjugiert zu magnetischen Perlen hinzufügen und 1 h bei 4 °C bei 50 Umdrehungen pro Minute inkubieren.
- Zentrifugieren Sie die Bindungsreaktion für 1 min bei 1.000 x g bei 4 °C. Entfernen Sie den Überstand (Durchfluss) und halten Sie das Pellet. Halten Sie 5% des Überstandes für Western Blot Analyse.
- 5 ml Waschpuffer in das Pellet geben, vorsichtig mischen, indem man das Rohr wirbelt, und dann zentrifugieren für 1 min bei 1.000 x g bei 4 °C. Entsorgen Sie den Überstand. Wiederholen Sie die Wäsche fünfmal. Bei Verwendung von Magnetperlen, sammeln Überstand und führen Sie Wärmittel mit einem magnetischen Ständer (Zentrifugationen sind nicht notwendig).
- Fügen Sie 50 L 2x Laemmli Puffer (oder 8 M Harnstoff/100 mM Glycin pH 2.9, um die Verwendung von SDS zu vermeiden) zu den Perlen und mischen durch Tippen oder Wirbel (vermeiden Pipettierung, weil Perlen an Pipettenspitzen befestigen können), und dann bei 95 °C für 5 min erhitzen. Zentrifuge für 10.000 x g für 1 min und sammeln Sie das Überstand (eluierte Proteine). Wiederholen Sie den Vorgang zweimal, um insgesamt drei Brüche zu sammeln.
- Eluat bei -80 °C einfrieren, ansonsten Proteine in SDS/PAGE trennen oder in Lösung für Diepinisierung und Massenspektrometrie-Analyse halten.
- Wachsen Sie T. brucei-Zellen bis zur Mid-Log-Phase und überwachen Sie die Zelllebensfähigkeit und -dichte.
- Trypsin-Verdauung von Proteinen für die Massenspektrometrie
HINWEIS: Zwei Varianten dieses Verfahrens sind für Abschnitt 2.2.1 (in Gel) oder Abschnitt 2.2.2 (in Lösung) Die Verdauung von Proteinen dargestellt. Proteinarme Bindungsröhrchen werden empfohlen, um Probenverluste zu verhindern. Konsultieren Sie mit einem analytischen Chemiker in der Proteomik-Anlage die Eignung des Protokolls für Proben und Massenspektrometer-Instrumente zur Verfügung.- In-Gel-Trypsinisierung von Proteinen
- Nach der Proteintrennung in SDS/PAGE das Gel kurz in hochreinem Wasser abspülen. Übertragen Sie das Gel auf eine saubere Glasplatte. Verbrauche Proteinbänder mit einer sauberen Klinge und vermeiden das Schneiden zusätzlicher Gel-Außenbänder. Die Gelstücke in kleine Stücke (d.h. ca. 1 mm Quadrat) schneiden und in ein 1 ml-Rohr geben. Verwenden Sie bei Bedarf eine Pipettenspitze, aber stellen Sie sicher, dass die Pipettespitze vor dem Gebrauch in Ethanol vorgespült wird.
HINWEIS: Verwenden Sie Handschuhe, um eine Gelkontamination zu vermeiden. Gelstücke können bei -20 °C gelagert werden. - Fügen Sie 100 l hochreines oder leistungsstarkes Flüssigchromatographiewasser zu Denröhrchen hinzu, um Gelstücke zu spülen. Entsorgen Sie das Wasser.
- Für Coomassie- oder Ruthenium-basierte fluoreszierende gebeizte Gelstücke die Gelstücke in Entsalzungslösung (25 mM NH4HCO3 in 50% Acetonitril) für 1 h inkubieren und dann entsorgen. Wiederholen Sie den Vorgang, bis die Färbung nicht sichtbar ist.
HINWEIS: Bereiten Sie Lösungen mit NH4HCO3 durch Verdünnung aus einer Lagerlösung bei 100 mM NH4HCO3, pH 7.8 vor.
VORSICHT: Acetonitril ist ein flüchtiges Lösungsmittel, entzündlich und giftig. NH4HCO3 kann Haut- oder Augenreizungen verursachen. Verwenden Sie Handschuhe und arbeiten Sie unter einer chemischen Haube. - Für silbergebeizte Gelstücke, Inkubationsgelstücke in 50 l Entsalzungslösung für Silberfleck (15 mM K3[Fe(CN)6], 50 mM Na2S2O3) in Wasser für 30 min. Entsorgen Sie die Lösung und waschen Gelstücke mit 200 l wasser. Waschen Sie fünfmal oder bis die gelbe Farbe des Gels nicht sichtbar ist.
VORSICHT: K3[Fe(CN)6] kann Haut- oder Augenreizungen verursachen. Verwenden Sie Handschuhe.
- Für Coomassie- oder Ruthenium-basierte fluoreszierende gebeizte Gelstücke die Gelstücke in Entsalzungslösung (25 mM NH4HCO3 in 50% Acetonitril) für 1 h inkubieren und dann entsorgen. Wiederholen Sie den Vorgang, bis die Färbung nicht sichtbar ist.
- Dehydrieren Sie die Gelstücke mit 200 l Acetonitril für 10 min bei RT. Entsorgen Sie dann die Lösung.
HINWEIS: Dehydrierte Gelstücke sind in Volumen kleiner, undurchsichtig und klebrig. Wenn mehrere Gelstücke in einem Rohr kombiniert werden, wiederholen Sie das Verfahren zur effizienten Austrocknung von Gelstücken. - Fügen Sie 50 l (oder genug Volumen, um die Gelstücke zu decken) der Reduktionslösung (10 mM Dithiothreitol [DTT] in 100 mM NH4HCO3) und inkubieren bei 56 °C für 1 h. Danach kühlen Sie die Rohre auf RT und entsorgen Sie den Überschuss der Reduktionslösung.
- Fügen Sie 50 l (oder genug Volumen, um die Gelstücke zu decken) der Alkylierungslösung (50 mM Iodoacetamid in 100 mM NH4HCO3) und inkubieren Sie für 30 min bei RT im Dunkeln. Danach entsorgen Sie den Überschuss der Alkylierungslösung.
- Die Gelstücke mit 200 l Acetonitril 10 min bei RT dehydrieren. Acetonitril entfernen und die Gelstücke mit 100 mM NH4HCO3 10 min bei RT hydratisieren.
- Die Gelstücke erneut mit 200 l Acetonitril für 10 min bei RT dehydrieren und den Überschuss der Lösung entsorgen.
- Fügen Sie 15 l Massespektrometrie Grade Trypsin in 50 mM NH4HCO3 Puffer verdünnt, oder genug Volumen, um hydratisierte Gelstücke zu decken und für 4 h oder über Nacht bei 37 °C zu brüten. Halten Sie die Gesamten der Trypsinmengen zwischen 100 und 500 ng (oder 20 ng Trypsin/g Protein).
- Kühlen Sie die Probe auf RT und Zentrifugen für 1 min bei 2.000 x g in einer Mikrozentrifuge. Fügen Sie 10-20 l 5% Ameisensäure in Wasser verdünnt und inkubieren für 10 min bei RT.
VORSICHT: Ameisensäure ist entzündlich, ätzend und giftig. Verwenden Sie Handschuhe und arbeiten Sie unter einer chemischen Haube. - Zentrifuge, wie in Schritt 2.2.1.9 angegeben, dann den Überstand (enthalten extrahierte Peptide) in einem anderen Rohr sammeln. Fügen Sie 20 l 5% Ameisensäure in 50-60% Acetonitril verdünnt in die Röhre und inkubieren für 10 min bei RT. Sammeln Sie extrahierte Peptidfraktionen in der gleichen Röhre.
- Trocknen Sie die Probe in einem Vakuumkonzentrator und rekonstituieren Sie sich in 10 l 0,5% Essigsäure und 2% Acetonitril für die Massenspektrometrieanalyse. Zentrifuge und sammeln die Lösung an der Unterseite des Rohres; Lösung bei -20 °C oder -80 °C lagern.
- Nach der Proteintrennung in SDS/PAGE das Gel kurz in hochreinem Wasser abspülen. Übertragen Sie das Gel auf eine saubere Glasplatte. Verbrauche Proteinbänder mit einer sauberen Klinge und vermeiden das Schneiden zusätzlicher Gel-Außenbänder. Die Gelstücke in kleine Stücke (d.h. ca. 1 mm Quadrat) schneiden und in ein 1 ml-Rohr geben. Verwenden Sie bei Bedarf eine Pipettenspitze, aber stellen Sie sicher, dass die Pipettespitze vor dem Gebrauch in Ethanol vorgespült wird.
- In Lösung Trypsinisierung von Proteinen
- Niederschlagen Sie Proteine, um das Probenvolumen, die Entsalzung und den Pufferaustausch zu reduzieren. Fügen Sie der Probe sechs Volumina von gekühltem (-20 °C) Aceton hinzu, z. B. 600 l Aceton bis 100 l Probe. Vortex und inkubieren bei -20 °C für 15 min bis 1 h. Die Lösung wird trüb oder bildet einen Niederschlag.
VORSICHT: Aceton ist giftig und entzündlich. Verwenden Sie Handschuhe und arbeiten Sie unter einer chemischen Haube. - Zentrifugenproben bei 4 °C für 30 min. Aceton dekantieren und das Pellet 15 min lang lufttrocknen.
- Fügen Sie 10 l 6-8 M Harnstoff oder 1% SDS in 50 mM NH4HCO3 hinzu, um das Pellet und den Wirbel zu mischen. Für größere Mengen an Proteinen (> 10 g) verwenden Sie bis zu 20 l Auflösungspuffer.
- Fügen Sie 5 L der reduzierenden Lösung und Wirbel. Drehen Sie das Volumen mit einer Mikrozentrifuge nach unten. Wenn Proben in Harnstoff verdünnt werden, dann für 1 h bei RT inkubieren. Wenn Proben in SDS verdünnt werden, dann für 1 h bei 56 °C inkubiert.
- Drehen Sie das Volumen mit einer Mikrozentrifuge nach unten. Fügen Sie 3 L Alkylierungslösung und Wirbel hinzu. Dann drehen Sie das Volumen nach unten und inkubieren sie im Dunkeln für 30 min bei RT.
- Fügen Sie 3 L der reduzierenden Lösung hinzu, um die Reaktion zu neutralisieren. Die Probe langsam auf 1 M Harnstoff oder 0,05% SDS mit 50 mM NH4HCO3verdünnen.
HINWEIS: Der Trypsin-Verdauungspuffer muss über Waschmittel oder Denaturierungsmittel verfügen. Konzentrationsgrenzwerte für Denaturierungsmittel sind: 0,05% SDS; 0,1% Octyl B-D-Glucopyranosid; 0,1% 4-Nonylphenyl-Polyethylenglycol; 0,1% t-Octylphenoxypolyethoxyethanol; 0,1% Polysorbat 20; 0,1% 3-[(3-cholamidopropyl)dimethylammonio]-1-propanesulfonat; < 1 M Harnstoff oder Thiourea. - Fügen Sie 5 l Massespektrometrie Grade Trypsin in 50 mM NH4HCO3 Puffer verdünnt und inkubieren für 4 h, oder über Nacht, bei 37 °C. Halten Sie die gesamten Trypsin-Mengen zwischen 100 und 500 ng (oder 20 ng Trypsin/g Protein).
- Kühlen Sie die Probe auf RT, und drehen Sie das Volumen mit einer Mikrozentrifuge nach unten. Fügen Sie 5% Essigsäure (oder 5% Ameisensäure in 50% Acetonitril) hinzu, um die Reaktion zu löschen.
- Trocknen Sie die Proben in einem Vakuumkonzentrator, wie in Schritt 2.2.1.11 angegeben. Proben bei -80 °C lagern. Desalt und Konzentrat Peptide mit einer umgekehrten Phasensäule wie C18 Zip-Tip und dann durch Massenspektrometrie analysieren.
- Niederschlagen Sie Proteine, um das Probenvolumen, die Entsalzung und den Pufferaustausch zu reduzieren. Fügen Sie der Probe sechs Volumina von gekühltem (-20 °C) Aceton hinzu, z. B. 600 l Aceton bis 100 l Probe. Vortex und inkubieren bei -20 °C für 15 min bis 1 h. Die Lösung wird trüb oder bildet einen Niederschlag.
- In-Gel-Trypsinisierung von Proteinen
Ergebnisse
Analyse der RAP1- und PI(3,4,5)P3-Wechselwirkung durch Affinitätschromatographie und Western Blotting
Dieses Beispiel veranschaulicht die Anwendung dieser Methode zur Analyse der Bindung von PIs durch RAP1 aus T. brucei lysate oder durch rekombinantes T. brucei RAP1-Protein. Lysate von T. brucei Blutbahnformen, die Hemagglutinin (HA)-getaggt RAP1 ausdrücken, wurden in Bindungstests verwendet. RAP1 ist ein Protein, das an der transkriptionellen Kontrolle der Varianten-Oberflächenglykoprotein -Gene (VSG)3,48beteiligt ist, die für Oberflächenproteine kodieren, die an der Parasiten-Immunflucht durch antigene Variation49beteiligt sind. RAP1 interagiert innerhalb eines telomerischen Proteinkomplexes mit dem Phosphatidylinositol-Enzym 5-Phosphatase (PIP5Pase)3, das auch bei der Kontrolle der VSG-Gentranskription1,3funktioniert. RAP1 hat eine N-terminale Brustkrebs-1-Carboxyl-Terminal-Domäne (BRCT), auf die eine Myeloblastose (Myb) DNA-Bindungsdomäne und eine C-terminale mybähnliche Domäne3,48folgen. Es fehlen jedoch kanonische PI-Bindungsdomänen. Bindungstests wurden mit PIs durchgeführt, die nicht phosphoryliert sind oder an verschiedenen Positionen des Inositolrings phosphoryliert sind, und mit nicht konjugierten Agaroseperlen. Die westliche Analyse zeigt, dass RAP1 bevorzugt an PI(3,4,5)P3-Perlen bindet (Abbildung 3A), aber es bindet auch in geringerem Maße an PI(4,5)P2-Perlen. Es bindete jedoch nicht an andere PIs oder Agarose-Perlen. Da RAP1 Teil eines Multiprotein-Komplexes3ist, ist seine Interaktion mit einigen PIs möglicherweise nicht direkt und daher resultiert aus rap1-HA Interaktion mit anderen zellulären Proteinen, die an PIs binden.
Um zu testen, ob RAP1 direkt an PIs bindet, wurde daher ein C-terminalmitiertes 6x-sein rekombinantes RAP1 (rRAP1)-Protein exprimiert und von E. coli3zur Homogenitätgereinigt. Das Protein wurde in bindungstests mit PI(3,4,5)P3-Perlen in Gegenwart konkurrierender Konzentrationen von PI(3,4,5)P3 oder PI(4,5)P2 verwendet. Western Blotting zeigt, dass steigende Konzentrationen von PI(3,4,5)P3, aber nicht PI(4,5)P2 die Wechselwirkung von rRAP1 mit PI(3,4,5)P3 (Abbildung 3B) hemmen. Darüber hinaus ist die Zugabe des T. brucei-gereinigten PIP5Pase-Enzyms zur Reaktion wiederhergestellte PI(3,4,5)P3-Bindung durch rRAP1, die auf pip5Pase-Dephosphorylierung freier PI(3,4,5)P33 zurückzuführen ist und somit darauf hinweist, dass das Phosphorylierungsmuster dieser Metabolit ist für die rRAP1-Bindung unerlässlich. Daher interagiert rRAP1 mit PI(3,4,5)P3 wie RAP1-HA von T. brucei lysates. Darüber hinaus zeigen die Daten, dass die Bindung von RAP1-HA von Lysat zu PI(4,5)P2 wahrscheinlich auf rap1-Wechselwirkungen mit anderen Proteinen im Komplex (z.B. PIP5Pase)3resultiert. Die Daten veranschaulichen die Komplementarität von Bindungsassays mit Zelllysaten und rekombinanten Proteinen. Es zeigt auch den Nutzen von wettbewerbsfähigen Bindungstests, um die Spezifität der Wechselwirkungen zwischen Proteinen und PIs zu bestimmen.
Identifizierung von Ins(1,4,5)P3-bindenden Proteinen durch Affinitätschromatographie und Massenspektrometrie
In diesem Beispiel wurde die Affinitätschromatographie gefolgt von der Massenspektrometrie verwendet, um T. brucei-Proteine zu identifizieren, die an Ins(1,4,5)P3 binden; Daher untersucht das Experiment potenzielle Ins(1,4,5)P3-bindende Proteine aus T. brucei Blutkreislaufformen. T. brucei lysat wurde mit Ins(1,4,5)P3 konjugiert zu Agaroseperlen oder mit nicht konjugierten Perlen (als Kontrolle verwendet) inkubiert und gebundene Proteine mit Laemmli-Probenpuffer eluiert. Die SDS/PAGE-Analyse zeigt die Anreicherung von Proteinen, die von Ins(1,4,5)P3-Perlen im Vergleich zu Proteinen, die aus den Kontrollagaroseperlen eluiert werden , eluiert werden (Abbildung 4A). Massenspektrometrie-Analyse von eluierten Proteinen identifiziert über 250 Proteine, von denen 84 mit Ins(1,4,5)P3 Perlen im Vergleich zu Kontrollperlen angereichert wurden (Abbildung 4B, Faltenänderung [FC] 2, p < 0,05). Die Anreicherung von Proteinen, die im Vergleich zu Kontrollperlen an Ins(1,4,5)P3 gebunden sind, korreliert mit dem von SDS/PAGE detektierten Proteinsignal. Die Daten umfassen Proteine, die validiert wurden, um Ins(1,4,5)P3 zu binden, und Proteine, deren Bindungsmechanismen an Ins(1,4,5)P3 unbekannt sind8. Darüber hinaus unterschied sich das Ins(1,4,5)P3-Bindungsproteom stark von dem von Ins(1,3,4,5)P4 und anderen PIs8, was darauf hindeutet, dass einige dieser Proteine die spezifische Phosphatkonfiguration von Ins(1,4,5)P3 erkennen. Daher kann biotin-markierte Ins(1,4,5)P3 für die Affinitätschromatographie gekoppelt an die Massenspektrometrie verwendet werden, um Proteine zu identifizieren, die an Ins(1,4,5)P3 binden. Der Ansatz kann untersucht werden, um Proteine zu identifizieren, die an andere IPs oder PIs binden3,8,46,47.

Abbildung 1 : Affinity Reagenzien für verbindliche Assays. (A) PI(3,4,5)P3 (oben) und Ins(1,4,5)P3 (unten) konjugiert mit Biotin in sn1 Position des Inositols. In PI(3,4,5)P3 wird das Biotin an der Sn1-Position des Inositols mit der Lipidkette konjugiert, während in Ins(1,4,5)P3 das Biotin an Position sn1 mit dem Phosphat konjugiert wird. (B) Ins(1,4,5)P3 wird mit Biotin konjugiert und über Bindung an Streptavidin konjugiert zu Perlen (z. B. Agarose oder Magnetperlen) gefangen. Variationen dieser Reagenzien mit benutzerdefinierten synthetisierten Linkern, die das Biotin ersetzen, sind ebenfalls möglich46. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
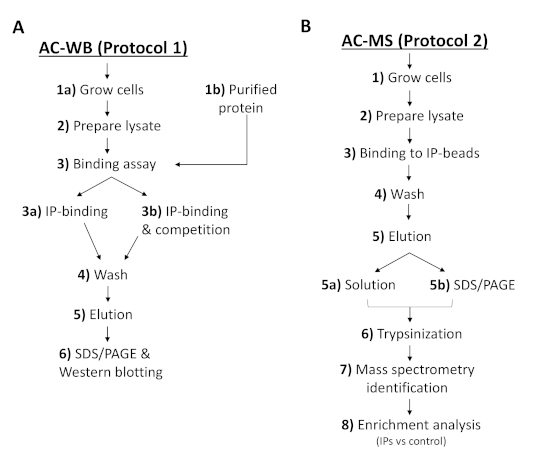
Abbildung 2 : Der Workflow der Protokolle beschreibt die Schritte zur Analyse der IP- oder PI-Affinitätsinteraktion mit Proteinen von T. brucei und der Detektion durch (A) Western Blotting oder (B) Massenspektrometrie. AC-WB, Affinitätschromatographie und Western Blotting; AC-MS, Affinitätschromatographie und Massenspektrometrie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3 : Bindung von T. brucei RAP1 an Phosphoinositide. (A) Lysate von T. brucei (5,0 x 107 Parasiten), die HA-markierte RAP1 ausdrücken, wurden für 2 h bei 4 °C mit 50 l PIs (jeweils 1 ml Agaroseperlen mit 10 nmol konjugierten PIs) oder Agarose (Ag) Beads inkubiert. Die Bindungsreaktion wurde mit 2 x Laemmli Probenpuffer gewaschen und eluiert und bei 95 °C für 5 min erhitzt. Proteine wurden in 4-20% SDS/PAGE abgetrennt, auf eine PVDF-Membran übertragen und mit monoklonalen Antikörpern Anti-HA (1:5.000, verdünnt in 6% PBS-Milch) untersucht. per Anti-Maus-IgG-HRP (1:5.000, verdünnt in 6% PBS-Milch) und durch Chemilumineszenz nachgewiesen. (B) Eine g rRAP1 wurde für 1 h bei RT mit 50 l PI(3,4,5)P3-Agarose-Perlen in Gegenwart oder Abwesenheit von 5 bis 50 m Dioctanoylglycerol (diC8) PI(3,4,5)P3, 20 bis 50 m diC8 PI(4,5)P2 oder 50 m diC8 PI(3,4,5)P3 und 250 ng PIP5Pase gereinigt von T. brucei Blutkreislauf formen3. Die Bindung wurde durch Western Blotting mit Mausanti-His HRP monoklonalen Antikörpern (1:2.000, verdünnt in 6% PBS-Milch) analysiert und durch Chemilumineszenz entwickelt. Diese Zahl wurde von Cestari et al.3geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4 : Affinitätschromatographie und Massenspektrometrieanalyse von T. brucei-Proteinen, die an Ins(1,4,5)P3 binden. (A) 10% SDS/PAGE-Analyse von T. brucei-Proteinen, die an Ins(1,4,5)P3-Perlen oder Agaroseperlen binden. Lysate von 5,0 x 109 Parasiten wurden bei 4 °C für 2 h mit 400 l Ins(1,4,5)P3 konjugiert zu Agaroseperlen oder mit Agaroseperlen ohne Ins(1,4,5)P3 [1 ml Perlen enthalten 10 Nmol konjugierter Ins(1,4,5)P3] inkubiert. Die Bindungsreaktion wurde gewaschen, in 2 x Laemmli Probenpuffer eluiert und 5 min bei 95 °C gekocht. Proteine wurden in 10% SDS/PAGE getrennt und mit Coomassie-Färbung(Materialtabelle)gefärbt. Pfeilspitzen zeigen Proteine, die in Ins(1,4,5)P3-Perlen im Vergleich zu Agaroseperlen angereichert sind; Kreise zeigen Proteine an, die in beiden Ins(1,4,5)P3-Perlen und Agaroseperlen vorhanden sind, und die Klammer zeigt Proteine an, die in beiden vorhanden sind, aber in Ins(1,4,5)P3-Perlen im Vergleich zu Agaroseperlen angereichert sind. (B) Dot-Plot zeigt Proteine, die durch Massenspektrometrie identifiziert werden und in Ins(1,4,5)P3-Perlen im Vergleich zu Agaroseperlen angereichert sind. Anreicherung definiert durch FC > 2 und p-Wert <0.05. Vier biologische Repliken wurden für Agarose-Perlen AC-MS und drei biologische Repliken für IP3-Perlen AC-MS verwendet. Faltenwechsel von Proteinen, die in IP3-Perlen im Vergleich zu Agarose-Perlen identifiziert wurden, wurde mit Peptidspektrenintensität mit MSstat50berechnet. Detaillierte Ergebnisse und Liste der Peptide sind verfügbar8. Massenspektrometrie-Rohdaten sind auch mit dem Identifikator PXD005907 über das ProteomeXchange Consortium über das PRIDE-Partner-Repository verfügbar. Diese Zahl wurde von Cestari et al.8geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die Identifizierung von Proteinen, die an IPs oder PIs binden, ist entscheidend, um die zelluläre Funktion dieser Metaboliten zu verstehen. Die Affinitätschromatographie, gekoppelt mit der Westlichen Fleck- oder Massenspektrometrie, bietet die Möglichkeit, IP- oder PI-interagierende Proteine zu identifizieren und so Einblicke in ihre regulatorische Funktion zu gewinnen. IPs oder PIs, die chemisch getaggt sind [z.B., Ins(1,4,5)P3, die chemisch mit Biotin verbunden sind] und über Streptavidin mit Agaroseperlen verbunden oder mit Streptavidin-Magnetperlen erfasst werden, ermöglichen die Isolierung interagierender Proteine, die dann durch Masse identifiziert werden können. Spektrometrie oder Western Blot. Die hier beschriebenen Protokolle wurden verwendet, um Proteine aus T. brucei3,8 und Säugetierzellen47 zu identifizieren, die an diese Metaboliten binden. Variationen des Ansatzes, der benutzerdefinierte Tags (außer Biotin) verwendet, wurden auch in Hefe46verwendet. Ein wichtiger Aspekt dieses Ansatzes ist die Verwendung von Kontrollen, um bestimmte von unspezifischen Interaktionen zu unterscheiden. Nicht konjugierte Perlen sind ein wesentliches Steuerelement, aber zusätzliche Kontrollen können nicht-phosphorylierte PIs oder Inositol konjugiert zu Perlen3umfassen. IPs oder PIs mit unterschiedlichen Phosphatkombinationen3,8 können auch verwendet werden, weil Proteinbindung an diese Metaboliten Domänen beinhalten kann, die die Phosphatkonfiguration des Inositols38 39,40,41. Darüber hinaus kann die Probenkomplexität die Empfindlichkeit des Ansatzes beeinflussen, und daher kann eine Verringerung der Probenkomplexität durch Probenfraktionierung den Nachweis von proteinarmen Proteinen in der Zelle unterstützen. Es gibt etablierte Protokolle für die Zellfraktionierung und Isolierung von Mitochondrion51, Kern52, Glykosom53und Flagellum54,55 von Trypanosomen. Beachten Sie, dass Puffer und Reagenzien, die in subzellulären Fraktionen verwendet werden, möglicherweise an die Kompatibilität mit Puffern und Reagenzien angepasst werden müssen, die in diesem Protokoll verwendet werden. Insbesondere hängt die Identifizierung von Proteinen, die durch Affinitätschromatographie an IPs oder PIs binden, wie hier angegeben, von der Protein- und Metabolitenaffinität der Interaktion ab, und daher können Proteine, die eine schwache Affinität zu IPs oder PIs aufweisen, möglicherweise nicht ohne weiteres nachgewiesen werden.
Die Analyse der IP- oder PI-Interaktion mit Proteinen aus Zelllysat kann auch zur Identifizierung von Proteinen führen, die nicht direkt an diese Metaboliten binden, aber aus einer Proteinassoziation in komplex mit anderen Proteinen resultieren, die an IPs binden. oder PIs. Diese Funktion wird in Abbildung 3Averanschaulicht, in der RAP1-HA aus T. brucei lysate an PI(3,4,5)P3-Perlen und PI(4,5)P2-Perlen zu binden scheint. Verbindliche Assays mit His-tagged rRAP1 zeigen jedoch, dass dieses Protein an PI(3,4,5)P3 bindet und nicht an PI(4,5)P23. Dies ist in Abbildung 3Bdargestellt, in der wettbewerbsorientierte Assays zeigen, dass freie PI(3,4,5)P3, aber nicht freie PI(4,5)P2 um rRAP1-seine Interaktion mit PI(3,4,5)P3-Perlen konkurriert. Die RAP1-scheinbare Wechselwirkung mit PI(4,5)P2-Perlen ist auf die RAP1-Assoziation innerhalb eines Komplexes mit Proteinen zurückzuführen, die PI(4,5)P2 (z.B. PIP5Pase)3binden. Daher können bindungsspezifische Assays von Zelllysaten Proteine identifizieren, die direkt oder indirekt an IPs oder PIs binden. Insbesondere unterscheiden sich indirekte Wechselwirkungen von unspezifischen Wechselwirkungen, da erstere eine biologische Funktion (im Kontext des Proteinkomplexes) haben können, die die Metabolitenbindung beeinflusst oder beeinflusst. Beispielsweise bindet Ins(1,4,5,6)P4 an den Multi-Subunit Co-Repressor Deacetylase-Komplex und steuert die komplexe Baugruppe und Aktivität13. Daher ist es wichtig, IP/PI-Wechselwirkungen mit Proteinen zu validieren. Die Validierung der Wechselwirkung kann Wettbewerbstests mit einem Überschuss an IPs oder PIs (wie in Abbildung 3B)3,8, Mutationen potenzieller Proteindomänen56, oder die Verwendung von gereinigten Proteinen direkte Wechselwirkungen bestimmen (Abbildung 3A,B)3.
Andere Methoden zur Untersuchung von Protein- und IP- oder PI-Wechselwirkungen umfassen die Bindung von Proteinen an radioaktiv markierte IPs oder PIs, die Verwendung von IPs oder PIs, die an hydrophobe Membranen als Matrizen für die Proteinaufnahme gebunden sind, oder die Bindung von Proteinen an in Liposomen integrierte PIs. 42 , 57 , 58. Wichtig ist, wenn die Proteininteraktion mit PIs Membranstrukturenerfordert 38, Liposomen-basierte Assays können als komplementärer Ansatz verwendet werden. Zu den Einschränkungen dieser Ansätze gehören geringer Durchsatz, geringe Empfindlichkeit, die unbekannte chemische Ausrichtung von IPs oder PIs Assoziation zu Matrizen58, oder die Verwendung radioaktiver Materialien42. Die hier beschriebene Methode ist empfindlich, liposomenfrei, nicht radioaktiv, und IP/PI-Perlen sind kommerziell erhältlich und erfordern daher keine kundenspezifische chemische Synthese. Darüber hinaus ist die Position des Biotins, das mit IPs oder PIs verknüpft ist, klar definiert, und es kann auch modifiziert werden46,58, was eine präzise Analyse der Protein- und Metaboliteninteraktion ermöglicht. Die hier beschriebene Methode kann auch mit quantitativen Massenspektrometrieansätzen wie der stabilen Isotopenkennzeichnung von Aminosäuren in der Zellkultur (SILAC)47kombiniert werden, mit denen dynamische Wechselwirkungen unter verschiedenen zellulären Behandlungen oder Bedingungen. Die Affinitätschromatographie, gekoppelt mit Western Blot oder Massenspektrometrie, hat dazu beigetragen, zahlreiche IP- oder PI-bindende Proteine aus T. brucei, Säugetierzellen und Hefe3,8,46, 47, einschließlich Proteine, die keine IP- oder PI-bindenden Domänen gekennzeichnet haben, und es hat auch zur Identifizierung neuartiger Bindungsdomänen beigetragen, z. B. PI(3,4,5)P3-Bindungsdomäne47.
Insgesamt können die hier beschriebenen Protokolle verwendet werden, um potenzielle IP- oder PI-interagierende Proteine von T. bruceizu untersuchen und die molekulare Wechselwirkung von Proteinen mit diesen Metaboliten zu untersuchen. Das Protokoll kann leicht angepasst werden, um IP- oder PI-bindende Proteine von anderen einzelligen Parasiten oder von anderen Organismen wie Densäuerzellen47 und Hefe46zu identifizieren, und es wird dazu beitragen, die biologische Funktion von IP und PIs in eukaryotes.
Offenlegungen
Der Autor hat nichts zu verraten.
Danksagungen
Diese Arbeit wurde vom Natural Sciences and Engineering Research Council of Canada (NSERC, RGPIN-2019-04658) unterstützt. NSERC Discovery Launch Supplement for Early Career Researchers (DGECR-2019-00081) und von der McGill University.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetone | Sigma-Aldrich | 650501 | Ketone |
| Acetonitrile | Sigma-Aldrich | 271004 | Solvent |
| Ammonium bicarbonate | Sigma-Aldrich | A6141 | Inorganic salt |
| Centrifuge Avanti J6-MI | Beckman Coulter | Avanti J6-MI | Centrifuge for large volumes (e.g., 1L) |
| Centrifuge botles | Sigma-Aldrich | B1408 | Bottles for centrifugation of 1L of culture |
| Control Beads | Echelon | P-B000-1ml | Affinity chromatography reagent - control |
| D-(+)-Glucose | Sigma-Aldrich | G8270 | Sugar, Added in PBS to keep cells viable |
| Dithiothreitol (DTT) | Bio-Rad | 1610610 | Reducing agent |
| Dynabeads M-270 Streptavidin | ThermoFisher Scientific | 65305 | Streptavidin beads for binding to biotin ligands |
| EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitors |
| Electrophoresis running buffer | Bio-Rad | 1610732 | 25 mM Tris, 192 mM glycine, 0.1% SDS, pH 8.3 |
| Falcon 15 mL Conical Centrifuge Tubes | Corning Life Sciences | 430052 | To centrifuge 10 mL cultures |
| Formic acid | Sigma-Aldrich | 106526 | Acid |
| Glycine | Sigma-Aldrich | G7126 | Amino acid |
| HMI-9 cell culture medium | ThermoFisher Scientific | ME110145P1 | Cell culture medium for T. brucei bloodstream forms |
| Imperial Protein Stain | ThermoFisher Scientific | 24615 | Coomassie staining for protein detection in SDS/PAGE |
| Ins(1,4,5)P3 Beads | Echelon | Q-B0145-1ml | Affinity chromatography reagent |
| Instant Nonfat Dry Milk | Thomas Scientific | C837M64 | Blocking reagent for Western blotting |
| Iodoacetamide | Sigma-Aldrich | I6125 | Alkylating reagent for cysteine proteins or peptides |
| Lab Rotator | Thomas Scientific | 1159Z92 | For binding assays |
| LoBind Microcentrifuge Tubes | ThermoFisher Scientific | 13-698-793 | Low protein binding tubes for mass spectrometry |
| Nonidet P-40 (Igepal CA-630) | Sigma-Aldrich | 21-3277 | Detergent |
| PBS, pH 7.4 | ThermoFisher Scientific | 10010031 | Physiological buffer |
| Peroxidase substrate for chemiluminescence | ThermoFisher Scientific | 32106 | Substrate for Western bloting detection of proteins |
| PhosSTOP Phosphatase Inhibitor Cocktail Tablets | Roche | 4906845001 | Phosphatase inhibitors |
| PI(3)P PIP Beads | Echelon | P-B003a-1ml | Affinity chromatography reagent |
| PI(3,4)P2 PIP Beads | Echelon | P-B034a-1ml | Affinity chromatography reagent |
| PI(3,4,5)P3 diC8 | Echelon | P-3908-1mg | Affinity chromatography reagent |
| PI(3,4,5)P3 PIP Beads | Echelon | P-B345a-1ml | Affinity chromatography reagent |
| PI(3,5)P2 PIP Beads | Echelon | P-B035a-1ml | Affinity chromatography reagent |
| PI(4)P PIP Beads | Echelon | P-B004a-1ml | Affinity chromatography reagent |
| PI(4,5)P2 diC8 | Echelon | P-4508-1mg | Affinity chromatography reagent |
| PI(4,5)P2 PIP Beads | Echelon | P-B045a-1ml | Affinity chromatography reagent |
| PI(5)P PIP Beads | Echelon | P-B005a-1ml | Affinity chromatography reagent |
| Ponceau S solution | Sigma-Aldrich | P7170 | Protein staining (0.1% [w/v] in 5% acetic acid) |
| Potassium hexacyanoferrate(III) | Sigma-Aldrich | 702587 | Potassium salt |
| PtdIns PIP Beads | Echelon | P-B001-1ml | Affinity chromatography reagent |
| PVDF Membrane | Bio-Rad | 1620177 | For Western blotting |
| Refrigerated centrifuge | Eppendorf | 5910 R | Microcentrifuge for small volumes (e.g., 1.5 mL) |
| Sodium dodecyl sulfate | Sigma-Aldrich | 862010 | Detergent |
| Sodium thiosulfate | Sigma-Aldrich | 72049 | Chemical |
| SpeedVac Vacuum Concentrators | ThermoFisher Scientific | SPD120-115 | Sample concentration (e.g., for mass spectrometry) |
| T175 flasks for cell culture | ThermoFisher Scientific | 159910 | To grow 50 mL T. brucei culture |
| Trypsin, Mass Spectrometry Grade | Promega | V5280 | Trypsin for protein digestion |
| Urea | Sigma-Aldrich | U5128 | Denaturing reagent |
| Vortex | Fisher Scientific | 02-215-418 | For mixing reactions |
| Western blotting transfer buffer | Bio-Rad | 1610734 | 25 mM Tris, 192 mM glycine, pH 8.3 with 20% methanol |
| Whatman 3 mm paper | Sigma-Aldrich | WHA3030861 | Paper for Wester transfer |
| 2-mercaptoethanol (14.2 M) | Bio-Rad | 1610710 | Reducing agent |
| 2x Laemmli Sample Buffer | Bio-Rad | 161-0737 | Protein loading buffer |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels | Bio-Rad | 4561094 | Gel for protein electrophoresis |
| 4x Laemmli Sample Buffer | Bio-Rad | 161-0747 | Protein loading buffer |
Referenzen
- Cestari, I., Stuart, K. Inositol phosphate pathway controls transcription of telomeric expression sites in trypanosomes. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2803-2812 (2015).
- Odom, A. R., Stahlberg, A., Wente, S. R., York, J. D. A role for nuclear inositol 1,4,5-trisphosphate kinase in transcriptional control. Science. 287 (5460), 2026-2029 (2000).
- Cestari, I., McLeland-Wieser, H., Stuart, K. Nuclear Phosphatidylinositol 5-Phosphatase Is Essential for Allelic Exclusion of Variant Surface Glycoprotein Genes in Trypanosomes. Molecular and Cellular Biology. 39 (3), (2019).
- Billcliff, P. G., et al. OCRL1 engages with the F-BAR protein pacsin 2 to promote biogenesis of membrane-trafficking intermediates. Molecular Biology of the Cell. 27 (1), 90-107 (2016).
- Brochet, M., et al. Phosphoinositide metabolism links cGMP-dependent protein kinase G to essential Ca(2)(+) signals at key decision points in the life cycle of malaria parasites. PLoS Biology. 12 (3), 1001806(2014).
- Azevedo, C., Szijgyarto, Z., Saiardi, A. The signaling role of inositol hexakisphosphate kinases (IP6Ks). Advances in Enzyme Regulation. 51 (1), 74-82 (2011).
- Szijgyarto, Z., Garedew, A., Azevedo, C., Saiardi, A. Influence of inositol pyrophosphates on cellular energy dynamics. Science. 334 (6057), 802-805 (2011).
- Cestari, I., Anupama, A., Stuart, K. Inositol polyphosphate multikinase regulation of Trypanosoma brucei life stage development. Molecular Biology of the Cell. 29 (9), 1137-1152 (2018).
- Bang, S., et al. AMP-activated protein kinase is physiologically regulated by inositol polyphosphate multikinase. Proceedings of the National Academy of Sciences of the United States of America. 109 (2), 616-620 (2012).
- Seeds, A. M., Tsui, M. M., Sunu, C., Spana, E. P., York, J. D. Inositol phosphate kinase 2 is required for imaginal disc development in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 112 (51), 15660-15665 (2015).
- Hamada, K., Miyatake, H., Terauchi, A., Mikoshiba, K. IP3-mediated gating mechanism of the IP3 receptor revealed by mutagenesis and X-ray crystallography. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), 4661-4666 (2017).
- Watson, P. J., et al. Insights into the activation mechanism of class I HDAC complexes by inositol phosphates. Nature Communications. 7, 11262(2016).
- Watson, P. J., Fairall, L., Santos, G. M., Schwabe, J. W. Structure of HDAC3 bound to co-repressor and inositol tetraphosphate. Nature. 481 (7381), 335-340 (2012).
- Irvine, R. F. Nuclear lipid signalling. Nature Reviews: Molecular Cell Biology. 4 (5), 349-360 (2003).
- Sobol, M., et al. UBF complexes with phosphatidylinositol 4,5-bisphosphate in nucleolar organizer regions regardless of ongoing RNA polymerase I activity. Nucleus. 4 (6), 478-486 (2013).
- Nascimbeni, A. C., et al. ER-plasma membrane contact sites contribute to autophagosome biogenesis by regulation of local PI3P synthesis. EMBO Journal. 36 (14), 2018-2033 (2017).
- Martin, K. L., Smith, T. K. The myo-inositol-1-phosphate synthase gene is essential in Trypanosoma brucei. Biochemical Society Transactions. 33, Pt 5 983-985 (2005).
- Matsu-ura, T., et al. Cytosolic inositol 1,4,5-trisphosphate dynamics during intracellular calcium oscillations in living cells. Journal of Cell Biology. 173 (5), 755-765 (2006).
- Blind, R. D., et al. The signaling phospholipid PIP3 creates a new interaction surface on the nuclear receptor SF-1. Proceedings of the National Academy of Sciences of the United States of America. 111 (42), 15054-15059 (2014).
- Lin, A., et al. The LINK-A lncRNA interacts with PtdIns(3,4,5)P3 to hyperactivate AKT and confer resistance to AKT inhibitors. Nature Cell Biology. 19 (3), 238-251 (2017).
- Steger, D. J., Haswell, E. S., Miller, A. L., Wente, S. R., O'Shea, E. K. Regulation of chromatin remodeling by inositol polyphosphates. Science. 299 (5603), 114-116 (2003).
- York, J. D., Odom, A. R., Murphy, R., Ives, E. B., Wente, S. R. A phospholipase C-dependent inositol polyphosphate kinase pathway required for efficient messenger RNA export. Science. 285 (5424), 96-100 (1999).
- Adams, R. L., Mason, A. C., Glass, L., Aditi,, Wente, S. R. Nup42 and IP6 coordinate Gle1 stimulation of Dbp5/DDX19B for mRNA export in yeast and human cells. Traffic. 18 (12), 776-790 (2017).
- Macbeth, M. R., et al. Inositol hexakisphosphate is bound in the ADAR2 core and required for RNA editing. Science. 309 (5740), 1534-1539 (2005).
- Dickson, E. J., Hille, B. Understanding phosphoinositides: rare, dynamic, and essential membrane phospholipids. Biochemical Journal. 476 (1), 1-23 (2019).
- Mellman, D. L., et al. A PtdIns4,5P2-regulated nuclear poly(A) polymerase controls expression of select mRNAs. Nature. 451 (7181), 1013-1017 (2008).
- Lee, Y. S., Mulugu, S., York, J. D., O'Shea, E. K. Regulation of a cyclin-CDK-CDK inhibitor complex by inositol pyrophosphates. Science. 316 (5821), 109-112 (2007).
- Nagy, A. I., et al. IP3 signalling regulates exogenous RNAi in Caenorhabditis elegans. EMBO Reports. 16 (3), 341-350 (2015).
- Hall, B. S., et al. TbVps34, the trypanosome orthologue of Vps34, is required for Golgi complex segregation. Journal of Biological Chemistry. 281 (37), 27600-27612 (2006).
- Rodgers, M. J., Albanesi, J. P., Phillips, M. A. Phosphatidylinositol 4-kinase III-beta is required for Golgi maintenance and cytokinesis in Trypanosoma brucei. Eukaryotic Cell. 6 (7), 1108-1118 (2007).
- Gilden, J. K., et al. The role of the PI(3,5)P2 kinase TbFab1 in endo/lysosomal trafficking in Trypanosoma brucei. Molecular and Biochemical Parasitology. 214, 52-61 (2017).
- Demmel, L., et al. The endocytic activity of the flagellar pocket in Trypanosoma brucei is regulated by an adjacent phosphatidylinositol phosphate kinase. Journal of Cell Science. 129 (11), 2285(2016).
- Gimenez, A. M., et al. Phosphatidylinositol kinase activities in Trypanosoma cruzi epimastigotes. Molecular and Biochemical Parasitology. 203 (1-2), 14-24 (2015).
- Hashimoto, M., et al. Inositol 1,4,5-trisphosphate receptor regulates replication, differentiation, infectivity and virulence of the parasitic protist Trypanosoma cruzi. Molecular Microbiology. 87 (6), 1133-1150 (2013).
- Cestari, I., Haas, P., Moretti, N. S., Schenkman, S., Stuart, K. Chemogenetic Characterization of Inositol Phosphate Metabolic Pathway Reveals Druggable Enzymes for Targeting Kinetoplastid Parasites. Cell Chemical Biology. 23 (5), 608-617 (2016).
- Lovett, J. L., Marchesini, N., Moreno, S. N., Sibley, L. D. Toxoplasma gondii microneme secretion involves intracellular Ca(2+) release from inositol 1,4,5-triphosphate (IP(3))/ryanodine-sensitive stores. Journal of Biological Chemistry. 277 (29), 25870-25876 (2002).
- McNamara, C. W., et al. Targeting Plasmodium PI(4)K to eliminate malaria. Nature. 504 (7479), 248-253 (2013).
- Tresaugues, L., et al. Structural basis for phosphoinositide substrate recognition, catalysis, and membrane interactions in human inositol polyphosphate 5-phosphatases. Structure. 22 (5), 744-755 (2014).
- Varnai, P., et al. Quantifying lipid changes in various membrane compartments using lipid binding protein domains. Cell Calcium. 64, 72-82 (2017).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Letters. 590 (15), 2454-2468 (2016).
- Cullen, P. J., Cozier, G. E., Banting, G., Mellor, H. Modular phosphoinositide-binding domains--their role in signalling and membrane trafficking. Current Biology. 11 (21), 882-893 (2001).
- Klarlund, J. K., et al. Signaling by phosphoinositide-3,4,5-trisphosphate through proteins containing pleckstrin and Sec7 homology domains. Science. 275 (5308), 1927-1930 (1997).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Gerasimaite, R., et al. Inositol Pyrophosphate Specificity of the SPX-Dependent Polyphosphate Polymerase VTC. ACS Chemical Biology. 12 (3), 648-653 (2017).
- Potapenko, E., et al. 5-Diphosphoinositol pentakisphosphate (5-IP7) regulates phosphate release from acidocalcisomes and yeast vacuoles. Journal of Biological Chemistry. 293 (49), 19101-19112 (2018).
- Wu, M., Chong, L. S., Perlman, D. H., Resnick, A. C., Fiedler, D. Inositol polyphosphates intersect with signaling and metabolic networks via two distinct mechanisms. Proceedings of the National Academy of Sciences of the United States of America. 113 (44), 6757-6765 (2016).
- Jungmichel, S., et al. Specificity and commonality of the phosphoinositide-binding proteome analyzed by quantitative mass spectrometry. Cell Reports. 6 (3), 578-591 (2014).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137 (1), 99-109 (2009).
- Cestari, I., Stuart, K. Transcriptional Regulation of Telomeric Expression Sites and Antigenic Variation in Trypanosomes. Current Genomics. 19 (2), 119-132 (2018).
- Clough, T., Thaminy, S., Ragg, S., Aebersold, R., Vitek, O. Statistical protein quantification and significance analysis in label-free LC-MS experiments with complex designs. BMC Bioinformatics. 13, Suppl 16 6(2012).
- Schneider, A., Charriere, F., Pusnik, M., Horn, E. K. Isolation of mitochondria from procyclic Trypanosoma brucei. Methods in Molecular Biology. 372, 67-80 (2007).
- DeGrasse, J. A., Chait, B. T., Field, M. C., Rout, M. P. High-yield isolation and subcellular proteomic characterization of nuclear and subnuclear structures from trypanosomes. Methods in Molecular Biology. 463, 77-92 (2008).
- Opperdoes, F. R. A rapid method for the isolation of intact glycosomes from Trypanosoma brucei by Percoll -gradient centrifugation in a vertical rotor. Molecular and Biochemical Parasitology. 3 (3), 181-186 (1981).
- Pereira, N. M., de Souza, W., Machado, R. D., de Castro, F. T. Isolation and properties of flagella of trypanosomatids. The Journal of protozoology. 24 (4), 511-514 (1977).
- Subota, I., et al. Proteomic analysis of intact flagella of procyclic Trypanosoma brucei cells identifies novel flagellar proteins with unique sub-localization and dynamics. Molecular and Cellular Proteomics. 13 (7), 1769-1786 (2014).
- Fukuda, M., Kojima, T., Kabayama, H., Mikoshiba, K. Mutation of the pleckstrin homology domain of Bruton's tyrosine kinase in immunodeficiency impaired inositol 1,3,4,5-tetrakisphosphate binding capacity. Journal of Biological Chemistry. 271 (48), 30303-30306 (1996).
- Knodler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. BioTechniques. 38 (6), (2005).
- Best, M. D. Global approaches for the elucidation of phosphoinositide-binding proteins. Chemistry and Physics of Lipids. 182, 19-28 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten