Method Article
Identificación de fosfato de inositol o proteínas que interactúan con fosfoinositida por cromatografía de afinidad acoplada a Western Blot o espectrometría de masas
En este artículo
Resumen
Este protocolo se centra en la identificación de proteínas que se unen a fosfatos de inositol o fosfoinosítidos. Utiliza cromatografía de afinidad con fosfatos de inositol biotinylados o fosfoinositidos que se inmovilizan a través de estreptavidina a agarosa o cuentas magnéticas. Las proteínas de unión de fosfato de inositol o fosfoinositida se identifican por la hincha occidental o espectrometría de masas.
Resumen
Fosfatos de inositol y fosfoinosítidos regulan varios procesos celulares en eucariotas, incluyendo la expresión génica, tráfico de vesículas, transducción de señal, metabolismo, y el desarrollo. Estos metabolitos realizan esta actividad reguladora uniéndose a proteínas, cambiando así la conformación de proteínas, la actividad catalítica y/o las interacciones. El método descrito aquí utiliza cromatografía de afinidad acoplado a espectrometría de masas o hincha occidental para identificar proteínas que interactúan con fosfatos de inositol o fosfoinositidas. Fosfatos de inositol o fosfoinositidas se etiquetan químicamente con biotina, que luego se captura a través de la estreptavidina conjugada con agarosa o cuentas magnéticas. Las proteínas se aíslan por su afinidad de unión al metabolito, luego se eluyen e identifican mediante espectrometría de masas o hincha occidental. El método tiene un flujo de trabajo simple que es sensible, no radioactivo, libre de liposomas y personalizable, apoyando el análisis de la interacción de proteínas y metabolitos con precisión. Este enfoque se puede utilizar en métodos de espectrometría cuantitativa de masas sin etiquetas o en aminoácidos para identificar interacciones proteína-metabolito en muestras biológicas complejas o utilizando proteínas purificadas. Este protocolo está optimizado para el análisis de proteínas de Trypanosoma brucei,pero se puede adaptar a parásitos protozoarios relacionados, levaduras o células de mamíferos.
Introducción
Los fosfatos de inositol (IPs) y los fosfoinositidos (PI) desempeñan un papel central en la biologíadel eucariota a través de la regulación de los procesos celulares como el control de la expresión génica 1,2,3, tráfico de vesículas 4, transducción de señal5,6, metabolismo7,8,9, y desarrollo8,10. La función reguladora de estos metabolitos resulta de su capacidad para interactuar con las proteínas y así regular la función proteica. Tras la unión por proteínas, las IPs y los INDICADORes de usuario pueden alterar la conformación de proteínas11,la actividad catalítica12o las interacciones13 y, por lo tanto, afectar la función celular. Las IPs y pIs se distribuyen en múltiples compartimentos subcelulares, tales como núcleo2,3,14,15, retículo endoplasmático16,17, plasma membrana1 y citosol18,ya sea asociadas a proteínas3,19 o con ARN20.
El escote de la membrana asociada a PI(4,5)P2 por fosfolipasa C da como resultado la liberación de Ins(1,4,5)P3, que puede ser fosforilado o desfosforilado por quinasas IP y fosfatasas, respectivamente. Las IPs son moléculas solubles que pueden unirse a las proteínas y ejercer funciones reguladoras. Por ejemplo, Ins(1,4,5)P3 en metazoan puede actuar como un segundo mensajero mediante la unión a los receptores IP3, que induce los cambios de conformación de receptores y por lo tanto la liberación de Ca2 + de los almacenes intracelulares11. Ins(1,3,4,5)P4 se une al complejo histona deacetilasa y regula el ensamblaje y la actividad del complejo proteico13. Otros ejemplos de la función reguladora de IPs incluyen el control de la organización de la cromatina21, el transporte de ARN22,23, la edición de ARN24y la transcripción1,2,3 . Por el contrario, los indicadores de calidad a menudo se asocian con el reclutamiento de proteínas en la membrana plasmática u membranas orgánulos25. Sin embargo, una propiedad emergente de los PE es la capacidad deasociarse con proteínas en un entorno no membranoso 3,15,19,26. Este es el caso del factor esteroideogénico del receptor nuclear, que la función de control transcripcional está regulada por PI(3,4,5)P319, y poli-A polimerasa que la actividad enzimática está regulada por PI nuclear(4,5)P226. Se ha demostrado un papel regulador para las IPs y los INDICADORes de poder en muchos organismos, entre ellos la levadura22,27, las células de mamíferos19,23, Drosophila10 y los gusanos28. De importancia es el papel de estos metabolitos en los tripanosomas, que divergieron temprano del linaje eucariota. Estos metabolitos desempeñan un papel esencial en el control transcripcional de Trypanosoma brucei 1,3, desarrollo8, biogénesis de organélteme y tráfico proteico29,30 , 31 , 32, y también participan en el control del desarrollo y la infección en los patógenos T. cruzi33,34,35,Toxoplasma36 y Plasmodium 5 , 37. Por lo tanto, comprender el papel de las iP y los indicadores de rendimiento en los tripanosomas puede ayudar a dilucidar una nueva función biológica para estas moléculas e identificar nuevos objetivos farmacológicos.
La especificidad de la proteína y la unión IP o PI depende de los dominios de interacción de proteínas y el estado de fosforilación del inositol13,38, aunque las interacciones con la parte lipídica de los indicadores de rendimiento también se producen19. La variedad de IPs y PIs y sus quinasas modificadoras y fosfaplanas proporciona un mecanismo celular flexible para controlar la función proteica que está influenciado por la disponibilidad y abundancia de metabolitos, el estado de fosforilación del inositol, y la proteína afinidad de interacción1,3,13,38. Aunque algunos dominios proteicos están bien caracterizados39,40,41, por ejemplo, dominio de homología de pleckstrina42 y SPX (SYG1/Pho81/XPR1) dominios43 ,44,45, algunas proteínas interactúan con IPs o PIs por mecanismos que permanecen desconocidos. Por ejemplo, la proteína represor-activadora 1 (RAP1) de T. brucei carece de dominios canónicos de unión PI, pero interactúa con PI(3,4,5)P3 y controla la transcripción de genes implicados en la variación antigénica3. Cromatografía de afinidad y análisis de espectrometría de masas de proteínas de interacción IP o PI a partir de células tripanosomasas, de levadura o de mamíferos identificados varias proteínas sin dominios conocidos de unión IP o PI8,46, 47. Los datos sugieren dominios proteicos adicionales no caracterizados que se unen a estos metabolitos. Por lo tanto, la identificación de proteínas que interactúan con IPs o PIs puede revelar nuevos mecanismos de interacción proteína-metabolito y nuevas funciones reguladoras celulares para estas moléculas pequeñas.
El método descrito aquí emplea cromatografía de afinidad acoplada a la hincha occidental o espectrometría de masas para identificar proteínas que se unen a iPs o PIs. Utiliza IP biotiniadas o PIs que están reticuladas a estreptavidina conjugadas con cuentas de agarosa o, alternativamente, capturadas a través de cuentas magnéticas conjugadas con estreptavidina (Figura1). El método proporciona un flujo de trabajo simple que es sensible, no radioactivo, libre de liposomas y es adecuado para detectar la unión de proteínas de los lysatos celulares o proteínas purificadas3 (Figura 2). El método se puede utilizaren proteínas de masa cuantitativa 47 sin etiquetas 8,46 o acopladas a espectrometría cuantitativa de masas con etiqueta de aminoácidos47 para identificar proteínas IP o PI-binding a partir de muestras biológicas complejas. Por lo tanto, este método es una alternativa a los pocos métodos disponibles para estudiar la interacción de IPs o PI con proteínas celulares y ayudará a entender la función reguladora de estos metabolitos en los tripanosomas y tal vez otros eucariotas.
Protocolo
1. Análisis de proteínas de unión IP o PI mediante cromatografía de afinidad y hincha occidental
- Crecimiento celular, lisis y cromatografía de afinidad
- Aumente las células de T. brucei hasta la fase de registro medio y supervise la viabilidad y densidad celular. Un total de 5.0 x 107 celdas es suficiente para un ensayo de unión.
- Para las formas del torrente sanguíneo, las células de crecimiento en medios hinmultimedia-9 complementadas conun 10% de suero bovino fetal (FBS) a 37 oC y un 5% de CO 2. Mantenga la densidad celular entre 8.0 x 105 a 1.6 x 106 celdas/mL.
NOTA: La densidad superior a 1,8 x 106 celdas/ml puede afectar a la viabilidad de las células. El tiempo de duplicación de la cepa T. brucei 427 cultivada in vitro es de entre 5,5 y 6,5 h. - Para las formas procyclicas, las células crecen en sdm-79 medio complementado con 10% FBS a 27 oC y mantener la densidad celular entre 1.0 x 107 y 3.0 x 107 células/ml.
- Para proteínas purificadas (p. ej., proteínas recombinantes), tomar de 0,5 a 1 g de proteína y diluir en 450 ml de tampón de unión (25 mM HEPES, 150 mM NaCl, 0,2% 4-nonil fenil-polietilenglicol, pH 7,4). Mantener el 5% de la proteína diluida (entrada) para el análisis de manchas occidentales. Continúe con el paso 1.1.7.
- Para las formas del torrente sanguíneo, las células de crecimiento en medios hinmultimedia-9 complementadas conun 10% de suero bovino fetal (FBS) a 37 oC y un 5% de CO 2. Mantenga la densidad celular entre 8.0 x 105 a 1.6 x 106 celdas/mL.
- Células centrífugas a 1.600 x g durante 10 min a temperatura ambiente (RT). Descarta el sobrenadante.
NOTA: Consulte el paso 2.1.2 para obtener información adicional sobre la centrifugación de grandes volúmenes de cultivo. - Resuspenda suavemente el pellet en 10 ml de pH salino tamponado de fosfato 7.4 complementado con glucosa de 6 mM (PBS-G) y precalentado a 37 oC para lavar las células. A continuación, centrifugar las células a 1.600 x g durante 5 minutos en RT. Repita el procedimiento dos veces.
- Resuspenda el pellet en 1 ml de PBS-G. A continuación, transfiera el volumen a un tubo de 1,5 ml y centrífuga a 1.600 x g durante 5 min. Deseche el sobrenadante.
NOTA: Los pellets celulares pueden congelarse en nitrógeno líquido y almacenarse a -80 oC o nitrógeno líquido. - Resuspender el pellet en 0,5 ml de tampón de lisis (25 mM HEPES, 150 mM NaCl, 1% t-octilphenoxypolyethoxyethanolethanol, pH 7.4) complementado con 1,5x cóctel inhibidor de la proteasa y 1 cóctel inhibidor de fosfatasa (Tablade materiales)preenfriado en hielo para atislar las células. Incubar el lisado durante 10 min girando a 50 rpm a 4oC.
NOTA: Este es un paso crítico porque las proteínas pueden degradarse si no se manejan como se indica. Compruebe la integridad del lisado proteico mediante electroforesis de gel de sulfato de sulfato de sodio-poliacrilamida (SDS/PAGE) si es necesario.
ADVERTENCIA: T-octylphenoxypolyethoxyethanol es tóxico y puede causar irritación de la piel y los ojos. Use guantes, protección para el parabrisas y parael parabrisas. - Centrifugar el lisado a 14.000 x g durante 10 min a 4oC. Recoja el sobrenadante en un nuevo tubo de 1,5 ml para ensayos de fijación. Mantenga el 5% del lisado total (entrada) para el análisis de hinchas occidentales. El sobrenadante contiene proteínas de parásito extraídas con tampón de lisis.
- Recoger 50 l de IPs o PIs conjugados con cuentas de agarosa (es decir, 50 l de lodo) o 50 l de perlas de agarosa, y centrifugar durante 1 min a 1.000 x g. Deseche el sobrenadante y resuspenda en 50 s de búfer de unión para equilibrar las perlas. Utilice cuentas no conjugadas como control. Utilice IP/PI-beads con diferentes configuraciones de fosfato, incluidas las formas no fosforiladas para controlar interacciones inespecíficas.
- Añadir 50 l de PERPi o PI a la célula de lisado o proteínas purificadas (cada 1 ml de perlas contiene 10 nmol de IP o PI conjugadas). Mantenga el volumen de IP- o PI-beads dentro del 10% del lisado total y, si es necesario, ajuste el volumen de reacción de unión con búfer de unión.
- Para los ensayos de competición, añada a la reacción vinculante diversas concentraciones de IP o PI no conjugadas (por ejemplo, un exceso molar de 1, 10, 10, 100 veces en comparación con las perlas IP o PI).
- Incubar la reacción durante 1 h, o durante la noche, a 4oC y girando a 50 rpm.
NOTA: Las reacciones de unión con proteínas purificadas se pueden realizar en RT dependiendo de la estabilidad de la proteína. Si el uso de direcciones IP o PI conjugados a biotina sólo proceda al paso 1.1.9.1, de lo contrario proceda al paso 1.1.10.- Añadir 50 sl de estreptavidina conjugada con perlas magnéticas a la reacción de unión e incubar durante 1 h a 4 oC girando a 50 rpm.
- Centrifugar la mezcla durante 1 min a 1.000 x g a 4oC. Retire el sobrenadante (flujo a través) y mantenga el pellet. Mantenga el 5% del sobrenadante para el análisis de manchas occidentales.
NOTA: Si utiliza perlas magnéticas, retire los sobrenadores y realice lavados posteriores con un soporte magnético (las centrifugaciones no son necesarias). - Añadir 1 ml de tampón de lavado (25 mM HEPES, 300 mM NaCl, 0,2% 4-nonil fenil-polietilenglicol, pH 7.4) y resuspender la resina tocando o arremolinando el tubo (no utilice una pipeta porque las perlas pueden unirse a las puntas de la pipeta). Centrifugar la reacción durante 1 min a 1.000 x g a 4oC y desechar el sobrenadante. Repita el procedimiento para un total de cinco lavados.
ADVERTENCIA: 4-nonyl fenil-polietilenglicol es tóxico y puede causar irritación de la piel y los ojos. Use guantes, protección para el parabrisas y parael parabrisas. - Añadir 50 l de 2x tampón De Laemmli complementado con 710 mM 2-mercaptoetanol a las perlas y mezclar tocando o vórtice para eluir las proteínas. Calentar a 95 oC durante 5 min, luego centrifugar durante 10.000 x g durante 1 min y recoger el sobrenadante (contener proteínas elutadas). Alternativamente, eluda proteínas con 8 M de urea/100 mM de pH de glicina 2.9 para evitar el uso de SDS. Congele el eluido a -80 oC, de lo contrario proceda al análisis de la mancha occidental.
ADVERTENCIA: 2-mercaptoetanol es tóxico y puede causar irritaciones de la piel, los ojos y las vías respiratorias. Use guantes y trabaje en la campana química.
- Aumente las células de T. brucei hasta la fase de registro medio y supervise la viabilidad y densidad celular. Un total de 5.0 x 107 celdas es suficiente para un ensayo de unión.
- Análisis de hinchas occidentales
- Mezclar muestras de 15 ml de entrada (del paso 1.1.6) o de flujo (del paso 1.1.10) con 5 ml de tampón De 4x Laemmli. Entrada de calor y muestras de flujo a través de 5 min a 95 oC. Para muestras eluidas en urea de 8 M/100 mM de glicina pH 2.9, mezclar 15 ml de eluido con 5 ml de tampón de laemmli de 4x y calentar durante 5 min a 95 oC.
NOTA: Este paso no es necesario para las muestras eluidas en 2 búfer laemmli. - Cargue los pozos de gel SDS/PAGE de 4-20% con 2,5 ml de entrada, flujo de 2,5 l y 20 ml de muestras elutadas, y cargue la escalera de proteínas de acuerdo con la recomendación del fabricante.
NOTA: Elija gel% según el peso molecular de la proteína de interés. - Ejecute SDS/PAGE a 150 V durante 30-45 minutos en tampón en funcionamiento, o hasta que el tinte azul del tampón Laemmli esté al final del gel.
NOTA: El tiempo de ejecución puede variar según el equipo de laboratorio. - Retire el gel de las placas de vidrio (o plástico) y remoje en el tampón de transferencia durante 15 minutos.
- Transfiera las proteínas a la membrana de difluoruro de polivinilideno (PVDF) o a la membrana de nitrocelulosa. Remoje las membranas y el papel filtrante de 3 mm en el tampón de transferencia. Ensamble un sándwich con tres hojas de papel de filtro, nitrocelulosa o membrana PVDF, gel y tres hojas adicionales de papel de filtro. Asegúrese de que no haya burbujas de aire atrapadas en el sándwich. Utilice un rodillo para eliminar las burbujas si es necesario. Ajuste la membrana en el cátodo y el gel en el lado del ánodo del cassette.
NOTA: Consulte las instrucciones del fabricante de la membrana para obtener información sobre la activación de la membrana o la preparación de manchas. - Coloque el cassette que contiene el sándwich en el tanque de transferencia con tampón de transferencia. Coloque el depósito en un cubo de hielo o a 4 oC (por ejemplo, en la cámara fría). Transfiera proteínas a 100 V durante 1 h (la corriente varía entre 200-400 mA). Alternativamente, transfiera durante la noche a una corriente constante de 15 mA a 4oC.
- Retire la membrana del cassette. Incubar la membrana en leche seca sin grasa al 6% diluida en PBS con 0,05% de polisorbato 20 (PBS-T), o solución de bloqueo compatible, durante 1 h a RT para bloquear la membrana.
NOTA: Antes de bloquear la membrana, la calidad de la transferencia se puede comprobar con la mancha de Ponceau S. Incubar la membrana durante 1 min en 15 ml de Ponceau S, enjuagar en agua y visualizar bandas. - Retire la solución de bloqueo e incubar la membrana durante 1 h a RT con rotación de 50 rpm en anticuerpos primarios diluidos en leche seca sin grasa al 6% diluida en PBS-T. Alternativamente, incubar la membrana durante la noche a 4 oC con rotación de 50 rpm.
NOTA: El tiempo de incubación puede variar según la calidad de los anticuerpos; sin embargo, la mayoría de los anticuerpos funcionarán con incubaciones de 1-3 h en RT. Siga la recomendación del fabricante para la concentración o dilución de anticuerpos. - Lavar la mancha incubando la membrana en PBS-T durante 5 min con rotación de 50 rpm en RT. Repita el procedimiento 3-5 veces. Es posible que se necesiten más lavados dependiendo de la calidad de los anticuerpos.
- Incubar la membrana en los anticuerpos secundarios conjugados por peroxidasa de rábano picante (HRP) durante 1 h a RT en 6% leche seca no grasa diluida en PBS-T con rotación de 50 rpm.
NOTA: Siga la recomendación del fabricante para la concentración o dilución de anticuerpos. - Lave la mancha como se indica en el paso 1.2.9.
- Añadir el sustrato quimioluminiscente para cubrir la membrana. Retire el exceso de sustrato e incubar durante 5 min a RT en la oscuridad.
NOTA: Consulte las instrucciones del fabricante para obtener recomendaciones sobre los reactivos de quimioluminiscencia. - Capture la señal quimioluminiscente utilizando un imager basado en cámara. Alternativamente, utilice una película de rayos X para capturar la señal quimioluminiscente.
- Mezclar muestras de 15 ml de entrada (del paso 1.1.6) o de flujo (del paso 1.1.10) con 5 ml de tampón De 4x Laemmli. Entrada de calor y muestras de flujo a través de 5 min a 95 oC. Para muestras eluidas en urea de 8 M/100 mM de glicina pH 2.9, mezclar 15 ml de eluido con 5 ml de tampón de laemmli de 4x y calentar durante 5 min a 95 oC.
2. Análisis de proteínas de unión IP/PI por cromatografía de afinidad y espectrometría de masas
- Crecimiento celular, lisis y cromatografía de afinidad
- Aumente las células de T. brucei hasta la fase de registro medio y supervise la viabilidad y densidad celular.
NOTA: Un total de 1.0 x 1010 celdas es suficiente para dos ensayos de unión. El uso de menos células de las indicadas aquí puede afectar a la detección de proteínas de baja abundancia por espectrometría de masas.- Para las formas del torrente sanguíneo de T. brucei, las células crecen en fase de registro medio (8,0 x 105-1,6 x 106 células/ml) en medios HMI-9 complementados con 10% de FBS a 37 oC y con 5% de CO2. Para la cepa 427, 5 L de cultivo producirá0.5-1.0 x 1010 células. Supervise el crecimiento celular para evitar una densidad superior a 1,8 x 106 células/ml que puede afectar la viabilidad celular. Mantenga el volumen del cultivo celular en 1/10 del volumen del matraz; de lo contrario, la tasa de crecimiento de las células se verá afectada debido a la mala aireación.
NOTA: La cepa 427 tiene un tiempo de duplicación de 5.5-6.5 h. - Para las formas procyclicas, las células crecen en sdm-79 medio complementado con 10% FBS a 27 oC y mantener la densidad celular entre 1.0 x 107 y 3.0 x 107 células/ml. 500 ml de cultivo producirán 0,5-1,5 x 1010 celdas.
- Para las formas del torrente sanguíneo de T. brucei, las células crecen en fase de registro medio (8,0 x 105-1,6 x 106 células/ml) en medios HMI-9 complementados con 10% de FBS a 37 oC y con 5% de CO2. Para la cepa 427, 5 L de cultivo producirá0.5-1.0 x 1010 células. Supervise el crecimiento celular para evitar una densidad superior a 1,8 x 106 células/ml que puede afectar la viabilidad celular. Mantenga el volumen del cultivo celular en 1/10 del volumen del matraz; de lo contrario, la tasa de crecimiento de las células se verá afectada debido a la mala aireación.
- Centrifugar las células a 1.600 x g durante 15 minutos a RT. Deseche el sobrenadante y resuspenda el pellet en 200 ml de PBS-G precalentado a 37oC. Utilice tubos de centrifugación de fondo redondo (Tablade materiales)porque los pellets de forma de flujo sanguíneo t. brucei se perturban fácilmente cuando se utilizan rotores de centrífuga de ángulo fijo. Centrifugar las células de nuevo a 1.600 x g durante 5 min en RT.
- Retire el sobrenadante y vuelva a suspender el pellet en 10 ml de PBS-G. Centrifugar las células a 1.600 x g durante 5 min en RT. Repita el procedimiento y después del lavado final, deseche el sobrenadante.
NOTA: Los pellets pueden congelarse en nitrógeno líquido y almacenarse a -80 oC o nitrógeno líquido. - Resuspender el pellet celular en 5 ml de tampón de lisis pre-enfriado en hielo y complementado con 1.5x cóctel inhibidor de la proteasa y 1cóctel inhibidor de fosfatasa (Tabla de Materiales). Incubar el lisado durante 10 min a 4oC girando a 50 rpm.
- Centrifugar el lisado a 10.000 x g durante 10 min a 4oC. Recoger el sobrenadante (proteínas solubilizadas) y diluirlo en 20 ml de tampón de unión.
- Recoger 400 l de DIRECCIONes IP o PIs conjugadas con perlas de agarosa (es decir, 400 l de lodos) o 400 l de perlas de control, y centrifugar durante 1 min a 1.000 x g en RT. Deseche el sobrenadante y vuelva a suspender en 400 oL de tampón de unión para equilibrar las perlas. Utilice cuentas de agarosa como un control negativo para determinar el enriquecimiento específico de las interacciones proteína-metabolito en comparación con interacciones inespecíficas (por ejemplo, proteínas que se unen no específicamente a las perlas). Utilice iPs o PIs con diferentes configuraciones de fosfato, incluidas formas no fosforiladas, para controlar interacciones inespecíficas debidas a cargas de fosfato o unión a la biotina.
- Añadir 400 l de IP/PI-perlas o perlas de control a 10 ml de lisado e incubados durante 1 h, o durante la noche, a 4 oC girando a 50 rpm. Si utiliza direcciones IP o PI conjugadas únicamente con biotina (sin perlas), proceda al paso 2.1.7.1, de lo contrario proceda al paso 2.1.8.
- Añadir 100 oC de estreptavidina conjugada con perlas magnéticas e incubar durante 1 h a 4 oC girando a 50 rpm.
- Centrifugar la reacción de unión durante 1 min a 1.000 x g a 4oC. Retire el sobrenadante (flujo a través) y mantenga el pellet. Mantenga el 5% del sobrenadante para el análisis de manchas occidentales.
- Añadir 5 ml de tampón de lavado al pellet, mezclar suavemente girando el tubo, y luego centrifugar durante 1 min a 1.000 x g a 4 oC. Descarta el sobrenadante. Repita el lavado cinco veces. Si utiliza perlas magnéticas, recoja los sobrenatantes y realice lavados con un soporte magnético (las centrifugaciones no son necesarias).
- Añadir 50 l de 2 x tampón De Laemmli (o 8 M de urea/100 mM de glicina pH 2.9, para evitar el uso de SDS) a las perlas y mezclar tocando o vórtice (evitar el pipeteo porque las perlas pueden unirse a las puntas de la pipeta), y luego calentar a 95 oC durante 5 min. Centrifuge durante 10.000 x g durante 1 min y recoger el sobrenadante (proteínas elutadas). Repita el procedimiento dos veces para recoger un total de tres fracciones.
- Congele el eluido a -80 oC, separen las proteínas en SDS/PAGE o mantengan en solución para la tripinización y el análisis de espectrometría de masas.
- Aumente las células de T. brucei hasta la fase de registro medio y supervise la viabilidad y densidad celular.
- Digestión de la trippsina de proteínas para espectrometría de masas
NOTA: Se muestran dos variaciones de este procedimiento para la sección 2.2.1 (en gel) o la sección 2.2.2 (en solución) de digestión de proteínas. Se recomiendan tubos de unión baja en proteínas para evitar pérdidas de muestras. Consulte con un químico analítico en la instalación proteómica sobre la idoneidad del protocolo para muestras e instrumentos de espectrómetro de masas disponibles.- Trippsinización en gel de proteínas
- Después de la separación de proteínas en SDS/PAGE, enjuague brevemente el gel en agua de alta pureza. Transfiera el gel a una placa de vidrio limpia. Exprime las bandas proteicas con una cuchilla limpia y evita cortar gel extra fuera de las bandas. Cortar las piezas de gel en trozos pequeños (es decir, aproximadamente 1 mm cuadrados) y transferirlas a un tubo de 1 ml. Utilice una punta de pipeta si es necesario, pero asegúrese de que la punta de la pipeta esté enjuagada en etanol antes de usarla.
NOTA: Use guantes para evitar la contaminación por gel. Las piezas de gel se pueden almacenar a -20 oC. - Añadir 100 l de agua de grado de cromatografía líquida de alta pureza o alto rendimiento a los tubos para enjuagar las piezas de gel. Deseche el agua.
- Para piezas de gel teñido fluorescente a base de coomassie o rutenio, incubar las piezas de gel en solución destaining (25 mM NH4HCO3 en 50% acetonitrilo) durante 1 h, luego deseche la solución. Repita el procedimiento hasta que la tinción no sea visible.
NOTA: Preparar soluciones que contengan NH4HCO3 mediante dilución a partir de una solución en stock a 100 mM NH4HCO3, pH 7,8.
ADVERTENCIA: El acetonitrilo es un disolvente volátil, inflamable y tóxico. NH4HCO3 puede causar irritación de la piel o de los ojos. Use guantes y trabaje bajo una capucha química. - Para piezas de gel teñido de plata, incubar piezas de gel en 50 l de solución de destaining para manchas de plata (15 mM K3[Fe(CN)6], 50 mM Na2S2O3) en agua durante 30 min. Deseche la solución y lave las piezas de gel con 200 ml de agua. Repita el lavado cinco veces o hasta que el color amarillo del gel no sea visible.
ADVERTENCIA: K3[Fe(CN)6] puede causar irritación de la piel o de los ojos. Usa guantes.
- Para piezas de gel teñido fluorescente a base de coomassie o rutenio, incubar las piezas de gel en solución destaining (25 mM NH4HCO3 en 50% acetonitrilo) durante 1 h, luego deseche la solución. Repita el procedimiento hasta que la tinción no sea visible.
- Deshidratar las piezas de gel usando 200 ml de acetonitrilo durante 10 min a RT. A continuación, deseche la solución.
NOTA: Las piezas de gel deshidratadas son más pequeñas en volumen, opacas y pegajosas. Si se combinan varias piezas de gel en un tubo, repita el procedimiento para la deshidratación eficiente de las piezas de gel. - Añadir 50 ml (o volumen suficiente para cubrir las piezas de gel) de la solución reductora (10 mM de ditiothreitol [TDT] en 100 mM NH4HCO3) e incubar a 56oC durante 1 h. Después, enfriar los tubos a RT y desechar el exceso de solución reductora.
- Añadir 50 l (o volumen suficiente para cubrir las piezas de gel) de la solución de alquilación (50 mM de yodoacetamida en 100 mM NH4HCO3) e incubar durante 30 minutos a RT en la oscuridad. Después, deseche el exceso de solución de alquilación.
- Deshidratar las piezas de gel con 200 ml de acetonitrilo durante 10 min a RT. Retire el acetonitrilo e hidrate las piezas de gel con 100 mM NH4HCO3 durante 10 min a RT.
- Deshidratar las piezas de gel de nuevo con 200 ml de acetonitrilo durante 10 minutos a RT y desechar el exceso de solución.
- Añadir 15 ml de tripsina de grado de espectrometría de masas diluido en un tampón NH4HCO3 de 50 mM, o un volumen suficiente para cubrir las piezas de gel hidratadas e incubar durante 4 h, o durante la noche, a 37 oC. Mantener cantidades totales de tripsina entre 100 y 500 ng (o 20 ng trypsin/g de proteína).
- Enfríe la muestra a RT y centrífuga durante 1 min a 2.000 x g en una microcentrífuga. Añadir 10-20 l de ácido fórmico al 5% diluido en agua e incubar durante 10 min a RT.
ADVERTENCIA: El ácido fórmico es inflamable, corrosivo y tóxico. Use guantes y trabaje bajo una capucha química. - Centrifugar como se indica en el paso 2.2.1.9, luego recoger el sobrenadante (contener péptidos extraídos) a un tubo diferente. Añadir 20 l de ácido fórmico 5% diluido en 50-60% acetonitrilo al tubo e incubar durante 10 minutos en RT. Recoger fracciones de péptido extraídas en el mismo tubo.
- Seque la muestra en un concentrador de vacío y reconstituya en 10 ml de ácido acético al 0,5% y 2% de acetonitrilo para el análisis de espectrometría de masas. Centrifugar y recoger la solución en la parte inferior del tubo; solución de almacenamiento a -20 oC o -80 oC.
- Después de la separación de proteínas en SDS/PAGE, enjuague brevemente el gel en agua de alta pureza. Transfiera el gel a una placa de vidrio limpia. Exprime las bandas proteicas con una cuchilla limpia y evita cortar gel extra fuera de las bandas. Cortar las piezas de gel en trozos pequeños (es decir, aproximadamente 1 mm cuadrados) y transferirlas a un tubo de 1 ml. Utilice una punta de pipeta si es necesario, pero asegúrese de que la punta de la pipeta esté enjuagada en etanol antes de usarla.
- En la solución de trippsinización de proteínas
- Precipita rinde proteínas para reducir el volumen de la muestra, la desalación y el intercambio de tampón. Añadir seis volúmenes de acetona refrigerada (-20 oC) a la muestra, por ejemplo, 600 ml de acetona a 100 s de muestra. Vórtice e incubar a -20oC durante 15 min a 1 h. La solución se volverá turbia o formará un precipitado.
ADVERTENCIA: La acetona es tóxica e inflamable. Use guantes y trabaje bajo una capucha química. - Muestras de centrífuga a 4oC durante 30 min. Decanta la acetona y seca al aire el pellet durante 15 min.
- Añadir 10 ml de urea de 6-8 M o 1% de SDS en 50 mM NH4HCO3 para disolver el pellet y el vórtice para mezclar. Para cantidades mayores de proteínas (> 10 g), utilice hasta 20 ml de tampón de disolución.
- Añadir 5 l de solución reductora y vórtice. Gire el volumen hacia abajo con una microcentrífuga. Si las muestras se diluyen en urea, incubar durante 1 h a RT. Si las muestras se diluyen en SDS, se incuban durante 1 h a 56 oC.
- Gire el volumen hacia abajo con una microcentrífuga. Añadir 3 l de solución de alquilación y vórtice. Luego, gire el volumen hacia abajo e incubar en la oscuridad durante 30 minutos en RT.
- Añadir 3 l de solución reductora para neutralizar la reacción. Diluir lentamente la muestra a 1 M de urea o 0,05% DeS con 50 mM NH4HCO3.
NOTA: El tampón de digestión de trippsina debe tener detergente o desnaturalizante. Los límites de concentración para desnaturalizantes son: 0.05% SDS; 0.1% octyl B-D-glucopyranoside; 0.1% 4-nonilfenilo-polietilenglicol; 0.1% t-octylphenoxypolyethoxyethanol; 0.1% polisorbato 20; 0.1% 3-[(3-cholamidopropyl)dimethylammonio]-1-propanesulfonato; < 1 M de urea o tiourea. - Añadir 5 l de tripsina de grado de espectrometría de masas diluido en 50 mM NH4HCO3 tampón e incubar durante 4 h, o durante la noche, a 37 oC. Mantener cantidades totales de tripsina entre 100 y 500 ng (o 20 ng trypsin/g de proteína).
- Enfríe la muestra a RT y gire el volumen hacia abajo con una microcentrífuga. Añadir 5% ácido acético (o 5% ácido fórmico en 50% acetonitrilo) para saciar la reacción.
- Seque las muestras en un concentrador de vacío como se indica en el paso 2.2.1.11. Almacene las muestras a -80 oC. Desalt y concentra péptidos usando una columna de fase invertida como C18 zip-tip y luego analizar por espectrometría de masas.
- Precipita rinde proteínas para reducir el volumen de la muestra, la desalación y el intercambio de tampón. Añadir seis volúmenes de acetona refrigerada (-20 oC) a la muestra, por ejemplo, 600 ml de acetona a 100 s de muestra. Vórtice e incubar a -20oC durante 15 min a 1 h. La solución se volverá turbia o formará un precipitado.
- Trippsinización en gel de proteínas
Resultados
Análisis de la interacción RAP1 y PI(3,4,5)P3 por cromatografía de afinidad y hincha occidental
Este ejemplo ilustra la aplicación de este método para analizar la unión de PIs por RAP1 de T. brucei lysate o por proteína recombinante T. brucei RAP1. Los lysates de T. brucei formas del torrente sanguíneo que expresan rap1 con etiqueta de hemaglutinina (HA) se utilizaron en ensayos de unión. RAP1 es una proteína implicada en el control transcripcional de genes de glicoproteína superficial variante (VSG)3,48, que codifican para las proteínas superficiales implicadas en la evasión inmune de parásitos por variación antigénica49. RAP1 interactúa dentro de un complejo proteico telomérico con la enzima fosfatidilinositol 5-fosfatasa (PIP5Pase)3, que también funciona en el control de la transcripción del gen VSG1,3. RAP1 tiene un dominio de n-terminal de cáncer de mama 1 carboxilo-terminal (BRCT) que es seguido por un dominio de unión al ADN mieloblastosis (myb) y un dominio similar a mib C-terminal3,48. Sin embargo, carece de dominios de enlace de PI canónicos. Los ensayos vinculantes se realizaron con PIs que no son fosforilados o están fosforilados en diferentes posiciones del anillo de inositol, y con cuentas de agarosa no conjugadas. El análisis occidental muestra que el RAP1 se une preferentemente a las cuentas PI(3,4,5)P3 (figura3A),pero también se vincula en menor medida a las cuentas PI(4,5)P2. Sin embargo, no se vinculó a ningún otro PIs o cuentas de agarosa. Debido a que RAP1 es parte de un complejo multiproteico3, su interacción con algunos PIs puede no ser directa y, por lo tanto, es el resultado de la interacción RAP1-HA con otras proteínas celulares que se unen a los PI.
Por lo tanto, para comprobar si RAP1 se une directamente a los PI, se expresó y purificó una proteína RAP1 recombinante (rRAP1) con etiqueta C-terminal 6x-his a partir de la homogeneidad de E. coli3. La proteína se utilizó en ensayos de unión con PERPI(3,4,5)P3-beads en presencia de concentraciones competidoras de PI(3,4,5)P3 o PI(4,5)P2. La hincha occidental muestra que el aumento de las concentraciones de PI(3,4,5)P3, pero no PI(4,5)P2 inhibe la interacción de rRAP1 con PI(3,4,5)P3 (Figura3B). Por otra parte, la adición de la enzima PIP5Pase purificada de T. brucei a la reacción restaurada PI(3,4,5)P3-unión por rRAP1, que se debe a la defosforilación PIP5Pase de PI libre PI(3,4,5)P33 y por lo tanto indica que el patrón de fosforilación de este metabolito es esencial para la unión de rRAP1. Por lo tanto, rRAP1 interactúa con PI(3,4,5)P3 como lo hace RAP1-HA de T. brucei lysates. Además, los datos muestran que la unión de RAP1-HA de lisado a PI(4,5)P2 probablemente resulta de la interacción rap1 con otras proteínas en el complejo (por ejemplo, PIP5Pase)3. Los datos ilustran la complementariedad de los ensayos de unión con los lysatos celulares y las proteínas recombinantes. También muestra la utilidad de los ensayos de unión competitivos para determinar la especificidad de las interacciones entre proteínas y pIS.
Identificación de proteínas de unión Ins(1,4,5)P3 por cromatografía de afinidad y espectrometría de masas
En este ejemplo, se utilizó cromatografía de afinidad seguida de espectrometría de masas para identificar proteínas T. brucei que se unen a Ins(1,4,5)P3; por lo tanto, el experimento examina las posibles proteínas de unión Ins(1,4,5)P3 de las formas del torrente sanguíneo de T. brucei. T. brucei lysate fue incubado con Ins(1,4,5)P3 conjugado a cuentas de agarosa o con cuentas no conjugadas (utilizadas como control), y las proteínas unidas se eluyó con tampón de muestra de Laemmli. El análisis SDS/PAGE muestra el enriquecimiento de proteínas eluyadas a partir de perlas Ins(1,4,5)P3 en comparación con las proteínas eluted de las perlas de agarosa de control (Figura4A). Análisis de espectrometría de masas de proteínas elutadas identificadas sobre más de 250 proteínas, de las cuales 84 se enriquecieron con perlas Ins(1,4,5)P3 en comparación con las cuentas de control (Figura4B,cambio de pliegue [FC] a 2, p < 0,05). El enriquecimiento de proteínas unidas a Ins(1,4,5)P3 en comparación con las perlas de control se correlaciona con la señal proteica detectada por SDS/PAGE. Los datos incluyen proteínas que fueron validadas para unir Ins(1,4,5)P3 y proteínas cuyos mecanismosde unión a Ins(1,4,5)P3 son desconocidos 8. Además, el proteoma de unión Ins(1,4,5)P3 difería en gran medida del de Ins(1,3,4,5)P4 y otros PIs8, lo que sugiere que algunas de estas proteínas reconocen la configuración específica de fosfato de Ins(1,4,5)P3. Por lo tanto, Ins etiquetado con biotina(1,4,5)P3 se puede utilizar para cromatografía de afinidad acoplada a espectrometría de masas para identificar proteínas que se unen a Ins(1,4,5)P3. El enfoque se puede explorar para identificar proteínas que se unen a otras IPs o PIs3,8,46,47.

Figura 1 : Reactivos de afinidad para ensayos de unión. (A) PI(3,4,5)P3 (arriba) e Ins(1,4,5)P3 (abajo) conjugado a la biotina en la posición sn1 del inositol. En PI(3,4,5)P3, la biotina se conjuga con la cadena lipídica en la posición sn1 del inositol, mientras que en Ins(1,4,5)P3 la biotina se conjuga con el fosfato en la posición sn1. (B) Ins(1,4,5)P3 se conjuga con biotina y se captura mediante unión a estreptavidina conjugada en cuentas (por ejemplo, agarosa o cuentas magnéticas). Las variaciones de estos reactivos utilizando enlaces sintetizados personalizados que sustituyen a la biotina también son posibles46. Haga clic aquí para ver una versión más grande de esta figura.
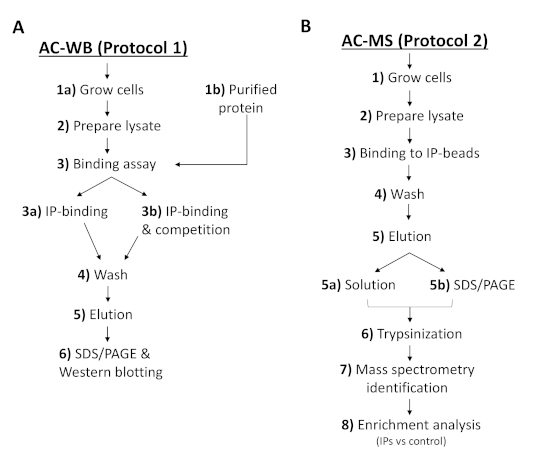
Figura 2 : El flujo de trabajo de los protocolos describe los pasos para el análisis de la interacción de afinidad IP o PI con proteínas de T. brucei y la detección por (A) hinchamiento occidental o (B) espectrometría de masas. AC-WB, cromatografía de afinidad y hincha occidental; AC-MS, cromatografía de afinidad y espectrometría de masas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Unión de T. brucei RAP1 a fosfoinositidos. (A) Los lysates de T. brucei (5.0 x 107 parásitos) que expresan RAP1 con etiqueta HA fueron incubados durante 2 h a 4 oC con 50 oC de PIs (cada uno 1 ml de perlas de agarosa que contienen 10 nmol de PIT conjugados) o perlas de agarosa (Ag). La reacción de unión se lavó y eluyó con 2 tampón de muestra de Laemmli y se calentó a 95 oC durante 5 min. Las proteínas se separaron en 4-20% de SDS/PAGE, se transfirieron a una membrana PVDF y se sondearon con anticuerpos monoclonales anti-HA (1:5.000, diluidos en 6% de pbstéo) seguidos en 6% de leche PBS) seguidos por IgG-HRP anti-ratón (1:5,000, diluido en 6% PBS-leche), y detectado por quimioluminiscencia. (B) Se incubaron durante 1 h una g de rRAP1 a RT con 50 ml de PERlas de PI(3,4,5)P3-agarose en presencia o ausencia de dioctanoylglycerol de 5 a 50 m (diC8) PI(3,4,5)P3, 20 a 50 m diC8 PI(4,5)P2, o 50 éM diC8 PI(3,4,5)P3 y 250 ng PIP5Pase purificado de T. brucei formas del torrente sanguíneo3. La unión fue analizada por la hinchada occidental con anticuerpos monoclonales anti-His HRP de ratón (1:2.000, diluido en 6% de leche PBS) y desarrollado por quimioluminiscencia. Esta cifra ha sido modificada de Cestari et al.3. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Cromatografía de afinidad y análisis de espectrometría de masas de proteínas T. brucei que se unen a Ins(1,4,5)P3. (A) 10% Análisis SDS/PAGE de proteínas T. brucei que se unen a Perlas Ins(1,4,5)P3 o perlas de agarosa. Los lysates de 5,0 x 109 parásitos se incubaron a 4 oC durante 2 h con 400 oC de Ins(1,4,5)P3 conjugados con perlas de agarosa o con perlas de agarosa sin Ins(1,4,5)P3 [1 mL de perlas contiene 10 nmol de Ins conjugados(1,4,5)P3]. La reacción de unión se lavó, se eluyó en 2 x tampón de muestra de Laemmli y se hierve durante 5 min a 95 oC. Las proteínas se separaron en 10% de SDS/PAGE y se tiñieron con tinción de coomassie (Tablade materiales). Las puntas de flecha muestran proteínas que se enriquecen en Ins(1,4,5)P3-beads en comparación con las perlas de agarosa; los círculos indican proteínas presentes tanto en Ins(1,4,5)P3-perlas y cuentas de agarosa, y el soporte indica proteínas que están presentes en ambos pero están enriquecidas en Ins(1,4,5)P3-perlas en comparación con las perlas de agarosa. (B) La gráfica de puntos muestra proteínas identificadas por espectrometría de masas que se enriquecen en Perlas Ins(1,4,5)P3 en comparación con las perlas de agarosa. Enriquecimiento definido por FC > 2 y valor p-<0.05. Se utilizaron cuatro réplicas biológicas para agarose-beads AC-MS, y se calculó tres réplicas biológicas para IP3-beads AC-MS. El cambio de proteínas identificado en IP3-perlas vs agarose-perlas se calculó utilizando la intensidad de espectros de péptidos utilizando MSstat50. Los resultados detallados y la lista de péptidos están disponibles8. Los datos sin procesar de espectrometría de masas también están disponibles con el identificador PXD005907 a través del Consorcio ProteomeXchange a través del repositorio de socios PRIDE. Esta cifra ha sido modificada de Cestari et al.8. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La identificación de proteínas que se unen a iPs o PIs es fundamental para entender la función celular de estos metabolitos. La cromatografía de afinidad acoplada a la mancha occidental o a la espectrometría de masas ofrece la oportunidad de identificar proteínas que interactúan con IP o PI y, por lo tanto, obtener información sobre su función reguladora. IPs o PIs químicamente etiquetados [por ejemplo, Ins(1,4,5)P3 químicamente vinculados a la biotina] y reticulados a cuentas agarose a través de estreptavidina o capturados por perlas magnéticas de estreptavidina permite el aislamiento de proteínas interactuantes que luego pueden ser identificadas por masa espectrometría o mancha occidental. Los protocolos descritos aquí se han utilizado para identificar proteínas de T. brucei3,8 y células de mamíferos47 que se unen a estos metabolitos. Variaciones del enfoque que utiliza etiquetas personalizadas (distintas de la biotina) también se han utilizado en la levadura46. Una consideración importante a este enfoque es el uso de controles para discriminar interacciones específicas de interacciones no específicas. Las cuentas no conjugadas son un control esencial, pero los controles adicionales pueden incluir pIs no fosforilados o inositol conjugados a cuentas3. IPs o PIs con diferentes combinaciones de fosfato3,8 también se pueden utilizar porque la unión de proteínas a estos metabolitos puede implicar dominios que discriminan la configuración de fosfato del inositol38, 39,40,41. Además, la complejidad de la muestra puede afectar la sensibilidad del enfoque, y por lo tanto disminuir la complejidad de la muestra por fraccionamiento de la muestra puede ayudar a la detección de proteínas de bajo abundante en la célula. Existen protocolos bien establecidos para el fraccionamiento celular y el aislamiento de mitocondrion51, núcleo52,glucosoma53y flagelo54,55 de los tripanosomas. Tenga en cuenta que es posible que sea necesario ajustar los búferes y reactivos utilizados en los fraccionamientos subcelulares para que sean de compatibilidad con búferes y reactivos utilizados en este protocolo. En particular, la identificación de proteínas que se unen a las IP o PI mediante cromatografía de afinidad como se indica aquí depende de la afinidad de interacción con proteínas y metabolitos, y por lo tanto las proteínas que tienen una afinidad débil por las IP o los indicadores de usuario pueden no detectarse fácilmente.
El análisis de la interacción IP o PI con proteínas del lisato celular también puede dar lugar a la identificación de proteínas que no se unen directamente a estos metabolitos, pero que la interacción resulta de la asociación de proteínas en complejo con otras proteínas que se unen a las IP o PIs. Esta característica se ejemplifica en la Figura 3A, en la que RAP1-HA de T. brucei lysate parece enlazarse a PI(3,4,5)P3-beads y PI(4,5)P2-beads. Sin embargo, los ensayos de unión con RRAP1 con etiqueta His muestran que esta proteína se une a PI(3,4,5)P3 y no pi(4,5)P23. Esto se ilustra en la Figura 3B, en la que los ensayos competitivos muestran que PI(3,4,5)P3 gratuito pero no PI(4,5)P2 libre compite por rRAP1-su interacción con PI(3,4,5)P3-beads. La interacción aparente rap1 con PI(4,5)P2-beads se debe a la asociación RAP1 dentro de un complejo con proteínas que unen PI(4,5)P2 (por ejemplo, PIP5Pase)3. Por lo tanto, los ensayos de unión de los lysaatos celulares pueden identificar proteínas que se unen directa o indirectamente a iPs o PIs. En particular, las interacciones indirectas son distintas de las interacciones inespecíficas, ya que las primeras pueden tener una función biológica (en el contexto del complejo proteico) que afecta o se ve afectada por la unión a metabolitos. Por ejemplo, Ins(1,4,5,6)P4 se une al complejo de deacetilasa co-represis multi-subunidad y controla el complejo complejo complejo de ensamblaje y actividad13. Por lo tanto, es esencial validar las interacciones IP/PI con proteínas. La validación de la interacción puede implicar ensayos de competencia con un exceso de IPs o PIs (como en la Figura 3B)3,8, mutaciones de dominios proteicos potenciales56,o el uso de proteínas purificadas para determinar las interacciones directas (Figura3A,B)3.
Otros métodos para estudiar las interacciones de proteínas e IP o PI incluyen la unión de proteínas a IP o PI radiomarcados, el uso de IP o PI unidos a membranas hidrófobas como matrices para la captura de proteínas, o la unión de proteínas a PI incorporadas en liposomas 42 , 57 , 58. Es importante que, si la interacción proteica con los IP requiere estructuras de membrana38, los ensayos a base de liposomas pueden utilizarse como un enfoque complementario. Las limitaciones de estos enfoques incluyen bajo rendimiento, baja sensibilidad, la orientación química desconocida de IPs o la asociación de Indicadores de Rendimiento a matrices58,o el uso de materiales radiactivos42. El método descrito aquí es sensible, sin liposomas, no radioactivo, y IP / PI-perlas están disponibles comercialmente, y por lo tanto no requieren síntesis química personalizada. Además, la posición de la biotina vinculada a IPs o IP está bien definida, y también se puede modificar46,58, lo que permite un análisis preciso de la interacción proteica y metabolito. El método descrito aquí también se puede combinar con enfoques cuantitativos de espectrometría de masas, como el etiquetado estable de isótopos de aminoácidos en el cultivo celular (SILAC)47,que se pueden utilizar para identificar interacciones dinámicas bajo diferentes tratamientos o afecciones. La cromatografía de afinidad acoplada a la mancha occidental o a la espectrometría de masas ha ayudado a identificar numerosas proteínas de unión IP o PI de T. brucei, células de mamíferos y levadura3,8,46, 47, incluidas las proteínas que no tienen dominios de unión IP o PI, y también ha ayudado a la identificación de nuevos dominios de unión, por ejemplo, PI(3,4,5)P3 dominio de unión47.
En general, los protocolos descritos aquí se pueden utilizar para examinar posibles proteínas de interacción IP o PI de T. brucei,y para estudiar la interacción molecular de las proteínas con estos metabolitos. El protocolo se puede adaptar fácilmente para identificar proteínas ip o PI de otros parásitos unicelulares o de otros organismos como las células de mamíferos47 y la levadura46,y ayudará a comprender mejor la función biológica de las IP y Pi en eucariotas.
Divulgaciones
El autor no tiene nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el Consejo de Investigación en Ciencias Naturales e Ingeniería de Canadá (NSERC, RGPIN-2019-04658); NSERC Discovery Launch Supplement for Early Career Researchers (DGECR-2019-00081) y por McGill University.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetone | Sigma-Aldrich | 650501 | Ketone |
| Acetonitrile | Sigma-Aldrich | 271004 | Solvent |
| Ammonium bicarbonate | Sigma-Aldrich | A6141 | Inorganic salt |
| Centrifuge Avanti J6-MI | Beckman Coulter | Avanti J6-MI | Centrifuge for large volumes (e.g., 1L) |
| Centrifuge botles | Sigma-Aldrich | B1408 | Bottles for centrifugation of 1L of culture |
| Control Beads | Echelon | P-B000-1ml | Affinity chromatography reagent - control |
| D-(+)-Glucose | Sigma-Aldrich | G8270 | Sugar, Added in PBS to keep cells viable |
| Dithiothreitol (DTT) | Bio-Rad | 1610610 | Reducing agent |
| Dynabeads M-270 Streptavidin | ThermoFisher Scientific | 65305 | Streptavidin beads for binding to biotin ligands |
| EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitors |
| Electrophoresis running buffer | Bio-Rad | 1610732 | 25 mM Tris, 192 mM glycine, 0.1% SDS, pH 8.3 |
| Falcon 15 mL Conical Centrifuge Tubes | Corning Life Sciences | 430052 | To centrifuge 10 mL cultures |
| Formic acid | Sigma-Aldrich | 106526 | Acid |
| Glycine | Sigma-Aldrich | G7126 | Amino acid |
| HMI-9 cell culture medium | ThermoFisher Scientific | ME110145P1 | Cell culture medium for T. brucei bloodstream forms |
| Imperial Protein Stain | ThermoFisher Scientific | 24615 | Coomassie staining for protein detection in SDS/PAGE |
| Ins(1,4,5)P3 Beads | Echelon | Q-B0145-1ml | Affinity chromatography reagent |
| Instant Nonfat Dry Milk | Thomas Scientific | C837M64 | Blocking reagent for Western blotting |
| Iodoacetamide | Sigma-Aldrich | I6125 | Alkylating reagent for cysteine proteins or peptides |
| Lab Rotator | Thomas Scientific | 1159Z92 | For binding assays |
| LoBind Microcentrifuge Tubes | ThermoFisher Scientific | 13-698-793 | Low protein binding tubes for mass spectrometry |
| Nonidet P-40 (Igepal CA-630) | Sigma-Aldrich | 21-3277 | Detergent |
| PBS, pH 7.4 | ThermoFisher Scientific | 10010031 | Physiological buffer |
| Peroxidase substrate for chemiluminescence | ThermoFisher Scientific | 32106 | Substrate for Western bloting detection of proteins |
| PhosSTOP Phosphatase Inhibitor Cocktail Tablets | Roche | 4906845001 | Phosphatase inhibitors |
| PI(3)P PIP Beads | Echelon | P-B003a-1ml | Affinity chromatography reagent |
| PI(3,4)P2 PIP Beads | Echelon | P-B034a-1ml | Affinity chromatography reagent |
| PI(3,4,5)P3 diC8 | Echelon | P-3908-1mg | Affinity chromatography reagent |
| PI(3,4,5)P3 PIP Beads | Echelon | P-B345a-1ml | Affinity chromatography reagent |
| PI(3,5)P2 PIP Beads | Echelon | P-B035a-1ml | Affinity chromatography reagent |
| PI(4)P PIP Beads | Echelon | P-B004a-1ml | Affinity chromatography reagent |
| PI(4,5)P2 diC8 | Echelon | P-4508-1mg | Affinity chromatography reagent |
| PI(4,5)P2 PIP Beads | Echelon | P-B045a-1ml | Affinity chromatography reagent |
| PI(5)P PIP Beads | Echelon | P-B005a-1ml | Affinity chromatography reagent |
| Ponceau S solution | Sigma-Aldrich | P7170 | Protein staining (0.1% [w/v] in 5% acetic acid) |
| Potassium hexacyanoferrate(III) | Sigma-Aldrich | 702587 | Potassium salt |
| PtdIns PIP Beads | Echelon | P-B001-1ml | Affinity chromatography reagent |
| PVDF Membrane | Bio-Rad | 1620177 | For Western blotting |
| Refrigerated centrifuge | Eppendorf | 5910 R | Microcentrifuge for small volumes (e.g., 1.5 mL) |
| Sodium dodecyl sulfate | Sigma-Aldrich | 862010 | Detergent |
| Sodium thiosulfate | Sigma-Aldrich | 72049 | Chemical |
| SpeedVac Vacuum Concentrators | ThermoFisher Scientific | SPD120-115 | Sample concentration (e.g., for mass spectrometry) |
| T175 flasks for cell culture | ThermoFisher Scientific | 159910 | To grow 50 mL T. brucei culture |
| Trypsin, Mass Spectrometry Grade | Promega | V5280 | Trypsin for protein digestion |
| Urea | Sigma-Aldrich | U5128 | Denaturing reagent |
| Vortex | Fisher Scientific | 02-215-418 | For mixing reactions |
| Western blotting transfer buffer | Bio-Rad | 1610734 | 25 mM Tris, 192 mM glycine, pH 8.3 with 20% methanol |
| Whatman 3 mm paper | Sigma-Aldrich | WHA3030861 | Paper for Wester transfer |
| 2-mercaptoethanol (14.2 M) | Bio-Rad | 1610710 | Reducing agent |
| 2x Laemmli Sample Buffer | Bio-Rad | 161-0737 | Protein loading buffer |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels | Bio-Rad | 4561094 | Gel for protein electrophoresis |
| 4x Laemmli Sample Buffer | Bio-Rad | 161-0747 | Protein loading buffer |
Referencias
- Cestari, I., Stuart, K. Inositol phosphate pathway controls transcription of telomeric expression sites in trypanosomes. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2803-2812 (2015).
- Odom, A. R., Stahlberg, A., Wente, S. R., York, J. D. A role for nuclear inositol 1,4,5-trisphosphate kinase in transcriptional control. Science. 287 (5460), 2026-2029 (2000).
- Cestari, I., McLeland-Wieser, H., Stuart, K. Nuclear Phosphatidylinositol 5-Phosphatase Is Essential for Allelic Exclusion of Variant Surface Glycoprotein Genes in Trypanosomes. Molecular and Cellular Biology. 39 (3), (2019).
- Billcliff, P. G., et al. OCRL1 engages with the F-BAR protein pacsin 2 to promote biogenesis of membrane-trafficking intermediates. Molecular Biology of the Cell. 27 (1), 90-107 (2016).
- Brochet, M., et al. Phosphoinositide metabolism links cGMP-dependent protein kinase G to essential Ca(2)(+) signals at key decision points in the life cycle of malaria parasites. PLoS Biology. 12 (3), 1001806(2014).
- Azevedo, C., Szijgyarto, Z., Saiardi, A. The signaling role of inositol hexakisphosphate kinases (IP6Ks). Advances in Enzyme Regulation. 51 (1), 74-82 (2011).
- Szijgyarto, Z., Garedew, A., Azevedo, C., Saiardi, A. Influence of inositol pyrophosphates on cellular energy dynamics. Science. 334 (6057), 802-805 (2011).
- Cestari, I., Anupama, A., Stuart, K. Inositol polyphosphate multikinase regulation of Trypanosoma brucei life stage development. Molecular Biology of the Cell. 29 (9), 1137-1152 (2018).
- Bang, S., et al. AMP-activated protein kinase is physiologically regulated by inositol polyphosphate multikinase. Proceedings of the National Academy of Sciences of the United States of America. 109 (2), 616-620 (2012).
- Seeds, A. M., Tsui, M. M., Sunu, C., Spana, E. P., York, J. D. Inositol phosphate kinase 2 is required for imaginal disc development in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 112 (51), 15660-15665 (2015).
- Hamada, K., Miyatake, H., Terauchi, A., Mikoshiba, K. IP3-mediated gating mechanism of the IP3 receptor revealed by mutagenesis and X-ray crystallography. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), 4661-4666 (2017).
- Watson, P. J., et al. Insights into the activation mechanism of class I HDAC complexes by inositol phosphates. Nature Communications. 7, 11262(2016).
- Watson, P. J., Fairall, L., Santos, G. M., Schwabe, J. W. Structure of HDAC3 bound to co-repressor and inositol tetraphosphate. Nature. 481 (7381), 335-340 (2012).
- Irvine, R. F. Nuclear lipid signalling. Nature Reviews: Molecular Cell Biology. 4 (5), 349-360 (2003).
- Sobol, M., et al. UBF complexes with phosphatidylinositol 4,5-bisphosphate in nucleolar organizer regions regardless of ongoing RNA polymerase I activity. Nucleus. 4 (6), 478-486 (2013).
- Nascimbeni, A. C., et al. ER-plasma membrane contact sites contribute to autophagosome biogenesis by regulation of local PI3P synthesis. EMBO Journal. 36 (14), 2018-2033 (2017).
- Martin, K. L., Smith, T. K. The myo-inositol-1-phosphate synthase gene is essential in Trypanosoma brucei. Biochemical Society Transactions. 33, Pt 5 983-985 (2005).
- Matsu-ura, T., et al. Cytosolic inositol 1,4,5-trisphosphate dynamics during intracellular calcium oscillations in living cells. Journal of Cell Biology. 173 (5), 755-765 (2006).
- Blind, R. D., et al. The signaling phospholipid PIP3 creates a new interaction surface on the nuclear receptor SF-1. Proceedings of the National Academy of Sciences of the United States of America. 111 (42), 15054-15059 (2014).
- Lin, A., et al. The LINK-A lncRNA interacts with PtdIns(3,4,5)P3 to hyperactivate AKT and confer resistance to AKT inhibitors. Nature Cell Biology. 19 (3), 238-251 (2017).
- Steger, D. J., Haswell, E. S., Miller, A. L., Wente, S. R., O'Shea, E. K. Regulation of chromatin remodeling by inositol polyphosphates. Science. 299 (5603), 114-116 (2003).
- York, J. D., Odom, A. R., Murphy, R., Ives, E. B., Wente, S. R. A phospholipase C-dependent inositol polyphosphate kinase pathway required for efficient messenger RNA export. Science. 285 (5424), 96-100 (1999).
- Adams, R. L., Mason, A. C., Glass, L., Aditi,, Wente, S. R. Nup42 and IP6 coordinate Gle1 stimulation of Dbp5/DDX19B for mRNA export in yeast and human cells. Traffic. 18 (12), 776-790 (2017).
- Macbeth, M. R., et al. Inositol hexakisphosphate is bound in the ADAR2 core and required for RNA editing. Science. 309 (5740), 1534-1539 (2005).
- Dickson, E. J., Hille, B. Understanding phosphoinositides: rare, dynamic, and essential membrane phospholipids. Biochemical Journal. 476 (1), 1-23 (2019).
- Mellman, D. L., et al. A PtdIns4,5P2-regulated nuclear poly(A) polymerase controls expression of select mRNAs. Nature. 451 (7181), 1013-1017 (2008).
- Lee, Y. S., Mulugu, S., York, J. D., O'Shea, E. K. Regulation of a cyclin-CDK-CDK inhibitor complex by inositol pyrophosphates. Science. 316 (5821), 109-112 (2007).
- Nagy, A. I., et al. IP3 signalling regulates exogenous RNAi in Caenorhabditis elegans. EMBO Reports. 16 (3), 341-350 (2015).
- Hall, B. S., et al. TbVps34, the trypanosome orthologue of Vps34, is required for Golgi complex segregation. Journal of Biological Chemistry. 281 (37), 27600-27612 (2006).
- Rodgers, M. J., Albanesi, J. P., Phillips, M. A. Phosphatidylinositol 4-kinase III-beta is required for Golgi maintenance and cytokinesis in Trypanosoma brucei. Eukaryotic Cell. 6 (7), 1108-1118 (2007).
- Gilden, J. K., et al. The role of the PI(3,5)P2 kinase TbFab1 in endo/lysosomal trafficking in Trypanosoma brucei. Molecular and Biochemical Parasitology. 214, 52-61 (2017).
- Demmel, L., et al. The endocytic activity of the flagellar pocket in Trypanosoma brucei is regulated by an adjacent phosphatidylinositol phosphate kinase. Journal of Cell Science. 129 (11), 2285(2016).
- Gimenez, A. M., et al. Phosphatidylinositol kinase activities in Trypanosoma cruzi epimastigotes. Molecular and Biochemical Parasitology. 203 (1-2), 14-24 (2015).
- Hashimoto, M., et al. Inositol 1,4,5-trisphosphate receptor regulates replication, differentiation, infectivity and virulence of the parasitic protist Trypanosoma cruzi. Molecular Microbiology. 87 (6), 1133-1150 (2013).
- Cestari, I., Haas, P., Moretti, N. S., Schenkman, S., Stuart, K. Chemogenetic Characterization of Inositol Phosphate Metabolic Pathway Reveals Druggable Enzymes for Targeting Kinetoplastid Parasites. Cell Chemical Biology. 23 (5), 608-617 (2016).
- Lovett, J. L., Marchesini, N., Moreno, S. N., Sibley, L. D. Toxoplasma gondii microneme secretion involves intracellular Ca(2+) release from inositol 1,4,5-triphosphate (IP(3))/ryanodine-sensitive stores. Journal of Biological Chemistry. 277 (29), 25870-25876 (2002).
- McNamara, C. W., et al. Targeting Plasmodium PI(4)K to eliminate malaria. Nature. 504 (7479), 248-253 (2013).
- Tresaugues, L., et al. Structural basis for phosphoinositide substrate recognition, catalysis, and membrane interactions in human inositol polyphosphate 5-phosphatases. Structure. 22 (5), 744-755 (2014).
- Varnai, P., et al. Quantifying lipid changes in various membrane compartments using lipid binding protein domains. Cell Calcium. 64, 72-82 (2017).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Letters. 590 (15), 2454-2468 (2016).
- Cullen, P. J., Cozier, G. E., Banting, G., Mellor, H. Modular phosphoinositide-binding domains--their role in signalling and membrane trafficking. Current Biology. 11 (21), 882-893 (2001).
- Klarlund, J. K., et al. Signaling by phosphoinositide-3,4,5-trisphosphate through proteins containing pleckstrin and Sec7 homology domains. Science. 275 (5308), 1927-1930 (1997).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Gerasimaite, R., et al. Inositol Pyrophosphate Specificity of the SPX-Dependent Polyphosphate Polymerase VTC. ACS Chemical Biology. 12 (3), 648-653 (2017).
- Potapenko, E., et al. 5-Diphosphoinositol pentakisphosphate (5-IP7) regulates phosphate release from acidocalcisomes and yeast vacuoles. Journal of Biological Chemistry. 293 (49), 19101-19112 (2018).
- Wu, M., Chong, L. S., Perlman, D. H., Resnick, A. C., Fiedler, D. Inositol polyphosphates intersect with signaling and metabolic networks via two distinct mechanisms. Proceedings of the National Academy of Sciences of the United States of America. 113 (44), 6757-6765 (2016).
- Jungmichel, S., et al. Specificity and commonality of the phosphoinositide-binding proteome analyzed by quantitative mass spectrometry. Cell Reports. 6 (3), 578-591 (2014).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137 (1), 99-109 (2009).
- Cestari, I., Stuart, K. Transcriptional Regulation of Telomeric Expression Sites and Antigenic Variation in Trypanosomes. Current Genomics. 19 (2), 119-132 (2018).
- Clough, T., Thaminy, S., Ragg, S., Aebersold, R., Vitek, O. Statistical protein quantification and significance analysis in label-free LC-MS experiments with complex designs. BMC Bioinformatics. 13, Suppl 16 6(2012).
- Schneider, A., Charriere, F., Pusnik, M., Horn, E. K. Isolation of mitochondria from procyclic Trypanosoma brucei. Methods in Molecular Biology. 372, 67-80 (2007).
- DeGrasse, J. A., Chait, B. T., Field, M. C., Rout, M. P. High-yield isolation and subcellular proteomic characterization of nuclear and subnuclear structures from trypanosomes. Methods in Molecular Biology. 463, 77-92 (2008).
- Opperdoes, F. R. A rapid method for the isolation of intact glycosomes from Trypanosoma brucei by Percoll -gradient centrifugation in a vertical rotor. Molecular and Biochemical Parasitology. 3 (3), 181-186 (1981).
- Pereira, N. M., de Souza, W., Machado, R. D., de Castro, F. T. Isolation and properties of flagella of trypanosomatids. The Journal of protozoology. 24 (4), 511-514 (1977).
- Subota, I., et al. Proteomic analysis of intact flagella of procyclic Trypanosoma brucei cells identifies novel flagellar proteins with unique sub-localization and dynamics. Molecular and Cellular Proteomics. 13 (7), 1769-1786 (2014).
- Fukuda, M., Kojima, T., Kabayama, H., Mikoshiba, K. Mutation of the pleckstrin homology domain of Bruton's tyrosine kinase in immunodeficiency impaired inositol 1,3,4,5-tetrakisphosphate binding capacity. Journal of Biological Chemistry. 271 (48), 30303-30306 (1996).
- Knodler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. BioTechniques. 38 (6), (2005).
- Best, M. D. Global approaches for the elucidation of phosphoinositide-binding proteins. Chemistry and Physics of Lipids. 182, 19-28 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados