Method Article
Identification du phosphate d'inositol ou du phosphoinositide Protéines interactatrices par chromatographie d'affinité couplée à la fente occidentale ou à la spectrométrie de masse
Dans cet article
Résumé
Ce protocole se concentre sur l'identification des protéines qui se lient aux phosphates inositol ou phosphoinositides. Il utilise la chromatographie d'affinité avec les phosphates d'inositol biotinylated ou les phosphoinositides qui sont immobilisés par l'intermédiaire de streptavidin à l'agarose ou des perles magnétiques. Les protéines de liaison du phosphate d'inositol ou du phosphoinositide sont identifiées par le ballonnement occidental ou la spectrométrie de masse.
Résumé
Les phosphates inositol et les phosphoinositides régulent plusieurs processus cellulaires dans les eucaryotes, y compris l'expression de gène, le trafic de vésicule, la transduction de signal, le métabolisme, et le développement. Ces métabolites exercent cette activité réglementaire en se liant aux protéines, modifiant ainsi la conformation des protéines, l'activité catalytique et/ou les interactions. La méthode décrite ici utilise la chromatographie d'affinité couplée à la spectrométrie de masse ou au ballonnement occidental pour identifier les protéines qui interagissent avec les phosphates inositol ou les phosphoinositides. Les phosphates inositol ou phosphoinositides sont chimiquement étiquetés avec de la biotine, qui est ensuite capturée par streptavidinie conjuguée à des perles d'agarose ou magnétiques. Les protéines sont isolées par leur affinité de liaison au métabolite, puis élucées et identifiées par spectrométrie de masse ou ballonnement occidental. La méthode a un flux de travail simple qui est sensible, non radioactif, sans liposome, et personnalisable, soutenant l'analyse de l'interaction de protéine et de métabolite avec précision. Cette approche peut être utilisée dans des méthodes de spectrométrie de masse quantitative sans étiquette d'étiquette ou d'acide aminé pour identifier les interactions protéine-métabolite dans des échantillons biologiques complexes ou à l'aide de protéines purifiées. Ce protocole est optimisé pour l'analyse des protéines de Trypanosoma brucei, mais il peut être adapté aux parasites protozoaires, aux levures ou aux cellules mammifères connexes.
Introduction
Les phosphates inositol (IP) et les phosphoinositides (IP) jouent un rôle central dans la biologie de l'eucaryote par la régulation des processus cellulaires tels que le contrôle de l'expression génique1,2,3, le trafic de vésicule 4, transduction de signal5,6, métabolisme7,8,9, et le développement8,10. La fonction de régulation de ces métabolites résulte de leur capacité à interagir avec les protéines et donc à réguler la fonction protéique. Lors de la liaison par les protéines, les IP et les IP peuvent modifier la conformation des protéines11, l'activité catalytique12, ou les interactions13 et donc affecter la fonction cellulaire. Les IP et les IP sont distribués dans plusieurs compartiments subcellulaires, tels que le noyau2,3,14,15, réticulum endoplasmique16,17, plasma membrane1 et cytosol18, soit associée aux protéines3,19 ou avec RNAs20.
Le clivage de l'IP associé à la membrane(4,5)P2 par la phospholipase C entraîne la libération d'Ins(1,4,5)P3, qui peut être phosphorylé ou déphosphorylé par des kinases IP et des phosphatases, respectivement. Les ADRESSEs IP sont des molécules solubles qui peuvent se lier aux protéines et exercer des fonctions de régulation. Par exemple, Ins(1,4,5)P3 dans le métazoan peut agir comme un deuxième messager en se liant aux récepteurs IP3, ce qui induit des changements conformationnels des récepteurs et donc la libération de Ca2 'des magasins intracellulaires11. Ins(1,3,4,5)P4 se lie au complexe histone deacetylase et régule l'assemblage complexe protéique et l'activité13. D'autres exemples de fonction réglementaire d'IPs incluent le contrôle de l'organisation de chromatine21,le transport d'ARN22,23, l'édition d'ARN24,et la transcription1,2,3 . En revanche, les PI sont souvent associés au recrutement de protéines à la membrane plasmatique ou aux membranes d'organites25. Cependant, une propriété émergente des PI est la capacité de s'associer avec des protéines dans un environnement non-membranaire3,15,19,26. C'est le cas du facteur stéroïdomique de récepteur nucléaire, dont la fonction de contrôle transcriptionnelle est réglée par PI(3,4,5)P319, et polymérase polyzymatique qui est réglée par PI nucléaire (4,5)P226. Un rôle réglementaire pour les IP et les IP a été montré dans de nombreux organismes, y compris la levure22,27, les cellules des mammifères19,23, Drosophila10 et les vers28. Le rôle de ces métabolites dans les trypanosomes, qui s'écartait tôt de la lignée eucaryote, est important. Ces métabolites jouent un rôle essentiel dans trypanosoma brucei contrôle transcriptionnel1,3, développement8, biogenèse organelle et le trafic protéique29,30 , 31 Ans, états-unis ( , 32, et sont également impliqués dans le contrôle du développement et de l'infection chez les pathogènes T. cruzi33,34,35,Toxoplasma36 et Plasmodium 5 Annonces , 37. Par conséquent, la compréhension du rôle des IP et des IP dans les trypanosomes peut aider à élucider une nouvelle fonction biologique pour ces molécules et à identifier de nouvelles cibles médicamenteuses.
La spécificité de la protéine et de la liaison IP ou PI dépend des domaines d'interaction des protéines et de l'état de phosphorylation de l'inositol13,38, bien que les interactions avec la partie lipidique des IP se produisent également19. La variété des IP et des IP et leurs kinases et phosphatases modifiantes fournit un mécanisme cellulaire flexible pour contrôler la fonction protéique qui est influencée par la disponibilité et l'abondance des métabolites, l'état de phosphorylation de l'inositol et les protéines affinité de l'interaction1,3,13,38. Bien que certains domaines protéiques soient bien caractérisés39,40,41, par exemple, domaine d'homologie pleckstrin42 et SPX (SYG1/Pho81/XPR1) domaines43 ,44,45, certaines protéines interagissent avec les IP ou LES IP par des mécanismes qui restent inconnus. Par exemple, la protéine répresseur-activateur 1 (RAP1) de T. brucei n'a pas de domaines canoniques de liaison PI mais interagit avec PI(3,4,5)P3 et contrôle la transcription des gènes impliqués dans la variation antigénique3. Chromatographie d'affinité et analyse de spectrométrie de masse des protéines ip ou PI interagissant des cellules trypanosome, levure, ou mammifères ont identifié plusieurs protéines sans domaines IP- ou PI-contraignants connus8,46, 47. Les données suggèrent d'autres domaines protéiques non caractérisés qui se lient à ces métabolites. Par conséquent, l'identification des protéines qui interagissent avec les IP ou les IP peut révéler de nouveaux mécanismes d'interaction protéine-métabolite et de nouvelles fonctions de régulation cellulaire pour ces petites molécules.
La méthode décrite ici utilise la chromatographie d'affinité couplée au ballonnement occidental ou à la spectrométrie de masse pour identifier les protéines qui se lient aux IP ou aux IP. Il utilise des IP biotinylated ou IP qui sont soit reliés à la streptavidine conjuguée à des perles d'agarose ou alternativement, capturés par l'intermédiaire de perles magnétiques conjuguées par streptavidin (figure1). La méthode fournit un flux de travail simple qui est sensible, non radioactif, sans liposome et convient pour détecter la liaison des protéines des lysates cellulaires ou des protéines purifiées3 (Figure 2). La méthode peut être utilisée dans l'étiquette-libre8,46 ou couplé à la spectrométrie de masse quantitative étiquetée acide aminé47 pour identifier la propriété IP ou PI-liaison des protéines à partir d'échantillons biologiques complexes. Par conséquent, cette méthode est une alternative aux quelques méthodes disponibles pour étudier l'interaction des IP ou des IP avec les protéines cellulaires et aidera à comprendre la fonction réglementaire de ces métabolites chez les trypanosomes et peut-être d'autres eucaryotes.
Protocole
1. Analyse des protéines liant IP ou PI par chromatographie d'affinité et ballonnement occidental
- Chromatographie de croissance cellulaire, de lyse et d'affinité
- Cultivez les cellules de T. brucei jusqu'à la phase médiane du journal et surveillez la viabilité et la densité des cellules. Un total de 5,0 x 107 cellules est suffisant pour un jeu d'enfant.
- Pour les formes de circulation sanguine, les cellules de croissance dans les milieux HMI-9 complétées par 10% de sérum bovin fœtal (FBS) à 37 oC et 5% de CO2. Maintenir la densité cellulaire entre 8,0 x 105 à 1,6 x 106 cellules/mL.
REMARQUE : Une densité supérieure à 1,8 x 106 cellules/mL peut affecter la viabilité des cellules. Le temps de doublement de la souche T. brucei 427 cultivée in vitro se situe entre 5,5 et 6,5 h. - Pour les formes procycliques, les cellules de croissance dans le milieu SDM-79 complété avec 10% FBS à 27 oC et de garder la densité cellulaire entre 1,0 x 107 et 3,0 x 107 cellules/mL.
- Pour les protéines purifiées (p. ex., protéines recombinantes), prendre 0,5 à 1 g de protéines et diluer dans 450 l'amorti de liaison (25 mM HEPES, 150 mM NaCl, 0,2 % 4-nonyl phenyl-polyéthylène glycol, pH 7,4). Conserver 5 % de la protéine diluée (entrée) pour l'analyse de la tache occidentale. Passez à l'étape 1.1.7.
- Pour les formes de circulation sanguine, les cellules de croissance dans les milieux HMI-9 complétées par 10% de sérum bovin fœtal (FBS) à 37 oC et 5% de CO2. Maintenir la densité cellulaire entre 8,0 x 105 à 1,6 x 106 cellules/mL.
- Cellules centrifugeuses à 1 600 x g pendant 10 min à température ambiante (RT). Jetez le supernatant.
REMARQUE : Voir l'étape 2.1.2 pour plus d'informations sur la centrifugation des grands volumes de culture. - Resuspendre doucement la pastille dans 10 ml de phosphate tamponné pH salin 7,4 complété avec 6 mM de glucose (PBS-G) et préchauffé à 37 oC pour laver les cellules. Ensuite, centrifuger les cellules à 1600 x g pendant 5 min à RT. Répétez la procédure deux fois.
- Resuspendre la pastille dans 1 ml de PBS-G. Ensuite, transférer le volume dans un tube de 1,5 ml et une centrifugeuse à 1 600 x g pendant 5 min. Jeter le supernatant.
REMARQUE : Les granulés de cellules peuvent être congelés en flash dans de l'azote liquide et stockés à -80 oC ou à l'azote liquide. - Resuspendre le granule en 0,5 ml de tampon de lyse (25 mM HEPES, 150 mM NaCl, 1% t-octylphenoxypolyethoxyethanol, pH 7.4) complété par 1,5x cocktail inhibiteur de la protéase et 1x cocktail inhibiteur de la phosphatase (Table of Materials) pré-réfrigéré dans la glace à lyse les cellules. Incuber le lysate pendant 10 min en rotation à 50 tr/min à 4 oC.
REMARQUE : Il s'agit d'une étape critique parce que les protéines peuvent se dégrader si elles ne sont pas manipulées comme indiqué. Vérifiez l'intégrité du lysate protéique par l'électrophores gel de sulfate de sulfate de polyacrylamide de sodium (SDS/PAGE) si nécessaire.
CAUTION: T-octylphenoxypolyethoxyethanol est toxique et peut causer une irritation de la peau et des yeux. Utilisez des gants, un pare-œil et une protection pour le visage. - Centrifuger le lysate à 14 000 x g pendant 10 min à 4 oC. Recueillir le supernatant dans un nouveau tube de 1,5 ml pour les essais de liaison. Conserver 5 % du lysate total (entrée) pour l'analyse des ballonnements occidentaux. Le supernatant contient des protéines parasites extraites avec un tampon de lyse.
- Recueillir 50 L de PI ou de IP conjugués à des perles d'agarose (c.-à-d. 50 l de lisier) ou 50 l de perles d'agarose, et centrifugeuse pendant 1 min à 1 000 x g. Jeter le supernatant et le resuspendre dans 50 l de tampon de liaison pour équilibrer les perles. Utilisez des perles non conjuguées comme un contrôle. Utilisez des perles IP/PI avec différentes configurations de phosphate, y compris des formes non phosphorylées, pour contrôler les interactions non spécifiques.
- Ajouter 50 l de perles IP ou PI au lysate cellulaire ou aux protéines purifiées (chaque 1 ml de perles contient 10 nmol d'IP ou de IP conjugués). Maintenez le volume de perles IP ou PI dans 10 % du lysate total et, si nécessaire, ajustez le volume de réaction de liaison avec un tampon de liaison.
- Pour les essais de concurrence, ajoutez à la réaction contraignante diverses concentrations d'IP ou d'IP non conjugués (p. ex., 1, 10, 100 fois l'excès de molaire par rapport aux perles IP ou PI).
- Incuber la réaction pendant 1 h, ou toute la nuit, à 4 oC et tourner à 50 tr/min.
REMARQUE : Les réactions de liaison avec des protéines purifiées peuvent être faites à RT selon la stabilité de la protéine. Si l'utilisation d'IP ou d'IP conjuguées à la biotine ne fait qu'aller à l'étape 1.1.9.1, passez autrement à l'étape 1.1.10.- Ajouter 50 lde de perles magnétiques conjuguées à la streptavidine à la réaction de liaison et incuber pendant 1 h à 4 oC en rotation à 50 tr/min.
- Centrifuger le mélange pendant 1 min à 1 000 x g à 4 oC. Retirez le supernatant (flux) et conservez le granule. Gardez 5% du supernatant pour l'analyse western blot.
REMARQUE : Si vous utilisez des perles magnétiques, retirez les supernatants et effectuez des lavages subséquents à l'aide d'un support magnétique (les centrifugations ne sont pas nécessaires). - Ajouter 1 ml de tampon de lavage (25 mM HEPES, 300 mM NaCl, 0,2 % 4-nonyl phényl-polyéthylène glycol, pH 7,4) et resuspendre la résine en tapant ou en faisant tourbillonner le tube (ne pas utiliser de pipette parce que les perles peuvent se fixer à des pointes de pipette). Centrifuger la réaction pendant 1 min à 1 000 x g à 4 oC et jeter le supernatant. Répétez la procédure pour un total de cinq lavages.
CAUTION: 4-nonyl phenyl-polyéthylène glycol est toxique et peut causer une irritation de la peau et des yeux. Utilisez des gants, un pare-œil et une protection pour le visage. - Ajouter 50 l de tampon Laemmli 2x complété de 710 mM 2-mercaptoéthanol aux perles et mélanger en tapant ou en vortex pour éliper les protéines. Chauffer à 95 oC pendant 5 min, puis centrifuger pendant 10 000 x g pendant 1 min et recueillir le supernatant (contenir des protéines élucées). Alternativement, élichez les protéines avec 8 M d'urée/100 mM de glycine pH 2.9 pour éviter d'utiliser sDS. Congeler l'éluate à -80 oC, sinon procéder à l'analyse de la tache occidentale.
CAUTION : Le 2-mercaptoéthanol est toxique et peut causer des irritations de la peau, des yeux et des voies respiratoires. Utilisez des gants et travaillez dans le capot chimique.
- Cultivez les cellules de T. brucei jusqu'à la phase médiane du journal et surveillez la viabilité et la densité des cellules. Un total de 5,0 x 107 cellules est suffisant pour un jeu d'enfant.
- Analyse de l'ouest de l'adrage
- Mélanger 15 échantillons d'entrée (à partir de l'étape 1.1.6) ou d'écoulement (à partir de l'étape 1.1.10) avec 5 'L de tampon 4x Laemmli. Chauffer les échantillons d'entrée et d'écoulement pendant 5 min à 95 oC. Pour les échantillons élifiés dans 8 M d'urée/100 mM de glycine pH 2,9, mélanger 15 l'l d'éluate avec 5 ll de tampon 4x Laemmli, et chauffer pendant 5 min à 95 oC.
REMARQUE: Cette étape n'est pas nécessaire pour les échantillons élutés dans 2x laemmli tampon. - Chargez les puits de 4 à 20 % de gel SDS/PAGE avec 2,5 l d'entrée, 2,5 l d'écoulement et 20 l d'échantillons élutés, et chargez l'échelle protéique selon la recommandation du fabricant.
REMARQUE: Choisissez gel% en fonction du poids moléculaire de la protéine d'intérêt. - Exécuter SDS/ PAGE à 150 V pendant 30-45 min en mémoire tampon en cours d'exécution, ou jusqu'à ce que le colorant bleu du tampon Laemmli soit à la fin du gel.
REMARQUE : L'heure de course peut varier selon l'équipement de laboratoire. - Retirer le gel des plaques en verre (ou en plastique) et tremper dans un tampon de transfert pendant 15 minutes.
- Transférer les protéines à la membrane de polyvinylidene difluorure (PVDF) ou à la membrane de nitrocellulose. Tremper les membranes et le papier filtre de 3 mm dans le tampon de transfert. Assembler un sandwich avec trois feuilles de papier filtre, de la nitrocellulose ou de la membrane PVDF, du gel et trois feuilles de papier filtre supplémentaires. Assurez-vous qu'aucune bulle d'air n'est emprisonnée dans le sandwich. Utilisez un rouleau pour enlever les bulles si nécessaire. Placez la membrane sur la cathode et le gel sur le côté anode de la cassette.
REMARQUE : Vérifiez les instructions du fabricant de la membrane pour obtenir des renseignements sur l'activation de la membrane ou la préparation de la tache. - Placez la cassette contenant le sandwich dans le réservoir de transfert avec le tampon de transfert. Placer le réservoir dans un seau à glace ou à 4 oC (p. ex., dans la chambre froide). Transférer les protéines à 100 V pendant 1 h (le courant varie entre 200-400 mA). Alternativement, transférer la nuit à un courant constant de 15 mA à 4 oC.
- Retirer la membrane de la cassette. Incuber la membrane dans 6% de lait sec non gras dilué dans PBS avec 0,05% de polysorbate 20 (PBS-T), ou solution de blocage compatible, pendant 1 h à RT pour bloquer la membrane.
REMARQUE: Avant de bloquer la membrane, la qualité du transfert peut être vérifiée à l'aide de la tache Ponceau S. Incuber la membrane pendant 1 min en 15 ml de Ponceau S, rincer à l'eau et visualiser les bandes. - Retirez la solution de blocage et incubez la membrane pendant 1 h à RT avec rotation de 50 tr/min dans les anticorps primaires dilués dans 6 % de lait sec non gras dilué dans LE PBS-T. Alternativement, incuber la membrane pendant la nuit à 4 oC avec rotation de 50 tr/min.
REMARQUE : Le temps d'incubation peut varier en fonction de la qualité des anticorps; cependant, la plupart des anticorps fonctionneront avec des incubations de 1-3 h à RT. Suivez la recommandation du fabricant pour la concentration ou la dilution des anticorps. - Laver la tache en couvant la membrane dans PBS-T pendant 5 min avec une rotation de 50 tr/min à RT. Répétez la procédure 3-5 fois. Plus de lavages peuvent être nécessaires selon la qualité des anticorps.
- Incuber la membrane dans le raifort peroxidase (HRP)-anticorps secondaires conjugués pendant 1 h à RT dans 6% de lait sec non gras dilué dans PBS-T avec rotation de 50 tr/min.
REMARQUE : Suivez la recommandation du fabricant concernant la concentration ou la dilution des anticorps. - Laver la tache comme indiqué à l'étape 1.2.9.
- Ajouter le substrat chemiluminescent pour couvrir la membrane. Retirer l'excès de substrat et couver pendant 5 min à RT dans l'obscurité.
REMARQUE : Consultez les instructions du fabricant pour obtenir des recommandations sur les réactifs de chemiluminescence. - Capturez le signal chemiluminescent à l'aide d'un imageur à base de caméra. Vous pouvez également utiliser un film radiographique pour capturer le signal chemiluminescent.
- Mélanger 15 échantillons d'entrée (à partir de l'étape 1.1.6) ou d'écoulement (à partir de l'étape 1.1.10) avec 5 'L de tampon 4x Laemmli. Chauffer les échantillons d'entrée et d'écoulement pendant 5 min à 95 oC. Pour les échantillons élifiés dans 8 M d'urée/100 mM de glycine pH 2,9, mélanger 15 l'l d'éluate avec 5 ll de tampon 4x Laemmli, et chauffer pendant 5 min à 95 oC.
2. Analyse des protéines liant IP/PI par chromatographie d'affinité et spectrométrie de masse
- Chromatographie de croissance cellulaire, de lyse et d'affinité
- Cultivez les cellules de T. brucei jusqu'à la phase médiane du journal et surveillez la viabilité et la densité des cellules.
REMARQUE : Un total de 1,0 x 1010 cellules est suffisant pour deux essais de liaison. L'utilisation de moins de cellules que ce qui est indiqué ici peut affecter la détection de protéines à faible abondance par spectrométrie de masse.- Pour les formes de circulation sanguine de T. brucei, les cellules de croissance à mi-log phase (8,0 x 105-1,6 x 106 cellules/mL) dans les milieux HMI-9 complétés par 10% FBS à 37 oC et avec 5% co2. Pour 427 souches, 5 L de culture donnera 0,5-1,0 x 1010 cellules. Surveillez la croissance cellulaire pour éviter une densité supérieure à 1,8 x 106 cellules/mL, ce qui peut affecter la viabilité cellulaire. Maintenir le volume de culture cellulaire à 1/10 du volume flasque; sinon, le taux de croissance des cellules sera affecté en raison d'une mauvaise aération.
REMARQUE: La souche 427 a un temps de doublement de 5,5-6,5 h. - Pour les formes procycliques, les cellules de croissance dans le milieu SDM-79 complété avec 10% FBS à 27 oC et de garder la densité cellulaire entre 1,0 x 107 et 3,0 x 107 cellules/mL. 500 ml de culture donnera 0,5-1,5 x 1010 cellules.
- Pour les formes de circulation sanguine de T. brucei, les cellules de croissance à mi-log phase (8,0 x 105-1,6 x 106 cellules/mL) dans les milieux HMI-9 complétés par 10% FBS à 37 oC et avec 5% co2. Pour 427 souches, 5 L de culture donnera 0,5-1,0 x 1010 cellules. Surveillez la croissance cellulaire pour éviter une densité supérieure à 1,8 x 106 cellules/mL, ce qui peut affecter la viabilité cellulaire. Maintenir le volume de culture cellulaire à 1/10 du volume flasque; sinon, le taux de croissance des cellules sera affecté en raison d'une mauvaise aération.
- Centrifuger les cellules à 1 600 x g pendant 15 min à RT. Jetez le supernatant et suspendez la pastille dans 200 ml de PBS-G préchauffé à 37 oC. Utilisez des tubes de centrifugation à fond rond (Tableof Materials) parce que les granulés de forme de circulation sanguine T. brucei sont facilement perturbés lors de l'utilisation de rotors de centrifugeuse à angle fixe. Centrifuger les cellules à nouveau à 1600 x g pendant 5 min à RT.
- Retirez le supernatant et suspendez la pastille dans 10 ml de PBS-G. Centrifuger les cellules à 1600 x g pendant 5 min à RT. Répétez la procédure et après le lavage final, jetez le supernatant.
REMARQUE : Les granulés peuvent être congelés au flash dans de l'azote liquide et stockés à -80 oC ou à l'azote liquide. - Resuspendre la pastille cellulaire dans 5 ml de tampon de lyse pré-réfrigéré dans la glace et complété par 1,5x cocktail inhibiteur de la protéase et 1x cocktail inhibiteur de la phosphatase (Table of Materials). Incuber le lysate pendant 10 min à 4 oC en rotation à 50 tr/min.
- Centrifuger le lysate à 10 000 x g pendant 10 min à 4 oC. Recueillir le supernatant (protéines solubilisées) et le diluer dans 20 ml de tampon de liaison.
- Recueillir 400 L d'IP ou de IP conjugués à des perles d'agarose (c.-à-d. 400 l de boue) ou 400 l de perles témoins, et centrifugeuse pendant 1 min à 1 000 x g à RT. Jetez le supernatant et resuspendez dans 400 l de tampon de liaison pour équilibrer les perles. Utilisez des perles d'agarose comme un contrôle négatif pour déterminer l'enrichissement spécifique des interactions protéine-métabolite par rapport aux interactions non spécifiques (par exemple, protéines qui se lient non spécifiquement aux perles). Utilisez des IP ou des IP avec différentes configurations de phosphate, y compris des formes non phosphorylées pour contrôler les interactions non spécifiques en raison de charges de phosphate ou de liaison à la biotine.
- Ajouter 400 l de perles IP/PI ou des perles de contrôle à 10 ml de lysate et incubé pendant 1 h, ou toute la nuit, à 4 oC tournant à 50 tr/min. Si l'utilisation d'IP ou d'IP conjuguées à de la biotine seulement (sans perles) passe à l'étape 2.1.7.1, sinon passez à l'étape 2.1.8.
- Ajouter 100 l de streptavidinie-conjugué aux perles magnétiques et couver pendant 1 h à 4 oC en rotation à 50 tr/min.
- Centrifuger la réaction de liaison pendant 1 min à 1 000 x g à 4 oC. Retirez le supernatant (flux) et conservez le granule. Gardez 5% du supernatant pour l'analyse western blot.
- Ajouter 5 ml de tampon de lavage à la pastille, mélanger délicatement en faisant tourbillonner le tube, puis centrifuger pendant 1 min à 1 000 x g à 4 oC. Jetez le supernatant. Répétez le lavage cinq fois. Si vous utilisez des perles magnétiques, collectez les supernatants et effectuez des lavages à l'aide d'un support magnétique (les centrifugations ne sont pas nécessaires).
- Ajouter 50 l de tampon Laemmli 2x (ou 8 M d'urée/100 mM de glycine pH 2,9, pour éviter d'utiliser le SDS) aux perles et mélanger en tapant ou en vortex (éviter la pipetage parce que les perles peuvent se fixer aux pointes de pipette), puis chauffer à 95 oC pendant 5 min. Centrifugeuse pendant 10 000 x g pendant 1 min et recueillir le supernatant (protéines élucées). Répétez la procédure deux fois pour recueillir un total de trois fractions.
- Congelez l'éluate à -80 oC, autrement séparer les protéines dans SDS/PAGE ou conservez-les en solution pour la trypsinisation et l'analyse de spectrométrie de masse.
- Cultivez les cellules de T. brucei jusqu'à la phase médiane du journal et surveillez la viabilité et la densité des cellules.
- Digestion de trypsine des protéines pour la spectrométrie de masse
REMARQUE : Deux variantes de cette procédure sont indiquées pour la section 2.2.1 (en gel) ou la section 2.2.2 (en solution) de digestion des protéines. Les tubes à faible liaison protéique s'entrouvent sont recommandés pour prévenir les pertes d'échantillons. Consulter un chimiste analytique à l'installation de protéomique la pertinence du protocole pour les échantillons et les instruments de spectromètre de masse disponibles.- Trypsinisation in-gel des protéines
- Après la séparation des protéines dans SDS/PAGE, rincez brièvement le gel dans de l'eau de haute pureté. Transférer le gel sur une plaque de verre propre. Excisez les bandes de protéines avec une lame propre et évitez de couper du gel supplémentaire à l'extérieur des bandes. Couper les morceaux de gel en petits morceaux (c.-à-d. environ 1 mm carré) et les transférer dans un tube de 1 ml. Utilisez une pointe de pipette si nécessaire, mais assurez-vous que la pointe de pipette est rincée à l'éthanol avant utilisation.
REMARQUE : Utilisez des gants pour éviter la contamination par le gel. Les morceaux de gel peuvent être stockés à -20 oC. - Ajouter 100 ldre d'eau de haute pureté ou de haute performance de qualité de chromatographie liquide aux tubes pour rincer les morceaux de gel. Jeter l'eau.
- Pour les morceaux de gel teinté fluorescent à base de Coomassie ou de ruthénium, incuber les morceaux de gel dans la solution de destaining (25 mM NH4HCO3 en 50% d'acétonitrile) pendant 1 h, puis jeter la solution. Répétez la procédure jusqu'à ce que la coloration ne soit pas visible.
REMARQUE : Préparer des solutions contenant NH4HCO3 par dilution à partir d'une solution de stock à 100 mM NH4HCO3, pH 7,8.
CAUTION : L'acétonitrile est un solvant volatil, inflammable et toxique. NH4HCO3 peut causer une irritation de la peau ou des yeux. Utilisez des gants et travaillez sous une hotte chimique. - Pour les morceaux de gel tachéd d'argent, incuber des morceaux de gel en 50 oL de solution de destaining pour la tache d'argent (15 mM K3[Fe(CN)6], 50 mM Na2S2O3) dans l'eau pendant 30 min. Jeter la solution et laver les morceaux de gel avec 200 l d'eau. Répétez le lavage cinq fois ou jusqu'à ce que la couleur jaune gel ne soit pas visible.
CAUTION: K3[Fe(CN)6] peut causer une irritation de la peau ou des yeux. Utilise des gants.
- Pour les morceaux de gel teinté fluorescent à base de Coomassie ou de ruthénium, incuber les morceaux de gel dans la solution de destaining (25 mM NH4HCO3 en 50% d'acétonitrile) pendant 1 h, puis jeter la solution. Répétez la procédure jusqu'à ce que la coloration ne soit pas visible.
- Déshydrater les morceaux de gel à l'aide de 200 l d'acétonitrile pendant 10 min à RT. Ensuite, jetez la solution.
REMARQUE : Les morceaux de gel déshydratés sont plus petits en volume, opaques et collants. Si plusieurs morceaux de gel sont combinés dans un tube, répétez la procédure de déshydratation efficace des morceaux de gel. - Ajouter 50 L (ou assez de volume pour couvrir les morceaux de gel) de la solution réducteur (10 mM dithiothreitol [DTT] en 100 mN NH4HCO3) et couver à 56 oC pendant 1 h. Ensuite, refroidir les tubes à RT et jeter l'excès de solution de réduction.
- Ajouter 50 L (ou assez de volume pour couvrir les morceaux de gel) de la solution d'alkylation (50 mM d'iodoacétéamide en 100 mM NH4HCO3) et couver pendant 30 min à RT dans l'obscurité. Ensuite, jetez l'excès de solution d'alkylation.
- Déshydrater les morceaux de gel avec 200 l d'acétonitrile pendant 10 min à RT. Retirez l'acétonitrile et hydratez les morceaux de gel avec 100 mNN4HCO3 pendant 10 min à RT.
- Déshydrater à nouveau les morceaux de gel avec 200 l d'acétonitrile pendant 10 min à RT et jeter l'excès de solution.
- Ajouter 15 ll de trypsine de qualité de spectrométrie de masse diluée dans un tampon NH4HCO3 de 50 m, ou assez de volume pour couvrir les morceaux de gel hydraté et couver pendant 4 h, ou pendant la nuit, à 37 oC. Gardez des quantités totales de trypsine entre 100 et 500 ng (ou 20 ng trypsin/g de protéines).
- Refroidir l'échantillon à RT, et centrifugeuse pendant 1 min à 2 000 x g dans un microcentrifugeur. Ajouter 10-20 'L d'acide formique de 5% dilué dans l'eau et couver pendant 10 min à RT.
CAUTION : L'acide formique est inflammable, corrosif et toxique. Utilisez des gants et travaillez sous une hotte chimique. - Centrifugeuse comme indiqué à l'étape 2.2.1.9, puis recueillir le supernatant (contiennent des peptides extraits) à un tube différent. Ajouter 20 ll d'acide formique dilué à 50-60% d'acétonitrile au tube et couver pendant 10 min à RT. Recueillir les fractions de peptide extraites dans le même tube.
- Séchez l'échantillon dans un concentrateur sous vide et reconstituez-le en 10 L d'acide acétique de 0,5 % et de 2 % d'acétonitrile pour l'analyse de la spectrométrie de masse. Centrifuge et recueillir la solution au fond du tube; solution de magasin à -20 oC ou -80 oC.
- Après la séparation des protéines dans SDS/PAGE, rincez brièvement le gel dans de l'eau de haute pureté. Transférer le gel sur une plaque de verre propre. Excisez les bandes de protéines avec une lame propre et évitez de couper du gel supplémentaire à l'extérieur des bandes. Couper les morceaux de gel en petits morceaux (c.-à-d. environ 1 mm carré) et les transférer dans un tube de 1 ml. Utilisez une pointe de pipette si nécessaire, mais assurez-vous que la pointe de pipette est rincée à l'éthanol avant utilisation.
- Dans la trypsinisation de solution des protéines
- Précipitez les protéines pour réduire le volume de l'échantillon, le dessalement et l'échange de tampons. Ajouter six volumes d'acétone réfrigéré (-20 oC) à l'échantillon, par exemple, 600 l d'acétone à 100 oL d'échantillon. Vortex et incuber à -20 oC pendant 15 min à 1 h. La solution deviendra trouble ou formera un précipité.
CAUTION: L'acétone est toxique et inflammable. Utilisez des gants et travaillez sous une hotte chimique. - Échantillons de centrifugeuse à 4 oC pendant 30 min. Décanter l'acétone et sécher à l'air le granule pendant 15 min.
- Ajouter 10 l d'urée de 6 à 8 M ou 1 % de SDS dans 50 mNNH4HCO3 pour dissoudre le granule et le vortex pour mélanger. Pour de plus grandes quantités de protéines (à 10 g), utilisez jusqu'à 20 l de tampon de dissolution.
- Ajouter 5 ll de solution de réduction et de vortex. Faites tourner le volume vers le bas à l'aide d'un microcentrifugeur. Si les échantillons sont dilués dans l'urée, puis incuber pendant 1 h à RT. Si les échantillons sont dilués dans le SDS, puis incubé pendant 1 h à 56 oC.
- Faites tourner le volume vers le bas à l'aide d'un microcentrifugeur. Ajouter 3 ll de solution d'alkylation et de vortex. Ensuite, faites tourner le volume vers le bas et incubez dans l'obscurité pendant 30 min à RT.
- Ajouter 3 ll de solution de réduction pour neutraliser la réaction. Diluer lentement l'échantillon à 1 M d'urée ou 0,05 % de SDS à l'aide de 50 mN NH4HCO3.
REMARQUE : Le tampon de digestion de trypsin doit avoir le détergent ou le dénaturant. Les limites de concentration pour les dénaturants sont les suivante : 0,05 % de SDD; 0,1 % d'octyl B-D-glucopyranoside; 0,1 % 4-nonlphényl-polyéthylène glycol; 0,1% t-octylphenoxypolyethoxyethanol; 0,1 % polysorbate 20; 0,1 % 3-[(3-cholamidopropyl)dimethylammonio]-1-propanesulfonate; 1 M d'urée ou de thiourea. - Ajouter 5 ll de trypsine de catégorie spectrométrie de masse diluée dans un tampon De 50 mNH4HCO3 et incuber pendant 4 h, ou toute la nuit, à 37 oC. Gardez des quantités totales de trypsine entre 100 et 500 ng (ou 20 ng trypsin/g de protéines).
- Refroidir l'échantillon à RT, et faire tourner le volume vers le bas à l'aide d'un microcentrifugeur. Ajouter 5% d'acide acétique (ou 5% d'acide formique dans 50% d'acétonitrile) pour étancher la réaction.
- Séchez les échantillons dans un concentrateur sous vide indiqué à l'étape 2.2.1.11. Entreposer les échantillons à -80 oC. Désaler et concentrer les peptides à l'aide d'une colonne de phase inversée comme la pointe zippée C18, puis analyser par spectrométrie de masse.
- Précipitez les protéines pour réduire le volume de l'échantillon, le dessalement et l'échange de tampons. Ajouter six volumes d'acétone réfrigéré (-20 oC) à l'échantillon, par exemple, 600 l d'acétone à 100 oL d'échantillon. Vortex et incuber à -20 oC pendant 15 min à 1 h. La solution deviendra trouble ou formera un précipité.
- Trypsinisation in-gel des protéines
Résultats
Analyse de rap1 et PI(3,4,5)P3 interaction par chromatographie d'affinité et de ballonnement occidental
Cet exemple illustre l'application de cette méthode pour analyser la liaison des IP par RAP1 à partir du lysate de T. brucei ou par la protéine recombinante T. brucei RAP1. Lysates de T. brucei formes de circulation sanguine qui expriment hémagglutinine (HA) marqué RAP1 ont été utilisés dans les essais de liaison. RAP1 est une protéine impliquée dans le contrôle transcriptionnel des gènes de glycoprotéine de surface variable (VSG)3,48, qui codent pour les protéines de surface impliquées dans l'évasion immunitaire parasite par variation antigénique49. RAP1 interagit au sein d'un complexe protéique télomérique avec le phosphatidylinositol 5-phosphatase (PIP5Pase) enzyme3, qui fonctionne également dans le contrôle de la transcription du gène VSG1,3. RAP1 a un n-terminal cancer du sein 1 carboxyl-terminal (BRCT) domaine qui est suivie par un myeloblastose (myb) domaine de liaison de l'ADN et un C-terminal myb-like domaine3,48. Cependant, il manque de domaines de liaison PI canoniques. Des essais de liaison ont été exécutés avec des OPI qui ne sont pas phosphorylés ou qui sont phosphorylés à différentes positions de l'anneau d'inositol, et avec des perles d'agarose non conjuguées. L'analyse occidentale montre que RAP1 se lie de préférence aux PI (3,4,5)Perles De P3 (Figure 3A), mais qu'il lie aussi dans une moindre mesure à PI(4,5)P2-perles. Cependant, il ne s'est lié à aucun autre PI ou perles d'agarose. Puisque RAP1 fait partie d'un complexe multiprotéine3,son interaction avec quelques PI peut ne pas être directe et résulte ainsi de l'interaction de RAP1-HA avec d'autres protéines cellulaires qui se lient aux PIs.
Par conséquent, pour vérifier si RAP1 se lie directement aux PI, une protéine 6x-son RAP1 recombinante étiquetée En phase C a été exprimée et purifiée à l'homogénéité de E. coli3. La protéine a été utilisée dans les essais de liaison avec PI(3,4,5)P3-perles en présence de concentrations concurrentes de PI(3,4,5)P3 ou PI(4,5)P2. Le ballonnement occidental montre que l'augmentation des concentrations d'IP(3,4,5)P3, mais pas PI(4,5)P2 inhibe l'interaction du rRAP1 avec L'IP(3,4,5)P3 (figure 3B). En outre, l'ajout de T. brucei purifié enzyme PIP5Pase à la réaction restauréPI(3,4,5)P3-contraignant par rRAP1, qui est due à la déphosphorylation PIP5Pase de PI libre(3,4,5)P33 et indique donc que le modèle de phosphorylation de cette le métabolite est essentiel pour la liaison rRAP1. Par conséquent, rRAP1 interagit avec PI(3,4,5)P3 comme il le fait RAP1-HA de T. brucei lysates. De plus, les données montrent que la liaison du RAP1-HA du lysate à l'IP(4,5)P2 résulte probablement de l'interaction RAP1 avec d'autres protéines du complexe (p. ex. PIP5Pase)3. Les données illustrent la complémentarité des essais de liaison avec des lysates cellulaires et des protéines recombinantes. Il montre également l'utilité d'essais contraignants concurrentiels pour déterminer la spécificité des interactions entre les protéines et les PI.
Identification des protéines de liaison Ins(1,4,5)P3 par chromatographie d'affinité et spectrométrie de masse
Dans cet exemple, la chromatographie d'affinité suivie de la spectrométrie de masse a été employée pour identifier des protéines de T. brucei qui se lient à Ins(1,4,5)P3 ; par conséquent, l'expérience étudie les protéines potentielles Ins(1,4,5)P3 de liaison des formes de circulation sanguine de T. brucei. Le lysate de T. brucei a été incubé avec Des Ins(1,4,5)P3 conjugués à des perles d'agarose ou à des perles non conjuguées (utilisées comme contrôle), et les protéines liées ont été élucées avec le tampon d'échantillon de Laemmli. L'analyse SDS/PAGE montre l'enrichissement des protéines élucées à partir d'Ins(1,4,5)Perles P3 par rapport aux protéines élucées par les perles d'agarose témoins (figure 4A). Analyse de spectrométrie de masse des protéines élunées identifiées sur plus de 250 protéines, dont 84 ont été enrichies avec des perles Ins(1,4,5)P3 par rapport aux perles témoins (Figure 4B, changement de pli [FC] 2, p lt; 0,05). L'enrichissement des protéines liées à Ins(1,4,5)P3 par rapport aux perles témoins est en corrélation avec le signal protéique détecté par SDS/PAGE. Les données comprennent les protéines qui ont été validées pour lier Ins(1,4,5)P3 et les protéines dont les mécanismes de liaison à Ins(1,4,5)P3 sont inconnus8. En outre, le protéome de liaison Ins(1,4,5)P3 différait considérablement de celui d'Ins(1,3,4,5)P4 et d'autres PIs8, ce qui suggère que certaines de ces protéines reconnaissent la configuration spécifique du phosphate d'Ins(1,4,5)P3. Par conséquent, la biotine-étiquetée Ins(1,4,5)P3 peut être employée pour la chromatographie d'affinité couplée à la spectrométrie de masse pour identifier des protéines qui se lient à Ins(1,4,5)P3. L'approche peut être explorée pour identifier les protéines qui se lient à d'autres IP ou IP3,8,46,47.

Figure 1 : Réagents d'affinité pour les essais de liaison. (A) PI (3,4,5)P3 (en haut) et Ins(1,4,5)P3 (en bas) conjugués à la biotine à la position sn1 de l'inositol. Dans PI(3,4,5)P3, la biotine est conjuguée à la chaîne lipidique à la position sn1 de l'inositol, tandis que dans Ins(1,4,5)P3 la biotine est conjuguée au phosphate à la position sn1. (B) Ins(1,4,5)P3 est conjugué à la biotine et capturé par liaison à la streptavidine conjuguée aux perles (p. ex. aurrose ou perles magnétiques). Variations de ces réactifs à l'aide de liaisons synthétisées personnalisées qui remplacent la biotine sont également possibles46. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
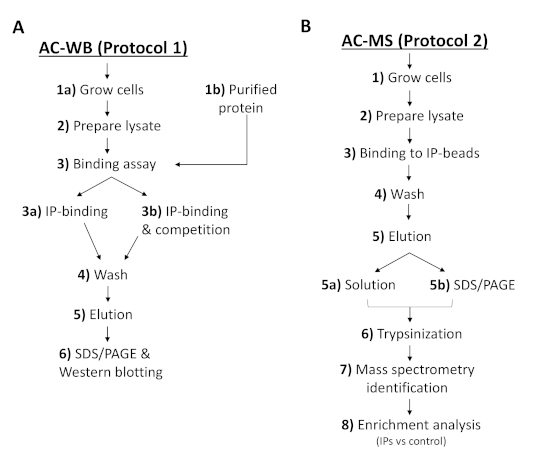
Figure 2 : Le flux de travail des protocoles décrit les étapes de l'analyse de l'interaction d'affinité IP ou PI avec les protéines de T. brucei et la détection par (A) le ballonnement occidental ou (B) spectrométrie de masse. AC-WB, chromatographie d'affinité et ballonnement occidental ; AC-MS, chromatographie d'affinité et spectrométrie de masse. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Liaison de T. brucei RAP1 aux phosphoinositides. (A) Lysates de T. brucei (5,0 x 107 parasites) qui expriment LE RAP1 étiqueté HA pendant 2 h à 4 oC avec 50 OL de PI (chacune 1 ml de perles d'agarose contenant 10 nmol de PI conjugués) ou de perles d'agarose (Ag). La réaction de liaison a été lavée et éludée avec 2 x tampon d'échantillon de Laemmli et chauffée à 95 oC pendant 5 min. Les protéines ont été séparées en 4-20% SDS/PAGE, transférées à une membrane PVDF, et sondées avec des anticorps monoclonaux anti-HA (1:5,000, dilué dans 6% PBS-milk) suivi par l'anti-souris IgG-HRP (1:5,000, dilué dans 6% PBS-lait), et détecté par chemiluminescence. (B) Un g de rRAP1 a été incubé pendant 1 h à RT avec 50 L de PI (3,4,5)Perles de P3-agarose en présence ou en absence de 5 à 50 M dioctanoylglycerol (diC8) PI(3,4,5)P3, P3, 20 à 50 M diC8 PI(4,5)P2, ou 50 M diC8 PI(3,4,5)P3 et 250 ng PIP5Pase purifié de T. brucei formes de circulation sanguine3. La liaison a été analysée par le ballonnement occidental avec la souris anti-His HRP monoclonal des anticorps (1:2,000, dilué dans 6% PBS-milk) et développé par chemiluminescence. Ce chiffre a été modifié à partir de Cestari et coll.3. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Analyse de la chromatographie d'affinité et de la spectrométrie de masse des protéines de T. brucei qui se lient à Ins(1,4,5)P3. (A) 10% SDS/PAGE analyse des protéines T. brucei qui se lient à Ins(1,4,5)P3-perles ou perles d'agarose. Lysates de 5,0 x 109 parasites ont été incubés à 4 oC pour 2 h avec 400 oL d'Ins(1,4,5)P3 conjugués à des perles d'agarose ou avec des perles d'agarose sans Ins(1,4,5)P3 [1 mL de perles contiennent 10 nmol d'Ins conjugués(1,4,5)P3]. La réaction de liaison a été lavée, éludée dans un tampon d'échantillon de 2 x Laemmli et bouillie pendant 5 min à 95 oC. Les protéines ont été séparées en 10% SDS/PAGE et tachées de taches Coomassie (Tableau des matériaux). Les pointes d'arrow montrent des protéines qui sont enrichies en Ins(1,4,5)Perles de P3 comparées aux perles d'agarose ; les cercles indiquent des protéines présentes dans les deux Ins(1,4,5)Perles de P3 et de perles d'agarose, et le support indique les protéines qui sont présentes dans les deux mais sont enrichies dans Ins(1,4,5)P3-perles comparées aux perles d'agarose. (B) L'intrigue ponctuelle montre les protéines identifiées par la spectrométrie de masse qui sont enrichies en Ins(1,4,5)Perles P3 par rapport aux perles d'agarose. Enrichissement défini par FC 'gt; 2 et p-valeur 'lt;0.05. Quatre répliques biologiques ont été utilisées pour les perles d'agarose AC-MS, et trois répliques biologiques pour les perles IP3 AC-MS. Le changement de pli des protéines identifiées dans les perles IP3 contre les perles d'agarose a été calculé à l'aide de l'intensité des spectres peptidiques à l'aide du MSstat50. Les résultats détaillés et la liste des peptides sont disponibles8. Les données brutes de spectrométrie de masse sont également disponibles avec l'identifiant PXD005907 par l'intermédiaire du Consortium ProteomeXchange via le référentiel partenaire PRIDE. Ce chiffre a été modifié à partir de Cestari et coll.8. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
L'identification des protéines qui se lient aux IP ou aux IP est essentielle pour comprendre la fonction cellulaire de ces métabolites. La chromatographie d'affinité couplée à la tache occidentale ou à la spectrométrie de masse offre l'occasion d'identifier les protéines interagissant IP ou PI et donc d'obtenir des informations sur leur fonction réglementaire. IPs ou IP chimiquement marqués [p. ex., Ins(1,4,5)P3 chimiquement liés à la biotine] et reliés entre des perles d'agarose par l'intermédiaire de streptavidin ou capturés par des perles magnétiques de streptavidin permet l'isolement des protéines interagissant qui peuvent alors être identifiées par masse spectrométrie ou tache occidentale. Les protocoles décrits ici ont été utilisés pour identifier les protéines de T. brucei3,8 et les cellules de mammifères47 qui se lient à ces métabolites. Des variantes de l'approche qui utilise des étiquettes personnalisées (autres que la biotine) ont également été utilisées dans la levure46. Une considération importante à cette approche est l'utilisation de contrôles pour discriminer spécifiquement des interactions non spécifiques. Les perles non conjuguées sont un contrôle essentiel, mais des contrôles supplémentaires peuvent inclure des PI non phosphorylés ou des inositol conjugués à des perles3. Les IP ou IP avec différentes combinaisons de phosphate3,8 peuvent également être utilisés parce que la liaison de protéines à ces métabolites peut impliquer des domaines qui discriminent la configuration de phosphate de l'inositol38, 39,40,41. En outre, la complexité de l'échantillon peut affecter la sensibilité de l'approche, et donc la diminution de la complexité de l'échantillon par fractionnement de l'échantillon peut aider à la détection de protéines faibles en abondance dans la cellule. Il existe des protocoles bien établis pour la fractionnement cellulaire et l'isolement de la mitochondrie51, noyau52, glycosome53, et flagellum54,55 de trypanosomes. Notez que les tampons et les réactifs utilisés dans les fractionnements subcellulaires peuvent devoir être ajustés pour la compatibilité avec les tampons et les réactifs utilisés dans ce protocole. Notamment, l'identification des protéines qui se lient aux IP ou aux IP par chromatographie d'affinité comme indiqué ici dépend de l'affinité de protéine et de métabolite de l'interaction, et ainsi les protéines qui ont une affinité faible pour des IP ou des PI peuvent ne pas être facilement détectées.
L'analyse de l'interaction IP ou PI avec les protéines du lysate cellulaire peut également entraîner l'identification de protéines qui ne se lient pas directement à ces métabolites, mais dont l'interaction résulte d'une association de protéines en complexe avec d'autres protéines qui se lient aux IP ou DES PI. Cette caractéristique est illustrée dans la figure 3A, dans laquelle RAP1-HA de T. brucei lysate semble se lier à PI(3,4,5)P3-perles et PI(4,5)P2-perles. Cependant, les essais de liaison avec son rRAP1 marqué montrent que cette protéine se lie à PI(3,4,5)P3 et non PI(4,5)P23. Ceci est illustré dans la figure 3B, dans laquelle les essais concurrentiels montrent que l'IP gratuit (3,4,5)P3 mais pas gratuit PI(4,5)P2 concurrence pour rRAP1-son interaction avec PI(3,4,5)P3-perles. L'interaction apparente RAP1 avec PI(4,5)P2-perles est due à l'association RAP1 au sein d'un complexe avec des protéines qui lient PI(4,5)P2 (par exemple, PIP5Pase)3. Par conséquent, les tests de liaison à partir de lysates cellulaires peuvent identifier les protéines qui se lient directement ou indirectement aux ADRESSEs IP ou IP. Notamment, les interactions indirectes sont distinctes des interactions non spécifiques puisque les premiers peuvent avoir une fonction biologique (dans le contexte du complexe protéique) qui affecte ou est affectée par la liaison des métabolites. Par exemple, Ins(1,4,5,6)P4 se lie au complexe multi-subunit co-répresseur deacetylase et contrôle l'assemblage complexe et l'activité13. Par conséquent, il est essentiel de valider les interactions IP/PI avec les protéines. La validation de l'interaction peut impliquer des essais de concurrence avec un excès d'IP ou de IP (comme dans la figure 3B)3,8, mutations de domaines protéiques potentiels56, ou l'utilisation de protéines purifiées pour déterminer les interactions directes (Figure 3A,B)3.
D'autres méthodes d'étude des interactions entre protéines et IP ou IP comprennent la liaison des protéines aux IP ou IP radio-étiquetés, l'utilisation de IP ou de IP liés aux membranes hydrophobes comme matrices pour la capture des protéines, ou la liaison des protéines aux IP incorporées dans les liposomes. 42 Ans, états-unis ( , 57 Annonces , 58. Il est important de noter que si l'interaction protéique avec les IP nécessite des structures membranaires38, les essais à base de liposome peuvent être utilisés comme approche complémentaire. Les limites de ces approches comprennent un faible débit, une faible sensibilité, l'orientation chimique inconnue des IP ou de l'association des IP vers les matrices58, ou l'utilisation de matières radioactives42. La méthode décrite ici est sensible, sans liposome, non-radioactive, et IP / PI-perles sont disponibles dans le commerce, et donc ils ne nécessitent pas de synthèse chimique personnalisée. En outre, la position de la biotine liée aux IP ou aux IP est bien définie, et elle peut également être modifiée46,58, ce qui permet une analyse précise de l'interaction des protéines et des métabolites. La méthode décrite ici peut également être combinée avec des approches quantitatives de spectrométrie de masse telles que l'étiquetage isotopique stable des acides aminés dans la culture cellulaire (SILAC)47, qui peut être utilisé pour identifier les interactions dynamiques sous différents cellulaires traitements ou conditions. La chromatographie d'affinité couplée à la tache occidentale ou à la spectrométrie de masse a aidé à identifier de nombreuses protéines IP- ou PI-contraignantes de T. brucei, de cellules de mammifères, et de levure3,8,46, 47, y compris les protéines qui n'ont pas caractérisé les domaines de liaison IP ou PI, et il a également contribué à l'identification de nouveaux domaines de liaison, par exemple, PI(3,4,5)P3 domaine de liaison47.
Dans l'ensemble, les protocoles décrits ici peuvent être utilisés pour étudier les protéines potentielles de T. bruceien matière d'IP ou d'IP, et pour étudier l'interaction moléculaire des protéines avec ces métabolites. Le protocole peut être facilement adapté pour identifier les protéines liant IP ou PI à partir d'autres parasites unicellulaires ou d'autres organismes tels que les cellules de mammifères47 et la levure46, et il aidera à mieux comprendre la fonction biologique des IP et PIs en eucaryotes.
Déclarations de divulgation
L'auteur n'a rien à révéler.
Remerciements
Ces travaux ont été appuyés par le Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG, RGPIN-2019-04658); Supplément de lancement de découverte du CRSNG pour les chercheurs en début de carrière (DGECR-2019-00081) et par l'Université McGill.
matériels
| Name | Company | Catalog Number | Comments |
| Acetone | Sigma-Aldrich | 650501 | Ketone |
| Acetonitrile | Sigma-Aldrich | 271004 | Solvent |
| Ammonium bicarbonate | Sigma-Aldrich | A6141 | Inorganic salt |
| Centrifuge Avanti J6-MI | Beckman Coulter | Avanti J6-MI | Centrifuge for large volumes (e.g., 1L) |
| Centrifuge botles | Sigma-Aldrich | B1408 | Bottles for centrifugation of 1L of culture |
| Control Beads | Echelon | P-B000-1ml | Affinity chromatography reagent - control |
| D-(+)-Glucose | Sigma-Aldrich | G8270 | Sugar, Added in PBS to keep cells viable |
| Dithiothreitol (DTT) | Bio-Rad | 1610610 | Reducing agent |
| Dynabeads M-270 Streptavidin | ThermoFisher Scientific | 65305 | Streptavidin beads for binding to biotin ligands |
| EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | Protease inhibitors |
| Electrophoresis running buffer | Bio-Rad | 1610732 | 25 mM Tris, 192 mM glycine, 0.1% SDS, pH 8.3 |
| Falcon 15 mL Conical Centrifuge Tubes | Corning Life Sciences | 430052 | To centrifuge 10 mL cultures |
| Formic acid | Sigma-Aldrich | 106526 | Acid |
| Glycine | Sigma-Aldrich | G7126 | Amino acid |
| HMI-9 cell culture medium | ThermoFisher Scientific | ME110145P1 | Cell culture medium for T. brucei bloodstream forms |
| Imperial Protein Stain | ThermoFisher Scientific | 24615 | Coomassie staining for protein detection in SDS/PAGE |
| Ins(1,4,5)P3 Beads | Echelon | Q-B0145-1ml | Affinity chromatography reagent |
| Instant Nonfat Dry Milk | Thomas Scientific | C837M64 | Blocking reagent for Western blotting |
| Iodoacetamide | Sigma-Aldrich | I6125 | Alkylating reagent for cysteine proteins or peptides |
| Lab Rotator | Thomas Scientific | 1159Z92 | For binding assays |
| LoBind Microcentrifuge Tubes | ThermoFisher Scientific | 13-698-793 | Low protein binding tubes for mass spectrometry |
| Nonidet P-40 (Igepal CA-630) | Sigma-Aldrich | 21-3277 | Detergent |
| PBS, pH 7.4 | ThermoFisher Scientific | 10010031 | Physiological buffer |
| Peroxidase substrate for chemiluminescence | ThermoFisher Scientific | 32106 | Substrate for Western bloting detection of proteins |
| PhosSTOP Phosphatase Inhibitor Cocktail Tablets | Roche | 4906845001 | Phosphatase inhibitors |
| PI(3)P PIP Beads | Echelon | P-B003a-1ml | Affinity chromatography reagent |
| PI(3,4)P2 PIP Beads | Echelon | P-B034a-1ml | Affinity chromatography reagent |
| PI(3,4,5)P3 diC8 | Echelon | P-3908-1mg | Affinity chromatography reagent |
| PI(3,4,5)P3 PIP Beads | Echelon | P-B345a-1ml | Affinity chromatography reagent |
| PI(3,5)P2 PIP Beads | Echelon | P-B035a-1ml | Affinity chromatography reagent |
| PI(4)P PIP Beads | Echelon | P-B004a-1ml | Affinity chromatography reagent |
| PI(4,5)P2 diC8 | Echelon | P-4508-1mg | Affinity chromatography reagent |
| PI(4,5)P2 PIP Beads | Echelon | P-B045a-1ml | Affinity chromatography reagent |
| PI(5)P PIP Beads | Echelon | P-B005a-1ml | Affinity chromatography reagent |
| Ponceau S solution | Sigma-Aldrich | P7170 | Protein staining (0.1% [w/v] in 5% acetic acid) |
| Potassium hexacyanoferrate(III) | Sigma-Aldrich | 702587 | Potassium salt |
| PtdIns PIP Beads | Echelon | P-B001-1ml | Affinity chromatography reagent |
| PVDF Membrane | Bio-Rad | 1620177 | For Western blotting |
| Refrigerated centrifuge | Eppendorf | 5910 R | Microcentrifuge for small volumes (e.g., 1.5 mL) |
| Sodium dodecyl sulfate | Sigma-Aldrich | 862010 | Detergent |
| Sodium thiosulfate | Sigma-Aldrich | 72049 | Chemical |
| SpeedVac Vacuum Concentrators | ThermoFisher Scientific | SPD120-115 | Sample concentration (e.g., for mass spectrometry) |
| T175 flasks for cell culture | ThermoFisher Scientific | 159910 | To grow 50 mL T. brucei culture |
| Trypsin, Mass Spectrometry Grade | Promega | V5280 | Trypsin for protein digestion |
| Urea | Sigma-Aldrich | U5128 | Denaturing reagent |
| Vortex | Fisher Scientific | 02-215-418 | For mixing reactions |
| Western blotting transfer buffer | Bio-Rad | 1610734 | 25 mM Tris, 192 mM glycine, pH 8.3 with 20% methanol |
| Whatman 3 mm paper | Sigma-Aldrich | WHA3030861 | Paper for Wester transfer |
| 2-mercaptoethanol (14.2 M) | Bio-Rad | 1610710 | Reducing agent |
| 2x Laemmli Sample Buffer | Bio-Rad | 161-0737 | Protein loading buffer |
| 4–20% Mini-PROTEAN TGX Precast Protein Gels | Bio-Rad | 4561094 | Gel for protein electrophoresis |
| 4x Laemmli Sample Buffer | Bio-Rad | 161-0747 | Protein loading buffer |
Références
- Cestari, I., Stuart, K. Inositol phosphate pathway controls transcription of telomeric expression sites in trypanosomes. Proceedings of the National Academy of Sciences of the United States of America. 112 (21), 2803-2812 (2015).
- Odom, A. R., Stahlberg, A., Wente, S. R., York, J. D. A role for nuclear inositol 1,4,5-trisphosphate kinase in transcriptional control. Science. 287 (5460), 2026-2029 (2000).
- Cestari, I., McLeland-Wieser, H., Stuart, K. Nuclear Phosphatidylinositol 5-Phosphatase Is Essential for Allelic Exclusion of Variant Surface Glycoprotein Genes in Trypanosomes. Molecular and Cellular Biology. 39 (3), (2019).
- Billcliff, P. G., et al. OCRL1 engages with the F-BAR protein pacsin 2 to promote biogenesis of membrane-trafficking intermediates. Molecular Biology of the Cell. 27 (1), 90-107 (2016).
- Brochet, M., et al. Phosphoinositide metabolism links cGMP-dependent protein kinase G to essential Ca(2)(+) signals at key decision points in the life cycle of malaria parasites. PLoS Biology. 12 (3), 1001806(2014).
- Azevedo, C., Szijgyarto, Z., Saiardi, A. The signaling role of inositol hexakisphosphate kinases (IP6Ks). Advances in Enzyme Regulation. 51 (1), 74-82 (2011).
- Szijgyarto, Z., Garedew, A., Azevedo, C., Saiardi, A. Influence of inositol pyrophosphates on cellular energy dynamics. Science. 334 (6057), 802-805 (2011).
- Cestari, I., Anupama, A., Stuart, K. Inositol polyphosphate multikinase regulation of Trypanosoma brucei life stage development. Molecular Biology of the Cell. 29 (9), 1137-1152 (2018).
- Bang, S., et al. AMP-activated protein kinase is physiologically regulated by inositol polyphosphate multikinase. Proceedings of the National Academy of Sciences of the United States of America. 109 (2), 616-620 (2012).
- Seeds, A. M., Tsui, M. M., Sunu, C., Spana, E. P., York, J. D. Inositol phosphate kinase 2 is required for imaginal disc development in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 112 (51), 15660-15665 (2015).
- Hamada, K., Miyatake, H., Terauchi, A., Mikoshiba, K. IP3-mediated gating mechanism of the IP3 receptor revealed by mutagenesis and X-ray crystallography. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), 4661-4666 (2017).
- Watson, P. J., et al. Insights into the activation mechanism of class I HDAC complexes by inositol phosphates. Nature Communications. 7, 11262(2016).
- Watson, P. J., Fairall, L., Santos, G. M., Schwabe, J. W. Structure of HDAC3 bound to co-repressor and inositol tetraphosphate. Nature. 481 (7381), 335-340 (2012).
- Irvine, R. F. Nuclear lipid signalling. Nature Reviews: Molecular Cell Biology. 4 (5), 349-360 (2003).
- Sobol, M., et al. UBF complexes with phosphatidylinositol 4,5-bisphosphate in nucleolar organizer regions regardless of ongoing RNA polymerase I activity. Nucleus. 4 (6), 478-486 (2013).
- Nascimbeni, A. C., et al. ER-plasma membrane contact sites contribute to autophagosome biogenesis by regulation of local PI3P synthesis. EMBO Journal. 36 (14), 2018-2033 (2017).
- Martin, K. L., Smith, T. K. The myo-inositol-1-phosphate synthase gene is essential in Trypanosoma brucei. Biochemical Society Transactions. 33, Pt 5 983-985 (2005).
- Matsu-ura, T., et al. Cytosolic inositol 1,4,5-trisphosphate dynamics during intracellular calcium oscillations in living cells. Journal of Cell Biology. 173 (5), 755-765 (2006).
- Blind, R. D., et al. The signaling phospholipid PIP3 creates a new interaction surface on the nuclear receptor SF-1. Proceedings of the National Academy of Sciences of the United States of America. 111 (42), 15054-15059 (2014).
- Lin, A., et al. The LINK-A lncRNA interacts with PtdIns(3,4,5)P3 to hyperactivate AKT and confer resistance to AKT inhibitors. Nature Cell Biology. 19 (3), 238-251 (2017).
- Steger, D. J., Haswell, E. S., Miller, A. L., Wente, S. R., O'Shea, E. K. Regulation of chromatin remodeling by inositol polyphosphates. Science. 299 (5603), 114-116 (2003).
- York, J. D., Odom, A. R., Murphy, R., Ives, E. B., Wente, S. R. A phospholipase C-dependent inositol polyphosphate kinase pathway required for efficient messenger RNA export. Science. 285 (5424), 96-100 (1999).
- Adams, R. L., Mason, A. C., Glass, L., Aditi,, Wente, S. R. Nup42 and IP6 coordinate Gle1 stimulation of Dbp5/DDX19B for mRNA export in yeast and human cells. Traffic. 18 (12), 776-790 (2017).
- Macbeth, M. R., et al. Inositol hexakisphosphate is bound in the ADAR2 core and required for RNA editing. Science. 309 (5740), 1534-1539 (2005).
- Dickson, E. J., Hille, B. Understanding phosphoinositides: rare, dynamic, and essential membrane phospholipids. Biochemical Journal. 476 (1), 1-23 (2019).
- Mellman, D. L., et al. A PtdIns4,5P2-regulated nuclear poly(A) polymerase controls expression of select mRNAs. Nature. 451 (7181), 1013-1017 (2008).
- Lee, Y. S., Mulugu, S., York, J. D., O'Shea, E. K. Regulation of a cyclin-CDK-CDK inhibitor complex by inositol pyrophosphates. Science. 316 (5821), 109-112 (2007).
- Nagy, A. I., et al. IP3 signalling regulates exogenous RNAi in Caenorhabditis elegans. EMBO Reports. 16 (3), 341-350 (2015).
- Hall, B. S., et al. TbVps34, the trypanosome orthologue of Vps34, is required for Golgi complex segregation. Journal of Biological Chemistry. 281 (37), 27600-27612 (2006).
- Rodgers, M. J., Albanesi, J. P., Phillips, M. A. Phosphatidylinositol 4-kinase III-beta is required for Golgi maintenance and cytokinesis in Trypanosoma brucei. Eukaryotic Cell. 6 (7), 1108-1118 (2007).
- Gilden, J. K., et al. The role of the PI(3,5)P2 kinase TbFab1 in endo/lysosomal trafficking in Trypanosoma brucei. Molecular and Biochemical Parasitology. 214, 52-61 (2017).
- Demmel, L., et al. The endocytic activity of the flagellar pocket in Trypanosoma brucei is regulated by an adjacent phosphatidylinositol phosphate kinase. Journal of Cell Science. 129 (11), 2285(2016).
- Gimenez, A. M., et al. Phosphatidylinositol kinase activities in Trypanosoma cruzi epimastigotes. Molecular and Biochemical Parasitology. 203 (1-2), 14-24 (2015).
- Hashimoto, M., et al. Inositol 1,4,5-trisphosphate receptor regulates replication, differentiation, infectivity and virulence of the parasitic protist Trypanosoma cruzi. Molecular Microbiology. 87 (6), 1133-1150 (2013).
- Cestari, I., Haas, P., Moretti, N. S., Schenkman, S., Stuart, K. Chemogenetic Characterization of Inositol Phosphate Metabolic Pathway Reveals Druggable Enzymes for Targeting Kinetoplastid Parasites. Cell Chemical Biology. 23 (5), 608-617 (2016).
- Lovett, J. L., Marchesini, N., Moreno, S. N., Sibley, L. D. Toxoplasma gondii microneme secretion involves intracellular Ca(2+) release from inositol 1,4,5-triphosphate (IP(3))/ryanodine-sensitive stores. Journal of Biological Chemistry. 277 (29), 25870-25876 (2002).
- McNamara, C. W., et al. Targeting Plasmodium PI(4)K to eliminate malaria. Nature. 504 (7479), 248-253 (2013).
- Tresaugues, L., et al. Structural basis for phosphoinositide substrate recognition, catalysis, and membrane interactions in human inositol polyphosphate 5-phosphatases. Structure. 22 (5), 744-755 (2014).
- Varnai, P., et al. Quantifying lipid changes in various membrane compartments using lipid binding protein domains. Cell Calcium. 64, 72-82 (2017).
- Lystad, A. H., Simonsen, A. Phosphoinositide-binding proteins in autophagy. FEBS Letters. 590 (15), 2454-2468 (2016).
- Cullen, P. J., Cozier, G. E., Banting, G., Mellor, H. Modular phosphoinositide-binding domains--their role in signalling and membrane trafficking. Current Biology. 11 (21), 882-893 (2001).
- Klarlund, J. K., et al. Signaling by phosphoinositide-3,4,5-trisphosphate through proteins containing pleckstrin and Sec7 homology domains. Science. 275 (5308), 1927-1930 (1997).
- Wild, R., et al. Control of eukaryotic phosphate homeostasis by inositol polyphosphate sensor domains. Science. 352 (6288), 986-990 (2016).
- Gerasimaite, R., et al. Inositol Pyrophosphate Specificity of the SPX-Dependent Polyphosphate Polymerase VTC. ACS Chemical Biology. 12 (3), 648-653 (2017).
- Potapenko, E., et al. 5-Diphosphoinositol pentakisphosphate (5-IP7) regulates phosphate release from acidocalcisomes and yeast vacuoles. Journal of Biological Chemistry. 293 (49), 19101-19112 (2018).
- Wu, M., Chong, L. S., Perlman, D. H., Resnick, A. C., Fiedler, D. Inositol polyphosphates intersect with signaling and metabolic networks via two distinct mechanisms. Proceedings of the National Academy of Sciences of the United States of America. 113 (44), 6757-6765 (2016).
- Jungmichel, S., et al. Specificity and commonality of the phosphoinositide-binding proteome analyzed by quantitative mass spectrometry. Cell Reports. 6 (3), 578-591 (2014).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137 (1), 99-109 (2009).
- Cestari, I., Stuart, K. Transcriptional Regulation of Telomeric Expression Sites and Antigenic Variation in Trypanosomes. Current Genomics. 19 (2), 119-132 (2018).
- Clough, T., Thaminy, S., Ragg, S., Aebersold, R., Vitek, O. Statistical protein quantification and significance analysis in label-free LC-MS experiments with complex designs. BMC Bioinformatics. 13, Suppl 16 6(2012).
- Schneider, A., Charriere, F., Pusnik, M., Horn, E. K. Isolation of mitochondria from procyclic Trypanosoma brucei. Methods in Molecular Biology. 372, 67-80 (2007).
- DeGrasse, J. A., Chait, B. T., Field, M. C., Rout, M. P. High-yield isolation and subcellular proteomic characterization of nuclear and subnuclear structures from trypanosomes. Methods in Molecular Biology. 463, 77-92 (2008).
- Opperdoes, F. R. A rapid method for the isolation of intact glycosomes from Trypanosoma brucei by Percoll -gradient centrifugation in a vertical rotor. Molecular and Biochemical Parasitology. 3 (3), 181-186 (1981).
- Pereira, N. M., de Souza, W., Machado, R. D., de Castro, F. T. Isolation and properties of flagella of trypanosomatids. The Journal of protozoology. 24 (4), 511-514 (1977).
- Subota, I., et al. Proteomic analysis of intact flagella of procyclic Trypanosoma brucei cells identifies novel flagellar proteins with unique sub-localization and dynamics. Molecular and Cellular Proteomics. 13 (7), 1769-1786 (2014).
- Fukuda, M., Kojima, T., Kabayama, H., Mikoshiba, K. Mutation of the pleckstrin homology domain of Bruton's tyrosine kinase in immunodeficiency impaired inositol 1,3,4,5-tetrakisphosphate binding capacity. Journal of Biological Chemistry. 271 (48), 30303-30306 (1996).
- Knodler, A., Mayinger, P. Analysis of phosphoinositide-binding proteins using liposomes as an affinity matrix. BioTechniques. 38 (6), (2005).
- Best, M. D. Global approaches for the elucidation of phosphoinositide-binding proteins. Chemistry and Physics of Lipids. 182, 19-28 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon