Method Article
Hibridização In Situ de Embriões Inteiros de Astyanax
Neste Artigo
Resumo
Este protocolo permite a visualização da expressão gênica em embrionárias Astyanax cavefish. Esta abordagem foi desenvolvida com o objetivo de maximizar o sinal de expressão do gene, minimizando a coloração de fundo específico.
Resumo
Nos últimos anos, foi lançado um genoma de projecto para o cego mexicano cavefish (Astyanax mexicanus), revelando as identidades de sequência para milhares de genes. Pesquisas anteriores neste sistema modelo emergente capitalizou investigações abrangentes de todo o genoma que identificaram numerosos loci de traço quantitativos (QTL) associado com vários fenótipos associados a caverna. No entanto, a capacidade de conectar os genes de interesse para a base hereditária para mudança fenotípica permanece um desafio significativo. Uma técnica que pode facilitar a compreensão mais profunda do papel do desenvolvimento na evolução troglomorphic é toda a montagem hibridação in situ. Esta técnica pode ser implementada para diretamente comparar a expressão gênica entre as formas e superfície-habitações na caverna, nomear candidato genes subjacentes QTL estabelecida, identificar os genes de interesse em estudos de sequenciamento de nova geração ou desenvolver outros abordagens de descoberta. Neste relatório, nós apresentamos um protocolo simples, apoiado por uma lista de verificação flexível, que pode ser amplamente adaptada para o uso bem além do sistema de estudo apresentado. Espera-se que este protocolo pode servir como um grande recurso para a comunidade de Astyanax e além.
Introdução
Hibridação in situ é um método comum para coloração de tecidos fixos para visualizar de padrões de expressão de gene1. Esta técnica tem sido realizada há anos em outros tradicionais2 e não-tradicionais3 sistemas de modelo, para uma variedade de estudos biológicos. No entanto, várias etapas e os reagentes são necessários para executar com êxito este procedimento. Para os investigadores que nunca tenham realizado esta técnica, iniciar o processo pode ser intimidante, devido as muitas etapas envolvidas. Além disso, a natureza longa deste procedimento presta-se a erros técnicos, que podem ser um desafio para solucionar problemas.
O objetivo geral deste artigo é apresentar um método simples e direto que irá processar esta técnica de hibridização acessível a um vasto público. Para reduzir a introdução de erros, nós apresentamos uma abordagem direta que produz coloração de expressão do gene de alta qualidade e minimiza o sinal de fundo específico. Este procedimento é semelhante a outras abordagens desenvolvidas em sistemas modelo tradicional, como Danio rerio4. Aqui, pretendemos facilitar a aplicação cuidadosa de cada etapa usando uma lista de verificação para download (arquivo suplementar 1), para promover a aplicação cuidadosa do protocolo. A justificativa para fazer isto é para facilitar a organização através dos muitos passos envolvidos neste procedimento. Este artigo é apropriado para pesquisadores interessados em realizar toda a montagem hibridização in situ no desenvolvimento de embriões, mas ainda não ter realizado o procedimento. A vantagem da abordagem escolhida por pesquisadores de Astyanax é que tem sido testada e comprovada em cavefish e morfos de peixe de superfície, facilitando a análise comparativa da expressão. O método apresentado pode ser usado por pesquisadores em estudos sobre Astyanax e outros sistemas.
Protocolo
Todos os métodos descritos aqui foram aprovados pelo cuidado institucional do Animal e Comissão de utilização (IACUC) da University of Cincinnati (protocolo #10-01-01-21).
1. fixação
- Isolar o número desejado de Astyanax mexicanus embriões de um tanque de reprodução e corrigir ~ 50 embriões de cada vez. Se os embriões são grandes e antigos, pode ser necessário fixar 25 ao mesmo tempo para garantir a fixação do mesmo.
- Dependendo da idade do embrião, utilize o método aprovado IACUC de anestesia. Para os mais antigos embriões com um funcionamento do sistema nervoso, sacrifica embriões através de overdose de anestésico. Nesse sentido, coloca embriões em uma solução de ~ 1% metanosulfonato (tamponada de pH 7,4) para minimizar a dor e desconforto para o organismo.
- Uma vez que os embriões não respondem ao toque, sistema de água contendo tricaina e adicionar ~ 1 mL de 1x tampão fosfato salino (PBS, pH 7,4).
- Remover a solução de PBS e adicionar 1 mL de paraformaldeído 4% (PFA). Fixar os embriões durante a noite a 4 ° C.
Atenção: PFA é perigosa (ou seja, é inflamável e é uma pele e irritantes do pulmão), manuseie com cuidado.
2. desidratação
- Para desidratar os embriões, retire a fixador solução e enxágue com 1 mL de PBS. Coloque os frascos contendo embriões em um ângulo (entre 30° e 45°), num agitador de plataforma durante o enxágue. Continue a lavar os embriões duas vezes, 5 min por lavagem.
- Se os embriões ainda têm um córion, coloque todos os 50 embriões em uma 100 x 25 mm placa de Petri e cuidadosamente, isolá-los dos chorions usando dois conjuntos de pinças de relojoeiro (por exemplo, #5 pinças) sob um microscópio.
Nota: Durante as etapas descritas abaixo e para o restante do protocolo, cuidadosamente retire todo o líquido do passo anterior usando limpa, pipetas de Pasteur de vidro antes de adicionar a seguinte solução - Desidrata-se os embriões em uma série de lavagens cada vez mais concentradas de metanol (MeOH) descrito abaixo. As diluições são baseadas em um volume total de 1 mL com 500 µ l de solução em cada frasco de vidro de 4 mL. Execute todas as etapas de desidratação em temperatura ambiente (RT) agitador de plataforma.
- Remova cuidadosamente a solução de PBS. Adicione uma solução de MeOH de 25% (250 µ l de MeOH) + 750 µ l de PBS. Agite suavemente num agitador de plataforma por 5 min.
- Remova cuidadosamente a solução de MeOH 25%. Adicione uma solução de 50% MeOH (500 µ l de MeOH) + 500 µ l de PBS. Agite suavemente num agitador de plataforma por 5 min.
- Remova cuidadosamente a solução de MeOH 50%. Adicione uma solução de MeOH 75% (750 µ l de MeOH) + 250 µ l de PBS. Agite suavemente num agitador de plataforma por 5 minutos.
- Remova cuidadosamente a solução de MeOH 75%. Adicione uma solução de MeOH 100% (1 mL de MeOH). Agite suavemente num agitador de plataforma 5 min. Repita este passo 3 vezes.
- Neste ponto, armazene embriões desidratados, conforme necessário, em seus frascos de vidro a-20 ° C (longo prazo). Alternativamente, vá direto ao dia 1 do protocolo.
3. dia 1: reidratação
- Obter embriões desidratados do congelador-20 ° C (ou proceder directamente da etapa 2.5).
- Classificar os embriões com uma pipeta Pasteur. Um pode classificar com base no Morfotipo (i.e., caverna e/ou superfície), e o número de genes avaliados em cada experimento. Há normalmente não mais que 12 embriões por frasco uma vez classificados. Para manter a organização, use fita colorida laboratório para designar os frascos e pipetas para cada gene. Os embriões vão ficar no mesmo frasco em todo o protocolo inteiro.
Nota: Lugar a ponta da pipeta Pasteur em 100% EtOH para esterilizar entre usa. - Defina um tremendo banho de água a 70 ° C, para ser usado em uma etapa posterior. Cuidadosamente, retire o MeOH em frascos de embriões classificados e substituir com 500 µ l de novo 100% MeOH. Lavagem brevemente (~ 1 min) agitador de plataforma.
- Reidratar embriões em uma concentração crescente de 1X PBS com Tween 20 (PBT, veja abaixo), agitador de plataforma. As diluições são baseadas em um volume de diluição final de 1 mL com 500 µ l em cada frasco.
- Adicione uma solução PBT de 25% (250 µ l de PBT, 750 µ l de MeOH). Agite suavemente num agitador de plataforma por 5 min.
- Remova cuidadosamente a solução PBT de 25%. Adicione uma solução PBT de 50% (500 µ l de PBT, 500 µ l de MeOH). Agite suavemente num agitador de plataforma por 5 min.
- Remova cuidadosamente a solução PBT de 50%. Adicione uma solução PBT de 75% (750 µ l de PBT, 250 µ l de MeOH). Agite suavemente num agitador de plataforma por 5 min.
- Remova cuidadosamente a solução PBT de 75%. Adicione uma solução PBT de 100% (1 mL de PBT). Agite suavemente num agitador de plataforma 5 min. Repita este passo 3 vezes.
4. dia 1: Digestão e fixação
- Prepare uma solução de K (PK) de proteinase adicionando 1 µ l de PK (20 mg/mL) a 2 mL de PBT.
- Em antecipação de etapas subsequentes, obter alíquotas congeladas de buffers de hibridação (Hyb - e Hyb +; ver suplementar arquivo 2 e 3 de arquivo suplementar) e PFA de armazenamento-20 ° C.
- Permitir que o PFA descongelar no RT
- Colocar alíquotas de Hyb - e Hyb + num banho rotativo-70 ° C. Coloque todos os reagentes e os frascos dentro de um pequeno "junta" com um fundo de malha, no interior do aparelho de banho de água flutuante. Isso permite que a simples adição e remoção de tubos e frascos do banho-maria a 70 ° C rotativa.
- Delicadamente, adicione solução de PK para o conj de embriões, garantindo a todos os tecidos são completamente cobertos com solução. Digerir embriões para ~ 12 min em solução de trabalho PK o agitador de plataforma.
Nota: O comprimento da digestão pode ser variado pelo investigador para garantir melhores resultados. - Delicadamente, retirar a solução PK e brevemente inundar o frasco com PBT para diluir qualquer restante PK
- Retirar a solução PBT e substituir com 500 µ l de PBT de novo. Permitir que a solução enxaguar o agitador de plataforma por 5 min.
- Trasfegar PBT e substituir com 500 µ l de descongelado 4% PFA. Permitir que os embriões incubar por 20 min o agitador de plataforma no RT
- Retirar os 4% PFA e brevemente inundar o frasco com PBT para diluir qualquer restante PFA. Trasfegar o PBT e substitua com 500 µ l de PBT fresco. Permitir que os embriões enxaguar durante 5 min o agitador de plataforma. Repita esta etapa 4 vezes mais.
5. dia 1: Prehybridization
- Lugar de 500 µ l de pré-aquecido Hyb-solução dentro do frasco. Cuidadosamente coloque o frasco na 70 ° C água banho (dentro de gaxetas) sem tremer, por 5 min.
- Trasfegar a Hyb-solução e inundar o frasco com 500 µ l de solução Hyb + previamente aquecida. Coloque o frasco volta em banho de água a 70 ° C com agitação (40 rpm). Incubar durante qualquer 4 h, ou uma noite.
Nota: Uma incubação de 4h renderá um protocolo in-situ completo que irá durar por 4 dias no total. Aqui, esta etapa é apresentada como uma incubação durante a noite, que renderá um protocolo durou 5 dias no total.
6. dia 2: hibridização
- Coloque uma parte alíquota do Hyb + do congelador-20 ° C para o tremendo banho de água quente por 5 min.
- Trasfegar o Hyb + do frasco e substituir com 500 µ l de pré-aquecido Hyb +. Para esta solução, cuidadosamente adicione 2 µ l de sonda RNA a cada frasco. Agite suavemente o frasco para assegurar uma distribuição uniforme da sonda.
- Incube a solução Hyb + (com sonda adicionada) em banho de água quente a 70 ° C durante a noite, agitando a 40 rpm.
Nota: Um pode re-utilizar solução Hyb + (com sonda). Para isso, ter Hyb + com sonda da primeira execução do congelador-20 ° C e coloque num banho de água quente durante 5 min. Replace Hyb + desde o dia 1 protocolo com Hyb + com sonda e permitir a incubação durante a noite no banho de água quente.
7. dia 3: Preparação de solução
- Prepare tubos microcentrifuga rotulados Hyb + com a sonda de RNA "gene de interesse". Prepare a série de diluições que serão usadas durante o dia 3.
- Usando 6 tubos separados, preparar a seguinte série de diluições do Hyb - e citrato de sódio salino (CCD, 0 a 100%) em um volume de 1 mL e lugá-los na 70 ° C a tremer água de banho: 1 = 100% de tubo Hyb-(1 mL de Hyb-): tubo 2 = 25% 2 x SSC (250 µ l de 2 x SSC, 750 µ l de Hyb-); Tubo 3 = 50% 2 x SSC (500 µ l de 2 x SSC, 500 µ l do Hyb-); Tubo 4 = 75% 2 x SSC (750 µ l de 2 x SSC, 250 µ l de Hyb-); Tubo de CCD de 2 x 5 = 100% (1 mL de 2 x SSC); Tubo de 6 = 100% x 0.2 SSC (2 mL de 0,2 x SSC).
Nota: Ser vigilante da concentração do SSC, como muda de 2x para 0.2 x. - Usando 4 separar os tubos, preparar a seguinte série de diluições de PBT e CCD em um volume de 1 mL e coloque no RT: tubo 1 = 25% PBT (250 µ l de PBT, 750 µ l de 0.2 x SSC); Tubo 2 = 50% PBT (500 µ l de PBT, 500 µ l de 0,2 x SSC); Tubo 3 = 75% PBT (750 µ l de PBT, 250 µ l de 0,2 x SSC); Tubo de PBT 4 = 100% (1mL de PBT).
- Prepare um tubo com 2 mL de tampão de ácido maleico, contendo solução de trabalho de Tween 20 (MABT).
- Prepare dois tubos cónico de 15 mL de solução de bloqueio. Em cada tubo, adicionar 0,2 g de reagente de bloqueio para 10 mL de MABT (ver 4 de arquivo suplementar). Coloque os dois tubos um misturador de oscilador (ou agitador de plataforma) até completamente dissolvido em solução (até 3 h).
- Usando 6 tubos separados, preparar a seguinte série de diluições do Hyb - e citrato de sódio salino (CCD, 0 a 100%) em um volume de 1 mL e lugá-los na 70 ° C a tremer água de banho: 1 = 100% de tubo Hyb-(1 mL de Hyb-): tubo 2 = 25% 2 x SSC (250 µ l de 2 x SSC, 750 µ l de Hyb-); Tubo 3 = 50% 2 x SSC (500 µ l de 2 x SSC, 500 µ l do Hyb-); Tubo 4 = 75% 2 x SSC (750 µ l de 2 x SSC, 250 µ l de Hyb-); Tubo de CCD de 2 x 5 = 100% (1 mL de 2 x SSC); Tubo de 6 = 100% x 0.2 SSC (2 mL de 0,2 x SSC).
8. dia 3: Remoção de sonda
- Trasfegar Hyb + (com sonda) solução com uma pipeta Pasteur de vidro e coloque-o em uma estéril, rotulado microcentrifuga tubo. Manter este tubo no congelador-20 ° C para uso futuro (se sonda-rotulagem é bem sucedida).
- Cuidadosamente, adicione 500 µ l das SSC quentes/Hyb-diluições (indicado abaixo). Incube em cada uma das seguintes soluções por 10 min em banho de água agitando a 70 ° C.
- Incubar em sequência com 100% Hyb-(1ml de Hyb-), 25% 2 x SSC (250 µ l de 2 x SSC, 750 µ l de Hyb-), 50% 2 x SSC (500 µ l de 2 x SSC, 500 µ l do Hyb-), 75% 2 x SSC (750 µ l de 2 x SSC, 250 µ l de Hyb-), 100% 2 x SSC (1ml de 2 x SSC) , 100% 0.2 x SSC (2 mL de 0.2 x SSC).
- Após a última etapa, incubar em cada uma das seguintes soluções para 10 min cada. Todos a incubação do seguintes ocorrem em RT o agitador de plataforma: 25% PBT (250 µ l de PBT, 750 µ l de 0.2 x SSC), 50% PBT (500 µ l de PBT, 500 µ l de 0.2 x SSC), 75% PBT (750 µ l de PBT, 250 µ l de 0.2 x SSC) , 100% PBT (1 mL de PBT).
- Após uma incubação de 10 min, retire o 100% PBT e adicione 500 µ l de MABT em cada frasco. Repita duas vezes por 5 min.
9. dia 3: bloqueio
- Remova MABT de cada frasco e inundação com pré-misturado solução bloqueio de um dos tubos (preparados na etapa 7.1.4). Coloque o frasco em um misturador de oscilador para ~ 4 h no RT
- Adicionar 2 µ l de DIG-AP-Fab fragmentos no segundo frasco de 10 mL de solução (preparada na etapa 7.1.4) e brevemente o vórtice de bloqueio.
- Encha cada frasco quase completamente com bloqueio (~ 5 mL) de solução e coloque no misturador de oscilador durante a noite no frigorífico a 4 ° C.
10. dia 4: MABT lavagens
- Preparar um frasco estoque de 10% de soro de cabra normal (NGS) em MABT (adicionar 100 µ l de NGS para 900 µ l de MABT).
- Retirar a solução de bloqueio em cada frasco e adicione 500 µ l de mistura NGS/MABT em cada frasco. Permitir que os embriões incubar durante 25 min no RT o agitador de plataforma.
- Substituir a mistura NGS/MABT com 500 µ l de 100% MABT. Incube durante 30 min à RT o agitador de plataforma. Execute este enxaguamento 11 vezes mais ao longo do dia cada 30 min.
- Encha o frasco com 100% MABT e coloque em um misturador de oscilador durante a noite em uma câmara de geladeira ou frigorífico a 4 ° C.
11. dia 5: Visualização da ponta de prova
- Prepare-se uma alíquota de 50 mL de tampão de fosfatase alcalina (AP) (ver 5 de arquivo suplementar). Combinar o seguinte em um tubo cônico de 50 mL, embrulhado em papel de alumínio para limitar a exposição à luz: 5 mL de 1 M Tris (pH 9,5), 5 mL de 50mm MgCl2, 5ml de 1% Tween 20, 5ml de 1 M NaCl, 30 mL de ddH2O.
- Remover MABT e substitua por 1 mL de tampão de AP (tubo envolvido em papel alumínio). Deixe lavar por 5 min. Fazer isso duas vezes para assegurar a remoção completa dos MABT.
- Retire o tampão do AP e substituir com 1 mL de tampão de AP com 5-bromo-4-chloro-3'-indolyphosphate 3.5 μL (BCIP) e 4.5 μL de nitro-azul de tetrazólio (NBT). Substitua com AP recentemente preparada amortecedor/NBT/BCIP uma vez a cada hora até a reação é completa. Acompanhar de perto, verificando a cada 15 minutos, para permitir que a reação de coloração ser feita até o nível desejado de coloração foi alcançado. Se o precipitado começa a se formar, substitua a solução mais cedo.
- Pare a reação de coloração enxaguando os embriões no buffer de AP 100% fresco (sem NBT/BCIP) por 5 min. Continue lavagens em PBT até que sejam alcançados os níveis ideais de sinal (com quantidades mínimas de coloração de fundo). Continue enxaguando espécimes com crescentes diluições de PBT no Buffer de AP da seguinte forma: 25% PBT (250 μL de PBT, 750 μL de tampão, AP), enxaguar por 5 min; 50% PBT (500 μL de PBT, 500 μL de tampão, AP), enxaguar por 5 min; 75% PBT (750 μL de PBT, 250 μL de tampão, AP), enxaguar por 5 min.
- Enxágue os embriões em ~ 5 mL de 100% PBT no oscilador misturador até desejada de coloração de fundo mínimo é alcançado. Alternar com PBT fresco várias vezes. Isto pode demorar até vários dias.
- Quando é uma lavagem completos, lavagem de embriões em 500 µ l de PBS estéril num agitador de plataforma. Execute este enxaguar duas vezes por 5 min. Depois lava-PBS, fixar os espécimes em 500 μL de 4% PFA por 1h no RT num agitador de plataforma. Alternativamente, consertar a noite em 1 mL de 4% PFA na geladeira a 4 ° C.
- Substitua o fixador fresco, PBS estéril. Executar este enxágue pelo menos duas vezes por 5 min. do lugar os embriões em ~ 4 mL de PBS estéril de 100% e armazenar a longo prazo a 4 ° C.
12. imaging
- Compõem uma placa de imagem em uma placa de Petri usando 3% de agarose e tampão TAE.
Nota: As quantidades dependem de quantas placas são necessárias. As placas podem ser reutilizadas várias vezes. É recomendável que um molde retangular raso é colocado na caixa de Petri, enquanto o gel está esfriando a fim de criar uma depressão por conter os embriões na placa. - Coloque os embriões na chapa na PBS.
Nota: Recomenda-se delicadamente despeje embriões na placa em vez de pipetagem-los porque verificou-se que eles vão ficar com o interior de pipetas de plástico. - Microscopia de luz de uso para visualizar cada embrião. Use uma ponta de prova contundente para manobrar os embriões para a posição desejada.
- Tirar uma imagem quando o embrião está na posição desejada. Observe que é importante ter imagens de embriões dentro de algumas semanas de coloração concluído para evitar a degradação do potencial da mancha.
Resultados
Neste relatório, nós fornecemos uma abordagem simples e direta para executar a rotulagem de espécimes de Astyanax embrionárias para análise de expressão do gene de alta qualidade. Esta técnica pode ser realizada em quatro ou cinco dias, e cada etapa principal do procedimento é representada em um fluxograma com codificação de cores (Figura 1). Depois de concluído, manchada de embriões devem abrigar um rótulo cromático roxo escuro em tecidos que expressa o gene específico de interesse. Nós implementamos com sucesso este protocolo em Pachón cavefish (Figura 2A–B, E) e embriões de peixes de superfície (Figura 2–D, F).
Os embriões cavefish foram corados por dois fatores de transcrição que tecidos de crista neural precoce rótulo, Sox9 e Tfap2a5,6. Embriões rotulados para expressão Sox9 demonstram rotulagem clara no desenvolvimento arcos branquial e barbatana peitoral (setas amarelas, Figura 2A). Observe que a coloração é praticamente ausente no saco vitelino ou as somitas em desenvolvimento no flanco (Figura 2A). Da mesma forma, a expressão de Tfap2a é evidente nas porções de cabeça em desenvolvimento, os primeiros migração células crista neural (Figura 2B, ponta de flecha) ao longo da região dorsal do flanco do embrião. O terceiro gene representante apresentado para embriões cavefish é Phf20a, um marcador de diferenciação de osteoblastos7. Observe a coloração positiva nas porções da mesoderme somitic e cabeça posterior que está destinadas a dar origem ao tecido ósseo (Figura 2E, pontas de seta).
Em embriões de peixe de superfície, nós analisados os genes CXCR, Adcyap1ae Sox10. CXCR codifica um receptor de membrana-limite G-proteína que se liga de quimiocinas CXC8. A rotulagem positivo está presente em regiões isoladas da cabeça e flanco (Figura 2F, pontas de seta), bem como algumas células individuais, sobrepondo-se o saco vitelino. O gene ativando adenilato ciclase polipeptídeo, Adcyap1a, manifesta-se nas regiões do sistema nervoso central, incluindo as células da hipófise. Observe a expressão altamente específica em clusters emparelhados, bilaterais de células no dorso do embrião; assim como uma maior região de expressão mediana (Figura 2, pontas de seta). Finalmente, apresentamos a expressão do Sox10, um fator de transcrição quais rótulos cedo crista neural e oligodendrocyte células10. Coloração positiva altamente específico é evidente como marcador precoce da crista neural à esquerda e à direita do embrião dorsal (Figura 2D, pontas de seta).
Apresentamos cada um dos dois tipos de confundimento questões que outros investigadores podem encontrar. A primeira questão que se encontra periodicamente é punctate manchas de rotulagem não-específica. Estas manchas podem surgir como precipitado desde as final MABT lavagens, ou o buffer de AP durante as reações de coloração. Um exemplo desse rótulo específico é evidente sobre o saco vitelino de um embrião de peixe de superfície manchado para expressão de Pnp4a. Este gene codifica uma enzima (fosforilase de purina nucleosídeo) que facilita a produção de pigmentação iridescente11. Este gene é primeiramente evidente nos olhos em desenvolvimento e a bexiga natatória. As partículas punctate observaram em algumas amostras de superfície (Figura 3A), foram eliminados por lavagens frequentes e substituição do AP Buffer + NBT/BCIP na fase final do protocolo (Figura 3B). Uma segunda questão que se encontra periodicamente é a expressão difusa, em grande parte não-específicos de genes que caso contrário iria produzir um padrão de expressão distintas. Um exemplo é o gene BMP4, que aparece como um padrão amplamente difuso com baixos níveis de cromogénio presente em toda a amostra (Figura 3). Em casos como estes, nós geralmente identificar uma região diferente do gene, amplificar em um vetor e realizar uma nova síntese de sonda (ver 6 de arquivo suplementar). O exemplo de uma amostra de controle (sem sonda) (Figura 3D) é fornecido para ilustrar a natureza difusa e inespecífica de nossa sonda BMP4 falhou.
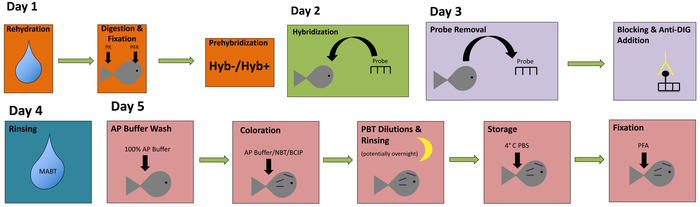
Figura 1: um fluxograma simples para hibridação in situ de toda a montagem. Este fluxograma utiliza a codificação de cores para ilustrar as etapas principais de hibridação in situ. Clique aqui para ver uma versão maior desta figura.
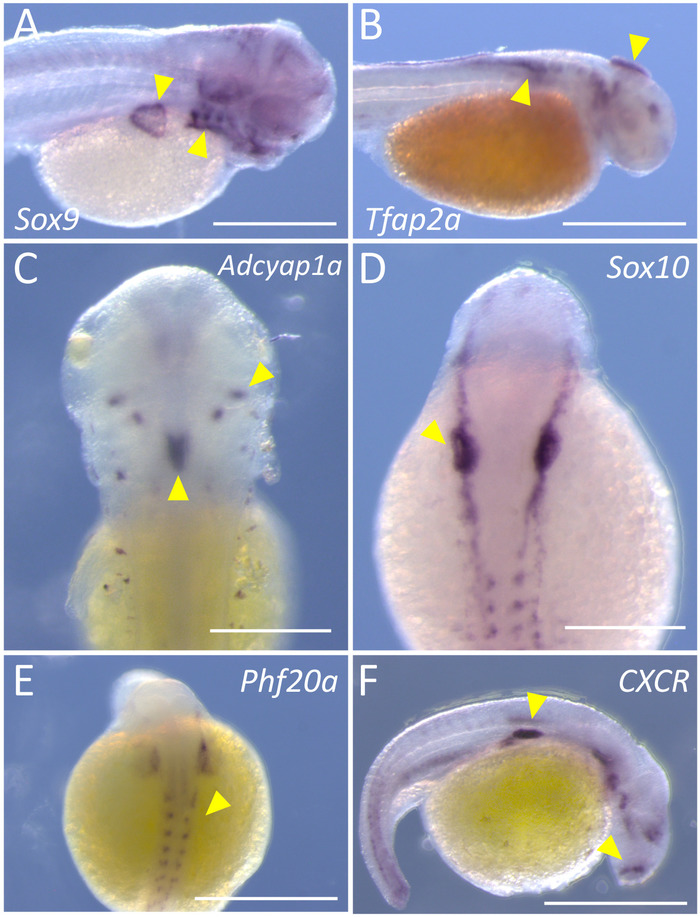
Figura 2: Coloração representativo para seis genes, usando tanto a caverna e a superfície morfos de Astyanax. Imagem do (A) mostra a coloração específica (setas amarelas) de Sox9 no lado lateral direita de uma pós-fertilização 72 h (hpf) Pachón cavefish (x 45). (B) coloração específica para Tfap2a é evidente no lado lateral direita de um 36 hpf Pachón cavefish (x 45). (C) Bilateral e linha média coloração (setas amarelas) de Adcyap1a são rotulados na região dorsal de um peixe de superfície hpf e 72 (100 x). (D) coloração de Sox10 em tecidos de crista neural de um peixe de superfície hpf e 24 (100 x). (E) Phf20a demonstra um fraco, mas claro, padrão de expressão na região dorsal de um 24 hpf Pachón cavefish (100 x). (F) o cytokine receptor, CXCR, é expressa em regiões distintas do lado lateral direita de um peixe de superfície hpf e 24 (100 x). Barras de escala em A, B, E, F = 0,5 mm; dimensionamento de barras em C, D = 2,5 mm. clique aqui para ver uma versão maior desta figura.
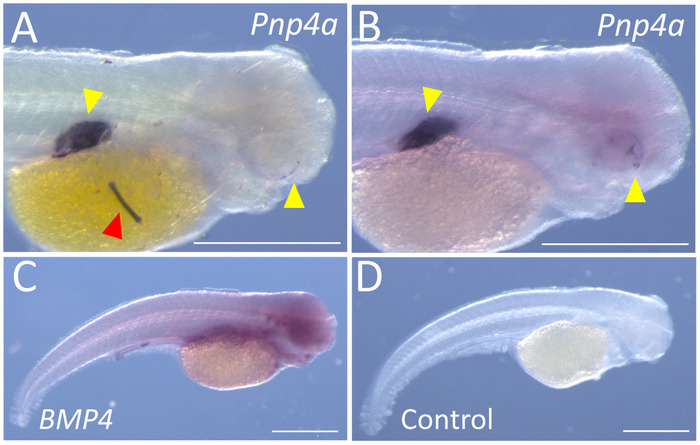
Figura 3: representativos exemplos de resultados abaixo do ideal para toda a montagem de hibridação in situ. (A) coloração específica é evidente ao lado específico precipitado e/ou detritos (seta vermelha) na face lateral direita de um 72 hpf Pachón cavefish (100 x). (B) a mesma sonda visualizada na, retratando a mesma coloração padrões sem precipitado ou plano de fundo em um 72 hpf Pachón cavefish (100 x). (C) o flanco lateral direita de um 72 hpf Pachón cavefish demonstra difusa, inespecífica mancha para BMP4 na ampliação x 45. (D) A 72 hpf Pachón cavefish sujeitos ao presente protocolo, sem a adição da sonda (x 45). Escala de barras = 0,5 mm. clique aqui para ver uma versão maior desta figura.
Discussão
Devido à vulnerabilidade do RNA, a degradação, um dos passos mais importantes no protocolo refere-se a síntese estéril da sonda RNA. No entanto, se uma sonda com cuidado é gerada e fornece bons resultados, ele pode ser reutilizado em reações de coloração subsequentes. Um segundo passo crucial é a produção cuidadosa de todos os reagentes utilizados em todo o protocolo. Uma vez que este protocolo envolve vários dias e muitos pequenos passos, é essencial que todos os reagentes são com precisão produzidos e armazenados de forma estéril. Além disso, é fundamental que o investigador mantém controle cuidadoso de cada etapa no protocolo. Nós encontramos que a lista de verificação fornecida de etapas pode ser extremamente úteis para garantir uma conclusão exata e precisa de cada aspecto do presente protocolo.
Nós muitas vezes não modifique o protocolo que apresentamos aqui. No entanto, os investigadores podem realizar incubação do sonda em temperaturas diferentes do que os sugeridos (i.e., 70 ° C). Pequenas mudanças em temperaturas de hibridização impactará vinculação de sondas de RNA, e portanto, buscando a temperatura ideal de hibridização pode impactar positivamente a qualidade da coloração. Com relação a resolução de problemas, recomendamos que outros investigadores para utilizar a lista de verificação fornecida com este artigo (suplementar 1 arquivo). Mantendo cuidado de registros é um primeiro passo necessário para assegurar a alta qualidade coloração. Sugere-se uma segunda pequena modificação é para realizar a reação de coloração final sem balanço (por exemplo, sem colocar em um agitador de plataforma ou nutator). A razão para isto é que periodicamente verificamos a produção do precipitado, que presumivelmente surge da solução-tampão de AP. Esse precipitado geralmente overstains para uma cor escura e cria punctate (específico) de fundo sobre o tecido manchado. Para minimizar a produção desse precipitado, preparamos o buffer de AP esterilizado antes de cada reação. Além disso, uma vez NBT e BCIP foram adicionados para o buffer, nós substitui-la com buffer de fresco e NBT/BCIP cada hora até que a reação de coloração é concluída.
Uma limitação para o método apresentado é que uma mancha cromática foi usada para visualização de expressão do gene. Preferimos esta abordagem, desde que é rentável e requer apenas a microscopia de luz para visualizar. Se um estava interessado em avaliar diferenças quantitativas, sugerimos que eles usam uma reação de coloração fluorescente. Isto irá permitir a quantificação semi, por exemplo, através da comparação de unidades fluorescentes relativas de expressão entre as experiências.
Protocolos para a hibridação in situ estão disponíveis amplamente na web12,13, bem como em publicações científicas. O protocolo que apresentamos foi desenvolvido especificamente para o nosso sistema de modelo, Astyanax mexicanus. Usamos este protocolo para manchar a expressão de várias dezenas de genes e sentir-se consistentemente fornece resultados de alta qualidade. Uma vantagem significativa do presente protocolo é a passo a passo lista de verificação de itens para permitir que o investigador realizar várias tarefas, garantindo a conclusão exata de cada uma das etapas do presente protocolo. Esperamos que este protocolo vai servir como um recurso útil para outros investigadores no campo e além e antecipar que essa técnica de laboratório comum oferecerá suporte a futuras descobertas ligando genótipo ao fenótipo no cavefish cego mexicano.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores desejam agradecer membros do laboratório Gross para comentários úteis sobre este manuscrito. Gostaríamos de reconhecer quatro estudantes do ensino médio que utilizaram este protocolo durante os estágios de verão em 2017 e 2018, incluindo David Nwankwo, Michael Warden, Aki Li e Christine Cao. HL foi apoiado por uma bolsa UC biologia tronco durante o verão de 2017. Este trabalho foi financiado por doações da Fundação Nacional da ciência (DEB-1457630 de JBG) e institutos nacionais de dentária e Craniofacial Research (NIH; DE025033 de JBG).
Materiais
| Name | Company | Catalog Number | Comments |
| 10 mL Serological Pipette | VWR | 89130-888 | |
| 1000 mL Filtration Unit | VWR | 89220-698 | |
| 15 mL Conical | VWR-Greiner | 82050-278 | |
| 25 mL Serological Pipette | VWR | 89130-890 | |
| 250 mL Filtration Unit | VWR | 89220-694 | |
| 5 mL Serological Pipette | VWR | 89130-886 | |
| 50 mL Conical | VWR-Falcon | 21008-940 | |
| 500 mL Filtration Unit | VWR | 89220-696 | |
| Anti-Digoxigenin-AP, Fab fragments | Sigma-Roche | 11093274910 | |
| BCIP | Sigma-Aldrich | B8503-1G | 1 g |
| Blocking Solution | Sigma-Roche | 11 096 176 001 | 50 g |
| Citric Acid | Fisher Scientific | A104-500 | 500 g |
| DIG RNA Labeling Kit (SP6/T7) | Sigma-Roche | 11175025910 | |
| Eppendorf Tubes | VWR | 20170-577 | |
| Ethanol | Fisher-Decon | 04-355-223 | 1 Gal |
| Formamide | Thermo Fisher Scientific | 17899 | 100 mL |
| Glass dram vials | VWR | 66011-041 | 1 Dr |
| Glass Pipettes | Fisher Scientific | 13-678-8A | |
| HCl | Thermal-ScientificPharmco-AAPER | 284000ACS | 500 mL |
| Heparin | Sigma | H3393-25KU | |
| Magnesium Chloride-crystalline | Fisher Scientific | M33-500 | 500 g |
| Maleic Acid | Sigma | M0375-100g | 100g |
| Methanol | Fisher Scientific | A452-4 | 4L |
| Molecular-grade Water (RNase-free) | VWR | 7732-18-5 | 500 mL |
| NaCl | Fisher Scientific | S271-3 | 3 kg |
| NaOH pellets | Fisher Scientific | S318-500 | 500 g |
| NBT Substrate powder | ThermoFisher Scientific | 34035 | 1 g |
| Normal Goat Serum | Fisher-Invitrogen | 31873 | |
| Nutating Mixer | VWR | 82007-202 | |
| Paraformaldehyde | Sigma | 158127-500g | 500 g |
| PBS 10x | Fisher Scientific | BP399-20 | 20L |
| Proteinase K (200mg/10ml) | Qiagen | 19133 | 10 mL |
| Plastic Pipettes | VWR-Samco | 14670-147 | |
| RNAse | Sigma | R2020-250mL | 250 mL |
| Shaking Water Bath 12 L | VWR | 10128-126 | 12 L |
| Standard Analog Shaker | VWR | 89032-092 | |
| Tris | Sigma Millipore-OmniPur | 9210-500GM | 500 g |
| tRNA Yeast | Fisher-Invitrogen | 15401011 | 25 mg |
| Tween 20 | Sigma | P9416-50mL | 50 mL |
| Vortex-Genie 2 | Fisher Scientific-Scientific Industries, Inc | 50-728-002 | |
| Lithium Chloride (LiCl) | Sigma-Aldrich | 203637-10G | 10 g |
Referências
- Valentino, K. L., Eberwine, J. H., Barchas, J. D. In situ hybridization: Application to neurobiology. , Oxford University Press. Oxford. (1987).
- Mugrauer, G., Alt, F. W., Ekblom, P. N-myc proto-oncogene expression during organogenesis in the developing mouse as revealed by in situ hybridization. The Journal of Cell Biology. 107 (4), 1325-1335 (1988).
- Kerney, R., Gross, J. B., Hanken, J. Early cranial patterning in the direct‐developing frog Eleutherodactylus coqui revealed through gene expression. Evolution & Development. 12 (4), 373-382 (2010).
- Thisse, C., Thisse, B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nature Protocols. 3 (1), 59-69 (2008).
- Cheung, M., Briscoe, J. Neural crest development is regulated by the transcription factor Sox9. Development. 130 (23), 5681-5693 (2003).
- Knight, R. D., Nair, S., Nelson, S. S., Afshar, A., Javidan, Y., Geisler, R., Rauch, G. J., Schilling, T. F. lockjaw encodes a zebrafish tfap2a required for early neural crest development. Development. 130 (23), 5755-5768 (2003).
- Yang, J. W., Jeong, B. C., Park, J., Koh, J. T. PHF20 positively regulates osteoblast differentiation via increasing the expression and activation of Runx2 with enrichment of H3K4me3. Scientific Reports. 7 (1), 8060(2017).
- Ganju, R. K., Brubaker, S. A., Meyer, J., Dutt, P., Yang, Y., Qin, S., Newman, W., Groopman, J. E. The α-chemokine, stromal cell-derived factor-1α, binds to the transmembrane G-protein-coupled CXCR-4 receptor and activates multiple signal transduction pathways. Journal of Biological Chemistry. 273 (36), 23169-23175 (1998).
- Cai, Y., Xin, X., Yamada, T., Muramatsu, Y., Szpirer, C., Matsumoto, K. Assignments of the genes for rat pituitary adenylate cyclase activating polypeptide (Adcyap1) and its receptor subtypes (Adcyap1r1, Adcyap1r2, and Adcyap1r3). Cytogenetic and Genome Research. 71 (2), 193-196 (1995).
- Stolt, C. C., Rehberg, S., Ader, M., Lommes, P., Riethmacher, D., Schachner, M., Bartsch, U., Wegner, M. Terminal differentiation of myelin-forming oligodendrocytes depends on the transcription factor Sox10. Genes & Development. 16 (2), 165-170 (2002).
- Kimura, T., Takehana, Y., Naruse, K. pnp4a is the causal gene of the Medaka Iridophore mutant guanineless. G3: Genes, Genomes, Genetics. 7 (4), 1357-1363 (2017).
- Monsoro-Burq, A. H. A rapid protocol for whole-mount in situ hybridization on Xenopus embryos. Cold Spring Harbor Protocols. (8), pp.pdb-prot4809(2007).
- Schulz, C. In situ hybridization to Drosophila testes. Cold Spring Harbor Protocols. (8), pp.pdb-prot4764(2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados