Method Article
Wholemount In Situ Hybridisierung für Astyanax Embryonen
In diesem Artikel
Zusammenfassung
Dieses Protokoll ermöglicht die Visualisierung der Genexpression in embryonalen Astyanax Cavefish. Dieser Ansatz wurde mit dem Ziel der Maximierung der gen-Expression-Signal, bei gleichzeitiger Minimierung der unspezifischen Hintergrundfärbung entwickelt.
Zusammenfassung
In den letzten Jahren wurde eine Entwurf Genom für die blinde mexikanischen Cavefish (Astyanax Mexicanus) freigegeben, enthüllt die Sequenz Identitäten für Tausende von Genen. Frühere Forschung in diesem aufstrebenden Modellsystem aktiviert auf umfassende genomweite Untersuchungen, die zahlreiche quantitative Trait Loci (QTL) verbunden mit verschiedenen Höhle verbundenen Phänotypen identifiziert haben. Die Möglichkeit, Gene von Interesse, die erbliche Grundlage für phänotypische Veränderung verbinden bleibt jedoch eine große Herausforderung dar. Eine Technik, die tieferes Verständnis für die Rolle der Entwicklung in der Troglomorphic Evolution erleichtern kann, ist vollständig-hängen in-situ-Hybridisierung. Diese Technik kann implementiert werden, um direkt vergleichen Genexpression zwischen Höhle und Oberfläche Wohnformen, Anwärter Gene zugrunde liegenden etablierten QTL, Gene Interesse von Next Generation Sequencing Studien zu identifizieren oder entwickeln andere Entdeckung-basierte Ansätze. In diesem Bericht stellen wir ein einfaches Protokoll, unterstützt durch eine flexible Checkliste, die für den Einsatz auch außerhalb des Studiensystems der vorgestellten weit angepasst werden kann. Es ist zu hoffen, dass dieses Protokoll für die Astyanax Gemeinschaft und darüber hinaus als eine umfassende Ressource dienen kann.
Einleitung
In-situ Hybridisierung ist eine gängige Methode zur Färbung von festen Gewebe um gen-Expression-Muster1zu visualisieren. Diese Technik ist seit Jahren in anderen traditionellen2 und nicht-traditionellen3 -Modellsysteme, für eine Vielzahl von biologischen Studien durchgeführt. Allerdings sind mehrere Schritte und Reagenzien notwendig, um dieses Verfahren erfolgreich durchführen. Für Forscher, die diese Technik noch nie durchgeführt haben, kann es aufgrund der vielen Schritte einschüchternd sein, den Prozess zu initiieren. Weiter, die langwierige Natur dieses Verfahren eignet sich für technische Fehler, die schwierig sein können, um zu beheben.
Das übergeordnete Ziel dieses Artikels ist es, eine einfache und unkomplizierte Methode vorstellen, die diese Hybridisierung-Technik für ein breites Publikum zugänglich machen wird. Um die Einführung von Fehlern zu reduzieren, stellen wir eine einfache Lösung, die liefert hochwertige gen Ausdruck Färbung und unspezifische Hintergrundsignal minimiert. Dieses Verfahren ist ähnlich wie andere Ansätze, die in traditionellen Modellsysteme wie Danio Rerio4entwickelt. Wir wollen hier, sorgfältige Umsetzung der einzelnen Schritte anhand einer herunterladbaren Checkliste (ergänzende Datei 1), sorgfältige Durchführung des Protokolls zu fördern. Die Begründung hierfür ist Organisation durch die vielen Schritte in diesem Verfahren zu erleichtern. Dieser Artikel eignet sich für Forscher interessiert bei der Durchführung von in-situ Hybridisierung vollständig-hängen bei der Entwicklung von Embryonen, aber noch nicht das Verfahren durchgeführt. Der Vorteil des gewählten Ansatzes für Astyanax Forscher ist, dass es getestet wurde und in Cavefish und Oberfläche Fisch verwandelt, wodurch vergleichende Expressionsanalysen bewährt. Die vorgestellte Methode kann durch Forscher in Studien über Astyanax und anderen Systemen verwendet werden.
Protokoll
Alle hier beschriebene Methoden wurden von den institutionellen Animal Care und Nutzung Committee (IACUC) von der University of Cincinnati (Protokoll Nr. 10-01-21-01) genehmigt.
1. Fixierung
- Isolieren Sie gewünschte Anzahl von Astyanax Mexicanus Embryonen aus einem Zucht-Tank zu und beheben Sie ~ 50 Embryonen zu einem Zeitpunkt. Wenn Embryonen groß und alt sind, kann es 25 gleichzeitig auch Fixierung darauf festzulegen sein.
- Je nach Alter des Embryos nutzen Sie die IACUC erprobte Methode der Anästhesie. Für ältere Embryonen mit ein funktionierendes Nervensystem Embryonen über Narkose Überdosierung zu opfern. Entsprechend legen Sie Embryonen in einer Lösung von ~ 1 % Tricaine (gepuffert pH 7.4) zur Minimierung von Schmerzen und Beschwerden für den Organismus.
- Sobald die Embryonen nicht zu berühren reagieren, ersetzen Sie System wasserhaltigen Tricaine zu, und ~ 1 mL 1 x Phosphat-gepufferte Kochsalzlösung (PBS, pH 7.4).
- Entfernen Sie die PBS-Lösung, und fügen Sie 1 mL 4 % Paraformaldehyd (PFA). Beheben von Embryonen über Nacht bei 4 ° C.
Achtung: PFA ist gefährlich (d. h. es ist brennbar und ist ein Haut- und Lungenkrebs Reizstoffe), pfleglich zu behandeln.
(2) Dehydratation
- Um die Embryonen zu entwässern, die Fixativ Lösung entfernen und Spülen mit 1 mL PBS. Legen Sie bei Spülen Embryo-haltigen Fläschchen in einem Winkel (zwischen 30° und 45°), auf einem Plattform-Shaker. Weiterhin die Embryonen zweimal, 5 min pro Waschgang waschen.
- Wenn Embryonen noch ein Chorion haben, setzen Sie alle 50 Embryonen in einem 100 mm x 25 mm Petrischale und isolieren Sie sorgfältig von den Chorions mit zwei Sätzen von Uhrmacher Pinzette (z. B. #5 Zangen) unter dem Mikroskop zu.
Hinweis: Während die Schritte unten, und für den Rest des Protokolls, entfernen Sie vorsichtig alle Flüssigkeit aus dem vorherigen Schritt mit sauberen, Glas Pasteur Pipetten, bevor Sie die nächste Lösung hinzufügen - Pausiert die Embryonen in einer Reihe von zunehmend konzentrierte wäscht von Methanol (MeOH) beschrieben. Die Verdünnungen basieren auf einem 1 mL Gesamtvolumen mit 500 µL Lösung in jedem 4 mL Glasflasche. Führen Sie alle Schritte der Dehydrierung bei Raumtemperatur (RT) auf Plattform Shaker.
- Entfernen Sie vorsichtig die PBS-Lösung. Fügen Sie eine 25 % ige MeOH Lösung (250 µL MeOH) + 750 µL PBS. Auf einer Plattform Shaker für 5 min schütteln.
- Entfernen Sie vorsichtig die 25 % MeOH Lösung. Fügen Sie eine 50 % ige MeOH Lösung (500 µL MeOH) + 500 µL PBS. Auf einer Plattform Shaker für 5 min schütteln.
- Entfernen Sie vorsichtig die 50 % MeOH Lösung. Fügen Sie eine 75 % ige MeOH Lösung (750 µL MeOH) + 250 µL PBS. Für 5 Minuten auf einer Plattform Shaker schütteln.
- Entfernen Sie vorsichtig die 75 % MeOH Lösung. Fügen Sie eine 100 % MeOH Lösung (1 mL MeOH). Auf einer Plattform Shaker für 5 min. Wiederholen Sie diese Schritt 3 Mal schütteln Sie vorsichtig.
- An diesem Punkt speichern Sie dehydrierte Embryonen Bedarf in ihre Glasfläschchen bei-20 ° C (langfristig). Alternativ gehen Sie direkt zu Tag 1 des Protokolls.
3. Tag 1: Rehydratation
- Erhalten Sie dehydrierte Embryonen von-20 ° C Gefrierschrank zu (oder gehen Sie direkt von Schritt 2.5).
- Sortieren Sie die Embryonen mit einer Pasteurpipette. Man kann anhand Morphotyp (d.h., Höhle und/oder Oberfläche) sortieren, und die Anzahl der Gene beurteilt jedes Experiment. Es gibt in der Regel nicht mehr als 12 Embryonen pro Fläschchen einmal sortiert. Um die Organisation zu erhalten, verwenden Sie farbige Lab Band, um Fläschchen und Pipetten für jedes Gen zu benennen. Embryonen bleiben in der gleichen Flasche in das gesamte Protokoll.
Hinweis: Statt der Pasteur Pipettenspitze in 100 % verwendet EtOH zwischen sterilisieren. - Stellen Sie schütteln Wasserbad auf 70 ° C in einem späteren Schritt verwendet werden. Vorsichtig, MeOH in Fläschchen von sortierten Embryonen herauszuziehen und ersetzen mit 500 µL des neuen 100 % MeOH. Waschen Sie kurz (~ 1 min) auf Plattform Shaker.
- Rehydrieren Sie Embryonen in eine zunehmende Konzentration von 1 X PBS mit Tween 20 (PBT, siehe unten) auf Plattform Shaker. Die Verdünnungen basieren auf einem Volume 1 mL Endverdünnung mit 500 µL gehen in jedes Fläschchen.
- Fügen Sie eine 25 % PBT Lösung (250 µL PBT, 750 µL MeOH). Auf einer Plattform Shaker für 5 min schütteln.
- Entfernen Sie vorsichtig die 25 % PBT-Lösung. Fügen Sie eine 50 % PBT Lösung (500 µL PBT, 500 µL MeOH). Auf einer Plattform Shaker für 5 min schütteln.
- Entfernen Sie vorsichtig die 50 % PBT-Lösung. Fügen Sie eine 75 % PBT Lösung (750 µL PBT, 250 µL MeOH). Auf einer Plattform Shaker für 5 min schütteln.
- Entfernen Sie vorsichtig die 75 % PBT-Lösung. Fügen Sie eine 100 % PBT-Lösung (1 mL PBT). Auf einer Plattform Shaker für 5 min. Wiederholen Sie diese Schritt 3 Mal schütteln Sie vorsichtig.
4. Tag 1: Verdauung und Fixierung
- Bereiten Sie eine Proteinase K (PK) Lösung durch Zugabe von 1 µL PK (20 mg/mL), 2 mL PBT.
- Erhalten Sie im Vorgriff auf spätere Schritte eingefrorenen aliquoten Hybridisierung Puffer (Hyb- und Hyb +; zusätzliche Datei 2 und zusätzliche Datei 3zu sehen) und PFA von-20 ° C Lagerung.
- Lassen Sie PFA bei RT Auftauen
- Legen Sie Aliquote Hyb - und Hyb + in einem rotierenden 70 ° C-Wasserbad. Legen Sie alle Reagenzien und Fläschchen in einem kleinen "Dichtung" mit einem Netz-Boden im Inneren der schwimmenden Wasser Bad Apparat. Dies ermöglicht einfaches hinzufügen und Entfernen von Röhren und Fläschchen aus dem rotierenden 70 ° C-Wasserbad.
- Fügen Sie sanft PK-Lösung für die Durchstechflasche(n) Embryonen gewährleisten allen Geweben sind komplett mit Lösung bedeckt. Embryonen für ~ 12 min in PK funktionierende Lösung auf der Plattform-Shaker zu verdauen.
Hinweis: Die Länge der Verdauung kann variiert werden, vom Prüfer um optimale Ergebnisse zu gewährleisten. - Die PK-Lösung ziehen Sie vorsichtig ab und kurz überschwemmen Sie das Fläschchen mit PBT, alle übrigen PK zu verdünnen
- Die PBT-Lösung ziehen Sie ab und ersetzen Sie durch neue PBT 500 µL. Lassen Sie die Lösung auf der Plattform Shaker für 5 min spülen.
- PBT abziehen und ersetzen mit 500 µL aufgetauten 4 % PFA. Ermöglichen Sie die Embryonen für 20 min auf der Plattform Shaker bei RT inkubieren
- Beziehen Sie die 4 % PFA, und kurz überschwemmen das Fläschchen mit PBT, alle übrigen PFA zu verdünnen. Die PBT abziehen und mit 500 µL frische PBT ersetzen. Lassen Sie Embryonen für 5 min auf der Plattform Shaker ausspülen. Wiederholen Sie diesen Schritt 4 weitere Male.
5. Tag 1: Prehybridization
- Platz 500 µL vorgewärmten Hyb-Lösung in die Flasche. Legen Sie vorsichtig das Fläschchen in der 70 ° C Wasser Bad (in Dichtungen) ohne schütteln, für 5 Minuten.
- Die Hyb-Lösung abziehen und das Fläschchen mit 500 µL vorgewärmten Hyb + Lösung zu überfluten. Platzieren Sie das Fläschchen zurück in die 70 ° C-Wasserbad mit schütteln (40 u/min). Inkubieren entweder 4 Stunden oder über Nacht.
Hinweis: Eine 4 h Inkubation erbringt ein komplettes in-situ-Protokoll, das für insgesamt 4 Tage dauern wird. Hier präsentiert sich dieser Schritt als eine Übernachtung Inkubation, die eine Dauer von 5 Tagen insgesamt Protokoll Ausbeute wird.
6. Tag 2: Hybridisierung
- Legen Sie eine Aliquote von Hyb + aus dem Tiefkühler-20 ° C in dem Schütteln heißen Wasserbad für 5 min.
- Ziehen Sie der Hyb ab + aus dem Fläschchen und mit 500 µL vorgewärmten Hyb + zu ersetzen. Diese Lösung fügen Sie vorsichtig 2 µL RNA-Sonde für jedes Fläschchen hinzu. Vorsichtig schwenken das Fläschchen um gleichmäßige Verteilung der Sonde zu gewährleisten.
- Inkubieren Sie die Hyb + (mit zusätzlichen Sonde) Lösung in der 70 ° C heißen Wasserbad über Nacht unter Schütteln bei 40 u/min.
Hinweis: Man kann Hyb + (mit Sonde) Lösung erneut verwenden. Dafür nehmen Sie Hyb + mit Sonde aus dem ersten Lauf aus dem Gefrierschrank-20 ° C und es in ein heißes Wasserbad für 5 min. ersetzen Hyb + ab dem Tag 1 Protokoll mit Hyb + mit Sonde und Inkubation über Nacht im heißen Wasserbad zu ermöglichen.
7. Tag 3: Vorbereitung der Lösung
- Bereiten Sie Mikrozentrifugenröhrchen beschriftet Hyb + mit der "gen-of-Interest" RNA-Sonde vor. Bereiten Sie die Serie von Verdünnungen, die tagsüber 3 verwendet werden.
- Mit 6 separaten Röhren, bereiten Sie die folgende Reihe von Verdünnungen von Hyb und Kochsalzlösung Natriumcitrat (SSC, 0 bis 100 %) in einer 1 mL Volumen und Stelle sie in den 70 ° C schütteln Wasserbad: Rohr 1 = 100 % Hyb-(1 mL Hyb-): 2 = 25 % Tube 2 X SSC (250 µL 2 X SSC, 750 µL Hyb-); 3 = 50 % 2 X SSC Rohr (2 X SSC, 500 µL Hyb - 500 µL); 4 = 75 % 2 X SSC Rohr (2 X SSC, 250 µL Hyb - 750 µL); Schlauch 5 = 100 % 2 X SSC (1 mL 2 X SSC); Rohr 6 = 100 % 0,2 X SSC (2 mL 0,2 X SSC).
Hinweis: Der Konzentration von SSC, wachsam sein, wie es ändert sich von 2 X 0,2 X. - Mit 4 Rohre zu trennen, die folgende Reihe von Verdünnungen von PBT und SSC in einem Volumen von 1 mL vorbereitet und legen Sie bei RT: Rohr 1 = 25 % PBT (250 µL PBT, 750 µL 0,2 X SSC); Rohr 2 = 50 % PBT (500 µL PBT, 500 µL 0,2 X SSC); Rohr 3 = 75 % PBT (750 µL PBT, 250 µL 0,2 X SSC); Rohr 4 = 100 % PBT (1mL PBT).
- Bereiten Sie ein Röhrchen mit 2 mL Maleic Acid-Puffer mit Tween 20 (MABT) funktionierende Lösung.
- Bereiten Sie zwei 15 mL konische Röhrchen Lösung zu blockieren. In jedem Röhrchen 0,2 g blocking Reagenz zu 10 mL MABT hinzufügen (siehe ergänzende Datei 4). Legen Sie beide Röhren auf ein Nutating Mixer (oder Plattform Shaker) bis vollständig in Lösung (max. 3 h) aufgelöst.
- Mit 6 separaten Röhren, bereiten Sie die folgende Reihe von Verdünnungen von Hyb und Kochsalzlösung Natriumcitrat (SSC, 0 bis 100 %) in einer 1 mL Volumen und Stelle sie in den 70 ° C schütteln Wasserbad: Rohr 1 = 100 % Hyb-(1 mL Hyb-): 2 = 25 % Tube 2 X SSC (250 µL 2 X SSC, 750 µL Hyb-); 3 = 50 % 2 X SSC Rohr (2 X SSC, 500 µL Hyb - 500 µL); 4 = 75 % 2 X SSC Rohr (2 X SSC, 250 µL Hyb - 750 µL); Schlauch 5 = 100 % 2 X SSC (1 mL 2 X SSC); Rohr 6 = 100 % 0,2 X SSC (2 mL 0,2 X SSC).
8. Tag 3: Sonde entfernen
- Hyb + (mit Sonde) Lösung mit einem Glas Pasteurpipette ziehen Sie ab und in eine sterile, mit der Bezeichnung Microcentrifuge Schlauch. Behalten Sie diese Röhre in den Gefrierschrank-20 ° C für eine spätere Verwendung (wenn Sonde Kennzeichnung erfolgreich ist).
- Fügen Sie sorgfältig 500 µL warmen SSC/Hyb-Verdünnungen (unten angegeben). In jedem der folgenden Lösungen für 10 min bei 70 ° C schütteln Wasserbad inkubieren.
- Mit 100 % gegenüber dem Vorquartal inkubieren Hyb-(1 mL Hyb-), 25 % 2 X SSC (250 µL 2 X SSC, 750 µL Hyb-), 50 % 2 X SSC (500 µL 2 X SSC, 500 µL Hyb-), 75 % 2 X SSC (750 µL 2 X SSC, 250 µL Hyb-), 100 % 2 X SSC (1 mL 2 X SSC) , 100 % 0,2 X SSC (2 mL 0,2 X SSC).
- Nach dem letzten Schritt in jedem der folgenden Lösungen für 10 min inkubieren. Alle der folgenden Inkubationen statt bei RT auf der Plattform-Shaker: 25 % PBT (250 µL PBT, 750 µL 0,2 X SSC), 50 % PBT (500 µL PBT, 500 µL 0,2 X SSC), 75 % PBT (750 µL PBT, 250 µL 0,2 X SSC) , 100 % PBT (1 mL PBT).
- Nach einer Inkubationszeit von 10 min, entfernen Sie die 100 % PBT, und fügen Sie 500 µL MABT in jedes Fläschchen. Wiederholen Sie diesen Schritt zweimal für 5 Minuten.
9. Tag 3: Sperrung
- Entfernen Sie MABT aus jedem Fläschchen und Flut mit vorgemischten blockierende Lösung eines der Rohre (vorbereitet in Schritt 7.1.4). Ort-Fläschchen auf ein Nutating Mixer für ~ 4 h bei RT
- Fügen Sie 2 µL Anti-DIG-AP Fab-Fragmente, das zweite Fläschchen 10 ml Lösung (in Schritt 7.1.4 vorbereitet) und kurz Wirbel blockieren.
- Füllen Sie jedes Fläschchen fast vollständig mit blockiert Lösung (ca. 5 mL) und auf Nutating Mixer über Nacht im Kühlschrank bei 4 ° C.
10. Tag 4: MABT Spülungen
- Bereiten Sie ein Lager Fläschchen mit 10 % normalem Ziegenserum (NGS) in MABT (100 µL NGS 900 µL MABT hinzufügen).
- Zeichnen Sie die blockierenden Lösung in jedes Fläschchen und fügen Sie 500 µL NGS/MABT Mischung in jedes Fläschchen. Lassen Sie die Embryonen für 25 min bei RT auf der Plattform Shaker auszubrüten.
- Ersetzen Sie die NGS/MABT Mischung mit 500 µL 100 % MABT. 30 min bei RT auf der Plattform Shaker inkubieren. Führen Sie diese spülen 11 Mal mehr tagsüber alle 30 Minuten.
- Füllen Sie das Fläschchen mit 100 % MABT und Platz auf einem Nutating Mixer über Nacht im Kühlschrank oder begehbare Kammer bei 4 ° C.
11. Tag 5: Sonde Visualisierung
- Bereiten Sie eine 50 mL Aliquot der alkalischen Phosphatase (AP) Puffer (siehe ergänzende Datei 5). Kombinieren Sie die folgenden in einem 50 mL konische Röhrchen eingewickelt in Alufolie, Belichtung zu begrenzen: 5 mL 1 M Tris (pH 9,5), 5 mL 50 mM MgCl2, 5 mL von 1 % Tween 20, 5 mL 1 M NaCl, 30 mL ddH2O.
- MABT entfernen und ersetzen mit 1 mL AP Puffer (Rohr in Folie gewickelt). Lassen Sie es für 5 min waschen. Tun Sie dies zweimal, um vollständige Entfernung von MABT zu gewährleisten.
- AP-Puffer zu entfernen und ersetzen Sie durch 1 mL der AP-Puffer mit 5-bromo-4-chloro-3'-indolyphosphate 3,5 μl (BCIP) und 4.5 μL der Nitro-blau Tetrazoliumsalz (NBT). Ersetzen Sie mit frisch zubereiteten AP Puffer/NBT/BCIP einmal stündlich bis Reaktion abgeschlossen ist. Überwachen Sie, prüfen alle 15 min, damit kann die Färbung Reaktion stattfinden, bis die gewünschte Färbung erreicht ist. Wenn Niederschlag zu bilden beginnt, ersetzen Sie früher die Lösung.
- Hören Sie auf, die Färbung Reaktion durch Spülen der Embryonen in frische 100 % AP Puffer (ohne NBT/BCIP) für 5 min. weiter Spülungen in PBT bis optimale Signal (mit minimalen Mengen an Hintergrundfärbung) erreicht werden. Weiterspülen Exemplare mit zunehmender Verdünnungen von PBT in AP Puffer wie folgt: 25 % PBT (250 μL der PBT, 750 μl des AP-Puffer), Spülen für 5 min; 50 % PBT (500 μL der PBT, 500 μL des AP-Puffer), Spülen für 5 min; 75 % PBT (750 μl des PBT, 250 μL des AP-Puffer), für 5 min spülen.
- Spülen Sie Embryonen in ~ 5 mL 100 % PBT auf Nutating Mixer bis minimale Hintergrundfärbung gewünscht erreicht wird. Wechseln Sie mit frischen PBT mehrmals. Dies kann bis zu mehreren Tagen dauern.
- Beim Spülen vollständig, waschen Embryonen in 500 μl steriler PBS auf einer Plattform-Shaker ist. Durchführen Sie diese Spülung zweimal für 5 min. Nachdem PBS wäscht, Post-fix die Exemplare in 500 μl 4 % PFA für 1 h bei RT auf einer Plattform-Shaker. Alternativ zu beheben über Nacht in 1 mL 4 % PFA im Kühlschrank bei 4 ° C.
- Ersetzen Sie das Fixiermittel mit frischen, sterilen PBS. Führen Sie diese spülen mindestens zweimal für 5 min. Platz die Embryonen in ~ 4 mL 100 % sterile PBS und lagern langfristig bei 4 ° c
12. Abbildung
- Eine Speicherfolie in einer Petrischale mit 3 % Agarose und TAE Puffer bilden.
Hinweis: Mengen hängen davon ab, wie viele Platten benötigt werden. Platten können mehrmals wiederverwendet werden. Es empfiehlt sich, eine flache rechteckige Form in der Petrischale gelegt wird, während das Gel abkühlt, um eine Depression zur Eindämmung der Embryonen auf der Platte zu schaffen. - Legen Sie die Embryonen auf dem Teller in der PBS.
Hinweis: Es empfiehlt sich, vorsichtig gießen Embryonen auf den Teller statt pipettieren sie aus, denn es festgestellt wurde, dass sie an der Innenseite der Kunststoff Pipetten haften. - Verwenden Sie Lichtmikroskopie, um jedem Embryo sichtbar zu machen. Verwenden Sie eine stumpfe Sonde Embryonen in gewünschte Position zu manövrieren.
- Nehmen Sie ein Bild, wenn der Embryo in der gewünschten Position ist. Beachten Sie, dass es wichtig ist, Bilder von Embryonen in ein paar Wochen der abgeschlossenen Färbung zur Vermeidung von möglichen Abbau von Fleck zu nehmen.
Ergebnisse
In diesem Bericht stellen wir einen einfachen und unkomplizierten Ansatz zur Kennzeichnung von embryonalen Astyanax Exemplare für qualitativ hochwertige gen Expressionsanalyse durchführen. Diese Technik kann in vier oder fünf Tagen durchgeführt werden, und jeder wesentlichen Schritt im Verfahren ist in eine farbcodierte Flussdiagramm (Abbildung 1) vertreten. Einmal abgeschlossen, sollte gebeizt Embryonen ein dunkles Lila chromatische Label in Geweben, die mit dem Ausdruck des Gens von Interesse hegen. Wir haben dieses Protokoll in Pachón Cavefish (Abbildung 2A–B, E) und Oberfläche (Abbildung 2–D, F) Fischembryonen erfolgreich umgesetzt.
Die Cavefish Embryonen wurden für zwei Transkriptionsfaktoren das Label frühen Neuralleiste Gewebe, Sox9 und Tfap2a5,6gebeizt. Embryonen, die mit der Bezeichnung für Sox9 Ausdruck zeigen klare Kennzeichnung in den Entwicklungsländern Branchial Bögen und Brustflosse (gelbe Pfeilspitzen, Abb. 2A). Beachten Sie, dass die Färbung ist praktisch nicht vorhanden in der Dottersack oder den entwickelnden Somiten an der Flanke (Abbildung 2A). Ebenso ist Tfap2a Ausdruck zeigt sich in die Teile der Entwicklung Kopf, sowie frühe Migration Neuralleiste Zellen (Abb. 2 b, Pfeilspitze entlang der dorsalen Flanke des Embryos). Der dritte vertretende gen präsentiert für Cavefish Embryonen ist Phf20a, eine Markierung von Osteoblasten Differenzierung7. Beachten Sie die positive Färbung in den Teilen des somitic Mesoderm und hinteren Kopf, die dazu bestimmt sind, zu Knochengewebe (Abbildung 2E, Pfeilspitzen) führen.
In Oberfläche Fischembryonen sondiert wir für die Gene CXCR, Adcyap1aund Sox10. CXCR kodiert einen G-Protein-Membrane-springen-Rezeptor, der CXC-Chemokine8bindet. Positive Kennzeichnung ist in abgelegenen Regionen des Kopfes sowie Flanke (Abbildung 2F, Pfeilspitzen) und ein paar einzelne Zellen über den Dottersack. Gen Adenylat-Cyclase-aktivierende Polypeptid, Adcyap1a, drückt sich in Regionen des zentralen Nervensystems, einschließlich Hypophyse Zellen. Beachten Sie die hochspezifischen Ausdruck in gekoppelten, bilaterale Gruppen von Zellen auf der dorsalen Aspekt des Embryos; sowie eine größere Region der Mittellinie Ausdruck (Abbildung 2, Pfeilspitzen). Schließlich präsentieren wir der Expression von Sox10, einen Transkriptionsfaktor Etiketten frühen Neuralleiste und Oligodendrozyt Zellen10. Hochspezifische positive Färbung zeigt sich als eine frühe Marker der Neuralleiste auf der linken und rechten Seiten des dorsalen Embryos (Abb. 2D, Pfeilspitzen).
Wir präsentieren jede der zwei Arten der verwirrende Fragen, die auftreten können, andere Forscher. Die erste Ausgabe, die man in regelmäßigen Abständen trifft ist punktförmige Flecken von nicht-spezifische Kennzeichnung. Diese Flecken können als Niederschlag aus der endgültigen MABT Spülungen oder den AP-Puffer während der Färbung Reaktionen auftreten. Ein Beispiel für diese unspezifische Bezeichnung ist offensichtlich auf dem Dottersack des Embryos Oberfläche Fisch gebeizt für Ausdruck der Pnp4a. Dieses Gen kodiert ein Enzym (Purin-Nukleosid-Phosphorylase), die Produktion von schillernden Pigmentierung11erleichtert. Dieses Gen zeigt sich zuerst im dritten Auge und die Schwimmblase. Die punktförmige Flecken beobachtet in einigen Oberfläche Proben (Abbildung 3A) schieden durch häufige Waschungen und Ersatz des AP-Puffer + NBT/BCIP in der Endphase des Protokolls (Abb. 3 b). Ein zweites Thema, bei dem man in regelmäßigen Abständen auftritt ist die diffuse, weitgehend unspezifische Expression von Genen, die ansonsten eine unterschiedliche Expressionsmuster produzieren würde. Ein Beispiel ist das Gen BMP4, erscheint als ein weitgehend diffusen Muster mit niedrigem Chromogen in der Probe (Abbildung 3). In solchen Fällen wir in der Regel eine andere Region des Gens zu identifizieren, in einen Vektor verstärken und führen eine neue Sonde-Synthese (siehe zusätzliche Datei-6). Das Beispiel einer Kontrolle (keine Sonde) Probe (Abbildung 3D) erfolgt um die diffuse und unspezifische Natur unserer gescheiterten BMP4 -Sonde zu illustrieren.
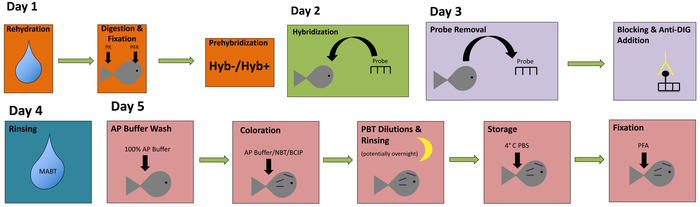
Abbildung 1: ein einfaches Flussdiagramm für die gesamte-Mount in-situ Hybridisierung. Dieses Flussdiagramm nutzt Farbcodierung veranschaulichen die wichtigsten Schritte der in-situ Hybridisierung. Bitte klicken Sie hier für eine größere Version dieser Figur.
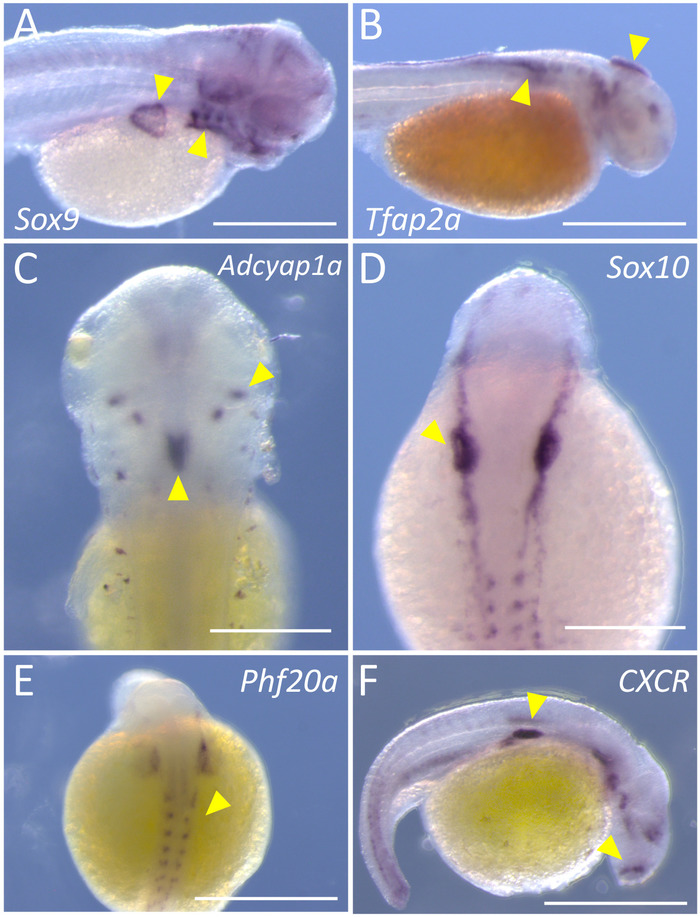
Abbildung 2: Repräsentative Färbung für sechs Gene, mit Höhle und Oberfläche Morphen von Astyanax. (A) Abbildung zeigt die spezifische Färbung (gelbe Pfeile) des Sox9 auf der rechts lateralen Seite der ein 72 h nach Befruchtung (hpf) Pachón Cavefish (45 X). (B) spezifische Färbung, für Tfap2a offensichtlich auf der rechts lateralen Seite eine 36er ist hpf Pachón Cavefish (45 X). (C) bilaterale und Mittellinie Färbung (gelbe Pfeilspitzen) des Adcyap1a sind in der dorsalen Region 72 hpf Oberfläche Fisch (100 X) gekennzeichnet. (D) Färbung des Sox10 in den Geweben der Neuralleiste 24 hpf Oberfläche Fisch (100 X). (E) Phf20a zeigt ein schwaches, aber klar, Muster der Ausdruck in der dorsalen Region ein 24 hpf Pachón Cavefish (100 X). (F) die Cytokin Rezeptor, CXCR, drückt sich in unterschiedlichen Regionen von der rechts lateralen Seite 24 hpf Oberfläche Fisch (100 X). Skala-bars in A, B, E, F = 0,5 mm; skalieren, Bars in C, D = 2,5 mm. Bitte klicken Sie hier für eine größere Version dieser Figur.
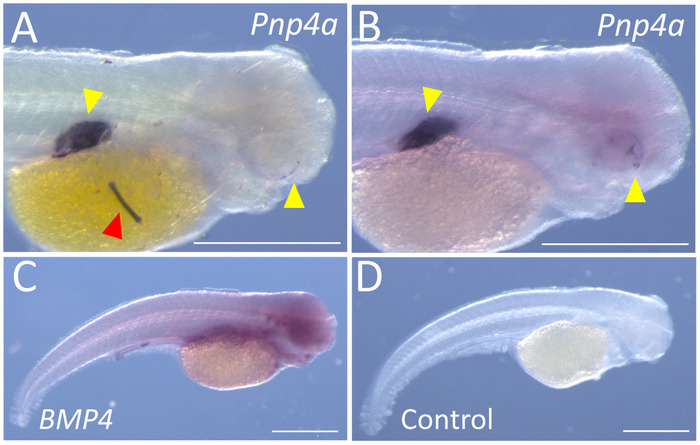
Abbildung 3: repräsentative Beispiele für Sub-optimale Ergebnisse für die gesamte-Mount in-situ Hybridisierung. (A) spezifische Färbung zeigt sich neben unspezifischen Niederschlag und/oder Trümmer (roter Pfeil) auf der rechten lateralen Seite des einen 72 hpf Pachón Cavefish (100 X). (B) der gleichen Sonde visualisiert in A, Darstellung der gleichen Färbung ohne Niederschlag oder Hintergrund in einem 72 Muster hpf Pachón Cavefish (100 X). (C) der rechten seitlichen Flanke eine 72 hpf Pachón Cavefish zeigt diffuse, unspezifische Färbung für BMP4 bei 45 X Vergrößerung. (D) A 72 hpf Pachón Cavefish dieses Protokolls, ohne Zusatz von Sonde (45 X) unterzogen. Skalieren von Balken = 0,5 mm. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Aufgrund der Anfälligkeit der RNA zu einer Verschlechterung betrifft einer der wichtigsten Schritte im Protokoll die sterile Synthese der RNA-Sonde. Jedoch wenn eine Sonde sorgfältig erzeugt wird und gute Ergebnisse liefert, kann es in folgenden befleckenden Reaktionen wiederverwendet werden. Ein zweiter wichtiger Schritt ist die sorgfältige Herstellung der alle Reagenzien im gesamten Protokoll. Da dieses Protokoll mehrere Tage und viele kleine Schritte umfasst, ist es wichtig, dass alle Reagenzien genau produziert und steril aufbewahrt. Darüber hinaus ist es grundlegend wichtig, dass die Ermittler vorsichtig Track der einzelnen Schritte im Protokoll hält. Wir haben festgestellt, dass die bereitgestellte Prüfliste mit folgenden Schritten in gewährleisten genaue und präzise Fertigstellung der einzelnen Aspekte dieses Protokolls sehr nützlich sein kann.
Wir verändern nicht oft das Protokoll, die, das wir hier vorgestellt. Jedoch können Ermittler Sonde Inkubationen bei unterschiedlichen Temperaturen als die vorgeschlagen durchführen (z.B. 70 ° C). Leichte Veränderungen in der Hybridisierung Temperaturen beeinträchtigt die Bindung der RNA-Sonden, und daher sucht die optimale Hybridisierung Temperatur kann positiv beeinflussen die Qualität der Färbung. In Bezug auf Fehlersuche, empfehlen wir andere Forscher, die Checkliste zur Verfügung gestellt, mit diesem Artikel (ergänzende Datei 1) zu nutzen. Die Aufrechterhaltung vorsichtig Records ist ein notwendiger erster Schritt bei der Sicherstellung hoher Qualität zu beflecken. Eine zweite kleine Änderung wird vorgeschlagen, die endgültige Färbung Reaktion ohne Schaukeln durchzuführen ist (z. B. ohne Platzierung auf einer Plattform Shaker oder Nutator). Der Grund dafür ist, dass wir in regelmäßigen Abständen die Produktion von Niederschlag, beachten Sie, die vermutlich aus der AP-Pufferlösung entsteht. Dieser Niederschlag in der Regel eine dunkle Farbe overstains und punctata (unspezifische) Hintergrund auf die gefärbten Gewebe schafft. Um die Produktion von diesem Niederschlag zu minimieren, bereiten wir sterilisiert AP Puffer kurz vor jeder Reaktion. Weiter, nachdem NBT und BCIP in den Puffer hinzugefügt wurden, ersetzen wir es mit frischen Puffer und NBT/BCIP jede Stunde, bis die Färbung Reaktion abgeschlossen ist.
Eine Beschränkung auf die vorgestellte Methode ist, dass ein chromatische Fleck für gen-Ausdruck-Visualisierung verwendet wurde. Dieser Ansatz werden von uns bevorzugt, da es kostengünstig ist und nur Lichtmikroskopie erfordert zu visualisieren. Wenn man bei der Bewertung von quantitativen Unterschiede interessiert waren, empfehlen wir, dass sie eine fluoreszierende Färbung Reaktion verwenden. Semi-Quantifizierung, wird z. B. durch Vergleich der relativen fluoreszierende Einheiten des Ausdrucks zwischen Experimente damit.
Protokolle für die in-situ Hybridisierung sind weit verbreitet auf der Web-12,13, sowie in wissenschaftlichen Publikationen erhältlich. Das Protokoll, das wir präsentieren wurde speziell für unsere Modellsystem, Astyanax Mexicanusentwickelt. Wir haben dieses Protokoll verwendet, um Flecken auf den Ausdruck von mehreren Dutzend Gene, und das Gefühl, dass sie konsequent qualitativ hochwertige Ergebnisse liefert. Ein wesentlicher Vorteil dieses Protokolls ist die schrittweise Checkliste von Elementen, die den Prüfer für mehrere Aufgaben gleichzeitig präzise Fertigstellung der einzelnen Schritte dieses Protokolls zu ermöglichen. Wir hoffen, dass dieses Protokoll als eine hilfreiche Ressource für andere Forscher auf dem Gebiet und darüber hinaus dient, und erwarten, dass diese allgemeine Labortechnik zukünftige Entdeckungen Verknüpfung Genotyp, Phänotyp in die blinde mexikanischen Cavefish unterstützen wird.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren möchte Mitglieder des Brutto-Labors für die hilfreichen Kommentare auf diesem Manuskript danken. Wir wollen vier Schülerinnen und Schüler zu würdigen, die dieses Protokoll während Sommerpraktika in 2017 und 2018, darunter Christine Cao, Michael Warden, Aki Li und David Nwankwo genutzt. HL wurde im Sommer 2017 durch ein UC-Biologie-STEM-Stipendium unterstützt. Diese Arbeit wurde unterstützt durch Zuschüsse von der National Science Foundation (DEB-1457630, JBG), National Institute of Dental und Craniofacial Forschung (NIH; DE025033, JBG).
Materialien
| Name | Company | Catalog Number | Comments |
| 10 mL Serological Pipette | VWR | 89130-888 | |
| 1000 mL Filtration Unit | VWR | 89220-698 | |
| 15 mL Conical | VWR-Greiner | 82050-278 | |
| 25 mL Serological Pipette | VWR | 89130-890 | |
| 250 mL Filtration Unit | VWR | 89220-694 | |
| 5 mL Serological Pipette | VWR | 89130-886 | |
| 50 mL Conical | VWR-Falcon | 21008-940 | |
| 500 mL Filtration Unit | VWR | 89220-696 | |
| Anti-Digoxigenin-AP, Fab fragments | Sigma-Roche | 11093274910 | |
| BCIP | Sigma-Aldrich | B8503-1G | 1 g |
| Blocking Solution | Sigma-Roche | 11 096 176 001 | 50 g |
| Citric Acid | Fisher Scientific | A104-500 | 500 g |
| DIG RNA Labeling Kit (SP6/T7) | Sigma-Roche | 11175025910 | |
| Eppendorf Tubes | VWR | 20170-577 | |
| Ethanol | Fisher-Decon | 04-355-223 | 1 Gal |
| Formamide | Thermo Fisher Scientific | 17899 | 100 mL |
| Glass dram vials | VWR | 66011-041 | 1 Dr |
| Glass Pipettes | Fisher Scientific | 13-678-8A | |
| HCl | Thermal-ScientificPharmco-AAPER | 284000ACS | 500 mL |
| Heparin | Sigma | H3393-25KU | |
| Magnesium Chloride-crystalline | Fisher Scientific | M33-500 | 500 g |
| Maleic Acid | Sigma | M0375-100g | 100g |
| Methanol | Fisher Scientific | A452-4 | 4L |
| Molecular-grade Water (RNase-free) | VWR | 7732-18-5 | 500 mL |
| NaCl | Fisher Scientific | S271-3 | 3 kg |
| NaOH pellets | Fisher Scientific | S318-500 | 500 g |
| NBT Substrate powder | ThermoFisher Scientific | 34035 | 1 g |
| Normal Goat Serum | Fisher-Invitrogen | 31873 | |
| Nutating Mixer | VWR | 82007-202 | |
| Paraformaldehyde | Sigma | 158127-500g | 500 g |
| PBS 10x | Fisher Scientific | BP399-20 | 20L |
| Proteinase K (200mg/10ml) | Qiagen | 19133 | 10 mL |
| Plastic Pipettes | VWR-Samco | 14670-147 | |
| RNAse | Sigma | R2020-250mL | 250 mL |
| Shaking Water Bath 12 L | VWR | 10128-126 | 12 L |
| Standard Analog Shaker | VWR | 89032-092 | |
| Tris | Sigma Millipore-OmniPur | 9210-500GM | 500 g |
| tRNA Yeast | Fisher-Invitrogen | 15401011 | 25 mg |
| Tween 20 | Sigma | P9416-50mL | 50 mL |
| Vortex-Genie 2 | Fisher Scientific-Scientific Industries, Inc | 50-728-002 | |
| Lithium Chloride (LiCl) | Sigma-Aldrich | 203637-10G | 10 g |
Referenzen
- Valentino, K. L., Eberwine, J. H., Barchas, J. D. . In situ hybridization: Application to neurobiology. , (1987).
- Mugrauer, G., Alt, F. W., Ekblom, P. N-myc proto-oncogene expression during organogenesis in the developing mouse as revealed by in situ hybridization. The Journal of Cell Biology. 107 (4), 1325-1335 (1988).
- Kerney, R., Gross, J. B., Hanken, J. Early cranial patterning in the direct‐developing frog Eleutherodactylus coqui revealed through gene expression. Evolution & Development. 12 (4), 373-382 (2010).
- Thisse, C., Thisse, B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nature Protocols. 3 (1), 59-69 (2008).
- Cheung, M., Briscoe, J. Neural crest development is regulated by the transcription factor Sox9. Development. 130 (23), 5681-5693 (2003).
- Knight, R. D., Nair, S., Nelson, S. S., Afshar, A., Javidan, Y., Geisler, R., Rauch, G. J., Schilling, T. F. lockjaw encodes a zebrafish tfap2a required for early neural crest development. Development. 130 (23), 5755-5768 (2003).
- Yang, J. W., Jeong, B. C., Park, J., Koh, J. T. PHF20 positively regulates osteoblast differentiation via increasing the expression and activation of Runx2 with enrichment of H3K4me3. Scientific Reports. 7 (1), 8060 (2017).
- Ganju, R. K., Brubaker, S. A., Meyer, J., Dutt, P., Yang, Y., Qin, S., Newman, W., Groopman, J. E. The α-chemokine, stromal cell-derived factor-1α, binds to the transmembrane G-protein-coupled CXCR-4 receptor and activates multiple signal transduction pathways. Journal of Biological Chemistry. 273 (36), 23169-23175 (1998).
- Cai, Y., Xin, X., Yamada, T., Muramatsu, Y., Szpirer, C., Matsumoto, K. Assignments of the genes for rat pituitary adenylate cyclase activating polypeptide (Adcyap1) and its receptor subtypes (Adcyap1r1, Adcyap1r2, and Adcyap1r3). Cytogenetic and Genome Research. 71 (2), 193-196 (1995).
- Stolt, C. C., Rehberg, S., Ader, M., Lommes, P., Riethmacher, D., Schachner, M., Bartsch, U., Wegner, M. Terminal differentiation of myelin-forming oligodendrocytes depends on the transcription factor Sox10. Genes & Development. 16 (2), 165-170 (2002).
- Kimura, T., Takehana, Y., Naruse, K. pnp4a is the causal gene of the Medaka Iridophore mutant guanineless. G3: Genes, Genomes, Genetics. 7 (4), 1357-1363 (2017).
- Monsoro-Burq, A. H. A rapid protocol for whole-mount in situ hybridization on Xenopus embryos. Cold Spring Harbor Protocols. (8), pp.pdb-prot4809 (2007).
- Schulz, C. In situ hybridization to Drosophila testes. Cold Spring Harbor Protocols. (8), pp.pdb-prot4764 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten