Method Article
Wholemount ibridazione In Situ per gli embrioni Astyanax
In questo articolo
Riepilogo
Questo protocollo permette la visualizzazione dell'espressione genica in embrionali Astyanax caverna. Questo approccio è stato sviluppato con l'obiettivo di massimizzare il segnale di espressione genica, riducendo al minimo una colorazione di fondo non specifici.
Abstract
Negli ultimi anni, è stato rilasciato un genoma di bozza per la caverna cieca messicano (Astyanax mexicanus), rivelando l'identità di sequenza per migliaia di geni. Ricerca priore in questo sistema modello emergente capitalizzati sulle indagini di genoma complete che hanno identificato numerosi loci di carattere ereditario quantitativo (QTL) associati con vari fenotipi di grotta-collegata. Tuttavia, la capacità di connettersi alla base ereditabile per cambiamento fenotipico di geni di interesse rimane una sfida significativa. Una tecnica che può facilitare la comprensione più profonda del ruolo di sviluppo in troglomorfiche evoluzione è intero-monta l'ibridazione in situ. Questa tecnica può essere implementata per confrontare l'espressione genica tra forme e superficie-caverne, nominare candidato geni alla base di QTL stabilito, identificare geni di interesse dagli studi di sequenziamento di nuova generazione o sviluppare altri direttamente approcci basati sulla scoperta. In questo rapporto, presentiamo un protocollo semplice, supportato da una lista di controllo flessibile, che può essere ampiamente adattato per l'uso ben oltre il sistema di studio presentato. Si spera che questo protocollo può servire come una vasta risorsa per la comunità di Astianatte e oltre.
Introduzione
L'ibridazione in situ è un metodo comune per la colorazione di tessuti fissi per visualizzare gene expression pattern1. Questa tecnica è stata eseguita per anni in altri tradizionali2 e sistemi di modello non tradizionali3 , per una varietà di studi biologici. Tuttavia, diversi passi e reagenti sono necessari per eseguire correttamente questa procedura. Per i ricercatori che non hanno mai eseguito questa tecnica, dando inizio al processo può essere intimidatorio a causa i molti passaggi. Ulteriormente, la natura lunga di questa procedura si presta ad errori tecnici, che possono essere difficile da risolvere.
L'obiettivo generale di questo articolo è di presentare un metodo semplice e diretto che renderà accessibile ad un vasto pubblico questa tecnica di ibridazione. Per ridurre l'introduzione di errori, vi presentiamo un approccio diretto che produce alta qualità gene espressione macchiatura e riduce al minimo segnale di fondo non specifici. Questa procedura è simile ad altri approcci sviluppati in sistemi modello tradizionale, come Danio rerio4. Qui, ci proponiamo di facilitare l'implementazione attenta di ogni passo usando una lista scaricabile (Supplemental File 1), per promuovere attenzione l'attuazione del protocollo. La spiegazione razionale per questa operazione è quello di facilitare organizzazione attraverso i molti passaggi di questa procedura. Questo articolo è adatto per i ricercatori interessati a condurre l'ibridazione in situ del intero-supporto nello sviluppo di embrioni, ma non hanno ancora eseguito la procedura. Il vantaggio dell'approccio scelto per i ricercatori Astyanax è che è stato testato e dimostrato in caverna e superficie pesce morph, facilitando in tal modo l'analisi dell'espressione comparativa. Il metodo presentato può essere utilizzato dai ricercatori negli studi su Astyanax e altri sistemi.
Protocollo
Tutti i metodi descritti qui sono stati approvati dal istituzionale Animal Care e uso Committee (IACUC) dell'Università di Cincinnati (protocollo n. 10-01-21-01).
1. fissazione
- Isolare il numero desiderato di Astyanax mexicanus embrioni da un serbatoio di allevamento e difficoltà ~ 50 embrioni alla volta. Se gli embrioni sono grandi e vecchi, può essere necessario fissare 25 alla volta per garantire anche fissazione.
- A seconda dell'età dell'embrione, utilizzare il metodo IACUC-approvato dell'anestesia. Per vecchi embrioni con un sistema nervoso funzionante, sacrificare embrioni tramite overdose di anestetico. Di conseguenza, posizionare gli embrioni in una soluzione di ~ 1% tricaina (tamponato a pH 7.4) per ridurre al minimo il dolore e il disagio per l'organismo.
- Una volta che gli embrioni sono insensibili al tatto, sistema acqua contenente tricaina di sostituire e aggiungere ~ 1 mL di tampone fosfato salino (PBS, pH 7.4): 1x.
- Rimuovere la soluzione di PBS e aggiungere 1 mL di paraformaldeide al 4% (PFA). Difficoltà embrioni durante la notte a 4 ° C.
Attenzione: PFA è pericoloso (vale a dire, è infiammabile ed è una pelle e polmone irritante), maneggiare con cura.
2. disidratazione
- Per disidratare gli embrioni, rimuovere la soluzione fissante e sciacquare con 1 mL di PBS. Posizionare fiale contenenti embrioni ad un angolo (tra 30° e 45°), su un agitatore di piattaforma durante il risciacquo. Continuare a lavare gli embrioni due volte, 5 min a lavaggio.
- Se gli embrioni hanno ancora un corion, collocare 50 tutti gli embrioni in un 100 mm x 25 mm piastra di Petri e attentamente isolarli da chorions utilizzando due set di Pinzette da orologiaio (ad es., #5 forcipe) sotto un microscopio.
Nota: Durante la procedura descritta qui sotto e per il resto del protocollo, con attenzione rimuovere tutto il liquido dal passaggio precedente utilizzando pulito, vetro Pasteur pipette prima di aggiungere la soluzione successiva - Disidratare gli embrioni in una serie di lavaggi sempre più concentrate di metanolo (MeOH) descritto di seguito. Le diluizioni sono basate su un volume totale di 1 mL con 500 µ l di soluzione andando in ogni flaconcino di vetro da 4 mL. Eseguire tutti i passaggi di disidratazione a temperatura ambiente (TA) su shaker a piattaforma.
- Rimuovere con attenzione la soluzione PBS. Aggiungere una soluzione al 25% MeOH (250 µ l di MeOH) + 750 µ l di PBS. Agitare delicatamente su un agitatore di piattaforma per 5 min.
- Rimuovere con attenzione la soluzione di MeOH 25%. Aggiungere una soluzione di 50% MeOH (500 µ l di MeOH) + 500 µ l di PBS. Agitare delicatamente su un agitatore di piattaforma per 5 min.
- Rimuovere con attenzione la soluzione di MeOH 50%. Aggiungere una soluzione di MeOH 75% (750 µ l di MeOH) + 250 µ l di PBS. Agitare delicatamente su un agitatore di piattaforma per 5 minuti.
- Rimuovere con attenzione la soluzione di MeOH 75%. Aggiungere una soluzione di MeOH 100% (1 mL di MeOH). Agitare delicatamente su un agitatore di piattaforma per 5 min, ripetere questo passo 3 volte.
- A questo punto, conservare gli embrioni disidratati, come necessario, in loro fiale di vetro a-20 ° C (lungo termine). In alternativa, procedere direttamente al giorno 1 del protocollo.
3. giorno 1: reidratazione
- Ottenere embrioni disidratati dal congelatore a-20 ° C (o procedere direttamente dal punto 2.5).
- Ordinare gli embrioni utilizzando una pipetta Pasteur. Uno può ordinare basata su morfotipo (cioè, grotta e/o superficie), e il numero di geni valutati in ogni esperimento. Non ci sono solitamente non più di 12 embrioni per flaconcino una volta ordinato. Per mantenere l'organizzazione, è possibile utilizzare nastro colorato laboratorio per designare i flaconcini e le pipette per ciascun gene. Gli embrioni rimarranno nel flacone stesso in tutto l'intero protocollo.
Nota: Posto la punta della pipetta Pasteur in 100% EtOH per sterilizzarlo tra utilizza. - Impostare un bagnomaria con agitazione a 70 ° C deve essere utilizzato in un passaggio successivo. Attentamente, tirare fuori il MeOH in fiale di embrioni ordinati e sostituire con 500 µ l di nuovo 100% MeOH. Lavare brevemente (~ 1 min) su shaker a piattaforma.
- Reidratare embrioni in una crescente concentrazione di 1X PBS con Tween 20 (PBT, vedi sotto) su shaker a piattaforma. Le diluizioni sono basate su un volume di diluizione finale di 1 mL con 500 µ l in ogni flaconcino.
- Aggiungere una soluzione al 25% PBT (250 µ l di PBT, 750 µ l di MeOH). Agitare delicatamente su un agitatore di piattaforma per 5 min.
- Rimuovere con attenzione la soluzione PBT 25%. Aggiungere una soluzione al 50% PBT (500 µ l di PBT, 500 µ l di MeOH). Agitare delicatamente su un agitatore di piattaforma per 5 min.
- Rimuovere con attenzione la soluzione di 50% PBT. Aggiungere una soluzione di 75% PBT (750 µ l di PBT, 250 µ l di MeOH). Agitare delicatamente su un agitatore di piattaforma per 5 min.
- Rimuovere con attenzione la soluzione PBT di 75%. Aggiungere una soluzione al 100% PBT (1 mL di PBT). Agitare delicatamente su un agitatore di piattaforma per 5 min, ripetere questo passo 3 volte.
4. giorno 1: Digestione e fissazione
- Preparare una soluzione di K (PK) proteinasi aggiungendo 1 µ l di PK (20 mg/mL) a 2 mL di PBT.
- In previsione di passaggi successivi, è possibile ottenere le aliquote congelate di buffer di ibridazione (IBRID - e Ibrid +; vedere Supplemental File 2 e 3 File supplementare) e PFA da stoccaggio-20 ° C.
- Consentire PFA scongelare a TA.
- Porre le aliquote di ibrid - e Ibrid + in un bagno di acqua 70 ° C rotante. Posizionare tutti i reagenti e fiale all'interno di un piccolo "guarnizione" con un fondo di rete, all'interno l'apparecchio del bagno d'acqua galleggianti. In questo modo semplice aggiunta e rimozione di tubi e flaconi dal bagno di acqua rotante di 70 ° C.
- Aggiungere delicatamente la soluzione PK per il o i flaconcini di embrioni garantendo tutti i tessuti sono completamente ricoperti di soluzione. Digerire gli embrioni per ~ 12 min in soluzione di lavoro di PK nell'agitatore di piattaforma.
Nota: La lunghezza di digestione può essere variata dallo sperimentatore per garantire risultati ottimali. - Sfilare delicatamente la soluzione PK e brevemente inondare il flaconcino con PBT per diluire qualsiasi restante PK.
- Erogare la soluzione PBT e sostituire con 500 µ l di nuovo PBT. Lasciare la soluzione di sciacquare l'agitatore di piattaforma per 5 min.
- Disegnare i PBT e sostituire con 500 µ l di scongelati 4% PFA. Consentire gli embrioni a incubare per 20 minuti sull'agitatore piattaforma a TA.
- Prelevare il 4% PFA e brevemente inondare il flaconcino con PBT per diluire qualsiasi PFA rimanente. Sfilare il PBT e sostituire con 500 µ l di PBT fresco. Consentire gli embrioni sciacquare per 5 minuti sull'agitatore piattaforma. Ripetere questo passaggio 4 volte di più.
5. giorno 1: Prehybridization
- Posto 500 µ l di pre-riscaldato IBRID-soluzione nel flaconcino. Con attenzione è possibile posizionare la cuvetta nei 70 ° C acqua bagno (all'interno di guarnizioni) senza agitazione, per 5 min.
- Erogare la soluzione di ibridazione e inondare il flaconcino con 500 µ l di soluzione Hyb + pre-riscaldato. Posizionare la cuvetta indietro nel bagno di acqua 70 ° C con agitazione (40 giri). Incubare per entrambi 4 h, o durante la notte.
Nota: Un'incubazione di 4 h produrrà un protocollo completo in situ che durerà per 4 giorni in totale. Qui, questo passo si presenta come un'incubazione overnight, che produrrà un protocollo di durata di 5 giorni in totale.
6. giorno 2: ibridazione
- Introdurre un'aliquota di ibrid + dal congelatore a-20 ° C in agitazione bagno acqua calda per 5 min.
- Sfilare il Hyb + dal flaconcino e sostituire con 500 µ l di pre-riscaldato Hyb +. A questa soluzione, aggiungere con cautela 2 µ l di sonda RNA per ogni flaconcino. Agitare delicatamente il flaconcino per garantire una distribuzione omogenea della sonda.
- Incubare la soluzione Hyb + (con sonda aggiunto) nel bagno d'acqua calda di 70 ° C per una notte mentre si stringono a 40 giri/min.
Nota: Si può riutilizzare soluzione Hyb + (con sonda). Per questo, prendere Hyb + con sonda dalla prima esecuzione dal congelatore a-20 ° C e posto in una vasca di acqua calda per 5 min, Sostituisci Hyb + dal giorno 1 protocollo con Hyb + con sonda e consentire l'incubazione durante la notte in bagno di acqua calda.
7. giorno 3: Preparazione soluzione
- Preparare le provette microcentrifuga etichettate Hyb + con la sonda RNA "gene di interesse". Preparare la serie di diluizioni che verranno utilizzati durante il giorno 3.
- Utilizzando 6 tubi separati, preparare la seguente serie di diluizioni di ibrid - e salino sodio citrato (SSC, 0-100%) in un volume di 1 mL e posto nei 70 ° C in agitazione acqua vasca: tubo 1 = 100% IBRID-(1 mL di ibrid-): tubo 2 = 25% 2 x SSC (250 µ l di 2 x SSC, 750 µ l di ibrid-); Tubo 3 = 50% 2 x SSC (500 µ l di 2X SSC, 500 µ l di ibrid-); Tubo 4 = 75% 2 x SSC (750 µ l di 2X SSC, 250 µ l di ibrid-); Tubo 5 = 100% 2 x SSC (1 mL di 2 x SSC); Tubo 6 = 100% 0,2 x SSC (2 mL di 0,2 x SSC).
Nota: Essere vigili della concentrazione di SSC, che cambia da 2x a 0.2 x. - Utilizzando 4 separare tubi, preparare la seguente serie di diluizioni di PBT e SSC in un volume di 1 mL e inserire a RT: tubo 1 = 25% PBT (250 µ l di PBT, 750 µ l di 0.2 x SSC); Tubo 2 = 50% PBT (500 µ l di PBT, 500 µ l di 0,2 x SSC); Tubo 3 = 75% PBT (750 µ l di PBT, 250 µ l di 0,2 x SSC); Tubo 4 = 100% PBT (1mL di PBT).
- Preparare una provetta da 2 mL di tampone acido maleico, contenente la soluzione di lavoro di Tween 20 (MABT).
- Preparare due provette coniche da 15 mL di soluzione di blocco. In ogni provetta, aggiungere 0,2 g di reagente bloccante a 10 mL di MABT (vedere Supplemental File 4). Posizionare entrambi i tubi un mixer nutante (o shaker a piattaforma) fino al completamente dissolto nella soluzione (fino a 3 h).
- Utilizzando 6 tubi separati, preparare la seguente serie di diluizioni di ibrid - e salino sodio citrato (SSC, 0-100%) in un volume di 1 mL e posto nei 70 ° C in agitazione acqua vasca: tubo 1 = 100% IBRID-(1 mL di ibrid-): tubo 2 = 25% 2 x SSC (250 µ l di 2 x SSC, 750 µ l di ibrid-); Tubo 3 = 50% 2 x SSC (500 µ l di 2X SSC, 500 µ l di ibrid-); Tubo 4 = 75% 2 x SSC (750 µ l di 2X SSC, 250 µ l di ibrid-); Tubo 5 = 100% 2 x SSC (1 mL di 2 x SSC); Tubo 6 = 100% 0,2 x SSC (2 mL di 0,2 x SSC).
8. giorno 3: Rimozione della sonda
- Erogare la soluzione Hyb + (con sonda) con un pipetta Pasteur di vetro e posizionarlo in una sterile, etichettato microcentrifuga. Conservare questo tubo nel congelatore di-20 ° C per un uso futuro (se la sonda-etichettatura è successo).
- Con attenzione aggiungere 500 µ l di calde SSC/IBRID-diluizioni (indicato sotto). Incubare in ciascuna delle seguenti soluzioni per 10 minuti ciascuno, a bagnomaria con agitazione i 70 ° C.
- Incubare in sequenza con 100% IBRID-(1 mL di ibrid-), 25% 2 x SSC (250 µ l di 2 x SSC, 750 µ l di ibrid-), 50% 2 x SSC (500 µ l di 2 x SSC, 500 µ l di ibrid-), 75% 2 x SSC (750 µ l di 2 x SSC, 250 µ l di ibrid-), 100% 2 x SSC (1 mL di 2 x SSC) , 100% 0,2 x SSC (2 mL di 0.2 x SSC).
- Dopo l'ultimo passo, Incubare in ciascuna delle seguenti soluzioni per 10 minuti ciascuno. Tutte le incubazioni seguente si terrà al RT il shaker a piattaforma: 25% PBT (250 µ l di PBT, 750 µ l di 0.2 x SSC), 50% PBT (500 µ l di PBT, 500 µ l di 0.2 x SSC), 75% PBT (750 µ l di PBT, 250 µ l di 0.2 x SSC) , 100% PBT (1 mL di PBT).
- Dopo un'incubazione di 10 min, rimuovere il 100% PBT e aggiungere 500 µ l di MABT in ogni flaconcino. Ripetere questo passaggio due volte per 5 min.
9. giorno 3: blocco
- Rimuovere MABT da ogni flaconcino e flood con soluzione premiscelata bloccante da uno dei tubi (preparati al punto 7.1.4). Flaconcino posto su un mixer nutante per ~ 4 h a TA.
- Aggiungere 2 µ l di Anti-DIG-AP Fab frammenti nel secondo flaconcino da 10 mL di soluzione (preparata al punto 7.1.4) e brevemente vortice di blocco.
- Riempire quasi completamente ogni flaconcino con soluzione (~ 5 mL) di blocco e nutante mixer durante la notte in frigorifero a 4 ° C.
10. giorno 4: MABT risciacqui
- Preparare un flaconcino stock di 10% siero di capra normale (NGS) nel MABT (aggiungere 100 µ l di NGS a 900 µ l di MABT).
- Erogare la soluzione bloccante in ogni fiala e aggiungere 500 µ l di miscela NGS/MABT in ogni flaconcino. Consentire gli embrioni a incubare per 25 min a RT sull'agitatore piattaforma.
- Sostituire la miscela NGS/MABT con 500 µ l di 100% MABT. Incubare per 30 min a RT sull'agitatore piattaforma. Eseguire questo risciacquo 11 più volte durante il giorno ogni 30 min.
- Riempire il flacone con 100% MABT e posto su un mixer nutante durante la notte in una camera frigorifero o cabina a 4 ° C.
11. giorno 5: Visualizzazione della sonda
- Preparare un'aliquota di 50 mL di tampone di fosfatasi alcalina (AP) (vedere Supplemental File 5). Combinare le operazioni seguenti in una provetta conica 50 mL avvolta nella carta stagnola per limitare l'esposizione alla luce: 5 mL di 1 M Tris (pH 9.5), 5 mL di 50 mM MgCl2, 5ml di 1% Tween 20, 5 mL di NaCl di 1m, 30 mL di ddH2O.
- Rimuovere MABT e sostituire con 1 mL di tampone di AP (tubo avvolto in carta stagnola). Lasciarlo a lavare per 5 min. Farlo due volte a garantire la rimozione completa di MABT.
- Buffer di AP di rimuovere e sostituire con 1 mL di tampone di AP con 3,5 μL 5-bromo-4-chloro-3'-indolyphosphate (BCIP) e 4,5 μL di nitro blu di tetrazolio (NBT). Sostituire con preparati AP buffer/NBT/BCIP una volta ogni ora fino a quando la reazione è completa. Monitorare attentamente, controllando ogni 15 minuti, per permettere la reazione di colorazione che si terrà fino a quando è stato raggiunto il livello desiderato di macchiatura. Se precipitato comincia a formarsi, sostituire la soluzione al più presto.
- Fermare la reazione di colorazione risciacquando gli embrioni nel buffer di AP 100% fresco (senza NBT/BCIP) per 5 min continua risciacqui in PBT fino al raggiungimento dei livelli ottimali del segnale (con quantità minime di colorazione di fondo). Continuare a sciacquare gli esemplari con l'aumento di diluizioni di PBT in AP Buffer come segue: 25% PBT (250 μL di PBT, 750 μL di tampone di AP), risciacquo per 5 min; 50% PBT (500 μL di PBT, 500 μL di tampone di AP), risciacquo per 5 min; 75% PBT (750 μL di PBT, 250 μL di tampone di AP), sciacquare per 5 min.
- Sciacquare gli embrioni in ~ 5 mL del 100% PBT nutante mixer fino a quando lo si desidera una colorazione di fondo minimo viene raggiunto. Interruttore fuori con PBT fresco più volte. Questo potrebbe richiedere fino a diversi giorni.
- Quando risciacquo è completi, lavare gli embrioni in 500 μL di PBS sterile su un agitatore di piattaforma. Eseguire questo risciacquo due volte per 5 min. Dopo lava PBS, post-fissare i campioni in 500 μL di 4% PFA per 1h a RT su un agitatore di piattaforma. In alternativa, difficoltà durante la notte in 1 mL di 4% PFA in frigorifero a 4 ° C.
- Sostituire il fissativo con fresco, PBS sterile. Eseguire questo risciacquo almeno due volte per 5 min posto gli embrioni in ~ 4 mL di PBS sterile al 100% e archiviare a lungo termine a 4 ° C.
12. imaging
- Compongono una piastra di imaging in una capsula Petri usando agarosio al 3% e tampone TAE.
Nota: Quantità dipendono da quanti piatti sono necessari. Piastre possono essere riutilizzati più volte. È consigliabile che uno stampo rettangolare poco profondo è disposto in di Petri mentre il gel è di raffreddamento al fine di creare una depressione per contenenti gli embrioni sulla piastra. - Posizionare gli embrioni sulla piastra in PBS.
Nota: Si consiglia di versare delicatamente gli embrioni nella piastra invece di pipettaggio fuori perché è stato trovato che che si attacchi all'interno delle pipette di plastica. - Utilizzare la microscopia chiara al fine di visualizzare ciascun embrione. Utilizzare una sonda smussata per manovrare gli embrioni nella posizione desiderata.
- Prendere un'immagine quando l'embrione è nella posizione desiderata. Si noti che è importante acquisire le immagini degli embrioni all'interno di un paio di settimane di macchiatura completato per evitare una potenziale riduzione della macchia.
Risultati
In questo rapporto, forniamo un approccio semplice e diretto per eseguire etichettatura delle embrionali Astyanax campioni per analisi di espressione genica di alta qualità. Questa tecnica può essere effettuata in quattro o cinque giorni, e ogni passaggio principale della procedura è rappresentata in un diagramma di flusso con codifica a colori (Figura 1). Una volta completato, macchiati embrioni dovrebbero harbor un'etichetta cromatica viola scura nei tessuti che esprimono il gene di interesse particolare. Abbiamo implementato con successo questo protocollo sia Pachón caverna (Figura 2A–B, E) in embrioni di pesce superficie (Figura 2–S, F).
Gli embrioni di caverna sono stati macchiati per due fattori di trascrizione che tessuti neurali della cresta in anticipo etichetta, Sox9 e Tfap2a5,6. Gli embrioni con l'etichetta per l'espressione di Sox9 dimostrano chiara etichettatura nella sviluppo archi branchiali e delle pinne pettorali (punte di freccia gialle, Figura 2A). Si noti che la colorazione è praticamente assente nel sacco vitellino o dei metameri in via di sviluppo sul fianco (Figura 2A). Allo stesso modo, Tfap2a espressione è evidente nelle parti della testa in via di sviluppo, come pure le cellule neurali della cresta migrazione precoce (Figura 2B, arrowhead) lungo la regione dorsale fianco dell'embrione. Il terzo gene rappresentanza ha presentato per gli embrioni caverna è Phf20a, un indicatore di differenziazione degli osteoblasti7. Si noti la macchiatura positiva nelle porzioni del mesoderma somitic e la testa posteriore che è destinati a dare origine a tessuto osseo (Figura 2E, punte di freccia).
Negli embrioni di pesce di superficie, abbiamo sondato per i geni CXCR, Adcyap1ae Sox10. CXCR codifica per un recettore di membrana-limitano G-proteina che lega di chemochine CXC8. Positivo di etichettatura è presente nelle regioni isolate della testa e fianco (Figura 2F, punte di freccia), così come alcune singole celle che ricopre il sacco vitellino. Gene d'attivazione dell'adenilato ciclasi polipeptide, Adcyap1a, si esprime nelle regioni del sistema nervoso centrale, compreso le cellule ipofisarie. Nota l'espressione altamente specifico in mazzi appaiati, bilaterali delle cellule sulla funzione dorsale dell'embrione; così come una più grande regione di espressione del midline (Figura 2, punte di freccia). Infine, presentiamo l'espressione di Sox10, un fattore di trascrizione quali etichette dalla cresta neurale e del oligodendrocyte cellule iniziali10. La macchiatura positiva altamente specifico è evidente come un indicatore in anticipo di cresta neurale a sinistra e a destra dell'embrione dorsale (Figura 2D, punte di freccia).
Presentiamo in ciascuno dei due tipi di confondimento problemi che potrebbero verificarsi altri investigatori. Il primo problema che si incontra periodicamente è punctate pagliuzze di contrassegno non specifico. Queste macchie possono sorgere come precipitato dai finali risciacqui MABT, o il buffer di AP durante le reazioni di colorazione. Un esempio di questo contrassegno non specifico è evidente il sacco vitellino di un embrione di pesce superficie macchiato per l'espressione di Pnp4a. Questo gene codifica per un enzima (fosforilasi del nucleoside della purina) che facilita la produzione di pigmentazione cangiante11. Questo gene è il primo evidente nell'occhio in via di sviluppo e la vescica natatoria. Le macchioline punctate osservato in alcuni esemplari di superficie (Figura 3A), sono stati eliminati dai lavaggi frequenti e sostituzione del Buffer AP + BCIP/NBT nelle fasi finali del protocollo (Figura 3B). Un secondo problema che si incontra periodicamente è l'espressione diffusa, in gran parte non-specifico di geni che altrimenti sarebbe produrre un pattern di espressione distinta. Un esempio è il gene BMP4, che appare come un modello ampiamente diffuso con bassi livelli di cromogeno presente in tutto il campione (Figura 3). In casi come questi, noi identificare una diversa regione del gene, amplificare in un vettore un generalmente ed eseguire una nuova sintesi di sonda (Vedi supplementare 6 File). L'esempio di un esemplare di controllo (senza sonda) (Figura 3D) viene fornito per illustrare la natura diffusa e non specifici della nostra sonda BMP4 fallito.
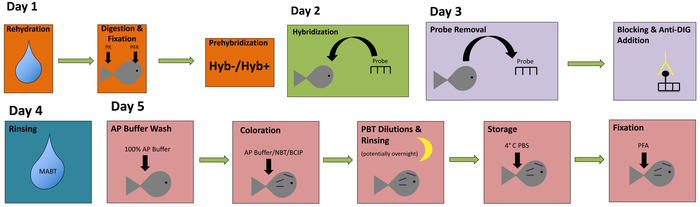
Figura 1: un semplice diagramma di flusso per intero-monta l'ibridazione in situ. Questo diagramma di flusso utilizza la codifica a colori per illustrare i passaggi principali dell'ibridazione in situ. Clicca qui per visualizzare una versione più grande di questa figura.
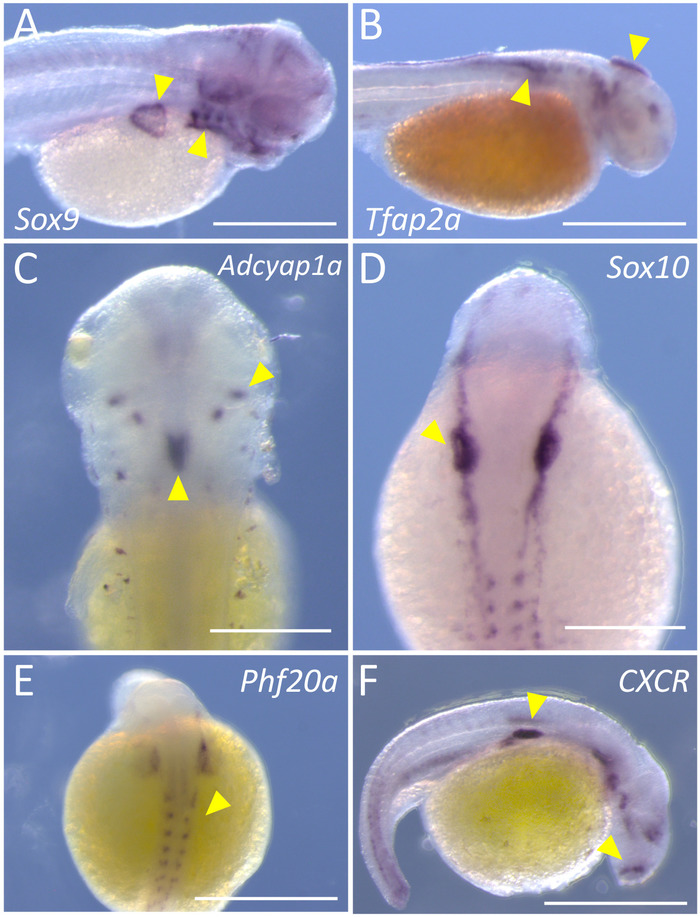
Figura 2: La macchiatura rappresentativo per sei geni, utilizzando sia la grotta che superficie morph di Astianatte. (A) immagine mostra la colorazione specifica (frecce gialle) di Sox9 sul lato laterale di destra di una post-fecondazione 72h (hpf) Pachón caverna (45x). (B) colorazione specifica per Tfap2a è evidente sul lato laterale di destra di un 36 hpf Pachón caverna (45x). (C) bilaterale e del midline colorazione (frecce gialle) delle Adcyap1a sono etichettati nella regione dorsale di un pesce di superficie hpf 72 (100x). (D) colorazione di Sox10 nei tessuti neurali della cresta di un pesce di superficie hpf 24 (100 x). (E) Phf20a di seguito viene illustrato un modello debole, ma chiaro, di espressione nella regione dorsale di un 24 hpf Pachón caverna (100x). (F), la citochina ricevitore, CXCR, è espresso in regioni distinte del lato laterale di destra di un pesce di superficie hpf 24 (100 x). Scala bar a, B, E, F = 0,5 mm; scala bar in C, D = 2,5 mm. per favore clicca qui per visualizzare una versione più grande di questa figura.
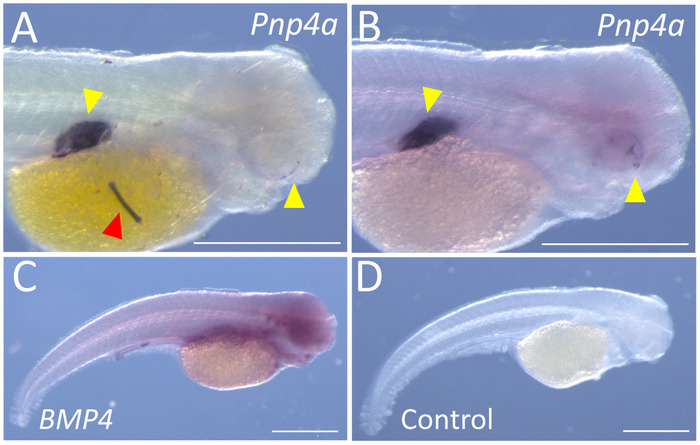
Figura 3: esempi rappresentativi di risultati non ottimali per intero-monta l'ibridazione in situ. (A) la colorazione specifica è evidente al fianco di precipitato non specifici e/o detriti (freccia rossa) sull'aspetto laterale di destra di un 72 hpf Pachón caverna (100x). (B) la stessa sonda visualizzata nella A, raffigurante la stessa colorazione modelli senza precipitato o di sfondo in un 72 hpf Pachón caverna (100x). (C) il fianco laterale di destra di un 72 hpf Pachón caverna dimostra diffusa, non specifico la macchiatura per BMP4 45 ingrandimenti. (D) A 72 hpf Pachón caverna sottoposto al presente protocollo, senza l'aggiunta della sonda (45x). Scala bar = 0,5 mm. per favore clicca qui per visualizzare una versione più grande di questa figura.
Discussione
A causa della vulnerabilità di RNA alla degradazione, una delle fasi più critiche nel protocollo riguarda la sintesi sterile della sonda RNA. Tuttavia, se una sonda è generata con attenzione e fornisce buoni risultati, può essere riutilizzato nelle successive reazioni di colorazione. Un secondo punto cruciale è la produzione accurata di tutti i reattivi utilizzati in tutto il protocollo. Poiché questo protocollo coinvolge diversi giorni e molti piccoli passi, è essenziale che tutti i reagenti sono accuratamente prodotte e archiviate in maniera sterile. Inoltre, è di fondamentale importanza che lo sperimentatore tiene traccia di ogni passo nel protocollo. Abbiamo trovato che l'elenco di controllo fornito di passaggi può essere estremamente utile per garantire il completamento accurato e preciso di ogni aspetto del presente protocollo.
Non abbiamo spesso modificare il protocollo che abbiamo presentato qui. Tuttavia, gli investigatori possono svolgere incubazioni di sonda a temperature diverse rispetto a quelle suggerite (cioè, 70 ° C). Lievi variazioni di temperature di ibridazione urterà associazione di sonde del RNA, e pertanto, cercando la temperatura di ibridazione ottimale può influire positivamente la qualità della colorazione. Per quanto riguarda la risoluzione dei problemi, consigliamo vivamente altri ricercatori di utilizzare l'elenco di controllo fornito con questo articolo (Supplemental File 1). Mantenere attenzione records è un primo passo necessario per garantire alta qualità di colorazione. Una seconda modifica minore è suggerita è quello di eseguire la reazione di colorazione finale senza dondolo (ad es., senza mettere su una piattaforma shaker o nutator). La ragione di questo è che periodicamente si nota la produzione di precipitato, che presumibilmente deriva dalla soluzione tampone AP. Questo precipitato solitamente overstains un colore scuro e crea punctata (aspecifici) sfondo sul tessuto macchiato. Per ridurre al minimo la produzione di questo precipitato, prepariamo il buffer di AP sterilizzati prima di ogni reazione. Ulteriormente, una volta NBT e BCIP aggiunti al buffer, sostituiremo con tampone fresco e BCIP/NBT ogni ora fino a quando non viene completata la reazione di colorazione.
Una limitazione per il metodo proposto è che una macchia cromatica è stata utilizzata per la visualizzazione di espressione genica. Noi preferiamo questo approccio poiché è conveniente e richiede solo la microscopia chiara di visualizzare. Se uno era interessato a valutare le differenze quantitative, suggeriamo che usano una reazione di colorazione fluorescente. Ciò consentirà la semi-quantificazione, ad esempio, attraverso il confronto di unità relative fluorescente di espressione tra esperimenti.
Protocolli per l'ibridazione in situ sono disponibili ampiamente sul web12,13, così come nelle pubblicazioni scientifiche. Il protocollo che presentiamo è stato sviluppato specificamente per il nostro sistema di modello, Astyanax mexicanus. Abbiamo usato questo protocollo per macchiare l'espressione di diverse decine di geni e sento che fornisce costantemente risultati di alta qualità. Un vantaggio significativo di questo protocollo è l'elenco di controllo dettagliata degli elementi per consentire al ricercatore di eseguire più attività garantendo nel contempo completamento accurata di tutti i passaggi del presente protocollo. Speriamo che questo protocollo servirà come una risorsa utile per altri ricercatori nel campo e di là e anticipare che questa tecnica di laboratorio comune sosterrà future scoperte che collegano il genotipo al fenotipo nella caverna cieca messicano.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori desiderano ringraziare i membri del laboratorio lordo per utili commenti su questo manoscritto. Vogliamo riconoscere quattro studenti delle scuole superiori che hanno utilizzato questo protocollo durante stage estivi nel 2017 e 2018, tra cui Christine Cao, Michael Warden, Aki Li e David Nwankwo. HL è stato sostenuto da una borsa di studio UC biologia staminali durante l'estate del 2017. Quest'opera è stata sostenuta da sovvenzioni da parte della National Science Foundation (DEB-1457630 a JBG) e il National Institutes of Dental e Craniofacial Research (NIH; DE025033 a JBG).
Materiali
| Name | Company | Catalog Number | Comments |
| 10 mL Serological Pipette | VWR | 89130-888 | |
| 1000 mL Filtration Unit | VWR | 89220-698 | |
| 15 mL Conical | VWR-Greiner | 82050-278 | |
| 25 mL Serological Pipette | VWR | 89130-890 | |
| 250 mL Filtration Unit | VWR | 89220-694 | |
| 5 mL Serological Pipette | VWR | 89130-886 | |
| 50 mL Conical | VWR-Falcon | 21008-940 | |
| 500 mL Filtration Unit | VWR | 89220-696 | |
| Anti-Digoxigenin-AP, Fab fragments | Sigma-Roche | 11093274910 | |
| BCIP | Sigma-Aldrich | B8503-1G | 1 g |
| Blocking Solution | Sigma-Roche | 11 096 176 001 | 50 g |
| Citric Acid | Fisher Scientific | A104-500 | 500 g |
| DIG RNA Labeling Kit (SP6/T7) | Sigma-Roche | 11175025910 | |
| Eppendorf Tubes | VWR | 20170-577 | |
| Ethanol | Fisher-Decon | 04-355-223 | 1 Gal |
| Formamide | Thermo Fisher Scientific | 17899 | 100 mL |
| Glass dram vials | VWR | 66011-041 | 1 Dr |
| Glass Pipettes | Fisher Scientific | 13-678-8A | |
| HCl | Thermal-ScientificPharmco-AAPER | 284000ACS | 500 mL |
| Heparin | Sigma | H3393-25KU | |
| Magnesium Chloride-crystalline | Fisher Scientific | M33-500 | 500 g |
| Maleic Acid | Sigma | M0375-100g | 100g |
| Methanol | Fisher Scientific | A452-4 | 4L |
| Molecular-grade Water (RNase-free) | VWR | 7732-18-5 | 500 mL |
| NaCl | Fisher Scientific | S271-3 | 3 kg |
| NaOH pellets | Fisher Scientific | S318-500 | 500 g |
| NBT Substrate powder | ThermoFisher Scientific | 34035 | 1 g |
| Normal Goat Serum | Fisher-Invitrogen | 31873 | |
| Nutating Mixer | VWR | 82007-202 | |
| Paraformaldehyde | Sigma | 158127-500g | 500 g |
| PBS 10x | Fisher Scientific | BP399-20 | 20L |
| Proteinase K (200mg/10ml) | Qiagen | 19133 | 10 mL |
| Plastic Pipettes | VWR-Samco | 14670-147 | |
| RNAse | Sigma | R2020-250mL | 250 mL |
| Shaking Water Bath 12 L | VWR | 10128-126 | 12 L |
| Standard Analog Shaker | VWR | 89032-092 | |
| Tris | Sigma Millipore-OmniPur | 9210-500GM | 500 g |
| tRNA Yeast | Fisher-Invitrogen | 15401011 | 25 mg |
| Tween 20 | Sigma | P9416-50mL | 50 mL |
| Vortex-Genie 2 | Fisher Scientific-Scientific Industries, Inc | 50-728-002 | |
| Lithium Chloride (LiCl) | Sigma-Aldrich | 203637-10G | 10 g |
Riferimenti
- Valentino, K. L., Eberwine, J. H., Barchas, J. D. . In situ hybridization: Application to neurobiology. , (1987).
- Mugrauer, G., Alt, F. W., Ekblom, P. N-myc proto-oncogene expression during organogenesis in the developing mouse as revealed by in situ hybridization. The Journal of Cell Biology. 107 (4), 1325-1335 (1988).
- Kerney, R., Gross, J. B., Hanken, J. Early cranial patterning in the direct‐developing frog Eleutherodactylus coqui revealed through gene expression. Evolution & Development. 12 (4), 373-382 (2010).
- Thisse, C., Thisse, B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nature Protocols. 3 (1), 59-69 (2008).
- Cheung, M., Briscoe, J. Neural crest development is regulated by the transcription factor Sox9. Development. 130 (23), 5681-5693 (2003).
- Knight, R. D., Nair, S., Nelson, S. S., Afshar, A., Javidan, Y., Geisler, R., Rauch, G. J., Schilling, T. F. lockjaw encodes a zebrafish tfap2a required for early neural crest development. Development. 130 (23), 5755-5768 (2003).
- Yang, J. W., Jeong, B. C., Park, J., Koh, J. T. PHF20 positively regulates osteoblast differentiation via increasing the expression and activation of Runx2 with enrichment of H3K4me3. Scientific Reports. 7 (1), 8060 (2017).
- Ganju, R. K., Brubaker, S. A., Meyer, J., Dutt, P., Yang, Y., Qin, S., Newman, W., Groopman, J. E. The α-chemokine, stromal cell-derived factor-1α, binds to the transmembrane G-protein-coupled CXCR-4 receptor and activates multiple signal transduction pathways. Journal of Biological Chemistry. 273 (36), 23169-23175 (1998).
- Cai, Y., Xin, X., Yamada, T., Muramatsu, Y., Szpirer, C., Matsumoto, K. Assignments of the genes for rat pituitary adenylate cyclase activating polypeptide (Adcyap1) and its receptor subtypes (Adcyap1r1, Adcyap1r2, and Adcyap1r3). Cytogenetic and Genome Research. 71 (2), 193-196 (1995).
- Stolt, C. C., Rehberg, S., Ader, M., Lommes, P., Riethmacher, D., Schachner, M., Bartsch, U., Wegner, M. Terminal differentiation of myelin-forming oligodendrocytes depends on the transcription factor Sox10. Genes & Development. 16 (2), 165-170 (2002).
- Kimura, T., Takehana, Y., Naruse, K. pnp4a is the causal gene of the Medaka Iridophore mutant guanineless. G3: Genes, Genomes, Genetics. 7 (4), 1357-1363 (2017).
- Monsoro-Burq, A. H. A rapid protocol for whole-mount in situ hybridization on Xenopus embryos. Cold Spring Harbor Protocols. (8), pp.pdb-prot4809 (2007).
- Schulz, C. In situ hybridization to Drosophila testes. Cold Spring Harbor Protocols. (8), pp.pdb-prot4764 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon