Method Article
Wholemount hibridación In Situ de embriones de Astyanax
En este artículo
Resumen
Este protocolo permite la visualización de la expresión génica en embrionario Astyanax cavefish. Este enfoque ha sido desarrollado con el objetivo de maximizar la señal de la expresión del gene, minimizando tinción de fondo inespecífica.
Resumen
En los últimos años, se ha publicado un genoma de proyecto para el cavefish mexicano ciego (Astyanax mexicanus), revelando las identidades de la secuencia de miles de genes. Investigaciones anteriores en este sistema de modelo emergente capitalizan completo genoma las investigaciones que han identificado numerosos loci de rasgos cuantitativos (QTL) asociados con diversos fenotipos asociados de cueva. Sin embargo, la capacidad de conectarse a genes de interés a la base hereditaria para cambio fenotípico sigue siendo un desafío significativo. Una técnica que puede facilitar la comprensión del papel del desarrollo en evolución troglomórphico es todo montaje hibridación in situ. Esta técnica puede implementarse directamente comparar la expresión génica entre las formas de vivienda de la cueva y la superficie, nominar a candidato genes subyacentes QTL establecido, identificar los genes de interés de los estudios de secuenciación de próxima generación, o desarrollar otros enfoques basados en el descubrimiento. En este informe, presentamos un protocolo simple, apoyado por una lista flexible, que puede ser ampliamente adaptada para su uso más allá del sistema de estudio presentados. Se espera que este protocolo puede servir como un recurso amplio para la comunidad de Astyanax y más allá.
Introducción
Hibridación in situ es un método común para la tinción de tejidos fijos para visualizar patrones de expresión de gen1. Esta técnica se ha realizado durante años en otros tradicionales2 y sistemas no tradicionales3 modelo para una variedad de estudios biológicos. Sin embargo, varios pasos y los reactivos son necesarios para realizar exitosamente este procedimiento. Para los investigadores que nunca han realizado esta técnica, iniciando el proceso puede ser intimidante debido a los muchos pasos necesarios. Además, la larga naturaleza de este procedimiento se presta a errores técnicos, que pueden ser difícil solucionar problemas.
El objetivo general de este artículo es presentar un método simple y sencillo que se puede representar esta técnica de hibridación accesible a un público más amplio. Para reducir la introducción de errores, presentamos un enfoque sencillo que produce alta calidad gene expresión manchas y reduce al mínimo la señal de fondo no específicas. Este procedimiento es similar a otros enfoques desarrollados en los sistemas de modelo tradicional, como Danio rerio4. Aquí, nuestro objetivo es facilitar la aplicación cuidadosa de cada paso usando una lista descargable (1 archivo suplementario), para promover el cuidado de aplicación del protocolo. La justificación para hacer esto es para facilitar la organización a través de los muchos pasos implicados en este procedimiento. Este artículo es apropiado para los investigadores interesados en realizar la hibridación in situ de montaje en conjunto en el desarrollo de embriones, pero aún no han realizado el procedimiento. La ventaja del enfoque elegido por los investigadores de Astyanax es que ha sido probado y comprobado en cavefish y morfos de peces de superficie, facilitando así el análisis comparativo de la expresión. El método presentado puede utilizarse por los investigadores en estudios sobre Astyanax y otros sistemas.
Protocolo
Todos los métodos aquí descritos han sido aprobados por el cuidado institucional del Animal y el Comité uso (IACUC) de la Universidad de Cincinnati (Protocolo nº 10-01-21-01).
1. fijación
- Número deseado de Astyanax mexicanus embriones de un tanque de cría de aislar y fijar ~ 50 embriones a la vez. Si los embriones son grandes y viejos, puede ser necesario fijar 25 a la vez para asegurar incluso la fijación.
- Dependiendo de la edad del embrión, utilizar el método aprobado por el IACUC de anestesia. Para embriones mayores con un sistema de los nervios, sacrificar embriones por sobredosis de anestésico. Por lo tanto, coloque los embriones en una solución de ~ 1% Metanosulfonato (tamponado a pH 7.4) para minimizar el dolor y las molestias para el organismo.
- Una vez que los embriones son insensibles al tacto, sistema de agua que contiene Metanosulfonato y se añade ~ 1 mL de 1 x de tampón fosfato salino (PBS, pH 7,4).
- Retirar la solución de PBS y añadir 1 mL de 4% paraformaldehido (PFA). Fijar los embriones durante la noche a 4 ° C.
PRECAUCIÓN: El PFA es peligroso (es decir, es inflamable y es una piel y pulmón irritante), manipúlelas con cuidado.
2. deshidratación
- Para deshidratar los embriones, quite la solución fijadora y enjuague con 1 mL de PBS. Coloque los frascos que contienen el embrión en un ángulo (entre 30° y 45°), en un agitador de plataforma durante el enjuague. Seguir lavar los embriones dos veces, 5 minutos por lavado.
- Si los embriones todavía tienen un corion, coloque todos los 50 embriones 100 mm x 25 mm placa de Petri y aislarlos cuidadosamente de las chorions con dos juegos de pinzas de relojero (p. ej., #5 pinzas) bajo un microscopio.
Nota: Durante los pasos descritos a continuación y para el resto del Protocolo, retire con cuidado todo el líquido del paso anterior usando limpia, pipetas de Pasteur de vidrio antes de agregar la solución de la siguiente - Deshidratar los embriones en una serie de lavados cada vez más concentradas de metanol (MeOH) se describe a continuación. Las diluciones se basan en un volumen total de 1 mL con 500 μl de solución en cada frasco de vidrio de 4 mL. Realizar todos los pasos de deshidratación a temperatura ambiente (RT) en el agitador de plataforma.
- Retire con cuidado la solución de PBS. Añadir una solución al 25% MeOH (250 μl de MeOH) + 750 μl de PBS. Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución al 25% MeOH. Agregar una solución de 50% MeOH (500 μl de MeOH) + 500 μl de PBS. Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución al 50% MeOH. Agregar una solución de MeOH 75% (750 μl de MeOH) + 250 μl de PBS. Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución de MeOH 75%. Añadir una 100% MeOH solución (1 mL de MeOH). Suavemente agite en una coctelera de plataforma por 5 min, repetir este paso 3 veces.
- En este punto, almacenar embriones deshidratados, según sea necesario, en los frascos de cristal a-20 ° C (largo plazo). Por otra parte, proceder directamente a día 1 del protocolo.
3. día 1: rehidratación
- Obtener embriones deshidratados de congelador de-20 ° C (o proceden directamente de paso 2.5).
- Clasificar los embriones utilizando una pipeta Pasteur. Uno puede clasificar en base a morfotipo (es decir, cueva o superficie), y el número de genes evaluados en cada experimento. Hay generalmente no más de 12 embriones por frasco una vez ordenados. Para mantener la organización, utilice cinta de color lab para designar los frascos y pipetas para cada gen. Embriones se quedarán en el mismo frasco en todo el conjunto del protocolo.
Nota: Lugar la punta de la pipeta Pasteur en 100% EtOH a esterilizar entre usa. - Establecer un baño a 70 ° C para ser utilizado en un paso posterior. Con cuidado, extraer el MeOH en viales de embriones clasificados y reemplazar con 500 μl de nuevo 100% MeOH. Lavado brevemente (~ 1 min) en el agitador de plataforma.
- Rehidratar los embriones en un aumento de la concentración de 1 x PBS con Tween 20 (PBT, ver más abajo) en el agitador de plataforma. Las diluciones se basan en un volumen de dilución final de 1 mL con 500 μL en cada frasco.
- Añadir una solución al 25% PBT (250 μl de PBT, 750 μl de MeOH). Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución al 25% PBT. Añadir una solución al 50% PBT (500 μl de PBT, 500 μl de MeOH). Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución PBT 50%. Agregar una solución de 75% de la PBT (750 μl de PBT, 250 μl de MeOH). Sacuda suavemente en un agitador de plataforma durante 5 minutos.
- Retire con cuidado la solución PBT de 75%. Agregar una solución PBT 100% (1 mL de PBT). Suavemente agite en una coctelera de plataforma por 5 min, repetir este paso 3 veces.
4. día 1: Digestión y fijación
- Preparar una solución de K (PK) de proteinasa añadiendo 1 μl de PK (20 mg/mL) y 2 mL de PBT.
- En previsión de realizar los siguientes pasos, obtener alícuotas congeladas de búferes de hibridación (Hyb - y Hyb +; ver archivo suplementario 2 y 3 de archivo suplementario) y PFA de almacenamiento-20 ° C.
- Permiten PFA descongelar a TA.
- Colocar alícuotas de Hyb - y Hyb + giratorio 70 º C baño María. Coloque todos los reactivos y los frascos dentro de una pequeña "Junta" con un fondo de malla, dentro del aparato de baño de agua flotante. Esto permite simple adición y eliminación de tubos y viales desde el baño de agua de 70 ° C rotación.
- Suavemente Agregar solución de PK para los frascos de embriones garantizando todos los tejidos están completamente cubiertos con la solución. Digerir los embriones por ~ 12 min en solución de trabajo de PK en el agitador de plataforma.
Nota: Se puede variar la longitud de la digestión por el investigador para asegurar resultados óptimos. - Extraer suavemente la solución PK y brevemente de la inundación el frasco con PBT para diluir cualquier PK restantes
- Extraer la solución PBT y reemplazar con 500 μl de PBT nuevo. Deje que la solución de enjuague en el agitador de plataforma durante 5 minutos.
- Extraer PBT y reemplazar con 500 μl de descongelado 4% PFA. Permitir que los embriones destinados a incubar durante 20 minutos en el agitador de plataforma a TA.
- Extraer el 4% del PFA y brevemente de la inundación el frasco con PBT para diluir cualquier PFA restantes. Extraer el PBT y reemplazar con 500 μl de PBT fresco. Permite embriones enjuagar durante 5 minutos en el agitador de plataforma. Repetir este paso 4 veces más.
5. día 1: Prehybridization
- Lugar 500 μl de solución de Hyb precalentada en el frasco. Coloque con cuidado el frasco en la 70 ° C agua baño (dentro de las juntas) sin agitación, durante 5 minutos.
- Extraer la solución Hyb y frasco con 500 μl de precalentado Hyb + solución de inundaciones. Coloque el frasco en el baño de agua de 70 ° C con agitación (40 rpm). Incubar durante cada 4 horas o toda la noche.
Nota: Un 4 h de incubación dará un protocolo completo in situ que tendrá una duración de 4 días en total. Aquí, este paso se presenta como una incubación durante la noche, que dará como resultado un protocolo de duración 5 días en total.
6. día 2: hibridación
- Colocar una alícuota de Hyb + del congelador de-20 ° C en el baño de agua caliente moviendo durante 5 minutos.
- Extraer el Hyb + del frasco y vuelva a colocar con 500 μl de Hyb precalentada. A esta solución, cuidadosamente añadir 2 μl de sonda de RNA a cada frasco. Agitar suavemente el frasco para asegurar una distribución uniforme de la sonda.
- Incubar durante una noche Hyb + (con sonda agregada) solución en el baño de agua caliente de 70 ° C y agitación a 40 rpm.
Nota: Uno puede volver a utilizar solución de Hyb + (con sonda). Para esto, tomar Hyb + con sonda de la primera ejecución desde el congelador de-20 ° C y lugar en un baño de agua caliente durante 5 minutos reemplazar Hyb + desde el día 1 de protocolo con Hyb + con sonda permite incubar durante la noche en baño de agua caliente.
7. día 3: Preparación de la solución
- Preparar los tubos de microcentrífuga etiquetados Hyb + con la sonda de RNA "gen de interés". Preparar la serie de diluciones que se utilizarán durante el día 3.
- 6 tubos separados, preparar la siguiente serie de diluciones de Hyb - y citrato salino de sodio (SSC, 0 a 100%) en un volumen de 1 mL y un lugar en los 70 ° C agitando baño de agua: tubo de 1 del = 100% Hyb-(1 mL de Hyb-): 2 = 25% de tubo 2 x SSC (250 μl de 2 x SSC, 750 μl de Hyb-); Tubo 3 = 50% 2 x SSC (500 μl de 2 x SSC, 500 μl de Hyb-); Tubo 4 = 75% 2 x SSC (750 μl de 2 x SSC, 250 μl de Hyb-); Tubo 5 = 100% 2 x SSC (1 mL de 2 x SSC); Tubo 6 = 100% 0,2 x SSC (2 mL de 0,2 x SSC).
Nota: Estar alerta de la concentración de SSC, como cambia de 2 x 0.2 x. - 4 separar los tubos, preparar la siguiente serie de diluciones de PBT y SSC en un volumen de 1 mL y colocar en RT: tubo 1 = 25% PBT (250 μl de PBT, 750 μl de 0.2 x SSC); Tubo 2 = 50% PBT (500 μl de PBT, 500 μl de 0,2 x SSC); Tubo 3 = 75% PBT (750 μl de PBT, 250 μl de 0,2 x SSC); Tubo 4 = 100% PBT (1mL de PBT).
- Preparar un tubo con 2 mL de tampón ácido maleico que contiene solución de Tween 20 (MABT) de trabajo.
- Preparar dos tubos cónicos de 15 mL de solución de bloqueo. En cada tubo, agregue 0.2 g de reactivo de bloqueo a 10 mL de MABT (ver 4 archivos suplementarios). Coloca ambos tubos en un mezclador oscilante (o agitador de plataforma) hasta totalmente disuelto en la solución (hasta 3 h).
- 6 tubos separados, preparar la siguiente serie de diluciones de Hyb - y citrato salino de sodio (SSC, 0 a 100%) en un volumen de 1 mL y un lugar en los 70 ° C agitando baño de agua: tubo de 1 del = 100% Hyb-(1 mL de Hyb-): 2 = 25% de tubo 2 x SSC (250 μl de 2 x SSC, 750 μl de Hyb-); Tubo 3 = 50% 2 x SSC (500 μl de 2 x SSC, 500 μl de Hyb-); Tubo 4 = 75% 2 x SSC (750 μl de 2 x SSC, 250 μl de Hyb-); Tubo 5 = 100% 2 x SSC (1 mL de 2 x SSC); Tubo 6 = 100% 0,2 x SSC (2 mL de 0,2 x SSC).
8. día 3: Retiro de la sonda
- Extraer Hyb + (con sonda) solución con un pipeta Pasteur de vidrio y colóquelo en un estéril, etiquetados tubo de microcentrífuga. Conservar este tubo en el congelador de-20 ° C para su uso futuro (si es acertado sondeo etiquetado).
- Cuidadosamente Añadir 500 μl de las SSC/Hyb-diluciones calientes (indicado abajo). Incubar en cada una de las siguientes soluciones para 10 min en el baño de 70 ° C.
- Incubar secuencialmente con 100% Hyb-(1 mL de Hyb-), 25% 2 x SSC (250 μl de 2 x SSC, 750 μl de Hyb-), 50% 2 x SSC (500 μl de 2 x SSC, 500 μl de Hyb-), 75% 2 x SSC (750 μl de 2 x SSC, 250 μl de Hyb-), 100% 2 x SSC (1 mL de 2 x SSC) , 100% 0.2 x SSC (2 mL de 0.2 x SSC).
- Tras el último paso, incubar en cada una de las siguientes soluciones para 10 minutos cada uno. Todas las incubaciones siguientes llevará a cabo en RT en el agitador de plataforma: 25% PBT (250 μl de PBT, 750 μl de 0.2 x SSC), 50% PBT (500 μl de PBT, 500 μl de 0.2 x SSC), 75% PBT (750 μl de PBT, 250 μl de 0.2 x SSC) , 100% PBT (1 mL de PBT).
- Después de una incubación de 10 minutos, retirar el 100% PBT y agregar 500 μl de MABT en cada frasco. Repita este paso dos veces durante 5 minutos.
9. día 3: bloqueo de
- Retire MABT cada vial e inundaciones con solución premezclada bloqueo de uno de los tubos (preparados en el paso 7.1.4). Coloque el frasco en un mezclador oscilante ~ 4 h a TA.
- Añadir 2 μl de Anti-DIG-AP Fab fragmentos al segundo frasco de 10 mL de solución (preparada en el paso 7.1.4) y brevemente de vórtice de bloqueo.
- Llenar cada frasco casi en su totalidad con bloqueo solución ~ 5 mL y coloque en el mezclador oscilante durante la noche en un refrigerador a 4 ° C.
10. día 4: MABT enjuagues
- Preparar un frasco común de 10% de suero normal de cabra (NGS) en MABT (añadir 100 μl de NGS a 900 μl de MABT).
- Extraer la solución de bloqueo en cada frasco y agregar 500 μl de mezcla NGS/MABT en cada frasco. Permitir que los embriones destinados a incubar durante 25 min a temperatura ambiente en el agitador de plataforma.
- Vuelva a colocar la mezcla NGS/MABT con 500 μl del 100% MABT. Incubar durante 30 min a temperatura ambiente en el agitador de plataforma. Realizar este enjuague 11 veces más durante el día cada 30 minutos.
- Llene el frasco con 100% MABT y el lugar en un mezclador oscilante durante la noche en un refrigerador o vestidor cámara a 4 º C.
11. día 5: Visualización de la sonda
- Preparar una alícuota de 50 mL de tampón de fosfatasa alcalina (AP) (ver 5 de archivo suplementario). Combinar los siguientes en un tubo cónico de 50 mL, envuelto en papel de aluminio para limitar la exposición a la luz: 5 mL de 1 M Tris (pH 9.5), 5 mL de 50 mM de MgCl2, 5 mL de 1% Tween 20, 5 mL de 1 M NaCl, 30 mL de ddH2O.
- MABT de quitar y reemplazar con 1 mL de tampón de AP (tubo envuelto en papel de aluminio). Que lavar durante 5 minutos. Hacer esto dos veces para asegurar el retiro completo del MABT.
- Elimine el AP de tampón y reemplazar con 1 mL de tampón de AP con 3,5 μL 5-bromo-4-chloro-3'-indolyphosphate (BCIP) y 4.5 μL de nitro azul de tetrazolio (NBT). Reemplazar con tampón/NBT/BCIP recién preparado de la AP una vez cada hora hasta que la reacción es completa. Seguir de cerca, comprobar cada 15 minutos, para permitir la reacción de coloración que tendrá lugar hasta conseguir el nivel deseado de la coloración. Precipitado se empieza a formar, reemplazar la solución cuanto antes.
- Detener la reacción de coloración de enjuague los embriones en fresco 100% buffer de AP (sin NBT/BCIP) para 5 minutos continuar enjuagues en PBT hasta que se alcancen niveles óptimos de señal (con cantidades mínimas de tinción de fondo). Continuar a enjuagar las muestras con el aumento de las diluciones de PBT en tampón de AP como sigue: 25% PBT (250 μL de PBT, 750 μL de tampón de AP), enjuague durante 5 minutos; 50% PBT (500 μL de PBT, 500 μL de tampón de AP), enjuague durante 5 minutos; 75% PBT (750 μL de PBT, 250 μL de tampón de AP), enjuague durante 5 minutos.
- Enjuague los embriones en ~ 5 mL de PBT en mezclador oscilante hasta que desea tinción de fondo mínima se alcanza el 100%. Interruptor hacia fuera con PBT fresca varias veces. Esto puede tomar hasta varios días.
- Cuando enjuague es completados, lave los embriones en 500 μL de PBS estéril en un agitador de plataforma. Realizar este enjuague dos veces durante 5 minutos. Después lava de la PBS, después fijar las muestras en 500 μL de 4% PFA por 1 h a temperatura ambiente en un agitador de plataforma. Alternativamente, fijar un día para otro en 1 mL de 4% PFA en el refrigerador a 4 ° C.
- Vuelva a colocar el fijador fresco, PBS estéril. Realizar este enjuague por lo menos dos veces por 5 min lugar los embriones en ~ 4 mL de PBS estéril 100% y almacenar a largo plazo a 4 ° C.
12. proyección de imagen de
- Hacer una placa de imagen en una placa Petri con agarosa al 3% y Tampón TAE.
Nota: Las cantidades dependen de cuantas placas se necesitan. Las placas pueden reutilizarse varias veces. Se recomienda que un molde rectangular de poca profundidad se coloca en la caja Petri, mientras que el gel es refrigeración con el fin de crear una depresión para contener los embriones en la placa. - Coloque los embriones en la placa en el PBS.
Nota: Es mejor verter suavemente los embriones en la placa en el lugar de pipeteo sacarlos porque se ha encontrado que se pegue en el interior de las pipetas de plástico. - Uso de la microscopia ligera con el fin de visualizar cada embrión. Utilizar una sonda Roma para maniobrar los embriones hasta la posición deseada.
- Tomar una imagen cuando el embrión está en la posición deseada. Tenga en cuenta que es importante tomar imágenes de embriones dentro de un par de semanas de tinción completa para evitar la potencial degradación de la mancha.
Resultados
En este informe, nos ofrece un enfoque sencillo para realizar etiquetado de embrionario Astyanax muestras para análisis de expresión génica de alta calidad. Esta técnica puede realizarse en cuatro o cinco días, y cada paso principal en el procedimiento se representa en un diagrama de flujo con códigos de color (figura 1). Una vez completado, embriones teñidos deben abrigar una etiqueta cromática púrpura oscurezca en tejidos que expresan el gen de interés particular. Con éxito hemos implementado este protocolo en cavefish Pachón (figura 2A–B, E) y embriones de peces de superficie (figura 2–D, F).
Los embriones cavefish fueron manchados para dos factores de transcripción los tejidos de la cresta neural temprana etiqueta, Sox9 y Tfap2a5,6. Embriones para la expresión de Sox9 demuestran un etiquetado claro en el desarrollo branquial arcos y la aleta pectoral (puntas de flecha amarillo, figura 2A). Tenga en cuenta que la coloración está prácticamente ausente en el saco vitelino o los somitas en desarrollo en el flanco (figura 2A). Del mismo modo, expresión Tfap2a es evidente en las porciones de la cabeza en desarrollo, así como la temprana migración de la cresta neural las células (figura 2B, punta de flecha) a lo largo de la región de flanco dorsal del embrión. El tercer gen representante presentado para embriones cavefish es Phf20a, un marcador de diferenciación de osteoblastos7. Tenga en cuenta la coloración positiva en las porciones del mesodermo somitic y cabeza posterior que está destinada para dar lugar al tejido óseo (Figura 2E, puntas de flecha).
En embriones de peces de superficie, hemos sondeado para los genes CXCR, Adcyap1ay Sox10. CXCR codifica un receptor de membrana-limite de proteína G que une a CXC quimioquinas8. Etiquetado positivo está presente en regiones aisladas de la cabeza y costado (figura 2F, puntas de flecha), así como algunas células individuales que cubre el saco vitelino. El gene del polipéptido activador de adenilato ciclasa, Adcyap1a, se expresa en las regiones del sistema nervioso central, incluyendo las células de la pituitarias. Nota la expresión altamente específica en grupos de pares, bilaterales de células en el aspecto dorsal del embrión; así como una región más grande de la expresión de la línea media (figura 2, las puntas de flecha). Por último, presentamos la expresión de Sox10, un factor de transcripción que etiquetas primeros nervios de la cresta y oligodendrocyte células10. Coloración positiva altamente específico es evidente como un marcador temprano de la cresta neural a la izquierda y derecha del embrión dorsal (Figura 2D, puntas de flecha).
Presentamos cada uno de los dos tipos de problemas de confusión que pueden encontrarse con otros investigadores. El primer problema se encuentra periódicamente es manchas punteadas de etiquetado no específicos. Estas manchas surgen como precipitado de los enjuagues MABT finales o el búfer de AP durante las reacciones de coloración. Un ejemplo de este etiquetado no específico es evidente en el saco vitelino de un embrión de pez superficie teñido para la expresión de Pnp4a. Este gen codifica una enzima (phosphorylase del nucleoside del purine) que facilita la producción de pigmentación iridiscente11. Este gen es primero evidente en el ojo en desarrollo y en la vejiga natatoria. Las manchas punteadas observaron en algunas muestras de superficie (Figura 3A), fueron eliminados por lavados frecuentes y reemplazo de la AP Buffer + NBT/BCIP en las etapas finales del Protocolo (figura 3B). Una segunda cuestión que uno se encuentra con periódicamente es el difusa, en gran parte no-específica de expresión de los genes que de lo contrario produciría un patrón de expresión distinto. Un ejemplo es el gen de la BMP4, que aparece como un patrón difuso en gran parte con bajos niveles de cromógeno presente a lo largo de la muestra (figura 3). En estos casos, generalmente identificamos una región diferente del gen, amplificar en un vector y realizar una nueva síntesis de la sonda (ver 6 archivos suplementarios). El ejemplo de una muestra control (sin sonda) (figura 3D) se proporciona para ilustrar la naturaleza difusa y no específica de la fallida sonda de BMP4 .
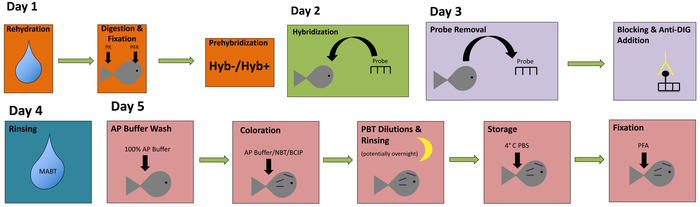
Figura 1: Diagrama de flujo simple para hibridación in situ de todo Monte. El diagrama de flujo utiliza codificación de color para ilustrar los principales pasos de hibridación in situ. Haga clic aquí para ver una versión más grande de esta figura.
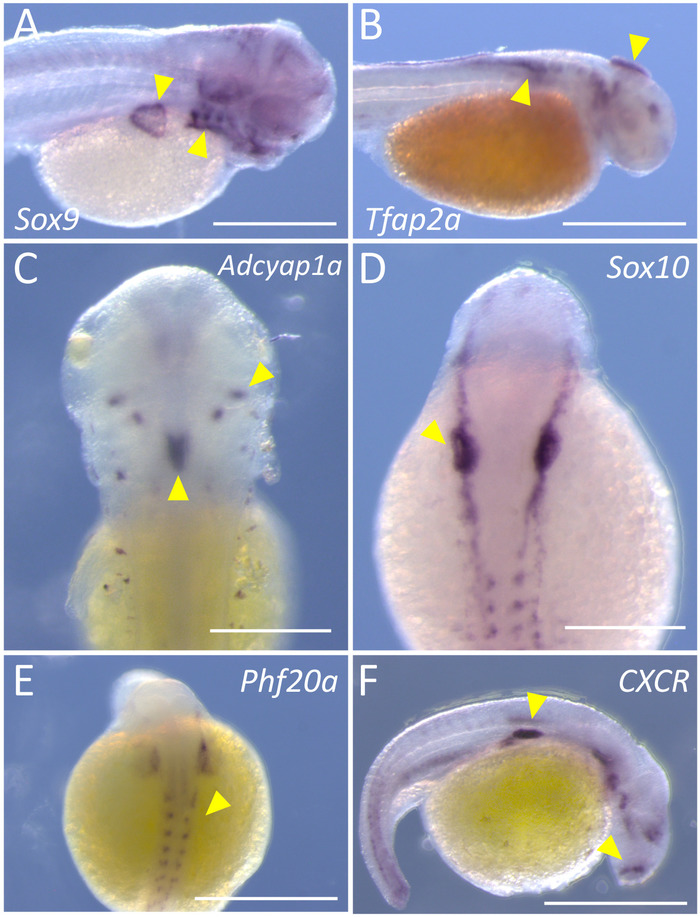
Figura 2: Representante de coloración para seis genes, con cueva y superficie morfos de Astyanax. Imagen de (A) muestra la tinción específica (flechas amarillas) de Sox9 en la parte lateral derecha de una fertilización después de 72 h (hpf) cavefish Pachón (45 x). (B) tinción específica para Tfap2a es evidente en el lado lateral derecho de una 36 hpf cavefish Pachón (45 x). Bilateral (C) y media (flecha amarilla) la coloración de Adcyap1a están marcados en la región dorsal de un pez superficie 72 de hpf (100 x). (D) tinción de Sox10 en los tejidos de la cresta neural de un pez superficie 24 de hpf (100 x). (E) Phf20a muestra un patrón débil, pero claro, de la expresión en la región dorsal de un 24 hpf cavefish Pachón (100 x). (F) la citocina receptor CXCR, se expresa en distintas regiones de la parte lateral derecha de un pez superficie 24 de hpf (100 x). Barras de escala en A, B, E, F = 0,5 mm; escala de barras en C, D = 2,5 mm. haga clic aquí para ver una versión más grande de esta figura.
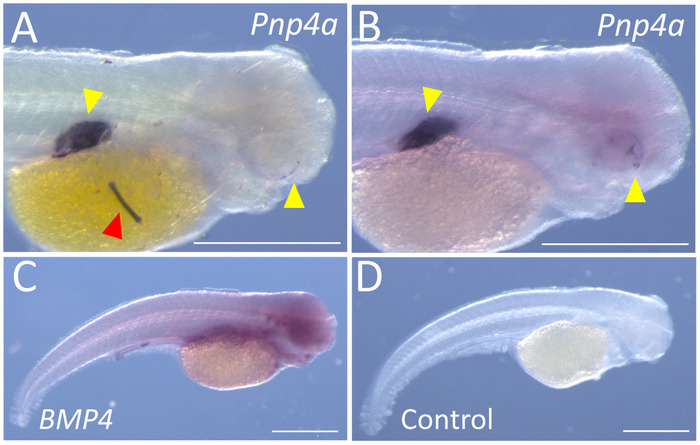
Figura 3: ejemplos representativos de resultados subóptimos para hibridación in situ de montaje conjunto. (A) la coloración específica es evidente junto con precipitado no específicos o desechos (flecha roja) en el aspecto lateral derecho de un 72 hpf cavefish Pachón (100 x). (B) la misma sonda visualizada en el A, que representa la misma tinción patrones sin precipitado o de fondo en un 72 hpf cavefish Pachón (100 x). (C) el flanco lateral derecho de una 72 hpf cavefish Pachón demuestra difusa, inespecífica tinción de BMP4 en el aumento de 45 x. (D) A 72 hpf Pachón cavefish sometidos a este protocolo, sin la adición de sondeo (45 x). Barras de escala = 0,5 mm. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Debido a la vulnerabilidad del ARN a la degradación, uno de los pasos más importantes en el protocolo refiere a la síntesis estéril de la sonda de RNA. Sin embargo, si una sonda se genera con cuidado y proporciona buenos resultados, pueden ser reutilizado en posteriores reacciones de tinción. Un segundo paso crucial es la cuidadosa producción de todos los reactivos utilizados en el protocolo. Puesto que este protocolo trata de varios días y muchos pequeños pasos, es esencial que todos los reactivos son exactamente producido y almacenado de manera estéril. Además, es fundamental que el investigador mantiene la pista cuidadosa de cada paso en el protocolo. Hemos encontrado que la lista proporcionada de pasos puede ser extremadamente útil para asegurar el cumplimiento exacto y preciso de cada aspecto de este protocolo.
A menudo no modificamos el protocolo que presentamos aquí. Sin embargo, los investigadores pueden realizar sonda incubaciones a distintas temperaturas que el sugerido (es decir, 70 ° C). Ligeros cambios en las temperaturas de hibridación impactara Unión de sondas de RNA, y por lo tanto, la buscando la temperatura óptima de hibridación puede impactar positivamente la calidad de la coloración. Con respecto a la solución de problemas, le recomendamos otros investigadores a utilizar la lista de verificación con este artículo (1 archivo suplementario). Mantener cuidado de registros es un primer paso necesario para garantizar la alta calidad de tinción. Una segunda modificación menor sugiere es para llevar a cabo la reacción de coloración final sin balancearse (p. ej., sin colocar sobre un agitador de plataforma o nutator). La razón de esto es que periódicamente se nota la producción de precipitado, que probablemente surge de la solución tampón de AP. Este precipitado generalmente overstains un color oscuro y crea punteada (no específicos) fondo del tejido y teñido. Para minimizar la producción de este precipitado, preparamos buffer de AP esterilizado antes de cada reacción. Además, una vez que se han añadido al buffer de NBT y BCIP, reemplazamos con almacenador intermediario fresco y NBT/BCIP cada hora hasta que se complete la reacción de coloración.
Una limitación al método presentado es que una mancha cromática fue utilizada para la visualización de expresión génica. Preferimos este método ya que es rentable y sólo requiere de microscopía de luz para visualizar. Si uno se interesa evaluar diferencias cuantitativas, sugerimos que utilizan una reacción de coloración fluorescente. Esto permitirá la cuantificación, por ejemplo, a través de la comparación de unidades relativas de fluorescentes de expresión entre experimentos.
Protocolos de hibridación in situ están disponibles ampliamente en la web12,13, así como en publicaciones científicas. El protocolo que presentamos fue desarrollado específicamente para nuestro sistema modelo, Astyanax mexicanus. Hemos utilizado este protocolo para la expresión de varias docenas de genes de la mancha y sentir constantemente proporciona resultados de alta calidad. Una ventaja importante de este protocolo es el paso a paso lista de elementos que permiten al investigador a realizar múltiples tareas al tiempo que garantiza la terminación exacta de cada uno de los pasos de este protocolo. Esperamos que este protocolo servirá como un recurso útil a otros investigadores en el campo y más allá y anticipar que esta técnica de laboratorio común apoyará a genotipo a fenotipo en el cavefish oculto mexicano los descubrimientos futuros.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a los miembros del laboratorio de bruto para comentarios de este manuscrito. Queremos reconocer a cuatro estudiantes de secundaria que utilizan este protocolo durante las pasantías de verano en 2017 y 2018, incluyendo Christine Cao, Michael Warden, Aki Li y David Nwankwo. HL fue apoyado por una beca de madre de Biología UC durante el verano de 2017. Este trabajo fue apoyado por subvenciones de la National Science Foundation (DEB-1457630 a la JBG) y el institutos nacionales de Dental e investigación craneofacial (NIH; DE025033 a la JBG).
Materiales
| Name | Company | Catalog Number | Comments |
| 10 mL Serological Pipette | VWR | 89130-888 | |
| 1000 mL Filtration Unit | VWR | 89220-698 | |
| 15 mL Conical | VWR-Greiner | 82050-278 | |
| 25 mL Serological Pipette | VWR | 89130-890 | |
| 250 mL Filtration Unit | VWR | 89220-694 | |
| 5 mL Serological Pipette | VWR | 89130-886 | |
| 50 mL Conical | VWR-Falcon | 21008-940 | |
| 500 mL Filtration Unit | VWR | 89220-696 | |
| Anti-Digoxigenin-AP, Fab fragments | Sigma-Roche | 11093274910 | |
| BCIP | Sigma-Aldrich | B8503-1G | 1 g |
| Blocking Solution | Sigma-Roche | 11 096 176 001 | 50 g |
| Citric Acid | Fisher Scientific | A104-500 | 500 g |
| DIG RNA Labeling Kit (SP6/T7) | Sigma-Roche | 11175025910 | |
| Eppendorf Tubes | VWR | 20170-577 | |
| Ethanol | Fisher-Decon | 04-355-223 | 1 Gal |
| Formamide | Thermo Fisher Scientific | 17899 | 100 mL |
| Glass dram vials | VWR | 66011-041 | 1 Dr |
| Glass Pipettes | Fisher Scientific | 13-678-8A | |
| HCl | Thermal-ScientificPharmco-AAPER | 284000ACS | 500 mL |
| Heparin | Sigma | H3393-25KU | |
| Magnesium Chloride-crystalline | Fisher Scientific | M33-500 | 500 g |
| Maleic Acid | Sigma | M0375-100g | 100g |
| Methanol | Fisher Scientific | A452-4 | 4L |
| Molecular-grade Water (RNase-free) | VWR | 7732-18-5 | 500 mL |
| NaCl | Fisher Scientific | S271-3 | 3 kg |
| NaOH pellets | Fisher Scientific | S318-500 | 500 g |
| NBT Substrate powder | ThermoFisher Scientific | 34035 | 1 g |
| Normal Goat Serum | Fisher-Invitrogen | 31873 | |
| Nutating Mixer | VWR | 82007-202 | |
| Paraformaldehyde | Sigma | 158127-500g | 500 g |
| PBS 10x | Fisher Scientific | BP399-20 | 20L |
| Proteinase K (200mg/10ml) | Qiagen | 19133 | 10 mL |
| Plastic Pipettes | VWR-Samco | 14670-147 | |
| RNAse | Sigma | R2020-250mL | 250 mL |
| Shaking Water Bath 12 L | VWR | 10128-126 | 12 L |
| Standard Analog Shaker | VWR | 89032-092 | |
| Tris | Sigma Millipore-OmniPur | 9210-500GM | 500 g |
| tRNA Yeast | Fisher-Invitrogen | 15401011 | 25 mg |
| Tween 20 | Sigma | P9416-50mL | 50 mL |
| Vortex-Genie 2 | Fisher Scientific-Scientific Industries, Inc | 50-728-002 | |
| Lithium Chloride (LiCl) | Sigma-Aldrich | 203637-10G | 10 g |
Referencias
- Valentino, K. L., Eberwine, J. H., Barchas, J. D. . In situ hybridization: Application to neurobiology. , (1987).
- Mugrauer, G., Alt, F. W., Ekblom, P. N-myc proto-oncogene expression during organogenesis in the developing mouse as revealed by in situ hybridization. The Journal of Cell Biology. 107 (4), 1325-1335 (1988).
- Kerney, R., Gross, J. B., Hanken, J. Early cranial patterning in the direct‐developing frog Eleutherodactylus coqui revealed through gene expression. Evolution & Development. 12 (4), 373-382 (2010).
- Thisse, C., Thisse, B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nature Protocols. 3 (1), 59-69 (2008).
- Cheung, M., Briscoe, J. Neural crest development is regulated by the transcription factor Sox9. Development. 130 (23), 5681-5693 (2003).
- Knight, R. D., Nair, S., Nelson, S. S., Afshar, A., Javidan, Y., Geisler, R., Rauch, G. J., Schilling, T. F. lockjaw encodes a zebrafish tfap2a required for early neural crest development. Development. 130 (23), 5755-5768 (2003).
- Yang, J. W., Jeong, B. C., Park, J., Koh, J. T. PHF20 positively regulates osteoblast differentiation via increasing the expression and activation of Runx2 with enrichment of H3K4me3. Scientific Reports. 7 (1), 8060 (2017).
- Ganju, R. K., Brubaker, S. A., Meyer, J., Dutt, P., Yang, Y., Qin, S., Newman, W., Groopman, J. E. The α-chemokine, stromal cell-derived factor-1α, binds to the transmembrane G-protein-coupled CXCR-4 receptor and activates multiple signal transduction pathways. Journal of Biological Chemistry. 273 (36), 23169-23175 (1998).
- Cai, Y., Xin, X., Yamada, T., Muramatsu, Y., Szpirer, C., Matsumoto, K. Assignments of the genes for rat pituitary adenylate cyclase activating polypeptide (Adcyap1) and its receptor subtypes (Adcyap1r1, Adcyap1r2, and Adcyap1r3). Cytogenetic and Genome Research. 71 (2), 193-196 (1995).
- Stolt, C. C., Rehberg, S., Ader, M., Lommes, P., Riethmacher, D., Schachner, M., Bartsch, U., Wegner, M. Terminal differentiation of myelin-forming oligodendrocytes depends on the transcription factor Sox10. Genes & Development. 16 (2), 165-170 (2002).
- Kimura, T., Takehana, Y., Naruse, K. pnp4a is the causal gene of the Medaka Iridophore mutant guanineless. G3: Genes, Genomes, Genetics. 7 (4), 1357-1363 (2017).
- Monsoro-Burq, A. H. A rapid protocol for whole-mount in situ hybridization on Xenopus embryos. Cold Spring Harbor Protocols. (8), pp.pdb-prot4809 (2007).
- Schulz, C. In situ hybridization to Drosophila testes. Cold Spring Harbor Protocols. (8), pp.pdb-prot4764 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados