Method Article
Um ensaio de HS-MRM para a quantificação de proteínas de célula hospedeira em biofármacos de proteínas por espectrometria de massa líquida cromatografia iônica mobilidade QTOF
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, descrevemos um ensaio cromatográfico, juntamente com a separação de mobilidade do íon de precursores de peptídeo seguido de alta resolução (~ 30.000) MS-detecção de fragmentos de peptídeo para a quantificação dos padrões de peptídeo cravado em um anticorpo monoclonal digerir.
Resumo
A análise de impurezas de proteína de baixo nível (1-100 ppm) (por exemplo, célula hospedeira proteínas (HCPs)) em proteína biotherapeutics é um ensaio desafiador que requerem alta sensibilidade e uma ampla faixa dinâmica. Ensaios de quantificação de espectrometria de massa base para proteínas normalmente envolvem a digestão de proteínas, seguido pela reação reação seletiva múltipla/monitoramento monitoramento quantificação (SRM/MRM) de peptídeos usando uma baixa resolução em tandem (Rs ~ 1.000) espectrômetro de massa quadrupolo. Uma das limitações desta abordagem é o fenômeno de interferência observado quando o peptídeo de interesse tem o "mesmo" precursor e fragmento de massa (em termos de valores m/z) como outros peptídeos co eluição presentes na amostra (dentro de uma janela de 1-Da). Para evitar este fenômeno, propomos uma abordagem alternativa de espectrometria de massa, um ensaio MRM de alta seletividade (HS) que combina a separação de mobilidade do íon de precursores de peptídeo com o de alta resolução (~ 30.000 Rs) MS deteção de fragmentos de peptídeo. Nós exploramos os recursos desta abordagem para quantificar padrões de baixa abundância peptídeo cravado em digerir um anticorpo monoclonal (mAb) e demonstrou que tem a sensibilidade e o alcance dinâmico (pelo menos 3 ordens de magnitude) normalmente alcançado no HCP análise. Todos os seis padrões de peptídeo foram detectados em concentrações tão baixas quanto 0.1 nM (1 femtomole carregado em uma coluna cromatográfica de 2,1 mm ID) na presença de um fundo de alta-abundância de peptídeo (2 µ g de uma digest carregado de mAb na coluna). Quando se considera o MW de fosforilase coelho (97,2 kDa), de que derivam os peptídeos cravados, o LOQ deste teste é inferior a 50 ppm. Desvios-padrão relativos (RSD) das áreas de pico (n = 4 repetições) foram menos de 15% em toda a gama de toda concentração investigada (0,1-100 nM ou 1-1.000 ppm) neste estudo.
Introdução
Quantificação de grandes biomoléculas (proteínas) em ambientes industriais é atualmente baseada em imunoensaios (por exemplo, ELISA), principalmente devido a diversas vantagens: sensibilidade, alta produtividade, facilidade de uso e baixo custo por amostra. Quando aplicado para analisar as impurezas de abundância de baixa proteína (1-100 ppm de proteínas da célula hospedeira (HCPs)) presentes na terapêutica de proteína, estes ensaios biológicos normalmente fornecem a concentração total do HCP (geralmente expressada em ppm ou ng mAb HCP/mg), mas eles Não consigo identificar e medir contaminantes individuais do HCP. Vários ensaios baseados em MS recentemente têm sido desenvolvidos para complementar o ELISA ou forneça informações que Elisa não oferecem1,2,3,4,5,6 , 7 , 8 , 9. por causa da complexidade da amostra e a exigência para detectar peptídeos HCP através de uma ampla faixa dinâmica em concentração (pelo menos 3 ordens de magnitude), métodos cromatográficos multidimensionais, fracionamento de amostra extensa de concurso têm tradicionalmente foi empregado para ajudar a identificar a baixa abundância HCPs1,2,3,4,5,6,7.
Um natural passo seguinte HCP identificação e validação é rastreamento HCP (monitoramento) entre vários lotes de produtos biofarmacêuticos. Nesta situação, os métodos de LC/MS de dimensão única têm sido propostos para melhorar a taxa de transferência de amostra8,9. No entanto, a precisão e alcance dinâmico do HCP medições podem ser afetadas em um ensaio de LC/MS 1D pela esmagadora presença de peptídeos biofarmacêutica. Comparado a uma separação multidimensional, o potencial de interferência sinal19,20,21,de22 é aumentado em uma separação cromatográfica de dimensão única porque a probabilidade de precursores de peptídeo mais ser co eluição é aumentada. A incorporação de meios ortogonais para a separação de precursores de peptídeo sem estender o tempo de separação cromatográfica claramente seria vantajosa. Viajar de mobilidade (TWIM) íon onda10 tem a capacidade de resolver espectros de MS congestionados em milissegundos. Aproximadamente 500 separações de mobilidade podem ser executadas durante a eluição de um peptídeo único, assumindo uma largura do pico cromatográfico completo de 10 s e considerando-se que o tempo de execução de uma separação de IM do instrumento de mobilidade do íon é 20 ms.
Espectrometria de massa ensaios para quantificação de proteína foram desenvolvidos com sucesso nas últimas década usando o bem aceita selecionado (vários) reação monitoramento abordagem (método SRM/MRM) implementada em tandem espectrómetros de massa11, 12,13,14,15,16,17,18,19,20,21 ,22,23. Uma das limitações deste teste baixa resolução e espectrometria de massa é a interferência fenômeno19,20,21,22 observada quando o peptídeo de interesse tem o "mesmo" precursor e fragmento de massa como outros peptídeos co eluição presentes na amostra (dentro de uma janela de 1-Da). Há duas maneiras para melhorar a precisão dos métodos SRM/MRM: uma opção envolve uma etapa extra de separação ao nível do precursor para remover íons interferentes de precursor, enquanto a outra opção é aumentar a resolução MS da detecção de precursor/fragmento Evite a sobreposição de MS de sinais. O modo de aquisição de MRM (HS) alta seletividade descrito aqui tira proveito de ambas essas abordagens pelo acoplamento a separação de mobilidade do íon de precursores de peptídeo com o de alta resolução (~ 30.000 Rs) MS deteção de fragmentos de peptídeo. O ensaio descrito aqui capas de pelo menos três ordens de magnitude, que é a gama dinâmica normalmente observadas em SRM/MRM proteomics experimentos17,18,24.
O utilitário do ensaio HS-MRM para quantificação de HCP foi demonstrado pelo monitoramento a linearidade do sinal produzido por seis padrões de peptídeo cravados em diferentes concentrações (faixa de 0,1 a 100 nM) em digerir um anticorpo monoclonal.
Protocolo
1. preparação de digerir o infliximab (procedimento de ~ 24 h)
- Prepare soluções novas de 50mm de amônio bicarbonato de sódio, surfactante aniônico 1% em 50mm NH4HCO3, 500mm ditiotreitol (DTT) em 50 milímetros NH4HCO3e iodoacetamide de 500 mM (IAM) em 50 milímetros NH4HCO3.
- A 750 µ l de 50mm NH4HCO3, adicione 200 µ l de infliximab mAb (10 mg/mL) e 50 µ l de solução de surfactante aniônico 1% e desnaturar a proteína por 15 min a 60 ° C, na presença de 0,05% de surfactante aniônico.
- Adicionar 40 µ l de 500 mM DTT e reduzir a amostra por 60 min a 60 ° C, na presença de 20 mM DTT.
- Adicionar 20 µ l de 500mm IAM e alquilada a amostra por 30 min à temperatura ambiente no escuro na presença de ~ 10mm IAM.
- Adicionar o conteúdo do tubo microcentrifuga para um frasco de vidro contendo 20 µ g de MS da série sequenciamento Lys C/tripsina e digerir a amostra por 3 h a 37 ° C.
- Adicionar uma segunda enzima alíquota (20 µ g), transferir a amostra digerida para outro frasco de vidro contendo 20 µ g de MS sequenciamento-grau Lys C/tripsina e digerir a amostra durante a noite (12-15 h) a 37 ° C.
- Após a proteólise durante a noite, adicionar 5 µ l de ácido fórmico a 100% (FA) e incube a digest por 30 min a 37 ° C para decompor o ácido lábil surfactante aniônico.
- Spin para baixo o resumo para 15 min a 4.000 x g em uma centrífuga para precipitar o componente insolúvel do surfactante aniônico.
- Recuperar ~ 1.000 µ l de digerir o infliximab e colocá-lo em um frasco de amostrador automático LC; a concentração de digerir deve ser ~ 2 mg/mL. Armazene o resumo em um congelador a-20 ° C até que o sistema de LC/MS está pronto para análise.
2. cravação de padrões de peptídeo (~ 30 min)
- Descongele o infliximab digest, colocando a amostra congelada no frasco de vidro em um banco na temperatura ambiente.
- Adicione 1 mL de 0.1% FA em frasco de digerir H2O para um coelho fosforilase b (PHO) para preparar 1 µM as soluções para todos os seis padrões de peptídeo do PHO contido no frasco (o frasco contém 1 nmol de cada peptídeo).
- Preparar diluições de 4 x 1 mL da solução estoque para obter soluções contendo, 0,1, 1, 10 e 100 peptídeos nM, utilizando 0,1% PHO FA como o solvente de diluição. Preparar todas as diluições em frascos de vidro (LC auto-sampler frascos) e adicionar 100 µ l do infliximab digest como a matriz de plano de fundo para todos os frascos. Carga de 2 µ g de digerir fundo na coluna com cada injeção de 10 µ l. Preparar uma amostra em branco que contém o resumo de infliximab diluído (a mesma 01:10 diluição), com nenhum peptídeos cravados.
3. configuração do método de aquisição de dados de LC/HDMS E
Nota: O fluxo de trabalho Resumindo as etapas necessárias para configurar uma aquisição de HS-MRM é retratado na Figura 1 e é descrito em detalhes nas seções 3-6. A aquisição de um conjunto de dados HDMSE dados independente é necessária para estabelecer o tempo de retenção de cada peptídeo monitorado, o pai de m/z do íon mais abundante do peptide após ionização electrospray, e o correspondente CCS (colisão Cruz seção) derivado da separação de mobilidade do íon. Além disso, o conjunto de dados independentes de dados fornece informações sobre a m/z dos três íons mais abundantes fragmento para cada precursor do peptídeo. Na segunda etapa do fluxo de trabalho (otimização de CE), a sensibilidade do ensaio é aumentada por meio do ajuste da energia de célula de colisão para obter a mais alta intensidade de íon para cada íon fragmento. Finalmente, na etapa final, todos os parâmetros descritos acima são introduzidos para o editor de método de HS-MRM para cada peptide monitorado.
- Realizar experiências de LC/MS em um quadrupolo tempo-de-voo (QTOF) espectrômetro de massa acoplado a um sistema de cromatografia líquida de ultra eficiência usando um método de aquisição de dados independente (HDMSE).
Nota: Mais detalhes sobre a configuração do instrumento são incluídos na seção de Material. Este aparelho utiliza um dispositivo de mobilidade de íon wave itinerante para a separação de peptídeos precursores10. - Preparar duas fases móveis contendo 0,1% de FA na água (solvente A) e 0,1% FA em acetonitrila (solvente B).
Nota: Utilize uma coluna de C18 híbrido superfície carregada (CSH) (2.1 x 150 mm, embalado com partículas de 1.7-µm) para a separação de digere o mAb cravado. - Do software de aquisição de dados, clique em "Create/análise método" e escolha "Gerar uma aquisição e aplicação do método". Digite o nome do método, navegue até a pasta do método (diretório) e clique em "Next".
- Para "Tipo de análise," escolher "peptídeo mapa (IMS)" e na aba "Instrumento de sistema", selecione o instrumento "Quaternário solvente Manager". Editar as configurações de gradientes para conseguir a separação de peptídeos com um caudal de 200 µ l/min. Use uma gradiente eluição de 1 a 40% de solvente B em 30min, seguido por uma lavagem da coluna 2-min e um equilibrio 9-min, com uma duração total de 50 min. introduzir estes experimental parâmetros no editor de método de LC.
- Na aba "Instrumento de sistema", selecione o instrumento de "gerente de amostra". Definir a temperatura da amostra para 10 ° C e a temperatura da coluna a 60 ° C.
- Opere o espectrômetro de massa QTOF em íon positivo ESI sensibilidade modo, com um poder de resolução típico de 30.000 FWHM.
Nota: LC/HDMSE é um modo de aquisição de dados independente (diâmetro) que opera através da recolha rapidamente digitalizações com a alternância de energias de colisão (na célula de colisão) entre uma baixa energia (6 V para varreduras de canal 1 de MS) e uma energia elevada (15 - 40 V rampa para produzir os espectros de fragmentação sem seleção de precursor em MS canal 2).- Digite os parâmetros de MS relacionados a fonte padrão do software de aquisição de dados: "Meu trabalho/do instrumento de sistema," Selecione a guia "Vion IMS Qtof" e no menu "Ferramentas", digite esses parâmetros: 3,0 kV de tensão capilar, temperatura de 100 ° C fonte, fonte de 100 V deslocamento, 50 L/h cone de fluxo de gás e tensão de cone 40 V. Regule a temperatura de desolvation a 250 ° C, a taxa de fluxo de gás desolvation a 500 L/h e a tensão de referência capilar para 3,0 kV.
- Do software de aquisição de dados, clique em "Create/análise método" e escolha "Gerar uma aquisição e aplicação do método". Digite o nome do método e navegue até a pasta do método (diretório) e clique em "Next".
- Para "Tipo de análise," escolher "peptídeo mapa (IMS)" e na aba "Instrumento de sistema", selecione o instrumento "Vion IMS QTof". Clique em "Concluir".
Nota: O método recém-criado de LC/HDMSE abre automaticamente na tela de "Configuração de método de instrumento". - Na guia "Configurações", insira esses parâmetros: 3,0 kV tensão capilar, temperatura fonte de 100 ° C, temperatura de 250 ° C desolvation, fluxo de gás de cone 50 L/h e fluxo de gás de desolvation de 500 L/h.
- Nas configurações do guia "Experimento", selecione "MSEde alta definição" e escolha "Runtime de método de análise." Na guia "Configurações de digitalização", insira esses parâmetros: baixa massa, 100; massa alta, 2.000; e tempo de varredura, 400 MS. na aba "CE", digite "6 V" para a configuração de baixa energia e escolher uma rampa alta energia de 15 a 40 V. Certifique-se de ter "Disable redução de dados" verificada.
- Administrar uma solução de 50 encefalina leucina ng/mL (LE) preparado em 50% acetonitrila com 0,1% FA a uma taxa de fluxo de 10 µ l/min. para calibração de bloqueio-massa durante a aquisição de dados.
Nota: Os dados de massa de bloqueio são adquiridos cada 5 min usando a mesma taxa de aquisição sobre o mesmo intervalo de massa.
- Analise a amostra de 10 nM, PHO-cravado usando o ensaio de LC/HDMSE acima descrito por injectar 10 µ l de amostra (montante carregado na coluna é fmol 100 para cada padrão de peptide).
4. configuração do método de otimização de energia (CE) colisão Tof-MRM
- Do conjunto de dados, LC/HDMSE obter o tempo de retenção, o precursor e o fragmento íon m/z para cada peptide PHO. Reter a m/z e o estado de carga para o mais abundante íon precursor e o correspondente mais abundante de três íons do fragmento.
Nota: Um exemplo de baixa energia e alta energia espectros normalmente por LC/HDMSE ensaio é apresentado na Figura 2. - Use a otimização de energia (CE) de colisão em um experimento SRM/MRM para obter o melhor sinal para cada peptídeo23.
- Do software de aquisição de dados, clique em "Create/análise método" e escolha "Gerar uma aquisição e aplicação do método". Digite o nome do método, navegue até a pasta do método (diretório) e clique em "Next".
- Para "Tipo de análise", escolha "Quantify." Escolha "Quantify ensaio cromatográfico de Tof 2D" e clique em "Next". Na aba "Instrumento de sistema", selecione "Vion IMS QTof" e clique em "Concluir".
Nota: O método de Tof-MRM recém-criado abre automaticamente na tela de "Configuração de método de instrumento". - Na guia "Configurações", insira esses parâmetros: 3,0 kV tensão capilar, temperatura fonte de 100 ° C, temperatura de 250 ° C desolvation, fluxo de gás de cone 50 L/h e fluxo de gás de desolvation de 500 L/h.
- Nas configurações do guia "Experimento", selecione "Tabela de função" e escolher "uma ou mais MS, MSMS ou MRM funções." Usando as setas para baixo, configurar uma função de "Tof-MRM" para cada peptide digitando três "transições" (combinações do precursor mais abundante e três dos seus íons mais abundantes do fragmento).
Nota: No modo de Tof-MRM, a mais alta sensibilidade é conseguida usando uma abordagem MRM agendada. - No editor de função "Tof-MRM", escolha uma janela de tempo de retenção para cada peptide (pelo menos de 1 min longa), organizadas de acordo com a ordem de eluição do peptide. No editor de função "Tof-MRM", insira o peptídeo precursor m/z e o produto de m/z; Selecione a janela quad isolamento como "Low" (4-Da janela) para o precursor do peptídeo; Selecione um tempo de verificação fixa de 100 ms; e escolher doze valores de energia de colisão diferente na faixa de 14-36 V, como segue: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34 e 36, conforme mostrado no exemplo da Figura 3.
Nota: Com o método de Tof-MRM acima descrito, pontos de dados de pelo menos 10 por pico cromatográfico são coletados para cada transição para cada CE especificado. Para reduzir o tamanho do arquivo de dados, selecione a opção "Largura" (6-Da janela) para o sinal do íon fragmento. Exemplos de valores de CE-otimizado são mostrados na tabela 2. - Analise a amostra de 10 nM, PHO-cravado usando o ensaio de Tof-MRM acima descrito por injectar 10 µ l de amostra (montante carregado na coluna é fmol 100 para cada padrão de peptide).
5. instalação do método de aquisição final do HS-MRM para quantificação de peptídeo usando separação de mobilidade iônica de precursores do peptide
- No menu "Investigar" do software de aquisição de dados, exibir todos os 11 traços cromatográficos gerados para cada CE para cada peptídeo e integrar os picos usando o botão "Integrar" na barra de ferramentas "processamento opções/operações"; a área do pico mais elevada indicará a melhor CE para cada transição.
- Visualmente comparar as melhores áreas de pico para os 3 fragmentos de cada peptídeo e reter apenas o melhor "transição" (o íon fragmento que produz o sinal mais intenso); A tabela 2 exibe o melhor "transições" obtidos de quatro peptídeos PHO.
- Configure um método de HS-MRM contendo apenas um íon fragmento por peptídeo. Use os valores de CCS de cada precursor do peptídeo do LC/HDMSE dataset.
- Modifique o método de Tof-MRM criado na seção anterior (seção 4), selecionando a função de "HS-MRM" em vez da função de "Tof-MRM".
- No editor de função "HS-MRM", insira os seguintes parâmetros relacionados ao precursor: m/z, quadrupolo resolução: baixa (4 Da), cobrar o estado, valor de CCS precursor e resolução de CCS precursor: baixa. Para o íon de produto, digite seu m/z, a energia de colisão ideal, escolher um tempo de varredura de 0,4 s e selecione uma configuração de espectro "Wide" (6 Da) para incluir todos seus isótopos. Um exemplo do método de aquisição de HS-MRM final para todos os quatro peptídeos é apresentado na Figura 4.
- Analisar todas as amostras usando o método de HS-MRM, começando com o mAb digest em branco (não-cravado amostra), seguidas por 4 injeções replicar (10 µ l de cada) das seguintes concentrações: 0.1, 1, 10 e 100 peptídeos PHO nM.
6. criação de um método de processamento para a análise do conjunto de dados HS-MRM
- Crie um método de processamento para a análise do conjunto de dados HS-MRM.
- No método de análise criado para o "HS-MRM" método de aquisição de dados, clique na guia "Propósito" e escolha "Gerenciar componentes".
- No menu suspenso em "Tipo de experimento", selecione a opção "HS MS/MS/HS-MRM".
- Clique em "Create/New Entry" e introduzir os parâmetros a seguir no editor de método de processamento: tempo de retenção do peptídeo (RT), estado de carga do precursor, precursor m/z e tempo de deriva do precursor (DT). Para o íon fragmento, insira seu estado m/z e carga e especifique o rastreamento "XIC" como o modo de extração.
- Na guia "Propósito", o mesmo método de aquisição "HS-MRM", clique em "Valores padrão" e digite as seguintes concentrações: 0.1, 1, 10 e 100 peptídeos PHO nM.
- Insira os seguintes parâmetros nas "Configurações de processamento e extração": tolerância de massa padrão, 10 ppm (para o m/z de todos os íons de fragmento) e padrão deriva de tempo, 5%; um exemplo de método de processamento de peptídeo PHO é mostrado na Figura 5.
- Para o tipo de calibração, escolha o encaixe de curva linear com 1 / X2 ponderação.
- Processe os dados de HS-MRM para integrar todos os cromatogramas e para construir as curvas de calibração de cada peptídeo PHO.
Resultados
As sequências individuais de seis fosforilase b peptídeo as normas contidas na mistura do peptide PHO são mostradas na tabela 1, juntamente com seus tempos de retenção e seus precursores mais abundantes observadas no HDMSE experimentar. O primeiro passo no desenvolvimento de um ensaio MRM (HS) alta seletividade é a aquisição de um conjunto de dados HDMSE estabelecer os tempos de eluição de cada peptídeo PHO, juntamente com sua correspondente mais abundante precursor e os três fragmentos mais abundantes. Figura 2 exibe os espectros HDMSE adquiridos por um dos peptides PHO (Pep 6) cravado na digest infliximab. Depois de estabelecer o melhor de 3 "transições" (combinações de massas precursor e fragmento) para cada peptídeo, um experimento de Tof-MRM é executado para encontrar a energia de colisão ideal para maximizar os sinais gerados para cada peptídeo. Os resultados do experimento otimização CE estão resumidos na tabela 2. O ensaio final do HS-MRM (ver Figura 4) retém apenas o melhor "transição" para cada peptídeo e é usado para analisar todas as amostras cravadas. Exemplos de cromatogramas HS-MRM gerados para 4 peptídeos PHO em todas as concentrações investigadas são apresentados na Figura 7. Quatro curvas de calibração obtidas para cada seguinte peptídeo a integração dos picos do HS-MRM destacada na Figura 7 são exibidas na Figura 8. Além disso, o pico área desvio-padrão relativo, calculado com base em 4 replicar as injeções, é resumido em 4 tabelas apresentadas na tabela 3.
| Peptídeo | Peptídeo | Retenção de | Estados de carga | ||||
| ID | Sequência de | tempo (min) | + 1 | + 2 | + 3 | + 4 | |
| Pep 1 | VLYPNDNFFEGK | 19,4 | 1442.6951 | 721.8512 | 481.5699 | 361.4292 | |
| Pep 2 | TCAYTNHTVLPEALER | 16,0 | 1874.9065 | 937.9569 | 625.6404 | 469.4821 | |
| Pep 3 | IGEEYISDLDQLRK | 18.9 | 1678.8646 | 839.9360 | 560.2931 | 420.4716 | |
| Pep 4 | LLSYVDDEAFIR | 21.1 | 1440.7369 | 720.8721 | 480.9172 | 360.9397 | |
| Pep 5 | LITAIGDVVNHDPVVGDR | 19,7 | 1890.0080 | 945.5076 | 630.6742 | 473.2574 | |
| Pep 6 | VFADYEEYVK | 17,7 | 1262.5939 | 631.8006 | 421.5362 | 316.4039 | |
Tabela 1. Padrões de peptídeo PHO contidos na mistura massa PREP cravada na digest infliximab. Tempos de retenção de peptídeo e seus precursores mais abundantes (destacadas em negrito) são exibidos em formato tabular.
| Peptídeo | Peptídeo | Retenção de | Precursor do peptídeo | Mais abundante fragmento íons/carga | Optimum | ||||
| ID | Sequência de | tempo (min) | m/z & carga | Tempo de deriva (ms) | Eu | II | III | CE (V) | |
| Pep 2 | TCAYTNHTVLPEALER | 16,0 | 625.6404 + (3) | 6.2 | 714.3781 (+ 1) | 807.4177 + (2) | 827.4621 (+ 1) | 24 | |
| Pep 4 | LLSYVDDEAFIR | 21.1 | 720.8721 + (2) | 7.5 | 865.4050 (+ 1) | 964.4734 (+ 1) | 1214.5688 (+ 1) | 22 | |
| Pep 5 | LITAIGDVVNHDPVVGDR | 19,7 | 630.6742 + (3) | 6.3 | 642.3570 (+ 1) | 689.8391 + (2) | 832.4236 + (2) | 20 | |
| Pep 6 | VFADYEEYVK | 17,7 | 631.8006 + (2) | 7.0 | 830.3931 (+ 1) | 945.4200 + (2) | 1016.4571 (+ 1) | 24 | |
Tabela 2. Os resultados do experimento otimização Tof-MRM: os três mais abundantes fragmentos de cada peptídeo PHO quantificados neste estudo são indicados, juntamente com a energia de colisão otimizado correspondente.
| Conc | Quantidade | Pep 2 áreas de pico (tabela 3A) | |||||
| (nM) | na coluna (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Quer dizer | RSD (%) |
| 0.1 | 1 | 490 | 439 | 462 | 431 | 456 | 5.8 |
| 1 | 10 | 5121 | 4842 | 5198 | 4842 | 5001 | 3.7 |
| 10 | 100 | 63853 | 64279 | 66111 | 62509 | 64188 | 2.3 |
| 100 | 1000 | 612392 | 605553 | 613229 | 611004 | 610545 | 0.6 |
| Conc | Quantidade | Pep 4 áreas de pico (tabela 3B) | |||||
| (nM) | na coluna (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Quer dizer | RSD (%) |
| 0.1 | 1 | 275 | 359 | 325 | 288 | 312 | 12.2 |
| 1 | 10 | 3559 | 3694 | 3287 | 3754 | 3574 | 5.8 |
| 10 | 100 | 45259 | 45775 | 42976 | 45548 | 44890 | 2.9 |
| 100 | 1000 | 459374 | 467927 | 436272 | 458994 | 455642 | 3.0 |
| Conc | Quantidade | Pep 5 áreas de pico (tabela 3) | |||||
| (nM) | na coluna (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Quer dizer | RSD (%) |
| 0.1 | 1 | 3194 | 3243 | 3202 | 3257 | 3224 | 1.0 |
| 1 | 10 | 31464 | 31150 | 31464 | 31433 | 31378 | 0,5 |
| 10 | 100 | 313638 | 320712 | 311943 | 311943 | 314559 | 1.3 |
| 100 | 1000 | 2845736 | 2840031 | 2882006 | 2864052 | 2857956 | 0.7 |
| Conc | Quantidade | Pep 6 áreas de pico (tabela 3D) | |||||
| (nM) | na coluna (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Quer dizer | RSD (%) |
| 0.1 | 1 | 490 | 583 | 440 | 440 | 488 | 13,8 |
| 1 | 10 | 6429 | 6429 | 6848 | 6623 | 6582 | 3.0 |
| 10 | 100 | 71295 | 70400 | 71563 | 70400 | 70915 | 0.9 |
| 100 | 1000 | 707640 | 707640 | 694461 | 729490 | 709808 | 2.0 |
Tabela 3. Tabela que contém as áreas de pico dos cromatogramas HS-MRM gravou 4 peptídeos PHO (Pep 2, 4, 5 e 6) para cada injecção de LC/MS (16 corridas de LC/MS e as 4 concentrações testadas).
O desvio-padrão relativo foi melhor do que 15% de todos os peptídeos na faixa de concentração todo investigado.

Figura 1. Diagrama de fluxo de trabalho, resumindo os três passos necessários para configurar um método de aquisição de HS-MRM. Clique aqui para ver uma versão maior desta figura.
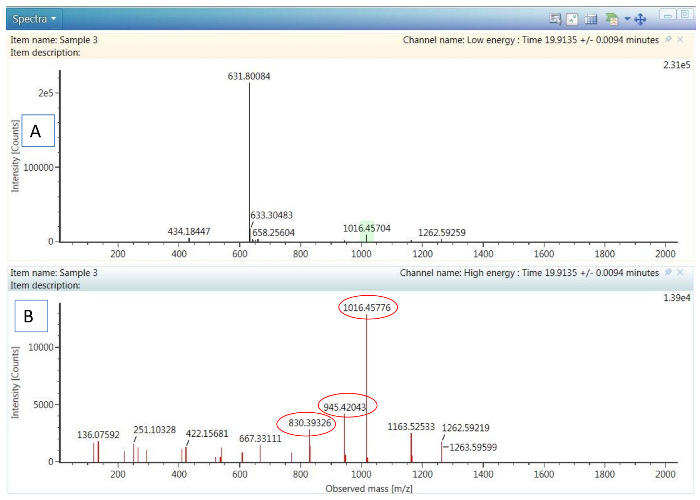
Figura 2. Exemplo de dados HDMSE:
(A) espectro de baixa energia mostrando o íon precursor do Pep 6. (B) espectro de fragmentação de alta energia do peptide mesmo, exibindo os top 3 mais abundante fragmento íons (circulados) selecionados para otimização de energia de colisão de Tof-MRM. Clique aqui para ver uma versão maior desta figura.

Figura 3. Parâmetros utilizados para a criação de uma otimização de Tof-MRM experimentam.
Para cada transição, testaram-se onze energias de colisão (na faixa de 16 a 36 V). Clique aqui para ver uma versão maior desta figura.
Figura 4. Exemplo do método HS-MRM final.
Vários parâmetros são necessários para cada "transição," incluindo o peptídeo precursor m/z, seu estado de carga e tempo de deriva de mobilidade do íon, a m/z do íon fragmento mais abundante, a energia de colisão ideal e o tempo de aquisição de MS. Clique aqui para ver uma versão maior desta figura.
Figura 5. Configurações usadas pelo método de processamento para analisar o dataset HS-MRM.
Cada peptídeo é monitorado por um único "transição", descrito pelo peptídeo precursor m/z, cobrar o estado e esperado o tempo de retenção, juntamente com a m/z de seu estado de carga e o fragmento mais abundante. Clique aqui para ver uma versão maior desta figura.

Figura 6. Diagrama do espectrômetro de massa de mobilidade de íons.
No modo de aquisição de HS-MRM, os precursores do peptide é ser quantificadas são separados de outros precursores de peptídeo co eluição (interferência) na célula de mobilidade de íon, isolado pelo quadrupolo e fragmentado, com uma energia de colisão fixo no célula de colisão. O sinal produzido por íons de fragmento de peptídeo é aprimorado ajustando a frequência de empurrador e quantificação de peptídeo é executada usando a MS de alta-resolução (> 30.000) sinais produzidos pelo íon fragmento mais intenso de cada peptídeo. Clique aqui para ver uma versão maior desta figura.

Figura 7. Cromatogramas HS-MRM gravado para 4 peptídeos PHO em 4 diferentes concentrações abrangendo 3 ordens de magnitude (0.1, 1, 10 e 100 nM).
(A) pep 2 cromatogramas, 4 (B) Pep cromatogramas, cromatogramas (C) Pep 5 e 6 (D) Pep cromatogramas. Clique aqui para ver uma versão maior desta figura.
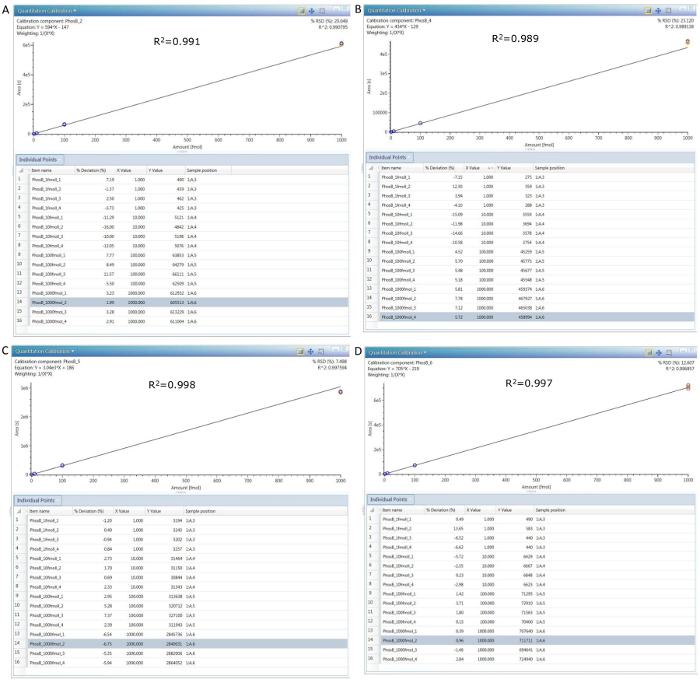
Figura 8. As curvas de calibração para 4 peptídeos PHO em 4 diferentes concentrações (0,1, 1, 10 e 100 nM).
As tabelas abaixo cada curva exibem as áreas de pico individual (valores de Y) registadas para cada injecção, enquanto a segunda coluna de cada tabela mostra o desvio percentual da esperada resposta linear. (A) pep 2 calibração, calibração de Pep (B) 4, calibração (C) Pep 5 e 6 (D) Pep calibração. Clique aqui para ver uma versão maior desta figura.
Discussão
De alta resolução (Rs > 20.000) espectrometria de massa é usada rotineiramente para caracterização estrutural de proteínas terapêuticas em uma variedade de plataformas de instrumento. Em contraste, quantificação de proteína baseada em MS é normalmente realizada por SRM/MRM em baixa resolução (Rs ~ 1.000) em tandem quadrupolo espectrómetros de massa usando assinatura peptides gerados pela clivagem enzimática de proteínas11,12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23. como separações cromatográficas de dimensão única não podem resolver totalmente as misturas do complexo peptídeo produzidas por digestão enzimática, eluição co peptídeo é uma ocorrência comum, mesmo no caso de um único-proteína digest. Para digere proteína muito complexa (por exemplo, para a quantificação de 1-100 ppm de HCPs na presença de um fundo rico em peptídeos produzidos pela proteína terapêutica), a sensibilidade, exatidão ou linearidade da SRM/MRM ensaio pode ser afetado por interferência.
Os ensaios SRM/MRM têm seletividade unidimensional, contando apenas com uma combinação de "exclusiva" de massas precursor/fragmento. Por esta razão, estes ensaios falharem em situações quando o fundo de peptídeo muda inesperadamente (por exemplo, para amostras de Biofarmacêutica obtidos de processos de purificação diferentes). Para superar essas limitações, propomos aqui um ensaio de alta seletividade (HS) MRM implementado em um íon habilitados para mobilidade de alta resolução quadrupolo tempo-de-voo (QTOF) híbrido espectrômetro de massa (para o diagrama do instrumento, veja a Figura 6).
O instrumento separa os precursores do peptide do interesse de outros precursores de peptídeo co eluição (interferência) na célula de mobilidade do íon, isola o envelope cheio isotópico do precursor no quadrupolo e fragmentos-lo com um CE fixo na célula de colisão. O sinal produzido por seu fragmento de peptídeo mais abundante é reforçado, ajustando a frequência de empurrador (realce de destino), que seletivamente empurra massa regiões de interesse para o tubo de voo, em vez de todos os íons, como com uma varredura completa. Quantificação de peptídeo é executada usando os sinais de alta-MS-resolução (Rs ~ 30.000) produzidos por este íon fragmento. Em comparação com os ensaios SRM/MRM, o ensaio de HS-MRM oferece dois níveis adicionais de seletividade: é fornecida pela separação de mobilidade do íon precursor-nível, enquanto o segundo é oferecido pela resolução massa aumentada do analisador TOF. Os resultados trazidos por estas melhorias de seletividade são visíveis os cromatogramas de HS-MRM exibidos na Figura 7, que são livres de interferências através de três ordens de magnitude.
Ao contrário dos ensaios SRM/MRM, existem vários parâmetros que podem ser ajustados para otimizar os ensaios de HS-MRM: a janela de RT em torno do precursor do peptídeo (normalmente definida em 0,2 min), a janela de isolamento de quadrupolo (4 Da), a janela de tempo de deriva em torno a precursor (± FWHM do pico do precursor do mobilogram de íon correspondente) e a resolução MS do íon fragmento (20.000-40.000). Os ensaios de HS-MRM são muito sensíveis: o montante mais baixo detectado para cada peptídeos PHO é 1 femtomole na coluna (ou 0.1 nM em termos de concentração de peptídeo). Quando se considera o peptídeo MW (ver tabela 1 para MWs precisos), a quantidade detectada na coluna é da ordem de 1-2-pg, enquanto que a coluna é carregada com uma quantidade significativamente maior (2 µ g) de peptídeos de fundo do mAb digest.
Tendo em conta o peso molecular da proteína PHO completo (97,2 kDa) de que derivam os peptídeos cravados, o ensaio é capaz de detectar 50 ppm de uma impureza de proteína na presença de íons de alta-abundância de fundo. Mais baixos limites de detecção (5-10 ppm) são realizáveis para menor peso molecular HCPs (10-20 kDa). O ensaio abrange três ordens de magnitude (conforme as curvas de calibração da Figura 8), o que significa que pode medir HCPs na faixa de 1-1.000 ppm. Também, a reprodutibilidade dos ensaios HS-MRM, ilustrado na tabela 3, combina muito bem com a reprodutibilidade dos ensaios SRM/MRM pequeno-molécula, com a área do pico RSD melhor do que 15%.
Nós explorou os recursos de um ensaio de romance para a quantificação dos padrões de peptídeo cravado em digerir um anticorpo monoclonal (mAb) e demonstrou a sua sensibilidade e utilidade para cobrir a ampla faixa dinâmica (pelo menos três ordens de magnitude) normalmente encontradas na análise do HCP. Todos os seis padrões de peptídeo foram detectados em concentrações tão baixas quanto 0.1 nM (1 femtomole carregado em uma coluna cromatográfica de 2,1 mm ID) na presença de um fundo de alta-abundância de peptídeo (2 µ g de uma digest carregado de mAb na coluna). Incorporando ambos HRMS alvo e separação de precursor de mobilidade do íon, o ensaio de HS-MRM tem um grande potencial para tornar-se um ensaio de controlo rápido, de alta produtividade para múltiplos HCPs através de vários lotes de produtos biofarmacêuticos.
Divulgações
Todos os autores são empregados da Waters Corporation, que é o produtor de vários reagentes e instrumentos utilizados neste artigo.
Agradecimentos
Os autores gostaria agradecer Lesley Malouin e Tony Catlin de águas Corporation para a preparação de figura 6 do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| Vion IMS Qtof mass spectrometer | Waters | 186009214 | |
| Acquity H-Class Quaternary solvent manager (QSM) | Waters | 186015041 | |
| Acquity H-Class FTN Sample Manager (SM) | Waters | 186015040 | |
| Acquity H-Class Column Manager (CM) | Waters | 186015043 | |
| Acetonitrile (ACN) | Fisher Chemical | A996-4 | |
| Ammonium bicarbonate | Sigma Aldrich | 40867-50G-F | |
| 2.1 x 150 mm CSH C18 UPLC column, 1.8 µm particles | Waters | 186005298 | |
| Dithiothreitol (DTT) | Sigma Aldrich | D5545-5G | |
| Formic acid, eluent additive for LC/MS | Sigma Aldrich | 56302-10X1ML-F | |
| Hi3 rabbit phosphorylase (PHO) MassPREP standard | Waters | 186006011 | |
| Iodoacetamide (IAM) | Sigma Aldrich | I-1149-5G | |
| LC vials (12x32 mm glass vials, screw neck) | Waters | 186000327c | |
| Leucine Enkephalin acetate salt hydrate | Sigma Aldrich | L9133-10MG | |
| Protein LoBind 2.0 mL tubes (2x50) | Eppendorf | 22431102 | |
| RapiGest SF | Waters | 186001861 | |
| Trypsin/Lys-C enzyme mix, mass spec grade (5 x 20 µg) | Promega | V5073 | |
| Water, LC/MS grade | Sigma Aldrich | 39253-1L-R |
Referências
- Doneanu, C. E., et al. Analysis of host-cell proteins in biotherapeutic proteins by comprehensive online two-dimensional liquid chromatography/mass spectrometry. MAbs. 4, 24-44 (2012).
- Schenauer, M. R., Flynn, G. C., Goetze, A. M. Identification of host-cell protein impurities in biotherapeutics using mass spectrometry. Anal Biochem. 428, 150-157 (2012).
- Zhang, Q., et al. Comprehensive tracking of host-cell proteins during monoclonal antibody purifications using mass spectrometry. MAbs. 6, 659-670 (2014).
- Farrell, A., et al. Quantitative host-cell protein analysis using two-dimensional data independent LC-MS(E). Anal Chem. 87, 9186-9193 (2015).
- Doneanu, C. E., et al. Enhanced detection of low-abundance host cell protein impurities in high-purity monoclonal antibodies down to 1 ppm using ion mobility mass spectrometry coupled with multidimensional liquid chromatography. Anal Chem. 87, 10283-10291 (2015).
- Doneanu, C. E., et al. Improved identification and quantification of host cell proteins (HCPs) in biotherapeutics using liquid chromatography mass spectrometry. Technologies for therapeutic monoclonal antibody characterization, vol. 3: Defining the next generation of analytical and biophysical techniques. , Washington DC. ACS Symposium series 1202 357-393 (2015).
- Zhang, Q., et al. Characterization of the co-elution of host-cell proteins with monoclonal antibodies during protein A purification. Biotechnol Prog. 32, 708-717 (2016).
- Reisinger, V., Toll, H., Mayer, R. E., Visser, J., Wolschin, F. A mass spectrometry based approach to host cell protein identification and its application in a comparatibility exercise. Anal Biochem. , 1-6 (2014).
- Madsen, J. A., et al. Toward the complete characterization of host-cell proteins in biotherapeutics via affinity depletions, LC/MS/MS and multivariate analysis. MAbs. 7, 1128-1137 (2015).
- Giles, K., et al. Applications of a trevelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18, 2401-2414 (2004).
- Anderson, V., Hunter, C. L. Quantitative mass spectrometric multiple reaction monitoring assays for major plasma proteins. Mol Cell Proteomics. 5, 573-588 (2006).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Mol Syst Biol. 4, 1-14 (2008).
- Picotti, P., et al. High-throughput generation of selected reaction-monitoring assays for proteins and proteomes. Nat Methods. 7, 43-46 (2010).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. (53), (2011).
- Liebler, D. C., Zimmerman, L. J. Targeted quantitation of proteins by mass spectrometry. Biochemistry. 52, 3797-3806 (2013).
- Manes, N. P., Mann, J. M., Nita-Lazar, A. Selected reaction monitoring mass spectrometry for absolute protein quantification. J Vis Exp. (102), (2015).
- Ebhardt, H. A., Sabido, E., Huttenhain, R., Collins, B., Aebersold, R. Range of protein detection by selected/multiple reaction monitoring mass spectrometry in an unfractioned human cell culture lysate. Proteomics. 12, 1185-1193 (2012).
- Picotti, P., Bodenmiller, B., Mueller, L. N., Domon, B., Aebersold, R. Full dynamic range proteome analysis of S. cerevisiae by targetted proteomics. Cell. 138, 795-806 (2009).
- Sherman, J., McKay, M. J., Ashman, K., Molloy, M. P. How specific is my SRM: the issue of precursor and product ion reductancy. Proteomics. 9, 1120-1123 (2009).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of innacurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Rost, H., Malmstrom, L., Aebersold, R. A computational tool to detect and avoid redundancy in selected reaction monitoring. Mol Cell Proteomics. 11, 540-549 (2012).
- Bao, Y., et al. Detection and correction of interference in SRM analysis. Methods. 61, 299-303 (2013).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Mbasu, R. J., et al. Advances in quadrupole and time-of-flight mass spectrometry for peptide MRM based translational reserch analysis. Proteomics. 16, 2206-2220 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados