Method Article
Ein HS-MRM-Assay für die Quantifizierung der Wirtszelle Proteine in Protein Biopharmazeutika durch flüssige Chromatographie Ion Mobility QTOF Massenspektrometrie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir einen chromatographischen Assay, gekoppelt mit der Ionen-Mobilität Trennung von Peptid-Vorstufen, gefolgt von den hochauflösenden (~ 30.000) MS-Detektion von Peptid-Fragmente für die Quantifizierung der Spike Peptid Standards in ein monoklonaler Antikörper Digest.
Zusammenfassung
Die Analyse der Low-Level (1-100 ppm) Protein Verunreinigungen (z. B. Wirtszelle Proteine (HCPs)) in Protein Biotherapeutics ist ein herausfordernde Assay erfordern hohe Empfindlichkeit und einen großen Dynamikbereich. Massenspektrometrie-basierte Quantifizierung Assays für die Proteine beinhalten in der Regel Proteinverdauung, gefolgt von der selektiven Reaktion Überwachung/Multiple Reaktion Überwachung (SRM/MRM) Quantifizierung von Peptiden mit niedriger Auflösung (~ 1.000 Rs) tandem Quadrupol-Massenspektrometer. Eine Einschränkung dieses Ansatzes ist die Einmischung Phänomen beobachtet, als das Peptid von Interesse anderen Co medikamentenfreisetzende Peptiden in der Probe (innerhalb von 1-Da) die "gleiche" Vorläufer und Fragment Masse (in Bezug auf m/Z-Werte) vorliegt. Um dieses Phänomen zu vermeiden, schlagen wir eine alternative massenspektrometrische Methode, eine hohe Selektivität (HS)-MRM-Assay, die die Ionen-Mobilität Trennung von Peptid-Vorstufen mit der hochauflösenden (Rs ~ 30.000) kombiniert MS Detektion von Peptid-Fragmente. Wir erkundeten die Fähigkeiten dieses Ansatzes zu quantifizieren Low-Fülle Peptid Standards versetzt in ein monoklonaler Antikörper (mAb) verdauen und bewiesen, dass es die Empfindlichkeit und Dynamikbereich (mindestens 3 Größenordnungen) in der Regel bei HCP erreicht hat Analyse. Alle sechs Peptid-Standards wurden festgestellt bei Konzentrationen von 0,1 nM (1 Femtomole geladen auf einer 2,1-mm ID chromatographischen Säule) in Anwesenheit hoher Fülle Peptid Hintergrund (2 µg einer mAb-Digest geladen auf Spalte). Wenn man MW von Kaninchen-Phosphorylase (97,2 kDa), von dem die Spikes Peptide abgeleitet wurden, ist die LOQ des Assays niedriger als 50 ppm. Relative Standardabweichung (RSD) von Peakflächen (n = 4 Wiederholungen) waren weniger als 15 % über den gesamten Konzentrationsbereich untersucht (0,1-100 nM oder 1-1.000 ppm) in dieser Studie.
Einleitung
Quantifizierung von großen Biomolekülen (Proteine) im industriellen Umfeld basiert derzeit auf Immunoassays (z. B. ELISA), vor allem durch mehrere Vorteile: Empfindlichkeit, Hochdurchsatz-Ease-of-Use und niedrigen Kosten pro Probe. Bei Anwendung auf die Low-Fülle Protein Verunreinigungen (1-100 ppm der Wirtszelle Proteine (HCPs)) im Protein Therapeutics analysieren, bieten diese biologische Assays in der Regel die Gesamtkonzentration HCP (normalerweise ausgedrückt in ppm oder ng HCP/mg mAb), aber Sie nicht identifizieren und Messen der einzelnen HCP-Verunreinigungen. Mehrere MS-basierte Tests haben vor kurzem entwickelt worden, um ELISAs zu ergänzen oder Informationen bereitstellen, die ELISAs scheitern,1,2,3,4,5,6 anzubieten , 7 , 8 , 9. wegen Probe Komplexität und die Anforderung zur Erkennung von HCP Peptide eines breiten dynamischen Bereichs in Konzentration (mindestens 3 Größenordnungen), multidimensionale Chromatographische Methoden umfangreiche Stichprobe Fraktionierung Ausschreibungen haben Traditionell wurde eingesetzt zur Identifizierung von niedrig-Fülle HCPs1,2,3,4,5,6,7.
Ein natürlicher Schritt nach HCP Identifizierung und Validierung ist HCP-Überwachung (monitoring) über mehrere Batches von Biopharmazeutika. In diesem Fall sind Single-Dimension-LC/MS-Methoden zur Probe Durchsatz8,9Verbesserung vorgeschlagen worden. Jedoch könnte die Genauigkeit und dynamischen Bereich der HCP-Messungen in einem 1D-LC/MS-Assay die überwältigende Präsenz von biopharmazeutischen Peptiden betroffen sein. Im Vergleich zu einer mehrdimensionalen Trennung, das Potenzial für Signal Störungen19,20,21,22 wird erhöht, in einer einzigen Dimension chromatographische Trennung da die Wahrscheinlichkeit für Weitere Peptid-Vorläufer Co medikamentenfreisetzende sein wird erhöht. Die Einbeziehung der orthogonalen Mittel zur Trennung von Peptid-Vorstufen ohne Verlängerung der chromatographischen Trennung wäre eindeutig vorteilhaft. Welle Ion Mobilität (TWIM)10 Reisen hat die Fähigkeit, überlastete MS Spektren in Millisekunden zu lösen. Rund 500 Mobilität Trennungen durchgeführt werden können, während der Elution von einem einzigen Peptid, unter der Annahme einer vollständigen chromatographischen Peak Breite von 10 s und bedenkt, dass die Laufzeit einer IM Trennung auf dem Ion Mobilität Instrument 20 ms ist.
Massenspektrometrische Assays für Protein Quantifizierung wurden erfolgreich entwickelt in den letzten Jahrzehnt mit der anerkannten ausgewählt (mehrfach-) Reaktion Überwachung Ansatz (SRM/MRM-Methode) auf Tandem Massenspektrometer11, implementiert 12,13,14,15,16,17,18,19,20,21 ,22,23. Eine Einschränkung dieser niedrig aufgelöste massenspektrometrische Assay ist die Einmischung Phänomen19,20,21,22 beobachtet, wenn das Peptid von Interesse hat die "gleiche" Vorläufer und Fragment präsentieren Masse andere Co medikamentenfreisetzende Peptide in der Probe (innerhalb von 1-Da). Es gibt zwei Möglichkeiten zur Verbesserung der Genauigkeit der SRM/MRM Methoden: eine Option beinhaltet einen zusätzliche Trennung Schritt Ebene der Vorläufer zu störenden Vorläufer Ionen zu entfernen, während die andere Möglichkeit besteht darin die MS Auflösung der Vorläufer/Fragment-Erkennung zu erhöhen vermeiden Sie überlappende MS-Signale. Die hohe Selektivität (HS) MRM Akquisitionsmodus beschrieben hier nutzt beide Ansätze durch die Kopplung der Ionen-Mobilität Trennung von Peptid-Vorstufen mit der hochauflösenden (Rs ~ 30.000) MS-Detektion von Peptid-Fragmente. Der Test beschriebenen hier umfasst mindestens drei Größenordnungen, die den dynamischen Bereich in der Regel in SRM/MRM Proteomics Experimente17,18,24beobachtet.
Das Dienstprogramm des HS-MRM-Assays für HCP Quantifizierung zeigte sich durch die Überwachung der Linearität des Signals produziert durch sechs Peptid-Standards bei verschiedenen Konzentrationen (0,1 - 100 nM-Bereich) in ein monoklonaler Antikörper Digest gespickt.
Protokoll
1. Vorbereitung des Infliximab Digest (~ 24 h-Verfahren)
- Bereiten Sie frische Lösungen von 50 mM Ammonium Bicarbonat, 1 % anionischen Tenside in 50 mM NH4HCO3500 mM Dithiothreitol (DTT) in 50 mM NH4HCO3und 500 mM Iodoacetamide (IAM) in 50 mM NH4HCO3.
- Fügen Sie um 750 µL 50 mM NH4HCO3200 µL Infliximab MAB (10 mg/mL) und 50 µL 1 % anionische Tensid-Lösung hinzu und Denaturieren Sie das Protein für 15 min bei 60 ° C in Gegenwart von 0,05 % anionische Tenside.
- Dazugeben Sie 40 µL von 500 mM DTT und reduzieren Sie die Probe für 60 min bei 60 ° C in Anwesenheit von 20 mM DTT.
- Fügen Sie 20 µL von 500 mM IAM und Alkylat Probe für 30 min bei Raumtemperatur im Dunkeln im Beisein von ~ 10 mM IAM.
- Fügen Sie den Inhalt der Microcentrifuge Schlauch zu einem Glasfläschchen mit 20 µg von MS Sequenzierung-Grade Lys C/Trypsin und verdauen der Probe 3 h bei 37 ° C.
- Fügen Sie ein zweites Enzym aliquoten (20 µg) durch die Übertragung der verdauten Probe auf ein weiteres Glasfläschchen mit 20 µg MS Sequenzierung-Grade Lys C/Trypsin und verdauen der Probe über Nacht (12-15 h) bei 37 ° C.
- Fügen Sie nach der Übernachtung Proteolyse 5 µL 100 % Ameisensäure (FA hinzu) und inkubieren Sie den Digest für 30 min bei 37 ° C, die saure labile anionische Tensid zu zerlegen.
- Spin-down der Digest für 15 min bei 4.000 x g in einer Zentrifuge, die unlösliche Bestandteil der anionischen Tenside auszufällen.
- Erholen Sie ~ 1.000 µL der Infliximab-Digest zu, und legen Sie es in einem LC-Auto-Sampler-Fläschchen; die Digest-Konzentration sollte ~ 2 mg/mL. Speichern des Digests im Gefrierschrank bei-20 ° C bis die LC/MS-System zur Analyse bereit ist.
(2) Spick Peptid Standards (~ 30 min)
- Tauen Sie den Infliximab-Digest indem man die gefrorenen Probe in der Glasflasche auf einer Bank bei Raumtemperatur auf.
- Fügen Sie 1 mL 0,1 % FA in H2O zu einem Kaninchen Phosphorylase b (PHO) verdauen Fläschchen vorzubereiten 1 µM auf lagerlösungen für alle sechs Peptid-Standards von der PHO in der Durchstechflasche enthaltenen (die Durchstechflasche enthält 1 Nmol jedes Peptid).
- Bereiten Sie 4 x 1-mL-Verdünnungen der PHO Vorratslösung zu Lösungen mit 0,1, 1, 10 und 100 nM Peptide, mit 0,1 % FA als Verdünnung Lösungsmittel. Bereiten Sie alle Verdünnungen in Glasfläschchen (LC-Auto-Sampler-Fläschchen) und alle Fläschchen fügen Sie 100 µL von Infliximab Digest als Hintergrund-Matrix hinzu. Last 2 µg Digest Hintergrund auf Spalte bei jeder 10-µL-Injektion. Bereiten Sie eine leere Probe, die den verdünnten Infliximab Digest enthält (die gleichen 01:10 Verdünnung), mit keine Spikes Peptiden.
3. Aufbau der Erwerbsmethode LC/HDMS E Daten
Hinweis: Der Workflow fasst die erforderlichen Schritte zum Einrichten einer HS-MRM-Akquisition ist in Abbildung 1 dargestellt und ausführlich in Kapitel 3-6. Der Erwerb eines Daten-unabhängige HDMSE Datasets ist erforderlich, um die Retentionszeit von jeder überwachten Peptid, das übergeordnete m/Z die am häufigsten vorkommende Peptid-ions nach Elektrospray-Ionisation, zu etablieren und die entsprechende CCS (Elektronenstruktur Kreuz Abschnitt) abgeleitet aus der Ion-Mobilität-Trennung. Darüber hinaus bietet das Daten-unabhängig-Dataset Informationen über die m/Z die drei am häufigsten vorkommende Fragment-Ionen für jedes Peptid-Vorläufer. Im zweiten Schritt des Workflows (CE-Optimierung) steigt die Empfindlichkeit des Tests durch tuning die Kollision-Zell-Energie um die höchste Intensität der Ionen-für jedes Fragment Ion zu erhalten. Im letzten Schritt werden schließlich alle Parameter, die oben beschriebenen HS-MRM Methodeneditor für jedes Peptid überwacht vorgestellt.
- Durchführen Sie LC/MS Experimente auf einem Quadrupol Time-of-Flight (QTOF) Massenspektrometer gekoppelt an ein Ultra-Performance liquid Chromatography-System mit einer Daten-unabhängige Erwerbsmethode (HDMSE).
Hinweis: Weitere Informationen über die Gerätekonfiguration sind im Bereich Material enthalten. Dieses Instrument verwendet ein Reisender Welle Ion Mobilität Gerät für die Trennung von Peptid Vorstufen10. - Bereiten Sie zwei mobile Phasen mit 0,1 % FA im Wasser (Lösungsmittel A) und 0,1 % FA in Acetonitril (Lösungsmittel B).
Hinweis: Verwenden einer geladenen Oberfläche Hybrid (CSH) C18 Spalte (2,1 x 150 mm, verpackt mit 1,7 µm Partikeln) für die Trennung von den Spike mAb-Übersichten. - Klicken Sie in der Datenerfassungs-Software auf "Create/Analyse-Methode" und wählen Sie "Erzeugen eine Akquisition und Verarbeitungsmethode." " Geben Sie den Namen der Methode, navigieren Sie zu der Methode Ordner (Verzeichnis), und klicken Sie auf "Weiter".
- Wählen Sie für"Analyse" "Peptid Karte (IMS)", und wählen Sie im Register "Instrumenten-System" das "Quartäre Solvent Manager" Instrument. Bearbeiten Sie die Steigungen Einstellungen zu Peptid Trennung mit einer Durchflussrate von 200 µL/min Nutzung eines Gradienten Elution von 1 auf 40 % Lösungsmittel B in 30min eine 2-min.-Spalte zu waschen und ein 9-min Gleichgewichtherstellung folgt zu erreichen, mit einer Gesamtlaufzeit von mind. 50 führen diese experimentelle Parameter im Methodeneditor LC.
- Wählen Sie im Reiter "Instrument System" die "Sample Manager" Instrument. Stellen Sie die Probentemperatur um 10 ° C und Säulentemperatur bis 60 ° C.
- Das QTOF-Massenspektrometer in positive Ionen ESI Empfindlichkeit-Modus, mit einem typischen Auflösungsvermögen von 30.000 FWHM zu betreiben.
Hinweis: LC/HDMSE ist ein Daten-unabhängige Übernahme (DIA)-Modus, der durch das Sammeln von rasch Scans mit abwechselnden Kollision Energien (in der Kollision Zelle) betreibt zwischen einem Niedrigenergie (6 V für MS Kanal 1 Scans) und eine erhöhte Energie (15 - 40 V Rampe produzieren Sie die Fragmentierung Spektren ohne Vorläufer Auswahl in MS Kanal 2).- Geben Sie die Standardparameter Quelle im Zusammenhang mit MS in der Datenerfassungs-Software: auf "Meine Arbeit/Instrument System," wählen Sie die Registerkarte "Vion IMS Qtof" im Menü "Extras" aufrufen und diese Parameter: 3,0 kV Kapillare Spannung, 100 ° C Quelltemperatur, 100 V-Quelle ausgeglichen, Kegel 50 L/h Gasstrom und 40 V-Kegel-Spannung. Stellen Sie die zugeführten Temperatur auf 250 ° C, die zugeführten Gas-Durchfluss bis 500 L/h und die Kapillare Referenzspannung bis 3,0 kV.
- Klicken Sie in der Datenerfassungs-Software auf "Create/Analyse-Methode" und wählen Sie "Erzeugen eine Akquisition und Verarbeitungsmethode." " Geben Sie den Namen der Methode und navigieren Sie zu der Methode Ordner (Verzeichnis) und klicken Sie dann auf "Weiter".
- Wählen Sie für"Analyse" "Peptid Karte (IMS)", und wählen Sie im Reiter "Instrument System", das "Vion IMS QTof" Instrument. Klicken Sie auf "Fertig stellen".
Hinweis: Die neu erstellte LC/HDMSE -Methode öffnet sich automatisch im Fenster "Methode Gerätekonfiguration". - In der Registerkarte "Einstellungen", geben Sie diese Parameter: 3,0 kV Kapillare Spannung, 100 ° C Quelltemperatur zugeführten Temperatur 250 ° C, 50 L/h Kegel Gasstrom und 500 L/h zugeführten Gasstrom.
- In den "Experiment" Registerkarte "Einstellungen wählen Sie"High-Definition-MSE"und"Analyse Methode Laufzeit." Im Register "Einstellungen" geben Sie diese Parameter: niedrige Masse, 100; Hochamt, 2.000; Scan-Zeit, 400 Ms. im Register "CE" Typ "6 V" für die niedrig-Energie-Einstellung und wählen Sie eine energiereiche Rampe aus 15 bis 40 V. Stellen Sie sicher, "Disable Data Reduction" aktiviert haben.
- Eine Lösung von 50 ng/mL Leucin Enkephalin (LE) in 50 % Acetonitril mit 0,1 % vorbereitet die Infusion FA bei einer Durchflussrate von 10 µL/min für Lock-Masse Kalibrierung während der Datenerfassung.
Hinweis: Die Sperre-Massendaten werden alle 5 min. mit der gleichen Erfassungsrate über den gleichen Massenbereich erworben.
- 10-nM, PHO-dotierten Probe des LC/HDMSE Tests beschriebenen durch Injektion 10 µL der Probe (die Spalte "Betrag geladen auf-" ist 100 Fmol für jedes Peptid standard) zu analysieren.
4. Setup der Tof-MRM-Methode für die Kollision Energieoptimierung (CE)
- Erhalten Sie aus dem Dataset LC/HDMSE die Retentionszeit, der Vorläufer und das Fragment Ionen m/Z für jedes PHO-Peptid. Behalten die m/Z und den Ladezustand für die am häufigsten vorkommende Vorläufer Ionen und die entsprechenden drei am häufigsten vorkommende fragment Ionen.
Hinweis: Ein Beispiel für Niedrigenergie- und energiereiche Spektren, die in der Regel von der LC/HDMSE Test aufgenommen wird in Abbildung 2 dargestellt. - Verwenden Sie Kollision Energieoptimierung (CE) in einem SRM/MRM-Experiment, um das beste Signal für jedes Peptid-23zu erhalten.
- Klicken Sie in der Datenerfassungs-Software auf "Create/Analyse-Methode" und wählen Sie "Erzeugen eine Akquisition und Verarbeitungsmethode." " Geben Sie den Namen der Methode, navigieren Sie zu der Methode Ordner (Verzeichnis), und klicken Sie auf "Weiter".
- Wählen Sie für"Analyse", "Quantify." Wählen Sie "Quantify Assay Tof 2D chromatographische" und klicken Sie auf "Weiter". Wählen Sie in der Registerkarte "Instrument System" "Vion IMS QTof" und klicken Sie auf "Fertig stellen".
Hinweis: Die neu erstellte Tof-MRM-Methode öffnet sich automatisch im Fenster "Methode Gerätekonfiguration". - In der Registerkarte "Einstellungen", geben Sie diese Parameter: 3,0 kV Kapillare Spannung, 100 ° C Quelltemperatur zugeführten Temperatur 250 ° C, 50 L/h Kegel Gasstrom und 500 L/h zugeführten Gasstrom.
- Wählen Sie in den "Experiment" Registerkarte "Einstellungen"Funktionstabelle"und wählen Sie"eine oder mehrere MS, MSM oder MRM Funktionen." Richten Sie die Symbolleistenschaltflächen verwenden, eine "Tof-MRM" Funktion für jedes Peptid durch Eingabe von drei "Übergänge" (Kombinationen von der am häufigsten vorkommende Vorläufer und drei der am häufigsten vorkommende Fragment-Ionen).
Hinweis: Im Tof-MRM-Modus wird die höchste Empfindlichkeit erreicht mit einem geplanten MRM-Ansatz. - Wählen Sie in den "Tof-MRM" Funktionseditor ein Zurückbehaltungsrecht Zeitfenster für jedes Peptid (mindestens 1 min lang), entsprechend der Peptid Elution Reihenfolge angeordnet. Setzen Sie in den "Tof-MRM" Funktionseditor Peptid Vorläufer m/Z und das Produkt m/Z; Wählen Sie das Quad Isolierung Fenster als "Low" (4-Da Fenster) für die Peptid-Vorstufe; Wählen Sie eine feste Scanzeit von 100 ms; und wählen Sie zwölf verschiedene Kollision Energie-Werte im Bereich von 14-36 V, wie folgt: 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34 und 36, wie in dem Beispiel in Abbildung 3 dargestellt.
Hinweis: Mit dem oben beschriebenen Tof-MRM-Verfahren sind mindestens 10 Datenpunkte pro chromatographischen Gipfel für jeden Übergang bei jeder CE angegeben gesammelt. Um die Größe der Daten-Datei zu reduzieren, die Option "Wide" (6-Da Fenster) für das Signal der Fragment-Ionen. Beispiele für die CE-optimierten Werte sind in Tabelle 2 dargestellt. - 10-nM, PHO-dotierten Probe der Tof-MRM-Assays beschriebenen durch Injektion 10 µL der Probe (die Spalte "Betrag geladen auf-" ist 100 Fmol für jedes Peptid standard) zu analysieren.
5. Einrichtung von der endgültigen HS-MRM Erwerbsmethode Peptid Quantifizierung mit Ionen-Mobilität Trennung von Peptid-Vorstufen
- Im Menü "Investigate" der Datenerfassungs-Software zeigt alle 11 chromatographische Spuren erzeugt für jedes CE für jedes Peptid und integrieren die Gipfeln mit dem "Integrieren" Button in der Symbolleiste "Processing Optionen/Operations"; die höchsten Peakfläche zeigt die besten CE für jeden Übergang.
- Visuell vergleichen Sie die besten Peakflächen für die 3 Fragmente des jedes Peptid und behalten Sie nur das beste "Übergang" (das Fragment Ion, die das intensivste Signal erzeugt); Tabelle 2 zeigt die besten "Übergänge" von vier PHO Peptide erhalten.
- Richten Sie eine HS-MRM-Methode, die nur ein Fragment-Ionen pro Peptid enthält. Verwenden Sie die CCS-Werte jedes Peptid-Vorstufe aus dem LC/HDMSE -Dataset.
- Ändern Sie die Tof-MRM-Methode erstellt, die im vorherigen Abschnitt (Abschnitt 4) durch Auswahl der Funktion "HS-MRM" statt der "Tof-MRM"-Funktion.
- In der "HS-MRM" Funktions-Editor, geben Sie die folgenden Vorläufer-bezogenen Parameter: m/Z, Quadrupol-Auflösung: niedrig (4 Da), Aufladen von Staat, Vorläufer CCS Wert und Vorläufer CCS Auflösung: Low. Geben Sie für das Produkt Ion, seine m/Z, die optimale Aufprallenergie, wählen Sie eine Scanzeit von 0,4 s und wählen Sie eine Einstellung "Spektrum" (6 Da), um alle seine Isotope enthalten. Ein Beispiel für die letzte HS-MRM-Erwerb-Methode für alle vier Peptide wird in Abbildung 4 dargestellt.
- Alle Proben mit der HS-MRM-Methode, beginnend mit der mAb-Digest leer (nicht dotierten Probe), gefolgt von 4 wiederholte Injektionen (10 µL) der folgenden Konzentrationen zu analysieren: 0,1, 1, 10 und 100 nM PHO Peptide.
6. Schaffung eine Verarbeitungsmethode für die Analyse des HS-MRM-Datasets
- Erstellen Sie eine Verarbeitungsmethode für die Analyse des HS-MRM-Datasets.
- Erstellt für die Erwerbsmethode "HS-MRM" Daten-Analyse-Methode klicken Sie auf die Registerkarte "Zweck" und wählen Sie "Manage-Komponenten."
- Dropdown-Menü unter "Experiment Typ" die Option "HS MS/MS/HS-MRM".
- Klicken Sie auf "Create/New Entry" und führen Sie die folgenden Parameter im Methodeneditor Verarbeitung: Peptid Verweilzeit (RT), Vorläufer Ladezustand, Vorläufer m/Z und Vorläufer Drift Zeit (DT). Für das Fragment Ion geben Sie seinen Zustand m/Z und kostenlos und die "XIC" Spur als des Extraktionsmodus.
- Im Register "Zweck" der gleiche "HS-MRM" Erwerbsmethode, klicken Sie auf"Standard" und geben Sie die folgenden Konzentrationen: 0,1, 1, 10 und 100 nM PHO Peptide.
- Geben Sie die folgenden Parameter in der "Verarbeitung/Extraktionseinstellungen": Masse Standardtoleranz, 10 ppm (für den m/Z alle Fragment-Ionen) und Standard drift Zeit, 5 %; ein Beispiel für die PHO Peptid Verarbeitungsmethode ist in Abbildung 5 dargestellt.
- Kalibrierung wählen Sie die lineare Kurvenanpassung mit 1 / X2 Gewichtung.
- Prozessdaten der HS-MRM, alle Chromatogramme integrieren und Eichkurven von jedem PHO-Peptid zu konstruieren.
Ergebnisse
Die einzelnen Sequenzen von sechs Phosphorylase b Peptid-Normen in die PHO-Peptid-Gemisch enthaltenen sind in Tabelle 1 gezeigt, zusammen mit ihre Retentionszeiten und experimentieren Sie ihre am häufigsten vorkommenden Vorstufen in der HDMSE beobachtet. Der erste Schritt in der Entwicklung eines hohen Selektivität (HS) MRM-Assays ist der Erwerb von einem Dataset HDMSE herstellen der Elution Zeiten jeder PHO-Peptid, zusammen mit ihren entsprechenden reichlich Vorläufer und die drei am häufigsten vorkommende Fragmente. Abbildung 2 zeigt die HDMSE -Spektren für eines der PHO Peptide (Pep 6) versetzt in der Infliximab-Digest erworben. Nach der Gründung der 3 Bestes "Übergänge" (Kombinationen von Vorstufe und Fragment Massen) für jedes Peptid, ist ein Tof-MRM-Experiment durchgeführt, um die optimale Aufprallenergie, die Signale generiert für jedes Peptid zu maximieren zu finden. Die Ergebnisse des Experiments CE Optimierung sind in Tabelle 2 zusammengefasst. Die letzte HS-MRM-Assay (siehe Abbildung 4) behält nur das beste "Übergang" für jedes Peptid und ist für die Analyse aller dotierten Proben verwendet. Beispiele für HS-MRM Chromatogramme generiert für 4 PHO Peptide über alle untersuchten Konzentrationen sind in Abbildung 7 dargestellt. Vier Kalibrierkurven erhalten für jedes Peptid folgt die Integration der HS-MRM Gipfel markiert in Abbildung 7 sind in Abbildung 8 dargestellt. Darüber hinaus ist Spitze Bereich relative Standardabweichung berechnet basierend auf 4 wiederholte Injektionen, in 4 in Tabelle 3 dargestellten Tabellen zusammengefasst.
| Peptid | Peptid | Aufbewahrung | Ladungszustände | ||||
| ID | Sequenz | Zeit (min) | + 1 | + 2 | + 3 | + 4 | |
| PEP 1 | VLYPNDNFFEGK | 19.4 | 1442.6951 | 721.8512 | 481.5699 | 361.4292 | |
| PEP 2 | TCAYTNHTVLPEALER | 16.0 | 1874.9065 | 937.9569 | 625.6404 | 469.4821 | |
| PEP 3 | IGEEYISDLDQLRK | 18.9 | 1678.8646 | 839.9360 | 560.2931 | 420.4716 | |
| PEP 4 | LLSYVDDEAFIR | 21.1 | 1440.7369 | 720.8721 | 480.9172 | 360.9397 | |
| PEP 5 | LITAIGDVVNHDPVVGDR | 19,7 | 1890.0080 | 945.5076 | 630.6742 | 473.2574 | |
| PEP 6 | VFADYEEYVK | 17.7 | 1262.5939 | 631.8006 | 421.5362 | 316.4039 | |
Tabelle 1. PHO Peptid Normen enthalten in der Masse PREP-Mischung in der Infliximab-Digest gespickt. Peptid Retentionszeiten und deren häufigsten Vorstufen (fett hervorgehoben) werden im Tabellenformat angezeigt.
| Peptid | Peptid | Aufbewahrung | Peptid-Vorläufer | Am häufigsten vorkommende Fragment-Ionen/Ladung | Optimale | ||||
| ID | Sequenz | Zeit (min) | m/Z & kostenlos | Drift-Zeit (ms) | Ich | II | III | CE (V) | |
| PEP 2 | TCAYTNHTVLPEALER | 16.0 | 625.6404 (+ 3) | 6.2 | 714.3781 (+ 1) | 807.4177 (+ 2) | 827.4621 (+ 1) | 24 | |
| PEP 4 | LLSYVDDEAFIR | 21.1 | 720.8721 (+ 2) | 7.5 | 865.4050 (+ 1) | 964.4734 (+ 1) | 1214.5688 (+ 1) | 22 | |
| PEP 5 | LITAIGDVVNHDPVVGDR | 19,7 | 630.6742 (+ 3) | 6.3 | 642.3570 (+ 1) | 689.8391 (+ 2) | 832.4236 (+ 2) | 20 | |
| PEP 6 | VFADYEEYVK | 17.7 | 631.8006 (+ 2) | 7.0 | 830.3931 (+ 1) | 945.4200 (+ 2) | 1016.4571 (+ 1) | 24 | |
Tabelle 2. Ergebnisse des Experiments Tof-MRM Optimierung: die drei am häufigsten vorkommende Fragmente von jeder PHO-Peptid in dieser Studie quantifiziert werden angezeigt, zusammen mit den entsprechenden optimierte Aufprallenergie.
| Conc | Betrag | PEP 2 Peakflächen (Tabelle 3A) | |||||
| (nM) | auf Spalte (Fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Meine | RSD (%) |
| 0.1 | 1 | 490 | 439 | 462 | 431 | 456 | 5.8 |
| 1 | 10 | 5121 | 4842 | 5198 | 4842 | 5001 | 3.7 |
| 10 | 100 | 63853 | 64279 | 66111 | 62509 | 64188 | 2.3 |
| 100 | 1000 | 612392 | 605553 | 613229 | 611004 | 610545 | 0,6 |
| Conc | Betrag | PEP 4 Peakflächen (Tabelle 3 b) | |||||
| (nM) | auf Spalte (Fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Meine | RSD (%) |
| 0.1 | 1 | 275 | 359 | 325 | 288 | 312 | 12.2 |
| 1 | 10 | 3559 | 3694 | 3287 | 3754 | 3574 | 5.8 |
| 10 | 100 | 45259 | 45775 | 42976 | 45548 | 44890 | 2.9 |
| 100 | 1000 | 459374 | 467927 | 436272 | 458994 | 455642 | 3.0 |
| Conc | Betrag | PEP 5 Peakflächen (Tabelle 3) | |||||
| (nM) | auf Spalte (Fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Meine | RSD (%) |
| 0.1 | 1 | 3194 | 3243 | 3202 | 3257 | 3224 | 1.0 |
| 1 | 10 | 31464 | 31150 | 31464 | 31433 | 31378 | 0,5 |
| 10 | 100 | 313638 | 320712 | 311943 | 311943 | 314559 | 1.3 |
| 100 | 1000 | 2845736 | 2840031 | 2882006 | 2864052 | 2857956 | 0,7 |
| Conc | Betrag | PEP 6 Peakflächen (Tabelle 3D) | |||||
| (nM) | auf Spalte (Fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Meine | RSD (%) |
| 0.1 | 1 | 490 | 583 | 440 | 440 | 488 | 13.8 |
| 1 | 10 | 6429 | 6429 | 6848 | 6623 | 6582 | 3.0 |
| 10 | 100 | 71295 | 70400 | 71563 | 70400 | 70915 | 0,9 |
| 100 | 1000 | 707640 | 707640 | 694461 | 729490 | 709808 | 2.0 |
Tabelle 3. Tabelle mit der Peakflächen von HS-MRM Chromatogramme aufgezeichnet für 4 PHO Peptide (Pep 2, 4, 5 und 6) für jede LC/MS-Injektion (16 LC/MS Durchläufe und 4 Konzentrationen getestet).
Die relative Standardabweichung war besser als 15 % für alle Peptide über den gesamten Konzentrationsbereich untersucht.

Abbildung 1: Arbeitsablauf-Diagramm fasst die drei Schritte zum Einrichten einer HS-MRM Erwerbsmethode. Bitte klicken Sie hier für eine größere Version dieser Figur.
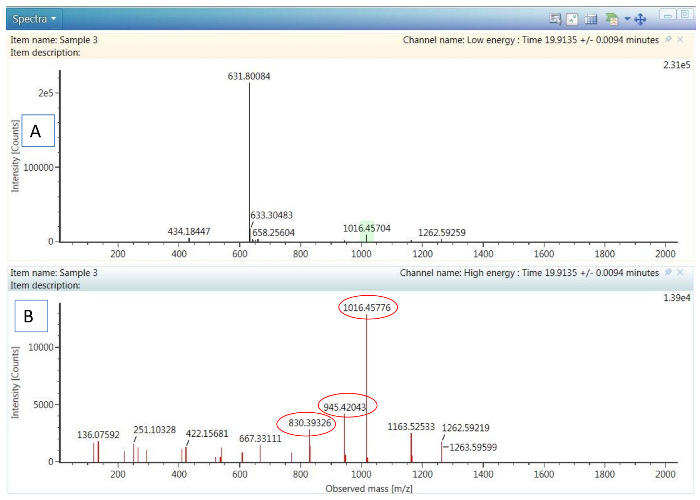
Abbildung 2. Beispiel für HDMSE Daten:
(A) Niedrig-Energie-Spektrum zeigt die Pep 6 Vorläufer Ionen. (B) hochenergetische Fragmentierung Spektrum des gleichen Peptids, Anzeige der Top 3 am häufigsten vorkommende Fragment-Ionen (eingekreist) für Tof-MRM Kollision Energieoptimierung ausgewählt. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Für die Einrichtung einer Tof-MRM Optimierung verwendeten Parameter experimentieren.
Für jeden Übergang wurden elf Kollision Energien (im Bereich von 16 bis 36 V) getestet. Bitte klicken Sie hier für eine größere Version dieser Figur.
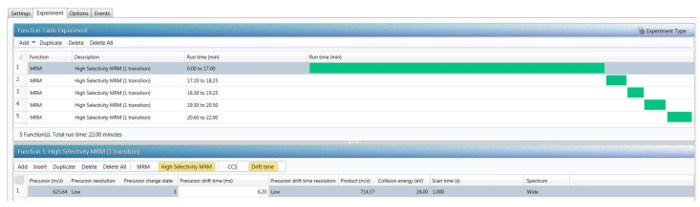
Abbildung 4. Beispiel für die endgültige HS-MRM-Methode.
Mehrere Parameter sind erforderlich für jeden "Übergang", darunter das Peptid Vorläufer m/Z, den Ladezustand und Ionen-Mobilität Drift Zeit, von der am häufigsten vorkommende Fragment-Ionen, die optimale Aufprallenergie und MS-Erfassungszeit m/Z. Bitte klicken Sie hier für eine größere Version dieser Figur.
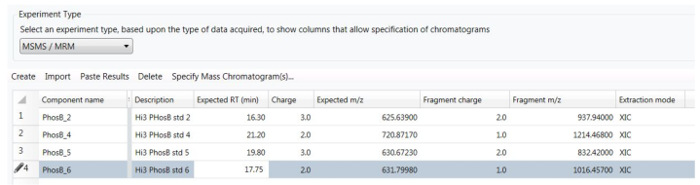
Abbildung 5. Einstellungen durch das Bearbeitungsverfahren für die Analyse der HS-MRM-Dataset verwendet.
Jedes Peptid wird überwacht durch einen einzigen "Übergang" von der Peptid-Vorläufer m/Z beschrieben, Ladezustand und erwartet Retentionszeit, zusammen mit der m/Z das am häufigsten vorkommende Fragment und den Ladezustand. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6. Schematische Darstellung der Masse Ionenmobilitätsspektrometer.
In der HS-MRM Akquisitionsmodus sind die Vorläufer des Peptids, die quantifiziert werden andere Co medikamentenfreisetzende (störenden) Peptid-Vorläufer in der Ionen-Mobilität Zelle, durch den Quadrupol isoliert und fragmentiert mit einem festen Aufprallenergie in getrennter der Kollision Zelle. Das Signal von der Peptid-Fragment-Ionen produziert wird durch Anpassung der Schieber, und Peptid Quantifizierung erfolgt mit Hilfe der hochauflösenden MS (> 30.000) Signale durch das intensivste Fragment Ion jedes Peptid produziert. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 7. HS-MRM Chromatogramme aufgezeichnet für 4 PHO Peptide in 4 verschiedenen Konzentrationen überspannt 3 Größenordnungen (0,1, 1, 10 und 100 nM).
(A) Pep 2 Chromatogramme, (B) Pep 4 Chromatogramme (C) Pep 5 Chromatogramme und Pep (D) 6 Chromatogramme. Bitte klicken Sie hier für eine größere Version dieser Figur.
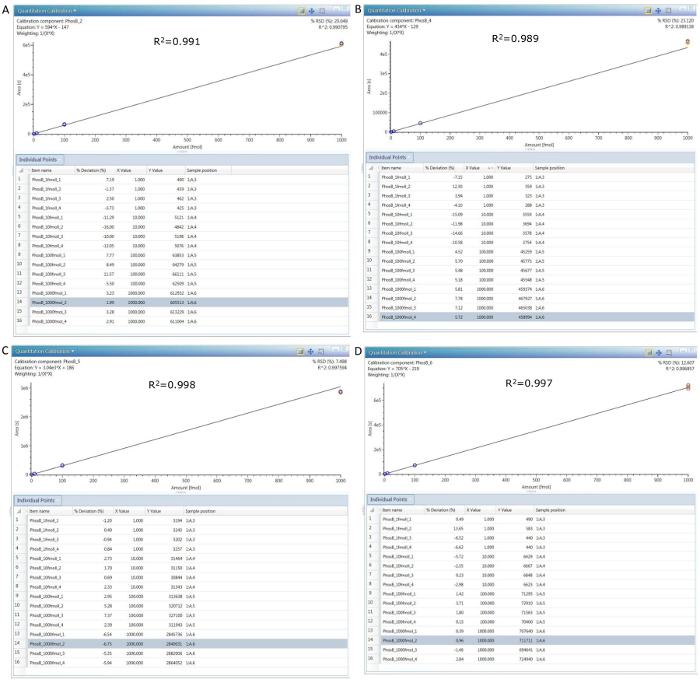
Abbildung 8. Kalibrierkurven für 4 PHO Peptide in 4 verschiedenen Konzentrationen (0,1, 1, 10 und 100 nM).
Die Tabellen unter jede Kurve zeigen die einzelnen Peakflächen (Y-Werte) für jede Injektion aufgezeichnet, während die zweite Spalte aus jeder Tabelle die prozentuale Abweichung von der erwartete lineare Reaktion zeigt. (A) Pep 2 Kalibrierung, (B) Pep 4 Kalibrierung (C) Pep 5 Kalibrierung und Pep (D) 6 Kalibrierung. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Hochauflösende (Rs > 20.000) Massenspektrometrie wird routinemäßig für die strukturelle Charakterisierung von therapeutischen Proteinen auf eine Vielzahl von Plattformen Instrument eingesetzt. Im Gegensatz dazu MS-basierte Protein Quantifizierung in der Regel von SRM/MRM mit niedriger Auflösung (~ 1.000 Rs) Tandem Quadrupol-Massenspektrometern mit Unterschrift Peptide erzeugt durch die enzymatische Spaltung von Proteinen11,12 erfolgt auf , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23. wie Single-Dimension chromatographische Trennungen komplexe Peptid-Mischungen hergestellt durch enzymatische Verdauung vollständig auflösen können, Co Elution Peptid ist ein gemeinsames auftreten, auch bei einer Single-Protein zu verdauen. Für sehr komplexe Eiweiß verdaut (z. B. für die Quantifizierung von 1-100 ppms von HCPs in Anwesenheit von einem Peptid-reichen Hintergrund durch das therapeutische Protein produziert), Empfindlichkeit, Genauigkeit oder Linearität der SRM/MRM kann Assay betroffen Störungen.
Die SRM/MRM-Assays sind eindimensional Selektivität, verlässt sich nur auf eine "einzigartige" Kombination der Vorläufer/Fragment Massen. Aus diesem Grund diese Assays fehlschlagen in Situationen, wenn die Peptid-Hintergrund ändert sich unerwartet (z. B. für biopharmazeutische Proben aus verschiedenen Reinigung Verfahren gewonnen). Um diese Einschränkungen zu überwinden, wir schlagen hier eine hohe Selektivität (HS) MRM-Assay umgesetzt auf eine Ionen-Mobilität ermöglicht hochauflösende Quadrupol Time-of-Flight (QTOF) Hybrid-Massenspektrometer (für das Instrument-Diagramm, siehe Abbildung 6).
Das Instrument trennt die Vorläufer des Peptids von Interesse aus anderen Co medikamentenfreisetzende (störenden) Peptid-Vorstufen in der Ionen-Mobilität Zelle, isoliert die volle isotopischen Umschlag der Vorstufe in die Quadrupol und mit einem festen CE in Fragmente der Kollision Zelle. Das Signal erzeugt durch seine am häufigsten vorkommende Peptid Fragment wird verstärkt durch Anpassung der Drücker (Target Enhancement), die selektiv Masse Regionen von Interesse in der Flug-Röhre, anstatt alle Ionen, wie bei einen vollständigen Scan drückt. Peptid-Quantifizierung erfolgt mit Hilfe der MS-hochauflösende (Rs ~ 30.000) Signale von diesem Fragment-Ionen produziert. Verglichen mit der SRM/MRM-Assays, die HS-MRM-Assay bietet zwei zusätzliche Ebenen der Selektivität: ist bestimmt durch die Vorläufer-Ebene Ion Mobilität Trennung, während die zweite durch die erhöhte Masse Auflösung des TOF Analyzer angeboten wird. Die Ergebnisse, die durch diese Selektivität Verbesserungen gebracht sind sichtbar in die HS-MRM Chromatogramme in Abbildung 7 dargestellt, die über drei Größenordnungen frei von Störungen sind.
Im Gegensatz zu der SRM/MRM-Assays, gibt es verschiedene Parameter, die angepasst werden können, um die HS-MRM-Assays zu optimieren: die RT-Fenster um die Peptid-Vorläufer (in der Regel bei 0,2 min eingestellt), Quadrupol Isolierung Fenster (4 Da), die Drift-Zeitfenster rund um die Vorläufer (± FWHM des Vorläufer Peak aus der entsprechenden Ionen-Mobilogram), und die MS Auflösung der Fragment-Ionen (20.000-40.000). Die HS-MRM-Assays sind sehr empfindlich: der niedrigste erkannten Betrag für jedes PHO Peptide ist 1 Femtomole auf-Spalte (oder 0,1 nM in Bezug auf die Peptid-Konzentration). Bei der Betrachtung von Peptid MW (siehe Tabelle 1 für genaue MWs), die Menge erkannt auf Spalte wird in der Größenordnung von 1-2-Pg, während die Spalte mit einem deutlich höheren Betrag (2 µg) Hintergrund Peptide aus dem mAb-Digest geladen wird.
Wenn man das Molekulargewicht des Full-Length PHO Proteins (97,2 kDa) dotierten Peptide abgeleitet wurden, ist der Test 50 ppm eine Protein-Verunreinigung in Gegenwart von hohen Fülle Hintergrund Ionen erkennen. Untergrenze der Erkennung (5-10 ppm) sind für geringere Molekulargewicht HCPs (10-20 kDa) erreichbar. Der Test umfasst drei Größenordnungen (siehe die Kalibrierkurven aus Abbildung 8), was bedeutet, dass es im Bereich von 1-1.000 ppm HCPs messen kann. Die Reproduzierbarkeit der HS-MRM-Assays, in Tabelle 3 dargestellten entspricht auch, sehr gut mit der Reproduzierbarkeit von klein-Molekül SRM/MRM Assays mit Peakfläche RSDs besser als 15 %.
Wir erforschten die Fähigkeiten eines neuartigen Assays für die Quantifizierung von Spike Peptid Normen in ein monoklonaler Antikörper (mAb) verdauen und demonstriert seine Empfindlichkeit und Dienstprogramm, um den Dynamikbereich (mindestens drei Größenordnungen) in der Regel decken begegnet in der HCP-Analyse. Alle sechs Peptid-Standards wurden festgestellt bei Konzentrationen von 0,1 nM (1 Femtomole geladen auf einer 2,1-mm ID chromatographischen Säule) in Anwesenheit hoher Fülle Peptid Hintergrund (2 µg einer mAb-Digest geladen auf Spalte). Durch den Einbau von gezielten HRMS und Ionen-Mobilität Vorläufer Trennung, hat die HS-MRM-Assay großes Potential für immer eine schnelle, Hochdurchsatz-Überwachung-Assay für mehrere HCPs über mehrere Batches von Biopharmazeutika.
Offenlegungen
Alle Autoren sind Mitarbeiter der Waters Corporation, die der Produzent von mehreren Reagenzien und Instrumenten, die in diesem Artikel verwendet wird.
Danksagungen
Die Autoren möchten Lesley Malouin und Tony Catlin von Waters Corporation für die Zubereitung von Abbildung 6 des Manuskripts danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Vion IMS Qtof mass spectrometer | Waters | 186009214 | |
| Acquity H-Class Quaternary solvent manager (QSM) | Waters | 186015041 | |
| Acquity H-Class FTN Sample Manager (SM) | Waters | 186015040 | |
| Acquity H-Class Column Manager (CM) | Waters | 186015043 | |
| Acetonitrile (ACN) | Fisher Chemical | A996-4 | |
| Ammonium bicarbonate | Sigma Aldrich | 40867-50G-F | |
| 2.1 x 150 mm CSH C18 UPLC column, 1.8 µm particles | Waters | 186005298 | |
| Dithiothreitol (DTT) | Sigma Aldrich | D5545-5G | |
| Formic acid, eluent additive for LC/MS | Sigma Aldrich | 56302-10X1ML-F | |
| Hi3 rabbit phosphorylase (PHO) MassPREP standard | Waters | 186006011 | |
| Iodoacetamide (IAM) | Sigma Aldrich | I-1149-5G | |
| LC vials (12x32 mm glass vials, screw neck) | Waters | 186000327c | |
| Leucine Enkephalin acetate salt hydrate | Sigma Aldrich | L9133-10MG | |
| Protein LoBind 2.0 mL tubes (2x50) | Eppendorf | 22431102 | |
| RapiGest SF | Waters | 186001861 | |
| Trypsin/Lys-C enzyme mix, mass spec grade (5 x 20 µg) | Promega | V5073 | |
| Water, LC/MS grade | Sigma Aldrich | 39253-1L-R |
Referenzen
- Doneanu, C. E., et al. Analysis of host-cell proteins in biotherapeutic proteins by comprehensive online two-dimensional liquid chromatography/mass spectrometry. MAbs. 4, 24-44 (2012).
- Schenauer, M. R., Flynn, G. C., Goetze, A. M. Identification of host-cell protein impurities in biotherapeutics using mass spectrometry. Anal Biochem. 428, 150-157 (2012).
- Zhang, Q., et al. Comprehensive tracking of host-cell proteins during monoclonal antibody purifications using mass spectrometry. MAbs. 6, 659-670 (2014).
- Farrell, A., et al. Quantitative host-cell protein analysis using two-dimensional data independent LC-MS(E). Anal Chem. 87, 9186-9193 (2015).
- Doneanu, C. E., et al. Enhanced detection of low-abundance host cell protein impurities in high-purity monoclonal antibodies down to 1 ppm using ion mobility mass spectrometry coupled with multidimensional liquid chromatography. Anal Chem. 87, 10283-10291 (2015).
- Doneanu, C. E., et al. Improved identification and quantification of host cell proteins (HCPs) in biotherapeutics using liquid chromatography mass spectrometry. Technologies for therapeutic monoclonal antibody characterization, vol. 3: Defining the next generation of analytical and biophysical techniques. , Washington DC. ACS Symposium series 1202 357-393 (2015).
- Zhang, Q., et al. Characterization of the co-elution of host-cell proteins with monoclonal antibodies during protein A purification. Biotechnol Prog. 32, 708-717 (2016).
- Reisinger, V., Toll, H., Mayer, R. E., Visser, J., Wolschin, F. A mass spectrometry based approach to host cell protein identification and its application in a comparatibility exercise. Anal Biochem. , 1-6 (2014).
- Madsen, J. A., et al. Toward the complete characterization of host-cell proteins in biotherapeutics via affinity depletions, LC/MS/MS and multivariate analysis. MAbs. 7, 1128-1137 (2015).
- Giles, K., et al. Applications of a trevelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18, 2401-2414 (2004).
- Anderson, V., Hunter, C. L. Quantitative mass spectrometric multiple reaction monitoring assays for major plasma proteins. Mol Cell Proteomics. 5, 573-588 (2006).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Mol Syst Biol. 4, 1-14 (2008).
- Picotti, P., et al. High-throughput generation of selected reaction-monitoring assays for proteins and proteomes. Nat Methods. 7, 43-46 (2010).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. (53), (2011).
- Liebler, D. C., Zimmerman, L. J. Targeted quantitation of proteins by mass spectrometry. Biochemistry. 52, 3797-3806 (2013).
- Manes, N. P., Mann, J. M., Nita-Lazar, A. Selected reaction monitoring mass spectrometry for absolute protein quantification. J Vis Exp. (102), (2015).
- Ebhardt, H. A., Sabido, E., Huttenhain, R., Collins, B., Aebersold, R. Range of protein detection by selected/multiple reaction monitoring mass spectrometry in an unfractioned human cell culture lysate. Proteomics. 12, 1185-1193 (2012).
- Picotti, P., Bodenmiller, B., Mueller, L. N., Domon, B., Aebersold, R. Full dynamic range proteome analysis of S. cerevisiae by targetted proteomics. Cell. 138, 795-806 (2009).
- Sherman, J., McKay, M. J., Ashman, K., Molloy, M. P. How specific is my SRM: the issue of precursor and product ion reductancy. Proteomics. 9, 1120-1123 (2009).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of innacurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Rost, H., Malmstrom, L., Aebersold, R. A computational tool to detect and avoid redundancy in selected reaction monitoring. Mol Cell Proteomics. 11, 540-549 (2012).
- Bao, Y., et al. Detection and correction of interference in SRM analysis. Methods. 61, 299-303 (2013).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Mbasu, R. J., et al. Advances in quadrupole and time-of-flight mass spectrometry for peptide MRM based translational reserch analysis. Proteomics. 16, 2206-2220 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten