Method Article
Un dosage de HS-MRM pour la Quantification des protéines de la cellule-hôte en protéine Biopharmaceuticals par chromatographie en phase liquide Ion Mobility QTOF spectrométrie de masse
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous décrivons ici une analyse chromatographique couplée avec la séparation de mobilité ionique des précurseurs de peptide suivie de la haute résolution (~ 30 000) MS-détection des fragments peptidiques pour la quantification des normes peptide enrichis à un anticorps monoclonal Digest.
Résumé
L’analyse des impuretés de protéine de bas niveau (1-100 ppm) (p.ex. : protéines de cellule hôte (PS)) en protéine biothérapeutique est un dosage difficile nécessitant une haute sensibilité et une gamme dynamique étendue. Essais de quantification basé sur la spectrométrie de masse de protéines comprennent habituellement la digestion des protéines, suivie de la réaction de surveillance/multiple réaction sélective suivi de quantification (SRM/MRM) des peptides utilisant un tandem (~ 1 000 Rs) basse résolution spectromètre de masse quadripolaire. Une des limites de cette approche est le phénomène d’interférence observé quand le peptide d’intérêt a la « même » précurseur et la masse de fragment (en termes de valeurs de m/z) comme autres peptides co élution présents dans l’échantillon (dans une fenêtre 1-Da). Pour éviter ce phénomène, nous vous proposons une autre approche par spectrométrie de masse, un essai MRM haute sélectivité (HS) qui allie la séparation de mobilité ionique des précurseurs de peptide à la haute résolution (~ 30 000 Rs) MS détection des fragments peptidiques. Nous avons exploré les capacités de cette approche pour quantifier les normes de faible abondance peptide fortifiés dans un condensé de l’anticorps monoclonal (ACM) et démontré qu’elle a de la sensibilité et la plage dynamique (au moins 3 ordres de grandeur), généralement réalisés dans HCP analyse. Tous les six étalons de peptide ont été détectés à des concentrations aussi faibles que 0,1 nM (1 femtomole chargées sur une colonne chromatographique de 2,1 mm ID) en présence d’un fond de peptide haute-abondance (2 µg d’une mAb digest chargé sur-colonne). Si l'on considère le MW de la phosphorylase de lapin (97,2 kDa), dont sont dérivés les peptides enrichis, la limite de dosage de ce test est inférieure à 50 ppm. Écart types relatifs (RSD) des domaines de pointe (n = 4 répétitions) ont moins de 15 % sur toute la gamme de concentration tout étudiée (0,1 à 100 nM ou 1 à 1 000 ppm) dans cette étude.
Introduction
Quantification des grandes molécules (protéines) dans les milieux industriels repose actuellement sur les immuno-essais (p. ex., ELISA), principalement en raison de plusieurs avantages : sensibilité, haut débit, facilité d’utilisation et faible coût par échantillon. Lorsqu’il est appliqué pour analyser les impuretés de la protéine de faible abondance (1 à 100 ppm de protéines (PS) de la cellule hôte) présentes dans les protéines thérapeutiques, ces dosages biologiques fournissent généralement la concentration totale de HCP (habituellement exprimée en ppm ou ng mAb HCP/mg), mais ils ne peut pas identifier et mesurer les contaminants HCP individuels. Plusieurs analyses MS récemment ont été développées pour compléter des tests ELISA ou des renseignements qui ELISA ne parviennent pas à offrir1,2,3,4,5,6 , 7 , 8 , 9. en raison de la complexité de l’échantillon et l’obligation de détecter des peptides HCP à travers une large gamme dynamique de concentration (au moins 3 ordres de grandeur), ont des méthodes de chromatographie multidimensionnelles d’appels d’offres de fractionnement vaste échantillon traditionnellement utilisé pour aider à identifier la faible abondance PS1,2,3,4,5,6,7.
Un naturel étape suite HCP identification et validation est suivi de HCP (surveillance) à travers plusieurs lots de produits biopharmaceutiques. Dans cette situation, unidimensionnel LC/MS méthodes ont été proposées pour améliorer l’échantillon débit8,9. Cependant, l’exactitude et les mensurations de la gamme dynamique du HCP pourraient être affectées dans un essai de LC/MS 1D par la présence écrasante de peptides de la biopharmaceutique. Par rapport à une séparation multidimensionnelle, le potentiel d’interférence de signal19,20,21,22 est augmenté dans une séparation chromatographique unidimensionnel parce que la probabilité pour plusieurs précurseurs de peptide d’être co élution est augmentée. L’incorporation de moyens orthogonales pour séparer les précurseurs peptidiques sans allonger le temps de la séparation chromatographique serait nettement plus avantageuse. Voyage de vague ion mobility (TWIM)10 a la capacité de résoudre des spectres MS encombrées en millisecondes. Environ 500 départs de mobilité peuvent être effectuées pendant l’élution d’un peptide unique, en supposant une largeur du pic chromatographique complète de 10 s et étant donné que le runtime d’une séparation IM sur l’instrument de mobilité ionique est de 20 ms.
Les dosages par spectrométrie de masse pour la quantification des protéines ont été développés avec succès au cours de la dernière décennie en utilisant le reconnus sélectionné (multiples) réaction méthode (méthode de SRM/MRM) mis en œuvre sur de spectromètres de masse en tandem11, de surveillance 12,13,14,15,16,17,18,19,20,21 ,22,23. Une des limites de ce dosage par spectrométrie de masse basse résolution est l’interférence phénomène19,20,21,22 observé lorsque le peptide d’intérêt a le « même » précurseur et fragment de masse que d’autres peptides co élution présentent dans l’échantillon (dans une fenêtre 1-Da). Il existe deux façons d’améliorer la précision des méthodes SRM/MRM : une option comporte une étape supplémentaire de séparation au niveau du précurseur pour enlever les ions interférents précurseur, tandis que l’autre option est d’augmenter la résolution MS de la détection de précurseur/fragment à éviter le chevauchement des signaux de MS. Le mode d’acquisition de MRM (HS) haute sélectivité décrit ici profite des deux de ces approches en couplant la séparation de mobilité ionique des précurseurs de peptide à la haute résolution (~ 30 000 Rs) MS détection des fragments peptidiques. Le test décrit ici couvre au moins trois ordres de grandeur, qui est la gamme dynamique typiquement observées chez Mrs/MRM protéomique expériences17,18,24.
L’utilité de l’essai du HS-MRM pour la quantification de HCP a été démontrée en surveillant la linéarité du signal produit par six étalons de peptide dopés à différentes concentrations (plage de 0,1 à 100 nM) à un anticorps monoclonal "Digest".
Protocole
1. préparation de l’empreinte de l’infliximab (procédure ~ 24 h)
- Préparer de nouvelles solutions de bicarbonate d’ammonium de 50 mM, 1 % tensioactif anionique en 50 mM NH4HCO3, 500 mM dithiothréitol (DTT) 50 mM NH4HCO3et 500 mM iodoacétamide (IAM) 50 mM NH4HCO3.
- À 750 µL de 50 mM NH4HCO3, ajouter 200 µL d’infliximab mAb (10 mg/mL) et 50 µL de solution à 1 % tensioactif anionique et dénaturer les protéines pendant 15 min à 60 ° C en présence de 0.05 % tensioactif anionique.
- Ajouter 40 µL de 500 mM DTT et réduire l’échantillon pendant 60 min à 60 ° C en présence de 20 mM TNT.
- Ajouter 20 µL de 500 mM IAM et alkylat l’échantillon pendant 30 min à température ambiante dans l’obscurité en présence de ~ 10 mM IAM.
- Ajouter le contenu du tube microcentrifuge un flacon en verre contenant 20 µg de MS séquençage-grade Lys C/la trypsine et de digérer l’échantillon pendant 3 h à 37 ° C.
- Ajouter une deuxième enzyme aliquote (20 µg) en transférant l’échantillon digéré un autre flacon en verre contenant 20 µg de MS séquençage-grade Lys C/la trypsine et de digérer l’échantillon pendant la nuit (12-15 h) à 37 ° C.
- Après la protéolyse durant la nuit, ajouter 5 µL de l’acide formique (FA) de 100 % et incuber le digest pendant 30 min à 37 ° C se décompose l’acide labile anionique.
- Tournez en bas le digest pendant 15 min à 4 000 x g dans une centrifugeuse à précipiter du composant insoluble du tensioactif anionique.
- Récupérer ~ 1 000 µL du digest infliximab et placez-le dans un flacon échantillonneur automatique de LC ; la concentration de digest devrait être environ 2 mg/mL. Entreposer le condensé dans un congélateur à-20 ° C jusqu'à ce que le système LC/MS est prêt pour l’analyse.
2. de fortification des normes de peptide (~ 30 min)
- Décongeler le digest de l’infliximab en plaçant l’échantillon congelé dans le flacon de verre sur un banc à température ambiante.
- Ajouter 1 mL de 0.1 % FA en flacon de recueil H2O à un lapin phosphorylase b (PHO) pour préparer les solutions 1 µM pour tous les six étalons de peptide de la PHO contenue dans le flacon (le flacon contient 1 nmol de chaque peptide).
- Préparer des dilutions de 4 x 1 mL de la solution mère de PHO pour obtenir des solutions contenant 0,1, 1, 10 et 100 peptides nM, à l’aide de 0,1 % FA comme le solvant de dilution. Préparer toutes les dilutions en fioles de verre (LC échantillonneur automatique fioles) et ajouter 100 µL d’infliximab digest comme la matrice de base à tous les flacons. Charger 2 µg de fond digest sur colonne avec chaque injection de 10 µL. Préparer un échantillon témoin qui contient l’empreinte de l’infliximab dilué (le même 01:10 dilution), avec aucun des peptides enrichis.
3. la configuration de la méthode d’acquisition de données LC/HDMS E
NOTE : Le flux de travail résumant les étapes requises pour mettre en place une acquisition HS-MRM est représenté dans la Figure 1 et est décrit en détail dans les Sections 3-6. L’acquisition d’un ensemble de données HDMSE indépendante des données est nécessaire pour établir le temps de rétention de chaque peptide surveillé, le parent m/z de l’ion de peptide plus abondant après ionisation par électronébulisation, et le correspondant CCS (collision Croix section) dérivé de la séparation de mobilité ionique. En outre, le dataset indépendante des données fournit des informations au sujet de la m/z des trois ions fragments plus abondantes pour chaque précurseur du peptide. Dans la deuxième étape du flux de travail (optimisation de la CE), la sensibilité du test est augmentée par le réglage de l’énergie de cellule de collision pour obtenir l’intensité maximale d’ion pour chaque ion de fragment. Enfin, dans l’étape finale, tous les paramètres décrits ci-dessus sont introduits dans l’éditeur de méthode d’HS-MRM pour chaque peptide surveillé.
- Réaliser des expériences de LC/MS sur un temps de vol (QTOF) spectromètre de masse quadripolaire couplé à un système de chromatographie liquide à ultra hautes performances à l’aide d’une méthode d’acquisition de données indépendantes (HDMSE).
NOTE : Plus de détails concernant la configuration de l’instrument sont incluses dans la section matérielle. Cet instrument utilise un dispositif de mobilité ionique vague itinérante pour la séparation des peptides précurseurs10. - Préparer deux phases mobiles contenant 0,1 % de FA dans l’eau (solvant A) et 0,1 % FA dans l’acétonitrile (solvant B).
Remarque : Utilisez une colonne C18 à hybride surface chargée (CSH) (2,1 x 150 mm, emballé avec des particules de 1,7 µm) pour séparer les condensés de mAb enrichis. - Dans le logiciel d’acquisition de données, cliquez sur « Create/analyse méthode » et choisissez « Générer une acquisition et méthode de traitement. » Tapez le nom de la méthode, naviguez jusqu’au dossier de la méthode (répertoire) et cliquez sur « Suivant ».
- Pour « Type d’analyse », choisir « Peptide carte (IMS) » et dans l’onglet « Instrument système », sélectionnez l’instrument « Gestionnaire de solvant quaternaire ». Modifier les paramètres de gradients pour atteindre la séparation des peptides à un débit de 200 µL/min. utiliser une dégradé élution de 1 à 40 % de solvant B à 30min suivie d’un lavage de colonne 2 min et une équilibration 9-min, avec une totale autonomie de 50 min. introduire ces expérimental paramètres dans l’éditeur de méthode LC.
- Dans l’onglet « Instrument système », sélectionnez l’instrument « Sample Manager ». Régler la température de l’échantillon à 10 ° C et la température de la colonne à 60 ° C.
- Exploiter le spectromètre de masse QTOF en ion positif ESI sensibilité mode, avec un pouvoir de résolution typique de 30 000 FWHM.
NOTE : LC/HDMSE est un mode d’acquisition de données indépendantes (DIA) qui opère en recueillant rapidement des numérisations avec en alternance des énergies de collision (dans la cellule de collision) entre une faible consommation d’énergie (6 V pour les scans de canal 1 MS) et une énergie élevée (15 à 40--v rampe à produire les fragmentation des spectres sans sélection de précurseur à MS canal 2).- Entrez les paramètres de MS axés sur la source par défaut dans le logiciel d’acquisition de données : « Mon travail/instrument système, » cliquez sur l’onglet « Vion IMS Qtof » et dans le menu « Outils », entrez ces paramètres : 3,0 kV de tension capillaire, 100 ° C température de la source, source de V 100 compenser, 50 L/h cône de débit de gaz et 40 V tension de cône. Régler la température de désolvatation à 250 ° C, le débit de gaz de désolvatation à 500 L/h et la tension capillaire de référence à 3,0 kV.
- Dans le logiciel d’acquisition de données, cliquez sur « Create/analyse méthode » et choisissez « Générer une acquisition et méthode de traitement. » Tapez le nom de la méthode et naviguez jusqu’au dossier (répertoire) de la méthode et puis cliquez sur « Suivant ».
- Pour « Type d’analyse », choisissez « Peptide carte (IMS) » et dans l’onglet « Instrument système », sélectionnez l’instrument « Vion IMS QTof ». Cliquez sur « Terminer ».
Remarque : La méthode LC/HDMSE nouvellement créée s’ouvre automatiquement dans l’écran « Configuration de la méthode Instrument ». - Dans l’onglet « Paramètres », entrez ces paramètres : 3,0 kV tension capillaire, 100 ° C température de la source, 250 ° C température de désolvatation, 50 L/h débit de gaz cône et débit de gaz de désolvatation 500 L/h.
- Dans les paramètres de tabulation « Expérience », sélectionnez « Haute définition MSE» et choisissez « Analyse méthode runtime. » Dans l’onglet « Paramètres de numérisation », entrez ces paramètres : à faible masse, 100 ; grand-messe, 2 000 ; et scan de temps, Mme 400 dans l’onglet « CE », tapez « 6 V » pour le réglage de basse énergie et choisir une rampe haute énergie de 15 à 40 c. Assurez-vous d’avoir la « Réduction des données Disable » vérifiée.
- Faire infuser une solution de 50 enképhaline de leucine ng/mL (LE) établi à 50 % d’acétonitrile avec 0,1 % FA à un débit de 10 µL/min pour l’étalonnage de la masse au cours de l’acquisition de données.
NOTE : Les données de masse s’acquièrent toutes les 5 min en utilisant le même taux d’acquisition au cours de la même gamme de masse.
- Analyser l’échantillon de 10 nM, PHO-dopés utilisant l’analyse de LC/HDMSE décrit ci-dessus en injecter 10 µL de l’échantillon (la montant chargé sur colonne est 100 fmol pour chaque peptide standard).
4. la configuration de la méthode de Tof-MRM pour optimisation énergétique (CE) collision
- Dans le dataset de LC/HDMSE , obtenir le temps de rétention, le précurseur et le fragment ion m/z pour chaque peptide PHO. Conserver la m/z et l’état de charge pour les plus abondants ion précurseur et les plus abondantes de trois correspondants fragmentent les ions.
Remarque : Un exemple de spectres de basse énergie et haute énergies généralement enregistrées par l’analyse de LC/HDMSE est présenté dans la Figure 2. - Utiliser l’optimisation énergétique (CE) collision lors d’une expérience SRM/MRM pour obtenir le meilleur signal pour chaque peptide23.
- Dans le logiciel d’acquisition de données, cliquez sur « Create/analyse méthode » et choisissez « Générer une acquisition et méthode de traitement. » Tapez le nom de la méthode, naviguez jusqu’au dossier de la méthode (répertoire) et cliquez sur « Suivant ».
- Pour « Type d’analyse », choisissez « Quantifier ». Choisissez « Quantifier Assay Tof 2D chromatographique » et cliquez sur « Suivant ». Dans l’onglet « Instrument système », sélectionnez « Vion IMS QTof » et cliquez sur « Terminer ».
Remarque : La méthode de Tof-MRM nouvellement créée s’ouvre automatiquement dans l’écran « Configuration de la méthode Instrument ». - Dans l’onglet « Paramètres », entrez ces paramètres : 3,0 kV tension capillaire, 100 ° C température de la source, 250 ° C température de désolvatation, 50 L/h débit de gaz cône et débit de gaz de désolvatation 500 L/h.
- Dans l’onglet Paramètres de « Expérience », sélectionnez « Fonction Table » et choisissez « une ou plusieurs MS, HSH ou MRM fonctions. » En utilisant les flèches vers le bas, mis en place une fonction de « Tof-MRM » pour chaque peptide en inscrivant trois « transitions » (combinaisons de précurseur plus abondant et trois de ses ions fragments plus abondantes).
Remarque : En mode MRM-Tof, la sensibilité la plus élevée est obtenue en utilisant une approche planifiée de MRM. - Dans l’éditeur de fonction « Tof-MRM », choisir une fenêtre de temps de rétention pour chaque peptide (au moins 1 min de long), disposées selon l’ordre d’élution de peptide. Dans l’éditeur de fonction « Tof-MRM », insérer le peptide précurseur m/z et le produit de m/z ; Sélectionnez la fenêtre isolation quad comme « Low » (fenêtre 4-Da) pour le précurseur du peptide ; Sélectionnez un temps de recherche fixe de 100 ms ; et choisir douze valeurs d’énergie de collision différent de l’ordre de 14 à 36 V, comme suit : 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34 et 36, comme illustré dans l’exemple de la Figure 3.
Remarque : Avec la méthode de Tof-MRM décrite ci-dessus, points de données au moins 10 par pic chromatographique sont collectées pour chaque transition à chaque EC spécifié. Pour réduire la taille de fichier de données, sélectionnez l’option « Wide » (fenêtre 6-Da) pour le signal de l’ion de fragment. Exemples des valeurs optimisé CE sont indiquées dans le tableau 2. - Analyser l’échantillon de 10 nM, PHO-dopés utilisant le test de Tof-MRM décrit ci-dessus en injecter 10 µL de l’échantillon (la montant chargé sur colonne est 100 fmol pour chaque peptide standard).
5. installation de la dernière méthode d’acquisition de HS-MRM pour la quantification de peptide par séparation mobilité ionique des précurseurs de peptide
- Dans le menu « enquêter » sur le logiciel d’acquisition de données, afficher tous les 11 traces chromatographiques générés pour chaque EC pour chaque peptide et intégrer les pics en utilisant le bouton « Intégrer » dans la barre d’outils « Processing Options/opérations » ; la surface du pic plus haute indiquera la meilleure CE pour chaque transition.
- Comparer les meilleures zones de pointe pour les 3 fragments de chaque peptide visuellement et de conserver uniquement le meilleur de « transition » (l’ion fragment qui produit le signal plus intense) ; Le tableau 2 affiche le meilleur « transitions » obtenus à partir de quatre peptides PHO.
- Configurer une méthode HS-MRM ne contenant qu’un seul fragment ion par peptide. Utilisez les valeurs de CCS de chaque précurseur du peptide du DataSet LC/HDMSE .
- Modifiez la méthode de Tof-MRM créée dans la section précédente (article 4) en sélectionnant la fonction « HS-MRM » au lieu de la fonction « Tof-MRM ».
- Dans l’éditeur de fonction « HS-MRM », entrez les paramètres suivants axés sur le précurseur : m/z, quadripolaire résolution : faible (4 Da), charger État, valeur de CCS précurseur et résolution de CCS précurseur : basse. Pour l’ion produit, entrez sa m/z, l’énergie de collision optimale, choisissez un temps d’acquisition de 0,4 s puis choisissez un paramètre de « gamme » (6 Da) à englober la totalité de ses isotopes. Un exemple de la méthode d’acquisition HS-MRM final pour tous les quatre peptides est présenté dans la Figure 4.
- Analyser tous les échantillons en utilisant la méthode HS-MRM, en commençant par le mAb digest vide (échantillon non enrichis), suivies de 4 injections répétées (10 µL de chaque) des concentrations suivantes : 0,1, 1, 10 et 100 nM PHO peptides.
6. création d’une méthode de traitement pour l’analyse de l’objet dataset HS-MRM
- Créez une méthode de traitement pour l’analyse de l’objet dataset HS-MRM.
- Dans la méthode d’analyse créé pour la méthode d’acquisition de données « HS-MRM », cliquez sur l’onglet « Objet » et choisissez « Gérer des composants. »
- Dans le menu déroulant sous « Type d’expérience », sélectionnez l’option « HS MS/MS/HS-MRM ».
- Cliquez sur « Create/New Entry » et présente les paramètres suivants dans l’éditeur de méthode de traitement : temps de rétention de peptide (RT), état de charge de précurseur, précurseur m/z et le précurseur dérive temps (DT). Pour l’ion fragment, entrez son état m/z et frais et spécifiez la trace « XIC » comme mode d’extraction.
- Dans l’onglet « Objet » de la même méthode d’acquisition « HS-MRM », cliquez sur « par défaut » et entrez les concentrations suivantes : 0,1, 1, 10 et 100 nM PHO peptides.
- Entrez les paramètres suivants dans les « paramètres de traitement / d’Extraction » : la masse tolérance par défaut, 10 ppm (pour m/z de tous les ions fragments) et par défaut bouge fois, 5 % ; un exemple de la méthode de traitement de peptide PHO est illustré à la Figure 5.
- Pour le type de calibrage, choisissez l’ajustement de courbe linéaire avec 1 /2 pondération X.
- Traiter les données de HS-MRM d’intégrer tous les chromatogrammes et de construire les courbes d’étalonnage de chaque peptide PHO.
Résultats
Les séquences individuelles de six phosphorylase b peptide normes contenues dans le mélange de peptides PHO figurent au tableau 1, ainsi que leurs temps de rétention et de leurs précurseurs plus abondants chez HDMSE experiment. La première étape dans le développement d’un test MRM (HS) haute sélectivité est l’acquisition d’un ensemble de données HDMSE d’établir le temps d’élution de chaque peptide PHO, ainsi que la plupart de ses correspondante précurseur abondante et les trois fragments plus abondants. La figure 2 affiche les spectres HDMSE acquis pour un des peptides PHO (Pep 6) dopés dans le digest de l’infliximab. Après avoir établi le 3 meilleur « transitions » (combinaisons des masses précurseur et fragment) pour chaque peptide, une expérience de Tof-MRM est effectuée pour trouver l’énergie de collision optimale afin de maximiser les signaux générés pour chaque peptide. Les résultats de l’expérience d’optimisation CE sont résumés dans le tableau 2. Le test final de HS-MRM (voir Figure 4) conserve seulement les meilleurs « transition » pour chaque peptide et est utilisé pour analyser tous les échantillons fortifiés. Exemples de chromatogrammes HS-MRM générés pour 4 PHO peptides à travers toutes les concentrations étudiées sont présentées dans la Figure 7. Quatre courbes d’étalonnage obtient pour chaque suite de peptide, l’intégration des pics HS-MRM a mis en évidence dans la Figure 7 sont affichés dans la Figure 8. En outre, la pointe zone écart-type relatif, calculé sur la base 4 injections répétées, est résumée en 4 tableaux reportés dans le tableau 3.
| Peptide | Peptide | Maintien en poste | États de charge | ||||
| ID | Séquence | temps (min) | + 1 | + 2 | + 3 | + 4 | |
| PEP 1 | VLYPNDNFFEGK | 19.4 | 1442.6951 | 721.8512 | 481.5699 | 361.4292 | |
| PEP 2 | TCAYTNHTVLPEALER | 16,0 | 1874.9065 | 937.9569 | 625.6404 | 469.4821 | |
| PEP 3 | IGEEYISDLDQLRK | 18,9 | 1678.8646 | 839.9360 | 560.2931 | 420.4716 | |
| PEP 4 | LLSYVDDEAFIR | 21.1 | 1440.7369 | 720.8721 | 480.9172 | 360.9397 | |
| PEP 5 | LITAIGDVVNHDPVVGDR | 19,7 | 1890.0080 | 945.5076 | 630.6742 | 473.2574 | |
| PPE-TSE 6 | VFADYEEYVK | 17,7 | 1262.5939 | 631.8006 | 421.5362 | 316.4039 | |
Tableau 1. Normes de peptide PHO contenus dans le mélange de masse PREP dopé dans le digest de l’infliximab. Temps de rétention de peptide et de leurs précurseurs plus abondants (indiqués en gras) sont affichées sous forme de tableau.
| Peptide | Peptide | Maintien en poste | Précurseur du peptide | Plus abondant fragment ions/charge | Optimum | ||||
| ID | Séquence | temps (min) | m/z & recharge | Temps de dérive (ms) | J’ai | II | III | CE (V) | |
| PEP 2 | TCAYTNHTVLPEALER | 16,0 | 625.6404 (+ 3) | 6.2 | 714.3781 (+ 1) | 807.4177 (+ 2) | 827.4621 (+ 1) | 24 | |
| PEP 4 | LLSYVDDEAFIR | 21.1 | 720.8721 (+ 2) | 7.5 | 865.4050 (+ 1) | 964.4734 (+ 1) | 1214.5688 (+ 1) | 22 | |
| PEP 5 | LITAIGDVVNHDPVVGDR | 19,7 | 630.6742 (+ 3) | 6.3 | 642.3570 (+ 1) | 689.8391 (+ 2) | 832.4236 (+ 2) | 20 | |
| PPE-TSE 6 | VFADYEEYVK | 17,7 | 631.8006 (+ 2) | 7.0 | 830.3931 (+ 1) | 945.4200 (+ 2) | 1016.4571 (+ 1) | 24 | |
Le tableau 2. Résultats de l’expérience d’optimisation de Tof-MRM : trois fragments plus abondants de chaque peptide PHO quantifié dans cette étude sont indiqués, ainsi que l’énergie de collision optimisés correspondant.
| Conc | Montant | Des pics de PEP 2 (tableau 3 a) | |||||
| (nM) | sur colonne (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Moyenne | RSD (%) |
| 0,1 | 1 | 490 | 439 | 462 | 431 | 456 | 5.8 |
| 1 | 10 | 5121 | 4842 | 5198 | 4842 | 5001 | 3.7 |
| 10 | 100 | 63853 | 64279 | 66111 | 62509 | 64188 | 2.3 |
| 100 | 1000 | 612392 | 605553 | 613229 | 611004 | 610545 | 0,6 |
| Conc | Montant | Des pics de PEP 4 (tableau 3 b) | |||||
| (nM) | sur colonne (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Moyenne | RSD (%) |
| 0,1 | 1 | 275 | 359 | 325 | 288 | 312 | 12.2 |
| 1 | 10 | 3559 | 3694 | 3287 | 3754 | 3574 | 5.8 |
| 10 | 100 | 45259 | 45775 | 42976 | 45548 | 44890 | 2.9 |
| 100 | 1000 | 459374 | 467927 | 436272 | 458994 | 455642 | 3.0 |
| Conc | Montant | Des pics de PEP 5 (tableau 3) | |||||
| (nM) | sur colonne (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Moyenne | RSD (%) |
| 0,1 | 1 | 3194 | 3243 | 3202 | 3257 | 3224 | 1.0 |
| 1 | 10 | 31464 | 31150 | 31464 | 31433 | 31378 | 0,5 |
| 10 | 100 | 313638 | 320712 | 311943 | 311943 | 314559 | 1.3 |
| 100 | 1000 | 2845736 | 2840031 | 2882006 | 2864052 | 2857956 | 0,7 |
| Conc | Montant | Des pics de PEP 6 (tableau 3D) | |||||
| (nM) | sur colonne (fmoles) | Rep01 | Rep02 | Rep03 | Rep04 | Moyenne | RSD (%) |
| 0,1 | 1 | 490 | 583 | 440 | 440 | 488 | 13,8 |
| 1 | 10 | 6429 | 6429 | 6848 | 6623 | 6582 | 3.0 |
| 10 | 100 | 71295 | 70400 | 71563 | 70400 | 70915 | 0,9 |
| 100 | 1000 | 707640 | 707640 | 694461 | 729490 | 709808 | 2.0 |
Tableau 3. Tableau contenant la surface des pics des chromatogrammes HS-MRM enregistré pour 4 peptides PHO (Pep 2, 4, 5 et 6) pour chaque injection de LC/MS (16 pistes de LC/MS et 4 concentrations testées).
L’écart type relatif était supérieure à 15 % pour tous les peptides sur la plage de concentration ensemble étudiée.

Figure 1. Diagramme de flux de travail résumant les trois étapes nécessaires pour mettre en place une méthode d’acquisition de HS-MRM. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
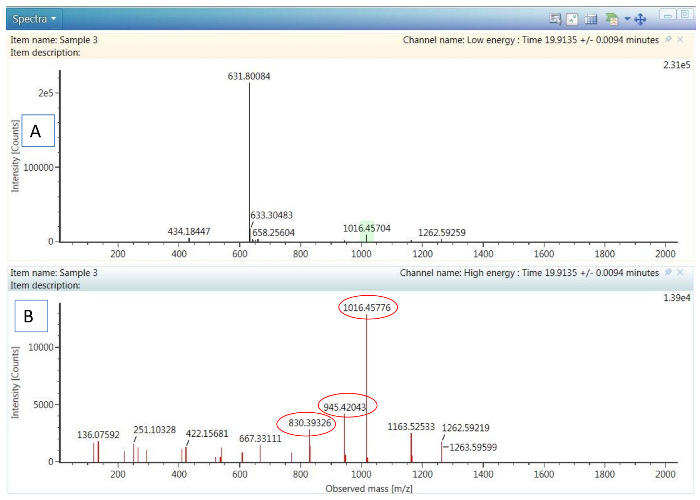
La figure 2. Exemple de données HDMSE :
(A) spectre de basse énergie montrant l’ion précurseur de Pep 6. (B) spectre de fragmentation à haute énergie du même peptide, affichant haut 3 ions les plus abondants fragment (encerclées) sélectionnées pour l’optimisation de l’énergie de collision Tof-MRM. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 3. Paramètres utilisés pour la mise en place d’une optimisation de Tof-MRM experiment.
Pour chaque transition, onze énergies de collision (de l’ordre de 16 à 36 V) ont été testés. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
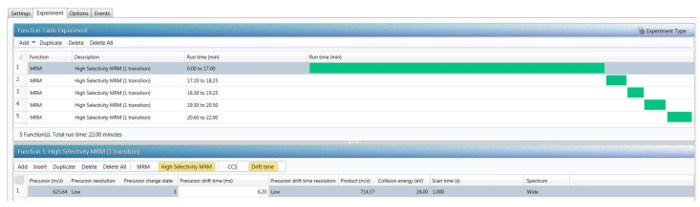
La figure 4. Exemple de la méthode de HS-MRM finale.
Plusieurs paramètres sont requis pour chaque « transition », y compris le peptide précurseur m/z, son état de charge et temps de dérive de mobilité ionique, le m/z de l’ion fragment plus abondante, l’énergie de collision optimale et le temps d’acquisition MS. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
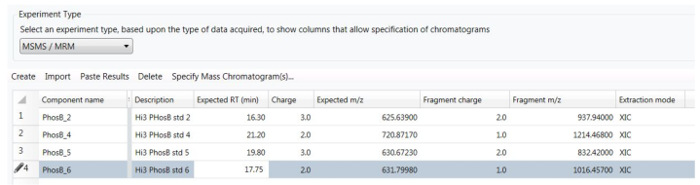
La figure 5. Paramètres utilisés par la méthode de traitement pour l’analyse de l’objet dataset HS-MRM.
Chaque peptide est surveillé par une seule « transition » décrite par le peptide précurseur m/z, état de charge et prévu des temps de rétention, au long avec le m/z le fragment plus abondant et son état de charge. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 6. Diagramme de la masse spectromètre à mobilité ionique.
Dans le mode d’acquisition de HS-MRM, les précurseurs du peptide qui est étant quantifiée sont séparés des autres précurseurs co élution de peptide (interférence) dans la cellule de mobilité ionique, isolée par le quadripôle et fragmentée et une énergie de collision fixe dans le cellule de collision. Le signal produit par les ions de fragments de peptide est renforcé en réglant la fréquence de pousseur et quantification de peptide est effectuée en utilisant le MS haute-résolution (> 30 000) signaux produits par l’ion fragment plus intense de chaque peptide. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

La figure 7. Les chromatogrammes de HS-MRM enregistrement pour 4 peptides PHO à 4 différentes concentrations s’étendant sur 3 ordres de grandeur (0,1, 1, 10 et 100 nM).
(A) chromatogrammes de pep 2, 4 (B) Pep chromatogrammes, chromatogrammes Pep (C) 5 et chromatogrammes Pep (D) 6. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
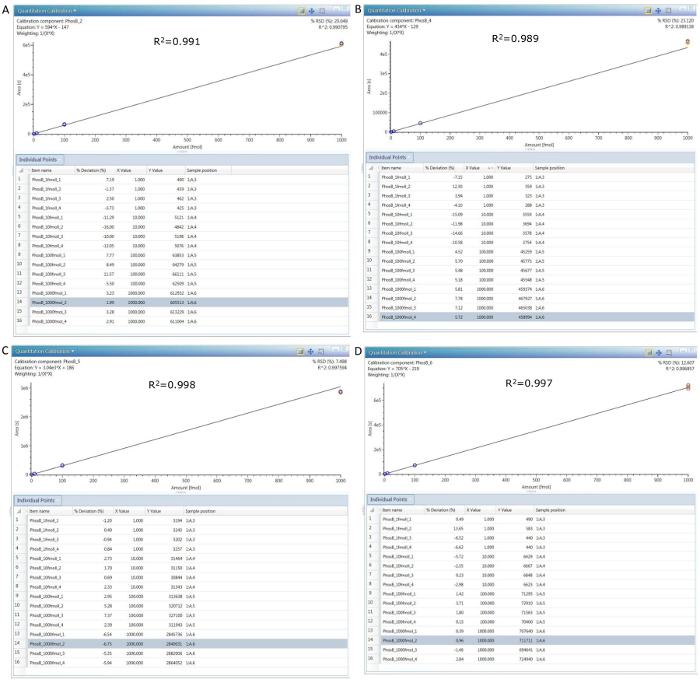
La figure 8. Courbes d’étalonnage pour 4 peptides PHO à travers 4 différentes concentrations (0,1, 1, 10 et 100 nM).
Les tableaux sous chaque courbe affichent la surface des pics individuels (valeurs Y) enregistrées pour chaque injection, tandis que la seconde colonne de chaque table montre l’écart en pourcentage de la réponse linéaire attendue. (A) pep 2 étalonnage, Pep (B) 4 étalonnage, Pep (C) 5 calibration et étalonnage de Pep (D) 6. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Haute résolution (Rs > 20 000) la spectrométrie de masse est couramment utilisée pour la caractérisation de protéines thérapeutiques sur une variété de plates-formes de l’instrument. En revanche, quantification basée sur MS protéine est généralement effectuée par SRM/MRM sur basse résolution (~ 1 000 Rs) tandem quadrupolaire spectromètres de masse à l’aide de peptides signature générées par le clivage enzymatique des protéines11,12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23. comme unidimensionnel séparations chromatographiques ne peut pas résoudre entièrement les mélanges complexes peptidiques produites par la digestion enzymatique, élution co peptide est un phénomène courant, même dans le cas d’un "Digest" single-protéine. Pour les digestions très complexe des protéines (par exemple, pour la quantification des ppms 1-100 de professionnels de la santé en présence d’un fond riche en peptide produite par la protéine thérapeutique), la sensibilité, précision ou de linéarité de la SRM/MRM dosage peut être affecté par interférence.
Les tests de la SRM/MRM ont une sélectivité unidimensionnelle, en se fondant uniquement sur une combinaison « unique » des masses précurseur/fragment. Pour cette raison, ces tests échouent dans des situations lorsque l’arrière-plan de peptide change de façon inattendue (par exemple, pour les échantillons de biopharmaceutique obtenu à partir des procédures de purification différent). Pour surmonter ces limites, nous vous proposons ici un test de haute sélectivité (HS) MRM mis en œuvre sur un ion mobilité compatible haute résolution temps de vol (QTOF) hybride spectromètre de masse quadripolaire (pour le diagramme de l’instrument, voir Figure 6).
L’instrument sépare les précurseurs du peptide d’intérêt d’autres précurseurs co élution de peptide (interférence) dans la cellule de mobilité ionique, isole l’enveloppe complète isotopique du précurseur dans le quadripôle et fragments d’elle avec un CE fixe dans le cellule de collision. Le signal produit par son fragment peptidique plus abondante est rehaussée en réglant la fréquence de poussoir (Target Enhancement), qui pousse sélectivement massives régions d’intérêt dans le tube de vol, plutôt que tous les ions, comme pour une analyse complète. Quantification de peptide est effectuée en utilisant les signaux de haute-résolution MS (~ 30 000 Rs) produites par cet ion de fragment. En comparaison avec les essais SRM/MRM, l’essai du HS-MRM propose deux niveaux de sélectivité supplémentaires : l’un est fourni par la séparation de mobilité ionique au niveau du précurseur, tandis que la seconde est offerte par la résolution de masse accrue de l’analyseur de TOF. Les résultats émanant de ces améliorations de sélectivité sont visibles dans les chromatogrammes de HS-MRM affichés à la Figure 7, qui sont libres d’interférences entre les trois ordres de grandeur.
À la différence des tests SRM/MRM, il y a plusieurs paramètres qui peuvent être ajustés afin d’optimiser les dosages HS-MRM : la fenêtre RT autour le précurseur du peptide (généralement fixée à 0,2 min), la fenêtre d’isolement quadripolaire (4 Da), la fenêtre de temps dérive entourant la précurseur (± FWHM du pic du précurseur de la mobilogram ion correspondant) et la résolution de MS de l’ion de fragment (20 000-40 000). Les dosages de HS-MRM sont très sensibles : le plus faible montant détecté pour chaque peptides PHO est 1 femtomole sur colonne (ou 0,1 nM en termes de concentration de peptide). Si l'on considère le peptide MW (voir tableau 1 pour le SMCLO précis), la quantité détectée sur colonne est l’ordre de 1-2 pg, tandis que la colonne est dotée d’un montant beaucoup plus élevé (2 µg) de peptides d’arrière-plan de la mAb digest.
En considérant le poids moléculaire de la protéine pleine longueur de PHO (97,2 kDa) dont sont dérivés les peptides enrichis, le test est capable de détecter des 50 ppm d’une impureté de protéines en présence d’ions de fond haute-abondance. Limite inférieure de détection (5-10 ppm) est réalisables pour un poids moléculaire inférieur PSS (10-20 kDa). L’analyse couvre les trois ordres de grandeur (comme indiqué dans les courbes d’étalonnage de la Figure 8), ce qui signifie qu’il peut mesurer des professionnels de la santé dans la gamme de 1 à 1 000 ppm. En outre, la reproductibilité des essais HS-MRM, illustré dans le tableau 3, correspond très bien avec la reproductibilité des essais de SRM/MRM de petites molécules, avec surface de pic RSDs supérieures à 15 %.
Nous avons exploré les capacités d’un nouveau dosage pour la quantification des normes de peptide enrichis dans un condensé de l’anticorps monoclonal (ACM) et démontré sa sensibilité et son utilité pour couvrir la plage dynamique étendue (au moins trois ordres de grandeur) généralement rencontrées dans l’analyse HCP. Tous les six étalons de peptide ont été détectés à des concentrations aussi faibles que 0,1 nM (1 femtomole chargées sur une colonne chromatographique de 2,1 mm ID) en présence d’un fond de peptide haute-abondance (2 µg d’une mAb digest chargé sur-colonne). En intégrant tant SGRH ciblé et ion mobilité précurseur séparation, l’essai du HS-MRM a un grand potentiel pour devenir un test de suivi rapide et à haut débit pour plusieurs professionnels de la santé à travers plusieurs lots de produits biopharmaceutiques.
Déclarations de divulgation
Tous les auteurs sont des employés de la société des eaux, qui est le producteur de plusieurs réactifs et instruments utilisés dans cet article.
Remerciements
Les auteurs tiens à remercier Lesley Malouin et Tony Catlin de Waters Corporation pour la préparation de la Figure 6 du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| Vion IMS Qtof mass spectrometer | Waters | 186009214 | |
| Acquity H-Class Quaternary solvent manager (QSM) | Waters | 186015041 | |
| Acquity H-Class FTN Sample Manager (SM) | Waters | 186015040 | |
| Acquity H-Class Column Manager (CM) | Waters | 186015043 | |
| Acetonitrile (ACN) | Fisher Chemical | A996-4 | |
| Ammonium bicarbonate | Sigma Aldrich | 40867-50G-F | |
| 2.1 x 150 mm CSH C18 UPLC column, 1.8 µm particles | Waters | 186005298 | |
| Dithiothreitol (DTT) | Sigma Aldrich | D5545-5G | |
| Formic acid, eluent additive for LC/MS | Sigma Aldrich | 56302-10X1ML-F | |
| Hi3 rabbit phosphorylase (PHO) MassPREP standard | Waters | 186006011 | |
| Iodoacetamide (IAM) | Sigma Aldrich | I-1149-5G | |
| LC vials (12x32 mm glass vials, screw neck) | Waters | 186000327c | |
| Leucine Enkephalin acetate salt hydrate | Sigma Aldrich | L9133-10MG | |
| Protein LoBind 2.0 mL tubes (2x50) | Eppendorf | 22431102 | |
| RapiGest SF | Waters | 186001861 | |
| Trypsin/Lys-C enzyme mix, mass spec grade (5 x 20 µg) | Promega | V5073 | |
| Water, LC/MS grade | Sigma Aldrich | 39253-1L-R |
Références
- Doneanu, C. E., et al. Analysis of host-cell proteins in biotherapeutic proteins by comprehensive online two-dimensional liquid chromatography/mass spectrometry. MAbs. 4, 24-44 (2012).
- Schenauer, M. R., Flynn, G. C., Goetze, A. M. Identification of host-cell protein impurities in biotherapeutics using mass spectrometry. Anal Biochem. 428, 150-157 (2012).
- Zhang, Q., et al. Comprehensive tracking of host-cell proteins during monoclonal antibody purifications using mass spectrometry. MAbs. 6, 659-670 (2014).
- Farrell, A., et al. Quantitative host-cell protein analysis using two-dimensional data independent LC-MS(E). Anal Chem. 87, 9186-9193 (2015).
- Doneanu, C. E., et al. Enhanced detection of low-abundance host cell protein impurities in high-purity monoclonal antibodies down to 1 ppm using ion mobility mass spectrometry coupled with multidimensional liquid chromatography. Anal Chem. 87, 10283-10291 (2015).
- Doneanu, C. E., et al. Improved identification and quantification of host cell proteins (HCPs) in biotherapeutics using liquid chromatography mass spectrometry. Technologies for therapeutic monoclonal antibody characterization, vol. 3: Defining the next generation of analytical and biophysical techniques. , Washington DC. ACS Symposium series 1202 357-393 (2015).
- Zhang, Q., et al. Characterization of the co-elution of host-cell proteins with monoclonal antibodies during protein A purification. Biotechnol Prog. 32, 708-717 (2016).
- Reisinger, V., Toll, H., Mayer, R. E., Visser, J., Wolschin, F. A mass spectrometry based approach to host cell protein identification and its application in a comparatibility exercise. Anal Biochem. , 1-6 (2014).
- Madsen, J. A., et al. Toward the complete characterization of host-cell proteins in biotherapeutics via affinity depletions, LC/MS/MS and multivariate analysis. MAbs. 7, 1128-1137 (2015).
- Giles, K., et al. Applications of a trevelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18, 2401-2414 (2004).
- Anderson, V., Hunter, C. L. Quantitative mass spectrometric multiple reaction monitoring assays for major plasma proteins. Mol Cell Proteomics. 5, 573-588 (2006).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Mol Syst Biol. 4, 1-14 (2008).
- Picotti, P., et al. High-throughput generation of selected reaction-monitoring assays for proteins and proteomes. Nat Methods. 7, 43-46 (2010).
- Zhao, L., et al. Quantification of proteins using peptide immunoaffinity enrichment coupled with mass spectrometry. J Vis Exp. (53), (2011).
- Liebler, D. C., Zimmerman, L. J. Targeted quantitation of proteins by mass spectrometry. Biochemistry. 52, 3797-3806 (2013).
- Manes, N. P., Mann, J. M., Nita-Lazar, A. Selected reaction monitoring mass spectrometry for absolute protein quantification. J Vis Exp. (102), (2015).
- Ebhardt, H. A., Sabido, E., Huttenhain, R., Collins, B., Aebersold, R. Range of protein detection by selected/multiple reaction monitoring mass spectrometry in an unfractioned human cell culture lysate. Proteomics. 12, 1185-1193 (2012).
- Picotti, P., Bodenmiller, B., Mueller, L. N., Domon, B., Aebersold, R. Full dynamic range proteome analysis of S. cerevisiae by targetted proteomics. Cell. 138, 795-806 (2009).
- Sherman, J., McKay, M. J., Ashman, K., Molloy, M. P. How specific is my SRM: the issue of precursor and product ion reductancy. Proteomics. 9, 1120-1123 (2009).
- Abbatiello, S. E., Mani, D. R., Keshishian, H., Carr, S. A. Automated detection of innacurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem. 56, 291-305 (2010).
- Rost, H., Malmstrom, L., Aebersold, R. A computational tool to detect and avoid redundancy in selected reaction monitoring. Mol Cell Proteomics. 11, 540-549 (2012).
- Bao, Y., et al. Detection and correction of interference in SRM analysis. Methods. 61, 299-303 (2013).
- Maclean, B., et al. Effect of collision energy optimization on the measurement of peptides by selected reaction monitoring (SRM) mass spectrometry. Anal Chem. 82, 10116-10124 (2010).
- Mbasu, R. J., et al. Advances in quadrupole and time-of-flight mass spectrometry for peptide MRM based translational reserch analysis. Proteomics. 16, 2206-2220 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon