Method Article
Caracterização de complexos multi-subunidade de proteína de humano utilizando MxA não desnaturante em gel de poliacrilamida-electroforese
Neste Artigo
Resumo
This article describes a simple and rapid protocol to evaluate the oligomeric state of the dynamin-like GTPase MxA protein from lysates of human cells using a combination of non-denaturing PAGE with western blot analysis.
Resumo
The formation of oligomeric complexes is a crucial prerequisite for the proper structure and function of many proteins. The interferon-induced antiviral effector protein MxA exerts a broad antiviral activity against many viruses. MxA is a dynamin-like GTPase and has the capacity to form oligomeric structures of higher order. However, whether oligomerization of MxA is required for its antiviral activity is an issue of debate. We describe here a simple protocol to assess the oligomeric state of endogenously or ectopically expressed MxA in the cytoplasmic fraction of human cell lines by non-denaturing polyacrylamide gel electrophoresis (PAGE) in combination with Western blot analysis. A critical step of the protocol is the choice of detergents to prevent aggregation and/or precipitation of proteins particularly associated with cellular membranes such as MxA, without interfering with its enzymatic activity. Another crucial aspect of the protocol is the irreversible protection of the free thiol groups of cysteine residues by iodoacetamide to prevent artificial interactions of the protein. This protocol is suitable for a simple assessment of the oligomeric state of MxA and furthermore allows a direct correlation of the antiviral activity of MxA interface mutants with their respective oligomeric states.
Introdução
A estrutura quaternária de uma proteína desempenha um papel crucial em muitos processos celulares. Vias de sinalização, a expressão do gene e enzima de activação / desactivação todos contam com a montagem adequada de complexos de proteínas 1-4. Este processo também conhecido como homo- ou hetero-oligomerização é devido à covalente irreversível ou interacções proteína-proteína electrostáticas e hidrofóbicas reversíveis. A oligomerização não só diversifica os diferentes processos celulares sem aumentar o tamanho do genoma, mas também fornece uma estratégia para construir proteínas de complexos estáveis que são mais resistentes à desnaturação e degradação no sentido 5. Defeitos na oligomerização ter um impacto sobre a função de proteínas e pode levar ao desenvolvimento de doenças. Por exemplo, a enzima fenilalanina hidroxilase forma um complexo tetramérico. Algumas mutações dentro do complexo de proteína pode enfraquecer a formação tetrâmero e conduzir à doença fenilcetonúria 6.
A proteína MxA humano é um interferão (IFN) induzida por proteína efectora antiviral exercendo uma ampla actividade antiviral contra diversos ARN, bem como vírus de ADN 7. Ela pertence à superfamília de grandes GTPases dinamina semelhante e tem a capacidade para formar grandes estruturas oligoméricas 8 in vitro. A oligomerização tem sido sugerido para proteger da degradação rápida MxA 9,10. Apesar dos intensos esforços por vários grupos de pesquisa, o mecanismo molecular de ação permanece difícil e o papel do estado de oligomerização de MxA para a sua função antiviral está em debate 9,11,12. A este respeito, Gao e colegas propuseram um modelo onde MxA exerce a sua actividade antiviral, interagindo com nucleoproteínas virais em forma de grandes estruturas oligoméricas em forma de anel 11. No entanto, mais recentemente, demonstrou-se que os dímeros MXA exibem actividade antiviral e interagir com a nucleoproteína do vírus influenza A 12. Bduziu sobre a estrutura de cristal de MxA, Gao e colaboradores identificaram vários resíduos de aminoácidos nas regiões de interface que são críticos para a sua oligomerização in vitro e a sua função antiviral 11,13. Portanto, a fim de elucidar qual oligomérica estado de MxA exerce atividade antiviral, procurou-se estabelecer um protocolo simples para determinar rapidamente o estado oligmeric de mutantes de interface MXA expressos em células humanas, bem como endógena MxA expressa após a estimulação IFN.
Embora existam muitas técnicas que são comumente usados para investigar a interação entre proteínas, tais como a proteína split-Verde Fluorescente (split-GFP) ensaio de complementação 14, ressonância de plasma de superfície 15 e Förster transferência de energia de ressonância (FRET) 16, eles não fornecem informações da estequiometria exacta de um complexo de proteína oligomérica. Para a investigação deste aspecto particular, tal como técnicasmulti-ângulo de espalhamento de luz (MALS) 17 e 18 de ultracentrifugação analítica são muito úteis. Geralmente, as proteínas analisadas usando estes métodos são proteínas purificadas. processos de oligomerização pode também depender de outros fatores celulares. Se estes factores são desconhecidas, a análise é mais difícil. Além disso, algumas proteínas são difíceis de expressar em E. coli e de purificar. Por conseguinte, estes métodos não são a escolha óptima para analisar a oligomerização de proteínas no meio celular. Além disso, essas técnicas exigem instrumentos dispendiosos que não estão facilmente disponíveis.
Não electroforese em gel desnaturante de poliacrilamida (PAGE), cromatografia de exclusão por tamanho ou por reticulação química convencional seguido de dodecil sulfato de sódio (SDS) -Página são ferramentas úteis para a caracterização da formação de oligómeros a partir de lisados de células 2,19,20. Estes métodos não requerem equipamento especializado e podem ser facilmente PErformed num laboratório padrão. Nós inicialmente avaliados vários protocolos químicos de reticulação que invariavelmente levaram a agregação e precipitação de MxA não específica. Portanto, o próximo testados protocolos PRINCIPAL não desnaturante. Como não-PAGE desnaturante exclui a utilização de SDS, a migração das proteínas depende da sua carga nativa. PAGE nativa utiliza-Azul de Coomassie G250 azul brilhante para carregar proteínas com uma carga global negativa, semelhante a SDS, mas não desnaturar a proteína de 21. Infelizmente, precipitados coomassie azul brilhante na presença de altas sais e catiões divalentes (por exemplo, Mg 2+) que são muitas vezes incluídos em tampões de lise. Dependendo dos tampões utilizados, pode ser difícil de analisar a amostra sem mais passos de optimização que podem ter um efeito sobre o complexo de proteína oligomérica.
Aqui é apresentado um protocolo simples baseado num método anteriormente publicado em 22 para determinar de oligomerizaçãoproteína MxA humana derivada a partir de lisados celulares usando não desnaturante PAGE.
Protocolo
NOTA: Este protocolo é baseado no não desnaturante protocolo de PAGE publicado anteriormente 12. Neste estudo, o estado oligomérico da proteína MxA foi avaliada utilizando quer de células Vero ou de células que sobre-expressam MxA A549 IFN-ct-estimuladas expressam MxA endógena. O protocolo abaixo descrito pode ser utilizado para analisar o estado oligomérico da proteína em qualquer além MxA. No entanto, uma maior optimização pode ser necessária.
1. Preparação de lisado celular para PAGE não desnaturante
NOTA: Para analisar o estado oligomérico da proteína MxA humana tanto em células Vero ou A549, 1,0 x 10 6 células foram colhidas. Dependendo do tipo de célula ou a abundância da proteína a analisar, o número de células deve ser ajustado. É também importante para proteger o tampão de lise da exposição à luz, logo que a iodoacetamida sensível à luz é adicionado.
- Semente de 0,3 x 10 6 ou Vero A549 células por poço em6 bem-pratos. Manter as células em 2 ml de meio de crescimento por poço (ver Tabela 1). Incubar as células durante a noite numa incubadora de cultura de células (37 ° C, 5% CO 2).
- Colher as células por lavagem com 1 ml de fosfato salino tamponado (PBS) e separar por adição de 0,5 ml de 0,25% de tripsina-ácido etilenodiaminotetracético (EDTA) 1 x solução durante aproximadamente 5 min à temperatura ambiente.
- Assim que as células destacar do prato, adicionar 0,5 ml de meio de crescimento e mistura-se cuidadosamente, pipetando para cima e para baixo.
- Transferir as células de cada poço para um tubo de 2 mL e o pellet-los usando uma centrifugadora de bancada (5000 x g, 4 ° C, 5 min).
- remover cuidadosamente o sobrenadante por pipetagem sem perturbar o sedimento celular.
- Lavam-se as células com PBS, 1 ml de gelo-frio, pipetando cuidadosamente a suspensão de células para cima e para baixo.
- células de pelotas em uma centrífuga de bancada (5.000 x g, 4 ° C, 5 min).
- Remova cuidadosamente o sobrenadante por pipetagem without separar o sedimento celular.
- Ressuspender as células em 200 de tampão gelado lise ul (ver Tabela 1) por pipetagem cima e para baixo e colocar no gelo.
- Imediatamente, proteger lisado proveniente de luz, cobrindo os tubos utilizando folha de alumínio e incubar durante 30 min em gelo.
NOTA: Após a incubação durante 30 min em gelo, já não é essencial para proteger o lisado proveniente de exposição à luz, uma vez que a protecção dos grupos tiol livres é irreversível. - Remover os detritos celulares por centrifugação numa centrifugadora de bancada de pré-arrefecida (13000 xg, 4 ° C, 20 min).
- Equilibrar colunas de diálise em tampão de diálise (Tabela 1) na câmara fria a 4 ° C durante 20 min durante o passo de centrifugação. Utilizar uma coluna com um peso molecular de corte de 10.000.
- Fixe as colunas a uma bóia flutuante e colocá-los num copo cheio com tampão de diálise. Para assegurar agitação suave, usar um agitador magnético. Não toque na membrana.
NOTA: Dialysé colunas podem ser comprados ou preparados a partir de tubos de 1,5 ml de acordo com o protocolo descrito por Fiala e colegas de trabalho 19.
- Fixe as colunas a uma bóia flutuante e colocá-los num copo cheio com tampão de diálise. Para assegurar agitação suave, usar um agitador magnético. Não toque na membrana.
- Remover as colunas do tampão de diálise e a bóia flutuante. Transfira os lisados límpidos para a coluna de diálise preparados por pipetagem sem tocar na membrana. Fixe as colunas a uma bóia flutuante e colocá-los de volta para o copo cheio com tampão de diálise.
- Dialisar o lisado num copo contendo tampão de diálise gelada (Tabela 1) durante pelo menos 4 horas (durante a noite ou, de preferência), a 4 ° C enquanto agitando cuidadosamente utilizando um agitador magnético. Utilize pelo menos 100 ml de tampão de diálise por um ligado de 200 mL.
- Transfira a amostra de dialisado para um tubo de 1,5 ml. Remover os precipitados por centrifugação numa centrífuga de bancada (13000 xg, 4 ° C, 20 min). Para evitar que a dissociação da proteína oligomérica complexos continuar com o protocolo (seção 2) imediatamente após a diálise. Não congeleos lisados preparados.
2. Eletroforese
NOTA: A electroforese foi realizada conforme descrito antes, com algumas modificações 22. No protocolo descrito abaixo, foram utilizados geles de gradiente pré-fundidos (4-15% gradiente). Alternativamente, os géis podem ser preparados em laboratório. É muito importante excluir qualquer agente desnaturante, como SDS para evitar a dissociação dos complexos de proteínas oligoméricas. Tempo de electroforese foi optimizada para diferentes estados oligoméricos da proteína MxA humano. No entanto, pode variar para outras proteínas, dependendo do tamanho do complexo oligomérico, bem como o intervalo de separação que é suposto ser alcançado para analisar o complexo. Portanto, o tempo óptimo de electroforese deve ser determinada empiricamente. Para melhor resolução dos oligómeros a ser analisado a corrente não deve ser superior a 25 mA.
- Montar o gel PAGE não-desnaturante em gel da câmara. Encha tele câmara interior e exterior com tampão de operação de pré-arrefecida (Tabela 1).
- Pré-correr o gel com o tampão de corrida previamente arrefecido a 25 mA por gel durante 15 minutos numa câmara fria a 4 ° C.
- Misturar 15 ul dos lisados preparados acima com 5 ul de tampão de amostra 4x (Tabela 1). Não ferva a amostra.
- Carregar 15 ul de amostra e um padrão de proteína nativa de escolha no gel. Submeter o gel a 25 mA durante 4 horas na câmara fria a 4 ° C.
NOTA: Para a análise semi-quantitativa de um protocolo de quantificação de proteínas (por exemplo, um ensaio de proteína de Bradford 23) pode ser realizada a fim de garantir um carregamento de quantidades iguais de proteína total por pista.
3. Western Blot
NOTA: descrito abaixo é o protocolo de um sistema de transferência de western molhado. Qualquer membrana de mata-borrão pode ser usado. Ative fluoreto de polivinilideno (PVDF) membranas em 100% de metanol antes de equilíbrio no mata-borrão buffer. A técnica de Western blot semi-seco pode ser usado como alternativa, mas tem que ser optimizado para grandes complexos oligoméricos.
- Desmontar o gel e transferi-la cuidadosamente em tampão de SDS (Tabela 1).
- Incubar durante 10 minutos à temperatura ambiente enquanto agitando suavemente.
- Prepare 2 esponjas, 4 folhas de papel de filtro de celulose e uma membrana absorvente por gel. Mergulhe-os em tampão de transferência (Tabela 1).
- Montar o sanduíche como se segue (de baixo para cima): uma esponja, duas folhas de papel de filtro de celulose, membranas, gel, duas folhas de papel de filtro de celulose, uma esponja.
- Coloque o sanduíche para o tanque blotting. Certifique-se de que a membrana enfrenta o pólo positivo, enquanto o gel enfrenta o pólo negativo.
- Encha o tanque mata-borrão com tampão de blotting pré-refrigerados.
- Seque a 90 mA durante a noite a 4 ° C para obter os melhores resultados de transferência de proteínas.
- Desmontar a sanduíche e visualizar o padrão de proteína, por incubação da membrana em Poncsolução eau S durante 5 min à temperatura ambiente.
- Destain a membrana com cuidado lavando fora do Ponceau S com água deionizada até que você pode ver claramente as bandas do padrão de proteína.
- Marcar as bandas do padrão de proteína, usando uma caneta.
NOTA: residual Ponceau S pode interferir com a imunocoloração. Para evitar isso, a membrana pode ser descorada adicional por incubação em NaOH 0,1 M durante 1 min e subsequente lavagem com água desionizada. - Bloquear a membrana com tampão de bloqueio (ver Tabela 1) durante pelo menos 1 h à temperatura ambiente ou durante a noite a 4 ° C.
- Visualizar proteína (s) de interesse por imunocoloração utilizando anticorpos dirigidos contra a proteína a ser analisada.
NOTA: A proteína humana MxA foi visualizada usando o anticorpo policlonal de coelho específico para Mx1 humano diluído 1: 1000 em tampão de bloqueio (Tabela 1). A solução de anticorpo foi incubada durante a noite a 4 ° C. Alternativamente, o anticorpo monoclonal umnti-MxA anticorpo (clone 143) pode ser utilizada (dados não mostrados) 24.
Resultados
Usando não-PAGE desnaturante, analisou-se o estado oligomérico do tipo selvagem humana MxA, os mutantes diméricas de interface MxA (R640A) e MxA (L617D), bem como o mutante de interface monomérica MxA (M527D) a partir de lisados celulares 12. As células foram lisadas num tampão contendo 1% de octilfenoxipolietoxietanol (NP-40) e iodoacetamida para assegurar a solubilização da proteína e a protecção dos grupos tiol livres (ver Figura 1). Conforme descrito antes, sal e pequenos metabólitos foram removidos por diálise 19. separação das proteínas foi realizada por PAGE não desnaturante. Para facilitar a eficiente transferência de Western, o gel foi incubado em tampão de SDS antes blotting. As proteínas foram visualizadas por MXA imunocoloração utilizando um anticorpo policlonal de coelho dirigido contra MxA. O fluxo de trabalho é descrito na Figura 2.
Para comparar o estado oligomérico da proteína MxA humana endógena a partir de IFN-α ; As células A549 estimuladas, transfectadas que as células Vero (que faltam MxA endógeno) com tipo selvagem recombinante, variantes MXA monomérica e dimérica. Estes tipo selvagem recombinante, monoméricos e diméricos MxA variantes formado tetrâmeros estáveis, monómeros e dímeros, respectivamente, quando comparada com um marcador de proteína nativa não coradas (Figura 3A). Portanto, utilizou-se estas proteínas recombinantes para avaliar o estado oligomérico da proteína MxA humana endógena derivada do IFN-α estimuladas células A549. A Figura 3B mostra que o tamanho de MxA em lisados de células A549 estimuladas IFN-ct-corresponde a um tetrâmero.
Tomados em conjunto, nós descrevemos um método para determinar o estado oligomérico da proteína MxA humano a partir de lisados de células. A nossa abordagem PAGE não desnaturante pode também ser utilizada para avaliar o estado oligomérico de outros complexos de proteínas oligoméricas.
83 / 54683fig1.jpg "/>
Figura 1:. Estrutura esquema de reacção e de iodoacetamida iodoacetamida irreversivelmente protege o grupo tiol de cisteínas livres através da formação de uma ligação tioéter. Esta modificação estáveis resulta da substituição nucleofílica do iodo com o átomo de enxofre da cisteína. Por favor clique aqui para ver uma versão maior desta figura.
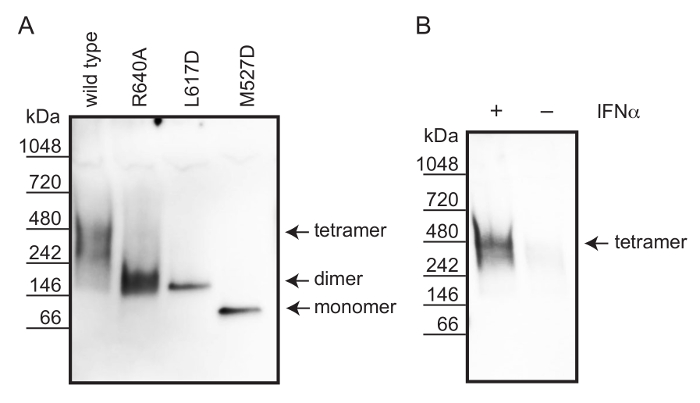
Figura 2:. Diagrama de fluxo de trabalho de PAGE não-desnaturante Uma representação sistemática da abordagem PAGE não desnaturante de lisados celulares. Durante a lise de células, detergentes solubilizar as proteínas e os grupos tiol estão protegidos por iodoacetamida para evitar a agregação de proteína. A diálise remove pequenos metabolitos e sais que podem interferir com os não-PAGE desnaturante 19. A separação complexo é realizada sob condições não desnaturantes. A detecção dos complexos oligoméricas é conseguido através de Western blot seguida de imunocoloração. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3: Determinação do estado oligomérico da proteína MxA humano utilizando PAGE não desnaturante e Western blotting (A) recombinante MxA variantes ectopicamente expressa em células Vero.. Os complexos de tipo selvagem MxA (tetrâmero) mutantes de interface MxA (R640A), MxA (L617D) (dímeros) e MxA (M527D) migrar os seus pesos moleculares esperados, confirmando o seu estado oligomérico. (B) As células A549 foram estimuladas com 1000 UI por ml de IFN-α para induzir a expressão MxA. O sh MxA endógenaOWS uma banda que corresponde à forma tetramérica. Por favor clique aqui para ver uma versão maior desta figura.
| nome tampão | Conteúdo | Comentários | ||
| O tampão de lise | Tris-HCl 20 (pH 7,5), 150 mM de NaCl, 5 mM de MgCl2, 100 uM iodoacetamida, NaF 50 mM, 1 mM de Na 3 VO 4, 1% de NP-40, mM β-glicerofosfato 50, 1 comprimido por 50 mL de tampão de lise de EDTA livre, cocktail inibidor de protease | Adicionar iodacetamide, NaF, Na 3 VO 4, octilfenoxipolietoxietanol (NP-40), β-glicerofosfato e protease inhibitor cocktail direita antes de lise celular Iodacetamide é sensível à luz e instável quando em solução. Evitar a exposição de luz e dissolver bem antes de usar. | ||
| tampão de diálise | Tris-HCl 20 (pH 6,8), 10% de glicerol, CHAPS a 0,1%, DTT 0,5 mM | CHAPS pode ser raplaced por outros detergentes não desnaturantes | ||
| tampão de corrida | Tris-HCl 25 (pH 8,3), glicina 192 mM, CHAPS a 0,1%, DTT 0,5 mM | A pH 8,3, a maioria das proteínas são carregadas negativamente. No entanto, para as proteínas básicas, um pH acídico deve ser usado. Caso contrário, as proteínas vai correr em sentido contrário e será perdido. CHAPS pode ser substituído por outros detergentes não desnaturantes | ||
| tampão de amostra | Tris-HCl 310 (pH 6,8), 0,05% de azul de bromofenol, 50% de glicerol | |||
| tampão SDS | Tris-HCl 25 (pH 8,3), glicina 192 mM, 0,1% de SDS | |||
| tampão absorvente | 25 mM de Tris, glicina 192 mM, 20% de metanol | Para grandes complexos, metanol pode ser omitido | ||
| tampão de bloqueio | Tris-HCl 50 (pH 7,4) 150 mM de NaCl 0,05% de Tween 20 5% de leite em pó | |||
| O meio de crescimento | meio modificado por Dulbecco 1x penicilina / estreptomicina 2 mM de glutamina 10% de Soro Fetal de Vitela | |||
Tabela 1: receitas buffer necessário para PAGE não desnaturante.
Discussão
Descrevemos aqui um método simples que permite a determinação rápida do estado oligomérico de proteínas expressas em células de mamíferos por PAGE não desnaturante seguido por análise Western Blot. A principal vantagem desta abordagem é que o estado oligomérico de uma dada proteína pode ser determinada a partir de lisados de células inteiras sem purificação de proteína antes. Isto pode ser importante para proteínas que oligomerizar ou exercem a sua função em associação com factores auxiliares. Além disso, as proteínas ainda estão no seu estado nativo e se ainda extraída a partir do gel, a actividade enzimática ou de outras funções da proteína pode ser determinada e correlacionada com o estado oligomérico.
Um aspecto crítico deste protocolo é a escolha de detergentes durante a preparação da amostra. Isto é de particular importância para as proteínas associadas com membranas celulares. MxA parece ser principalmente associado com as membranas do retículo endoplasmático liso 25 . Para a lise das células do detergente NP40 não iónico era óptima, evitando a precipitação de MxA. Após troca de tampão e a remoção de impurezas de baixo peso molecular dos lisados por diálise como descrito anteriormente 19, a presença de 0,1% de 3 - [(3-colamidopropil) dimetilamónio] -1-propanossulfonato (CHAPS) foi necessária para evitar a precipitação de MxA durante gel electroforese. Além disso, CHAPS a não interferir com a actividade enzimática de MxA conforme determinado com a proteína recombinante purificada MxA expressa em E. coli 12. É de grande importância que o detergente não desnaturar as proteínas ou interrompa interacções proteína-proteína durante a lise. detergentes não desnaturantes, tais como NP40, octoxinol-9, digitonina e CHAPS são adequadas para a solubilização. A escolha do detergente e a sua concentração deve ser determinada empiricamente. O detergente também pode influenciar experiências a jusante, por exemplo para certos ensaios do detergente precisa serremovido o que é facilmente conseguido com CHAPS por diálise, mas não com octoxinol-9, 26.
Uma vez que o método descrito análises de proteínas provenientes de lisados de células em condições não desnaturantes, sem prévia purificação das proteínas é necessária. Isto é uma vantagem uma vez que a purificação de proteínas recombinantes por vezes requer optimizações tampão através da adição de sais elevados e outros aditivos para evitar que a proteína de agregação ou precipitação. No entanto, estes aditivos e as elevadas concentrações de sal podem ter um impacto sobre a oligomerização da proteína que pode não necessariamente assemelham seu estado natural. Especialmente no caso de a proteína MxA humano, a concentração de sal e a presença de nucleótidos desempenham um papel crucial na formação de estados oligoméricos superiores 27. Em lisados de células, as proteínas são mais facilmente estabilizado desde os fatores de estabilização celulares ainda estão presentes. Por isso, é possível analisar PRotein complexos em condições fisiológicas (por exemplo, mais de células concentrações salinas fisiológicas, e de pH). Este protocolo deverá também ser aplicada a outras proteínas que formam oligómeros, por exemplo para a MxB estruturalmente relacionados ou dynamin 8.
Outro aspecto importante do protocolo é a protecção dos grupos tiol livres para evitar a formação de pontes dissulfureto artificiais de proteínas citoplasmáticas durante a lise (Figura 1). As experiências iniciais mostraram que a adição de 1,4-ditiotreitol (DTT) ou β-mercaptethanol não é suficiente para evitar a formação de ligações dissulfureto artificiais durante a preparação da amostra. Ambos os agentes redutores proteger o grupo tiol reversivelmente a formação de pontes de dissulfureto. Esta protecção reversível pode não ser suficiente para proteger todos os grupos tiol permanentemente. No caso de a proteína MxA, isto conduz a uma agregação irreversível da proteína. No entanto, a adição de iodoacetamida que irreversibly protege os grupos tiol livres de cisteinas grandemente reduzida agregação da proteína MxA Além disso, o tratamento de iodoacetamida de lisados preparados a partir de MxA expressando células de mamíferos não tiveram nenhuma influência sobre a actividade de GTPase de MxA imunoprecipitado quando comparado com MxA a partir de lisados tratados com DTT (dados não mostrados ).
Outras considerações importantes para a determinação exacta do número de protómeros em um oligómero são a escolha da gama de concentração de poliacrilamida assim como a referência de peso molecular de proteínas. A gama de concentração de poliacrilamida deve ser estabelecido pela primeira vez para permitir uma separação máxima das bandas a massas moleculares esperadas de oligómeros. Não desnaturante PAGE não contém qualquer SDS. Na falta de SDS, a carga, massa molecular e a forma da proteína determina a sua mobilidade electroforética. Portanto, a escolha do marcador de peso molecular da proteína é crucial. Idealmente, uma referência de peso molecular seria arecombinant forma purificada da proteína de interesse com os estados oligoméricos conhecidos. Uma vez que esta nem sempre está disponível, um marcador de proteína não desnaturada ou nativa deve ser usado. Desde MxA foi mostrado para associar com outras proteínas celulares, tais como nucleoproteínas ou UAP56 virais de Thogotovirus, La Crosse vírus ou vírus da gripe 12,28-30, também testado por imunocoloração utilizando anticorpos específicos se UAP56 ou nucleoproteína influenza A que co-separado com MxA sobre os géis não desnaturantes PA. No entanto, não encontrou nenhuma evidência para a formação de MXA hetero-oligômeros. Isto provavelmente deve-se ao facto de as interacções MxA-nucleoproteína MxA-UAP56 e são de baixa afinidade 12,24. Além disso, a fracção de proteína associar-se com MxA UAP56 nucleoproteínas ou virais pode ser muito baixa e, portanto, difíceis de detectar por este método.
Além disso, é importante ter em consideração o valor pKa da proteína a ser analisada. Na descrito não denatprotocolo de PAGE urante, as proteínas são separadas por electroforese a pH 8,3. A maioria das proteínas são carregadas negativamente a este pH. No entanto, as proteínas básicas apresentam uma carga líquida positiva a pH 8,3 e, portanto, vai correr em sentido contrário. Como consequência, é importante, para a análise de proteínas básicas para ajustar o pH do tampão de corrida para assegurar uma carga negativa global da proteína.
Tomados em conjunto que aqui apresentamos um protocolo para a rápida avaliação do estado oligomérico de proteínas expressas em células de mamíferos sem a necessidade da sua purificação prévia.
Divulgações
The authors have nothing to disclose.
Agradecimentos
This work was funded by a Grant from the Swiss National Science foundation (Grant nr. 31003A_143834) to JP.
Materiais
| Name | Company | Catalog Number | Comments |
| Slide-A-Lyzer MINI Dialysis Units, 10K MWCO, 0.5 ml | Thermo Fisher Scientific | 69570 | Pre-equilibrate in dialysis buffer (if Glycerol removal is desired) Can be self-made according to Fiala et al. 2011 |
| 4–15% Mini-PROTEAN TGX Precast Protein Gels, 10-well, | Bio-Rad | 456-1083 | Pre-run in running buffer to adjust buffer system |
| cOmplete, Mini, EDTA-free | Roche | 11836170001 | use 1 tablet per 50 ml |
| PBS, pH 7.4 bottle a 500 ml Gibco | Thermo Fisher Scientific | 14190-094 | |
| Ponceau S solution | Sigma-Aldrich | P7170 | TOXIC wear gloves and protect eyes |
| NativeMark Unstained Protein Standard 50 µl | Invitrogen | P/N 57030 | load 5 µl/well |
| A549 cells | ATCC | ATCC CCL185 | Grow in growth medium (see Table 1) |
| Vero cells | ATCC | ATCC CCL81 | Grow in growth medium (see Table 1) |
| anti-Mx1 antibody | Novus Biologicals | H00004599_D01P | Use at a 1:1,000 dilution |
| ECL Anti-rabbit IgG, Horseradish Peroxidase linked whole antibody (from donkey) | GE-Healthcare | NA934V | Use at a 1:10,000 dilution |
| 0.5% Trypsin-EDTA (1x) Life Technologies | Thermo Fisher | 15400-054 | |
| Iodoacetamide 5 g | Sigma-Aldrich | I-6125 | stock 100 mM |
| Bromphenolblue | Sigma-Aldrich | B0126-25G | |
| DMEM +4.5g/l Gluc,+L-Glut,+Pyruvate life technologies | Thermo Fisher Scientific | 41966-029 | |
| Pen Strep 100 x 100ml life technologies | Thermo Fisher Scientific | 15140 - 130 | |
| Glutamax 100x Stock, 100 ml life technologies | Thermo Fisher Scientific | 350500-038 | |
| Fetal Bovine Serum, Dialyzed , US Origin 500 ml Gibco Lot:42G9552K | Thermo Fisher Scientific | 10270-106 | |
| Cellulose filter paper | Bio-Rad | 1703965 | |
| PVDF blotting membrane | GE-Healthcare | 10600022 | |
| Tris(hydroxymethyl)aminomethane | Biosolve | 0020092391BS | |
| sodium fluoride (NaF) | Sigma Aldrich | S-7920 | |
| NP-40 | Calbiochem | 492015 | |
| cOmplete, Mini, EDTA-free | Roche | 11836170001 | |
| Tween 20 | Calbiochem | 6555204 | |
| CHAPS 10% solution | Amresco | N907 | |
| DL-Dithiothreitol (DTT) | Sigma Aldrich | 43819 | |
| Glycine | Biosolve | 0007132391BS | |
| sodium orthovanadate (Na3VO4) | Sigma Aldrich | 450243 | |
| Glycerol | Sigma Aldrich | G7757 | |
| β-Glycerophospate | Sigma Aldrich | G9422 | |
| Milk powder | Migros/Switzerland | ||
| Methanol | Millipore | 1.06009 | |
| sodium cloride (NaCl) | Sigma Aldrich | 71380 | |
| magnesium chloride (MgCl2) | Amresco | 288 | |
| Sodium dodecyl sulphate (SDS) | Sigma Aldrich | L4509 | |
| sodium hydroxide (NaOH) | Sigma Aldrich | S-8045 |
Referências
- Baisamy, L., Jurisch, N., Diviani, D. Leucine zipper-mediated homo-oligomerization regulates the Rho-GEF activity of AKAP-Lbc. J Biol Chem. 280, 15405-15412 (2005).
- Chen, C. P., Posy, S., Ben-Shaul, A., Shapiro, L., Honig, B. H. Specificity of cell-cell adhesion by classical cadherins: Critical role for low-affinity dimerization through beta-strand swapping. Proc Natl Acad Sci U S A. 102, 8531-8536 (2005).
- Jackson-Fisher, A. J., Chitikila, C., Mitra, M., Pugh, B. F. A role for TBP dimerization in preventing unregulated gene expression. Mol Cell. 3, 717-727 (1999).
- Torshin, I. Activating oligomerization as intermediate level of signal transduction: analysis of protein-protein contacts and active sites in several glycolytic enzymes. Front Biosci. 4, 557-570 (1999).
- Goodsell, D. S., Olson, A. J. Structural symmetry and protein function. Annu Rev Biophys Biomol Struct. 29, 105-153 (2000).
- Flydal, M. I., Martinez, A. Phenylalanine hydroxylase: function, structure, and regulation. IUBMB Life. 65, 341-349 (2013).
- Haller, O., Kochs, G. Human MxA protein: an interferon-induced dynamin-like GTPase with broad antiviral activity. J Interferon Cytokine Res. 31, 79-87 (2011).
- Haller, O., Staeheli, P., Schwemmle, M., Kochs, G. Mx GTPases: dynamin-like antiviral machines of innate immunity. Trends Microbiol. 23, 154-163 (2015).
- Di Paolo, C., Hefti, H. P., Meli, M., Landis, H., Pavlovic, J. Intramolecular backfolding of the carboxyl-terminal end of MxA protein is a prerequisite for its oligomerization. J Biol Chem. 274, 32071-32078 (1999).
- Janzen, C., Kochs, G., Haller, O. A monomeric GTPase-negative MxA mutant with antiviral activity. J Virol. 74, 8202-8206 (2000).
- Gao, S., et al. Structure of myxovirus resistance protein a reveals intra- and intermolecular domain interactions required for the antiviral function. Immunity. 35, 514-525 (2011).
- Nigg, P. E., Pavlovic, J. Oligomerization and GTP-binding Requirements of MxA for Viral Target Recognition and Antiviral Activity against Influenza A Virus. J Biol Chem. 290, 29893-29906 (2015).
- Gao, S., et al. Structural basis of oligomerization in the stalk region of dynamin-like MxA. Nature. 465, 502-506 (2010).
- Ghosh, I., Hamilton, A. D., Regan, L. Antiparallel leucine zipper-directed protein reassembly: Application to the green fluorescent protein. J Am Chem Soc. 122, 5658-5659 (2000).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterisation of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta. 1838, 43-55 (2014).
- Kenworthy, A. K. Imaging protein-protein interactions using fluorescence resonance energy transfer microscopy. Methods. 24, 289-296 (2001).
- Wyatt, P. J. Light-Scattering and the Absolute Characterization of Macromolecules. Analytica Chimica Acta. 272 (93), 1-40 (1993).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr Opin Chem Biol. 10, 430-436 (2006).
- Fiala, G. J., Schamel, W. W., Blumenthal, B. Blue native polyacrylamide gel electrophoresis (BN-PAGE) for analysis of multiprotein complexes from cellular lysates. J Vis Exp. , (2011).
- Zou, H., Li, Y., Liu, X., Wang, X. An APAF-1.cytochrome c multimeric complex is a functional apoptosome that activates procaspase-9. J Biol Chem. 274, 11549-11556 (1999).
- Schagger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal Biochem. 217, 220-230 (1994).
- Walker, J. M. Nondenaturing polyacrylamide gel electrophoresis of proteins. Methods Mol Biol. 32, 17-22 (1994).
- Stoscheck, C. M. Quantitation of protein. Methods Enzymol. 182, 50-68 (1990).
- Wisskirchen, C., Ludersdorfer, T. H., Muller, D. A., Moritz, E., Pavlovic, J. Interferon-induced antiviral protein MxA interacts with the cellular RNA helicases UAP56 and URH49. J Biol Chem. 286, 34743-34751 (2011).
- Stertz, S., et al. Interferon-induced, antiviral human MxA protein localizes to a distinct subcompartment of the smooth endoplasmic reticulum. J Interferon Cytokine Res. 26, 650-660 (2006).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: not just a soap opera. Biochim Biophys Acta. 1666, 105-117 (2004).
- Kochs, G., Haener, M., Aebi, U., Haller, O. Self-assembly of human MxA GTPase into highly ordered dynamin-like oligomers. J Biol Chem. 277, 14172-14176 (2002).
- Kochs, G., Haller, O. GTP-bound human MxA protein interacts with the nucleocapsids of Thogoto virus (Orthomyxoviridae). J Biol Chem. 274, 4370-4376 (1999).
- Reichelt, M., Stertz, S., Krijnse-Locker, J., Haller, O., Kochs, G. Missorting of LaCrosse virus nucleocapsid protein by the interferon-induced MxA GTPase involves smooth ER membranes. Traffic. 5, 772-784 (2004).
- Wisskirchen, C., Ludersdorfer, T. H., Muller, D. A., Moritz, E., Pavlovic, J. The cellular RNA helicase UAP56 is required for prevention of double-stranded RNA formation during influenza A virus infection. J Virol. 85, 8646-8655 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados