Method Article
Caratterizzazione di complessi multi-subunità proteiche di MxA umano per mezzo di non-denaturare poliacrilammide gel elettroforesi
In questo articolo
Riepilogo
This article describes a simple and rapid protocol to evaluate the oligomeric state of the dynamin-like GTPase MxA protein from lysates of human cells using a combination of non-denaturing PAGE with western blot analysis.
Abstract
The formation of oligomeric complexes is a crucial prerequisite for the proper structure and function of many proteins. The interferon-induced antiviral effector protein MxA exerts a broad antiviral activity against many viruses. MxA is a dynamin-like GTPase and has the capacity to form oligomeric structures of higher order. However, whether oligomerization of MxA is required for its antiviral activity is an issue of debate. We describe here a simple protocol to assess the oligomeric state of endogenously or ectopically expressed MxA in the cytoplasmic fraction of human cell lines by non-denaturing polyacrylamide gel electrophoresis (PAGE) in combination with Western blot analysis. A critical step of the protocol is the choice of detergents to prevent aggregation and/or precipitation of proteins particularly associated with cellular membranes such as MxA, without interfering with its enzymatic activity. Another crucial aspect of the protocol is the irreversible protection of the free thiol groups of cysteine residues by iodoacetamide to prevent artificial interactions of the protein. This protocol is suitable for a simple assessment of the oligomeric state of MxA and furthermore allows a direct correlation of the antiviral activity of MxA interface mutants with their respective oligomeric states.
Introduzione
La struttura quaternaria di una proteina gioca un ruolo fondamentale in molti processi cellulari. Vie di segnalazione, l'espressione genica, e l'enzima di attivazione / disattivazione tutti si basano sul corretto montaggio di complessi proteici 1-4. Questo processo noto anche come omo- o etero-oligomerizzazione è dovuta a covalente irreversibile o reversibile interazioni proteina-proteina elettrostatiche e idrofobiche. Oligomerizzazione non solo diversifica i diversi processi cellulari senza aumentare le dimensioni del genoma, ma fornisce anche una strategia di proteine per costruire complessi stabili che sono più resistenti verso denaturazione e degradazione 5. Difetti di oligomerizzazione hanno un impatto sulla funzione delle proteine e può portare allo sviluppo di malattie. Ad esempio, l'enzima fenilalanina idrossilasi forma un complesso tetrameric. Alcune mutazioni all'interno del complesso proteico possono indebolire la formazione tetramero e portare alla fenilchetonuria malattia 6.
La proteina MxA umano è un interferone (IFN) indotta antivirale proteine effettrici esercitando una vasta attività antivirale contro vari RNA così come virus a DNA 7. Essa appartiene alla superfamiglia delle grandi GTPasi Dynamin-like e ha la capacità di formare grandi strutture oligomeriche in vitro 8. L'oligomerizzazione è stato suggerito per proteggere MxA dalla rapida degradazione 9,10. Nonostante intensi sforzi da molti gruppi di ricerca, il meccanismo molecolare di azione rimane in gran parte sfuggente e il ruolo dello Stato oligomerizzazione di MxA per la sua funzione antivirale è in discussione 9,11,12. A questo proposito, Gao e colleghi hanno proposto un modello in cui MxA esercita la sua attività antivirale interagendo con nucleoproteine virali in forma di grandi strutture oligomeriche anulari 11. Tuttavia, più di recente, abbiamo dimostrato che dimeri MxA mostrano attività antivirale e interagiscono con la nucleoproteina del virus A 12. Based sulla struttura cristallina di MxA, Gao e colleghi hanno individuato diversi residui di amminoacidi nelle regioni di interfaccia che sono critiche per la sua oligomerizzazione in vitro e la sua funzione antivirale 11,13. Pertanto, al fine di chiarire che oligomerica stato di MxA esercita attività antivirale, abbiamo cercato di creare un semplice protocollo per determinare rapidamente lo stato di oligmeric MxA mutanti interfaccia espressi nelle cellule umane, così come endogena MxA espresso dopo la stimolazione IFNα.
Anche se ci sono molte tecniche che vengono comunemente utilizzati per studiare l'interazione tra le proteine come la proteina split-verde fluorescente (split-GFP) saggio di complementazione 14, risonanza plasmonica di superficie 15 e Förster trasferimento di energia di risonanza (FRET) 16, non forniscono informazioni dell'esatta stechiometria di un complesso proteico oligomerica. Per indagare questo particolare aspetto, tecniche comemulti-angolo di diffusione della luce (MALS) 17 e ultracentrifugazione analitica 18 sono molto utili. Di solito, le proteine analizzate utilizzando questi metodi sono proteine purificate. processi di oligomerizzazione può dipendere anche da altri fattori cellulari. Se questi fattori non sono noti, l'analisi è più difficile. Inoltre, alcune proteine sono difficili da esprimere in E. coli e purificare. Pertanto, questi metodi non sono la scelta ottimale per analizzare oligomerizzazione proteina nel ambiente cellulare. Inoltre, queste tecniche richiedono strumenti costosi che non sono facilmente disponibili.
Non denaturante elettroforesi su gel di poliacrilammide (PAGE), cromatografia ad esclusione sterica o reticolazione chimica seguita da convenzionale sodio dodecil solfato (SDS) -pagina sono strumenti utili per la caratterizzazione della formazione di oligomeri da lisati cellulari 2,19,20. Questi metodi non richiedono attrezzature specializzate e possono essere facilmente performed in un laboratorio standard. Inizialmente abbiamo valutato vari protocolli di reticolazione chimici che invariantly hanno portato alla non-specifica aggregazione e la precipitazione di MxA. Pertanto, la prossima testato protocolli PAGE non denaturazione. Come non-denaturazione PAGE esclude l'uso di SDS, la migrazione delle proteine dipende dalla loro carica nativa. Blue-native PAGE utilizza brillante Coomassie G250 blu per caricare proteine con una carica negativa complessiva, simile a SDS, ma non denatura la proteina 21. Purtroppo, brillante Coomassie precipitati blu in presenza di alte sali e cationi bivalenti (ad esempio Mg 2+) che sono spesso inclusi nei buffer di lisi. A seconda dei buffer utilizzati, può essere difficile analizzare il campione senza ulteriore ottimizzazione di passi che potrebbero avere un effetto sul complesso proteico oligomerica.
Qui vi presentiamo un protocollo semplice basato su un metodo precedentemente pubblicato 22 per determinare oligomerizzazione diproteina umana MxA derivate da lisati cellulari utilizzando non-denaturazione PAGE.
Protocollo
NOTA: Questo protocollo si basa sul precedentemente pubblicati non-denaturazione PAGE protocollo 12. In questo studio, lo stato oligomerico della proteina MxA è stata valutata utilizzando sia cellule Vero overexpressing MxA o cellule A549 IFN-alfa-stimolato esprimono endogeno MxA. Il protocollo descritto di seguito può essere utilizzato per analizzare lo stato oligomerico di qualsiasi proteina in aggiunta a MxA. Tuttavia, può essere necessaria un'ulteriore ottimizzazione.
1. Preparazione del lisato per i non-denaturazione PAGINA
NOTA: per analizzare lo stato oligomerico della proteina MxA umana sia in cellule Vero o A549, 1,0 x 10 6 cellule sono state raccolte. A seconda del tipo di cellula o l'abbondanza della proteina da analizzare, il numero della cella deve essere regolata. E 'anche importante proteggere il tampone di lisi da esposizione alla luce, non appena viene aggiunto il iodoacetamide fotosensibile.
- Seed 0,3 x 10 6 cellule A549 o Vero per bene ina 6 e piatti-. Mantenere le cellule in 2 ml di terreno di coltura per bene (vedi tabella 1). Incubare le cellule durante la notte in un incubatore di coltura cellulare (37 ° C, 5% CO 2).
- Raccogliere le cellule di lavaggio con 1 ml di tampone fosfato salino (PBS) e separare aggiungendo acido etilendiamminotetraacetico tripsina-0,5 ml di 0,25% (EDTA) 1x soluzione per circa 5 min a temperatura ambiente.
- Non appena le cellule si staccano dal piatto, aggiungere 0,5 ml di mezzo di crescita e miscelare accuratamente pipettando su e giù.
- Trasferire le cellule di ogni pozzetto in una provetta 2 ml e pellet usando una centrifuga da tavolo (5.000 xg, a 4 ° C, 5 min).
- rimuovere con attenzione il surnatante pipettando senza disturbare il pellet.
- Lavare le cellule con 1 ml di PBS ghiacciato pipettando accuratamente la sospensione cellulare su e giù.
- Cellule pellet in una tabella centrifuga (5000 XG, 4 ° C, 5 min).
- rimuovere con attenzione il surnatante pipettando wisarie, senza staccare il pellet di cellule.
- Risospendere le cellule in 200 tampone di lisi ghiacciata ml (vedi tabella 1) pipettando su e giù e mettere in ghiaccio.
- Immediatamente, proteggere dalla luce lisato coprendo i tubi con carta stagnola e incubare per 30 min in ghiaccio.
NOTA: Dopo incubazione per 30 min in ghiaccio, non è più essenziale per proteggere il lisato da esposizione alla luce, poiché la protezione dei gruppi tiolici liberi è irreversibile. - Rimuovere detriti cellulari mediante centrifugazione in una centrifuga da tavolo pre-raffreddata (13.000 xg, a 4 ° C, 20 min).
- Equilibrare colonne dialisi in tampone di dialisi (Tabella 1) in camera fredda a 4 ° C per 20 minuti durante la fase di centrifugazione. Utilizzare una colonna con un peso molecolare di 10.000 tagliato.
- Fissare le colonne ad una boa galleggiante e metterli in un bicchiere pieno di buffer di dialisi. Per garantire leggera agitazione, utilizzare un agitatore magnetico. Non toccare la membrana.
NOTA: Dialysè colonne possono essere acquistati o preparati da 1,5 ml tubi secondo il protocollo descritto da Fiala e collaboratori 19.
- Fissare le colonne ad una boa galleggiante e metterli in un bicchiere pieno di buffer di dialisi. Per garantire leggera agitazione, utilizzare un agitatore magnetico. Non toccare la membrana.
- Rimuovere le colonne dal buffer di dialisi e la boa galleggiante. Trasferire i lisati eliminato nella colonna di dialisi preparata pipettando senza toccare la membrana. Fissare le colonne ad una boa galleggiante e metterli nuovamente dentro il bicchiere pieno di buffer di dialisi.
- Dializzare il lisato in un becher contenente tampone di dialisi ghiacciata (Tabella 1) per almeno 4 ore (o preferibilmente durante la notte) a 4 ° C mentre accuratamente agitazione con un agitatore magnetico. Utilizzare almeno 100 ml di tampone di dialisi per un lisato 200 microlitri.
- Trasferire il campione dializzato in una provetta da 1,5 ml. Rimuovere precipitati per centrifugazione in una centrifuga da tavolo (13.000 xg, a 4 ° C, 20 min). Per impedire la dissociazione della proteina oligomerica complessi continuano con il protocollo (sezione 2) immediatamente dopo la dialisi. Non congelarelisati preparati.
2. elettroforesi
NOTA: L'elettroforesi è stata eseguita come descritto prima con alcune modifiche 22. Nel protocollo descritto di seguito, prefabbricati gel gradiente sono stati utilizzati (4-15% di pendenza). In alternativa, i gel possono essere preparati in laboratorio. E 'molto importante escludere qualsiasi agente denaturante come SDS impedire la dissociazione dei complessi proteici oligomerici. Tempo di elettroforesi è stata ottimizzata per i diversi stati oligomerici della proteina MxA umana. Tuttavia, può variare per altre proteine, a seconda delle dimensioni del complesso oligomerico così come l'intervallo di separazione, che dovrebbe essere raggiunto per analizzare il complesso. Pertanto, il tempo ottimale di elettroforesi deve essere determinato empiricamente. Per una risoluzione ottimale degli oligomeri da analizzare la corrente non deve superare i 25 mA.
- Montare il gel PAGE non denaturazione nella camera di gel. riempire tegli camera interna e esterna con tampone di corsa pre-raffreddata (Tabella 1).
- Pre-eseguire il gel con tampone di corsa pre-raffreddata a 25 mA per gel per 15 minuti in camera fredda a 4 ° C.
- Mescolare 15 ml di lisati sopra preparato con 5 ml di tampone campione 4x (Tabella 1). Non bollire il campione.
- Caricare 15 ml di campione e uno standard proteina nativa di scelta sul gel. Eseguire il gel a 25 mA per 4 ore in cella a 4 ° C.
NOTA: Per le analisi semi-quantitativa, un protocollo proteina quantificazione (ad esempio un Metodo di Bradford 23) può essere effettuata in modo da garantire il caricamento di quantità uguali di proteine totali per corsia.
3. Western Blot
NOTA: vedere più avanti è il protocollo di un sistema di Western Blot bagnato. Qualsiasi membrana assorbente può essere utilizzato. Attivare fluoruro di polivinile (PVDF) membrane in 100% di metanolo prima di equilibrio in buf assorbentefer. La tecnica Western Blot semi-secco può essere utilizzato in alternativa, ma deve essere ottimizzato per grandi complessi oligomerici.
- Smontare il gel e trasferire con cautela in SDS tampone (Tabella 1).
- Incubare per 10 minuti a temperatura ambiente, mentre scuotendo delicatamente.
- Preparare 2 spugne, 4 fogli di carta filtro di cellulosa e una membrana assorbente per gel. Ammollo in assorbente tampone (Tabella 1).
- Montare il panino come segue (basso verso l'alto): 1 spugna, 2 cellulosa fogli di carta filtro, membrana, gel, 2 di cellulosa fogli di carta filtro, 1 spugna.
- Mettere il sandwich nel serbatoio blotting. Assicurarsi che la membrana si affaccia sul polo positivo, mentre il gel di fronte al polo negativo.
- Riempire il serbatoio tamponando con tampone assorbente pre-raffreddata.
- Tamponare a 90 mA notte a 4 ° C per i migliori risultati di trasferimento proteina.
- Smontare il panino e visualizzare il livello di proteine incubando la membrana in Poncsoluzione eau S per 5 min a temperatura ambiente.
- Decolorare la membrana con attenzione lavare via il Ponceau S con acqua deionizzata fino a quando si può vedere chiaramente le bande dello standard di proteine.
- Segnare le bande dello standard proteina con una penna.
NOTA: residua Ponceau S può interferire con il immunocolorazione. Per evitare questo, la membrana può essere decolorato ulteriormente mediante incubazione in 0,1 M NaOH per 1 min e successivo lavaggio con acqua deionizzata. - Bloccare la membrana con tampone di bloccaggio (vedi Tabella 1) per almeno 1 ora a temperatura ambiente o per una notte a 4 ° C.
- Visualizza proteina (s) di interesse mediante immunocolorazione con anticorpi diretti contro la proteina da analizzare.
NOTA: La proteina MxA umana è stata visualizzata utilizzando l'anticorpo policlonale di coniglio specifico per Mx1 umano diluito 1: 1000 in tampone di bloccaggio (Tabella 1). La soluzione di anticorpi è stata incubata per una notte a 4 ° C. In alternativa, il monoclonale unnti-MxA anticorpi (clone 143) può essere utilizzato (dati non mostrati) 24.
Risultati
L'utilizzo non denaturazione PAGE, abbiamo analizzato lo stato oligomerico di tipo selvatico umano MxA, i mutanti dimerici interfaccia MxA (R640A) e MxA (L617D), così come l'interfaccia mutante monomerico MxA (M527D) da lisati cellulari 12. Le cellule sono state lisate in un tampone contenente 1% ottilfenossipolietossietanolo (NP-40) e iodoacetamide per assicurare solubilizzazione delle proteine e la protezione di gruppi tiolici liberi (vedi Figura 1). Come descritto prima, sale e piccoli metaboliti sono stati rimossi mediante dialisi 19. separazione delle proteine è stata effettuata da non denaturazione PAGE. Per facilitare efficiente western blotting, il gel è stato incubato in tampone SDS prima assorbente. Le proteine sono state visualizzate mediante MxA immunocolorazione utilizzando un anticorpo policlonale di coniglio diretto contro MxA. Il flusso di lavoro è descritto nella Figura 2.
Per confrontare lo stato oligomerico di proteine MxA umana endogena da IFN-α ; cellule A549 stimolate, abbiamo transfettate cellule Vero (mancano endogena MxA) con di tipo selvatico ricombinante, varianti monomerici e dimerici MxA. Questi tipo selvaggio ricombinante, monomero e dimero MxA varianti formata tetrameri stabili, monomeri e dimeri, rispettivamente, rispetto a un marcatore proteina nativa senza macchia (Figura 3A). Pertanto, abbiamo usato queste proteine ricombinanti per valutare lo stato oligomerico endogeno proteine MxA umano ottenuto da IFN-α stimolato cellule A549. Figura 3B mostra che la dimensione di MxA in lisati di cellule A549 IFN-alfa-stimolato corrisponde ad un tetramero.
Presi insieme, si descrive un metodo per determinare lo stato oligomerico della proteina MxA umana dal lisato cellulare. Il nostro approccio PAGE non denaturante può anche essere utilizzato per valutare lo stato oligomerico di complessi di proteine oligomeriche.
83 / 54683fig1.jpg "/>
Figura 1:. Struttura e reazione schema di Iodoacetamide Iodoacetamide protegge in maniera irreversibile il gruppo tiolo di cisteine libere formando un legame tioetere. Questa stalla modifica deriva dalla sostituzione nucleofila dello iodio con l'atomo di zolfo dalla cisteina. Clicca qui per vedere una versione più grande di questa figura.
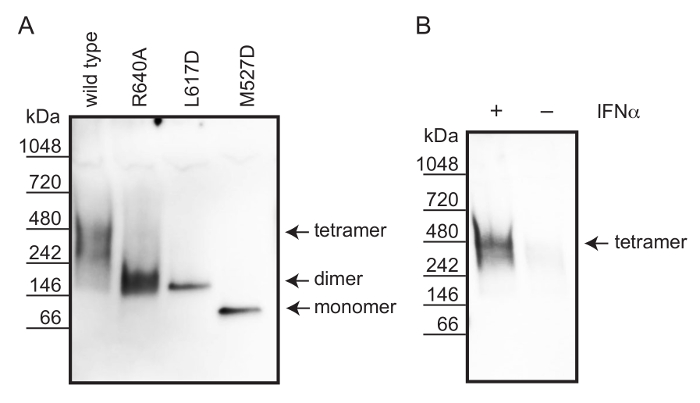
Figura 2:. Workflow schema della non-denaturazione PAGE Una rappresentazione sistematica dell'approccio PAGE non denaturante di lisati cellulari. Durante lisi cellulare, detergenti solubilizzano le proteine e gruppi tiolici sono protetti da iodoacetamide per prevenire l'aggregazione della proteina. La dialisi rimuove piccoli metaboliti e sali che potrebbero interferire con i non-denaturazione PAGINA 19. La separazione complesso viene eseguita in condizioni non denaturanti. Rilevazione dei complessi oligomerici si ottiene con Western Blot seguito da immunocolorazione. Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 3: Determinazione dello stato oligomerico della proteina MxA umana usando non-denaturazione PAGE e Western blotting (A) ricombinante MxA varianti ectopicamente espresso in cellule Vero.. I complessi di tipo selvaggio MxA (tetramero) mutanti interfaccia MxA (R640A), MxA (L617D) (dimeri) e MxA (M527D) migrano i loro pesi molecolari attese, confermando il loro stato oligomerico. (B), le cellule A549 sono state stimolate con 1.000 UI per ml di IFN-α per indurre l'espressione MxA. Il sh MxA endogenaOWS una band che corrisponde alla forma tetramerica. Clicca qui per vedere una versione più grande di questa figura.
| nome Buffer | contenuto | Commenti | ||
| tampone di lisi | 20 mM Tris-HCl (pH 7,5), 150 mM NaCl, 5 mM MgCl 2, 100 iodoacetamide micron, 50 mM NaF, 1 mM Na 3 VO 4, 1% NP-40, 50 mM β-glicerofosfato, 1 compressa per 50 ml Lysis buffer di EDTA libero, cocktail inibitore della proteasi | Aggiungere iodacetamide, NaF, Na 3 VO 4, ottilfenossipolietossietanolo (NP-40), β-glicerofosfato e proteasi inibizionetor cocktail a destra prima la lisi cellulare Iodacetamide è fotosensibile e instabile quando in soluzione. Evitare l'esposizione alla luce e sciogliere destra prima dell'uso. | ||
| tampone di dialisi | 20 mM Tris-HCl (pH 6,8), 10% glicerolo, 0,1% CHAPS, 0.5 mM DTT | CHAPS possono essere raplaced da altri detergenti non denaturanti | ||
| tampone di corsa | 25 mM Tris-HCl (pH 8,3), 192 mm glicina, 0,1% CHAPS, 0.5 mM DTT | A pH 8.3, la maggior parte delle proteine sono caricati negativamente. Tuttavia, per le proteine di base, un pH acido deve essere utilizzato. In caso contrario, le proteine verrà eseguito nella direzione opposta e saranno perduti. CHAPS possono essere sostituiti da altri detergenti non denaturanti | ||
| tampone campione | 310 mm Tris-HCl (pH6.8), bromofenolo blu 0,05%, 50% glicerolo | |||
| tampone SDS | 25 mM Tris-HCl (pH 8,3), 192 mm glicina, 0,1% SDS | |||
| tampone assorbente | 25 mM Tris, 192 mm glicina, 20% Metanolo | Per molto grandi complessi, metanolo può essere omesso | ||
| tampone di bloccaggio | 50 mM Tris-HCl (pH 7,4) 150 mM NaCl 0,05% Tween 20 5% Latte in polvere | |||
| mezzo di crescita | mezzo modificato di Dulbecco 1x penicillina / streptomicina 2 mM glutammina 10% siero fetale di vitello | |||
Tabella 1: ricette buffer necessari per non denaturazione PAGE.
Discussione
Qui si descrive un semplice metodo che permette la rapida determinazione dello stato oligomerico di proteine espresse in cellule di mammifero mediante PAGE non denaturante seguita da analisi Western blot. Il principale vantaggio di questo approccio è che lo stato oligomerico di una data proteina può essere determinata da lisati cellulari totali senza purificazione della proteina prima. Ciò può essere importante per proteine che oligomerize o esercitano la loro funzione in associazione con fattori ausiliari. Inoltre, le proteine sono ancora nel loro stato nativo e se ulteriormente estratta dal gel, l'attività enzimatica o altre funzioni della proteina possono essere determinate e correlati allo stato oligomerico.
Un aspetto critico di questo protocollo è la scelta dei detergenti durante la loro preparazione. Questo è di particolare importanza per le proteine associate alle membrane cellulari. MxA sembra essere principalmente associata con membrane del reticolo endoplasmatico liscio 25 . Per la lisi cellulare il detersivo NP40 non ionico è stato ottimale, impedendo la precipitazione di MxA. Dopo scambio buffer e rimozione delle impurezze a basso peso molecolare dei lisati di dialisi come precedentemente descritto 19 la presenza di 0,1% 3 - [(3-cholamidopropyl) dimethylammonio] -1-propanesulfonate (CHAPS) è necessario per impedire la precipitazione di MxA durante gel elettroforesi. Inoltre, CHAPS non interferisce con l'attività enzimatica di MxA determinato con purificata proteina ricombinante MxA espressa in E. coli 12. E 'di grande importanza che il detergente non denatura le proteine o interrompe le interazioni proteina-proteina durante la lisi. detergenti non denaturanti, come NP40, Octoxinol 9, digitonina e CHAPS sono adatti per solubilizzazione. La scelta del detersivo e la sua concentrazione deve essere determinato empiricamente. Il detersivo potrebbe anche influenzare gli esperimenti a valle, ad esempio per alcuni test il detergente deve essererimosso che è facilmente ottenibile con CHAPS per dialisi, ma non con Octoxinol 9 26.
Poiché il metodo descritto analizza proteine derivate da lisati cellulari in condizioni non denaturanti, non è necessaria alcuna purificazione preventiva delle proteine. Questo è un vantaggio poiché la purificazione di proteine ricombinanti volte richiede ottimizzazioni tampone con l'aggiunta di alte sali ed altri additivi per impedire la proteina dall'aggregazione o precipitazione. Tuttavia, tali additivi e le elevate concentrazioni saline potrebbero avere un impatto sulla oligomerizzazione della proteina che potrebbe non necessariamente assomigliare suo stato naturale. Specialmente nel caso della proteina MxA umana, la concentrazione di sale e la presenza di nucleotidi giocano un ruolo cruciale nella formazione di elevate stati oligomerici 27. In lisati cellulari, le proteine sono più facilmente stabilizzate in quanto i fattori di stabilizzazione cellulari sono ancora presenti. Pertanto è possibile analizzare prcomplessi otein in condizioni fisiologiche più cellulari (ad esempio concentrazioni saline fisiologiche, e pH). Questo protocollo dovrebbe essere applicabile anche per le altre proteine formando oligomeri, ad esempio per la strutturalmente correlato MxB o dynamin 8.
Un altro aspetto importante del protocollo è la protezione di gruppi tiolici liberi per evitare la formazione di ponti disolfuro artificiali di proteine citoplasmatiche durante lisi (Figura 1). esperimenti iniziali hanno mostrato che l'aggiunta di 1,4-ditiotreitolo (DTT) o β-mercaptethanol non è sufficiente per evitare la formazione di legami disolfuro artificiali durante la loro preparazione. Entrambi gli agenti riducenti proteggono il gruppo tiolico reversibilmente formazione di ponti disolfuro. Questa protezione reversibile, potrebbe non essere sufficiente per proteggere tutti i gruppi tiolici in modo permanente. Nel caso della proteina MxA, questo porta all'aggregazione irreversibile della proteina. Tuttavia, l'aggiunta di iodoacetamide che irreversprotegge ibly i gruppi tiolici liberi di cisteine notevolmente ridotta aggregazione della proteina MxA Inoltre, trattamento iodoacetamide di lisati preparati da MxA esprimere cellule di mammifero avuto alcuna influenza sull'attività GTPasi di immunoprecipitato MxA rispetto a MxA da lisati trattati con DTT (dati non mostrati ).
Altre considerazioni cruciali per l'esatta determinazione del numero di protomers in un oligomero sono la scelta della gamma di concentrazione poliacrilammide così come la proteina molecolare peso di riferimento. La gamma della concentrazione poliacrilammide dovrebbe essere stabilito prima di consentire la separazione massima delle bande alle masse molecolari attesi di oligomeri. Non denaturazione pagina non contiene alcuna SDS. Mancando SDS, la carica, la massa molecolare e la forma della proteina determina la sua mobilità elettroforetica. Pertanto, la scelta della proteina marker di peso molecolare è cruciale. Idealmente, un riferimento peso molecolare sarebbe arforma purificata ecombinant della proteina di interesse con note stati oligomerici. Poiché questo non è sempre disponibile, un marcatore proteina non denaturato o nativa dovrebbe essere usato. Dal MxA ha dimostrato di associarsi con altre proteine cellulari come UAP56 o nucleoproteine virali di Thogotovirus, La Crosse virus o virus influenzale 12,28-30, abbiamo testato anche da immunocolorazione con anticorpi specifici sia UAP56 o influenza A nucleoproteico avrebbe co-separata con MxA sui gel non denaturante PA. Tuttavia abbiamo trovato alcuna prova per la formazione di MxA etero-oligomeri. Questo probabilmente è dovuto al fatto che MxA-UAP56 e le interazioni MxA-nucleoproteici sono di bassa affinità 12,24. Inoltre, la frazione di MxA proteine associarli UAP56 o nucleoproteine virali potrebbe essere molto bassa e quindi difficile da rilevare con questo metodo.
Inoltre, è importante prendere in considerazione la pKa della proteina da analizzare. Nel non-denat descrittaprotocollo PAGE urante, le proteine sono separati mediante elettroforesi a pH 8,3. La maggior parte delle proteine vengono caricate negativamente a questo pH. Tuttavia, le proteine di base mostrano una carica netta positiva a pH 8,3 e quindi verrà eseguito nella direzione opposta. Di conseguenza, è importante, per l'analisi di proteine basiche per regolare il pH del tampone di corsa per assicurare una carica negativa complessiva della proteina.
Presi insieme presentiamo un protocollo per la valutazione rapida dello stato oligomerico di proteine espresse in cellule di mammifero, senza la necessità della sua purificazione prima.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work was funded by a Grant from the Swiss National Science foundation (Grant nr. 31003A_143834) to JP.
Materiali
| Name | Company | Catalog Number | Comments |
| Slide-A-Lyzer MINI Dialysis Units, 10K MWCO, 0.5 ml | Thermo Fisher Scientific | 69570 | Pre-equilibrate in dialysis buffer (if Glycerol removal is desired) Can be self-made according to Fiala et al. 2011 |
| 4–15% Mini-PROTEAN TGX Precast Protein Gels, 10-well, | Bio-Rad | 456-1083 | Pre-run in running buffer to adjust buffer system |
| cOmplete, Mini, EDTA-free | Roche | 11836170001 | use 1 tablet per 50 ml |
| PBS, pH 7.4 bottle a 500 ml Gibco | Thermo Fisher Scientific | 14190-094 | |
| Ponceau S solution | Sigma-Aldrich | P7170 | TOXIC wear gloves and protect eyes |
| NativeMark Unstained Protein Standard 50 µl | Invitrogen | P/N 57030 | load 5 µl/well |
| A549 cells | ATCC | ATCC CCL185 | Grow in growth medium (see Table 1) |
| Vero cells | ATCC | ATCC CCL81 | Grow in growth medium (see Table 1) |
| anti-Mx1 antibody | Novus Biologicals | H00004599_D01P | Use at a 1:1,000 dilution |
| ECL Anti-rabbit IgG, Horseradish Peroxidase linked whole antibody (from donkey) | GE-Healthcare | NA934V | Use at a 1:10,000 dilution |
| 0.5% Trypsin-EDTA (1x) Life Technologies | Thermo Fisher | 15400-054 | |
| Iodoacetamide 5 g | Sigma-Aldrich | I-6125 | stock 100 mM |
| Bromphenolblue | Sigma-Aldrich | B0126-25G | |
| DMEM +4.5g/l Gluc,+L-Glut,+Pyruvate life technologies | Thermo Fisher Scientific | 41966-029 | |
| Pen Strep 100 x 100ml life technologies | Thermo Fisher Scientific | 15140 - 130 | |
| Glutamax 100x Stock, 100 ml life technologies | Thermo Fisher Scientific | 350500-038 | |
| Fetal Bovine Serum, Dialyzed , US Origin 500 ml Gibco Lot:42G9552K | Thermo Fisher Scientific | 10270-106 | |
| Cellulose filter paper | Bio-Rad | 1703965 | |
| PVDF blotting membrane | GE-Healthcare | 10600022 | |
| Tris(hydroxymethyl)aminomethane | Biosolve | 0020092391BS | |
| sodium fluoride (NaF) | Sigma Aldrich | S-7920 | |
| NP-40 | Calbiochem | 492015 | |
| cOmplete, Mini, EDTA-free | Roche | 11836170001 | |
| Tween 20 | Calbiochem | 6555204 | |
| CHAPS 10% solution | Amresco | N907 | |
| DL-Dithiothreitol (DTT) | Sigma Aldrich | 43819 | |
| Glycine | Biosolve | 0007132391BS | |
| sodium orthovanadate (Na3VO4) | Sigma Aldrich | 450243 | |
| Glycerol | Sigma Aldrich | G7757 | |
| β-Glycerophospate | Sigma Aldrich | G9422 | |
| Milk powder | Migros/Switzerland | ||
| Methanol | Millipore | 1.06009 | |
| sodium cloride (NaCl) | Sigma Aldrich | 71380 | |
| magnesium chloride (MgCl2) | Amresco | 288 | |
| Sodium dodecyl sulphate (SDS) | Sigma Aldrich | L4509 | |
| sodium hydroxide (NaOH) | Sigma Aldrich | S-8045 |
Riferimenti
- Baisamy, L., Jurisch, N., Diviani, D. Leucine zipper-mediated homo-oligomerization regulates the Rho-GEF activity of AKAP-Lbc. J Biol Chem. 280, 15405-15412 (2005).
- Chen, C. P., Posy, S., Ben-Shaul, A., Shapiro, L., Honig, B. H. Specificity of cell-cell adhesion by classical cadherins: Critical role for low-affinity dimerization through beta-strand swapping. Proc Natl Acad Sci U S A. 102, 8531-8536 (2005).
- Jackson-Fisher, A. J., Chitikila, C., Mitra, M., Pugh, B. F. A role for TBP dimerization in preventing unregulated gene expression. Mol Cell. 3, 717-727 (1999).
- Torshin, I. Activating oligomerization as intermediate level of signal transduction: analysis of protein-protein contacts and active sites in several glycolytic enzymes. Front Biosci. 4, 557-570 (1999).
- Goodsell, D. S., Olson, A. J. Structural symmetry and protein function. Annu Rev Biophys Biomol Struct. 29, 105-153 (2000).
- Flydal, M. I., Martinez, A. Phenylalanine hydroxylase: function, structure, and regulation. IUBMB Life. 65, 341-349 (2013).
- Haller, O., Kochs, G. Human MxA protein: an interferon-induced dynamin-like GTPase with broad antiviral activity. J Interferon Cytokine Res. 31, 79-87 (2011).
- Haller, O., Staeheli, P., Schwemmle, M., Kochs, G. Mx GTPases: dynamin-like antiviral machines of innate immunity. Trends Microbiol. 23, 154-163 (2015).
- Di Paolo, C., Hefti, H. P., Meli, M., Landis, H., Pavlovic, J. Intramolecular backfolding of the carboxyl-terminal end of MxA protein is a prerequisite for its oligomerization. J Biol Chem. 274, 32071-32078 (1999).
- Janzen, C., Kochs, G., Haller, O. A monomeric GTPase-negative MxA mutant with antiviral activity. J Virol. 74, 8202-8206 (2000).
- Gao, S., et al. Structure of myxovirus resistance protein a reveals intra- and intermolecular domain interactions required for the antiviral function. Immunity. 35, 514-525 (2011).
- Nigg, P. E., Pavlovic, J. Oligomerization and GTP-binding Requirements of MxA for Viral Target Recognition and Antiviral Activity against Influenza A Virus. J Biol Chem. 290, 29893-29906 (2015).
- Gao, S., et al. Structural basis of oligomerization in the stalk region of dynamin-like MxA. Nature. 465, 502-506 (2010).
- Ghosh, I., Hamilton, A. D., Regan, L. Antiparallel leucine zipper-directed protein reassembly: Application to the green fluorescent protein. J Am Chem Soc. 122, 5658-5659 (2000).
- Patching, S. G. Surface plasmon resonance spectroscopy for characterisation of membrane protein-ligand interactions and its potential for drug discovery. Biochim Biophys Acta. 1838, 43-55 (2014).
- Kenworthy, A. K. Imaging protein-protein interactions using fluorescence resonance energy transfer microscopy. Methods. 24, 289-296 (2001).
- Wyatt, P. J. Light-Scattering and the Absolute Characterization of Macromolecules. Analytica Chimica Acta. 272 (93), 1-40 (1993).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr Opin Chem Biol. 10, 430-436 (2006).
- Fiala, G. J., Schamel, W. W., Blumenthal, B. Blue native polyacrylamide gel electrophoresis (BN-PAGE) for analysis of multiprotein complexes from cellular lysates. J Vis Exp. , (2011).
- Zou, H., Li, Y., Liu, X., Wang, X. An APAF-1.cytochrome c multimeric complex is a functional apoptosome that activates procaspase-9. J Biol Chem. 274, 11549-11556 (1999).
- Schagger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal Biochem. 217, 220-230 (1994).
- Walker, J. M. Nondenaturing polyacrylamide gel electrophoresis of proteins. Methods Mol Biol. 32, 17-22 (1994).
- Stoscheck, C. M. Quantitation of protein. Methods Enzymol. 182, 50-68 (1990).
- Wisskirchen, C., Ludersdorfer, T. H., Muller, D. A., Moritz, E., Pavlovic, J. Interferon-induced antiviral protein MxA interacts with the cellular RNA helicases UAP56 and URH49. J Biol Chem. 286, 34743-34751 (2011).
- Stertz, S., et al. Interferon-induced, antiviral human MxA protein localizes to a distinct subcompartment of the smooth endoplasmic reticulum. J Interferon Cytokine Res. 26, 650-660 (2006).
- Seddon, A. M., Curnow, P., Booth, P. J. Membrane proteins, lipids and detergents: not just a soap opera. Biochim Biophys Acta. 1666, 105-117 (2004).
- Kochs, G., Haener, M., Aebi, U., Haller, O. Self-assembly of human MxA GTPase into highly ordered dynamin-like oligomers. J Biol Chem. 277, 14172-14176 (2002).
- Kochs, G., Haller, O. GTP-bound human MxA protein interacts with the nucleocapsids of Thogoto virus (Orthomyxoviridae). J Biol Chem. 274, 4370-4376 (1999).
- Reichelt, M., Stertz, S., Krijnse-Locker, J., Haller, O., Kochs, G. Missorting of LaCrosse virus nucleocapsid protein by the interferon-induced MxA GTPase involves smooth ER membranes. Traffic. 5, 772-784 (2004).
- Wisskirchen, C., Ludersdorfer, T. H., Muller, D. A., Moritz, E., Pavlovic, J. The cellular RNA helicase UAP56 is required for prevention of double-stranded RNA formation during influenza A virus infection. J Virol. 85, 8646-8655 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon