Method Article
Empregando Digital Gota PCR para detectar mutações BRAF V600E em parafina-encaixados Linhas Celulares de Referência Padrão fixadas em formalina
Neste Artigo
Resumo
O objetivo deste vídeo é para demonstrar como executar extração de DNA automatizado a partir de linhas fixas em formalina e embebidos em parafina (FFPE) de referência padrão de células e de gotículas digital de análise de PCR (ddPCR) para detectar mutações raras em um ambiente clínico. Detecção de mutações em amostras de FFPE demonstra a utilidade clínica de ddPCR em amostras FFPE.
Resumo
ddPCR is a highly sensitive PCR method that utilizes a water-oil emulsion system. Using a droplet generator, an extracted nucleic acid sample is partitioned into ~20,000 nano-sized, water-in-oil droplets, and PCR amplification occurs in individual droplets. The ddPCR approach is in identifying sequence mutations, copy number alterations, and select structural rearrangements involving targeted genes. Here, we demonstrate the use of ddPCR as a powerful technique for precisely quantitating rare BRAF V600E mutations in FFPE reference standard cell lines, which is helpful in identifying individuals with cancer. In conclusion, ddPCR technique offers the potential to precisely profile the specific rare mutations in different genes in various types of FFPE samples.
Introdução
A acumulação de mutações genéticas nos genes reguladores chave altera a programação de células normais como a proliferação celular, diferenciação, sobrevivência e, levando a um cancro. A via RAS-RAF-cinase de MAP-medeia as respostas celulares a sinais de crescimento. Mutações BRAF oncogênico pode resultar de mutações driver no gene BRAF, que podem causar o BRAF oncoprotein para tornar-se hiperativa 2. As mutações no gene BRAF também resultar na sinalização a jusante através hiperactiva MEK e ERK 3, que, por sua vez, conduz ao crescimento celular e proliferação excessiva de forma independente do crescimento mediada pelo factor de regulação 4-6.
Várias ferramentas estão disponíveis para a mutação de DNA profiling, como em tempo real mutações BRAF V600E quantitativas na fixados em formol, embebidos em parafina (FFPE) de referência linhas de células normais por ddPCR. ddPCR é um método baseado na PCR, para a quantificação absolutaoferecendo maior precisão em comparação com em tempo real quantitativa de PCR convencional (qPCR) 7,8. ddPCR também oferece maior potência e precisão de resolução para a detecção de mutações raras em modelos de DNA, permitindo a pesquisa de cancro mais informativo e diagnóstico 9. Vantagens adicionais do ddPCR sobre qPCR convencional incluem a sua sensibilidade e precisão melhorada quando se estuda número de cópias baixo de modelo 10-12. Nisto, um protocolo de extracção automática de ADN a partir de linhas celulares FFPE padrão de referência, seguido pela determinação da presença ou ausência de mutações BRAF V600E por ddPCR é demonstrada. O uso de software para análise de dados e uma representação gráfica dos resultados são também descritos. Todo o procedimento é relativamente simples e completamente depende do número de amostras a serem perfiladas e o número de máquinas de PCR e ddPCR convencionais disponíveis.
O protocolo seguinte descreve os procedimentos padrão para BR Linhas celulares AF V600E-positivos FFPE de referência padrão (HD598, HD593, HD617, HD273 e do tipo selvagem (WT)) é realizado em um instrumento totalmente automatizada usando a preparação do sistema (TPS) protocolo de tecido. Subsequentemente, as amostras de ADN isoladas são analisadas quanto à presença de mutações de BRAF V600E usando o sistema ddPCR. Análise da mutação alvo é realizada após todas as amostras foram perfilado e os dados terem sido carregados no software de análise de dados. Dependendo do número de amostras / grupos estudados, a análise dos dados pode exigir a partir de uma a várias horas. O componente experimental da metodologia requer precisão na manipulação de ADN eneurológica Pipettes e Pipetadores, um vídeo de Educação Científica JoVE explicando mais sobre sobre o contexto de pipetagem "> pipetando para placas de 96 poços, enquanto a análise dos dados é realizada utilizando software.
Protocolo
1. Extração de DNA de linhas celulares FFPE Referência Padrão
Nota: Para este procedimento, a extracção do ADN foi realizada a partir de linhas celulares padrão de referência (FFPE HD598, HD593, HD617, HD273 e de tipo selvagem (WT)) usando o kit de isolamento de DNA de tecidos FFPE como descrito no protocolo abaixo. Extracção automática do DNA foi conseguida seguindo as instruções do fabricante para o isolamento de DNA total.
- Usando um micrótomo eo bloco FFPE original, preparar manualmente seções frescas antes da extração de DNA e análise, de acordo com os procedimentos estabelecidos. Assegurar que a amostra de entrada para a secção (s) tecido analisado não exceda uma espessura total de 10 um e que a área de superfície que não excede 600 mm2 para uma única secção.
1.2 protocolo TPS
Nota: As quantidades indicadas na Tabela 1 correspondem ao mínimo necessária para processar 48 amostras, e o procedimentoapresentadas estão de acordo com as diretrizes do TPS. Antes de iniciar o experimento estabelecer-se as amostras de FFPE em o e-tubo de centrifugação a 600 xg, para evitar a perda de amostras durante o programa automatizado.
- Ative o instrumento isolamento de ADN automatizada e computador. Abra o software de controle Run e inserir uma bandeja de carregamento automático em área de plataforma de carga do TPS.
- Distribuir reagentes em suas depressões correspondentes, como mostrado na Figura 1.
- Coloque as amostras (4 BRAF WT, BRAF V600E 50%, 5% e 1%) nas cremalheiras porta-amostras.
- Coloque as caixas de ponta nos cochos nas colunas 2 e 3 e confira as dicas para a presença de mais de um filtro por ponta, que irá abortar a execução.
- Assegurar a mistura adequada de tampão de lise e o tampão de lavagem, invertendo-lhes 3-5 vezes e carregá-los para as respectivas calhas na coluna 4 (Figura 1).
- Após a inversão de algumas vezes ou vort leveexing, carregar o tampão de eluição, esferas magnéticas, e tampão de FFPE em pequenas depressões na coluna 5, deixando um espaço vazio onde indicado (Figura 1, Tabela 1).
- Coloque 2 ml de uma placa de profundidade bem (DWP) sobre o suporte da placa (Figura 1).
- Execute os seguintes passos finais antes de iniciar uma execução:
- Uncap todos os tubos e as calhas reagentes. Confirme que a capacidade suficiente disponível no frasco de resíduos líquidos. Confirme se o frasco de resíduos sólidos está vazio e forrado com um saco de risco biológico. Confirme se a placa de ejeção de ponta é centrado na montagem de resíduos.
- Feche a tampa frontal.
- Inicie o software. Abra o arquivo NA_Prep_Main_MLSTARlet.med.
- Clique em "Iniciar". O status do instrumento vai mudar de marcha lenta até execução.
- Digite o número de amostras para essa execução. Escolha o método desejado para essa execução (DNA = 0). Introduza a posição da primeira alta volume ponta, a seleção de "1" se todas as bandejas estão cheias. Introduza a posição da primeira dica volume padrão, selecionando "1" se todas as bandejas estão cheias.
Nota: O instrumento será executado através de etapas automatizadas, sem intervenção do usuário. Um fluxo de trabalho detalhado é mostrado na Figura 2. Uma vez que o experimento é terminada, os resíduos de reagentes e as pontas são injectados para dentro do conjunto de resíduos. - Quantificar ADN genómico purificado, utilizando um método fluorométrico.
Nota: As amostras de DNA que contêm uma concentração mínima de 3,3 ng / mL são submetidos à análise ddPCR (seção 2).
2. Mutação DNA Profiling: ddPCR Protocol
Nota: O protocolo de mutação de DNA profiling consiste em 3 etapas principais: 1) geração gota, 2) amplificação convencional PCR, 3) leitura da gota e 4) mutação do DNA profiling.
2.1. Geração de gotículas
Nota: ddPCR é Supermixrecomendado para ddPCR, como esta mistura contém os reagentes necessários para a geração de gotículas.
- Para evitar a contaminação, siga as precauções padrão, como o uso de luvas, usando um capuz limpo PCR, pipetas limpas, e tubos de baixa ligação às proteínas.
- Assegure-se que a concentração mínima da amostra de DNA genómico humano é de 3,3 ng / uL. Nota: A quantidade de concentração purificado amostra de DNA varia de acordo com a análise de detecção de frequência de mutação%.
- Montar misturas reaccionais de PCR em placas de 96 poços. Descongelar e equilibrar os componentes da reacção à temperatura ambiente. Prepare reações de PCR, combinando 2X ddPCR supermix (10 ul) e 20 primers (frente e verso, a 900 nm) e sonda (250 milhas náuticas) com cada amostra de ADN purificado (66ng / 2 ul) fazer até 20 l com água destilada (como por o protocolo gerador de gota)
- Vortex a mistura cuidadosamente para assegurar a homogeneidade e centrifugar para recolher rapidamente os conteúdos ao fundo do tubo antes de dispensar. Carga 20 & #181, L para o cartucho para a formação de gotículas.
- A operação do gerador de gotícula de, acordo com o protocolo recomendado pelo fabricante
- Insira o cartucho no suporte com o entalhe do cartucho posicionado no lado superior esquerdo do titular. Adicionar 20 ul de mistura reaccional contendo amostras para o meio, e 70 ul de óleo de gerador para os poços de fundo.
- Fixar a junta em toda a parte superior do cartucho. Certifique-se de que a junta está firmemente viciado em ambas as extremidades do titular; de outra forma, uma pressão suficiente para a geração de gotículas não serão alcançados.
- Abra o gerador de gotículas ao pressionar o botão verde na parte superior do aparelho e inserir o cartucho. Quando o titular está na posição correta, tanto o poder (da esquerda para direita) e titular (médio direito) luzes indicadoras são verdes.
- Pressione o botão de cima do instrumento novamente para fechar a porta e iniciar a geração de gotículas. Nota: Depois de pressionar a button, ele próprio um múltiplas posições ao longo dos poços de saída, desenho petróleo e amostras através dos canais microfluídicos, onde gotículas são criados. Gotas fluir para a gota bem, onde se acumulam. A luz indicadora de gota (à direita) pisca em verde após 10 segundos para indicar que a geração de gota está em andamento.
- Quando a geração de gota está completa, todos os 3 luzes indicadoras de mudar para verde sólido; abrir a porta, pressionando o botão, e remova o suporte da unidade. Remover a vedação descartável do titular e descartá-lo. Nota: Os poços superior do cartucho conter gotículas, e os poços e inferiores são médias quase vazia, com uma pequena quantidade de óleo residual.
2.2. Preparação para a PCR
- Para cada amostra, pipetar a 40 ul do conteúdo das gotículas a partir do topo bem os cartuchos num único poço de uma placa de 96 poços de PCR recomendado como indicado no protocolo do aparelho do fabricante. NãoTE: Usando uma pipeta multi-canal é ideal para a transferência de gotas de emulsões. Aspiração lenta e suave de gotículas é recomendado para minimizar corte gotícula durante as transferências.
- Selar a placa PCR com a folha imediatamente após a transferência de gotas para evitar a evaporação. Use selos perfuráveis placa de folha que são compatíveis com o vedante de placa de PCR e as agulhas no leitor de gotícula. Siga as instruções do manual da placa PCR instrução selador.
- Ajustar a temperatura vedante de placa a 180 ° C e o tempo de 5 segundos.
- Toque na seta para abrir a porta da bandeja. Posicione o bloco de suporte na bandeja com o lado de 96 poços voltada para cima. Colocar a placa de 96 poços para o bloco de suporte e assegurar que todos os poços da placa são alinhados com o bloco de suporte.
- Cobrir a placa de 96 poços com uma folha de selagem de folha metálica perfurável. Uma vez que a placa de 96 poços é fixada no bloco de suporte e coberta com a folha de vedação perfurável, tocar no botão de vedação. O tabuleiro fecharáe vedação de calor irá iniciar.
- Quando selagem térmica estiver concluída, a porta se abre automaticamente; remova a placa do bloco de calor de ciclos térmicos e, em seguida, remover o bloco de calor.
- Assegurar que todos os poços da placa são vedados através da verificação de que as depressões na folha são facilmente aparentes ao longo cada poço. Uma vez selado, a placa está pronta para o ciclo térmico.
- Uma vez que as gotículas são removidos, pressionar as patilhas de suporte do cartucho para abri-la. Remova o cartucho vazio e descartá-lo.
- Realizar a amplificação por PCR convencional, seguindo os parâmetros descritos na Tabela 2.
2.3 leitura gota (conforme o protocolo recomendado pelo fabricante)
Nota: Após amplificação por PCR do ácido nucleico alvo nas gotículas, o instrumento leitor gota analisa cada gotícula individual utilizando um sistema de detecção de 2-13 cor. Nós normalmente definido para detectar um FAMd VIC fluorophores repórter.
- Clique em "sistema nivelado" para preparar o leitor gota e torná-lo pronto para a análise ddPCR.
- Coloque a placa no leitor de gota e clique em "Iniciar". O leitor gota aspira cada amostra, singulates as gotas, e córregos-los em um único arquivo passado um detector de 2 cores. O detector lê as gotículas para enumerar as amostras positivas e negativas.
- Quando leitura gota está completa, abra a porta e retire o suporte da placa da unidade. Remova a placa de 96 poços de PCR a partir do suporte e descartá-lo.
- Para uma manutenção adequada, substitua o óleo leitor gota e esvaziar o recipiente de resíduos, conforme necessário. Adicionar 50 ml de 10% de água sanitária para o frasco de resíduos para evitar o crescimento microbiano.
2.4 ADN mutação perfis (de acordo com o protocolo recomendado pelo fabricante)
Nota: Gotas de PCR-positivas e negativas por PCR são contados para fornecer Quantific absolutação de mutações BRAF V600E-alvo de DNA em forma digital, utilizando software de análise de dados.
- Clique em Configurar para inserir informações sobre as amostras, ensaios e experimentos.
- Na janela de configuração, coloque uma placa (filename.qlp), em seguida, clique em Analisar para abrir e analisar os dados. A interface de análise de dados é separado em três janelas: Resultados da Mesa, Bem Seletor e processou dados / gráfica de vídeo.
- Na janela de dados processados, os dados de concentração para cada alvo aparecem nos poços da placa mapa e estão tabelados na Tabela de Resultados. Clique Concentração de visualizar dados em parcelas de concentração.
Resultados
Pela nossa análise ddPCR, estudamos as linhas de células-padrão de referência mutação BRAF V600E FFPE. O leitor gota se conecta a um laptop software de análise de dados do computador de corrida. Cada gotícula individual é definido com base na amplitude fluorescente como sendo positivos ou negativos. O software fornecido pelo fabricante também permite um corte definido pelo usuário para ser inscrito para definir o limite entre as gotículas positivos e negativos. O número de gotículas de positivos e negativos em uma amostra é utilizada para calcular a concentração de alvo em termos de cópias / mL.
A fluorescência foi detectada e processada em um visor gráfico de dispersão bidimensional, software personalizado foi usada para desenhar portas apropriadas para cada agrupamento terminal de gotículas, e o número de gotículas dentro de cada porta foi contado. Como mostrado na Figura 3A, as gotas representado por pontos azuis (sinal de fluorescência de FAM) acima da linha de corte para todos SAMPLes (linha rosa) foram positivos para mutação BRAF V600E. Gotas representados por pontos azuis na BRAF WT (NTC; pista 2) amostra poderia ser devido a um sinal não específico (falsos positivos). Sinais falsos positivos (BRAF WT) foram normalizados com outras amostras de mutação. Tal como mostrado na Figura 3B, as gotas de WT BRAF são representados por pontos verde (sinal de fluorescência VIC). Em ambos os gráficos, os pontos cinzentos na parte inferior são considerados como a fluorescência de fundo. As frequências de alelos mutantes totais foram calculados usando os dados mostrados na Figura 3C, com base nas percentagens relativas de WT BRAF V600E no gene BRAF e modelos detectados. Resultados ddPCR obtidas contêm as contagens de eventos gota e calculado de tipo selvagem e a molécula de DNA mutante conta para o V600E BRAF (50%, 10%, 5%, 1%, 0,5%, 0,1% e 0,05%) as amostras calculadas usando a seguir fórmula mencionada.
% = Frequência de mutante (mutantecopy / (do tipo selvagem + cópia Mutant)) x 100
Consequentemente, mutações BRAF V600E foram identificadas e verificadas com padrão de referência (BRAF WT). BRAF V600E mutação definida frequências alélicas de 50%, 10%, 5%, 1%, 0,5%, 0,1% e 0,05% foram usadas para testar a sensibilidade e reprodutibilidade do sistema ddPCR. De nossa análise com concentrações das amostras conhecidas, confirmamos que ddPCR é capaz de detectar um preço tão baixo quanto 0,05% de mutação BRAF V600E. A detecção de contagem mutante falso positivo em NTC ou WT pode ser devido à hidrólise da sonda não específica conforme relatado anteriormente 14. A detecção de mais do que duas cópias de uma amostra foi considerada positiva em tecido tumoral 15.

Figura 1. Representação esquemática descrevendo reagente e carregamento de amostras em preparação para aut instrumento de extração de DNA omated. Colocar as amostras em um prateleiras de operadoras e dispensar os reagentes em depressões correspondentes como mencionado. Empregando protocolo TPS automatizado que oferece suporte a vários tipos de amostras, entrega precisas e resultados confiáveis com o máximo de produtividade. Re-impresso com a permissão da Siemens Healthcare Diagnostics. (cortesia da Siemens Healthcare Diagnostics).

Figura 2. Representação esquemática do tecido de preparação do sistema de fluxo de trabalho para a extração automatizada de DNA. Procedimento de isolamento de ADN totalmente automatizado para FFPE tecidos seções, incluindo medidas negativas de seleção de parafina, remoção de restos de tecido e as etapas de selecção positiva de ligação e eluição são mostrados. Re-impresso com a permissão da Siemens Healthcare Diagnostics. (cortesia da Siemens Healthcare Diagnostics).
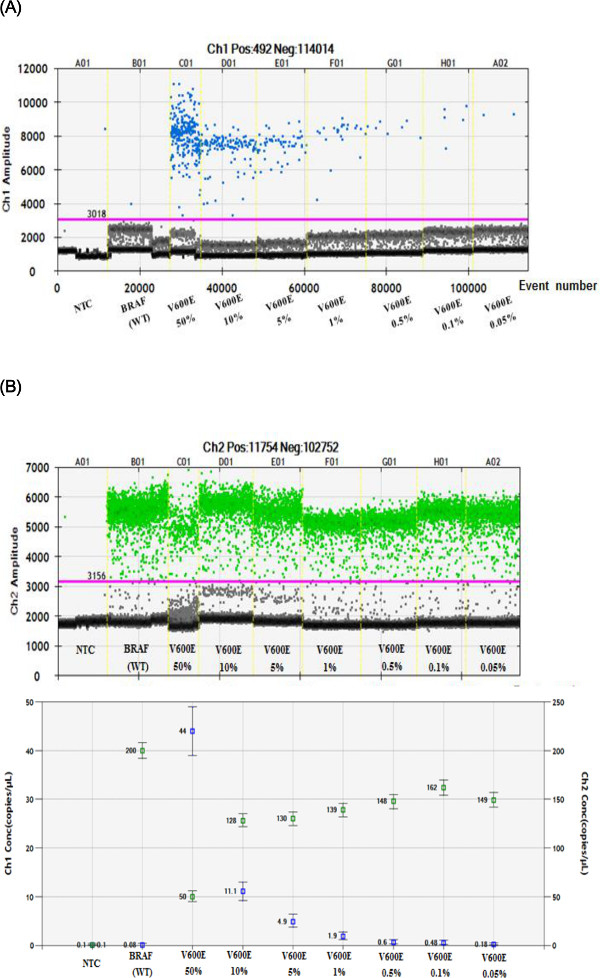
Figura 3. A utilização do sistema ddPCR para quantificação precisa do BRAF V600E mutação FFPE em amostras de referência da linha de células padrão. (A, B) Visualização das amplitudes de fluorescência positivos em parcelas 1D (1dot -1droplet). Os pontos azuis (A, FAM positivo) representam gotas V600E-positivo BRAF mutante, enquanto os pontos verdes (B, VIC positivo) representam WT BRAF gotículas -positivas. Esta determinação permite a quantificação precisa mutação em linhas celulares FFPE padrão de referência. A linha-de-rosa é o limiar de discriminação entre sinais positivos e negativos das gotículas. (C) A trama abundância fraccionada mostra marcadores azuis, que indicam a concentração (cópias / mL) de mutação BRAF V600E, eos marcadores verdes indicam a concentração (cópias / mL) de BRAF (WT). Todas as barras de erro gerados pelo software de análise de dados representam o intervalo de confiança de 95%.
| Reagentes | Volume (ml) |
| Tampão de Lise | 106 ml |
| Wash Buffer 1 | 101 ml |
| Wash Buffer 2 | 72 ml |
| Tampão de lavagem 3 | 106 ml |
| Tampão de Eluição | 19 ml |
| Esférulas magnéticas | 8 ml |
| Tampão FFPE | 15 ml |
| Proteinase K | 3,3 ml |
Tabela 1. O volume total de reagentes (kit TPS), necessários para a extração de DNA com 48 amostras.
| Ciclismo Etapa | Temperatura | Tempo | # Cycles |
| Activação da enzima | 95 ° C | 10 min | 1 |
| Desnaturação | 94 ° C | 30 seg | 40 |
| Recozimento / extensão | 60 ° C | 1 minuto * | |
| Aguarde | 98 ° C | 10 min | 1 |
| Aguarde | 4 ° C | Para sempre | 1 |
| * Ajuste as configurações da taxa de rampa de 2-2,5 ° C / seg. Usar uma tampa aquecida ajustado para 105 ° C e definir o volume da amostra de 40 ul | |||
Tabela 2. PCR convencional condições termociclagem
Discussão
Aqui, destaca-se a aplicabilidade do ddPCR e isolamento de DNA a partir de amostras de linhas celulares FFPE padrão de referência para uma avaliação específica mutação genética. Neste estudo, a TPS método de isolamento de DNA automatizado é utilizado, que pode ser facilmente adaptado, automatizada, e pode acomodar até 48 amostras diferentes simultaneamente, permitindo experiências escala maior e menor variabilidade. Uma das limitações do isolamento de ADN no presente trabalho é que todas as amostras FFPE é único, e irá variar um do outro na contaminantes da superfície, a flora microbiana, e / ou os fundos genéticos humanos. Em geral, a qualidade e quantidade do DNA extraído e o sucesso de amplificação de ADN genómico total estão dependentes de vários parâmetros, antes, durante e depois da extracção. Estes incluem, tipo e quantidade de tecido, tipo de fixador utilizado para a preservação do tecido, duração da fixação, da idade das condições de armazenamento e de blocos de parafina, bem como o comprimento do segmento de ADN desejado a ser analisado 16. Remoção de parafina do tecido é o passo mais importante para extração bem sucedida como a parafina não dissolvido leva a má qualidade da amostra. Durante a geração de gotículas, deve ser tomado cuidado para prevenir a formação de bolhas - este é um passo crítico para a detecção de mutações bem sucedidas. Considerando-se a amostra para amostra variação que possa surgir entre as populações da amostra e com base no motivo do experimento, certas modificações no procedimento pode ser necessária para obter resultados desejados.
Outra vantagem é que o isolamento de ADN e ddPCR é conduzida usando sistemas automatizados neste protocolo, e, portanto, não há erro insignificante e intervenção do usuário necessária é mínima. Isolamento de todo o genoma-DNA amplificado a partir de tecido / células embebidas em parafina foi obtido usando o sistema de TPS. Uma das desvantagens no uso de sistema de isolamento de DNA automatizado que é, ele não é eficiente para usar pequeno número de amostras. Em vez deste ste automatizadoP, outro procedimento de isolamento de ADN convencional também pode ser realizada para amostras limitadas
Um estudo recente indicou que o uso de gotícula de PCR digital (ddPCR) é capaz de determinar o número de loci em relação cópia genómica específica mesmo na presença de tecido normal entremeado obtido a partir de tecidos FFPE. Ao utilizar uma série de diluições de controlo, Nadauld, L. et al., Determinaram os limites de detecção (LOD) para o ensaio ddPCR e relataram a sua sensibilidade melhorada em quantidades mínimas de ADN em comparação com o padrão PCR em tempo real 17. Aqui, as linhas celulares padrão FFPE de referência são usados para demonstrar a capacidade de detecção de mutação do sistema ddPCR. Os resultados do sistema de ddPCR indicaram a possibilidade de detectar as frequências alélicas mutação rara mutação até 0,05%. Colectivamente, estes dados indicam que o sistema ddPCR também permite a análise quantitativa da percentagem de vários alelos mutantes e as diferenças relativas na amostra de tumor clínica heterozigóticas. Um grande número de amostras de FFPE pode ser analisado quanto a mutações de genes específicos em simultâneo e isto é uma técnica optimizada para largura estudos genéticos populacionais.
Finalmente, deve ser tido em conta que as frequências de mutação são aqui representados são quantificação absoluta e não devem ser considerados como valores relativos da taxa de mutação ou de frequência. ddPCR leitura fornece quantificação absoluta de mutação do DNA alvo. Estes valores podem ser usados para a validação de frequências de mutação de amostras preparadas sob a mesma condição, e sequenciados através da mesma região. No entanto, estes valores absolutos são reprodutíveis e pode ser utilizado para comparação quantitativa da distribuição de frequências de mutação e quando os parâmetros óptimos são controlados. Em conclusão, ddPCR emergiu recentemente como uma ferramenta robusta que dá a quantificação absoluta de ácidos nucleicos em amostras de biópsias e FFPE e também pode ser duplex com os ensaios de referência para a determinação de ambos os nenhumAs concentrações de transcrição rmalized ou do número de cópias de DNA.
Divulgações
Myung Ryuri Oh, Si Kim Eun, e Young Kim DEUG são funcionários da ABION CRO.
Agradecimentos
Esta pesquisa foi apoiada pelo Programa de P & D para a Sociedade da Fundação Nacional de Pesquisa (NRF), financiado pelo Ministério da Ciência, TIC e Planejamento Futuro (Grant No. 2013M3C8A1075908).
Materiais
| Name | Company | Catalog Number | Comments |
| Hamilton MICROLAB STARlet IVD instrument | Siemens | 10701001 | Automated DNA isolation instrument |
| QX200 Droplet Generator | Bio-Rad | 772BR1119 | |
| QX200 Droplet Reader | Bio-Rad | 771BR1497 | |
| Conventional PCR machine capable of ramp-time adjustment | 621BR17718 | ||
| PX1 PCR plate sealer | Bio-Rad | 770BR1575 | |
| QuantaSoft software | Bio-Rad | ||
| DNA isolation kit | |||
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632398 | |
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632399 | |
| CO-RE tips | Siemens | ||
| ddPCR mutation analysis | |||
| ddPCR Supermix | Bio-Rad | BR186-3010 | 2X concentration |
| DG8 cartridge | Bio-Rad | BR186-4008 | |
| Droplet Generator oil | Bio-Rad | BR-186-3005 | |
| Gasket | Bio-Rad | BR186-3006 | |
| Droplet reader oil | Bio-Rad | BR-186-3004 |
Referências
- Vogelstein, B., et al. Genetic alterations during colorectal-tumor development. The New England journal of medicine. 319, 525-532 (1988).
- Davies, H., et al. Mutations of the BRAF gene in human cancer. Nature. 417, 949-954 (2002).
- Solit, D. B., et al. BRAF mutation predicts sensitivity to MEK inhibition. Nature. 439, 358-362 (2006).
- Wong, K. K. Recent developments in anti-cancer agents targeting the Ras/Raf/ MEK/ERK pathway. Recent patents on anti-cancer drug discovery. 4, 28-35 (2009).
- Brose, M. S., et al. BRAF and RAS mutations in human lung cancer and melanoma. Cancer research. 62, 6997-7000 (2002).
- Wang, L., et al. BRAF mutations in colon cancer are not likely attributable to defective DNA mismatch repair. Cancer research. 63, 5209-5212 (2003).
- Hindson, B. J., et al. High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal Chem. 83, 8604-8610 (1021).
- Pinheiro, L. B., et al. Evaluation of a Droplet Digital Polymerase Chain Reaction Format for DNA Copy Number Quantification. Anal Chem. 84, 1003-1011 (1021).
- Jones, M., et al. Low copy target detection by Droplet Digital PCR through application of a novel open access bioinformatic pipeline, 'definetherain'. Journal of virological methods. 202, 46-53 (2014).
- Strain, M. C., et al. Highly precise measurement of HIV DNA by droplet digital PCR. PloS one. 8, e55943(2013).
- Miotke, L., Lau, B. T., Rumma, R. T., Ji, H. P. High sensitivity detection and quantitation of DNA copy number and single nucleotide variants with single color droplet digital PCR. Anal Chem. 86, 2618-2624 (2014).
- Bizouarn, F. Clinical applications using digital PCR. Methods in molecular biology. 1160, 189-214 (2014).
- McDermott, G. P., et al. Multiplexed target detection using DNA-binding dye chemistry in droplet digital PCR. Anal Chem. 85, 11619-11627 (2013).
- Milbury, C. A., et al. Determining lower limits of detection of digital PCR assays for cancer-related gene mutations. Biomolecular detection and quantification. 1, 8-22 (2014).
- Zhu, G., et al. Highly Sensitive Droplet Digital PCR Method for Detection of EGFR Activating Mutations in Plasma Cell-Free DNA from Patients with Advanced Non-Small Cell Lung Cancer. The Journal of molecular diagnostics: JMD. , (2015).
- Fan, H., Gulley, M. L. DNA extraction from paraffin-embedded tissues. Methods in molecular medicine. 49, 1-4 (2001).
- Nadauld, L., et al. Quantitative and Sensitive Detection of Cancer Genome Amplifications from Formalin Fixed Paraffin Embedded Tumors with Droplet Digital PCR. Translational medicine. 2, (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados