Method Article
Verwendung digitaler Droplet PCR zu BRAF V600E Mutationen in Formalin-fixierte Paraffin Reference Standard-Zelllinien Detect
In diesem Artikel
Zusammenfassung
Das Ziel dieses Video ist zu zeigen, wie automatisierte DNA-Extraktion aus Formalin fixierten und in Paraffin eingebetteten (FFPE) Referenz-Standard-Zelllinien und digitale Tröpfchen PCR (ddPCR) Analyse seltene Mutationen in einer klinischen Umgebung detektieren auszuführen. Nachweis von Mutationen in FFPE-Proben zeigt, die den klinischen Nutzen ddPCR in FFPE-Proben.
Zusammenfassung
ddPCR is a highly sensitive PCR method that utilizes a water-oil emulsion system. Using a droplet generator, an extracted nucleic acid sample is partitioned into ~20,000 nano-sized, water-in-oil droplets, and PCR amplification occurs in individual droplets. The ddPCR approach is in identifying sequence mutations, copy number alterations, and select structural rearrangements involving targeted genes. Here, we demonstrate the use of ddPCR as a powerful technique for precisely quantitating rare BRAF V600E mutations in FFPE reference standard cell lines, which is helpful in identifying individuals with cancer. In conclusion, ddPCR technique offers the potential to precisely profile the specific rare mutations in different genes in various types of FFPE samples.
Einleitung
Die Akkumulation von genetischen Mutationen in regulatorischen Schlüsselgenen verändert normalen Zellprogrammierung wie Zellproliferation, Differenzierung und das Überleben, die zu Krebs führen. 1 Die RAS-RAF-MAP-Kinase-Weg vermittelt zellulären Reaktionen auf Wachstumssignale. Onkogenen BRAF-Mutationen können vom Fahrer Mutationen des BRAF-Gens, der die BRAF oncoprotein verursachen können überaktiven 2 zu werden, zur Folge haben. Mutationen im BRAF-Gen auch in überaktiven nachgeschalteten Signal über MEK und ERK 3, die wiederum führt zu einer übermäßigen Zellwachstum und -proliferation unabhängig Wachstumsfaktor-vermittelten Regulation 4-6 führen.
Mehrere Werkzeuge sind für die DNA-Mutation Profilierung zur Verfügung, wie quantitative Echtzeit-BRAF-V600E-Mutationen in Formalin fixierten und in Paraffin eingebetteten (FFPE) Referenz-Standard-Zelllinien durch ddPCR. ddPCR ist eine PCR-basierte Methode zur absoluten Quantifizierungbietet eine höhere Genauigkeit im Vergleich zu herkömmlichen quantitative Echtzeit-PCR (qPCR) 7,8. ddPCR auch höheren Auflösungsvermögen und die Genauigkeit für die Detektion von seltenen Mutationen in DNA-Matrizen, so dass mehr informative Krebsforschung und Diagnose 9. Weitere Vorteile gegenüber herkömmlichen ddPCR qPCR sind seine verbesserte Empfindlichkeit und Genauigkeit bei der Untersuchung niedrigen Vorlage Kopienzahlen 10-12. Hierin ist ein Protokoll für die automatische Extraktion von DNA aus FFPE Referenzstandard Zellinien, gefolgt von der Bestimmung der Anwesenheit oder Abwesenheit von Mutationen durch BRAF V600E ddPCR demonstriert. Die Verwendung von Software für die Datenanalyse und eine grafische Darstellung der Ergebnisse werden ebenfalls beschrieben. Das gesamte Verfahren ist relativ einfach und hängt von der Anzahl von Proben, die profiliert werden und die Anzahl der konventionellen PCR und ddPCR Maschinen erhältlich völlig.
Das folgende Protokoll beschreibt Standardverfahren für die BR AF V600E-positive FFPE Referenz-Standard-Zelllinien (HD598, HD593, HD617, HD273 und Wildtyp (WT)) in einem vollautomatischen Instrument mit Hilfe des Tissue Preparation System (TPS) Protokoll. Anschließend werden isolierte DNA-Proben auf die Anwesenheit von Mutationen unter Verwendung BRAF V600E ddPCR System analysiert. Gezielte Mutation Analyse durchgeführt wird, nachdem alle Proben profiliert und die Daten in der Datenanalyse-Software geladen ist. Abhängig von der Anzahl der Proben / Gruppen untersucht, Datenanalyse kann von einem bis zu mehreren Stunden dauern. Die experimentelle Komponente der Methodik erfordert Genauigkeit bei der Handhabung DNA undlogischer Pipetten und Pipetten, ein JoVE Science Education Video erklärt mehr über über die Kontext Pipettieren "> Pipettieren in 96-Well-Platten, während der Datenanalyse wird unter Verwendung von Software.
Protokoll
1. DNA-Extraktion aus FFPE Reference Standard-Zelllinien
Hinweis: Bei diesem Verfahren wurde die DNA-Extraktion aus FFPE Referenz-Standard-Zelllinien (HD598, HD593, HD617, HD273 und Wildtyp (WT)) unter Verwendung der FFPE Gewebe-DNA-Isolation Kit, wie in der unten beschriebenen Protokoll durchgeführt. Automatisierte DNA-Extraktion wurde nach Angaben des Herstellers für die gesamte DNA-Isolierung erreicht.
- Verwendung eines Mikrotoms und die ursprüngliche FFPE Block manuell frische Abschnitte vor der DNA-Extraktion und Analyse vorzubereiten, nach festgelegten Verfahren. Dass die Probe-Eingang für den Gewebeabschnitt (en) analysiert nicht eine kombinierte Gesamtdicke von 10 & mgr; m überschreitet und die Fläche nicht 600 mm 2 für einen einzigen Abschnitt überschreiten.
1.2 TPS-Protokoll
Hinweis: Die in Tabelle 1 angegebenen Mengen entsprechen der erforderlich ist, um 48 Proben zu verarbeiten Minimum, und das Verfahrendargestellt ist in Übereinstimmung mit den TPS-Richtlinien. Vor dem Start des Experiments nieder die FFPE-Proben in der E-Rohr durch Zentrifugation bei 600 · g, um den Verlust von Proben während der automatisiertes Programm zu vermeiden.
- Schalten Sie die automatisierte DNA-Isolierung Instrument und Computer. Öffnen Sie den Run Steuerungssoftware und legen Sie eine automatische Last Fach in den TPS-Deck Ladefläche.
- Verzichtet Reagenzien in die entsprechenden Rinnen wie in Abbildung 1 dargestellt.
- Legen Sie die 4 Proben (BRAF-WT, BRAF V600E 50%, 5% und 1%) in den Probenträger-Racks.
- Setzen Sie die Spitze-Boxen in den Trögen in den Spalten 2 und 3 und überprüfen Sie die Tipps für die Anwesenheit von mehr als einem Filter pro Spitze, die den Lauf abbrechen wird.
- Stellen Sie sicher, die richtige Mischung des Lysepuffer und Waschpuffer durch Umdrehen sie 3-5 mal und laden Sie sie in die entsprechenden Mulden in Spalte 4 (Abbildung 1).
- Nach Invertierung für einige Male oder mild vortExing, laden Sie das Elutionspuffer, magnetische Kügelchen, und FFPE-Puffer in den kleinen Mulden in Spalte 5, wobei 1-Slot leer ist, wo angegeben (Abbildung 1, Tabelle 1).
- Legen Sie eine 2 ml Deep Well Platte (DWP) auf den Plattenträger (Abbildung 1).
- Führen Sie die folgenden letzten Schritte vor dem Start eines Lauf:
- Uncap alle Rohre und Reagenzien Tröge. Bestätigen Sie, dass ausreichende Kapazitäten in der flüssigen Abfallflasche erhältlich. Bestätigen, dass die Abfallflasche leer ist und mit einem Biohazard Tasche ausgekleidet. Bestätigen Sie, dass die Spitze Auswurfplatte wird in der Abfalleinheit zentriert ist.
- Schließen Sie die vordere Abdeckung.
- Starten Sie die Software. Öffnen Sie die NA_Prep_Main_MLSTARlet.med Datei.
- Klicken Sie auf "Start". Der Gerätestatus wird vom Leerlauf zum Laufen wechseln.
- Geben Sie die Anzahl der Proben für diesen Lauf. Wählen Sie die gewünschte Methode für diesen Lauf (DNA = 0). Geben Sie die Position des ersten Hoch volume Spitze, die Auswahl von "1", wenn alle Fächer voll sind. Geben Sie die Position des ersten Standard-Volumenspitze, die Auswahl von "1", wenn alle Fächer voll sind.
Hinweis: Das Gerät wird dann durch automatisierte Schritte laufen ohne Benutzereingriff. Eine detaillierte Arbeitsablauf ist in Abbildung 2 dargestellt. Wenn das Experiment beendet ist, werden Reagenz Abfälle und Tipps in den Abfallanordnung injiziert. - Quantifizierung gereinigter genomischer DNA unter Verwendung eines fluorometrischen Verfahrens.
Anmerkung: DNA-Proben, die eine Mindestkonzentration von 3,3 ng / & mgr; l enthalten, werden ddPCR Analyse (Teil 2) unterworfen.
2. DNA-Mutation Profilierung: ddPCR Protocol
Hinweis: Das Protokoll für DNA-Mutation Profilierung aus 3 Hauptschritten: 1) Tröpfchenerzeugung, 2) Konventionelle PCR-Amplifikation, 3) Tröpfchen Lesen und 4) DNA-Mutation Profilierung.
2.1. Tröpfchenerzeugung
Hinweis: ddPCR Supermix istempfohlen für ddPCR, da diese Mischung enthält Reagenzien für Tröpfchenerzeugung erforderlich.
- Um Verunreinigungen zu vermeiden, befolgen Sie Standard-Sicherheitsvorkehrungen, wie das Tragen von Handschuhen, mit einem sauberen PCR Haube, saubere Pipetten und low-Protein-bindenden Rohre.
- Sicherzustellen, daß die Mindestkonzentration von humanen genomischen DNA-Probe beträgt 3,3 ng / ul. Anmerkung: Die Menge an gereinigter DNA-Probenkonzentration wird auf der Analyse% Mutationsfrequenz Detektions variieren.
- Montieren Reaktionsmischungen in 96-Well-PCR-Platten. Auftauen und ins Gleichgewicht die Reaktionskomponenten auf RT. Bereiten PCR-Reaktionen durch die Kombination von 2X ddPCR Supermix (10 ul) und 20 Primer (vorwärts und rückwärts, 900 nM) und Sonde (250 nm) mit jedem gereinigten DNA-Probe (66ng / 2 ul) machen bis zu 20 & mgr; l mit destilliertem Wasser (als pro Tröpfchenerzeugers Protokoll)
- Vortexen Mischung gründlich bis zur Homogenität und kurz zentrifugieren, sicherzustellen, um den Inhalt am Boden des Rohres vor der Ausgabe zu sammeln. Last 20 & #181; l auf die Kartusche für die Tröpfchenbildung.
- Betrieb des Tropfengenerators, der nach Hersteller empfohlenen Protokoll
- Setzen Sie die Patrone in den Halter mit der Kerbe in der Patrone auf der oberen linken Seite des Halters positioniert. In 20 ul Reaktionsmischung, die Proben in der Mitte, und 70 & mgr; l-Generator Öl in den Boden Brunnen.
- Bringen Sie die Dichtung auf der Oberseite der Patrone. Stellen Sie sicher, dass die Dichtung fest eingehakt an beiden Enden des Inhabers; andernfalls wird ein ausreichender Druck zur Tropfenerzeugung nicht erreicht werden.
- Öffnen Sie den Tropfengenerator durch Drücken der grünen Taste auf der Oberseite des Gerätes und setzen Sie die Patrone. Wenn der Halter in der richtigen Position, sowohl die Leistung (links nach rechts) und Halter (Mitte rechts) leuchtet grün.
- Drücken Sie die obere Taste auf dem Gerät erneut, um die Tür zu schließen und zu initiieren Tropfenerzeugung. Hinweis: Nach dem Drücken der button, einem Verteiler positioniert sich über die Austrittsbohrungen, Ziehöl und Proben durch die mikrofluidischen Kanälen, in denen Tröpfchen erzeugt werden. Droplets fließen in den Tröpfchen gut, wo sie akkumulieren. Die Tröpfchenkontrollleuchte (rechts) blinkt grün, nach 10 Sekunden, um anzuzeigen, dass Tropfenerzeugung ist im Gange.
- Wenn Tröpfchenerzeugung abgeschlossen ist, wechseln alle 3-Anzeige leuchtet dauerhaft grün; öffnen Sie die Tür durch Drücken der Taste, und nehmen Sie die Halterung vom Gerät ab. Entfernen Sie die Einwegdichtung aus der Halterung und entsorgen Sie sie. Hinweis: Die oberen Vertiefungen der Patrone enthalten, Tröpfchen, und die mittleren und unteren Brunnen sind fast leer, mit einer kleinen Menge an Restöl.
2.2. Vorbereitung für die PCR
- Für jede Probe pipettieren Sie 40 ul der Tröpfchen Inhalt von oben auch die Patronen in einer einzigen Vertiefung einer empfohlenen 96-Well-PCR-Platte wie in Instrumentenprotokoll des Herstellers angegeben. Neinte: Unter Verwendung einer Mehrkanalpipette ist ideal zum Übertragen der Tröpfchen Emulsionen. Langsam und sanft Aspiration von Tröpfchen wird empfohlen, Tropfenscher bei der Übertragung zu minimieren.
- Verschließen Sie die PCR-Platte mit Folie sofort nach dem Übertragen von Tröpfchen, um Verdunstung zu vermeiden. Verwenden durchstechbare Folie Platte Dichtungen, die mit dem PCR Abklebefolie und den Nadeln in der Tröpfchen Reader kompatibel sind. Folgen Sie den Anweisungen in der PCR-Plattenversiegelung Gebrauchsanleitung.
- Stellen Sie die Plattenversiegelung Temperatur bis 180 ° C und die Zeit, um 5 Sek.
- Tippen Sie auf den Pfeil, um das Fach Tür zu öffnen. Positionieren Sie den Trägerblock auf dem Tablett mit dem 96-Loch-Seite nach oben. Legen Sie die 96-Lochplatte auf dem Trägerblock und sicherzustellen, dass alle Plattenvertiefungen werden mit dem Trägerblock ausgerichtet ist.
- Decken Sie die Platte mit 96 Vertiefungen mit 1 Blatt durchstechbare Foliendichtung. Sobald die 96-Lochplatte auf dem Trägerblock befestigt ist und mit dem durchstechbaren Siegelfolie abgedeckt, berühren Sie die Dichtung Taste. Die Schublade wird geschlossenund Heißsiegeln wird zu initiieren.
- Beim Heißsiegeln abgeschlossen ist, öffnet sich die Tür automatisch; entfernen Sie die Platte aus dem Wärmeblock für Temperaturwechsel und dann den Wärmeblock.
- Sicherstellen, dass alle Vertiefungen in der Platte werden durch Überprüfen, dass Vertiefungen in der Folie gibt über jedem offensichtlich gut abgedichtet. Einmal versiegelt ist die Platte bereit für thermische Zyklen.
- Sobald Tröpfchen entfernt werden, drücken Sie die Verriegelungen an den Patronenhalter, um sie zu öffnen. Entfernen Sie die leere Kartusche und entsorgen Sie sie.
- Führen herkömmliche PCR-Amplifikation durch Befolgen der in Tabelle 2 beschriebenen Parametern.
2.3 Droplet Lesung (gemäß dem vom Hersteller empfohlenen Protokoll)
Anmerkung: Nach der PCR-Amplifikation der Zielnukleinsäure in den Tröpfchen, analysiert die Tröpfchenleser Instrument jedes Tröpfchen einzeln mit einem 2-Farbdetektionssystem 13. Wir in der Regel eingestellt, um eine FAM erkennend VIC Reporter Fluorophore.
- Klicken Sie auf "System spülen" prime die Tröpfchen Leser und machen sie bereit für ddPCR Analyse.
- Legen Sie die Platte in die Tröpfchen Leser und klicken Sie auf "Start". Der Tröpfchen Leser saugt jede Probe, die vereinzelt Tropfen und Ströme sie im Gänsemarsch vorbei an einer 2-Farben-Detektor. Der Detektor liest die Tröpfchen auf positive und negative Proben aufzuzählen.
- Wenn Tröpfchen Lesen abgeschlossen ist, die Tür öffnen und die Plattenhalter aus dem Gerät zu entfernen. Entfernen Sie den 96-Well-PCR-Platte aus der Halterung und entsorgen Sie sie.
- Für eine ordnungsgemäße Wartung, ersetzen Sie die Tröpfchen Leser Öl und leeren Sie den Abfallbehälter nach Bedarf. In 50 ml 10% Bleichmittel auf die Abfallflasche, um mikrobielles Wachstum zu verhindern.
2.4 DNA-Mutation Profilierung (gemäß dem vom Hersteller empfohlenen Protokoll)
Hinweis: PCR-positiv und PCR-negative Tröpfchen werden gezählt, um absolute quantific liefernation von Ziel BRAF V600E DNA-Mutationen in digitaler Form unter Verwendung der Datenanalyse-Software.
- Klicken Sie auf Setup, um Informationen über die Proben, Tests und Experimente geben.
- Im Setup-Fenster, laden Sie eine Platte (filename.qlp), klicken Sie dann auf Analysieren Sie zu öffnen und die Daten analysieren. Ergebnisse Tabelle, Well Selector und verarbeiteten Daten / Grafische Darstellung: Die Analyse der Daten-Schnittstelle ist in drei Fenster getrennt.
- In der verarbeiteten Daten-Fenster, Konzentrationsdaten für jedes Ziel werden in den Vertiefungen in der Platte Karte und werden in der Ergebnistabelle aufgeführt. Klicken Konzentration, um Daten in der Konzentration Plots visualisieren.
Ergebnisse
Für unsere ddPCR Analyse, untersuchten wir die BRAF-V600E-Mutation FFPE Referenz-Standard-Zelllinien. Die Tröpfchen Leser verbindet sich mit einem Laptop-Computer mit Datenanalyse-Software. Jeder einzelne Tropfen wird auf der Basis der Fluoreszenzamplitude als entweder positiv oder negativ definiert. Die vom Hersteller bereitgestellten Software ermöglicht außerdem ein benutzerdefinierter Grenz eingegeben werden, um den Grenzbereich zwischen der positiven und negativen Tröpfchen zu definieren. Die Anzahl der positiven und negativen Tröpfchen in einer Probe verwendet wird, um die Konzentration der Zielsequenz in Bezug auf die Kopien / ul zu berechnen.
Die Fluoreszenz wurde erfasst und in ein zweidimensionales Streudiagramm-Anzeige verarbeitet, kundenspezifische Software wurde verwendet, um entsprechende Gates für jedes Tröpfchen Endpunkt Cluster zu ziehen, und die Anzahl der Tröpfchen in jedem Tor wurde gezählt. Wie in 3A gezeigt, Tröpfchen durch blaue Punkte (FAM Fluoreszenzsignal) über dem Cut-off-Linie für alle sampl vertretenES (rosa Linie) waren positiv für mutierte BRAF V600E. Tröpfchen durch blaue Punkte im BRAF WT dargestellt (NTC; Spur 2) Probe konnte aufgrund einer unspezifischen Signal sein (false positive). Falsch positive Signale (BRAF WT) wurden mit anderen Mutation Proben normalisiert. Wie in 3B gezeigt, sind WT BRAF Tröpfchen durch grüne Punkte (VIC Fluoreszenzsignal) repräsentiert. In beiden Stellplätzen werden die Grau Punkte unten als Fluoreszenzhintergrund betrachtet. Die Gesamt Mutante Allelhäufigkeiten wurden unter Verwendung der in Figur 3C gezeigten Daten, basierend auf den relativen Prozentsätze der WT BRAF und BRAF V600E Schablonen detektiert. Erhalten ddPCR Ergebnisse enthalten die Tröpfchen Event zählt und berechnet Wildtyp und mutierten DNA-Molekül, zählt für die BRAF-V600E (50%, 10%, 5%, 1%, 0,5%, 0,1% und 0,05%) Proben mit Hilfe der nachstehend berechnet genannten Formel.
% Der Mutantenhäufigkeit = (MutantKopieren / (Wildtyp + Mutant Kopie)) x 100
Dementsprechend wurden BRAF V600E-Mutationen identifiziert und mit Referenzstandard (BRAF WT) verifiziert. BRAF-Mutation V600E definiert Allelhäufigkeiten von 50%, 10%, 5%, 1%, 0,5%, 0,1% und 0,05% wurden verwendet, um die Empfindlichkeit und Reproduzierbarkeit des ddPCR System zu testen. Aus unserer Analyse mit bekannten Probenkonzentrationen, wir bestätigt, dass ddPCR der Lage ist, so niedrig wie 0,05% des BRAF-V600E-Mutation zu erkennen. Die Detektion von falsch-positiven Mutanten-Zählung in NTC oder WT könnte möglicherweise aufgrund nicht-spezifischen Sonde Hydrolyse sein, wie bereits berichtet 14. Nachweis von mehr als zwei Kopien in einer Probe als positiv in Tumorgewebe 15 betrachtet.

Abbildung 1. Schematische Darstellung der Beschreibung Reagenz und Probenbeladung in der Vorbereitung für aut omated DNA-Extraktion Instrument. Setzen Sie die Proben in einem Trägergestelle und verzichten die Reagenzien in entsprechende Mulden, wie erwähnt. Der Einsatz automatisierter TPS-Protokoll, das mehrere Probentypen unterstützt, liefert genaue und zuverlässige Ergebnisse bei maximaler Produktivität. Mit freundlicher Genehmigung von Siemens Healthcare Diagnostics neu gedruckt. (mit freundlicher Genehmigung von Siemens Healthcare Diagnostics).

Abbildung 2. Schematische Darstellung der Gewebepräparation Systemworkflow für die automatisierte DNA-Extraktion. Voll automatisierte DNA-Isolierung Verfahren für FFPE-Geweben Abschnitte einschließlich der negativen Selektionsschritte von Paraffin, Gewebereste entfernen und positiven Selektionsschritten der Bindung und Elution werden angezeigt. Mit freundlicher Genehmigung von Siemens Healthcare Diagnostics neu gedruckt. (mit freundlicher Genehmigung von Siemens Healthcare Diagnostics).
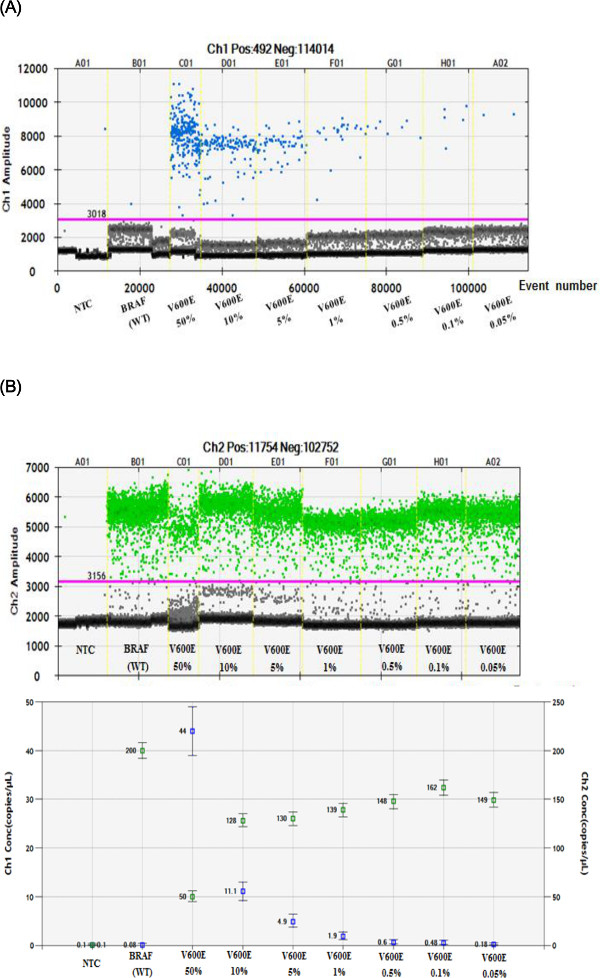
Abbildung 3. Verwendung des ddPCR System für genaue Quantifizierung des BRAF V600E Mutation in FFPE Referenzstandard Zelllinie Proben. (A, B) Visualisierung von positiven Fluoreszenzamplituden in 1D Grundstücke (1dot -1droplet). Blaue Punkte (A, FAM positiv) stellen mutierten BRAF V600E-positive Tröpfchen, während grüne Punkte (B, VIC positiv) stellen WT BRAF-positiven Tröpfchen. Diese Bestimmung ermöglicht eine präzise Quantifizierung Mutation in FFPE Referenz-Standard-Zelllinien. Die rosa Linie ist die Unterscheidungsschwelle zwischen positiven und negativen Signalen der Tröpfchen. (C) Der Bruch Fülle Plot zeigt blaue Markierungen, die die Konzentration anzugeben (Kopien / ul) des BRAF-V600E-Mutation, unddie grünen Markierungen zeigen die Konzentration (Kopien / ul) BRAF (WT). Alle Fehlerbalken von Datenanalyse-Software generiert stellen die 95% Konfidenzintervall.
| Reagenzien | Volumen (ml) |
| Lysepuffer | 106 ml |
| Wash Buffer 1 | 101 ml |
| Wash Buffer 2 | 72 ml |
| Waschpuffer 3 | 106 ml |
| Elution Buffer | 19 ml |
| Magnetische Kügelchen | 8 ml |
| FFPE-Puffer | 15 ml |
| Proteinase K | 3,3 ml |
Tabelle 1 Gesamtvolumen der Reagenzien (TPS-Kit) für die DNA-Extraktion mit 48 Proben erforderlich.
| Radfahren Schritt | Temperatur | Zeit | # Cycles |
| Enzymaktivierung | 95 ° C | 10 min | 1 |
| Denaturierung | 94 ° C | 30 sec | 40 |
| Annealing / Erweiterung | 60 ° C | 1 Minute * | |
| Halten | 98 ° C | 10 min | 1 |
| Halten | 4 ° C | Ewig | 1 |
| * Stellen Rampenrate Einstellungen um 2-2,5 ° C / s. Verwenden Sie einen Heizdeckel bis 105 ° C eingestellt und stellen Sie den Probenvolumen auf 40 ul | |||
Tabelle 2. Herkömmliche PCR Thermocycling-Bedingungen
Diskussion
Hier beleuchten wir die Anwendbarkeit der ddPCR und DNA-Isolierung aus FFPE Referenzstandard Zelllinie Proben für eine bestimmte Genmutation Beurteilung. In dieser Studie wird TPS automatischen DNA-Isolierungsverfahren verwendet werden, der leicht angepasst werden kann, automatisiert, und kann bis zu 48 verschiedene Proben gleichzeitig unterzubringen, so dass für größeren Maßstab Experimente und niedrigere Variabilität. Eine der Beschränkungen der DNA-Isolierung in der vorliegenden Arbeit ist, dass jedes FFPE Probe ist einzigartig und werden einander in Oberflächenverunreinigungen, mikrobielle Flora zu variieren, und / oder menschlichen genetischen Hintergründen. Im allgemeinen wird DNA extrahiert Qualität und -quantität und der Erfolg der gesamten genomischen DNA-Amplifikation hängt von verschiedenen Parametern vor, während und nach der Extraktion. Hierzu gehören Art und Menge des Gewebes, der Art des Fixiermittels zur Gewebekonservierung, der Dauer der Fixierung, Alter der Paraffinblock und Lagerbedingungen, sowie die Länge des gewünschten DNA-Segments zu analysierende 16. Entfernen von Paraffin aus dem Gewebe ist der kritischste Schritt für eine erfolgreiche Extraktion als ungelöste Paraffin führt zu einer schlechten Qualität der Proben. Während der Tröpfchenerzeugung, muss darauf geachtet werden, um eine Blasenbildung zu vermeiden - dies ist ein weiterer wichtiger Schritt für eine erfolgreiche Detektion von Mutationen. In Anbetracht der Probe, um Variation, die zwischen Probenpopulationen entstehen, und basierend auf dem Motiv des Experiments könnte probieren, könnten bestimmte Änderungen in der Vorgehensweise erforderlich, um gewünschte Ergebnis zu erhalten.
Ein weiterer Vorteil besteht darin, dass die DNA-Isolierung und ddPCR wird unter Verwendung automatisierter Systeme in diesem Protokoll durchgeführt, und daher besteht vernachlässigbaren Fehler und Eingreifen des Benutzers erforderlich ist, ist sehr gering. Isolierung des gesamten Genoms-amplifizierte DNA aus Paraffin eingebettetem Gewebe / Zellen wurde unter Verwendung von TPS-System erhalten. Einer der Nachteile bei der Verwendung automatisierter DNA-Isolationssystems ist, dass es nicht kosteneffizient, kleine Anzahl von Proben verwendet werden. Anstelle dieser automatisierten step, andere Standard-DNA-Isolierungsverfahren könnte auch für begrenzte Proben durchgeführt werden,
Eine neuere Studie festgestellt, dass unter Verwendung von Tröpfchen digitalen PCR (ddPCR) ist in der Lage, um die relative Kopienzahl von bestimmten Loci, selbst in Gegenwart von vermischten von FFPE-Geweben erhalten normalem Gewebe zu bestimmen. Durch die Verwendung eines Steuerverdünnungsreihe Nadauld, L. et al. Bestimmt die Nachweisgrenzen (LOD) für die ddPCR Assay und berichtet ihre verbesserte Empfindlichkeit auf geringe Mengen von DNA im Vergleich zu Standard-Echtzeit-PCR 17. Hier werden FFPE Referenzstandard Zelllinien verwendet, um die Mutation Erfassungsfähigkeit des ddPCR System demonstrieren. Die System Ergebnisse ddPCR angegeben die Möglichkeit, seltene Mutation Allelfrequenzen bis zu 0,05% Mutation zu erkennen. Zusammen zeigen diese Daten, dass die ddPCR System ermöglicht auch die quantitative Analyse der Prozentsätze der verschiedenen mutanten Allelen und relativen Unterschiede in heterozygoten klinischen Tumorprobes. Vielzahl von FFPE-Proben können für spezielle Genmutationen gleichzeitig analysiert werden, und dies ist die optimale Technik zur Bevölkerung breite genetische Studien.
Schließlich ist zu berücksichtigen, dass die Mutation Frequenzen werden hier absolute Quantifizierung dargestellt und sollte nicht als relativen Wert der Mutationsrate oder Frequenz erwogen werden. ddPCR Auslese bietet absolute Quantifizierung der Ziel-DNA-Mutation. Diese Werte können zum Validieren Mutationshäufigkeiten von Proben unter den gleichen Bedingungen hergestellt wurde, verwendet werden kann und über den gleichen Bereich sequenziert. Jedoch sind diese Absolutwerte reproduzierbar und kann zum quantitativen Vergleich der Mutation Verteilung und Frequenz verwendet werden, wenn die optimalen Parameter gesteuert werden. Im Ergebnis hat ddPCR kürzlich als ein robustes Werkzeug die absolute Quantifizierung von Nukleinsäuren gibt in FFPE und Biopsieproben und auch unter Bezugnahme Assays zur Bestimmung von entweder geduplext werden entstand keinrmalized Transkript-Konzentrationen oder DNA-Kopienzahl.
Offenlegungen
Myung Ryuri Oh, Si Eun Kim und Junge Deug Kim sind Mitarbeiter von ABION CRO.
Danksagungen
Diese Arbeit wurde von der F & E-Programm für die Gesellschaft der National Research Foundation (NRF), durch das Ministerium für Wissenschaft, IKT und Zukunftsplanung (Grant No 2013M3C8A1075908) finanziert unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Hamilton MICROLAB STARlet IVD instrument | Siemens | 10701001 | Automated DNA isolation instrument |
| QX200 Droplet Generator | Bio-Rad | 772BR1119 | |
| QX200 Droplet Reader | Bio-Rad | 771BR1497 | |
| Conventional PCR machine capable of ramp-time adjustment | 621BR17718 | ||
| PX1 PCR plate sealer | Bio-Rad | 770BR1575 | |
| QuantaSoft software | Bio-Rad | ||
| DNA isolation kit | |||
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632398 | |
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632399 | |
| CO-RE tips | Siemens | ||
| ddPCR mutation analysis | |||
| ddPCR Supermix | Bio-Rad | BR186-3010 | 2X concentration |
| DG8 cartridge | Bio-Rad | BR186-4008 | |
| Droplet Generator oil | Bio-Rad | BR-186-3005 | |
| Gasket | Bio-Rad | BR186-3006 | |
| Droplet reader oil | Bio-Rad | BR-186-3004 |
Referenzen
- Vogelstein, B., et al. Genetic alterations during colorectal-tumor development. The New England journal of medicine. 319, 525-532 (1988).
- Davies, H., et al. Mutations of the BRAF gene in human cancer. Nature. 417, 949-954 (2002).
- Solit, D. B., et al. BRAF mutation predicts sensitivity to MEK inhibition. Nature. 439, 358-362 (2006).
- Wong, K. K. Recent developments in anti-cancer agents targeting the Ras/Raf/ MEK/ERK pathway. Recent patents on anti-cancer drug discovery. 4, 28-35 (2009).
- Brose, M. S., et al. BRAF and RAS mutations in human lung cancer and melanoma. Cancer research. 62, 6997-7000 (2002).
- Wang, L., et al. BRAF mutations in colon cancer are not likely attributable to defective DNA mismatch repair. Cancer research. 63, 5209-5212 (2003).
- Hindson, B. J., et al. High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal Chem. 83, 8604-8610 (1021).
- Pinheiro, L. B., et al. Evaluation of a Droplet Digital Polymerase Chain Reaction Format for DNA Copy Number Quantification. Anal Chem. 84, 1003-1011 (1021).
- Jones, M., et al. Low copy target detection by Droplet Digital PCR through application of a novel open access bioinformatic pipeline, 'definetherain'. Journal of virological methods. 202, 46-53 (2014).
- Strain, M. C., et al. Highly precise measurement of HIV DNA by droplet digital PCR. PloS one. 8, e55943(2013).
- Miotke, L., Lau, B. T., Rumma, R. T., Ji, H. P. High sensitivity detection and quantitation of DNA copy number and single nucleotide variants with single color droplet digital PCR. Anal Chem. 86, 2618-2624 (2014).
- Bizouarn, F. Clinical applications using digital PCR. Methods in molecular biology. 1160, 189-214 (2014).
- McDermott, G. P., et al. Multiplexed target detection using DNA-binding dye chemistry in droplet digital PCR. Anal Chem. 85, 11619-11627 (2013).
- Milbury, C. A., et al. Determining lower limits of detection of digital PCR assays for cancer-related gene mutations. Biomolecular detection and quantification. 1, 8-22 (2014).
- Zhu, G., et al. Highly Sensitive Droplet Digital PCR Method for Detection of EGFR Activating Mutations in Plasma Cell-Free DNA from Patients with Advanced Non-Small Cell Lung Cancer. The Journal of molecular diagnostics: JMD. , (2015).
- Fan, H., Gulley, M. L. DNA extraction from paraffin-embedded tissues. Methods in molecular medicine. 49, 1-4 (2001).
- Nadauld, L., et al. Quantitative and Sensitive Detection of Cancer Genome Amplifications from Formalin Fixed Paraffin Embedded Tumors with Droplet Digital PCR. Translational medicine. 2, (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten