Method Article
Employant numérique Droplet PCR pour détecter des mutations BRAF V600E dans de paraffine standard de référence lignées cellulaires fixés au formol
Dans cet article
Résumé
Le but de cette vidéo est de montrer comment effectuer une extraction d'ADN automatisée à partir des lignes de formol paraffine (FFPE) référence standards cellulaires et gouttelettes numérique PCR (ddPCR) analyse pour détecter des mutations rares dans un cadre clinique. Détecter des mutations dans des échantillons FFPE démontre l'utilité clinique de ddPCR dans les échantillons FFPE.
Résumé
ddPCR is a highly sensitive PCR method that utilizes a water-oil emulsion system. Using a droplet generator, an extracted nucleic acid sample is partitioned into ~20,000 nano-sized, water-in-oil droplets, and PCR amplification occurs in individual droplets. The ddPCR approach is in identifying sequence mutations, copy number alterations, and select structural rearrangements involving targeted genes. Here, we demonstrate the use of ddPCR as a powerful technique for precisely quantitating rare BRAF V600E mutations in FFPE reference standard cell lines, which is helpful in identifying individuals with cancer. In conclusion, ddPCR technique offers the potential to precisely profile the specific rare mutations in different genes in various types of FFPE samples.
Introduction
L'accumulation de mutations génétiques dans les gènes régulateurs clés modifie la programmation de la cellule normale, comme la prolifération cellulaire, la différenciation et la survie, menant au cancer 1. La voie de RAS-RAF kinase-MAP médie la réponse cellulaire aux signaux de croissance. Mutations oncogènes BRAF peuvent résulter de mutations du pilote dans le gène BRAF, qui peuvent causer l'oncoprotéine BRAF pour devenir hyperactive 2. Des mutations dans le gène BRAF se traduisent également par la signalisation en aval par l'intermédiaire d'hyperactivité de MEK et ERK 3, qui, à son tour, conduit à une croissance cellulaire excessive et la prolifération indépendamment de la régulation de la croissance médiée par le facteur 6/4.
Plusieurs outils sont disponibles pour les mutations de l'ADN profilage, tels que quantitatives en temps réel des mutations BRAF V600E dans de formol, inclus en paraffine (FFPE) référence des lignes de cellules standard par ddPCR. ddPCR est une méthode basée sur la PCR pour la quantification absolue-offrant une plus grande précision par rapport à quantitative en temps réel classique de PCR (qPCR) 7,8. ddPCR fournit également une plus grande puissance et de la précision de la résolution de la détection des mutations rares dans des matrices d'ADN, permettant recherche sur le cancer et le diagnostic plus informative 9. Des avantages supplémentaires de ddPCR sur qPCR conventionnelle incluent sa sensibilité accrue et la précision lors de l'étude des nombres de copies bas de modèle 10-12. Ici, un protocole d'extraction automatique d'ADN à partir de lignées de cellules standards de référence FFPE, suivie par détermination de la présence ou de l'absence de mutations BRAF V600E par ddPCR est démontrée. L'utilisation du logiciel d'analyse de données et une représentation graphique des résultats sont également décrits. L'ensemble de la procédure est relativement simple et dépend du nombre d'échantillons à être profilées et le nombre de machines PCR et ddPCR classiques disponibles totalement.
Le protocole suivant décrit les procédures standard pour BR Des lignées de cellules positives AF-V600E FFPE référence standards (HD598, HD593, HD617, HD273 et de type sauvage (WT)) est réalisée dans un instrument entièrement automatisé en utilisant le système de préparation tissulaire (TPS) protocole. Par la suite, des échantillons d'ADN isolés sont analysés pour la présence de mutations BRAF V600E utilisant le système ddPCR. Analyse de mutation ciblée est réalisée après que tous les échantillons ont été profilée et les données ont été chargées dans le logiciel d'analyse des données. Selon le nombre d'échantillons / groupes étudiés, l'analyse des données peut exiger d'une à plusieurs heures. Le composant expérimentale de la méthode nécessite une précision dans la manipulation de l'ADN etrologique Pipettes et pipettes, une vidéo JoVE enseignement des sciences en expliquant plus sur sur le contexte de pipetage "> pipetage en plaques à 96 puits, tandis que l'analyse des données est effectuée en utilisant le logiciel.
Protocole
1. extraction d'ADN à partir de la FFPE standard de référence des lignées cellulaires
Remarque: Pour cette procédure, l'extraction d'ADN a été réalisée à partir de lignées de cellules standards de référence FFPE (HD598, HD593, HD617, HD273 et de type sauvage (WT)) en utilisant le kit d'isolement d'ADN de tissu FFPE de la manière décrite dans le protocole ci-dessous. Extraction de l'ADN automatisé a été réalisé en suivant les instructions du fabricant pour l'isolement de l'ADN total.
- En utilisant un microtome et le bloc FFPE original, préparer de nouvelles sections manuellement avant l'extraction et l'analyse de l'ADN, selon les procédures établies. Assurez-vous que l'entrée d'échantillon pour la section (s) de tissu analysé ne dépasse pas une épaisseur totale combinée de 10 um et que la surface spécifique ne dépasse pas 600 mm 2 pour un seul article.
1.2 protocole TPS
Remarque: Les volumes indiqués dans le tableau 1 correspondent au minimum requis pour traiter les 48 échantillons, et la procédureindiqué est en conformité avec les lignes directrices de TPS. Avant de commencer l'expérience installer les échantillons FFPE dans l'e-tubes par centrifugation à 600 xg, pour éviter la perte des échantillons durant le programme automatisé.
- Allumez l'instrument et l'ordinateur automatisé d'isolement d'ADN. Ouvrez le logiciel de commande Exécuter et insérez un plateau de chargement automatique dans la zone pont de chargement TPS.
- Distribuer des réactifs dans leurs creux correspondants comme représenté sur la figure 1.
- Placez les 4 échantillons (BRAF WT, BRAF V600E 50%, 5% et 1%) dans les racks de support de l'échantillon.
- Placez les boîtes de pointe dans les creux des colonnes 2 et 3 et de vérifier les conseils de la présence de plus d'un filtre par la pointe, ce qui annulerait la course.
- Assurer le bon mélange du tampon de lyse et tampon de lavage en les inversant 3-5 fois et de les charger dans les creux respectifs de la colonne 4 (Figure 1).
- Après avoir inversé pour quelques heures ou vort légèreExing, charger le tampon d'élution, des billes magnétiques, et le tampon de la FFPE dans les petits creux dans la colonne 5, en laissant 1 emplacement vide où indiquée (Figure 1, Tableau 1).
- Chargez une plaque de puits profond de 2 ml (DWP) sur le support de plaque (Figure 1).
- Effectuez les dernières étapes suivantes avant de commencer une course:
- Déboucher tous les tubes et les creux de réactifs. Assurez-vous que la capacité suffisante est disponible dans la bouteille de déchets liquides. Assurez-vous que la bouteille de déchets solides est vide et munie d'un sac de Biohazard. Assurez-vous que la plaque pointe d'éjection est centré dans l'ensemble des déchets.
- Fermez le capot avant.
- Démarrez le logiciel. Ouvrez le fichier NA_Prep_Main_MLSTARlet.med.
- Cliquez sur "Démarrer". L'état de l'instrument passe du ralenti à la course.
- Entrez le nombre d'échantillons pour cette course. Choisissez la méthode souhaitée pour cette course (ADN = 0). Entrez la position du premier vol hauteume pointe, la sélection de "1" si tous les bacs sont pleins. Entrez la position de la première pointe de volume standard, sélectionner "1" si tous les bacs sont pleins.
Remarque: L'instrument sera alors courir à travers étapes automatisées, sans intervention de l'utilisateur. Un flux de travail détaillé est présenté à la figure 2. Une fois que l'expérience est terminée, les déchets de réactif et des conseils sont injectés dans l'ensemble des déchets. - Quantifier l'ADN génomique purifié à l'aide d'un procédé fluorimétrique.
Note: Des échantillons d'ADN qui contiennent une concentration minimale de 3,3 ng / ul sont soumis à une analyse ddPCR (section 2).
2. mutation de l'ADN Profilage: Protocole ddPCR
Remarque: Le protocole de mutation de l'ADN profilage se compose de 3 grandes étapes: 1) la génération des gouttelettes, 2) l'amplification par PCR conventionnelle, 3) la lecture des gouttelettes et 4) mutation de l'ADN profilage.
2.1. Génération Droplet
Remarque: ddPCR supermix estrecommandé pour ddPCR, que ce mélange contient les réactifs nécessaires pour la production de gouttelettes.
- Pour éviter la contamination, suivre les précautions standard, telles que le port de gants, en utilisant une hotte propre PCR, pipettes propres, et des tubes à faible liaison aux protéines.
- Assurez-vous que la concentration minimale de l'échantillon d'ADN génomique humain est de 3,3 ng / ul. Note: La quantité de concentration de l'échantillon d'ADN purifiée varie en fonction de l'analyse de détection de fréquence de mutation%.
- Assembler mélanges de réaction dans des plaques de PCR à 96 puits. Décongeler et équilibrer les composants de la réaction à la température ambiante. Préparer les réactions PCR en combinant 2X ddPCR supermix (10 pi) et 20 amorces (avant et arrière, 900 nM) et d'une sonde (250 nM) pour chaque échantillon d'ADN purifié (66ng / 2 pi) faire jusqu'à 20 pi avec de l'eau distillée (comme par le protocole de générateur de gouttelettes)
- Vortexer le mélange soigneusement pour assurer l'homogénéité et centrifuger brièvement pour rassembler le contenu au fond du tube avant la distribution. Charge & # 20181; l sur la cartouche pour la formation de gouttelettes.
- Le fonctionnement du générateur de gouttelettes, selon le protocole recommandé par le fabricant
- Insérer la cartouche dans le support avec l'encoche de la cartouche positionnée sur le côté supérieur gauche de la porte. Ajouter 20 pi de mélange réactionnel contenant des échantillons dans le milieu, et 70 ul d'huile de générateur dans les puits de fond.
- Attacher le joint d'étanchéité dans la partie supérieure de la cartouche. Assurez-vous que le joint soit correctement accroché sur les deux extrémités du support; Sinon, la pression suffisante pour la génération de gouttelettes ne sera pas atteint.
- Ouvrez le générateur de gouttelettes en appuyant sur le bouton vert sur le dessus de l'appareil et insérez la cartouche. Lorsque le titulaire est dans la bonne position, à la fois la puissance (de gauche à droite) et titulaire (au milieu à droite) voyants sont au vert.
- Appuyez sur le bouton en haut sur l'instrument à nouveau pour fermer la porte et d'initier la génération de gouttelettes. Remarque: Après avoir appuyé sur le button, a positions multiples se sur les puits de sortie, dessin pétrole et des échantillons à travers les canaux microfluidiques, où les gouttelettes sont créés. Gouttelettes se jettent dans la gouttelette bien, où ils accumulent. L'indicateur de gouttelettes de lumière (à droite) clignote en vert après 10 secondes pour indiquer que la génération de gouttelettes est en cours.
- Lorsque la génération des gouttelettes est terminée, tous les 3 voyants changent au vert solide; ouvrir la porte en appuyant sur le bouton, et retirez le support de l'unité. Retirez le joint du support jetable et jetez-le. Remarque: Les meilleurs puits de la cartouche contiennent des gouttelettes, et les puits moyenne et inférieure sont presque vides, avec une petite quantité d'huile résiduelle.
2.2. Préparation pour la PCR
- Pour chaque échantillon, pipette à 40 ul du contenu de gouttelettes à partir du haut et les cartouches dans un seul puits d'une plaque à 96 puits PCR recommandé comme indiqué dans le protocole de l'instrument par le fabricant. Nonte: aide d'une pipette multi-canal est idéal pour transférer les gouttelettes des émulsions. Aspiration lente et douce de gouttelettes est recommandée afin de minimiser le cisaillement de gouttelettes pendant les transferts.
- Sceller la plaque PCR avec du papier immédiatement après le transfert de gouttelettes pour éviter l'évaporation. Utilisation perforable joints de plaques de feuille qui sont compatibles avec la plaque PCR et scellant les aiguilles dans le lecteur de gouttelettes. Suivez les instructions dans le manuel d'instruction de scellant pour plaques PCR.
- Régler la température de la plaque de scellement à 180 ° C et le temps de 5 sec.
- Appuyez sur la flèche pour ouvrir la porte du bac. Placez le bloc de support sur le plateau avec le côté 96 puits vers le haut. Placer la plaque à 96 puits sur le bloc de support et de sorte que tous les puits de la plaque sont alignés avec le bloc de support.
- Recouvrir la plaque de 96 puits avec 1 feuille de papier d'aluminium percé. Une fois la plaque 96 puits est fixé sur le bloc de support et couvert avec le papier d'aluminium percé, appuyez sur le bouton d'étanchéité. Le plateau se refermeet la chaleur étanchéité va initier.
- Lorsque le thermoscellage est terminée, la porte ouvre automatiquement; retirer la plaque du bloc thermique pour le cyclage thermique, puis retirez le bloc thermique.
- Veiller à ce que tous les puits de la plaque sont scellés en vérifiant que des dépressions dans la feuille sont facilement visibles sur chaque puits. Une fois scellé, la plaque est prêt pour le cyclage thermique.
- Une fois gouttelettes sont supprimés, appuyez sur les loquets sur le support de la cartouche pour l'ouvrir. Retirez la cartouche vide et le jeter.
- Effectuer une amplification par PCR conventionnelle en suivant les paramètres détaillés dans le tableau 2.
2.3 lecture Droplet (selon le protocole recommandé par le fabricant)
Note: Après amplification par PCR de la cible d'acide nucléique dans les gouttelettes, l'appareil de lecteur de gouttelettes analyse chaque gouttelette individuellement en utilisant un système de détection 2-couleur 13. Nous généralement configuré pour détecter un FAMd VIC fluorophores rapporteurs.
- Cliquez sur "système de rinçage» pour amorcer le lecteur de gouttelettes et de le rendre prêt pour l'analyse ddPCR.
- Chargez la plaque dans le lecteur de gouttelettes et cliquez sur "Démarrer". Le lecteur de gouttelettes aspire chaque échantillon, singulates les gouttelettes et les flux en file indienne devant un détecteur 2-couleur. Le détecteur lit les gouttelettes d'énumérer les échantillons positifs et négatifs.
- Lorsque gouttelettes lecture est terminée, ouvrez la porte et retirez le support de plaque de l'unité. Retirer la plaque PCR 96 puits de la porte et le jeter.
- Pour un bon entretien, remplacer l'huile de lecteur de gouttelettes et vider la poubelle, au besoin. Ajouter 50 ml d'eau de Javel à 10% à la bouteille de déchets pour empêcher la croissance microbienne.
2.4 mutation de l'ADN profilage (selon le protocole recommandé par le fabricant)
Remarque: gouttelettes PCR-positives et PCR-négatifs sont comptés pour fournir quantific absolueation des cibles mutations BRAF V600E ADN sous forme numérique, en utilisant un logiciel d'analyse de données.
- Cliquez sur Configuration pour entrer des informations sur les échantillons, des essais et des expériences.
- Dans la fenêtre de configuration, charger une plaque (filename.qlp), puis cliquez sur Analyser pour ouvrir et analyser les données. L'interface d'analyse de données est séparé en trois fenêtres: Résultats Tableau, le sélecteur de puits et des données traitées / affichage graphique.
- Dans la fenêtre données traitées, les données de concentration pour chaque cible apparaissent dans les puits dans le plan de la plaque et sont indiquées dans le tableau des résultats. Cliquez Concentration de visualiser les données dans les parcelles de concentration.
Résultats
Pour notre analyse ddPCR, nous avons étudié les références mutation FFPE lignes de cellules standard V600E BRAF. Le lecteur de gouttelettes se connecte à un ordinateur portable un logiciel d'analyse de données informatique de fonctionnement. Chaque gouttelette individuelle est définie sur la base de l'amplitude de fluorescence comme étant soit positif ou négatif. Le logiciel fourni par le fabricant permet également à un seuil défini par l'utilisateur à saisir pour définir le seuil entre les gouttelettes positives et négatives. Le nombre de gouttelettes positives et négatives dans un échantillon est utilisée pour calculer la concentration de la cible en fonction de copies / pl.
La fluorescence a été détectée et traitée dans un affichage de l'intrigue dispersion en deux dimensions, le logiciel personnalisé a été utilisé pour dessiner des portes appropriées pour chaque groupe d'extrémité de gouttelettes, et le nombre de gouttelettes dans chaque porte a été compté. Comme le montre la figure 3A, les gouttelettes représentés par des points bleus (signal de fluorescence FAM) au-dessus de la ligne de coupure pour tous samples (ligne rose) étaient positifs pour muté V600E BRAF. Gouttelettes représentés par des points bleus dans le BRAF WT (NTC; Lane 2) échantillons pourraient être dus à un signal non spécifique (faux positif). Faux signaux positifs (BRAF WT) ont été normalisés avec d'autres échantillons de mutation. Comme représenté sur la figure 3B, BRAF WT gouttelettes sont représentées par des points verts (signal de fluorescence VIC). Dans les deux parcelles, les points gris en bas sont considérés comme l'arrière-plan de fluorescence. Les fréquences globales allèle mutant ont été calculées en utilisant les données de la figure 3C, sur la base des pourcentages relatifs de BRAF WT et modèles BRAF V600E de détectés. Résultats ddPCR obtenus contiennent les comptages d'événements de gouttelettes et de type sauvage calculée et de la molécule d'ADN mutant compte pour l'V600E BRAF (50%, 10%, 5%, 1%, 0,5%, 0,1% et 0,05%) échantillons calculé en utilisant le ci-dessous formule mentionnée.
% De la fréquence Mutant = (Mutantcopie / (type sauvage + copie Mutant)) x 100
En conséquence, les mutations BRAF V600E ont été identifiées et vérifiées à la norme de référence (BRAF WT). Défini BRAF V600E mutation fréquences alléliques de 50%, 10%, 5%, 1%, 0,5%, 0,1% et 0,05% ont été utilisés pour tester la sensibilité et la reproductibilité du système ddPCR. De notre analyse avec des concentrations d'échantillons connus, nous avons confirmé que ddPCR est capable de détecter aussi bas que 0,05% de BRAF V600E mutation. La détection de faux comte mutant positif dans NTC ou WT pourrait être dû à la sonde hydrolyse non spécifique comme précédemment indiqué 14. La détection de plus de deux copies dans un échantillon a été considéré comme positif dans le tissu tumoral 15.

Figure 1. Représentation schématique décrivant réactif et de chargement de l'échantillon en préparation pour aut omated instrument d'extraction d'ADN. Placer les échantillons dans un porte-racks et de distribuer les réactifs dans des creux correspondants comme indiqué. Employant automatisé protocole TPS qui prend en charge plusieurs types d'échantillons, offre précise, et des résultats fiables avec une productivité maximale. Réimprimé avec la permission de Siemens Healthcare Diagnostics. (courtoisie de Siemens Healthcare Diagnostics).

Figure 2. Représentation schématique de la Préparation des tissus système de workflow pour l'extraction d'ADN automatisé. Entièrement automatisé de procédure d'isolement de l'ADN pour FFPE tissus sections, y compris des mesures négatives de sélection de la paraffine, l'enlèvement des débris de tissu et les étapes de sélection positive de liaison et d'élution sont indiqués. Réimprimé avec la permission de Siemens Healthcare Diagnostics. (courtoisie de Siemens Healthcare Diagnostics).
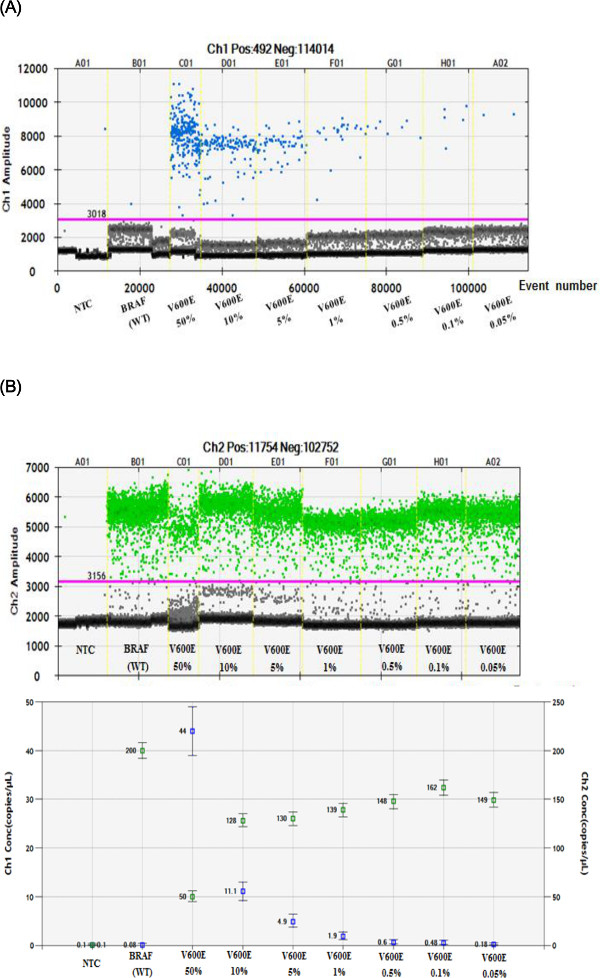
Figure 3. Utilisation du système ddPCR pour la quantification précise de la BRAF V600E mutation dans des échantillons de lignées cellulaires norme de référence FFPE. (A, B) Visualisation des amplitudes de fluorescence positifs dans les parcelles 1D (1dot -1droplet). Les points bleus (A, FAM positif) représentent BRAF V600E gouttelettes de positif mutantes, tandis que les points verts (B, VIC positif) représente WT BRAF gouttelettes -positifs. Cette détermination permet une quantification précise de la mutation dans les lignées cellulaires standards de référence FFPE. La ligne rose est le seuil de discrimination entre les signaux positifs et négatifs des gouttelettes. (C) Le terrain de l'abondance fractionnée montre marqueurs bleus qui indiquent la concentration (copies / pi) de BRAF V600E mutation, etles marqueurs verts indiquent la concentration (copies / pi) de BRAF (WT). Toutes les barres d'erreur générés par le logiciel d'analyse de données représentent l'intervalle de confiance de 95%.
| Réactifs | Volume (ml) |
| Lysis Buffer | 106 ml |
| Wash Buffer 1 | 101 ml |
| Wash Buffer 2 | 72 ml |
| Wash Buffer 3 | 106 ml |
| Elution Buffer | 19 ml |
| Billes magnétiques | 8 ml |
| Tampon FFPE | 15 ml |
| Proteinase K | 3,3 ml |
Tableau 1. Le volume total de réactifs (kit TPS) nécessaire à l'extraction de l'ADN de 48 échantillons.
| Cyclisme Étape | Température | Temps | # Cycles |
| Activation Enzyme | 95 ° C | 10 min | 1 |
| Dénaturation | 94 ° C | 30 sec | 40 |
| Recuit / prolongation | 60 ° C | 1 min * | |
| Tenir | 98 ° C | 10 min | 1 |
| Tenir | 4 ° C | Pour toujours | 1 |
| * Réglez les paramètres de taux de rampe à 2-2,5 ° C / sec. Utilisez un couvercle chauffant réglé à 105 ° C et régler le volume de l'échantillon de 40 ul | |||
Tableau 2. PCR conventionnelle conditions de thermocyclage
Discussion
Ici, nous mettons en évidence l'applicabilité de ddPCR et l'isolement de l'ADN à partir d'échantillons de lignées cellulaires standards de référence FFPE une évaluation spécifique de la mutation du gène. Dans cette étude, TPS méthode d'isolement d'ADN automatisé est utilisé qui peut être facilement adapté, automatisé, et peut accueillir jusqu'à 48 échantillons différents simultanément, permettant de plus grandes expériences à grande échelle et une plus faible variabilité. Une des limites de l'isolement de l'ADN dans le présent ouvrage est que chaque échantillon FFPE est unique, et variera entre eux dans les contaminants de surface, la flore microbienne, et / ou antécédents génétiques humaines. En général, on l'extrait qualité et la quantité d'ADN et le succès de l'ensemble de l'amplification d'ADN génomique dépendent de divers paramètres, avant, pendant et après l'extraction. Ceux-ci comprennent le type et la quantité de tissu, du type de fixateur utilisé pour la préservation du tissu, de la durée de fixation, l'âge des conditions des blocs de paraffine et de stockage, ainsi que la longueur du segment d'ADN désiré à analyser 16. Enlèvement de la paraffine à partir du tissu est l'étape la plus critique pour l'extraction réussie que la paraffine non dissous conduit à une mauvaise qualité de l'échantillon. Au cours de la génération de gouttelettes, des précautions doivent être prises pour éviter la formation de bulles - ce qui est une autre étape essentielle pour la détection de mutation réussie. Considérant l'échantillon à la variation qui pourrait surgir entre les échantillons de population et fondée sur le motif de l'expérience de l'échantillon, certaines modifications dans la procédure pourraient être nécessaires pour obtenir le résultat souhaité.
Un autre avantage est que l'isolement de l'ADN et ddPCR est effectuée en utilisant des systèmes automatisés dans ce protocole, et donc il ya erreur négligeable et intervention de l'utilisateur est très minime. Isolement de l'ADN du génome entier amplifié à partir de cellules / tissus inclus dans la paraffine ont été obtenus en utilisant le système TPS. L'un des inconvénients en utilisant un système automatisé d'isolement d'ADN qui est, elle n'a pas rentable d'utiliser petit nombre d'échantillons. Au lieu de cela ste automatisép, autre procédure d'isolement d'ADN standard pourrait également être effectuée pour les échantillons limités
Une étude récente a indiqué que l'utilisation de gouttelettes PCR numérique (ddPCR) est en mesure de déterminer le nombre de copies relative des loci génomiques spécifiques, même en présence d'un tissu normal entremêlés obtenu à partir de tissus FFPE. En utilisant une série de dilution de contrôle, Nadauld, L. et al. Déterminé les limites de détection (LOD) pour le dosage ddPCR et rapporté sa sensibilité améliorée sur des quantités minimes de l'ADN par rapport au temps réel PCR standard 17. Ici, les lignées cellulaires standards de référence FFPE sont utilisés pour démontrer la capacité de détection de la mutation du système ddPCR. Les résultats du système ddPCR indiqué la possibilité de détecter rares fréquences alléliques de mutation jusqu'à 0,05% de mutation. Collectivement, ces données indiquent que le système ddPCR permet également une analyse quantitative des pourcentages des divers alleles mutants et les différences relatives de l'échantillon clinique de la tumeur hétérozygotes. Grand nombre d'échantillons FFPE peut être analysée pour les mutations de gènes spécifiques simultanément et cela est une technique optimale pour de larges études génétiques de la population.
Enfin, il faut tenir compte du fait que les fréquences de mutation sont représentés ici sont quantification absolue et ne doivent pas être considérés comme valeur relative des taux de mutation ou de la fréquence. ddPCR lecture fournit une quantification absolue d'ADN cible mutation. Ces valeurs peuvent être utilisées pour valider la fréquence des mutations d'échantillons préparés dans les mêmes conditions, et séquences sur la même région. Toutefois, ces valeurs absolues sont reproductibles et peuvent être utilisés pour la comparaison quantitative de la distribution et de la fréquence de mutation lorsque les paramètres optimaux sont contrôlées. En conclusion, ddPCR a récemment émergé comme un outil robuste qui donne la quantification absolue des acides nucléiques dans des échantillons de biopsie et FFPE et peut également être recto verso avec les tests de référence pour la détermination de soit pasconcentrations de transcription rmalized ou le nombre de copies d'ADN.
Déclarations de divulgation
Myung Ryuri Oh, Eun Kim Si, et Young Kim Deug sont des employés de ABION CRO.
Remerciements
Cette recherche a été financée par le Programme de R & D pour la Société de la National Research Foundation (NRF), financé par le ministère de la Science, les TIC et la planification future (Grant No. 2013M3C8A1075908).
matériels
| Name | Company | Catalog Number | Comments |
| Hamilton MICROLAB STARlet IVD instrument | Siemens | 10701001 | Automated DNA isolation instrument |
| QX200 Droplet Generator | Bio-Rad | 772BR1119 | |
| QX200 Droplet Reader | Bio-Rad | 771BR1497 | |
| Conventional PCR machine capable of ramp-time adjustment | 621BR17718 | ||
| PX1 PCR plate sealer | Bio-Rad | 770BR1575 | |
| QuantaSoft software | Bio-Rad | ||
| DNA isolation kit | |||
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632398 | |
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632399 | |
| CO-RE tips | Siemens | ||
| ddPCR mutation analysis | |||
| ddPCR Supermix | Bio-Rad | BR186-3010 | 2X concentration |
| DG8 cartridge | Bio-Rad | BR186-4008 | |
| Droplet Generator oil | Bio-Rad | BR-186-3005 | |
| Gasket | Bio-Rad | BR186-3006 | |
| Droplet reader oil | Bio-Rad | BR-186-3004 |
Références
- Vogelstein, B., et al. Genetic alterations during colorectal-tumor development. The New England journal of medicine. 319, 525-532 (1988).
- Davies, H., et al. Mutations of the BRAF gene in human cancer. Nature. 417, 949-954 (2002).
- Solit, D. B., et al. BRAF mutation predicts sensitivity to MEK inhibition. Nature. 439, 358-362 (2006).
- Wong, K. K. Recent developments in anti-cancer agents targeting the Ras/Raf/ MEK/ERK pathway. Recent patents on anti-cancer drug discovery. 4, 28-35 (2009).
- Brose, M. S., et al. BRAF and RAS mutations in human lung cancer and melanoma. Cancer research. 62, 6997-7000 (2002).
- Wang, L., et al. BRAF mutations in colon cancer are not likely attributable to defective DNA mismatch repair. Cancer research. 63, 5209-5212 (2003).
- Hindson, B. J., et al. High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal Chem. 83, 8604-8610 (1021).
- Pinheiro, L. B., et al. Evaluation of a Droplet Digital Polymerase Chain Reaction Format for DNA Copy Number Quantification. Anal Chem. 84, 1003-1011 (1021).
- Jones, M., et al. Low copy target detection by Droplet Digital PCR through application of a novel open access bioinformatic pipeline, 'definetherain'. Journal of virological methods. 202, 46-53 (2014).
- Strain, M. C., et al. Highly precise measurement of HIV DNA by droplet digital PCR. PloS one. 8, e55943(2013).
- Miotke, L., Lau, B. T., Rumma, R. T., Ji, H. P. High sensitivity detection and quantitation of DNA copy number and single nucleotide variants with single color droplet digital PCR. Anal Chem. 86, 2618-2624 (2014).
- Bizouarn, F. Clinical applications using digital PCR. Methods in molecular biology. 1160, 189-214 (2014).
- McDermott, G. P., et al. Multiplexed target detection using DNA-binding dye chemistry in droplet digital PCR. Anal Chem. 85, 11619-11627 (2013).
- Milbury, C. A., et al. Determining lower limits of detection of digital PCR assays for cancer-related gene mutations. Biomolecular detection and quantification. 1, 8-22 (2014).
- Zhu, G., et al. Highly Sensitive Droplet Digital PCR Method for Detection of EGFR Activating Mutations in Plasma Cell-Free DNA from Patients with Advanced Non-Small Cell Lung Cancer. The Journal of molecular diagnostics: JMD. , (2015).
- Fan, H., Gulley, M. L. DNA extraction from paraffin-embedded tissues. Methods in molecular medicine. 49, 1-4 (2001).
- Nadauld, L., et al. Quantitative and Sensitive Detection of Cancer Genome Amplifications from Formalin Fixed Paraffin Embedded Tumors with Droplet Digital PCR. Translational medicine. 2, (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon