Method Article
El empleo de la gotita de PCR digital para detectar mutaciones BRAF V600E en embebidos en parafina líneas celulares de referencia estándar fijos-formalina
En este artículo
Resumen
El objetivo de este video es demostrar cómo llevar a cabo la extracción de ADN automatizado de parafina embebido (FFPE) referencia las líneas celulares estándar fijado en formol y gotita digital de PCR (ddPCR) análisis para detectar mutaciones raras en un entorno clínico. La detección de mutaciones en muestras de FFPE demuestra la utilidad clínica de ddPCR en muestras FFPE.
Resumen
ddPCR is a highly sensitive PCR method that utilizes a water-oil emulsion system. Using a droplet generator, an extracted nucleic acid sample is partitioned into ~20,000 nano-sized, water-in-oil droplets, and PCR amplification occurs in individual droplets. The ddPCR approach is in identifying sequence mutations, copy number alterations, and select structural rearrangements involving targeted genes. Here, we demonstrate the use of ddPCR as a powerful technique for precisely quantitating rare BRAF V600E mutations in FFPE reference standard cell lines, which is helpful in identifying individuals with cancer. In conclusion, ddPCR technique offers the potential to precisely profile the specific rare mutations in different genes in various types of FFPE samples.
Introducción
La acumulación de mutaciones genéticas en genes reguladores clave altera la programación normal de las células como la proliferación celular, la diferenciación y la supervivencia, que conduce al cáncer 1. La vía RAS-RAF quinasa MAP media las respuestas celulares a las señales de crecimiento. Mutaciones oncogénicas BRAF pueden resultar de mutaciones del conductor en el gen BRAF, que pueden causar la BRAF oncoproteína para convertirse hiperactiva 2. Las mutaciones en el gen BRAF también dan lugar a la señalización aguas abajo hiperactiva MEK y ERK 3, que, a su vez, conduce a un crecimiento celular excesivo y la proliferación independiente de factor de crecimiento mediada por la regulación 4-6.
Varias herramientas están disponibles para perfilar mutación del ADN, como en tiempo real mutaciones BRAF V600E cuantitativos en fijado en formol, embebidos en parafina (FFPE) referencia las líneas celulares estándar por ddPCR. ddPCR es un método basado en la PCR para la cuantificación absoluta-ofreciendo una mayor precisión en comparación con el tiempo real convencional de PCR cuantitativa (qPCR) 7,8. ddPCR también proporciona mayor poder de resolución y la precisión para la detección de mutaciones raras en plantillas de ADN, lo que permite la investigación del cáncer más informativo y diagnóstico 9. Las ventajas adicionales de ddPCR sobre qPCR convencional incluyen su mayor sensibilidad y precisión cuando se estudia el número de copias bajo plantilla 10-12. En este documento, un protocolo para la extracción automática de ADN de referencia FFPE líneas celulares estándar, seguido por la determinación de la presencia o ausencia de mutaciones BRAF V600E por ddPCR se demuestra. También se describen el uso de software para el análisis de datos y una representación gráfica de los resultados. Todo el procedimiento es relativamente simple y totalmente depende del número de muestras que se perfilados y el número de máquinas de PCR y ddPCR convencionales disponibles.
El siguiente protocolo describe los procedimientos estándar para BR FFPE de referencia líneas celulares estándar AF V600E-positivos (HD598, HD593, HD617, HD273 y de tipo salvaje (WT)) se realiza en un instrumento totalmente automatizado usando el protocolo Tissue Sistema de Preparación de (TPS). Posteriormente, se analizaron muestras de ADN aisladas de la presencia de mutaciones BRAF V600E utilizando sistema ddPCR. Análisis de la mutación se realiza después se han perfilado todas las muestras y los datos se ha cargado en el software de análisis de datos. Dependiendo del número de muestras / grupos estudiados, análisis de datos puede requerir de una a varias horas. El componente experimental de la metodología requiere precisión en la manipulación de ADN yrológicos Pipetas y pipetas, un video JoVe Educación Ciencia explicar más acerca sobre el contexto de pipeteo "> pipeteo en placas de 96 pocillos, mientras que el análisis de datos se realiza mediante software.
Protocolo
1. Extracción de ADN de líneas celulares FFPE norma de referencia
Nota: Para este procedimiento, la extracción de ADN se realizó a partir de líneas celulares FFPE estándar de referencia (HD598, HD593, HD617, HD273 y de tipo salvaje (WT)) con el kit de aislamiento de ADN de tejidos FFPE como se describe en el protocolo a continuación. Extracción de ADN automatizada se logró siguiendo las instrucciones del fabricante para el aislamiento de ADN total.
- Usando un microtomo y el bloque de FFPE original, preparar manualmente secciones frescas antes de la extracción y el análisis de ADN, de acuerdo con los procedimientos establecidos. Asegúrese de que la muestra de entrada para la sección (s) tejido analizado no exceda un espesor total combinado de 10 micras y que el área superficial no excede de 600 mm 2 para una sola sección.
1.2 protocolo de TPS
Nota: Los volúmenes mostrados en la Tabla 1 corresponden al mínimo requerido para procesar 48 muestras, y el procedimiento demostrado es de conformidad con las directrices de TPS. Antes de iniciar el experimento asentarse las muestras FFPE en la dirección de tubo por centrifugación a 600 xg, para evitar la pérdida de las muestras durante el programa automatizado.
- Encienda el instrumento de aislamiento de ADN automático y el ordenador. Abra el software de control de ejecución e inserte una bandeja de carga automática en el área de la cubierta de carga TPS.
- Dispensar reactivos en sus correspondientes canales como se muestra en la Figura 1.
- Colocar las 4 muestras (BRAF WT, BRAF V600E 50%, 5% y 1%) en los bastidores de soporte de la muestra.
- Coloque las cajas de propina en los valles en las columnas 2 y 3 y comprobar los consejos para la presencia de más de un filtro por la punta, lo que abortar la ejecución.
- Asegurar una mezcla adecuada del tampón de lisis y tampón de lavado mediante la inversión de ellos 3-5 veces y cargarlos en las respectivas depresiones en la columna 4 (Figura 1).
- Después de invertir para el par de veces o vort leveexing, cargue el tampón de elución, perlas magnéticas, y tampón FFPE en las canaletas en la columna 5, dejando 1 ranura vacía que se indique (Figura 1, Tabla 1).
- Cargue una placa de pozo profundo 2 ml (DWP) sobre la placa de soporte (Figura 1).
- Lleve a cabo los siguientes pasos finales antes de iniciar una carrera:
- Destape todos los tubos y valles de reactivos. Confirme que suficiente capacidad disponible en la botella de residuos líquidos. Confirme que la botella de residuos sólidos está vacía y forrado con una bolsa de riesgo biológico. Confirme que la placa de expulsión de punta se centra en la asamblea de residuos.
- Cierre la cubierta frontal.
- Inicie el software. Abra el archivo NA_Prep_Main_MLSTARlet.med.
- Haga clic en "Start". El estado del instrumento cambiará desde el ralentí hasta correr.
- Introduce el número de muestras para esta ejecución. Elegir el método deseado para esta ejecución (ADN = 0). Introduzca la posición de la primera alta volume punta, la selección de "1" si todas las bandejas están llenas. Introduzca la posición del primer consejo de volumen estándar, la selección de "1" si todas las bandejas están llenas.
Nota: El instrumento se ejecutará a través de pasos automatizados sin intervención del usuario. Un flujo de trabajo detallado se muestra en la Figura 2. Una vez que se terminó el experimento, los residuos de reactivos y consejos se inyectan en el conjunto de residuos. - Cuantificar el ADN genómico purificado mediante el uso de un método fluorométrico.
Nota: las muestras de ADN que contienen una concentración mínima de 3,3 ng / l se someten a análisis ddPCR (sección 2).
2. ADN Mutación Profiling: Protocolo ddPCR
Nota: El protocolo para el perfilado mutación del ADN consta de 3 pasos principales: 1) la generación de gotas, 2) amplificación por PCR convencional, 3) la lectura de la gotita y 4) de perfiles de mutación del ADN.
2.1. Generación Gotita
Nota: ddPCR supermix esrecomendado para ddPCR, ya que esta mezcla contiene los reactivos necesarios para la generación de gotitas.
- Para evitar la contaminación, siga las precauciones estándar, tales como el uso de guantes, con una capucha limpia PCR, pipetas limpias, y los tubos de unión de baja en proteínas.
- Asegúrese de que la concentración mínima de muestra de ADN genómico humano es de 3,3 ng / l. Nota: La cantidad de concentración de la muestra de ADN purificado variará basado en el análisis de detección de frecuencia% mutación.
- Montar mezclas de reacción de PCR en placas de 96 pocillos. Descongelar y equilibrar los componentes de la reacción a RT. Preparar las reacciones de PCR mediante la combinación de 2X ddPCR Supermix (10 l) y 20 cebadores (directo e inverso, 900 nM) y la sonda (250 nM) con cada muestra de ADN purificado (66ng / 2 l) hacer hasta 20 l con agua destilada (como se por el protocolo generador de gotitas)
- Vórtex con la mezcla a fondo para asegurar la homogeneidad y centrifugar brevemente para recoger el contenido en la parte inferior del tubo antes de la dispensación. Cargar 20 & #181; l en el cartucho para la formación de gotas.
- El funcionamiento del generador de gotas, según el protocolo recomendado por el fabricante
- Insertar el cartucho en el soporte con la muesca en el cartucho situado en el lado superior izquierdo del soporte. Añadir 20 l de mezcla de reacción que contiene muestras en el medio, y 70 l de aceite de generador en los pocillos de fondo.
- Coloque la junta en la parte superior del cartucho. Asegúrese de que la junta esté firmemente enganchado en ambos extremos del titular; de lo contrario, no se logrará una presión suficiente para la generación de gotitas.
- Abra el generador de gotas pulsando el botón verde en la parte superior del instrumento e inserte el cartucho. Cuando el soporte está en la posición correcta, tanto el poder (de izquierda a la derecha) y el soporte (centro derecha) luces indicadoras son de color verde.
- Presione el botón superior en el instrumento de nuevo para cerrar la puerta e iniciar la generación de gotas. Nota: Después de pulsar el button, a sí mismo múltiples posiciones en los pozos de salida, dibujo petróleo y las muestras a través de los canales de microfluidos, donde se crean gotas. Las gotas fluyan hacia la gota así, donde se acumulan. La luz indicadora de gota (a la derecha) parpadea en verde después de 10 segundos para indicar que la generación de gotas está en curso.
- Cuando la generación de gotas es completa, los 3 indicadores luminosos cambian de color verde; abrir la puerta pulsando el botón y retire el soporte de la unidad. Retire la junta desechable del soporte y descartarlo. Nota: Los pocillos superiores del cartucho contienen gotitas, y los pocillos medio e inferior son casi vacío, con una pequeña cantidad de aceite residual.
2.2. Preparación para la PCR
- Para cada muestra, pipetear a cabo 40 l de los contenidos de gotitas desde la parte superior así los cartuchos en un solo pocillo de una placa de 96 pocillos PCR recomendadas tal como se indica en el protocolo del fabricante del instrumento. Sinte: Con una pipeta multicanal es ideal para la transferencia de las emulsiones de gotitas. Se recomienda la aspiración lenta y suave de gotas para minimizar esquila de gota durante los traslados.
- Selle la placa de PCR con papel inmediatamente después de la transferencia de las gotas para evitar la evaporación. Utilice sellos de placa lámina perforables que son compatibles con el sellador de placas PCR y las agujas en el lector de gotas. Siga las instrucciones del manual de instrucciones sellador de placas PCR.
- Ajuste la temperatura de la placa selladora a 180 ° C y el tiempo de 5 seg.
- Toque la flecha para abrir la puerta de la bandeja. Coloque el bloque de soporte en la bandeja con la cara de 96 pocillos para arriba. Coloque la placa de 96 pozos en el bloque de ayuda y asegurarse de que todos los pocillos de la placa están alineados con el bloque de soporte.
- Cubra la placa de 96 pocillos con 1 hoja de papel de aluminio sellado perforable. Una vez que la placa de 96 pocillos se fija en el bloque de soporte y se cubre con el sello de aluminio que se puede perforar, toque el botón del sello. La bandeja se cerraráy termosellado iniciará.
- Cuando termosellado es completa, la puerta se abre de forma automática; retire la placa del bloque de calor de ciclos térmicos y luego retire el bloque de calor.
- Asegúrese de que todos los pozos de la placa están sellados comprobando que las depresiones en la lámina son evidentes sobre cada pocillo. Una vez sellado, el plato está listo para el ciclo térmico.
- Una vez que se eliminan las gotas, presione los pestillos en el soporte del cartucho para abrirla. Retire el cartucho vacío y deséchelo.
- Realizar la amplificación por PCR convencional siguiendo los parámetros detallados en la Tabla 2.
2.3 la lectura de la gotita (según el protocolo recomendado por el fabricante)
Nota: Después de la amplificación por PCR del ácido nucleico diana en las gotitas, el instrumento lector de gotita analiza cada gotita de forma individual utilizando un sistema de detección de 2 colores 13. Normalmente nos propusimos detectar un FAMfluoróforos reportero d VIC.
- Haga clic en "sistema Flush" para cebar el lector de gota y que quede listo para el análisis ddPCR.
- Cargue la placa en el lector de la gotita y haga clic en "start". El lector gotita aspira cada muestra, singulariza las gotas, y los arroyos en fila junto a un detector de 2 colores. El detector lee las gotitas para enumerar muestras positivas y negativas.
- Cuando la lectura de gota, abra la puerta y retire el soporte de la placa de la unidad. Retire la placa de PCR de 96 pocillos del soporte y descartarlo.
- Para el mantenimiento adecuado, reemplace el aceite lector de gota y vaciar el recipiente de residuos, según sea necesario. Añadir 50 ml de 10% de lejía a la botella de residuos para prevenir el crecimiento microbiano.
2.4 ADN perfiles mutación (según el protocolo recomendado por el fabricante)
Nota: Las gotas de PCR-positivos y PCR-negativos se cuentan para proporcionar quantific absolutaación de las mutaciones del ADN diana BRAF V600E en forma digital, utilizando el software de análisis de datos.
- Haga clic en Configuración para introducir información sobre las muestras, ensayos y experimentos.
- En la ventana de configuración, cargue una placa (filename.qlp), a continuación, haga clic en Analizar para abrir y analizar los datos. La interfaz de análisis de datos se divide en tres ventanas: Resultados de mesa, Selector Bueno y Procesado de Datos / pantalla gráfica.
- En la ventana de datos procesados, los datos de concentración para cada objetivo aparecen en los pozos en el mapa plato y se tabulan en la Tabla de Resultados. Haga clic Concentración de visualizar datos en parcelas de concentración.
Resultados
Para nuestro análisis ddPCR, estudiamos las de referencia FFPE mutación líneas celulares estándar BRAF V600E. El lector de gota se conecta a un software de análisis de datos portátil ordenador en funcionamiento. Cada gotita individual se define sobre la base de la amplitud fluorescente como positivo o negativo. El software proporcionado por el fabricante también permite un punto de corte definido por el usuario a introducir para definir el umbral entre las gotitas positivos y negativos. El número de gotitas positivos y negativos en una muestra se utiliza para calcular la concentración de objetivo en términos de copias / l.
La fluorescencia se detecta y se procesa en un display gráfico de dispersión bidimensional, de software personalizado se utiliza para dibujar puertas apropiados para cada clúster punto final gotita, y se contó el número de gotas dentro de cada puerta. Como se muestra en la Figura 3A, gotitas representado por puntos azules (señal de fluorescencia FAM) por encima de la línea de corte para toda samples (línea rosa) fueron positivas para mutación BRAF V600E. Gotitas representados por puntos azules en la BRAF WT (NTC; carril 2) muestra podrían ser debido a una señal no específica (falso positivo). Señales positivas falsas (BRAF WT) se normalizaron con otras muestras de mutación. Como se muestra en la Figura 3B, las gotas de WT BRAF están representados por puntos verdes (señal de fluorescencia VIC). En ambas parcelas, los puntos grises en la parte inferior son considerados como la fluorescencia de fondo. Las frecuencias globales alelo mutante se calcularon utilizando los datos mostrados en la Figura 3C, en base a los porcentajes relativos de WT BRAF V600E BRAF y plantillas detectados. Obtenido resultados ddPCR contienen los recuentos de eventos gotita y calculado de tipo salvaje y molécula de ADN mutante que cuenta para la V600E BRAF (50%, 10%, 5%, 1%, 0,5%, 0,1% y 0,05%) muestras calculadas utilizando el siguiente fórmula mencionada.
% De la frecuencia Mutant = (Mutantcopiar / (Wildtype + copia mutante)) x 100
Por consiguiente, se identificaron y se verifican con patrón de referencia (BRAF WT) mutaciones BRAF V600E. Mutación V600E BRAF Definido frecuencias alélicas de 50%, 10%, 5%, 1%, 0,5%, 0,1% y 0,05% se utilizaron para probar la sensibilidad y reproducibilidad del sistema ddPCR. De nuestro análisis con concentraciones de las muestras conocidas, nos confirmó que ddPCR es capaz de detectar un precio tan bajo como 0.05% de BRAF V600E mutación. La detección de falsos positivos en la cuenta mutante NTC o WT, posiblemente, podría ser debido a la hidrólisis de la sonda no específica como se informó anteriormente 14. La detección de más de dos copias en una muestra ha sido considerado como positivo en el tejido tumoral 15.

Figura 1. Representación esquemática que describe reactivo y carga de la muestra en la preparación para aut instrumento de extracción de ADN omated. Colocar las muestras en unos bastidores portadores y dispensar los reactivos en los correspondientes canales como se ha mencionado. Empleando protocolo automatizado TPS que soporta múltiples tipos de muestras, entrega precisa y resultados fiables con la máxima productividad. Re-impreso con el permiso de Siemens Healthcare Diagnostics. (cortesía de Siemens Healthcare Diagnostics).

Figura 2. Representación esquemática de la Tissue Sistema de Preparación de flujo de trabajo para la extracción de ADN automatizado. Procedimiento de aislamiento de ADN totalmente automatizado para tejidos FFPE secciones, incluyendo los pasos de selección negativos de parafina, eliminación de restos de tejido y los pasos positivos de selección de unión y la elución se muestran. Re-impreso con el permiso de Siemens Healthcare Diagnostics. (cortesía de Siemens Healthcare Diagnostics).
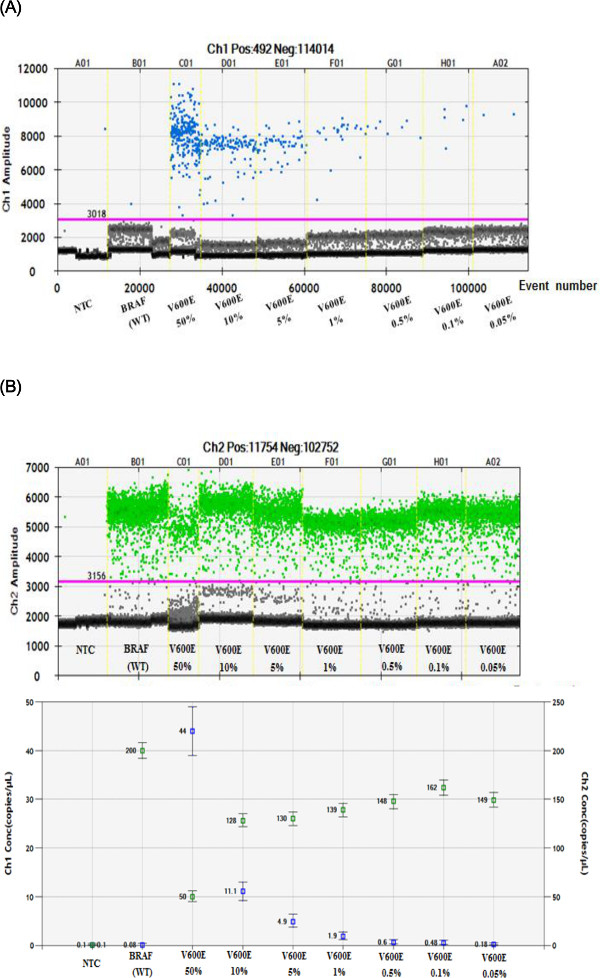
Figura 3. El uso del sistema ddPCR para la cuantificación precisa de la BRAF V600E mutación en muestras de línea celular estándar FFPE de referencia. (A, B) Visualización de amplitudes de fluorescencia positivos en parcelas 1D (1dot -1droplet). Puntos azules (A, FAM positivos) representan BRAF V600E gotas positivo mutantes, mientras que los puntos verdes (B, VIC positivo) representan WT BRAF gotas -positivos. Esta determinación permite la cuantificación precisa mutación en FFPE de referencia líneas celulares estándar. La línea rosa es el umbral de discriminación entre las señales positivas y negativas de las gotas (C) La trama abundancia fraccional muestra marcadores azules que indican la concentración (copias / l) de la mutación BRAF V600E, y.los marcadores verdes indican la concentración (copias / l) de BRAF (WT). Todas las barras de error generados por el software de análisis de datos representan el intervalo de confianza del 95%.
| Reactivos | Volumen (ml) |
| Tampón de Lisis | 106 ml |
| Tampón de lavado 1 | 101 ml |
| Tampón de lavado 2 | 72 ml |
| Wash Buffer 3 | 106 ml |
| Tampón de elución | 19 ml |
| Las perlas magnéticas | 8 ml |
| Búfer FFPE | 15 ml |
| Proteinasa K | 3,3 ml |
Tabla 1. Volumen total de reactivos (kit TPS) requerido para la extracción de ADN con 48 muestras.
| Ciclismo Paso | La temperatura | Hora | # Ciclos |
| Activación de la enzima | 95 ° C | 10 minutos | 1 |
| La desnaturalización | 94 ° C | 30 sec | 40 |
| El recocido / extensión | 60 ° C | 1 minuto * | |
| Mantener | 98 ° C | 10 minutos | 1 |
| Mantener | 4 ° C | Eternidad | 1 |
| * Ajustar la configuración del tipo de rampa a 2-2,5 ° C / seg. Use una tapa térmica ajustado a 105 ° C y ajuste el volumen de la muestra de 40 l | |||
Tabla 2. Condiciones de termociclado de PCR convencional
Discusión
En este sentido, destacamos la aplicabilidad de ddPCR y el aislamiento de ADN a partir de muestras de líneas celulares estándar FFPE de referencia para una evaluación específica de mutación genética. En este estudio, se utiliza TPS método de aislamiento de ADN automatizado que se puede adaptar fácilmente y automatizado, y tiene capacidad para un máximo de 48 muestras diferentes de forma simultánea, lo que permite más grandes experimentos a escala y una menor variabilidad. Una de las limitaciones del aislamiento del ADN en el presente trabajo es que todas las muestras FFPE es única, y variará entre sí de contaminantes de la superficie, la flora microbiana, y / o antecedentes genéticos humanos. En general, la calidad del ADN extraído y la cantidad y el éxito de la amplificación de ADN genómico entero dependen de varios parámetros antes, durante y después de la extracción. Estos incluyen, el tipo y cantidad de tejido, tipo de fijador utilizado para la conservación de tejidos, duración de la fijación, la edad de las condiciones de bloques de parafina y de almacenamiento, así como la longitud del segmento de ADN deseado para ser analizados 16. La eliminación de la parafina de los tejidos es el paso más crítico para la extracción exitosa como parafina no disuelto conduce a la mala calidad de la muestra. Durante la generación de gotas, se debe tener cuidado para evitar la formación de burbujas - este es otro paso fundamental para la detección de mutaciones éxito. Teniendo en cuenta la muestra a otra variación que pudiera surgir entre las poblaciones de la muestra y se basa en el motivo del experimento, ciertas modificaciones en el procedimiento podrían ser necesarias para obtener el resultado deseado.
Otra ventaja es que el aislamiento de ADN y ddPCR se lleva a cabo mediante sistemas automatizados en este protocolo, y por lo tanto hay un error insignificante y la intervención del usuario requerida es mínima. Aislamiento de ADN de todo el genoma-amplificado a partir de células / tejidos embebidos en parafina se obtuvo mediante el uso de sistema de TPS. Uno de los inconvenientes en el uso de sistema de aislamiento de ADN automatizado es que, no es rentable utilizar pequeño número de muestras. En lugar de esto ste automatizadop, otro procedimiento de aislamiento de ADN estándar también se podría realizar para muestras limitadas
Un estudio reciente indica que el uso de gotita PCR digital (ddPCR) es capaz de determinar el número de copias relativo de loci genómico específico incluso en presencia de tejido normal entremezclados obtenido a partir de tejidos FFPE. Mediante el uso de una serie de diluciones de control, Nadauld, L. et al. Determina los límites de detección (LOD) para el ensayo ddPCR y reportó su sensibilidad mejorada en cantidades mínimas de ADN en comparación con el tiempo real de PCR estándar 17. Aquí, FFPE de referencia líneas celulares estándar se utilizan para demostrar la capacidad de detección de la mutación del sistema ddPCR. Los resultados del sistema ddPCR indicaron la posibilidad de detectar las frecuencias alélicas rara mutación hasta 0,05% mutación. Colectivamente, estos datos indican que el sistema ddPCR también permite el análisis cuantitativo de los porcentajes de varios alelos mutantes y diferencias relativas en la muestra de tumor clínico heterocigotoss. Gran número de muestras FFPE se puede analizar de mutaciones de genes específicos simultáneamente y esta es una técnica óptima para estudios genéticos en toda la población.
Finalmente, debe tenerse en cuenta que las frecuencias de mutación están representados aquí están cuantificación absoluta y no deben ser considerados como valor relativo de la tasa de mutación o frecuencia. ddPCR lectura proporciona una cuantificación absoluta de la mutación de ADN diana. Estos valores se pueden utilizar para la validación de las frecuencias de mutación de las muestras preparadas bajo las mismas condiciones, y se secuenciaron sobre la misma región. Sin embargo, estos valores absolutos son reproducibles y pueden ser utilizados para la comparación cuantitativa de la distribución y la frecuencia de mutación cuando se controlan los parámetros óptimos. En conclusión, ddPCR ha surgido recientemente como una herramienta robusta que da cuantificación absoluta de los ácidos nucleicos en muestras de FFPE y biopsia y también puede ser a doble cara con los ensayos de referencia para la determinación de o bien noconcentraciones de transcripción rmalized o el número de copias de ADN.
Divulgaciones
Myung Ryuri Oh, Si Eun Kim, y Young Deug Kim son empleados de ABION CRO.
Agradecimientos
Esta investigación fue apoyada por el Programa de Investigación y Desarrollo para la Sociedad de la Fundación Nacional de Investigación (NRF), financiado por el Ministerio de Ciencia, TIC y planificación de futuro (Grant No. 2013M3C8A1075908).
Materiales
| Name | Company | Catalog Number | Comments |
| Hamilton MICROLAB STARlet IVD instrument | Siemens | 10701001 | Automated DNA isolation instrument |
| QX200 Droplet Generator | Bio-Rad | 772BR1119 | |
| QX200 Droplet Reader | Bio-Rad | 771BR1497 | |
| Conventional PCR machine capable of ramp-time adjustment | 621BR17718 | ||
| PX1 PCR plate sealer | Bio-Rad | 770BR1575 | |
| QuantaSoft software | Bio-Rad | ||
| DNA isolation kit | |||
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632398 | |
| VERSANT Tissue Preparation Reagents Box 1 | Siemens | 10632399 | |
| CO-RE tips | Siemens | ||
| ddPCR mutation analysis | |||
| ddPCR Supermix | Bio-Rad | BR186-3010 | 2X concentration |
| DG8 cartridge | Bio-Rad | BR186-4008 | |
| Droplet Generator oil | Bio-Rad | BR-186-3005 | |
| Gasket | Bio-Rad | BR186-3006 | |
| Droplet reader oil | Bio-Rad | BR-186-3004 |
Referencias
- Vogelstein, B., et al. Genetic alterations during colorectal-tumor development. The New England journal of medicine. 319, 525-532 (1988).
- Davies, H., et al. Mutations of the BRAF gene in human cancer. Nature. 417, 949-954 (2002).
- Solit, D. B., et al. BRAF mutation predicts sensitivity to MEK inhibition. Nature. 439, 358-362 (2006).
- Wong, K. K. Recent developments in anti-cancer agents targeting the Ras/Raf/ MEK/ERK pathway. Recent patents on anti-cancer drug discovery. 4, 28-35 (2009).
- Brose, M. S., et al. BRAF and RAS mutations in human lung cancer and melanoma. Cancer research. 62, 6997-7000 (2002).
- Wang, L., et al. BRAF mutations in colon cancer are not likely attributable to defective DNA mismatch repair. Cancer research. 63, 5209-5212 (2003).
- Hindson, B. J., et al. High-Throughput Droplet Digital PCR System for Absolute Quantitation of DNA Copy Number. Anal Chem. 83, 8604-8610 (1021).
- Pinheiro, L. B., et al. Evaluation of a Droplet Digital Polymerase Chain Reaction Format for DNA Copy Number Quantification. Anal Chem. 84, 1003-1011 (1021).
- Jones, M., et al. Low copy target detection by Droplet Digital PCR through application of a novel open access bioinformatic pipeline, 'definetherain'. Journal of virological methods. 202, 46-53 (2014).
- Strain, M. C., et al. Highly precise measurement of HIV DNA by droplet digital PCR. PloS one. 8, e55943(2013).
- Miotke, L., Lau, B. T., Rumma, R. T., Ji, H. P. High sensitivity detection and quantitation of DNA copy number and single nucleotide variants with single color droplet digital PCR. Anal Chem. 86, 2618-2624 (2014).
- Bizouarn, F. Clinical applications using digital PCR. Methods in molecular biology. 1160, 189-214 (2014).
- McDermott, G. P., et al. Multiplexed target detection using DNA-binding dye chemistry in droplet digital PCR. Anal Chem. 85, 11619-11627 (2013).
- Milbury, C. A., et al. Determining lower limits of detection of digital PCR assays for cancer-related gene mutations. Biomolecular detection and quantification. 1, 8-22 (2014).
- Zhu, G., et al. Highly Sensitive Droplet Digital PCR Method for Detection of EGFR Activating Mutations in Plasma Cell-Free DNA from Patients with Advanced Non-Small Cell Lung Cancer. The Journal of molecular diagnostics: JMD. , (2015).
- Fan, H., Gulley, M. L. DNA extraction from paraffin-embedded tissues. Methods in molecular medicine. 49, 1-4 (2001).
- Nadauld, L., et al. Quantitative and Sensitive Detection of Cancer Genome Amplifications from Formalin Fixed Paraffin Embedded Tumors with Droplet Digital PCR. Translational medicine. 2, (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados