Method Article
A detecção in situ de bactérias dentro dos tecidos de parafina-encaixados Usando um marcado com Digoxina DNA Probe Segmentação 16S rRNA
Neste Artigo
Resumo
Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled 16S rRNA-targeting DNA probes has been described. This protocol can be applied to study the role of bacteria in various diseases such as periodontitis, cancers, and inflammatory immune diseases.
Resumo
The presence of bacteria within the pocket epithelium and underlying connective tissue in gingival biopsies from patients with periodontitis has been reported using various methods, including electron microscopy, immunohistochemistry or immunofluorescence using bacteria-specific antibodies, and fluorescent in situ hybridization (FISH) using a fluorescence-labeled oligonucleotide probe. Nevertheless, these methods are not widely used due to technical limitation or difficulties. Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled DNA probes has been introduced. The paraffin-embedded tissues are the most common form of biopsy tissues available from pathology banks. Bacteria can be detected either in a species-specific or universal manner. Bacterial signals are detected as either discrete forms (coccus, rod, fusiform, and hairy form) of bacteria or dispersed forms. The technique allows other histological information to be obtained: the epithelia, connective tissue, inflammatory infiltrates, and blood vessels are well distinguished. This method can be used to study the role of bacteria in various diseases, such as periodontitis, cancers, and inflammatory immune diseases.
Introdução
As bactérias desempenham um papel na etiologia de diversas doenças orais, tais como periodontite, pulpite, pericoronite, celulite, osteomielite e. A fim de compreender o papel das bactérias na patogénese da doença e para monitorizar o efeito de tratamentos, localização de bactérias dentro do tecido é importante. A presença de bactérias no interior do tecido gengival a partir de pacientes com periodontite foi demonstrado usando vários métodos, incluindo microscopia electrónica de 1,2, imuno-histoquímica e imunofluorescência utilizando anticorpos específicos para cada bactéria 3-7, e hibridação in situ fluorescente (FISH) 8 utilizando um fluorescence- sonda oligonucleotídica marcada segmentação 16S rRNA. No entanto, estes métodos não são amplamente utilizados devido a limitações técnicas ou dificuldades. Comparado com os anticorpos, as sondas alvo rRNA 16S são fáceis de produzir e alcançar espécie-especificidade. FISH tem provado ser uma excelente ferramenta para a visualização de bactériaeria em seus ambientes naturais, como biofilme placa. No entanto, a aplicação de FISH para amostras de tecidos é limitado devido a autofluorescência de vários componentes do tecido. Por exemplo, o forte autofluorescência das células vermelhas do sangue, muitas vezes dificulta a aplicação da tecnologia de fluorescência para tecidos inflamados quando elas envolvem sangramento 9.
De forma a localizar as bactérias dentro dos tecidos gengivais inflamadas, por conseguinte, um método de hibridação in situ, utilizando uma digoxigenina (DIG) sonda de DNA marcado com foi desenvolvido e aplicado com sucesso 10,11. Aqui, um protocolo detalhado para a localização de bactérias no interior de tecidos embebidos em parafina, utilizando P. gingivalis sondas eubacterianas espec�icos e universais tem sido descrita. É particularmente focado na padronização do método de modo que resultados semelhantes podem ser reproduzidos em outros laboratórios. Este protocolo permite a localização de bactérias dentro de seu contexto histológica e do results são altamente reprodutível. O protocolo descrito pode ser utilizado para localizar bactérias ou de uma forma específica para a espécie ou universal em diversos tecidos. A sonda universal é especialmente útil para detectar bactérias em doenças polimicrobianas e para estudar o potencial papel da bactéria, em doenças em que o papel de bactérias específicas que não é conhecido.
Protocolo
1. Preparação Probe
- A amplificação por PCR da sonda
- Para o P. sonda gingivalis -específica, amplificar um fragmento de DNA 343-bp do P. gingivalis 16S rRNA por PCR utilizando o DNA genómico de P. gingivalis e os seguintes iniciadores: 5'TGC AAC TTG CCT TAC AGA GG-3 'e 5' AGIR CGT ATC CGT TAT GCC TC-3 '10. Realizar amplificações com as seguintes condições de ciclos: 35 ciclos a 95 ° C durante 30 seg, 60 ° C durante 30 seg, e 72 ° C durante 1 min 20 seg seguido de uma extensão de 5 min a 72 ° C 10,11.
- Amplificar a sonda eubacterial usando um 70-bp fragmento de DNA (5'-CAGGTGCTGCATGGCTGTCGTCAGCTCGTGTTGTGAAATGTTGGGTTAAG
TCCCGCAACGAGCGCAACCC-3 ') sintetizado como um oligonucleótido e os seguintes iniciadores: 5'-CTG CAG GTR GGY CMT-3' e 5'-AGG TTG GCG CTC TTG-3 '11. Realizar amplificações com as seguintes condições de ciclos: 40 ciclosa 9 4 ° C durante 30 seg, 60 ° C durante 30 seg e 72 ° C durante 1 min 20 s, seguido de uma extensão de 5 min a 72 ° C 11,12. - Precipitar os produtos de PCR (≥500 ul) pela adição de acetato de sódio 3M (pH 5,2) e etanol a 100% (a uma 10: 1: 2 ratio) e incubação a -20 ° C durante 2 horas.
- Centrífuga-se a mistura a 14000 xg durante 15 min, ressuspender o sedimento em 100 ul de tampão TE, e medir a concentração de DNA por densidade óptica a 260 nm.
- DIG-rotulagem utilizando rotulagem e detecção de DNA kit DIG
- Misturar 3 ug da sonda com água para um volume final de 15 ul.
- Desnaturar o ADN, a 95 ° C durante 10 min e relaxar rapidamente em gelo.
- Adicionar 2 mL de 10x mistura hexanucleotide, 2 ul de mistura de dNTP a rotulagem, e 1 ul da enzima Klenow para 15 ul do ADN desnaturado.
- Misturar e incubar a 37 ° C durante 24 h a 48 h e parar de reacção por aquecimento a 65 ° C.
- Verifique a sensibilidade da sonda marcada com DIG usando um kit de marcação e detecção de ADN DIG
- Carregar manualmente 1 ml de diluições em série da sonda de controlo positivo fornecido com o kit e 1 ng de sonda marcada com DIG na membrana de nylon e seco durante 5 min à TA.
- Resumidamente mergulhar a membrana no tampão 2 x SSC (NaCl 0,3 M, citrato de sódio 30 mM, pH 7,0) e incuba-se a membrana a 80 ° C durante 1 h para imobilizar o ADN.
- Resumidamente mergulhar a membrana no tampão de ácido maleico (MAB, 0,1 M de ácido maleico, 0,15 M de NaCl, pH 7,5) e incuba-se com fosfatase alcalina anti-DIG (AP) anticorpo conjugado a diluída (1: 5000) em 6 ml de solução de bloqueio, previsto no kit à TA durante 30 min.
- Lava-se a membrana com MABT (MAB contendo 1% de Tween 20) e aplicam-se 40 ul da solução de NBT / BCIP misturado com tampão de detecção de 2 ml (0,1 M de Tris-HCl, 0,1 M de NaCl, 0,05 M de MgCl2, pH 9,5) para o membrana durante 1 min. Se a sensibilidade da sonda marcada énão satisfatória, repita os passos a partir de 1.1.3 para 1.3.4.
- Medir a concentração de DNA da sonda marcada (DO260) e ajustar a concentração de 100 ng mL -1.
- Verifique a especificidade da sonda marcada com DIG
- Desnaturar amostras de ADN genómico (25 ng de 3 ul -1 por cada amostra) a partir de várias espécies bacterianas, incluindo a bactéria alvo da sonda específica, a 95 ° C durante 10 min e relaxar rapidamente em gelo.
- Carregar manualmente o ADN desnaturado na membrana de nylon e seco durante 20 min à TA.
- Resumidamente mergulhar a membrana no tampão 2 x SSC e incubar a membrana a 80 ° C durante 2 horas para imobilizar o ADN.
- Bloquear a membrana com uma solução de bloqueio à temperatura ambiente durante 1 h.
- Dilui-se a sonda a 1 ng mL -1 em tampão de hibridação, desnaturar as sondas diluído, e aplicam-se sobre a membrana.
- Incubar a membrana a 45 ° C durante 1 h.
- Lava-se a membrana trêsvezes com tampão 2 x SSC.
- Resumidamente mergulhar a membrana no MAB.
- Incubar com anticorpo anti-DIG-AP conjugado diluído (1: 2000) em 6 ml de solução de bloqueio à temperatura ambiente durante 1 h.
- Lava-se a membrana com MABT e aplicar 40 ul da solução de NBT / BCIP misturado com tampão de detecção de 2 ml.
- Quando a sonda não conseguir a especificidade esperada, aumentar a temperatura até que a especificidade de hibridação é observado.
2. Hibridação In Situ
Nota: Para evitar a secagem das amostras e reagentes, execute todas as incubações em câmara úmida forrada com papel-toalha molhada.
- De-parafinização e re-hidratação de secções de tecido
- Prepare a 4 mm de espessura secções de tecido a partir de blocos embebidos em parafina. Formalina, paraformaldeído, e fixador à base de zinco são compatíveis com este protocolo.
- Prepare todos os reagentes que utilizam água tratada com DEPC.
- Lâminas de calor num forno seco a 60 ° C durante 30 min e incubar à TA durante 30 min.
- Imergir as lâminas em 100% de xileno durante 5 minutos três vezes; use xileno fresco cada vez.
Nota: Faça isto procedimento de depilação de em um exaustor química. - Imergir as lâminas em etanol a 100% durante 5 min, duas vezes, com etanol fresco de cada vez, e re-hidratar as amostras em série de 90%, 80% e soluções de etanol a 70% durante 5 min cada.
- Imergir as lâminas em água tratada com DEPC para 1 min e lavagem das lâminas em PBS tratado com DEPC (pH 7,4) durante 6 min.
- Os pré-tratamentos de cortes de tecido
- Imergir as lâminas em ácido clorídrico 0,1 N durante 20 minutos e lavagem das lâminas em água tratada com DEPC para 30 seg.
- Desenhe uma barreira hidrofóbica ao redor espécimes usando uma caneta cera.
Nota: O tamanho da barreira hidrofóbica depende do tamanho das amostras. Por conseguinte, os volumes de reagentes utilizados nos passos seguintes variam dependendo do tamanho das amostras, mas tem que preencher o HYdrophobic barreira (50 ul para pequenas amostras e 400 ul para grandes amostras). - Tratar as amostras com 50 a 400 ul de proteinase K (1 a 10 ug ml -1 em PBS tratado com DEPC) num forno seco a 37 ° C durante 30 minutos e lavagem das lâminas em tratada com DEPC 1x PBS durante 1 min.
- Soak lâminas em paraformaldeído a 4% em PBS tratado com DEPC durante 10 min, em seguida, lavagem das lâminas em PBS tratado com DEPC a 1 min.
- Soak lâminas em trietanolamina 0,1 M-HCl (pH 8,0) contendo 0,5% de anidrido acético durante 20 minutos e lavagem das lâminas em água tratada com DEPC para 30 seg.
- A hibridação com sonda e lavagem
- Imergir as lâminas em tampão SSC 2x para 20 min.
- Dilui-se a sonda a 1 ng mL -1 em tampão de hibridação (SSC 4x, 50% [vol / vol] formamida, solução de Denhardt 1x, 10% [peso / vol] de sulfato de dextrano, 0,1% [peso / vol] de sulfato de dodecilo de sódio, 0,4 mg ml -1 de DNA de esperma de salmão). Para controlos negativos, misturar com a sonda marcadauma quantidade em excesso de 10 vezes de sonda não marcada.
- Desnaturar as sondas diluído e aplicam 50 a 400 ul para as secções de tecido. Coloque uma lamela sobre a lâmina e selar com unha polonês.
- Lâminas de calor na máquina de PCR a 90 ° C durante 10 min e incubar em uma câmara humidificada O / N a 45 ° C ou à temperatura óptima determinada no passo 1.4.11.
- Arrefecer as lâminas a 4 ° C durante 30 min, remover lamelas, e mergulhar as lâminas num tampão SSC série como seguintes: 4x SSC durante 10 min, pré-aquecido (45 ° C) 2x SSC durante 20 min, 2 x SSC durante 10 min, e 0,2x SSC durante 10 min.
- A detecção com anticorpo anti-DIG-AP conjugado
- Lavar as lâminas em MABT por 5 min.
- Bloco com 50 a 400 uL de solução com 1% de Tween 20 de bloqueio durante 20 minutos.
- Adicionar 50 a 400 ul de anticorpo anti-DIG-AP diluídos em solução (1: 1000) de bloqueio anld incubar a 37 ° C durante 90 min.
- Lavar as lâminas em MABT para 10 min.
- Imergir as lâminas em tampão de detecção contendo 1% de Tween 20 durante 5 min.
- Trata-se com 50 a 400 μ de levamisol 1 mM (em tampão de detecção contendo 1% de Tween 20) durante 5 min, para inactivar a fosfatase alcalina endógena.
- Distribuir 50 a 400 ul da solução pré-misturada de NBT / BCIP (diluída em tampão de detecção a 1: 100) para cada amostra e incubar numa câmara humidificada à temperatura ambiente durante 2 a 3 horas até que o sinal desenvolvido é satisfatória.
- Lavar as lâminas com água desionizada para parar a visualização e aplicam 50 a 400 pi de 0,05% [peso / vol] verde de metilo como um contador de manchas a 37 ° C durante 10 min.
- Lavar as lâminas com água desionizada, desidratar em 100% de etanol, e mergulhar em xileno.
- Aplicar 80 ml de solução de montagem em cada slide e cobrir com lamelas.
Resultados
A Figura 1 mostra dot blot de sondas marcada com DIG em comparação com a sonda de controlo positivo fornecido com o kit para determinar a sua sensibilidade. A 343 pb P. sonda espec�ico gingivalis é de 25 vezes mais sensível do que a sonda eubacteriana 70 pb. A Figura 2 mostra a hibridização in situ de tecidos gengivais obtidos a partir de pacientes com periodontite crónica para a detecção de P. gingivalis e eubactérias. Os sinais bacterianos, mostrado na violeta, foram detectadas no âmbito do epitélio, lamina própria e de biofilme localizado fora do tecido. No entanto, a distribuição de P. gingivalis e eubactérias dentro do tecido gengival foi diferente. Em alta ampliação (1000X), várias formas (cocos, haste, fusiforme, e forma cabeludo) de bactérias eram visíveis utilizando a sonda eubacterial, enquanto apenas haste em forma de bactérias foram detectadas pelo P. gingivalis espec�ico sonda. As formas dispersas de sinais bacterianosForam também observadas.
Hajishengallis et al. demonstraram o papel essencial de bactérias comensais no desenvolvimento de P. gingivalis induzida pela periodontite experimental em ratos 14. A aplicação de sulfato de sódio dextrano (DSS), uma junção apertado (TJ) -disrupting química, na mucosa gengival periodontite induzida e perda óssea alveolar em camundongos 13. A sonda detectada com sucesso eubacteriana bactérias dentro dos tecidos gengivais do grupo de DSS que não foram detectadas pelo P. gingivalis espec�ico sonda (Figura 3).
A sonda eubacteriana é útil para o estudo do potencial envolvimento de bactérias em outras doenças. Dado que a Agência Internacional de Investigação do Cancro designado Helicobacter pylori como grau I carcinógeno para o câncer gástrico 15, potencial envolvimento de bactérias no desenvolvimento de outros tipos de câncer tem sido amplamente estudado. Quando seCÇÕES de biópsias pulmonares diagnosticados com carcinoma de células escamosas foram hibridados in situ com a sonda de eubactérias, as bactérias foram detectadas tanto no interior das células tumorais e entre o estroma circundante / células infiltrantes (Figura 4). A hibridação in situ utilizando a sonda eubacteriana foi também aplicado a explorar a presença de bactérias nas lesões de líquen plano oral, uma doença imune inflamatória de etiologia desconhecida. Curiosamente, foram detectados sinais bacterianos não só dentro do epitélio mas também na lâmina própria, onde foi observada a infiltração de banda. Sob alta ampliação, os sinais bacterianos foram detectados nos núcleos de muitas células (Figura 5).

Figura 1. Teste de Sensibilidade das sondas DIG-rotulados. Sondas marcadas com personalizados diluída em série e um contr positivosonda ol fornecida no kit foram ponto colocados a hibridar com anticorpo anti-DIG-AP conjugado. Após a adição do substrato, o borrão com o P. gingivalis sonda espec�ico foi desenvolvido durante 30 segundos, enquanto o blot com a sonda eubacterial foi desenvolvido para 1 min. Por favor clique aqui para ver uma versão maior desta figura.
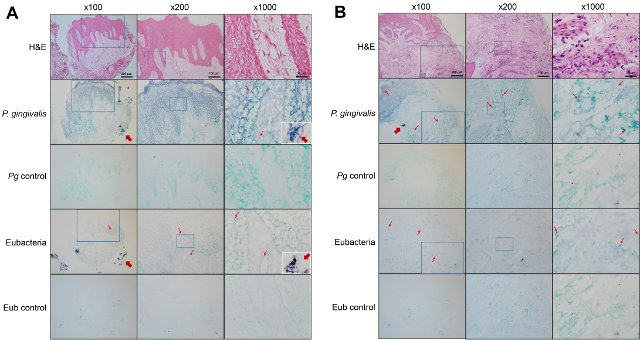
Figura 2. A detecção de bactérias dentro dos tecidos gengivais obtidos a partir de pacientes com periodontite crónicas. Secções de biópsias gengivais do local saudável (A) e o local doente (B) de um paciente com periodontite foram submetidas à coloração de hematoxilina-eosina (H & E) coloração ou A hibridação in situ com ou P. gingivalis sonda espec�ico ou a sonda eubacterial. Negative controlos foram hibridadas com a sonda misturado com 10 vezes o excesso de sonda não marcada. A área ampliada é indicado com uma caixa quadrada. Sinais positivos representativos, mostrado em violeta, estão marcados com setas finas. Setas grossas indicam biofilme placa fora do tecido. O biofilme placa examinados com a ampliação de 1000X são mostrados em inserções. Por favor clique aqui para ver uma versão maior desta figura.
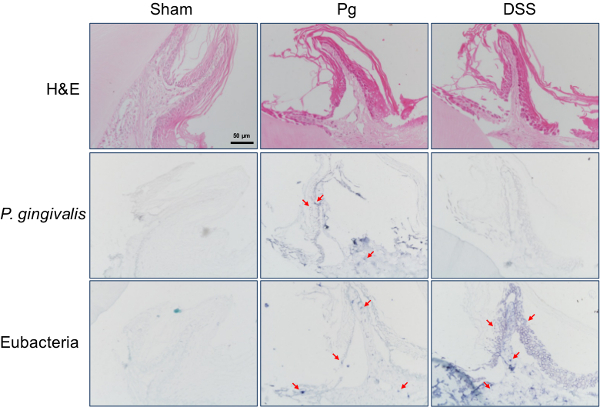
Figura 3. Detecção de bactérias comensais orais no interior do tecido gengival de rato. Periodontite experimental foi induzida em ratinhos BALB / c por inoculação oral de P. gingivalis (Pg) ou aplicação de DSS em mucosa gengival. Secções gengivais de ratos foram submetidos a coloração com H & E ou hibridação in situ, quer com o < em> P. gingivalis sonda espec�ico ou a sonda eubacterial. Sinais positivos representativos, mostrado em violeta, estão marcados com setas. Ampliação original é x400. Por favor clique aqui para ver uma versão maior desta figura.
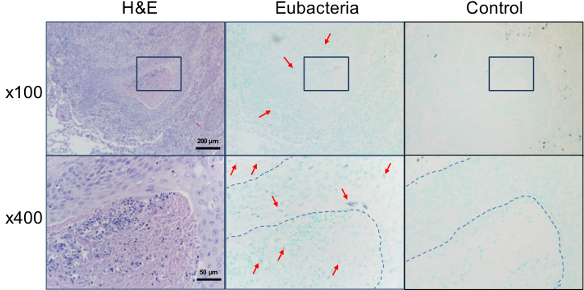
Figura 4. Detecção de bactérias dentro lesão câncer. As secções de biópsias pulmonares de pacientes com diagnóstico de carcinoma de células escamosas foram coradas com H & E ou in situ hibridizado com a sonda ou eubacterial universal ou a sonda de controlo negativo. A área ampliada é indicado com uma caixa quadrada. Sinais positivos representativos, mostrado em violeta, estão marcados com setas. Beira das células tumorais são indicados com linhas pontilhadas.arget = "_ blank"> Clique aqui para ver uma versão maior desta figura.
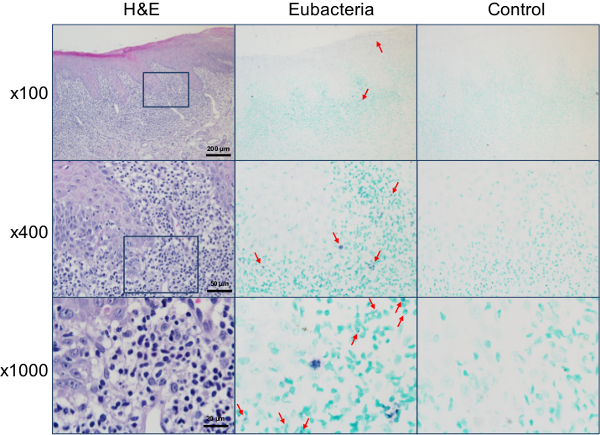
Figura 5. Detecção de bactérias dentro das lesões de líquen plano bucal. As seções de biópsia de mucosa bucal diagnosticados como líquen plano bucal foram coradas com H & E ou in situ hibridizado com a sonda ou eubacterial universal ou a sonda de controlo negativo. As áreas ampliadas são indicados com uma caixa quadrada. Sinais positivos representativos, mostrado em violeta, estão marcados com setas. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
Aqui um protocolo para localizar bactérias dentro dos tecidos embebidos em parafina, utilizando uma sonda de DNA marcada com DIG tem sido descrita. A sonda tem como alvo as moléculas de ADN ou ARN do gene 16S do rRNA bacteriano, e as sondas de rRNA 16S-alvo pode ser concebido como uma ou outra espécie específica, ou universal. A hibridação específica da P. gingivalis espec�ico sonda para P. gingivalis, mas não a outras bactérias orais tem sido mostrado previamente 10. Em contraste, a sonda hibridada com eubacteriana todo o ADN genómico bacteriano testadas (17 espécies diferentes), embora a eficiência de hibridação variou 12. O número de t-nucleotídeos dentro da sonda determina a eficiência da DIG-rotulagem. Produção de uma sonda marcada com DIG com alta sensibilidade é o passo mais crítico para o sucesso da implementação do protocolo descrito. Tratamento das amostras com ácido clorídrico melhora a relação sinal-ruído através da extracção de proteínas e parcial hydrolysis de sequências alvo. Um aumento na concentração de proteinase K aumenta a acessibilidade alvo de sondas, mas reduz a preservação do tecido. Portanto, recomenda-se a testar várias concentrações de proteinase K, dependendo dos tipos de tecido. O passo de acetilação diminui a ligação de fundo da sonda carregada negativamente para as proteínas do tecido por acetilação dos grupos amino carregados positivamente. Após uma série de etapas de preparação, as secções de tecido rasgar facilmente, deste modo, têm de ser manuseados com cuidado. O fundo é mínima em comparação com peixes. No entanto, os sinais a partir de tecidos com actividade de fosfatase alcalina endógena elevada, tais como ossos e glândulas mucosas deve ser interpretado de forma crítica.
As sondas de ADN amplificados por PCR são fáceis de produzir e estável em comparação com sondas de ARN. Detecção imunohistoquímica de sondas hibridizadas permite a identificação de estruturas histológicas através da comparação com secções H & E manchados: o epitélio,tecido conjuntivo, infiltrados inflamatórios, e os vasos sanguíneos são bem distinto. Uma das limitações do protocolo descrito, é a incapacidade de aplicar várias sondas ao mesmo tempo. Além disso, a exclusão dos sinais de contaminantes bacterianos na superfície das secções é limitado.
Este protocolo pode ser adaptado para detectar outros transcritos bacterianos dentro de secções de tecido 16. Além disso, este protocolo pode ser estendido a dupla marcação imuno-histoquímica com a detecção de um marcador de célula hospedeira.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este estudo foi apoiado por uma bolsa (2013R1A1A3005669) da Fundação Nacional da Coreia Investigação e uma subvenção (HI13C0016) do coreano Saúde Tecnologia R & D Projeto, Ministério da Saúde e Bem-Estar.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetic anhydride | Sigma | 6404 | |
| 50% Dextran sulfate solution | Millipore | S4030 | |
| 50X Denhardt’s solution | Sigma | D2532 | |
| DEPC | Sigma | P159220 | |
| DIG DNA labeling and detection kit | Roche | 11 093 657 910 | |
| Formamide | Sigma | F9037 | |
| ImmEdge™ Pen | Dako | H-400 | |
| Levamisole | Vector | SP-5000 | |
| Magnesium chloride | Sigma | 246964 | |
| Maleic acid | Sigma | M0375 | |
| Methyl green | Sigma | M6776 | |
| Paraformaldehyde | Sigma | P1648 | |
| Permount | Fisher | SP15-500 | |
| Salmon sperm DNA solution | Invitrogen | #15632-011 | |
| Sodium chloride | Sigma | S9625 | |
| Sodium citrate | Duksan | D1420 | |
| Sodium dodecyl sulfate | Amresco | 227 | |
| Triethanolamine-HCl | Sigma | 90279 | |
| Tris-HCl | Research organics | 3098T |
Referências
- Takarada, H., Cattoni, M., Sugimoto, A., Rose, G. G. Ultrastructural studies of human gingiva. II. The lower part of the pocket epithelium in chronic periodontitis. J. Periodontol. 45 (3), 155-169 (1974).
- Allenspach-Petrzilka, G. E., Guggenheim, B. Bacterial invasion of the periodontium; an important factor in the pathogenesis of periodontitis. J. Clin. Periodontol. 10 (6), 609-617 (1983).
- Saglie, F. R., et al. The presence of bacteria in the oral epithelium in periodontal disease. II. Immunohistochemical identification of bacteria. J. Periodontol. 57 (8), 492-500 (1986).
- Christersson, L. A., Albini, B., Zambon, J. J., Wikesjö, U. M., Genco, R. J. Tissue localization of Actinobacillus actinomycetemcomitans. in human periodontitis. I. Light, immunofluorescence and electron microscopic studies. J. Periodontol. 58 (8), 529-539 (1987).
- Saglie, F. R., Pertuiset, J., Rezende, M. T., Nestor, M., Marfany, A., Cheng, J. In situ. correlative immuno-identification of mononuclear infiltrates and invasive bacteria in diseased gingiva. J. Periodontol. 59 (10), 688-696 (1988).
- Rautemaa, R., et al. Intracellular localization of Porphyromonas gingivalis. thiol proteinase in periodontal tissues of chronic periodontitis patients. Oral Dis. 10 (5), 298-305 (2004).
- Marttila, E., et al. Intracellular localization of Treponema denticola. chymotrypsin-like proteinase in chronic periodontitis. J. Oral Microbiol. 6, (2014).
- Colombo, A. V., da Silva, C. M., Haffajee, A., Colombo, A. P. Identification of intracellular oral species within human crevicular epithelial cells from subjects with chronic periodontitis by fluorescence in situ. hybridization. J. Periodontal Res. 42 (3), 236-243 (2007).
- Abrams, K., Caton, J., Polson, A. Histologic comparisons of interproximal gingival tissues related to the presence or absence of bleeding. J. Periodontol. 55 (11), 629-632 (1984).
- Kim, Y. C., et al. Presence of Porphyromonas gingivalis. and plasma cell dominance in gingival tissues with periodontitis. Oral Dis. 16 (4), 375-381 (2010).
- Choi, Y. S., et al. Porphyromonas gingivalis. and dextran sodium sulfate induce periodontitis through the disruption of physical barriers. Eur. J. Inflammation. 10, 419-431 (2013).
- Choi, Y. S., Kim, Y. C., Ji, S., Choi, Y. Increased bacterial invasion and differential expression of tight junction proteins, growth factors, and growth factor receptors in periodontal lesions. J. Periodontol. 85 (8), e313-e322 (2014).
- Baker, G. C., Smith, J. J., Cowan, D. A. Review and re-analysis of domain-specific 16S primers. J. Microbiol. Methods. 55 (3), 541-555 (2003).
- Hajishengallis, G., et al. A Low-abundance biofilm species orchestrates inflammatory periodontal disease through the commensal microbiota and the complement. Cell Host Microbe. 10 (5), 497-506 (2011).
- Axon, A. Gastric cancer and Helicobacter pylori. Aliment. Pharmacol. Ther. 16 (suppl. 4), 83-88 (2002).
- Fenhalls, G., et al. In situ. detection of Mycobacterium tuberculosis transcripts in human lung granulomas reveals differential gene expression in necrotic lesions. Infect. Immun. 70 (11), 6330-6338 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados