Method Article
In Situ rilevamento di batteri all'interno dei tessuti in paraffina utilizzando una sonda DNA Digossina marcato targeting 16S rRNA
In questo articolo
Riepilogo
Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled 16S rRNA-targeting DNA probes has been described. This protocol can be applied to study the role of bacteria in various diseases such as periodontitis, cancers, and inflammatory immune diseases.
Abstract
The presence of bacteria within the pocket epithelium and underlying connective tissue in gingival biopsies from patients with periodontitis has been reported using various methods, including electron microscopy, immunohistochemistry or immunofluorescence using bacteria-specific antibodies, and fluorescent in situ hybridization (FISH) using a fluorescence-labeled oligonucleotide probe. Nevertheless, these methods are not widely used due to technical limitation or difficulties. Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled DNA probes has been introduced. The paraffin-embedded tissues are the most common form of biopsy tissues available from pathology banks. Bacteria can be detected either in a species-specific or universal manner. Bacterial signals are detected as either discrete forms (coccus, rod, fusiform, and hairy form) of bacteria or dispersed forms. The technique allows other histological information to be obtained: the epithelia, connective tissue, inflammatory infiltrates, and blood vessels are well distinguished. This method can be used to study the role of bacteria in various diseases, such as periodontitis, cancers, and inflammatory immune diseases.
Introduzione
I batteri svolgono un ruolo nell'eziologia di varie malattie orali come la parodontite, pulpite, pericoronite, cellulite e osteomielite. Per comprendere il ruolo dei batteri nella patogenesi della malattia e per monitorare l'effetto dei trattamenti, localizzazione di batteri all'interno del tessuto è importante. La presenza di batteri all'interno del tessuto gengivale di pazienti con periodontite è stato dimostrato utilizzando vari metodi, tra cui la microscopia elettronica 1,2, immunoistochimica e immunofluorescenza usando specifici anticorpi batteri-3-7 e ibridazione in situ fluorescente (FISH) 8 utilizzando un fluorescence- sonda oligonucleotide etichettato mira 16S rRNA. Tuttavia, questi metodi non sono ampiamente utilizzati a causa di limitazioni o difficoltà tecniche. Rispetto anticorpi, sonde destinate 16S rRNA sono facili da produrre e realizzare specie specificità. FISH ha dimostrato di essere un ottimo strumento per la visualizzazione di bacteria nei loro ambienti naturali, come la placca biofilm. Tuttavia, l'applicazione di FISH a campioni di tessuto è limitata a causa di autofluorescenza di varie componenti del tessuto. Ad esempio, il forte autofluorescenza di globuli rossi spesso ostacola l'applicazione della tecnologia di fluorescenza ai tessuti infiammati quando coinvolgono sanguinamento 9.
Per localizzare batteri all'interno dei tessuti gengivali infiammati, quindi, un metodo di ibridazione in situ usando un digossigenina (DIG) sonda di DNA -labeled è stato sviluppato e applicato con successo 10,11. Qui un protocollo dettagliato per la localizzazione di batteri nei tessuti inclusi in paraffina utilizzando P. gingivalis sonde eubatteriche specifico d'e universali è stata descritta. È particolarmente focalizzato sulla standardizzazione del metodo in modo che risultati simili possono essere riprodotti in altri laboratori. Questo protocollo permette la localizzazione dei batteri all'interno del loro contesto istologica e il resuLTS sono altamente riproducibili. Il protocollo descritto può essere utilizzato per localizzare batteri sia in modo specie-specifico o universale in vari tessuti. La sonda universale è particolarmente utile per rilevare batteri nelle malattie polimicrobiche e studiare un potenziale ruolo dei batteri nelle malattie dove il ruolo di batteri specifici non è noto.
Protocollo
1. Preparazione della sonda
- Amplificazione PCR della sonda
- Per la P. Sonda gingivalis -specific, amplificare un frammento di DNA 343 bp di P. gingivalis 16S rRNA mediante PCR utilizzando DNA genomico di P. gingivalis e le seguenti primer: 5'TGC AAC TTG CCT TAC AGA GG-3 'e 5'AGIRE CGT ATC GCC CGT TAT TC-3' 10. Eseguire amplificazioni con le seguenti condizioni ciclo: 35 cicli a 95 ° C per 30 sec, 60 ° C per 30 sec e 72 ° C per 1 min 20 sec seguita da un'estensione di 5 min a 72 ° C 10,11.
- Amplificare la sonda eubatterica con un 70-bp frammento di DNA (5'-CAGGTGCTGCATGGCTGTCGTCAGCTCGTGTTGTGAAATGTTGGGTTAAG
TCCCGCAACGAGCGCAACCC-3 ') sintetizzato come oligonucleotide ed i seguenti primer: 5'-CTG CAG GTR CMT GGY-3' e 5'-GTT AGG GCG CTC GTT-3 '11. Eseguire amplificazioni con le seguenti condizioni ciclo: 40 ciclia 9 4 ° C per 30 sec, 60 ° C per 30 sec e 72 ° C per 1 min 20 s seguita da un'estensione 5 min a 72 ° C 11,12. - Precipitare i prodotti di PCR (≥500 microlitri) aggiungendo 3 M acetato di sodio (pH 5.2) e 100% etanolo (a 10: 1: 2) e incubando a -20 ° C per 2 ore.
- Centrifugare la miscela a 14.000 xg per 15 min, risospendere il pellet in 100 ml di tampone TE, e misurare la concentrazione di DNA dalla densità ottica a 260 nm.
- DIG-etichettatura utilizzando un sistema di etichettatura e il rilevamento kit DNA DIG
- Mescolare 3 ug della sonda con acqua ad un volume finale di 15 microlitri.
- Denaturare il DNA a 95 ° C per 10 minuti e raffreddare rapidamente sul ghiaccio.
- Aggiungere 2 ml di 10x hexanucleotide mix, 2 ml di dNTP etichettatura mix, e 1 ml di Klenow enzima per 15 ml di DNA denaturato.
- Mescolare e incubare a 37 ° C per 24 ore a 48 ore e interrompere la reazione mediante riscaldamento a 65 ° C.
- Controllare la sensibilità della sonda DIG marcato utilizzando una etichettatura e Detection Kit DNA DIG
- Caricare manualmente 1 ml di diluizioni seriali della sonda di controllo positivo previste nel kit e 1 ng di sonda DIG marcato sulla membrana di nylon e asciutto per 5 minuti a temperatura ambiente.
- Brevemente immergere la membrana in tampone 2x SSC (0,3 M NaCl, 30 mM citrato di sodio, pH 7,0) e incubare la membrana a 80 ° C per 1 ora per immobilizzare il DNA.
- Brevemente immergere la membrana in tampone acido maleico (MAB, 0,1 M di acido maleico, 0,15 M di NaCl, pH 7,5) e incubare con DIG anti-fosfatasi alcalina (AP) anticorpale coniugata diluito (1: 5.000) in 6 ml di soluzione di blocco, previsto nel kit a temperatura ambiente per 30 min.
- Lavare la membrana con MABT (MAB contenente 1% Tween 20) e applicare 40 microlitri della soluzione NBT / BCIP mescolato con 2 ml di tampone di rilevamento (0,1 M Tris-HCl, 0,1 M NaCl, 0,05 M MgCl 2, pH 9,5) sulla Membrana per 1 min. Se la sensibilità della sonda marcata ènon soddisfacente, ripetere i punti da 1.1.3 a 1.3.4.
- Misurare la concentrazione di DNA della sonda marcata (OD260) e regolare la concentrazione di 100 ng ml -1.
- Controllare la specificità della sonda marcata con DIG
- Denaturare campioni di DNA genomico (25 ng 3 microlitri -1 per ogni campione) da varie specie batteriche, tra il batterio bersaglio dalla sonda specifica, a 95 ° C per 10 minuti e raffreddare rapidamente sul ghiaccio.
- Caricare manualmente il DNA denaturato sulla membrana di nylon e asciugare per 20 minuti a RT.
- Brevemente immergere la membrana in tampone 2x SSC e incubare la membrana a 80 ° C per 2 ore per immobilizzare il DNA.
- Bloccare la membrana con una soluzione di saturazione a temperatura ambiente per 1 ora.
- Diluire la sonda 1 ng ml -1 in tampone di ibridazione, denaturare le sonde diluito, e applicare sulla membrana.
- Incubare la membrana a 45 ° C per 1 ora.
- Lavare la membrana a trevolte con tampone 2x SSC.
- Brevemente immergere la membrana in MAB.
- Incubare con anticorpo anti-DIG AP-coniugato diluito (1: 2.000) in 6 ml di soluzione bloccante a temperatura ambiente per 1 ora.
- Lavare la membrana con MABT e applicare 40 microlitri della soluzione NBT / BCIP mescolato con 2 ml di tampone di rilevamento.
- Quando la sonda non raggiunge la specificità previsto, aumentare la temperatura di ibridazione fino si osserva la specificità.
2. In Situ Ibridazione
Nota: per evitare l'asciugatura dei campioni e dei reagenti, eseguire tutte le incubazioni in una camera umidificata foderato con carta da cucina bagnato.
- De-paraffinization e reidratazione di sezioni di tessuto
- Preparare 4 micron sezioni di tessuto di spessore dai blocchi inclusi in paraffina. Formalina, paraformaldeide e fissativo a base di zinco sono compatibili con questo protocollo.
- Preparare tutti i reagenti con acqua DEPC-trattata.
- Diapositive calore in un forno a secco a 60 ° C per 30 min e incubare vetrini a temperatura ambiente per 30 min.
- Immergere i vetrini in 100% xilene per 5 minuti tre volte; utilizzare xilene fresco ogni volta.
Nota: Effettuare questa procedura de-ceretta in una cappa. - Immergere i vetrini in etanolo al 100% per 5 minuti due volte, con l'etanolo nuovo ogni volta, e reidratare i campioni in serie il 90%, 80% e le soluzioni di etanolo 70% per 5 minuti ciascuno.
- Immergere i vetrini in acqua DEPC trattati per 1 min e lavare i vetrini in DEPC trattati con PBS (pH 7.4) per 6 min.
- Pretrattamenti di sezioni di tessuto
- Immergere i vetrini in acido cloridrico 0,1 N per 20 minuti e lavare i vetrini in acqua DEPC trattati per 30 sec.
- Disegnare una barriera idrofobica campioni utilizzando una penna cera circostante.
Nota: Le dimensioni della barriera idrofoba dipende dalle dimensioni dei campioni. Pertanto, i volumi dei reagenti usati nei seguenti passaggi variano a seconda delle dimensioni dei provini ma devono riempire il hybarriera drophobic (50 ml per i piccoli campioni e 400 ml per i grandi campioni). - Trattare campioni con 50 a 400 ml di proteinasi K (da 1 a 10 mcg ml -1 in DEPC trattati con PBS) in un forno a secco a 37 ° C per 30 minuti e lavare i vetrini in DEPC trattati 1x PBS per 1 min.
- Immergere i vetrini in paraformaldeide al 4% in DEPC trattati con PBS per 10 minuti, poi lavare i vetrini in DEPC trattati con PBS per 1 min.
- Immergere i vetrini in 0,1 M trietanolammina-HCl (pH 8.0) contenente 0,5% di anidride acetica per 20 minuti e lavare i vetrini in acqua DEPC trattati per 30 sec.
- Ibridazione con sonda e lavaggio
- Immergere i vetrini in tampone SSC 2x per 20 min.
- Diluire la sonda a 1 ng ml -1 a tampone di ibridazione (4x SSC, il 50% [vol / vol] formammide, la soluzione di 1x Denhardt, il 10% [peso / volume] destrano solfato, 0.1% [peso / volume] solfato di sodio dodecil, 0,4 mg ml -1 sperma di salmone DNA). Per i controlli negativi, mescolare la sonda marcata conun 10-fold quantità in eccesso di sonda non marcata.
- Denaturare le sonde diluito e applicare 50 a 400 ml su sezioni di tessuto. Posizionare un vetrino sul vetrino e sigillare con smalto.
- Diapositive calore in macchina PCR a 90 ° C per 10 minuti e incubare vetrini in una camera umidificata O / N a 45 ° C o la temperatura ottimale determinato al punto 1.4.11.
- Raffreddare i vetrini a 4 ° C per 30 min, rimuovere vetrini ed immergere i vetrini in tampone SSC seriale come seguenti: 4x SSC per 10 minuti, pre-riscaldato (45 ° C) 2x SSC per 20 minuti, 2x SSC per 10 min, e 0.2x SSC per 10 min.
- Detection con anticorpo anti-DIG AP-coniugato
- Lavare i vetrini in MABT per 5 min.
- Blocco con 50 a 400 ml di soluzione 1% Tween 20 blocco per 20 min.
- Aggiungere 50 a 400 ml di anticorpo anti-DIG-AP diluito in soluzione bloccante (1: 1.000) anld incubare a 37 ° C per 90 min.
- Lavare i vetrini in MABT per 10 min.
- immergere i vetrini in tampone rilevamento contenente 1% Tween 20 per 5 min.
- Trattare con 50 a 400 μ di 1 levamisolo mM (in tampone rilevamento contenente 1% Tween 20) per 5 minuti per inattivare fosfatasi alcalina endogena.
- Distribuire 50 a 400 microlitri della soluzione premiscelata NBT / BCIP (diluito in tampone di rilevamento a 1: 100) su ciascun campione e incubare vetrini in una camera umidificata a temperatura ambiente per 2 o 3 ore finché il segnale sviluppato è soddisfacente.
- Risciacquare i vetrini con acqua deionizzata per fermare la visualizzazione e applicare 50 a 400 ml di 0,05% [wt / vol] verde metile come una macchia contatore a 37 ° C per 10 min.
- Risciacquare i vetrini con acqua deionizzata, disidratare in etanolo al 100%, ed immergere in xilene.
- Applicare 80 ml di soluzione di montaggio su ogni vetrino e coprire con coprioggetto.
Risultati
La figura 1 mostra puntino assorbente di sonde DIG-marcate rispetto con la sonda di controllo positivo fornito nel kit per determinare la loro sensibilità. Il 343 bp P. Sonda gingivalis -specific è 25 volte più sensibile rispetto alla sonda eubatterica 70 bp. La figura 2 mostra l'ibridazione in situ dei tessuti gengivali ottenuti da pazienti con parodontite cronica per il rilevamento di P. gingivalis e eubatteri. I segnali batteriche, mostrati in viola, sono stati rilevati nel epiteli, la lamina propria, e biofilm situato all'esterno del tessuto. Tuttavia, la distribuzione di P. gingivalis e eubacteria all'interno del tessuto gengivale era diverso. Ad alto ingrandimento (1,000X), varie forme (cocco, canna, fusiforme, e la forma pelosa) di batteri erano visibili utilizzando la sonda eubatterica, mentre i batteri a forma solo asta sono stati rilevati dal P. Sonda gingivalis -specific. Le forme disperse di segnali batterichesono stati anche osservati.
Hajishengallis et al. dimostrato il ruolo fondamentale dei batteri commensali nello sviluppo di P. gingivalis indotta parodontite sperimentale nei topi 14. L'applicazione di destrano solfato di sodio (DSS), una giunzione a tenuta (TJ) -disrupting chimica, sulla gengiva mucosa periodontite indotta e la perdita di osso alveolare nei topi 13. La sonda ha rilevato con successo eubatterica batteri all'interno dei tessuti gengivali del gruppo DSS che non sono stati rilevati dal P. gingivalis Sonda -specific (Figura 3).
La sonda eubatterica è utile per lo studio del potenziale coinvolgimento di batteri in altre malattie. Dal momento che l'Agenzia Internazionale per la Ricerca sul Cancro designato Helicobacter pylori come grado I agente cancerogeno per cancro gastrico 15, potenziale coinvolgimento di batteri nello sviluppo di altri tumori è stato ampiamente studiato. Quando seZIONI di biopsie polmonari con diagnosi di carcinoma a cellule squamose in situ erano ibridato con la sonda eubatterica, i batteri sono stati rilevati sia all'interno delle cellule tumorali e tra le stromali circostante / infiltrante cellule (Figura 4). L'ibridazione in situ utilizzando la sonda eubatterica è stata applicata anche a esplorare la presenza di batteri nelle lesioni di lichen planus orale, una malattia immunitaria infiammatoria con eziologia sconosciuta. È interessante notare, segnali batterici sono stati rilevati non solo all'interno epiteli ma anche nella lamina propria in cui è stata osservata a fascia infiltrazione. In alto ingrandimento, i segnali di batteri sono stati rilevati nei nuclei delle molte cellule (Figura 5).

Figura 1. Sensibilità prova delle sonde DIG-etichettati. Sonde personalizzate marcato serialmente diluiti e un contr positivaSonda ol fornito nel kit sono stati dot cancellato con l'anticorpo anti-DIG-AP-coniugato. Dopo aver aggiunto il substrato, il blot con l'P. Sonda gingivalis -specific stato sviluppato per 30 secondi, mentre la macchia con la sonda eubatterica stato sviluppato per 1 min. Clicca qui per vedere una versione più grande di questa figura.
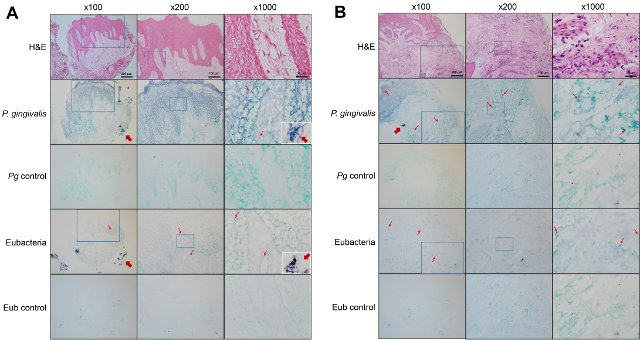
Figura 2. Rilevamento di batteri all'interno dei tessuti gengivali ottenuti da pazienti con periodontite cronica. Sezioni di biopsie gengivali dal sito sani (A) e il sito malati (B) di un paziente con parodontite sono stati sottoposti a ematossilina-eosina (H & E) macchie o ibridazione in situ sia con la P. gingivalis sonda -specific o della sonda eubatterica. Negative controlli sono stati ibridati con la sonda mescolato con 10 volte la sonda senza etichetta in eccesso. L'area ingrandita è indicata con una scatola quadrata. Segnali positivi rappresentativi, mostrati in viola, sono contrassegnati con frecce sottili. Frecce spesse indicano placca biofilm di fuori del tessuto. Il biofilm targa esaminato a 1,000X ingrandimento sono mostrati in inserti. Clicca qui per vedere una versione più grande di questa figura.
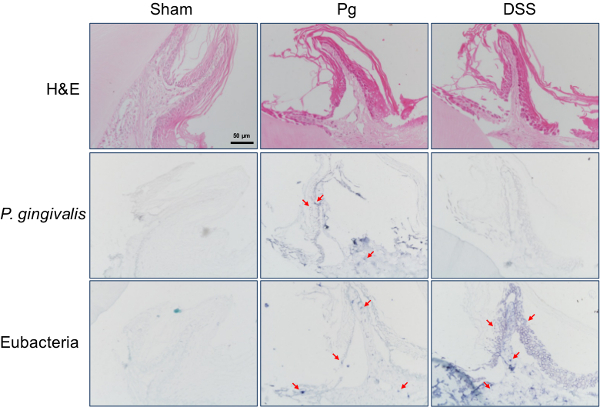
Figura 3. Rilevamento di batteri commensali orali all'interno del tessuto gengivale del mouse. Periodontite sperimentale è stata indotta in topi BALB / c per inoculazione orale di P. gingivalis (Pg) o l'applicazione del DSS sulla mucosa gengivale. Sezioni gengivali di topi sono stati sottoposti a colorazione H & E o ibridazione in situ con i tag HTML < em> P. gingivalis sonda -specific o della sonda eubatterica. Segnali positivi rappresentativi, mostrati in viola, sono contrassegnate con le frecce. Originale è l'ingrandimento x400. Cliccare qui per vedere una versione più grande di questa figura.
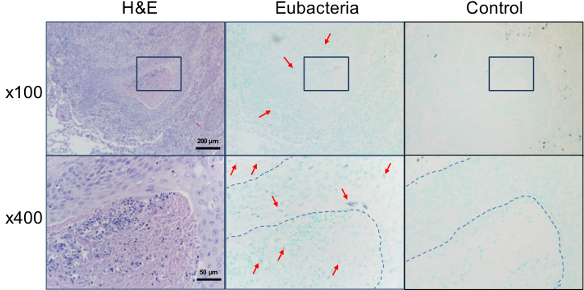
Figura 4. Individuazione dei batteri all'interno della lesione cancro. Le sezioni di biopsie polmonari di pazienti con diagnosi di carcinoma a cellule squamose sono state colorate con H & E o in situ ibridazione sia con la sonda eubatterica universale o la sonda di controllo negativo. L'area ingrandita è indicata con una scatola quadrata. Segnali positivi rappresentativi, mostrati in viola, sono contrassegnate con le frecce. Bordo delle cellule tumorali sono indicati con linee tratteggiate.Arget = "_ blank"> Clicca qui per vedere una versione più grande di questa figura.
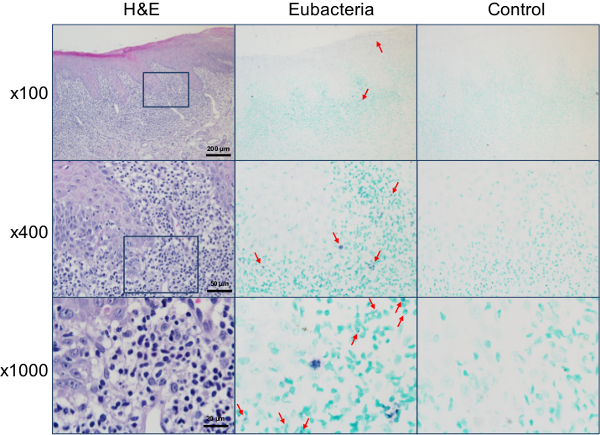
Figura 5. Individuazione dei batteri all'interno delle lesioni del lichen planus orale. Le sezioni di biopsia di mucosa buccale diagnosticati come lichen planus orale sono state colorate con H & E o in situ ibridazione sia con la sonda eubatterica universale o la sonda di controllo negativo. Le aree ingrandite sono indicate con una scatola quadrata. Segnali positivi rappresentativi, mostrati in viola, sono contrassegnate con le frecce. Cliccate qui per vedere una versione più grande di questa figura.
Discussione
Qui un protocollo per localizzare i batteri all'interno dei tessuti inclusi in paraffina utilizzando una sonda di DNA DIG marcato è stato descritto. La sonda obiettivi dei DNA o RNA molecole batterica gene 16S rRNA, e le sonde 16S rRNA-mirate può essere progettato come entrambe le specie-specifico o universale. L'ibridazione specifica del P. Sonda gingivalis -specific a P. gingivalis ma non ad altri batteri orali è stato mostrato in precedenza 10. Al contrario, la sonda eubatterica ibridato a tutti batterica DNA genomico testato (17 specie diverse), sebbene l'efficienza di ibridazione variata 12. Il numero di T-nucleotidi all'interno della sonda determina l'efficienza di DIG-etichettatura. Produzione di una sonda DIG marcato con l'alta sensibilità è la fase più critica per l'efficace attuazione del protocollo descritto. Trattamento dei campioni con acido cloridrico migliora il rapporto segnale-rumore attraverso estrazione di proteine e hydrolysi parzialis di sequenze bersaglio. Un aumento della concentrazione di proteinasi K aumenta bersaglio accessibilità delle sonde ma riduce conservazione dei tessuti. Pertanto, si raccomanda di testare diverse concentrazioni di proteinasi K, a seconda del tipo di tessuto. Il passo acetilazione diminuisce sfondo legame della sonda carica negativa alle proteine del tessuto acetilando gruppi amminici caricati positivamente. Dopo una serie di queste fasi di preparazione, le sezioni di tessuto strappare facilmente, quindi, devono essere maneggiati con cautela. Sfondo è minimo rispetto a FISH. Tuttavia, i segnali provenienti da tessuti con alta attività di fosfatasi alcalina endogena come ossa e ghiandole muco dovrebbero essere interpretate in modo critico.
Sonde di DNA PCR amplificati sono facili da produrre e stabile rispetto alle sonde di RNA. Individuazione immunoistochimica di sonde ibridate consente l'identificazione di strutture istologiche attraverso il confronto con le sezioni H & E macchiato: gli epiteli,del tessuto connettivo, infiltrati infiammatori, e vasi sanguigni sono ben distinti. Una delle limitazioni del protocollo descritto è l'incapacità di applicare più sonde contemporaneamente. Inoltre, l'esclusione dei segnali da contaminanti batterici sulla superficie delle sezioni è limitata.
Questo protocollo può essere adattato per rilevare altri trascritti batteriche all'interno delle sezioni di tessuto 16. Inoltre, questo protocollo può essere esteso a doppia colorazione immunoistochimica con rilevamento di un marcatore cellula ospite.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato sostenuto da una sovvenzione (2013R1A1A3005669) dal National Research Foundation di Corea e una borsa di studio (HI13C0016) del coreano Salute Technology R & S del progetto, Ministero della Salute e del Welfare.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetic anhydride | Sigma | 6404 | |
| 50% Dextran sulfate solution | Millipore | S4030 | |
| 50X Denhardt’s solution | Sigma | D2532 | |
| DEPC | Sigma | P159220 | |
| DIG DNA labeling and detection kit | Roche | 11 093 657 910 | |
| Formamide | Sigma | F9037 | |
| ImmEdge™ Pen | Dako | H-400 | |
| Levamisole | Vector | SP-5000 | |
| Magnesium chloride | Sigma | 246964 | |
| Maleic acid | Sigma | M0375 | |
| Methyl green | Sigma | M6776 | |
| Paraformaldehyde | Sigma | P1648 | |
| Permount | Fisher | SP15-500 | |
| Salmon sperm DNA solution | Invitrogen | #15632-011 | |
| Sodium chloride | Sigma | S9625 | |
| Sodium citrate | Duksan | D1420 | |
| Sodium dodecyl sulfate | Amresco | 227 | |
| Triethanolamine-HCl | Sigma | 90279 | |
| Tris-HCl | Research organics | 3098T |
Riferimenti
- Takarada, H., Cattoni, M., Sugimoto, A., Rose, G. G. Ultrastructural studies of human gingiva. II. The lower part of the pocket epithelium in chronic periodontitis. J. Periodontol. 45 (3), 155-169 (1974).
- Allenspach-Petrzilka, G. E., Guggenheim, B. Bacterial invasion of the periodontium; an important factor in the pathogenesis of periodontitis. J. Clin. Periodontol. 10 (6), 609-617 (1983).
- Saglie, F. R., et al. The presence of bacteria in the oral epithelium in periodontal disease. II. Immunohistochemical identification of bacteria. J. Periodontol. 57 (8), 492-500 (1986).
- Christersson, L. A., Albini, B., Zambon, J. J., Wikesjö, U. M., Genco, R. J. Tissue localization of Actinobacillus actinomycetemcomitans. in human periodontitis. I. Light, immunofluorescence and electron microscopic studies. J. Periodontol. 58 (8), 529-539 (1987).
- Saglie, F. R., Pertuiset, J., Rezende, M. T., Nestor, M., Marfany, A., Cheng, J. In situ. correlative immuno-identification of mononuclear infiltrates and invasive bacteria in diseased gingiva. J. Periodontol. 59 (10), 688-696 (1988).
- Rautemaa, R., et al. Intracellular localization of Porphyromonas gingivalis. thiol proteinase in periodontal tissues of chronic periodontitis patients. Oral Dis. 10 (5), 298-305 (2004).
- Marttila, E., et al. Intracellular localization of Treponema denticola. chymotrypsin-like proteinase in chronic periodontitis. J. Oral Microbiol. 6, (2014).
- Colombo, A. V., da Silva, C. M., Haffajee, A., Colombo, A. P. Identification of intracellular oral species within human crevicular epithelial cells from subjects with chronic periodontitis by fluorescence in situ. hybridization. J. Periodontal Res. 42 (3), 236-243 (2007).
- Abrams, K., Caton, J., Polson, A. Histologic comparisons of interproximal gingival tissues related to the presence or absence of bleeding. J. Periodontol. 55 (11), 629-632 (1984).
- Kim, Y. C., et al. Presence of Porphyromonas gingivalis. and plasma cell dominance in gingival tissues with periodontitis. Oral Dis. 16 (4), 375-381 (2010).
- Choi, Y. S., et al. Porphyromonas gingivalis. and dextran sodium sulfate induce periodontitis through the disruption of physical barriers. Eur. J. Inflammation. 10, 419-431 (2013).
- Choi, Y. S., Kim, Y. C., Ji, S., Choi, Y. Increased bacterial invasion and differential expression of tight junction proteins, growth factors, and growth factor receptors in periodontal lesions. J. Periodontol. 85 (8), e313-e322 (2014).
- Baker, G. C., Smith, J. J., Cowan, D. A. Review and re-analysis of domain-specific 16S primers. J. Microbiol. Methods. 55 (3), 541-555 (2003).
- Hajishengallis, G., et al. A Low-abundance biofilm species orchestrates inflammatory periodontal disease through the commensal microbiota and the complement. Cell Host Microbe. 10 (5), 497-506 (2011).
- Axon, A. Gastric cancer and Helicobacter pylori. Aliment. Pharmacol. Ther. 16 (suppl. 4), 83-88 (2002).
- Fenhalls, G., et al. In situ. detection of Mycobacterium tuberculosis transcripts in human lung granulomas reveals differential gene expression in necrotic lesions. Infect. Immun. 70 (11), 6330-6338 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon