Method Article
La detección in situ de las bacterias dentro de los tejidos embebidos en parafina utilizando un ADN sonda digoxina marcada focalización 16S rRNA
En este artículo
Resumen
Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled 16S rRNA-targeting DNA probes has been described. This protocol can be applied to study the role of bacteria in various diseases such as periodontitis, cancers, and inflammatory immune diseases.
Resumen
The presence of bacteria within the pocket epithelium and underlying connective tissue in gingival biopsies from patients with periodontitis has been reported using various methods, including electron microscopy, immunohistochemistry or immunofluorescence using bacteria-specific antibodies, and fluorescent in situ hybridization (FISH) using a fluorescence-labeled oligonucleotide probe. Nevertheless, these methods are not widely used due to technical limitation or difficulties. Here a method to localize bacteria within paraffin-embedded tissues using DIG-labeled DNA probes has been introduced. The paraffin-embedded tissues are the most common form of biopsy tissues available from pathology banks. Bacteria can be detected either in a species-specific or universal manner. Bacterial signals are detected as either discrete forms (coccus, rod, fusiform, and hairy form) of bacteria or dispersed forms. The technique allows other histological information to be obtained: the epithelia, connective tissue, inflammatory infiltrates, and blood vessels are well distinguished. This method can be used to study the role of bacteria in various diseases, such as periodontitis, cancers, and inflammatory immune diseases.
Introducción
Las bacterias desempeñan un papel en la etiología de diversas enfermedades orales tales como periodontitis, pulpitis, pericoronitis, celulitis, y osteomielitis. Con el fin de comprender el papel de las bacterias en la patogénesis de la enfermedad y para controlar el efecto de los tratamientos, la localización de las bacterias dentro del tejido es importante. La presencia de bacterias en el tejido gingival de pacientes con periodontitis se ha demostrado usando varios métodos, incluyendo microscopía electrónica de 1,2, inmunohistoquímica e inmunofluorescencia utilizando anticuerpos específicos de bacterias 3-7, y la hibridación fluorescente in situ (FISH) usando un 8 por fluorescencia sonda de oligonucleótido marcada orientación 16S rRNA. Sin embargo, estos métodos no son ampliamente utilizados debido a la limitación técnica o dificultades. En comparación con anticuerpos, sondas dirigidas a 16S rRNA son fáciles de producir y lograr especificidad de especie. FISH ha demostrado ser una excelente herramienta para la visualización de bacteria en sus entornos naturales como el biofilm de la placa. Sin embargo, la aplicación de FISH a las muestras de tejido es limitado debido a la autofluorescencia de los diversos componentes del tejido. Por ejemplo, el fuerte autofluorescencia de las células rojas de la sangre a menudo dificulta la aplicación de la tecnología de fluorescencia para los tejidos inflamados cuando implican sangrado 9.
Con el fin de localizar las bacterias dentro de los tejidos gingivales inflamados, por lo tanto, un método de hibridación in situ utilizando un digoxigenina (DIG) sonda de ADN marcada con se ha desarrollado y aplicado con éxito 10,11. Aquí un protocolo detallado para la localización de las bacterias dentro de los tejidos incluidos en parafina utilizando P. gingivalis sondas específicos de eubacterial y universales se ha descrito. Se centra sobre todo en la estandarización del método de manera que resultados similares se pueden reproducir en otros laboratorios. Este protocolo permite la localización de las bacterias dentro de su contexto histológico y el results son altamente reproducible. El protocolo descrito se puede utilizar para localizar las bacterias ya sea de una manera específica de la especie o universal en varios tejidos. La sonda universal es especialmente útil para detectar bacterias en enfermedades polimicrobianas y para estudiar un posible papel de las bacterias en enfermedades en las que no se conoce el papel de las bacterias específicas.
Protocolo
1. Preparación de la sonda
- La amplificación por PCR de la sonda
- Para el P. sonda gingivalis específico de, amplificar un fragmento de ADN de 343 pb de P. gingivalis 16S rRNA mediante PCR utilizando el ADN genómico de P. gingivalis y los siguientes cebadores: 5'-TGC AAC TTG CCT TAC AGA GG-3 'y 5' ACTUAR CGT ATC GCC CGT TAT TC-3 '10. Realizar amplificaciones con las siguientes condiciones de ciclo: 35 ciclos a 95 ° C durante 30 seg, 60 ° C durante 30 seg, y 72 ° C durante 1 min 20 seg seguido por una extensión de 5 min a 72 ° C 10,11.
- Amplificar la eubacteriana usando un 70-bp fragmento de ADN (5'-CAGGTGCTGCATGGCTGTCGTCAGCTCGTGTTGTGAAATGTTGGGTTAAG
TCCCGCAACGAGCGCAACCC-3 ') sintetizado como un oligonucleótido y los siguientes cebadores: 5'-CTG CAG GTR CMT GGY-3' y 5'-AGG GTT GCG CTC GTT-3 '11. Realizar amplificaciones con las siguientes condiciones de ciclo: 40 ciclos9 a 4 ° C durante 30 seg, 60 ° C durante 30 seg y 72 ° C durante 1 min 20 s, seguido de una extensión de 5 min a 72 ° C 11,12. - Precipitar los productos de PCR (≥500 mu l) mediante la adición de acetato de sodio 3 M (pH 5,2) y etanol 100% (a un 10: 1: 2 ratio) e incubando a -20 ° C durante 2 hr.
- Centrifugar la mezcla a 14.000 xg durante 15 min, resuspender el precipitado en 100 l de tampón TE, y medir la concentración de ADN mediante densidad óptica a 260 nm.
- DIG etiquetado utilizando un kit de etiquetado y detección de ADN DIG
- Mezclar 3 g de la sonda con agua hasta un volumen final de 15 l.
- Desnaturalizar el ADN a 95 ° C durante 10 minutos y enfriar rápidamente en hielo.
- Añadir 2 l de 10x mezcla hexanucleotide, 2 l de mezcla de dNTP-etiquetado, y 1 l de enzima Klenow a 15 l de la DNA desnaturalizado.
- Mezclar e incubar a 37 ° C durante 24 horas a 48 horas y detener la reacción por calentamiento a 65 ° C.
- Compruebe la sensibilidad de la sonda DIG-etiquetados utilizando un kit de etiquetado y detección de ADN DIG
- Cargar manualmente 1 l de diluciones seriadas de la sonda de control positivo en el kit y 1 ng de la sonda marcada con DIG en la membrana de nylon y seco durante 5 minutos a temperatura ambiente.
- Brevemente empapar la membrana en tampón 2x SSC (NaCl 0,3 M, citrato de sodio 30 mM, pH 7,0) y se incuba la membrana a 80 ° C durante 1 hora para inmovilizar el ADN.
- Brevemente empapar la membrana en tampón de ácido maleico (MAB, 0,1 M de ácido maleico, NaCl 0,15 M, pH 7,5) y se incuba con anti-DIG fosfatasa alcalina (AP) conjugada con anticuerpo diluido (1: 5000) en 6 ml de solución de bloqueo proporcionado en el kit a TA durante 30 min.
- Se lava la membrana con MABT (MAB que contiene 1% de Tween 20) y aplicar 40 l de la solución de NBT / BCIP mezclado con 2 ml de tampón de detección (0,1 M Tris-HCl, NaCl 0,1 M, 0,05 M MgCl 2, pH 9,5) en el membrana durante 1 min. Si la sensibilidad de la sonda marcada esno satisfactoria, repita los pasos de 1.1.3 a 1.3.4.
- Medir la concentración de ADN de la sonda marcada (OD260) y ajustar la concentración a 100 ng l -1.
- Compruebe la especificidad de la sonda marcada con DIG
- Desnaturalizar muestras de ADN genómico (25 ng de 3 l -1 por cada muestra) a partir de diversas especies bacterianas, incluyendo la bacteria blanco de la sonda específica, a 95 ° C durante 10 min y enfriar rápidamente en hielo.
- Cargar manualmente el ADN desnaturalizado en la membrana de nylon y seco durante 20 min a TA.
- Brevemente empapar la membrana en tampón 2x SSC y se incuba la membrana a 80 ° C durante 2 horas para inmovilizar el ADN.
- Bloquear la membrana con una solución de bloqueo a TA durante 1 hr.
- Diluir la sonda a 1 ng l -1 en tampón de hibridación, desnaturalizar las sondas diluidas, y aplicar sobre la membrana.
- Incubar la membrana a 45 ° C durante 1 hr.
- Lavar la membrana de tresveces con tampón SSC 2x.
- Brevemente remojo la membrana en el MAB.
- Incubar con el anticuerpo anti-DIG AP-conjugado diluido (1: 2000) en 6 ml de solución de bloqueo a TA durante 1 hr.
- Se lava la membrana con MABT y aplicar 40 l de la solución de NBT / BCIP mezclado con 2 ml de tampón de detección.
- Cuando la sonda no logra la especificidad esperada, aumentar la temperatura de hibridación hasta que se observa la especificidad.
2. Hibridación in situ
Nota: Para evitar el secado de las muestras y reactivos, realice todas las incubaciones en una cámara húmeda llena de toallas de papel húmedas.
- De-paraffinization y la rehidratación de secciones de tejido
- Preparar 4 micras secciones de tejidos gruesos de los bloques incluidos en parafina. Formalina, paraformaldehído y fijador a base de zinc son compatibles con este protocolo.
- Preparar todos los reactivos con agua tratada con DEPC.
- Diapositivas de calor en un horno seco a 60 ° C durante 30 min y Incubar los portaobjetos a TA durante 30 min.
- Sumergir los portaobjetos en 100% xileno durante 5 minutos tres veces; utilizar xileno nuevo cada vez.
Nota: Realice este procedimiento de-encerado en una campana de extracción química. - Sumergir los portaobjetos en etanol al 100% durante 5 min dos veces, el uso de etanol fresco cada vez, y rehidratar especímenes en serie 90%, 80% y 70% de soluciones de etanol durante 5 min cada uno.
- Sumergir los portaobjetos en agua tratada con DEPC por 1 min y lavar los portaobjetos en PBS tratada con DEPC (pH 7,4) durante 6 min.
- Pretratamientos de secciones de tejido
- Sumergir los portaobjetos en ácido clorhídrico 0,1 N durante 20 minutos y lavar los portaobjetos en agua tratada con DEPC durante 30 segundos.
- Dibuja una barrera hidrofóbica rodea especímenes utilizando un lápiz de cera.
Nota: El tamaño de la barrera hidrofóbica depende del tamaño de las muestras. Por lo tanto, los volúmenes de reactivos utilizados en los siguientes pasos varían dependiendo del tamaño de las muestras, pero tienen que llenar el HYbarrera drophobic (50 l para los pequeños especímenes y 400 l de grandes ejemplares). - Tratar especímenes con 50 a 400 l de proteinasa K (1 a 10 mg ml -1 en PBS tratada con DEPC) en un horno seco a 37 ° C durante 30 min y lavar los portaobjetos en PBS tratada con DEPC 1x durante 1 min.
- Remojar los portaobjetos en 4% de paraformaldehído en PBS tratada con DEPC durante 10 min, a continuación, lavar los portaobjetos en PBS tratada con DEPC por 1 min.
- Remojar las diapositivas en 0,1 M de trietanolamina-HCl (pH 8,0) que contenía anhídrido acético 0,5% durante 20 min y lavar los portaobjetos en agua tratada con DEPC para 30 seg.
- La hibridación con la sonda y el lavado
- Sumergir los portaobjetos en tampón SSC 2x durante 20 min.
- Diluir la sonda a 1 ng l -1 en tampón de hibridación (4x SSC, 50% [vol / vol] formamida, 1X solución de Denhardt, 10% [p / v] sulfato de dextrano, 0,1% [peso / vol] de dodecil sulfato de sodio, 0,4 mg ml -1 de ADN de esperma de salmón). Para los controles negativos, mezclar la sonda marcada conuna cantidad en exceso de 10 veces de sonda no marcada.
- Desnaturalizar las sondas diluidas y aplicar 50 a 400 l en secciones de tejido. Coloque una hoja de la cubierta de la diapositiva y sellar con esmalte de uñas.
- Diapositivas de calor en la máquina PCR a 90 ° C durante 10 minutos e incubar los portaobjetos en una cámara húmeda O / N a 45 ° C o temperatura óptima determinada en el paso 1.4.11.
- Enfriar las diapositivas a 4 ° C durante 30 minutos, retire cubreobjetos, y sumergir los portaobjetos en un tampón SSC serie como followings: 4x SSC durante 10 minutos, precalentado (45 ° C) 2x SSC durante 20 minutos, 2x SSC durante 10 min, y 0,2x SSC durante 10 min.
- Detección con anticuerpo anti-DIG conjugado AP-
- Lave las diapositivas en MABT durante 5 min.
- Bloque con 50 a 400 l de solución de bloqueo con 1% de Tween 20 durante 20 min.
- Añadir 50 a 400 l de anticuerpo anti-DIG-AP diluidos en solución de bloqueo (1: 1000) anld incubar a 37 ° C durante 90 min.
- Lave las diapositivas en MABT durante 10 min.
- Sumergir los portaobjetos en tampón de detección que contiene 1% de Tween 20 durante 5 min.
- Tratar con 50 a 400 μ de levamisol 1 mM (en tampón de detección que contiene 1% de Tween 20) durante 5 min para inactivar la fosfatasa alcalina endógena.
- Distribuir 50 a 400 l de la solución premezclada NBT / BCIP (diluido en tampón de detección a 1: 100) en cada muestra e incubar los portaobjetos en una cámara humidificada a temperatura ambiente durante 2 a 3 horas hasta que la señal desarrollado es satisfactoria.
- Enjuagar portaobjetos con agua desionizada para detener la visualización y aplicar 50 a 400 l de 0,05% [peso / vol] verde de metilo como una mancha contador a 37 ° C durante 10 min.
- Enjuague diapositivas con agua desionizada, se deshidratan en etanol al 100%, y sumergirse en xileno.
- Aplique 80 l de solución de montaje en cada diapositiva y cubrir con cubreobjetos.
Resultados
La Figura 1 muestra dot blotting de sondas marcadas con DIG en comparación con la sonda de control positivo proporcionado en el kit para determinar su sensibilidad. El 343 pb P. sonda específico de gingivalis es 25 veces más sensible que la sonda eubacterial 70 pb. La Figura 2 muestra la hibridación in situ de los tejidos gingivales obtenidas de pacientes con periodontitis crónica para la detección de P. gingivalis y eubacterias. Las señales bacterianas, que se muestran en violeta, se detectaron dentro de los epitelios, la lámina propia, y biofilm situado fuera del tejido. Sin embargo, la distribución de P. gingivalis y eubacterias dentro del tejido gingival era diferente. A mayor aumento (1.000X), diversas formas (coccus, barra, fusiforme, y la forma peluda) de bacterias eran visibles usando la sonda eubacterial, mientras que las bacterias en forma de varilla se detectaron solamente por el P. sonda gingivalis específico de. Las formas dispersas de señales bacterianastambién se observaron.
Hajishengallis et al. demostrado el papel esencial de las bacterias comensales en el desarrollo de P. gingivalis inducida periodontitis experimental en ratones 14. La aplicación de dextrano sulfato de sodio (DSS), una unión estrecha (TJ) química -disrupting, hacia gingival periodontitis inducida mucosa y la pérdida de hueso alveolar en ratones 13. La sonda eubacterial detecta con éxito las bacterias dentro de los tejidos gingivales del grupo de DSS que no fueron detectados por el P. gingivalis sonda específico de (Figura 3).
La sonda eubacterial es útil para el estudio de la posible participación de bacterias en otras enfermedades. Desde la Agencia Internacional para la Investigación sobre el Cáncer designado por Helicobacter pylori como un grado I carcinógeno para el cáncer gástrico 15, la participación potencial de las bacterias en el desarrollo de otros tipos de cáncer ha sido ampliamente estudiada. Cuando secciones de biopsias de pulmón diagnosticados con carcinoma de células escamosas in situ eran hibridado con la sonda eubacterial, las bacterias se detectaron tanto dentro de las células tumorales y entre el estroma circundante / infiltración de células (Figura 4). La hibridación in situ usando la sonda eubacterial también se aplicó a explorar la presencia de bacterias en las lesiones de liquen plano oral, una enfermedad inmune inflamatoria de etiología desconocida. Curiosamente, se detectaron señales bacterianas no sólo dentro de los epitelios, sino también en la lámina propia en donde se observó infiltración similar a una banda. Bajo la alta ampliación, se detectaron las señales bacterianas en los núcleos de muchas células (Figura 5).

Figura 1. Prueba de sensibilidad de las sondas marcadas con DIG. Sondas personalizados marcado diluidas en serie y un contr positivosonda ol proporcionado en el kit se dot borrado con el anticuerpo anti-DIG-AP-conjugado. Después de añadir el sustrato, el blot con el P. sonda gingivalis específico de se desarrolló durante 30 segundos, mientras que la mancha con el eubacteriana fue desarrollado durante 1 minuto. Por favor, haga clic aquí para ver una versión más grande de esta figura.
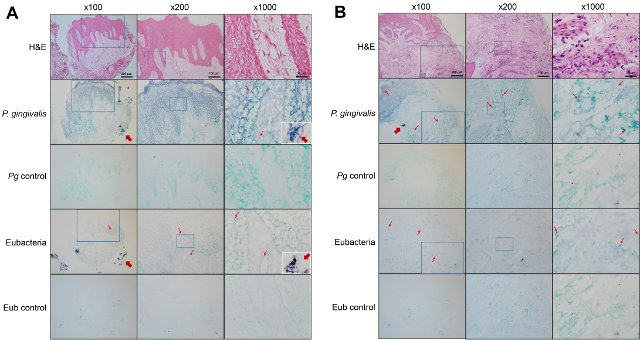
Figura 2. Detección de bacterias dentro de los tejidos gingivales obtenidas de pacientes con periodontitis crónica. Secciones de biopsias gingivales desde el sitio sano (A) y el sitio enfermo (B) de un paciente con periodontitis se sometieron a hematoxilina-eosina (H & E) tinción o hibridación in situ, ya sea con el P. gingivalis sonda específico de la sonda o eubacterial. Negativcontroles e se hibridaron con la sonda se mezcla con 10 veces el exceso de sonda no marcada. El área ampliada se indica con una caja cuadrada. Señales positivas representativas, que se muestran en violeta, están marcados con flechas finas. Flechas gruesas indican biofilm de la placa fuera del tejido. El biofilm de la placa examinado con una ampliación 1.000X se muestran en recuadros. Por favor, haga clic aquí para ver una versión más grande de esta figura.
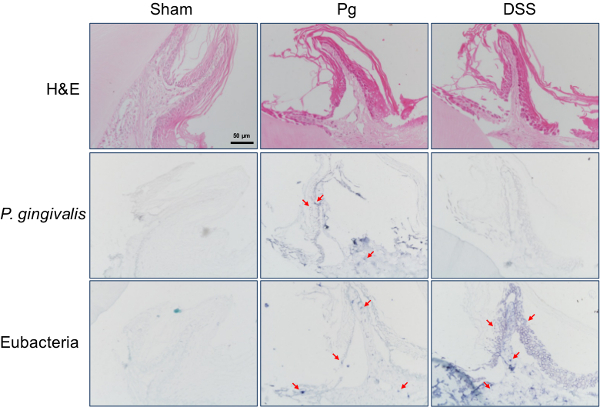
Figura 3. Detección de bacterias comensales orales dentro del tejido gingival de ratón. Periodontitis experimental fue inducida en ratones BALB / c por inoculación oral de P. gingivalis (Pg) o la aplicación de DSS en la mucosa gingival. Gingivales secciones de los ratones se sometieron a tinción H & E o hibridación in situ, ya sea con la < em> P. gingivalis sonda específico de la sonda o eubacterial. Señales positivas representativas, que se muestran en violeta, están marcados con flechas. Aumento original es x400. Por favor, haga clic aquí para ver una versión más grande de esta figura.
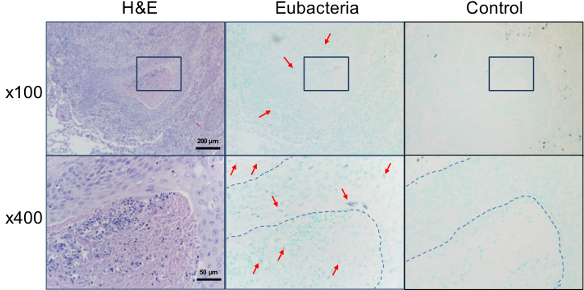
Figura 4. Detección de bacterias dentro de la lesión cáncer. Las secciones de biopsias de pulmón de pacientes con diagnóstico de carcinoma de células escamosas fueron teñidas con H & E o in situ hibridado con la sonda ya sea eubacterial universal o la sonda de control negativo. El área ampliada se indica con una caja cuadrada. Señales positivas representativas, que se muestran en violeta, están marcados con flechas. Frontera de las células tumorales se indican con líneas de puntos.arget = "_ blank"> Haga clic aquí para ver una versión más grande de esta figura.
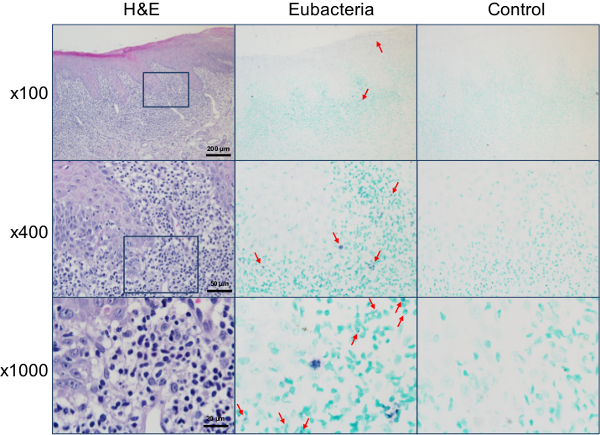
Figura 5. La detección de bacterias en las lesiones de liquen plano oral. Las secciones de la biopsia de la mucosa bucal diagnosticada como liquen plano oral fueron teñidas con H & E o in situ hibridado tanto con la sonda eubacterial universal o la sonda de control negativo. Las áreas ampliadas se indican con una caja cuadrada. Señales positivas representativas, que se muestran en violeta, están marcados con flechas. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
Aquí un protocolo para localizar las bacterias dentro de los tejidos embebidos en parafina utilizando una sonda de ADN marcada con DIG que se ha descrito. La sonda se dirige a las moléculas de ADN o ARN de origen bacteriano gen 16S rRNA, y las sondas de ARNr 16S-direccionamiento puede ser diseñado como cualquiera de las especies-específico o universal. La hibridación específica de la P. sonda específico de gingivalis a P. gingivalis pero no a otras bacterias orales se ha demostrado previamente 10. En contraste, la sonda hibridó con eubacterial todo el ADN genómico bacteriano probado (17 especies diferentes), aunque la eficiencia de hibridación varió 12. El número de T-nucleótidos dentro de la sonda determina la eficiencia de DIG-etiquetado. La producción de una sonda marcada con DIG con alta sensibilidad es el paso más crítico para la exitosa implementación del protocolo descrito. El tratamiento de las muestras con ácido clorhídrico mejora la relación señal-ruido a través de la extracción de proteínas y hydrolysi parcials de secuencias diana. Un aumento en la concentración de proteinasa K aumenta la accesibilidad objetivo de sondas, pero reduce la conservación de tejidos. Por lo tanto, se recomienda para poner a prueba varias concentraciones de proteinasa K, dependiendo de los tipos de tejido. La etapa de acetilación disminuye la unión de fondo de la sonda cargada negativamente a las proteínas del tejido por la acetilación de los grupos amino cargados positivamente. Después de una serie de estos pasos de preparación, las secciones de tejido romper con facilidad, por lo tanto, tiene que ser manejado con precaución. El fondo es mínima en comparación con FISH. Sin embargo, las señales procedentes de tejidos con alta actividad de la fosfatasa alcalina endógena, tales como huesos y glándulas mucosas deben ser interpretados de manera crítica.
Sondas de ADN amplificados por PCR son fáciles de producir y estable en comparación con sondas de ARN. La detección inmunohistoquímica de sondas hibridadas permite la identificación de las estructuras histológicas través de la comparación con las secciones de H & E manchadas: los epitelios,tejido conectivo, infiltrados inflamatorios, y los vasos sanguíneos están bien distinguidos. Una de las limitaciones del protocolo descrito es la incapacidad de aplicar múltiples sondas al mismo tiempo. Además, la exclusión de las señales procedentes de los contaminantes bacterianos en la superficie de las secciones es limitado.
Este protocolo puede ser adaptado para detectar otras transcripciones bacterianas dentro de secciones de tejido 16. Además, este protocolo se puede extender a doble tinción con detección inmunohistoquímica de un marcador de célula huésped.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por una beca (2013R1A1A3005669) de la Fundación Nacional de Investigación de Corea y una subvención (HI13C0016) de la coreana Salud Tecnología Proyecto I + D del Ministerio de Salud y Bienestar Social.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetic anhydride | Sigma | 6404 | |
| 50% Dextran sulfate solution | Millipore | S4030 | |
| 50X Denhardt’s solution | Sigma | D2532 | |
| DEPC | Sigma | P159220 | |
| DIG DNA labeling and detection kit | Roche | 11 093 657 910 | |
| Formamide | Sigma | F9037 | |
| ImmEdge™ Pen | Dako | H-400 | |
| Levamisole | Vector | SP-5000 | |
| Magnesium chloride | Sigma | 246964 | |
| Maleic acid | Sigma | M0375 | |
| Methyl green | Sigma | M6776 | |
| Paraformaldehyde | Sigma | P1648 | |
| Permount | Fisher | SP15-500 | |
| Salmon sperm DNA solution | Invitrogen | #15632-011 | |
| Sodium chloride | Sigma | S9625 | |
| Sodium citrate | Duksan | D1420 | |
| Sodium dodecyl sulfate | Amresco | 227 | |
| Triethanolamine-HCl | Sigma | 90279 | |
| Tris-HCl | Research organics | 3098T |
Referencias
- Takarada, H., Cattoni, M., Sugimoto, A., Rose, G. G. Ultrastructural studies of human gingiva. II. The lower part of the pocket epithelium in chronic periodontitis. J. Periodontol. 45 (3), 155-169 (1974).
- Allenspach-Petrzilka, G. E., Guggenheim, B. Bacterial invasion of the periodontium; an important factor in the pathogenesis of periodontitis. J. Clin. Periodontol. 10 (6), 609-617 (1983).
- Saglie, F. R., et al. The presence of bacteria in the oral epithelium in periodontal disease. II. Immunohistochemical identification of bacteria. J. Periodontol. 57 (8), 492-500 (1986).
- Christersson, L. A., Albini, B., Zambon, J. J., Wikesjö, U. M., Genco, R. J. Tissue localization of Actinobacillus actinomycetemcomitans. in human periodontitis. I. Light, immunofluorescence and electron microscopic studies. J. Periodontol. 58 (8), 529-539 (1987).
- Saglie, F. R., Pertuiset, J., Rezende, M. T., Nestor, M., Marfany, A., Cheng, J. In situ. correlative immuno-identification of mononuclear infiltrates and invasive bacteria in diseased gingiva. J. Periodontol. 59 (10), 688-696 (1988).
- Rautemaa, R., et al. Intracellular localization of Porphyromonas gingivalis. thiol proteinase in periodontal tissues of chronic periodontitis patients. Oral Dis. 10 (5), 298-305 (2004).
- Marttila, E., et al. Intracellular localization of Treponema denticola. chymotrypsin-like proteinase in chronic periodontitis. J. Oral Microbiol. 6, (2014).
- Colombo, A. V., da Silva, C. M., Haffajee, A., Colombo, A. P. Identification of intracellular oral species within human crevicular epithelial cells from subjects with chronic periodontitis by fluorescence in situ. hybridization. J. Periodontal Res. 42 (3), 236-243 (2007).
- Abrams, K., Caton, J., Polson, A. Histologic comparisons of interproximal gingival tissues related to the presence or absence of bleeding. J. Periodontol. 55 (11), 629-632 (1984).
- Kim, Y. C., et al. Presence of Porphyromonas gingivalis. and plasma cell dominance in gingival tissues with periodontitis. Oral Dis. 16 (4), 375-381 (2010).
- Choi, Y. S., et al. Porphyromonas gingivalis. and dextran sodium sulfate induce periodontitis through the disruption of physical barriers. Eur. J. Inflammation. 10, 419-431 (2013).
- Choi, Y. S., Kim, Y. C., Ji, S., Choi, Y. Increased bacterial invasion and differential expression of tight junction proteins, growth factors, and growth factor receptors in periodontal lesions. J. Periodontol. 85 (8), e313-e322 (2014).
- Baker, G. C., Smith, J. J., Cowan, D. A. Review and re-analysis of domain-specific 16S primers. J. Microbiol. Methods. 55 (3), 541-555 (2003).
- Hajishengallis, G., et al. A Low-abundance biofilm species orchestrates inflammatory periodontal disease through the commensal microbiota and the complement. Cell Host Microbe. 10 (5), 497-506 (2011).
- Axon, A. Gastric cancer and Helicobacter pylori. Aliment. Pharmacol. Ther. 16 (suppl. 4), 83-88 (2002).
- Fenhalls, G., et al. In situ. detection of Mycobacterium tuberculosis transcripts in human lung granulomas reveals differential gene expression in necrotic lesions. Infect. Immun. 70 (11), 6330-6338 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados